Method Article
IR-TEx: Un outil d'intégration de données Open Source pour la transcriptionomique Big Data conçu pour le vecteur antipaludique Anopheles gambiae
Dans cet article
Résumé
IR-TEx explore les profils transcriptionnels liés à la résistance aux insecticides chez l'espèce Anopheles gambiae. Fourni ici sont des instructions complètes pour l'utilisation de l'application, des modifications pour explorer plusieurs ensembles de données transcriptomiques, et en utilisant le cadre pour construire une base de données interactive pour les collections de données transcriptomiques de n'importe quel organisme, généré dans n'importe quelle plate-forme.
Résumé
IR-TEx est une application écrite dans Shiny (un paquet R) qui permet d'explorer l'expression des transcriptions (ainsi que d'attribuer des fonctions) dont l'expression est associée à des phénotypes de résistance aux insecticides chez les moustiques Anopheles gambiae. L'application peut être utilisée en ligne ou téléchargée et utilisée localement par n'importe qui. L'application locale peut être modifiée pour ajouter de nouveaux ensembles de données de résistance aux insecticides générés à partir de plusieurs plates-formes-omics. Ce guide montre comment ajouter de nouveaux jeux de données et gérer les données manquantes. En outre, IR-TEx peut être complètement et facilement recodé pour utiliser des ensembles de données d'omics à partir de toutes les données expérimentales, ce qui en fait une ressource précieuse pour de nombreux chercheurs. Le protocole illustre l'utilité de l'IR-TEx dans l'identification de nouveaux candidats à la résistance aux insecticides en utilisant le transfert microsomique du glutathion, GSTMS1, à titre d'exemple. Cette transcription est régulée dans de multiples populations résistantes aux pyréthrinoïdes de Côte d'Ivoire et du Burkina Faso. L'identification des transcriptions cocorridées fournit un aperçu plus approfondi des rôles putatifs de ce gène.
Introduction
La capacité de mesurer l'expression d'un grand nombre de transcriptions simultanément à l'aide de plates-formes de microréseaux et de la technologie RNAseq a permis de générer de vastes ensembles de données associant l'expression de la transcription à un phénotype particulier dans les organismes modèles et non modèles. Ces jeux de données sont une ressource extrêmement riche pour les chercheurs, dont la puissance peut être augmentée en combinant les ensembles pertinents dans une approche d'intégration de Big Data. Cependant, cette méthodologie est limitée à ceux qui ont des compétences particulières en bioinformatique. Décrit ici est un programme, IR-TEx (précédemment publié par Ingham et al.1) qui est écrit dans un paquet R appelé Shiny2 et permet aux utilisateurs avec peu de formation en bioinformatique d'intégrer et d'interroger ces ensembles de données avec une relative facilité.
IR-TEx, trouvé à http://www.lstmed.ac.uk/projects/IR-TEx, a été écrit pour explorer les transcriptions associées à la résistance aux insecticides dans Anopheles gambiae, le principal vecteur africain du paludisme1. Le paludisme est une maladie parasitaire causée par les espèces de Plasmodium, transmise entre les humains par les piqûres de moustiques anophèles femelles. Cibler le moustique vecteur avec des insecticides s'est avéré être le moyen le plus efficace de prévenir la morbidité et la mortalité liées au paludisme en Afrique. La mise à l'échelle des outils (c'est-à-d. les moustiquaires insecticides de longue durée) a également joué un rôle crucial dans la réduction spectaculaire des cas de paludisme depuis 20003. Avec un nombre très limité d'insecticides disponibles, il y a une forte pression évolutive sur les moustiques, et la résistance est maintenant répandue dans les vecteurs de paludisme africains4.
En outre, les mutations du site cible5 et le dégagement métabolique des insecticides6,7 restent les principaux mécanismes étudiés de résistance, mais d'autres mécanismes résistants puissants sont en train d'émerger1. Bon nombre de ces nouveaux mécanismes n'ont pas été associés auparavant à la résistance aux insecticides, mais ont été détectés en recherchant des modèles communs d'expression génique dans de multiples populations résistantes à l'aide de l'application IR-TEx et par la suite validées fonctionnellement par des approches génomiques1.
Décrit ici est une approche étape par étape à l'aide ir-TEx, à la fois sur le web et lorsqu'il est installé localement. Le protocole décrit comment de nouveaux ensembles de données sur la résistance aux insecticides peuvent être intégrés dans le paquet existant et explique comment fonctionner avec les données manquantes. Enfin, il décrit comment utiliser ce logiciel avec d'autres jeux de données -omics qui ne sont pas liés à la résistance aux insecticides, combinant ainsi des données provenant d'approches différentes -omics tout en fonctionnant avec des valeurs manquantes et la normalisation de sorte que les données sont comparables.
Protocole
1. Utilisation de l'application web IR-TEx
- Exécution de l'application dans un navigateur Web
- Ouvrez l'application web IR-TEx en suivant le lien au bas de la page trouvée à http://www.lstmed.ac.uk/projects/IR-TEx.
- Une fois la page Web paraphée, cliquez sur le bouton Application en haut de la page, qui affichera l'application et les sorties associées.
- Lisez chaque sortie liée à l'entrée par défaut de AGAP008212-RA (CYP6M2) dans la boîte d'iD de transcription avec les conditions suivantes : Ensembles de données An. coluzzii qui sont (i) exposés aux insecticides pyréthrinoïdes ou (ii) non exposés à une classe d'insecticide, et transcriptions associées avec une corrélation de 'r' 0,98 euros.
- Explorer l'expression d'une transcription d'intérêt
- Pour sélectionner une transcription d'intérêt, saisiz l'ID de transcription dans la boîte d'iD de transcription, se souvenant que les transcriptions se terminent dans -RX dépendant de l'isoforme d'intérêt.
- Sélectionnez les jeux de données à interroger en cochant les cases pertinentes pour (i) pays; (ii) Statut d'exposition, (iii) Espèces d'intérêt; et (iv) catégorie d'insecticides d'intérêt, tout en veillant à ce que ces critères donnent lieu à un ensemble de données inclus dans le cadre de l'ensemble de données (voir le tableau supplémentaire 1 dans Ingham et al.,1).
REMARQUE : (iii) fait référence au membre du complexe d'espèces An. gambiae qui intéresse l'utilisateur. Actuellement, les données sont disponibles pour An. coluzzii et An. arabiensis. - Cliquez sur Update View en bas du menu de sélection ou appuyez sur Retour, en ignorant la valeur de corrélation absolue (pour l'instant).
- Donnez à l'application le temps de la mise à jour.
- Lisez le premier graphique comme suit : journal2 changement de pli entre une population résistante et une population de moustiques sensibles au laboratoire de la transcription d'intérêt dans chaque ensemble de données qui répond aux critères choisis à l'étape 1.2 (Figure 1). Les détails de tous les jeux de données peuvent être trouvés dans Ingham et al.1.
- Lisez les informations ci-dessous le graphique comme suit : le pli change entre les moustiques résistants et sensibles pour chaque jeu de données pertinent, en plus des valeurs p corrigées (Q). Chaque ligne représente des sondes individuelles sur le microréseau. La méthodologie pour l'affichage graphique a été rapportée précédemment1.
- Lisez le tableau supplémentaire ci-dessous comme le nombre d'expériences dans lesquelles la transcription d'intérêt est importante ainsi que le nombre total d'expériences correspondant aux critères sélectionnés à l'étape 1.2.
- Pour télécharger les données en format séparé tab, cliquez sur le bouton Télécharger sous les deux tables. Cela permet à l'utilisateur d'explorer les données d'une manière plus facile à l'aide d'un programme tel qu'Excel.
- Interpréter la carte comme suit : chaque point représente les sites de collecte approximatifs de moustiques résistants dans chaque jeu de données dans lequel la transcription de l'intérêt est exprimée différemment. Les couleurs suivent un système de feux de circulation qui est expliqué dans l'application (Figure 2).
- Pour les étapes 1.2.5 et 1.2.8, enregistrez les sorties graphiques en cliquant à droite, en cliquant sur Enregistrer l'image comme..., et en choisissant un dossier approprié.
REMARQUE : Dans le cas d'une erreur de sortie par l'application, il est probable qu'aucun ensemble de données ne corresponde aux critères saisis. Vérifiez le tableau supplémentaire 1 à Ingham et al.1 si cela se produit.
- Identifier les fonctions putatives/voies de transcription d'intérêt
- Les corrélations (valeur minimale r2 saisies) des modèles d'expression des transcriptions à travers plusieurs ensembles de données peuvent être utilisées pour prédire la fonction de transcription et potentiellement élucider les transcriptions coréglementées de la même voie. En utilisant l'exemple d'Ingham et coll.1 (AGAP001076-RA; CYP4G16), suivez les étapes 1.2.1-1.2.2 dans la section ci-dessus, en sélectionnant tous les jeux de données pour la puissance maximale.
- Avant de cliquer sur Update View, déplacez le curseur de valeur de corrélation absolue à 0,85, et cliquez sur Update View ou appuyez sur Return.
- Examinez le tableau de corrélation (tableau le plus bas) pour trouver les transcriptions multiples qui sont maintenant affichées et qui sont corrélées (0,85 euros) avec la transcription saisie.
- Manipulez le curseur de valeur de corrélation absolue et observez tout changement dans le graphique et le tableau les plus bas ; les sorties de l'étape 1.3.2 resteront inchangées. Comme le montre la figure 3 (à l'adresse suivante : 0,9 , 0,8 ), l'abaissement de la rigueur de la valeur de corrélation affichera plus de transcriptions, mais introduira plus de bruit.
- Lisez le tableau ci-dessous de la sortie graphique, qui (en plus des paramètres décrits à l'étape 1.2.6) contient la valeur de corrélation pour chaque transcription.
- Pour télécharger les données dans un format séparé par onglet, cliquez sur le bouton Télécharger.
- L'analyse d'enrichissement fonctionnel peut être effectuée sur la liste d'iD de transcription téléchargée à l'aide de l'analyse DAVID8. Une fois sur le site web de DAVID (trouvé à https://david.ncifcrf.gov/),sélectionnez Analyse Fonctionnelle. Collez la liste complète des gènes, en utilisant des ID génétiques [identifiant sans le -RX, qui peut être fait en excellant en insérant une colonne à droite de l'ID systématique et en tapant 'LEFT(X1,10), où X1 est la cellule d'identification systématique]. Sélectionnez l'identifiant comme VectorBase_ID et liste de gènes et cliquez sur Soumettre la liste.
- Cliquez sur le bouton De clustering d'annotation fonctionnelle pour donner un aperçu des enrichissements trouvés dans ce réseau de corrélation, ce qui permet d'attribuer une fonction potentielle à la transcription. Explorez les enrichissements en profondeur en examinant les différentes catégories et en cliquant sur les boutons de l'apo pour chacun d'eux et en cliquant ensuite sur graphique.
2. Téléchargement et mise en œuvre d'IR-TEx localement
- Téléchargement et exécution d'IR-TEx
- Aller au lien trouvé à http://github.com/LSTMScientificComputing/IR-TEx; et cliquez sur Clone ou téléchargez Télécharger Zip. Dirigez-vous vers un dossier de choix et décompresser le fichier dans ce dossier.
- Téléchargez la dernière version du logiciel R pour le système d'exploitation approprié à partir du lien trouvé à http://cran.r-project.org/mirrors.html. Installez le programme.
- Télécharger et installer le dernier logiciel R Studio, encore une fois pour le système d'exploitation approprié à partir du lien trouvé à http://www.rstudio.com/products/rstudio/download/.
- Une fois installé, ouvrez R Studio Code supplémentaire Fichier 1 et exécutez chaque ligne pour configurer le système pour IR-TEx.
- Une fois que tous les paquets sont installés et mis à jour avec succès au besoin, rendez-vous sur File Ouvrez,localisez IR-TEx.R, mettez en évidence, et ouvrez. Cela devrait maintenant être visible dans la fenêtre supérieure de R Studio.
- Pour exécuter l'application, appuyez sur le bouton Run App en haut à droite de la fenêtre, et une deuxième fenêtre apparaîtra dans laquelle l'application se chargera. Une fois le chargement terminé, pour une fonctionnalité complète, cliquez sur Ouvrez dans le navigateur situé en haut à droite de la fenêtre chargée.
- Ajout de jeux de données de résistance à IR-TEx (généré à l'aide du tableau Anopheles gambiae 15k Agilent)
- Pour ajouter un nouveau jeu de données analysé généré sur la même plate-forme de microarray (A-MEXP-2196) au jeu de données disponible, téléchargez l'application et localisez le dossier décompressé téléchargé dans la section 2.1.
- Ouvrez le fichier supplémentaire 1, qui représente une sortie d'une analyse de limma sur A-MEXP-2196 1. En utilisant Excel, dans la colonne H1, écrivez Fold_Change, et en H2, écrivez 2 'B2, dans lequel B2 est le changement de pli de journal. Appliquez ceci tout au long de la colonne H pour produire des changements de pli cru.
- Disposez le fichier supplémentaire 1 de telle sorte que la colonne A est l'ID, la colonne B est la modification de pli de la colonne H (copier la colonne H, mettre en évidence la colonne B, puis clic droit et coller les valeurs) et la colonne C est la valeur p ajustée. Supprimez toutes les autres colonnes et enregistrez-la sous la forme d'un fichier délimité par onglet.
- Ouvrez le fichier de codage supplémentaire 2 et exécutez à l'aide de la feuille délimitée par onglet produite à l'étape 2.2.3.
NEWFILE_FC 'COUNTRY', 'EXPOSURE STATUS','SPECIES','INSECTICIDE')
NEWFILE_Q 'COUNTRY','EXPOSURE STATUS','SPECIES','INSECTICIDE')
REMARQUE : Les champs à l'intérieur de guillemets uniques doivent être modifiés pour refléter les informations du nouvel ensemble de données. L'état d'exposition se réfère à la question de savoir si des échantillons ont été prélevés à la suite d'une exposition aux insecticides (exposés/non exposés). Insecticide : si « non exposé », n'en utilisez pas. Voir Fold_Changes.txt. pour les métadonnées d'autres échantillons. Assurez-vous que l'orthographe est cohérente. - Ouvrez geography.txt, faites défiler vers la ligne finale occupée, et sélectionnez ci-dessous. Tapez le nom de l'ensemble de données, suivi de Q et NEWFILE_Q dans la colonne 1, de la latitude du site de collecte d'échantillons dans la colonne 2 et de la longitude dans la colonne 3. Enregistrer les modifications.
- Si des entrées nouvelles sont utilisées (c.-à-d. la Gambie), qui ne sont pas disponibles pour la sélection dans l'ensemble de données (voir Ingham et al. Tableau supplémentaire 11), celles-ci devront être ajoutées au code. Pour ce faire, ouvrez IR-TEx.R dans RStudio et localisez la ligne 26 comme indiqué par RStudio, à quel point ce qui suit devrait commencer :
'sidebarPanel(....'.
REMARQUE : Chacune des lignes d'instance se rapporte à un élément de métadonnées entrées dans les lignes sous le nom du jeu de données à Fold_Changes.txt à l'étape 2.2.5. - Pour ajouter les nouvelles métadonnées, faites défiler jusqu'à la fin de la ligne des métadonnées de choix et localisez le terme « sélectionné ». Immédiatement après cela devrait être une virgule et un support fermé; à ce stade, cliquez sur le curseur dans le support fermé. Après l'apostrophe finale, tapez une virgule, suivie d'une apostrophe, suivie des nouvelles métadonnées (p. ex. « Gambie »), et enregistrez les modifications. Voir ci-dessous pour un exemple.
checkboxGroupInput('CountryInput','Select Relevant Countries',c('Burkina Faso','Cote d'Ivoire','Cameroun','Equatorial Guinea','Zambia','Tanzania','Sudan','Uganda','Togo', 'Gambie',sélection'c('Burkina Faso','Côte d'Ivoire','Cameroun','Guinée équatoriale', 'Zambie', 'Tanzanie','Soudan','Ouganda','Togo')) - Exécutez l'application. La nouvelle entrée de métadonnées doit apparaître sous la rubrique pertinente sous la rubrique pertinente. Si l'utilisateur souhaite qu'il soit sélectionné, il doit être ajouté après la sélection (..., comme indiqué ci-dessous:
checkboxGroupInput('CountryInput','Select Relevant Countries',c('Burkina Faso','Cote d'Ivoire','Cameroun','Equatorial Guinea','Zambia','Tanzania','Sudan','Uganda','Togo', 'Gambie',sélectionc('Burkina Faso','Côte d'Ivoire','Cameroun', 'Guinée équatoriale', 'Zambie', 'Tanzanie','Soudan','Ouganda','Togo', 'Gambie')) - Pour ajouter des ensembles de données de résistance non exécutés sur A-MEXP-2196, voir la section 3.
3. Modification de l'IR-TEx pour une utilisation avec différents jeux de données
- Utilisation sur plusieurs plates-formes-omics et procédant avec les données manquantes
- Pour procéder à "0" dans les jeux de données: consulter la source de l'ensemble de données pour le sens spécifique de "0". Il est recommandé que "0" soit (conservateur) remplacé par "NA". Comme pour les changements de plis bruts (B/A), "0" indique un signal non détecté dans l'état expérimental B. Dans le cas où l'état expérimental A présente une expression substantielle, l'utilisateur peut appliquer une petite valeur de changement de pli.
- Ouvrez le fichier supplémentaire 2.txt, un fichier RNAseq adapté de Uyhelji et al.9. Ce fichier représente le modèle dans lequel les nouvelles données doivent être basées : colonne A ' identificateur, colonne B 'changement de pli brut, et colonne C 'valeur p ajustée. Utilisez ce fichier pour parcourir les étapes ci-dessous.
- Exécutez le code R pour faire correspondre les identificateurs dans un seul fichier délimité par onglet sur les plates-formes, puis organisez et normalisez les données (Fichier de codage supplémentaire 2). Les instructions sont contenues dans le fichier. Tout FILEPATH sera séparé par "/" pour MacOS ou "//" pour Windows (changez-les de "" ", comme ils apparaîtront).
- Sortie du fichier produit à la fin du fichier de codage supplémentaire 2 à un endroit de choix pour une utilisation à l'étape 3.1.5. Le fichier de codage supplémentaire 2 produira un nouveau fichier Fold_Changes.txt. Sauvegarder le fichier d'origine.
- Exécutez le code contenu dans le fichier de codage supplémentaire 3. Trouvez le fichier de sortie nommé FC_distribPlot.png dans le dossier spécifié comme FILEPATH. Vérifiez les distributions de la modification du journal2 pli pour vérifier que les distributions de changement de journal2 sont presque identiques entre les jeux de données.
- Suivez les instructions de l'étape 2.2.6 pour modifier des fichiers supplémentaires et assurer la compatibilité de la nouvelle Fold_Changes.txt.
- Modification de l'IR-TEx pour une utilisation avec des jeux de données entièrement nouveaux
- Ouvrez IR-TEx.R dans RStudio et localisez les lignes (23-34) en commençant par :
'tabPanel('
et se terminant par:
soumettreButton ("Update View", icône ("refresh"))
), - Modifier l'AGAP008212-RA trouvé dans les lignes ci-dessous à une transcription de l'intérêt pour les nouvelles données.
textInput('textInput','Transcript ID',value'AGAP008212-RA'), - Localisez les quatre options en commençant par :
case à cocherGroupInput(
Ces options peuvent être modifiées pour représenter des métadonnées importantes que l'utilisateur souhaite filtrer les nouvelles données par. Dans chaque cas, l'utilisateur doit modifier les pays pertinents sélectionnés; Sélectionnez statut d'exposition; Sélectionnez les espèces pertinentes; et sélectionnez la classe d'insecticide pour être représentative des données (c.-à-d. Sélectionnezle type de tissu ; Sélectionnez Sexe; Sélectionnez Age Bracket; Sélectionnez l'état de la maladie). - Identifier les métadonnées associées au jeu de données et l'entrée pour remplacer les options existantes immédiatement après le premier c('. Dans chaque cas, les options seront contenues dans les marques de la parole et séparées de la prochaine sélection par une virgule. Après la sélection finale, le support doit être fermé. Voici un exemple pour Sélectionner l'état de la maladie :
c('Infecté', 'Uninfected', 'Unknown') - Choisissez laquelle de ces métadonnées sera sélectionnée lors de l'ouverture de l'application. Celles-ci peuvent être modifiées en modifiant les options après avoir été sélectionnées.'. Voici un exemple pour Sélectionner l'état de la maladie :
sélectionnés ('Infecté', 'Non infecté')
Cela demandera à l'application de sélectionner uniquement les jeux de données correspondant à ces critères lors du chargement initial. - Pour créer une nouvelle table de données, suivez la mise en page trouvée dans Fold_Changes.txt et les instructions de la section 2. Modifier les métadonnées à chaque modification respective décrite à l'étape 3.2.4, exactement comme indiqué dans le code (R est sensible aux cas). Dans la colonne de désintoxication, les noms des gènes d'entrée, et dans la colonne de type de transcription, les descriptions de gènes d'entrée pour chaque transcription. Suivez la section 3.2 lors de l'ajout de nouveaux jeux de données.
- Si la cartographie n'est pas pertinente aux exigences expérimentales, localisez les lignes de code suivantes et placez le mot « ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' ' '
Lignes 49 à 51:
br(),br(),
avecSpinner (plotOutput("Géographie")),
textOutput ('Geography_legend'),
Lignes 493 à partir de:
sortie$Géographie 'lt;- renderPlot(
Pour la ligne 602 se terminant:
sortie$Geography_legend 'lt;- renderText(
pâte ("Transcriptions significatives seulement (p", as.expression ("lt; "), "0,05): FC 'gt; 5 ' Rouge, FC 'gt; 1 ' Amber, FC 'lt; 1 ' Vert,sep '")
})
- Ouvrez IR-TEx.R dans RStudio et localisez les lignes (23-34) en commençant par :
Résultats
En utilisant le fichier Fold_Changes.txt inclus avec IR-TEx, nous avons comparé les transcriptions qui ont été exprimées de manière significative de manière significative dans les ensembles de données résistants Anopheles coluzzii et Anopheles gambiae aux contrôles sensibles de la Côte d'Ivoire et du Burkina Faso. Cela a donné 18 transcriptions d'intérêt(tableau 1; cette recherche peut être effectuée à l'aide d'Excel, R, ou d'autres programmes). Deux d'entre eux, un ATPase (AGAP006879) et un '-crystallin (AGAP007160), ont été précédemment rapportés, le premier ayant un effet significatif sur la résistance des pyrèthrines1. En plus de ces deux transcriptions, deux transcriptions de désintoxication, GSTMS1 (FC- 1,95 et 1,85) et UGT306A2 (FC- 2,29 et 2,28) étaient présents.
qPCR validation de deux de ces transcriptions(GSTMS1, une transcription de désintoxication; et AGAP009110-RA, une transcription inconnue, spécifique aux moustiques contenant un domaine de liaison de 1,3-glucan) ont été effectuées comme précédemment décrit1. L'analyse a été effectuée à l'aide d'ensembles d'amorces décrits dans le fichier 3 additionnel et a montré que ces transcriptions étaient significativement régulées dans une population multirésistante de Côte d'Ivoire (Tiassalé) et d'une autre du Burkina Faso (Banfora), par rapport au N'Gousso(figure 4A).
Comme les deux transcriptions ont montré une augmentation significative de la réglementation dans chacune des populations résistantes, le knockdown induit par l'ARNi a été effectué sur les moustiques de la colonie de Tiassalé, laboratoire du LSTM. Cette colonie est originaire de Côte d'Ivoire et est résistante à toutes les grandes classes d'insecticides utilisés en santé publique, comme nous l'avons déjà décrit1,10. L'atténuation de l'expression de gstMS1 a entraîné une augmentation significative (p - 0,021) de la mortalité après l'exposition à la deltaméthrine par rapport aux témoins injectés par le GFP, ce qui démontre l'importance de cette transcription dans la résistance aux pyréthrinoïdes (figure 4B). Inversement, le renversement de l'AGAP009110-RA n'a entraîné aucun changement significatif (p - 0,082) de la mortalité après l'exposition (figure 4B).
GSTMS1 est une TPS microsomique et est l'un des trois trouvés dans les moustiques A. gambiae 11. Bien que les membres des classes d'epsilon et de delta des GST aient été précédemment impliqués dans la désintoxication d'insecticide12,13,14,c'est la première évidence à notre connaissance pour un rôle des GST microsomiques dans la résistance des pyréthrinoïdes15. Pour explorer la fonction putative de cette transcription dans les moustiques sl de Anopheles gambiae, l'expression et la corrélation dans IR-TEx ont été identifiées. GSTMS1 a été largement surexprimée dans 20 des 21 ensembles de données disponibles pour ces espèces, à l'exception de l'île Bioko. Dans chaque endroit, la surexpression était inférieure de cinq fois supérieure à celle des populations sensibles (figure 5).
Comme les TSG microsomiques ont été largement ignorés en tant que désintoxiqueurs potentiels d'insecticide, on sait peu de choses sur leur rôle dans la résistance aux insecticides15. En explorant la co-corrélation d'autres transcriptions, les fonctions putatives peuvent être élucidées par l'hypothèse de la corégulation ou de la participation aux mêmes voies. Afin de maximiser la puissance du réseau de corrélation, tous les ensembles de données sur microréseaux présents dans IR-TEx ont été sélectionnés, ainsi qu'un de '0.75 a été sélectionnée. Le tableau 2 montre la sortie d'IR-TEx.
Ces transcriptions sont enrichies dans l'activité d'oxioreductase et le métabolisme de glucose/hydrate de carbone dans l'outil fonctionnel d'annotation de DAVID8. Le glucose-6-phosphate dehydrogénase et le cytathione gamma-lyase maintiennent le niveau de glutathion dans les cellules mammifères16,17 et donc directement liés à GSTMS1, un glutathion-S-transferase. Catalase est un intervenant de stress oxydatif à action rapide qui protège les cellules contre les dommages réactifs des espèces d'oxygène, un sous-produit de l'exposition aux pyréthrinoïdes. Valacyclovir hydrolase est un hydrolase qui peut jouer un rôle dans la désintoxication dans les cellules de mammifères18. CYP4H17 est également présent dans le réseau de corrélation. Les p450 cytochromes sont des métabolisateurs directs d'insecticides pyréthrinoïdes, et ces produits de dégradation peuvent être métabolisés par les TSG. Enfin, CYP4H17 a été impliqué dans la résistance des pyréthrinoïdes dans A. funestus19. Prises ensemble, ces données appuient fortement le rôle de GSTMS1 dans la désintoxication xénobiotique.
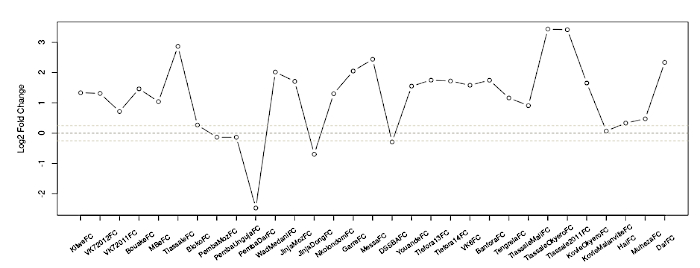
Figure 1 : Changement de pli de journal2 d'AGAP002865-RA dans tous les jeux de données. L'axe x détaille les différents ensembles de données, dont les informations peuvent être trouvées dans le tableau 1 supplémentaire dans une publication précédente1, et l'axe y montre le changement de journal2 fois dans la transcription d'intérêt. Les lignes pointillées gris clair indiquent des seuils approximatifs pour l'importance, prises ici pour être un changement de pli de 'lt;0.8 ou le changement de pli de 'gt;1.2. La ligne noire pointillée indique un changement de pli de 1 (c.-à-d., aucune différence d'expression entre les populations résistantes et sensibles). Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
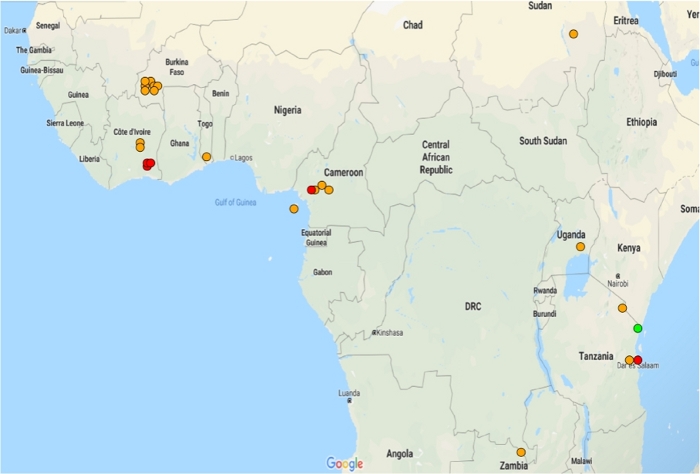
Figure 2 : Répartition des microréseaux montrant une expression différentielle significative de l'AGAP002865-RA dans les populations résistantes. Les changements de pliage sont représentés dans un système de feux de circulation : changement de pli vert de l'il1, changement de pli orange de 1, et changement de pli rouge de 'gt;5. Seuls les jeux de données avec une expression différentielle significative (p 0,05) sont affichés. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
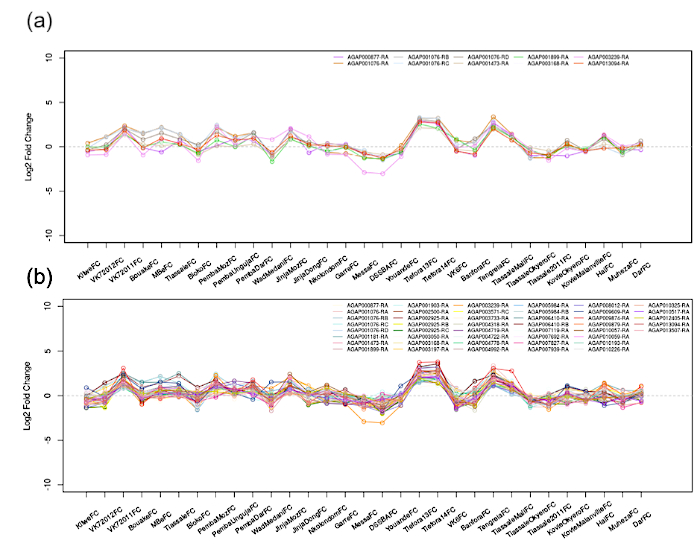
Figure 3 : Réseaux de corrélation de AGAP001076-RA (CYP4G16). Les corrélations paires sont calculées sur toutes les transcriptions des 31 ensembles de données microarray, avec une coupure définie par l'utilisateur appliquée. Montré ici est (A) 0,9 et (B) 0,8. Toutes les transcriptions affichées sur le graphique répondent à ce critère et suivent les changements d'expression de AGAP001076-RA. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
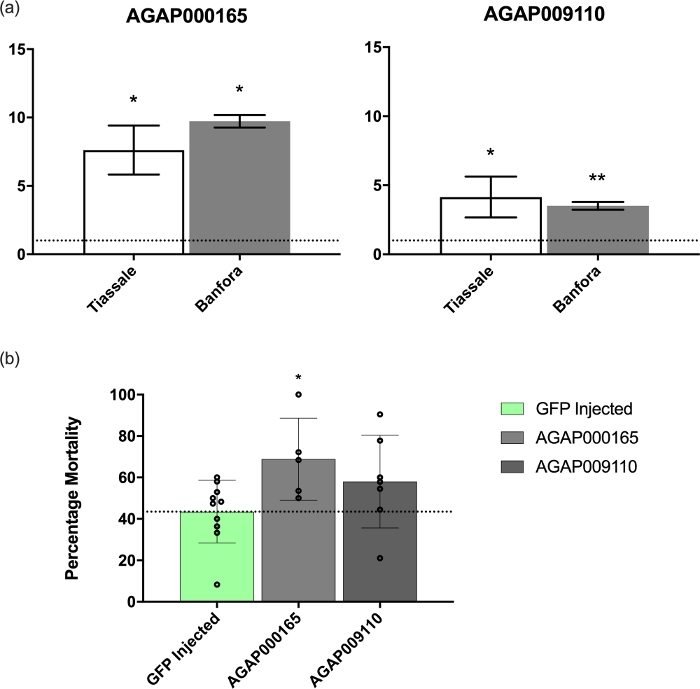
Figure 4 : expression de l'ARNm et phénotype sur atténuation de GSTMS1 et AGAP009110-RA. (A) expression de l'ARNm de GSTMS1 et AGAP009110-RA dans deux populations multirésistantes d'An. coluzzii de Côte d'Ivoire et du Burkina Faso, respectivement. Les niveaux ont été comparés à l'An. coluzzii N'Gousso, qui est sensible au laboratoire. Niveaux d'importance calculés par ANOVA avec un test post-hoc de Dunnett. (B) L'atténuation induite par l'ARNi des deux transcriptions par rapport aux contrôles injectés par GFP. L'atténuation de la TPSM1 montre une augmentation significative de la mortalité après l'exposition à la deltaméthrine (calculée par ANOVA avec un test Tukey post-hoc; 'p '0,05, 'p '0,01). Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
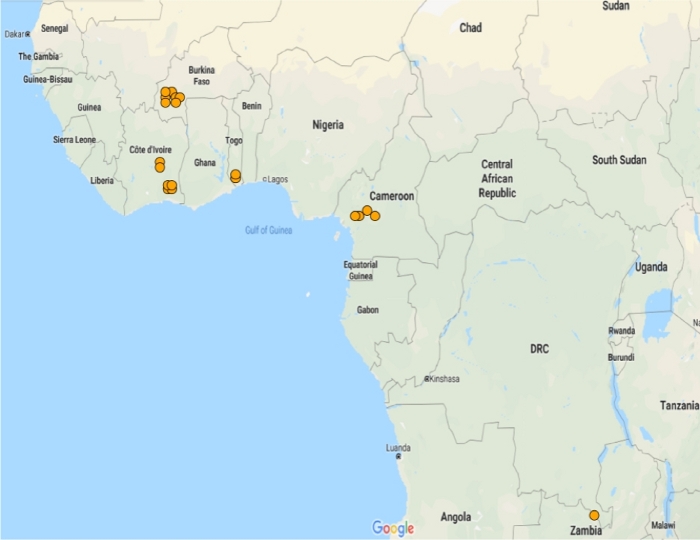
Figure 5 : Expression de la TPSMS1 dans les populations d'Anopheles gambiae et d'Anopheles coluzzii. Carte montrant l'expression significativement différente de GSTMS1 dans les ensembles de données disponibles sur les microréseaux. Il a été constaté que la TPSMS1 était significativement différentiele dans 20 des 21 ensembles de données sur microréseaux. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
| ID de transcription | Description | Burkina Faso | Côte D'Ivoire |
| AGAP006879-RA | Atpase | 27.94 | 43.05 |
| AGAP007160-RB | a-crystallin | 11.49 | 10.58 |
| AGAP007160-RC | a-crystallin | 11.14 | 10.38 |
| AGAP007160-RA | a-crystallin | 9.78 | 9.84 |
| AGAP009110-RA | Inconnu | 9.26 | 5.96 |
| AGAP007780-RA | Déshydrogénase NADH | 10.49 | 3.77 |
| AGAP006383-RA | oligosaccharyltransferase complexe sous-unité bêta | 3.69 | 5.57 |
| AGAP007249-RB | Flightin (en) | 4.61 | 3.86 |
| AGAP003357-RA | Protéine RAG1-activant 1-like protéine | 4.31 | 4.05 |
| AGAP007249-RA | Flightin (en) | 4.48 | 3.46 |
| AGAP001998-RA | mRpS10 (en) | 3.46 | 2.85 |
| AGAP007589-RA | UGT306A2 UGT306A2 UGT306A2 | 2.29 | 2.28 |
| AGAP000165-RA | GSTMS1 (en) | 1.95 | 1.85 |
| AGAP002101-RA | synthétase isoleucyl-tRNA | 0.57 | 0.59 |
| AGAP002969-RA | synthétase asparaginyl-tRNA | 0.45 | 0.45 |
| AGAP004199-RA | famille de porteurs de solute 5 (transporteur monocarboxylate couplé au sodium), membre 8 | 0.35 | 0.48 |
| AGAP004684-RA | protéine de traitement de rRNA CGR1 | 0.36 | 0.22 |
| AGAP006414-RA | Cht8 (Cht8) | 0.024 | 0.36 |
Tableau 1 : Transcriptions significativement différentieles dans la même direction de changement de pli dans les populations du Burkina Faso et de la Côte d'Ivoire. ID de transcription, description de gène, et changement moyen de pli pour chaque ensemble de données des deux pays représentant des populations d'An. coluzzii et d'An. gambiae.
| Corrélation | Nom systématique | Type de transcription |
| 1 | AGAP000165-RA | GSTMS1 (en) |
| 0.82 | AGAP004904-RA | Catalase |
| 0.76 | AGAP007243-RA | 26S protease sous-unité réglementaire 8 |
| 0.79 | AGAP008358-RA | CYP4H17 |
| 0.76 | AGAP009436-RA | Hydrolase de Valacyclovir |
| 0.75 | AGAP010739-RA | Glucose-6-phosphate 1-déshydrogénase |
| 0.85 | AGAP011172-RA | cystathionine gamma-lyase |
| 0.76 | AGAP012678-RA | Glucose-6-phosphate 1-déshydrogénase |
Tableau 2 : Transcriptions coliées avec GSTMS1. Le tableau montre la sortie du réseau de corrélation pour GSTMS1 sur IR-TEx avec de 0,75 euros. Le tableau montre la corrélation du Spearman, l'ID de transcription, et la description de gène pour chaque transcription co-corliée.
Fichier supplémentaire 1 : Fichier de sortie du tableau A-MEXP-2196 analysé sur limma. Le fichier provient d'un knockdown Met par rapport à un tableau de contrôle GFP, décrit plus en détail dans ArrayExpress (E-MTAB-4043) et une autre publication précédente1. Les colonnes représentent l'identifiant AGAP (SystematicName), le changement de pli de journal (logFC), les valeurs d'expression de journal (AveExpr), t-statistique (t), la valeur p non corrigée (P.Value), la valeur p ajustée (adj. P.Val), et B statistique (B)20. Aux fins de ce fichier, les moustiques sont des anophèles coluzzi de Côte d'Ivoire et ne sont pas exposés aux insecticides, avec une latitude de collecte et une longitude de -5,4 et 6,0, respectivement. S'il vous plaît cliquez ici pour voir ce fichier (Clic droit pour télécharger).
Fichier supplémentaire 2 : Fichier de sortie de l'expérience RNAseq. Analyse de RNAseq tirée d'Uyhelji et coll.9 décrivant les changements dans le transcriptome des moustiques anophèles lorsqu'ils sont exposés à une salinité de 50 %. Ce fichier est adapté du tableau S2 de la publication et comprend l'identifiant AGAP (SystematicID), le changement de pli brut (Fold_Change) et la valeur p ajustée (q_value). S'il vous plaît cliquez ici pour voir ce fichier (Clic droit pour télécharger).
Dossier supplémentaire 3 : Liste d'apprêt pour les résultats représentatifs. Identificateur AGAP, nom de gène, dsRNA en avant, dsRNA inversé, qPCR en avant, et qPCR ensembles d'apprêt inversé pour chaque transcription. S'il vous plaît cliquez ici pour voir ce fichier (Clic droit pour télécharger).
Code supplémentaire Fichier 1. S'il vous plaît cliquez ici pour voir ce fichier (Clic droit pour télécharger).
Code supplémentaire Fichier 2. S'il vous plaît cliquez ici pour voir ce fichier (Clic droit pour télécharger).
Code supplémentaire Fichier 3. S'il vous plaît cliquez ici pour voir ce fichier (Clic droit pour télécharger).
Discussion
La transcriptomique des données volumineuses produit des listes de milliers de transcriptions qui sont exprimées différemment pour chaque condition expérimentale. Bon nombre de ces expériences sont réalisées sur des organismes et des phénotypes apparentés et sont presque exclusivement analysées sous forme d'expériences indépendantes. L'utilisation de ces sources de données riches en examinant les données de manière holistique et sans hypothèses théoriques 1) conduira à l'identification de nouvelles transcriptions des candidats et 2) empêchera l'abandon de données précieuses simplement parce qu'il y a trop d'informations à valider in vivo1.
IR-TEx offre aux utilisateurs un bagage bioinformatique limité avec la possibilité d'examiner facilement plusieurs jeux de données, de visualiser les changements dans les jeux de données et de télécharger les informations associées1. Bien qu'IR-TEx ne prend pas en charge la recherche de plus d'une transcription dans chaque recherche, les utilisateurs peuvent examiner les fichiers associés Fold_Changes.txt simplement en utilisant Excel, R, ou d'autres programmes appropriés. L'utilité de l'IR-TEx découle de l'utilisation de réseaux de corrélation pour prédire la fonction de transcription, l'entrée de protéines hypothétiques ou de transcriptions ayant des fonctions inconnues et l'utilisation de logiciels en aval pour rechercher des enrichissements1.
Dans l'exemple démontré dans ce protocole, IR-TEx est utilisé en fonction de sa fonction d'origine. Ici, il permet d'explorer les transcriptions associées à la résistance aux insecticides et la visualisation de la distribution de la surexpression et de la sous-expression à travers des graphiques cartographiques. Les transcriptions d'intérêt sont validées in vivo pour déterminer si la surexpression ou la sous-expression des transcriptions données contribue à un phénotype1 observé (p. ex., résistance aux insecticides). Il a été démontré ici, comme indiqué précédemment1, qu'un ensemble de données peut être utilisé dans une approche fondée sur des hypothèses pour identifier les transcriptions d'intérêt sur une base spécifique à chaque pays. IR-TEx peut ensuite être utilisé pour 1) explorer l'expression de la transcription et 2) contextualiser la fonction de la transcription en appliquant un réseau de corrélation en deux sens pour toutes les transcriptions contenues dans chaque jeu de données -omics. En l'espèce, il a été démontré que gstMS1 était en corrélation avec un certain nombre d'autres transcriptions impliquées dans la désintoxication. Ces données (ainsi que le renversement de la transcription qui a eu comme conséquence une augmentation significative de la mortalité après exposition d'insecticide) démontrent l'importance de cette transcription dans le dégagement xénobiotique.
IR-TEx représente une ressource précieuse pour explorer les transcriptions liées à la résistance aux insecticides sur le Web ou à l'aide d'applications locales. Ce protocole montre comment modifier IR-TEx pour différentes plates-formes-omics ainsi que des données complètement nouvelles. Le guide illustre comment utiliser IR-TEx pour intégrer les données de plusieurs plates-formes et jeux de données avec des données manquantes ainsi que la façon de recoder IR-TEx simplement afin qu'il soit utile pour toute personne faisant des recherches sur les ensembles de données transcriptomiques.
Déclarations de divulgation
Les auteurs n'ont rien à révéler.
Remerciements
Ce travail a été financé par une bourse de perfectionnement des compétences du CRM à V.I. (MR/R024839/1) et par la Royal Society Challenge Grant (CH160059) à H.R.
matériels
| Name | Company | Catalog Number | Comments |
| Laptop with browser | Any | - | - |
| R Program | The R Project for Statistical Computing | - | https://www.r-project.org/ |
| R Studio | R Studio | - | https://www.rstudio.com/ |
Références
- Ingham, V. A., Wagstaff, S., Ranson, H. Transcriptomic meta-signatures identified in Anopheles gambiae populations reveal previously undetected insecticide resistance mechanisms. Nature Communications. 9 (1), 5282(2018).
- Chang, W., Cheng, J., Allaire, J., Xie, Y., McPherson, J. shiny: Web Application Framework for R. , (2017).
- Bhatt, S., et al. The effect of malaria control on Plasmodium falciparum in Africa between 2000 and 2015. Nature. 526 (7572), 207-211 (2015).
- Ranson, H., Lissenden, N. Insecticide Resistance in African Anopheles Mosquitoes: A Worsening Situation that Needs Urgent Action to Maintain Malaria Control. Trends in Parasitology. 32 (3), 187-196 (2016).
- Donnelly, M. J., et al. Does kdr genotype predict insecticide-resistance phenotype in mosquitoes. Trends in Parasitology. 25 (5), 213-219 (2009).
- Stevenson, B. J., et al. Cytochrome P450 6M2 from the malaria vector Anopheles gambiae metabolizes pyrethroids: Sequential metabolism of deltamethrin revealed. Insect Biochemistry and Molecular Biology. 41 (7), 492-502 (2011).
- Müller, P., et al. Field-Caught Permethrin-Resistant Anopheles gambiae Overexpress CYP6P3, a P450 That Metabolises Pyrethroids. PLoS Genetics. 4 (11), 1000286(2008).
- Huang, D., et al. The DAVID Gene Functional Classification Tool: a novel biological module-centric algorithm to functionally analyze large gene lists. Genome Biology. 8 (9), 183(2007).
- Uyhelji, H. A., Cheng, C., Besansky, N. J. Transcriptomic differences between euryhaline and stenohaline malaria vector sibling species in response to salinity stress. Molecular Ecology. 25 (10), 2210-2225 (2016).
- Edi, C. V., Benjamin, K. G., Jones, C. M., Weetman, D., Ranson, H. Multiple-Insecticide Resistance in Anopheles gambiae Mosquitoes, Southern Côte d’Ivoire. Emerging Infectious Diseases. 18 (9), 1508-1511 (2012).
- Ding, Y., Ortelli, F., Rossiter, L., Hemingway, J., Ranson, H. The Anopheles gambiae glutathione transferase supergene family: annotation, phylogeny and expression profiles. BMC Genomics. 4 (1), 1-16 (2003).
- Enayati, A. A., Ranson, H., Hemingway, J. Insect glutathione transferases and insecticide resistance. Insect Molecular Biology. 14 (1), 3-8 (2005).
- Ranson, H., et al. Identification of a novel class of insect glutathione S-transferases involved in resistance to DDT in the malaria vector Anopheles gambiae. The Biochemical Journal. 359, 295-304 (2001).
- Riveron, J. M., et al. A single mutation in the GSTe2 gene allows tracking of metabolically based insecticide resistance in a major malaria vector. Genome Biology. 15 (2), 27(2014).
- Pavlidi, N., Vontas, J., Van Leeuwen, T. The role of glutathione S-transferases (GSTs) in insecticide resistance in crop pests and disease vectors. Current Opinion in Insect Science. 27, 97-102 (2018).
- Salvemini, F., et al. Enhanced glutathione levels and oxidoresistance mediated by increased glucose-6-phosphate dehydrogenase expression. Journal of Biological Chemistry. 274 (5), 2750-2757 (1999).
- Deplancke, B., Gaskins, H. R. Redox control of the transsulfuration and glutathione biosynthesis pathways. Current Opinion in Clinical Nutrition & Metabolic Care. 5 (1), (2002).
- Puente, X. S., López-Otn, C. Cloning and expression analysis of a novel human serine hydrolase with sequence similarity to prokaryotic enzymes involved in the degradation of aromatic compounds. Journal of Biological Chemistry. 270 (21), 12926-12932 (1995).
- Riveron, J. M., et al. Genome-wide transcription and functional analyses reveal heterogeneous molecular mechanisms driving pyrethroids resistance in the major malaria vector Anopheles funestus across Africa. G3: Genes, Genomes, Genetics. 7 (6), 1819-1832 (2017).
- Smyth, G. K. Linear models and empirical bayes methods for assessing differential expression in microarray experiments. Statistical Applications in Genetics and Molecular Biology. 3 (1), 3(2004).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon