Method Article
使用 UMD 软件包分析 Ab Initio 分子动力学模拟中的熔体和流体
摘要
熔体和流体是自然系统中无处不在的质量传递载体。我们开发了一个开源软件包来分析此类系统的分子动力学模拟。我们计算结构(键合,聚类,化学形态),传递(扩散,粘度)和热力学性质(振动光谱)。
摘要
我们开发了一个基于Python的开源包来分析从流体的分子动力学模拟中产生的结果。该封装最适合自然系统上的应用,如硅酸盐和氧化物熔体、水基流体和各种超临界流体。该软件包是Python脚本的集合,其中包括两个处理文件格式和晶体学的主要库。所有脚本都在命令行运行。我们提出了一种简化的格式来存储模拟的原子轨迹和相关热力学信息,这些信息保存在UMD文件中,代表通用分子动力学。UMD封装允许计算一系列结构,传输和热力学特性。从成对分布函数开始,它定义键长,构建原子间连接矩阵,并最终确定化学物种形成。确定化学物质的寿命允许运行完整的统计分析。然后,专用脚本计算原子和化学物质的均方位移。对原子速度实施的自相关分析得出扩散系数和振动谱。对应力进行相同的分析得出粘度。该软件包可通过GitHub网站及其ERC IMPACT项目自己的专用页面作为开放访问软件包获得。
引言
流体和熔体是自然环境中的活性化学和物理传递载体。原子扩散速率的升高有利于化学交换和反应,低粘度加上不同的浮力有利于较大的质量传递,而晶体 - 熔体密度关系有利于行星体内部的分层。由于没有周期性晶格,达到熔融状态所需的典型高温以及淬火的难度使得一系列明显特性(如密度、扩散和粘度)的实验测定极具挑战性。这些困难使得替代计算方法强大而有用的工具可用于研究这类材料。
随着计算能力的出现和超级计算机的可用性,目前采用两种主要的数值原子模拟技术来研究非结晶原子系统的动力学状态,蒙特卡罗1 和分子动力学(MD)1,2。在蒙特卡罗模拟中,配置空间是随机采样的;蒙特卡罗方法显示并行化中的线性缩放,如果所有采样观测值彼此独立。结果的质量取决于随机数生成器的质量和抽样的代表性。蒙特卡罗方法显示并行化中的线性缩放,如果采样彼此独立。在分子动力学(MD)中,构型空间是通过瞬态原子轨迹采样的。从给定的配置开始,原子轨迹是通过积分牛顿运动方程来计算的。原子间力可以使用模型原子间电位(在经典MD中)或使用第一性原理方法(从头开始或第一性原理,MD)来计算。结果的质量取决于轨迹的长度及其不被局部最小值吸引的能力。
分子动力学模拟包含大量信息,所有这些都与系统的动力学行为有关。热力学平均属性,如内能、温度和压力,是相当标准的计算。它们可以从模拟的输出文件中提取并取平均值,而与原子的运动及其相互关系直接相关的量需要在提取原子位置和速度后计算。
因此,人们已经付出了很多努力来可视化结果,并且今天可以在不同的平台上使用各种软件包,无论是否开源[Ovito3,VMD4,Vesta5,Travis6等]。所有这些可视化工具都可以有效地处理原子间距离,因此,它们允许有效地计算成对分布函数和扩散系数。执行大规模分子动力学模拟的各种小组拥有专有软件来分析模拟产生的各种其他属性,有时以共享软件或其他形式的有限访问社区,有时仅限于范围和使用某些特定软件包。在其中一些包中开发和实现用于提取有关原子间键合,几何图案和热力学信息的复杂算法3,4,5,6,7等。
在这里,我们提出了 UMD包 - 一个用Python编写的开源包,用于分析分子动力学模拟的输出。UMD 封装允许计算各种结构、动力学和热力学特性(图 1)。该软件包可通过GitHub网站(https://github.com/rcaracas/UMD_package)和ERC IMPACT项目的专用页面(http://moonimpact.eu/umd-package/)作为开放获取软件包获得。
为了使其具有通用性并且更易于处理,我们的方法是首先从实际分子动力学运行的输出文件中提取与热力学状态和原子轨迹相关的所有信息。此信息存储在专用文件中,其格式独立于运行仿真的原始 MD 包。我们将这些文件命名为"umd"文件,代表通用分子动力学。通过这种方式,我们的UMD软件包可以被任何ab initio组使用任何软件,所有这些都可以以最小的适应工作进行。使用当前软件包的唯一要求是将特定MD软件的输出中的相应解析器写入umd文件格式(如果尚不存在)。目前,我们为VASP8 和QBox9 软件包提供这样的解析器。

图 1:UMD 库的流程图。
物理属性为蓝色,主要 Python 脚本及其选项为红色。 请点击此处查看此图的放大版本。
umd文件是ASCII文件;典型的扩展是"umd.dat",但不是强制性的。所有分析组件都可以读取 umd 格式的 ASCII 文件,而不管实际的扩展名如何。但是,一些旨在对多个模拟执行快速大规模统计信息的自动脚本专门查找扩展名为 umd.dat 的文件。每个物理属性都用一行表示。每行都以关键字开头。通过这种方式,格式具有高度的适应性,并允许将新属性添加到umd文件中,同时在整个版本中始终保持其可读性。下面讨论中使用的热膨胀石在4.6 GPa和3000 K下模拟的umd文件的前30行 如图2所示。

图 2:umd 文件的开头,描述了在 4.6 GPa 和 3000 K 下模拟液态热浮石的过程。
标头后跟每个快照的说明。每个属性都写在一行上,包含物理属性的名称、值和单位,全部用空格分隔。 请点击此处查看此图的放大版本。
所有 umd 文件都包含描述模拟单元内容的标题:原子数、电子数和原子类型,以及每个原子的详细信息,例如其类型、化学符号、价电子数及其质量。空行标记标头的末尾,并将其与 umd 文件的主要部分分开。
然后详细介绍模拟的每个步骤。首先,给出瞬时热力学参数,每个参数在不同的线上,指定(i)参数的名称,如能量,应力,等效静水压力,密度,体积,晶格参数等,(ii)其值和(iii)其单位。接下来是描述原子的表格。标题行给出了不同的度量,如笛卡尔位置,速度,电荷等,以及它们的单位。然后每个原子在一行上详细介绍。通过对应于三个 x,y,z 轴的三组,条目是:减少的位置,折叠到模拟单元中的笛卡尔位置,笛卡尔位置(正确考虑了原子在模拟过程中可以穿越多个单元单元的事实),原子速度和原子力。最后两个条目是标量:电荷和磁矩。
两个主要的库确保了整个软件包的正常运行。 umd_process.py 库处理 umd 文件,如读取和打印。 crystallography.py 库处理与实际原子结构相关的所有信息。 crystallography.py 库的基本理念是将格视为向量空间。单元单元参数及其方向表示基向量。"空间"具有一系列标量属性(特定体积,密度,温度和特定原子数),热力学属性(内能,压力,热容等)和一系列张量属性(应力和弹性)。原子填充了这个空间。"格"类定义了这个集合,以及各种简短的计算,如比体积,密度,从直接格获得倒数格等。"原子"类定义原子。它们的特点是一系列标量性质(名称,符号,质量,电子数等)和一系列矢量性质(空间中的位置,相对于格类中描述的向量基,或相对于通用笛卡尔坐标,速度,力等)。除了这两个类之外, crystallography.py 库还包含一系列函数,用于执行各种测试和计算,例如原子距离或单元格乘法。元素周期表也作为字典包含在内。
umd 包的各个组件写入多个输出文件。作为一般规则,它们都是ASCII文件,它们的所有条目都由选项卡分隔,并且它们尽可能不言自明。例如,它们总是清楚地指示物理属性及其单位。 umd.dat 文件完全符合此规则。
研究方案
1. 分子动力学运行分析
注意:该软件包可通过 GitHub 网站(https://github.com/rcaracas/UMD_package)和 ERC IMPACT 项目的专用页面(http://moonimpact.eu/umd-package/)作为开放获取软件包获得。
- 使用包中的一个或多个专用 Python 脚本提取每组特定的物理属性。在命令行运行所有脚本;它们都使用一系列标志,这些标志在一个脚本到另一个脚本之间尽可能一致。 表 1 总结了标志、其含义和默认值。
| 旗 | 意义 | 使用它的脚本 | 默认值 |
| -h | 简短帮助 | 都 | |
| -f | UMD 文件名 | 都 | |
| -i | 要丢弃的热化步骤 | 都 | 0 |
| -i | 包含原子间键的输入文件 | 物种形成 | 债券.输入 |
| -秒 | 频率采样 | MSD, 物种形成 | 1(考虑每一步) |
| -一 | 原子或阴离子列表 | 物种形成 | |
| -c | 阳离子列表 | 物种形成 | |
| -l | 键长 | 物种形成 | 2 |
| -t | 温度 | 振动、流变学 | |
| -v | 轨迹采样窗口宽度的离散化,用于均方位移分析 | MSD | 20 |
| -z | 轨迹采样窗口开始的离散化,用于均方位移分析 | MSD | 20 |
表 1:UMD 包中使用的最常见标志及其最常见的意义。
- 首先将在第一原则代码(如 VASP8 或 QBox9)中执行的 MD 仿真输出转换为 UMD 文件。
- 如果 MD 模拟是在 VASP 中完成的,则在命令行中键入:
VaspParser.py -f -i <初始步骤>
其中 –f 标志定义 VASP OUTCAR 文件的名称,–i 定义热化长度。
注意:由 –i 定义的初始步骤允许丢弃表示热化的模拟的第一步。在典型的分子动力学运行中,计算的第一部分表示热化,即系统所有原子描述温度的高斯式分布所需的时间,以及整个系统围绕平衡值表现出温度,压力,能量等的波动。在分析流体的统计特性时,不应考虑模拟的这一热化部分。
- 如果 MD 模拟是在 VASP 中完成的,则在命令行中键入:
- 转换 .将 文件转换为 .xyz 文件,便于在各种其他软件包(如 VMD4 或 Vesta5)上进行可视化。在命令行类型处:
umd2xyz.py -f -i -s
其中 –f 定义 的名称。umd 文件 –i 定义了要丢弃的热化周期,–s 定义了存储在 中的轨迹的采样频率。umd 文件。默认值为 –i 0 –s 1,即考虑模拟的所有步骤,而不会丢弃任何步骤。 - 使用 umd2poscar.py 脚本将 umd 文件反转为 VASP 类型的 POSCAR 文件;可以使用预定义的频率选择模拟的快照。在命令行类型处:
umd2poscar.py -f -i -l -s
其中 –l 表示要转换为 POSCAR 文件的最后一步。默认值为 -i 0 -l 10000000 -s 1。此值 –l 足以覆盖典型的整个轨迹。
2. 执行结构分析
- 运行gofrs_umd.py脚本以计算所有原子类型 A 和 B 对的对分布函数 (PDF) gᴀʙ(r)(图 3)。输出写在一个 ASCII 文件中,以制表符分隔,扩展名为 gofrs.dat。在命令行类型处:
gofrs_umd.py -f -s < Sampling_Frequency > -d <离散化中断> -i <初始步骤>
注:默认值为Sampling_Frequency(对轨迹进行采样的频率)= 1步;离散化间距(用于绘制 g(r)) = 0.01 Å;初始步骤(轨迹开始时丢弃的步骤数)= 0。径向PDF,gᴀʙ(r)是半径为r的球形壳内距离d_ᴀʙ的B型原子的平均数量,厚度dr以A型原子为中心(图3):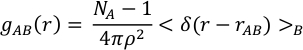
ρ为原子密度,NA和NB为A型和B型原子数,δ(r−rᴀʙ)如果原子A和B位于r和r + dr之间的距离,则delta函数等于1。gᴀʙ(r)的第一个最大值的横坐标给出了A型和B型原子之间的最高概率键长,这是最接近我们可以确定的平均键距离。第一个最小值限定了第一个协调范围的范围。因此,PDF上直到第一个最小值的积分给出了平均协调数。对于所有原子类型 A 和 B 的对,gᴀʙ(r) 的傅里叶变换之和产生流体的衍射图谱,这是用衍射仪通过实验获得的。然而,在现实中,由于gᴀʙ(r)中经常缺少高阶配位球,因此衍射图无法完整地获得。

图 3:对分布函数的确定。
(a) 对于一个物种(例如红色)的每个原子,协调物种的所有原子(例如灰色和/或红色)都算作距离的函数。(b) 每个快照的距离分布图,在此阶段只是δ函数的集合,然后对所有原子和所有快照进行平均,并按理想气体分布加权,以生成(c)连续的对分布函数。 g(r) 的第一个最小值是第一个配位球体的半径,稍后在物种形成分析中使用。 请点击此处查看此图的放大版本。
- 提取平均原子间键距离作为第一个配位球的半径。为此,请确定 gᴀʙ(r) 函数的第一个最大值的位置:在电子表格应用程序中绘制 gofrs.dat 文件,并搜索每对原子的最大值和最小值。
- 使用电子表格软件将第一个协调球体的半径标识为 PDF 的第一个最小值 gᴀʙ(r)。这是流体整个结构分析的基础;PDF产生流体中原子的平均键合状态。
- 提取第一个最小值(即横坐标)的距离,并将它们写入一个单独的文件,例如,称为 bonds.input。或者,运行 UMD 包的analyze_gofr脚本之一,以标识 gᴀʙ(r) 函数的最大值和最小值。在命令行类型处:
analyze_gofr_semi_automatic.py - 单击程序打开的图形中显示的 gᴀʙ(r) 函数的最大值和最小值的位置。该脚本会自动扫描当前文件夹,识别所有 gofrs.dat 文件,并对其中每个文件执行分析。每次脚本需要有根据的初始猜测时,再次单击窗口中的最大值和最小值。
- 打开并查看自动生成的名为 bonds.input 的文件,其中包含原子间键距离。
3. 执行形态分析
- 使用图论中的连通性概念计算原子之间键合的拓扑:原子是节点,原子间键是路径。 speciation_umd.py 脚本需要 bond.input 文件中定义的原子间键距。
注意:连通矩阵是在每个时间步长处构建的:两个原子的距离小于其相应的第一配位球半径,被认为是键合的,即连接的。通过将原子视为图中的节点来构建各种原子网络,其连接由此几何标准定义。这些网络是原子物种,它们的集合定义了该特定流体中的原子物种形成(图4)。
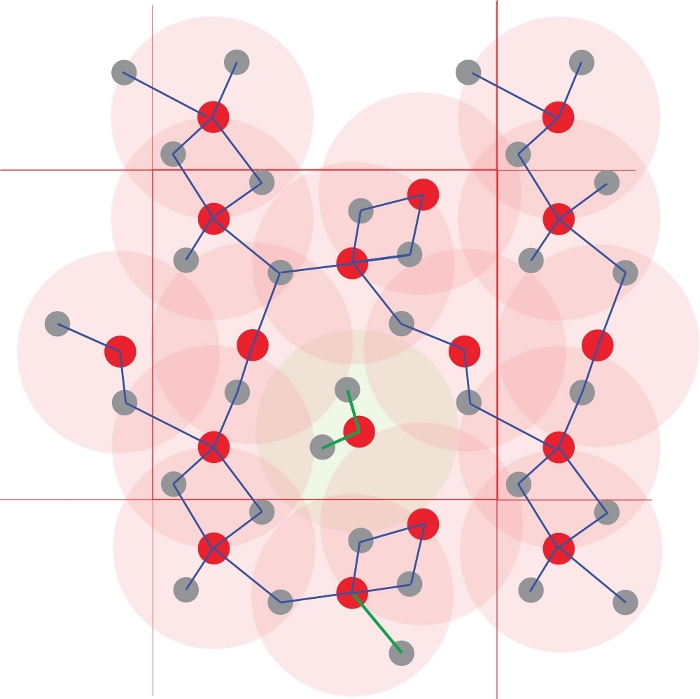
图 4:原子团簇的标识。
配位多面体使用原子间距离定义。距离小于指定半径的所有原子都被认为是键合的。此处的阈值对应于图 1 中定义的第一个协调球体(浅红色圆圈)。聚合和化学物质是从键合原子的网络中获得的。注意中心的Red1Grey2团簇,它与其他原子分离,形成无限的聚合物。请点击此处查看此图的放大版本。
- 运行形态形成脚本以获得连通矩阵并获得配位多面体或聚合。在命令行类型处:
speciation_umd.py -f -s -i -l -c -a <阴离子> -m -r
其中 -i 标志为文件提供了原子间键距离,例如在上一步中生成了该距离。或者,对 -l 标志定义的所有绑定使用一个长度运行脚本。
注意:-c 标志指定中心原子,-a 标志指定配体。中心原子和配体都可以是不同类型的;在这种情况下,它们必须用逗号分隔。-m 标志给出了在分析中考虑物种必须存活的最短时间。默认情况下,此最短时间为零,在最终分析中计算所有匹配项。- 运行带有标志 –r 0 的speciation_umd.py脚本,该脚本在第一级对连通性图进行采样以标识协调多面体。例如,表示为 阳离子 的中心原子可以被一个或多个 阴离子 包围(图4)。物种形成脚本识别每一个配位多面体。所有配位多面体的加权平均值给出了配位数,与从PDF积分中获得的配位数相同。在命令行类型处:
speciation_umd.py -f -i -c -a <阴离子> -r 0
注:流体中的平均配位数是小数。这种分数来自协调的平均特征。基于物种形成的定义可以更直观,更翔实地表示流体的结构,其中不同物种的相对比例(即配位)被量化。 - 使用标志 –r 1 运行 speciation_umd.py 脚本,该脚本在所有深度级别对连接图进行采样以获得聚合。通过原子图的网络具有一定的深度,因为原子被更远地键合到其他键(例如,在交替阳离子和阴离子的序列中)(图4)。
- 运行带有标志 –r 0 的speciation_umd.py脚本,该脚本在第一级对连通性图进行采样以标识协调多面体。例如,表示为 阳离子 的中心原子可以被一个或多个 阴离子 包围(图4)。物种形成脚本识别每一个配位多面体。所有配位多面体的加权平均值给出了配位数,与从PDF积分中获得的配位数相同。在命令行类型处:
- 打开两个文件。人口.dat 和.统计.dat 连续;它们构成了物种形成脚本的输出。每个簇都写在一行上,指定其化学式,形成时间,死亡时间,寿命,矩阵,其中包含形成该簇的原子列表。绘制模拟中发现的所有化学物质的每个原子簇的寿命,如 .popul.dat 文件所示(图 5)。
- 绘制种群分析与每个物种的丰度,如 所示。统计.dat 文件。该分析,绝对和相对,对应于情况-r 0的配位多面体的实际统计量;对于聚合的情况,对于-r 1,这需要仔细处理,因为可能需要对相对数量的原子施加一些归一化。丰度对应于一生中的积分。这。stat.dat 文件还列出了每个簇的大小,即形成它的原子数量。
4. 计算扩散系数
- 提取原子的均方位移(MSD)作为时间的函数,以获得自扩散率。MSD的标准配方为:

其中前置因子是重整化。使用MSD工具,有不同的方法来分析流体的动力学方面。
注意:T 是模拟的总时间,N α是α类型的原子数。初始时间 t0 是任意的,跨越了模拟的前半部分。Ninit 是初始时间的次数。τ 是计算 MSD 的时间间隔的宽度;其最大值是模拟时间长度的一半。在典型的 MSD 实现中,每个窗口都从前一个窗口的末尾开始。但是,稀疏采样可以加快MSD的计算速度,而不会改变MSD的斜率。为此,第 i 个窗口从时间 t0(i) 开始,但 (i+1) -第 5 个窗口从时间 t0(i) + τ + v 开始,其中 v 的值是用户定义的。类似地,窗口的宽度以用户定义的离散步长增加,如下所示:τ(i) = τ(i-1) + z。z("水平步长")和v("垂直步长")的值为正或零;两者的默认值均为 20。 - 使用一系列 msd_umd 脚本计算 MSD。它们的输出打印在 .msd.dat 文件,其中每个原子类型、原子或簇的 MSD 作为时间的函数打印在一列上。
- 计算每种原子类型的平均 MSD。为每个原子计算MSD,然后为每个原子类型取平均值。输出文件包含每个原子类型的一列。在命令行类型处:
msd_umd.py -f -z -v -b - 计算每个原子的MSD。为每个原子计算MSD,然后为每个原子类型取平均值。输出文件包含模拟中每个原子的一列,然后每个原子类型包含一列。此功能允许识别在两种不同环境中扩散的原子,例如液体和气体,或两种液体。在命令行类型处:
msd_all_umd.py -f -z -v -b - 计算化学物质的 MSD。使用用物种形成脚本标识并打印在 中的聚类群体。popul.dat 文件。MSD 是为每个单独的群集计算的。输出文件包含每个群集的一列。为避免考虑大规模聚合物,请对簇的大小施加限制;它的默认值为 20 个原子。在命令行类型处:
msd_cluster_umd.py -f -p -s -b -c
注意:默认值为:–b 100 –s 1 –c 20。
- 计算每种原子类型的平均 MSD。为每个原子计算MSD,然后为每个原子类型取平均值。输出文件包含每个原子类型的一列。在命令行类型处:
- 使用基于电子表格的软件绘制 MSD(图 6)。在 MSD 与时间的对数-对数表示中,确定斜率变化。分开第一部分,通常很短,它代表 弹道 制度,即碰撞后原子速度的守恒。第二个较长的部分表示 扩散 状态,即碰撞后原子速度的散射。
- 从 MSD 的斜率计算扩散系数为:
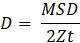
其中 Z 是自由度数(Z = 2 表示平面上的扩散,Z = 3 表示空间中的扩散),t 是时间步长。
5. 时间相关函数
- 使用一般公式计算时间相关性函数作为系统惯性的度量:

A 可以是各种时间相关变量,例如原子位置,原子速度,应力,极化等,每个变量都通过绿色 - Kubo关系产生不同的物理性质12,13,有时经过进一步的变换。 - 分析原子速度,得到液体的振动光谱和原子自扩散系数的替代表达式。
- 运行 vibr_spectrum_umd.py 脚本以计算每种原子类型的原子速度-速度自相关 (VAC) 函数,并执行其快速傅里叶变换。在命令行类型处:
vibr_spectrum_umd.py -f -t <温度>
其中 –t 是用户必须定义的温度。该脚本将打印两个文件:.vels.scf.dat 具有每种原子类型的 VAC 函数的文件,以及 .vibr.dat 文件,其中包含分解的振动光谱和每个原子物种的总值。 - 打开并阅读 vels.scf.dat。使用类似电子表格的软件从 vels.scf.dat 文件绘制 VAC 函数。
- 保留傅里叶VAC的真实部分。这就是产生振动频谱的原因,作为频率的函数:

其中 m 是原子质量。 - 使用类似电子表格的软件从 振动.dat 文件中绘制振动频谱(图7)。确定 ω=0 处的有限值,该值对应于流体的扩散特性和有限频率下频谱的各种峰值。确定每种原子类型对振动谱的参与。
注意:原子类型的分解表明,不同的原子具有不同的 ω= 0贡献,对应于它们的扩散系数。光谱的一般形状比相应的实体更平滑,特征更少。 - 在壳体上,读取振动光谱上的积分,该积分产生每种原子物种的扩散系数。
注意:热力学性质可以通过从振动光谱积分获得,但应谨慎使用结果,因为有两个近似值:积分在准谐波近似中有效,这在高温下不一定成立;并且需要丢弃与扩散相对应的光谱的气体样部分。然后,集成应仅在频谱的晶格状部分上进行。但这种分离通常需要几个进一步的后处理步骤和计算14,而这些步骤和计算在本 UMD 包中未涵盖。
- 运行 vibr_spectrum_umd.py 脚本以计算每种原子类型的原子速度-速度自相关 (VAC) 函数,并执行其快速傅里叶变换。在命令行类型处:
- 运行 viscosity_umd.py 脚本来分析组件应力张量的自相关,以估计熔体的粘度。在命令行类型处:
viscosity_umd.py -f -i -s -o -l
注意:此功能是探索性的,必须谨慎对待任何结果。首先,彻底检查粘度相对于模拟长度的收敛性。- 从应力张量的自相关推导出流体的粘度15 为:

其中 V 和 T 分别是体积和温度,κB 是玻尔兹曼常数,σ ij 是应力张量的 ij 对角线外分量,以笛卡尔坐标表示。 - 使用更合适的拟合来获得更稳健的粘度估计值15,16 ,并避免应力-张量自相关函数的噪声,该噪声可能由模拟的有限大小和有限持续时间引起。对于应力张量的自相关函数,请使用以下函数形式15,16 ,该函数形式会产生良好的结果:
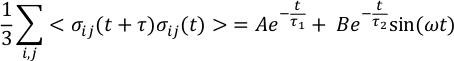
其中 A、B、τ1、τ2 和 ω 是拟合参数。积分后,粘度的表达式变为: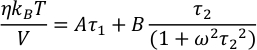
- 从应力张量的自相关推导出流体的粘度15 为:
6. 仿真产生的热力学参数。
- 运行 averages.py ,从 umd 文件中提取压力、温度、密度和内部能量的平均值和点差(作为标准差)。在命令行类型处:
averages.py -f -s
默认值为 –s 0。 - 使用阻塞方法计算平均值的统计误差。
注意:此方法有多种风格。根据 Allen 和 Tildesley2 的工作,通常对长度越来越长的时间块序列进行平均,并估计相对于算术平均值的标准偏差17。当采样不相关时,可以在多个且足够长的块大小的限制下达到收敛。尽管收敛的实际阈值通常需要手动选择。- 使用减半方法18:从初始数据样本开始,在每个步骤 κ 处,通过对上一步 κ−1 中每两个相应的连续样本进行平均,将样本数量减半:
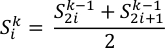
- 运行 fullaverages.py 脚本以执行完整的统计分析,包括均值的误差。在命令行类型处:
fullaverages.py -s -u <单位>
注意:该脚本已自动执行,以搜索当前目录中的所有 .umd.dat 文件,并对所有文件执行分析。默认值为 –s 0 –u 0。对于 -u 0,输出是最小的,对于 -u 1 输出是完整的,并打印出几个替代单元。此脚本需要图形支持,因为它会创建一个图形图像,用于检查收敛以估计均值上的误差。
- 使用减半方法18:从初始数据样本开始,在每个步骤 κ 处,通过对上一步 κ−1 中每两个相应的连续样本进行平均,将样本数量减半:
结果
Pyrolite 是一种模型式多组分硅酸盐熔体(0.5Na2O 2CaO 1.5Al2O3 4FeO 30MgO 24SiO2),最能接近散装硅酸盐地球的成分 - 地球化学平均值或我们的整个星球,除了铁基核心19。早期的地球被一系列大规模的融化事件所主导20,最后一次可能在原月盘凝结之后吞没了整个星球21。热浮岩代表了这种行星级岩浆海洋组成的最佳近似值。因此,我们通过 VASP 实施中的从头开始分子动力学模拟,广泛研究了高温岩熔体在 3,000\u2012,000 K 温度范围和 0\u2012150 GPa 压力范围内的物理性质。这些热力学条件完全是地球上最极端的岩浆海洋条件的特征。我们的研究是成功使用UMD封装进行熔体深入分析的一个很好的例子22。我们计算了分布和平均键长,我们追踪了阳离子氧配位的变化,并将我们的结果与先前对各种成分的无定形硅酸盐的实验和计算研究进行了比较。我们的深入分析有助于将标准配位数分解为其基本成分,概述熔体中外来配位多面体的存在,并提取所有配位多面体的寿命。它还概述了模拟中采样的重要性,包括轨迹的长度以及建模系统中存在的原子数量。至于后处理,UMD分析独立于这些因素,但是,在解释UMD包提供的结果时应考虑这些因素。在这里,我们展示了一些如何使用UMD包提取熔体的几个特征的例子,并应用于熔融热浮石。
从gofrs_umd.py脚本获得的Si-O对分布函数表明,第一个配位球体的半径(g(r)函数的第一个最小值)在T = 3000 K和P = 4.6 GPa时约为2.5埃。g(r) 的最大值为 1.635 Å,这是弯曲长度的最佳近似值。长尾巴是由于温度。使用这个极限作为Si-O键距离,形态分析表明,SiO4单元可以持续长达几皮秒,主导着熔体(图5)。熔体中有一个重要部分显示部分聚合,如Si2O7等二聚体和Si3Ox单元等三聚体的存在所反映的那样。它们相应的生存期是皮秒级的。高阶聚合物的使用寿命都大大缩短。
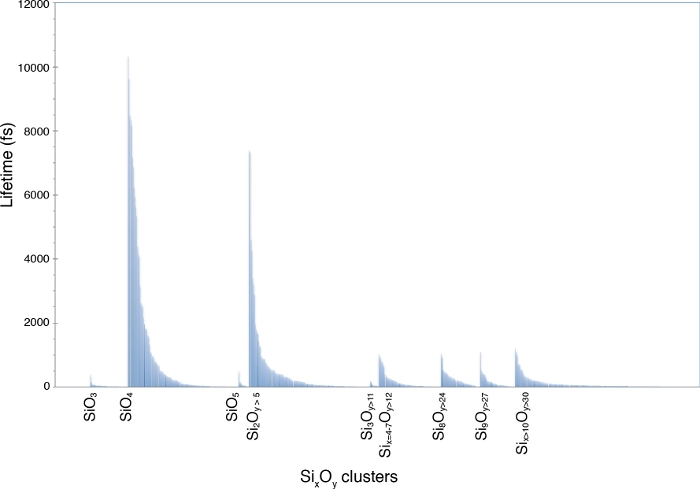
图5:Si-O化学物质的寿命。
在4.6 GPa和3000 K的多组分熔体中鉴定物种形成。标签上标记了 SiO3、SiO4 和 SiO5 单体以及各种 SixOy 聚合物。请点击此处查看此图的放大版本。
垂直和水平步长的不同值(由上面的 –z 和 –v 标志定义)产生 MSD 的各种采样(图 6)。即使z和v的值很大,也足以定义斜率,从而定义不同原子的扩散系数。当获得 z 和 v 的大值时,后处理的时间增益非常显著。MSD为模拟质量提供了非常强大的验证标准。如果MSD的扩散部分不够长,则表明模拟太短,并且无法达到统计意义上的流体状态。MSD扩散部分的最低要求在很大程度上取决于系统。可以要求所有原子在熔体结构中至少改变一次其位点,以便将其视为流体10。在行星科学中应用的一个很好的例子是复杂的硅酸盐熔体在接近甚至低于其液相线11的高压下熔体。Si原子是主要的网络形成阳离子,在二十多个皮秒之后交换位点。如果仿真时间短于此阈值,则会对可能的配置空间进行大量采样。然而,由于配位阴离子,即O原子,比中心Si原子移动得更快,它们可以补偿Si的一部分慢迁移率。因此,整个系统确实可以覆盖比仅从Si位移中假设的更好的配置空间采样。
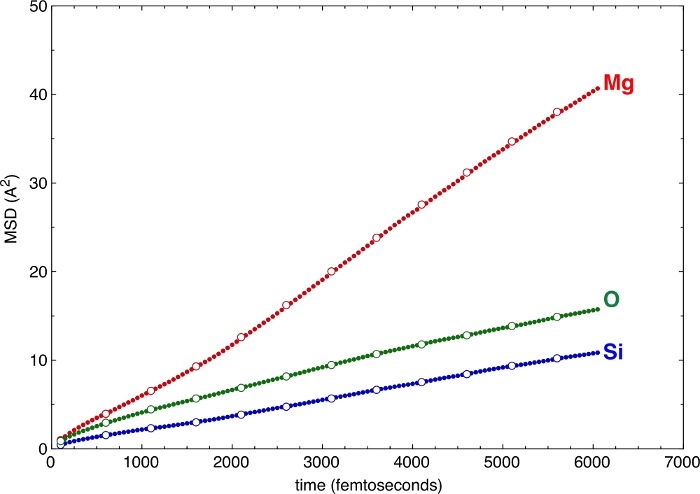
图 6:均方位移 (MSD)。
MSD用于多组分硅酸盐熔体的几种原子类型。使用各种水平和垂直步长(z 和 v)进行采样可产生一致的结果。实心圆:-z 50 –v 50。开环: -z 250 –v 500. 请点击此处查看此图的放大版本。
最后,原子VAC函数产生熔体的振动谱。 图7 显示了与上述相同的压力和温度条件下的光谱。我们代表了Mg,Si和O原子的贡献,以及总值。在零频率下,光谱有一个有限的值,对应于熔体的扩散特性。从振动光谱中提取热力学性质需要从零中去除这种类似气体的扩散特性,但也要适当考虑其在较高频率下的衰减。

图7:热熔体的振动光谱。
原子速度-速度自相关函数的傅里叶变换的实部产生振动谱。 这里计算了多组分硅酸盐熔体的光谱。流体在零频率下具有非零气体样扩散特性。 请点击此处查看此图的放大版本。
讨论
UMD 包旨在更好地与 ab initio 模拟配合使用,其中快照数量通常限制为数十到数十万个快照,每个单元单元只有几百个原子。如果运行后处理的计算机具有足够的活动内存资源,则还可以处理较大的模拟。该代码通过其可以计算的各种属性及其开源许可证来区分自己。
umd.dat 文件适用于在整个模拟过程中保持粒子数不变的融合。UMD 包可以读取来自模拟盒形状和体积变化的计算产生的文件。这些涵盖了最常见的计算,如NVT和NPT,其中粒子的数量,N,温度T,体积,V和/或压力P保持恒定。
对于开始的时间,对分布函数以及所有需要估计原子间距离的脚本(如物种形成脚本)仅适用于正交单元单元,这意味着对于立方,四方和正交单元,其中轴之间的角度为90°。
2.0版的主要开发路线是消除距离的正交性限制,并为物种形成脚本添加更多特征:分析单个化学键,分析原子间角,并实现第二个配位球体。在外部协作的帮助下,我们正在努力将代码移植到GPU上,以便在更大的系统中更快地进行分析。
披露声明
作者没有什么可透露的。
致谢
这项工作得到了欧洲研究理事会(ERC)在欧盟地平线2020研究和创新计划(资助协议编号681818 IMPACT to RC)下的支持,由深碳天文台极端物理和化学理事会以及挪威研究理事会通过其卓越中心资助计划(项目编号223272)提供支持。我们承认通过stl2816系列eDARI计算授权访问GENCI超级计算机,通过PRACE RA4947项目访问Irene AMD超级计算机,以及通过UNINETT Sigma2 NN9697K访问Fram超级计算机。金融服务得到了Marie Skłodowska-Curie项目的支持(赠款协议ABISSE No.750901)。
材料
| Name | Company | Catalog Number | Comments |
| getopt library | open-source | ||
| glob library | open-source | ||
| matplotlib library | open-source | ||
| numpy library | open-source | ||
| os library | open-source | ||
| Python software | The Python Software Foundation | Version 2 and 3 | open-source |
| random library | open-source | ||
| re library | open-source | ||
| scipy library | open-source | ||
| subprocess library | open-source | ||
| sys library | open-source |
参考文献
- Frenkel, D., Smit, B. . Understanding Molecular Simulation. From Algorithms to Applications. , (2001).
- Allen, M. P., Tildesley, D. J., Allen, T. . Computer Simulation of Liquids. , (1989).
- Zepeda-Ruiz, L. A., Stukowski, A., Oppelstrup, T., Bulatov, V. V. Probing the limits of metal plasticity with molecular-dynamics simulations. Nature Publishing Group. 550 (7677), 492-495 (2017).
- Humphrey, W., Dalke, A., Schulten, K. VMD: Visual molecular dynamics. Journal of Molecular Graphics & Modeling. 14 (1), 33-38 (1996).
- Momma, K., Izumi, F. VESTA3 for three-dimensional visualization of crystal, volumetric and morphology data. Journal of Applied Crystallography. 44 (6), 1272-1276 (2011).
- Brehm, M., Kirchner, B. TRAVIS - A free Analyzer and Visualizer for Monte Carlo and Molecular Dynamics Trajectories. Journal of Chemical Information and Modeling. 51 (8), 2007-2023 (2011).
- Stixrude, L. Visualization-based analysis of structural and dynamical properties of simulated hydrous silicate melt. Physics and Chemistry of Minerals. 37 (2), 103-117 (2009).
- Kresse, G., Hafner, J. Ab initio Molecular-Dynamics for Liquid-Metals. Physical Review B. 47 (1), 558-561 (1993).
- Gygi, F. Architecture of Qbox: A scalable first-principles molecular dynamics code. IBM Journal of Research and Development. 52 (1-2), 137-144 (2008).
- Harvey, J. P., Asimow, P. D. Current limitations of molecular dynamic simulations as probes of thermo-physical behavior of silicate melts. American Mineralogist. 100 (8-9), 1866-1882 (2015).
- Caracas, R., Hirose, K., Nomura, R., Ballmer, M. D. Melt-crystal density crossover in a deep magma ocean. Earth and Planetary Science Letters. 516, 202-211 (2019).
- Green, M. S. Markoff Random Processes and the Statistical Mechanics of Time-Dependent Phenomena. II. Irreversible Processes in Fluids. The Journal of Chemical Physics. 22 (3), 398-413 (1954).
- Kubo, R. Statistical-Mechanical Theory of Irreversible Processes. I. General Theory and Simple Applications to Magnetic and Conduction Problems. Journal of the Physical Society of Japan. 12 (6), 570-586 (1957).
- Lin, S. T., Blanco, M., Goddard, W. A. The two-phase model for calculating thermodynamic properties of liquids from molecular dynamics: Validation for the phase diagram of Lennard-Jones fluids. The Journal of Chemical Physics. 119 (22), 11792-11805 (2003).
- Meyer, E. R., Kress, J. D., Collins, L. A., Ticknor, C. Effect of correlation on viscosity and diffusion in molecular-dynamics simulations. Physical Review E. 90 (4), 1198-1212 (2014).
- Soubiran, F., Militzer, B., Driver, K. P., Zhang, S. Properties of hydrogen, helium, and silicon dioxide mixtures in giant planet interiors. Physics of Plasmas. 24 (4), 041401-041407 (2017).
- Flyvbjerg, H., Petersen, H. G. Error estimates on averages of correlated data. The Journal of Chemical Physics. 91 (1), 461-466 (1989).
- Tuckerman, M. E. . Statistical mechanics: theory and molecular simulation. , (2010).
- McDonough, W. F., Sun, S. S. The composition of the Earth. Chemical Geology. 120, 223-253 (1995).
- Elkins-Taton, L. T. Magma oceans in the inner solar system. Annual Review of Earth and Planetary Sciences. 40, 113-139 (2012).
- Lock, S. J., et al. The origin of the Moon within a terrestrial synestia. J. Geophysical Research: Planets. 123, 910-951 (2018).
- Solomatova, N. V., Caracas, R. Pressure-induced coordination changes in a pyrolitic silicate melt from ab initio molecular dynamics simulations. Journal of Geophysical Research: Solid Earth. 124, 11232-11250 (2019).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。