Method Article
Analyse des fusions et des fluides à partir de simulations de dynamique moléculaire Ab Initio avec le package UMD
Dans cet article
Résumé
Les fusions et les fluides sont des vecteurs omniprésents de transport de masse dans les systèmes naturels. Nous avons développé un progiciel open source pour analyser les simulations de dynamique moléculaire ab initio de tels systèmes. Nous calculons les propriétés structurelles (liaison, clusterisation, spéciation chimique), de transport (diffusion, viscosité) et thermodynamiques (spectre vibratoire).
Résumé
Nous avons développé un paquet open-source basé sur Python pour analyser les résultats issus de simulations de dynamique moléculaire ab initio de fluides. L’emballage est le mieux adapté aux applications sur les systèmes naturels, comme les fondus de silicate et d’oxyde, les fluides à base d’eau et divers fluides supercritiques. Le paquet est une collection de scripts Python qui incluent deux bibliothèques majeures traitant des formats de fichiers et de la cristallographie. Tous les scripts sont exécutés sur la ligne de commande. Nous proposons un format simplifié pour stocker les trajectoires atomiques et les informations thermodynamiques pertinentes des simulations, qui sont enregistrées dans des fichiers UMD, pour Universal Molecular Dynamics. Le package UMD permet le calcul d’une série de propriétés structurelles, de transport et thermodynamiques. En commençant par la fonction de distribution par paires, il définit les longueurs de liaison, construit une matrice de connectivité interatomique et détermine finalement la spéciation chimique. La détermination de la durée de vie de l’espèce chimique permet d’effectuer une analyse statistique complète. Ensuite, des scripts dédiés calculent les déplacements moyens-carrés pour les atomes ainsi que pour les espèces chimiques. L’analyse d’autocorrélation mise en œuvre des vitesses atomiques donne les coefficients de diffusion et le spectre vibratoire. La même analyse appliquée sur les contraintes donne la viscosité. Le package est disponible via le site Web GitHub et via sa propre page dédiée du projet ERC IMPACT en tant que package en libre accès.
Introduction
Les fluides et les fondus sont des vecteurs de transport chimiques et physiques actifs dans les environnements naturels. Les taux élevés de diffusion atomique favorisent les échanges et les réactions chimiques, la faible viscosité associée à une flottabilité variable favorise un transfert de masse important, et les relations de densité cristal-fusion favorisent la superposition à l’intérieur des corps planétaires. L’absence de réseau périodique, les températures élevées typiques requises pour atteindre l’état fondu et la difficulté de trempe rendent la détermination expérimentale d’une série de propriétés évidentes, telles que la densité, la diffusion et la viscosité, extrêmement difficile. Ces difficultés font des méthodes de calcul alternatives des outils solides et utiles pour étudier cette classe de matériaux.
Avec l’avènement de la puissance de calcul et la disponibilité des superordinateurs, deux techniques majeures de simulations atomistiques numériques sont actuellement utilisées pour étudier l’état dynamique d’un système atomistique non cristallin, Monte Carlo1 et la dynamique moléculaire (MD)1,2. Dans les simulations de Monte Carlo, l’espace de configuration est échantillonné de manière aléatoire ; Les méthodes de Monte Carlo montrent une mise à l’échelle linéaire en parallélisation si toutes les observations d’échantillonnage sont indépendantes les unes des autres. La qualité des résultats dépend de la qualité du générateur de nombres aléatoires et de la représentativité de l’échantillonnage. Les méthodes de Monte Carlo montrent une mise à l’échelle linéaire en parallélisation si l’échantillonnage est indépendant les uns des autres. En dynamique moléculaire (MD), l’espace de configuration est échantillonné par des trajectoires atomiques dépendantes du temps. A partir d’une configuration donnée, les trajectoires atomiques sont calculées en intégrant les équations newtoniennes du mouvement. Les forces interatomiques peuvent être calculées à l’aide de potentiels interatomiques modèles (dans la DM classique) ou à l’aide de méthodes de premiers principes (in ab initio, ou premiers principes, MD). La qualité des résultats dépend de la longueur de la trajectoire et de sa capacité à ne pas être attirée par les minima locaux.
Les simulations de dynamique moléculaire contiennent une pléthore d’informations, toutes liées au comportement dynamique du système. Les propriétés moyennes thermodynamiques, comme l’énergie interne, la température et la pression, sont plutôt standard à calculer. Ils peuvent être extraits du ou des fichiers de sortie des simulations et moyennés, tandis que les quantités directement liées au mouvement des atomes ainsi qu’à leur relation mutuelle doivent être calculées après extraction des positions atomiques et des vitesses.
Par conséquent, beaucoup d’efforts ont été consacrés à la visualisation des résultats, et divers paquets sont disponibles aujourd’hui, sur différentes plateformes, open source ou non [Ovito3, VMD4, Vesta5, Travis6, etc.]. Tous ces outils de visualisation traitent efficacement les distances interatomiques et, en tant que tels, ils permettent le calcul efficace des fonctions de distribution des paires et des coefficients de diffusion. Divers groupes effectuant des simulations de dynamique moléculaire à grande échelle disposent d’un logiciel propriétaire pour analyser diverses autres propriétés résultant des simulations, parfois dans des sharewares ou d’autres formes d’accès limité à la communauté, et parfois limités dans la portée et l’utilisation à certains paquets spécifiques. Des algorithmes sophistiqués pour extraire des informations sur la liaison interatomique, les motifs géométriques et la thermodynamique sont développés et mis en œuvre dans certains de ces packages3,4,5,6,7, etc.
Nous proposons ici le paquet UMD - un paquet open-source écrit en Python pour analyser le résultat des simulations de dynamique moléculaire. Le package UMD permet de calculer un large éventail de propriétés structurelles, dynamiques et thermodynamiques (Figure 1). Le package est disponible via le site Web GitHub (https://github.com/rcaracas/UMD_package) et via une page dédiée (http://moonimpact.eu/umd-package/) du projet ERC IMPACT en tant que package en libre accès.
Pour le rendre universel et plus facile à manipuler, notre approche consiste d’abord à extraire toutes les informations relatives à l’état thermodynamique et aux trajectoires atomiques du fichier de sortie de l’exécution de la dynamique moléculaire réelle. Ces informations sont stockées dans un fichier dédié, dont le format est indépendant du package MD d’origine où la simulation a été exécutée. Nous nommons ces fichiers « umd » files, ce qui signifie Universal Molecular Dynamics. De cette façon, notre package UMD peut être facilement utilisé par n’importe quel groupe ab initio avec n’importe quel logiciel, le tout avec un minimum d’effort d’adaptation. La seule exigence pour utiliser le package actuel est d’écrire l’analyseur approprié à partir de la sortie du logiciel MD particulier dans le format de fichier umd, si celui-ci n’existe pas encore. Pour le moment, nous fournissons de tels analyseurs pour les packages VASP8 et QBox9 .

Figure 1 : Organigramme de la bibliothèque UMD.
Les propriétés physiques sont en bleu, et les principaux scripts Python et leurs options sont en rouge. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Les fichiers umd sont des fichiers ASCII ; l’extension typique est « umd.dat » mais pas obligatoire. Tous les composants d’analyse peuvent lire les fichiers ASCII du format umd, quelle que soit l’extension de nom réelle. Cependant, certains des scripts automatiques conçus pour effectuer des statistiques rapides à grande échelle sur plusieurs simulations recherchent spécifiquement des fichiers avec l’extension umd.dat. Chaque propriété physique est exprimée sur une ligne. Chaque ligne commence par un mot-clé. De cette façon, le format est très adaptable et permet d’ajouter de nouvelles propriétés au fichier umd, tout en préservant sa lisibilité tout au long des versions. Les 30 premières lignes du fichier umd de la simulation de pyrolite à 4,6 GPa et 3000 K, utilisées ci-dessous dans la discussion, sont illustrées à la figure 2.

Figure 2 : Début du fichier umd décrivant la simulation de pyrolite liquide à 4,6 GPa et 3000 K.
L’en-tête est suivi de la description de chaque instantané. Chaque propriété est écrite sur une ligne, contenant le nom de la propriété physique, la ou les valeurs et les unités, toutes séparées par des espaces. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Tous les fichiers umd contiennent un en-tête décrivant le contenu de la cellule de simulation : le nombre d’atomes, d’électrons et de types atomiques, ainsi que des détails pour chaque atome, tels que son type, son symbole chimique, le nombre d’électrons de valence et sa masse. Une ligne vide marque la fin de l’en-tête et le sépare de la partie principale du fichier umd.
Ensuite, chaque étape de la simulation est détaillée. Tout d’abord, les paramètres thermodynamiques instantanés sont donnés, chacun sur une ligne différente, en spécifiant (i) le nom du paramètre, comme l’énergie, les contraintes, la pression hydrostatique équivalente, la densité, le volume, les paramètres du réseau, etc., (ii) sa ou ses valeurs, et (iii) ses unités. Un tableau décrivant les atomes vient ensuite. Une ligne d’en-tête donne les différentes mesures, comme les positions cartésiennes, les vitesses, les charges, etc., et leurs unités. Ensuite, chaque atome est détaillé sur une ligne. Par groupes de trois, correspondant aux trois axes x, y, z , les entrées sont : les positions réduites, les positions cartésiennes repliées dans la cellule de simulation, les positions cartésiennes (qui tiennent correctement compte du fait que les atomes peuvent traverser plusieurs cellules unitaires au cours d’une simulation), les vitesses atomiques et les forces atomiques. Les deux dernières entrées sont des scalaires : charge et moment magnétique.
Deux grandes bibliothèques assurent le bon fonctionnement de l’ensemble du paquet. La bibliothèque umd_process.py traite des fichiers umd, comme la lecture et l’impression. La bibliothèque crystallography.py traite de toutes les informations relatives à la structure atomique réelle. La philosophie sous-jacente de la bibliothèque crystallography.py est de traiter le réseau comme un espace vectoriel. Les paramètres de la cellule unitaire ainsi que leur orientation représentent les vecteurs de base. L'« Espace » possède une série d’attributs scalaires (volume spécifique, densité, température et nombre spécifique d’atomes), des propriétés thermodynamiques (énergie interne, pression, capacité thermique, etc.) et une série de propriétés tensorielles (contrainte et élasticité). Les atomes peuplent cet espace. La classe « Lattice » définit cet ensemble, à côté de quelques calculs courts, comme le volume spécifique, la densité, l’obtention du réseau réciproque à partir du réseau direct, etc. La classe « Atoms » définit les atomes. Ils sont caractérisés par une série de propriétés scalaires (nom, symbole, masse, nombre d’électrons, etc.) et une série de propriétés vectorielles (la position dans l’espace, soit par rapport à la base vectorielle décrite dans la classe de Réseau, soit par rapport aux coordonnées cartésiennes universelles, vitesses, forces, etc.). En dehors de ces deux classes, la bibliothèque crystallography.py contient une série de fonctions pour effectuer une variété de tests et de calculs, tels que les distances atomiques ou la multiplication cellulaire. Le tableau périodique des éléments est également inclus sous forme de dictionnaire.
Les différents composants du package umd écrivent plusieurs fichiers de sortie. En règle générale, ce sont tous des fichiers ASCII, toutes leurs entrées sont séparées par des onglets et elles sont rendues aussi explicites que possible. Par exemple, ils indiquent toujours clairement la propriété physique et ses unités. Les fichiers umd.dat sont entièrement conformes à cette règle.
Protocole
1. Analyse des cycles de dynamique moléculaire
REMARQUE: Le package est disponible via le site Web GitHub (https://github.com/rcaracas/UMD_package) et via une page dédiée (http://moonimpact.eu/umd-package/) du projet ERC IMPACT en tant que package en libre accès.
- Extrayez chaque ensemble spécifique de propriétés physiques à l’aide d’un ou plusieurs scripts Python dédiés à partir du package. Exécutez tous les scripts sur la ligne de commande ; ils utilisent tous une série de drapeaux, qui sont aussi cohérents que possible d’un script à l’autre. Les indicateurs, leur signification et les valeurs par défaut sont tous résumés dans le tableau 1.
| Drapeau | Signification | Script l’utilisant | Valeur par défaut |
| -h | Aide courte | tout | |
| -f | Nom du fichier UMD | tout | |
| -i | Étapes de thermalisation à jeter | tout | 0 |
| -i | Fichier d’entrée contenant les liaisons interatomiques | spéciation | bonds.input |
| -s | Échantillonnage de la fréquence | msd, spéciation | 1 (chaque étape est prise en compte) |
| -a | Liste des atomes ou anions | spéciation | |
| -c | Liste des cations | spéciation | |
| -l | Longueur de la liaison | spéciation | 2 |
| -t | Température | vibrations, rhéologie | |
| -v | Discrétisation de la largeur de la fenêtre d’échantillonnage de la trajectoire pour l’analyse du déplacement moyen-carré | Msd | 20 |
| -z | Discrétisation du début de la fenêtre d’échantillonnage de la trajectoire pour l’analyse du déplacement moyen-carré | Msd | 20 |
Tableau 1 : Indicateurs les plus couramment utilisés dans le package UMD et leur signification la plus courante.
- Commencez par transformer la sortie de la simulation MD effectuée dans un code de premiers principes, comme VASP8 ou QBox9, en un fichier UMD.
- Si les simulations MD ont été effectuées dans VASP, alors au niveau de la ligne de commande, tapez :
VaspParser.py -f -i < Étape initiale>
où –f flag définit le nom du fichier VASP OUTCAR et le –i la longueur de thermalisation.
REMARQUE: L’étape initiale, définie par –i permet de rejeter les premières étapes des simulations, qui représentent la thermalisation. Dans une course typique de dynamique moléculaire, la première partie du calcul représente la thermalisation, c’est-à-dire le temps qu’il faut au système pour que tous les atomes décrivent une distribution gaussienne de la température, et pour que l’ensemble du système présente des fluctuations de la température, de la pression, de l’énergie, etc. autour des valeurs d’équilibre. Cette partie thermalisation de la simulation ne doit pas être prise en compte lors de l’analyse des propriétés statistiques du fluide.
- Si les simulations MD ont été effectuées dans VASP, alors au niveau de la ligne de commande, tapez :
- Transformez le fichier . umd dans . xyz pour faciliter la visualisation sur divers autres paquets, comme VMD4 ou Vesta5. Au niveau de la ligne de commande, tapez :
umd2xyz.py -f -i -s
où –f définit le nom du fichier . umd , –i définit la période de thermalisation à écarter, et –s la fréquence de l’échantillonnage de la trajectoire stockée dans le fichier . umd fichier. Les valeurs par défaut sont –i 0 –s 1, c’est-à-dire en tenant compte de toutes les étapes de la simulation, sans qu’aucune ne soit ignorée. - Inverser le fichier umd en fichiers POSCAR de type VASP à l’aide du script umd2poscar.py ; les instantanés des simulations peuvent être sélectionnés avec une fréquence prédéfinie. Au niveau de la ligne de commande, tapez :
umd2poscar.py -f -i -l -s
où –l représente la dernière étape à transformer en fichier POSCAR. Les valeurs par défaut sont -i 0 -l 10000000 -s 1. Cette valeur de –l est suffisamment grande pour couvrir une trajectoire entière typique.
2. Effectuer l’analyse structurelle
- Exécutez le script gofrs_umd.py pour calculer la fonction de distribution de paires (PDF) gᴀʙ(r) pour toutes les paires de types atomiques A et B (Figure 3). La sortie est écrite dans un fichier ASCII, séparé par des tabulations, avec l’extension gofrs.dat. Au niveau de la ligne de commande, tapez :
gofrs_umd.py -f -s < Sampling_Frequency > -d -i
REMARQUE: Les valeurs par défaut sont Sampling_Frequency (la fréquence d’échantillonnage de la trajectoire) = 1 étape; DiscrétisationInterval (pour tracer le g(r)) = 0,01 Å; InitialStep (nombre d’étapes au début de la trajectoire qui sont ignorées) = 0. Le PDF radial, gᴀʙ(r) est le nombre moyen d’atomes de type B à une distance d_ᴀʙ dans une coquille sphérique de rayon r et d’épaisseur dr centrée sur les atomes de type A (Figure 3) :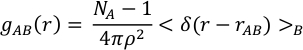
avec ρ la densité atomique, NA et NB le nombre d’atomes de type A et B, et δ(r−rᴀʙ) la fonction delta qui est égale à 1 si les atomes A et B se trouvent à une distance comprise entre r et r+dr. L’abscisse du premier maximum de gᴀʙ(r) donne la longueur de liaison de probabilité la plus élevée entre les atomes de type A et B, qui est la plus proche d’une distance de liaison moyenne que nous pouvons déterminer. Le premier minimum délimite l’étendue de la première sphère de coordination. Par conséquent, l’intégrale sur le PDF jusqu’au premier minimum donne le nombre moyen de coordination. La somme des transformées de Fourier du gᴀʙ(r) pour toutes les paires de types atomiques A et B donne le motif de diffraction du fluide, tel qu’obtenu expérimentalement avec un diffractomètre. Cependant, en réalité, comme souvent les sphères de coordination d’ordre élevé sont absentes du gᴀʙ(r), le modèle de diffraction ne peut pas être obtenu dans son intégralité.

Figure 3 : Détermination de la fonction de distribution des paires.
a) Pour chaque atome d’une espèce (par exemple rouge), tous les atomes de l’espèce coordinatrice (par exemple gris et/ou rouge) sont comptés en fonction de la distance. (b) Le graphique de distribution de distance résultant pour chaque instantané, qui à ce stade n’est qu’un ensemble de fonctions delta, est ensuite moyenné sur tous les atomes et tous les instantanés et pondéré par la distribution de gaz idéale pour générer (c) la fonction de distribution de paire qui est continue. Le premier minimum de g(r) est le rayon de la première sphère de coordination, utilisé plus tard dans l’analyse de spéciation. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Extraire les distances moyennes des liaisons interatomiques comme les rayons des premières sphères de coordination. Pour cela, identifiez la position du premier maximum des fonctions gᴀʙ(r) : tracez le fichier gofrs.dat dans un tableur et recherchez les maxima et minima pour chaque paire d’atomes.
- Identifiez le rayon de la première sphère de coordination, comme le premier minimum du PDF, gᴀʙ(r), à l’aide d’un tableur. C’est la base de toute l’analyse structurelle du fluide; le PDF donne l’état de liaison moyen des atomes dans le fluide.
- Extrayez les distances des premiers minima, c’est-à-dire l’abscisse, et écrivez-les dans un fichier séparé, appelé, par exemple, bonds.input. Vous pouvez également exécuter l’un des scripts analyze_gofr du package UMD pour identifier les maxima et les minima des fonctions gᴀʙ(r). Au niveau de la ligne de commande, tapez :
analyze_gofr_semi_automatic.py - Cliquez sur la position du maximum et du minimum de la fonction gᴀʙ(r) affichée dans le graphique ouvert par le programme. Le script analyse automatiquement le dossier actif, identifie tous les gofrs.dat fichiers et effectue l’analyse pour chacun d’eux. Cliquez à nouveau sur le maximum et le minimum dans la fenêtre chaque fois que le script a besoin d’une première supposition éclairée.
- Ouvrez et regardez le fichier généré automatiquement appelé bonds.input qui contient les distances de liaison interatomique.
3. Effectuer l’analyse de spéciation
- Calculer la topologie de liaison entre les atomes, en utilisant le concept de connectivité dans la théorie des graphes: les atomes sont les nœuds et les liaisons interatomiques sont les chemins. Le script speciation_umd.py a besoin des distances de liaison interatomique définies dans le fichier bonds.input .
REMARQUE: La matrice de connectivité est construite à chaque pas de temps: deux atomes qui se trouvent à une distance inférieure au rayon de leur première sphère de coordination correspondante sont considérés comme liés, c’est-à-dire connectés. Divers réseaux atomiques sont construits en traitant les atomes comme des nœuds dans un graphe dont les connexions sont définies par ce critère géométrique. Ces réseaux sont les espèces atomiques, et leur ensemble définit la spéciation atomique dans ce fluide particulier (Figure 4).
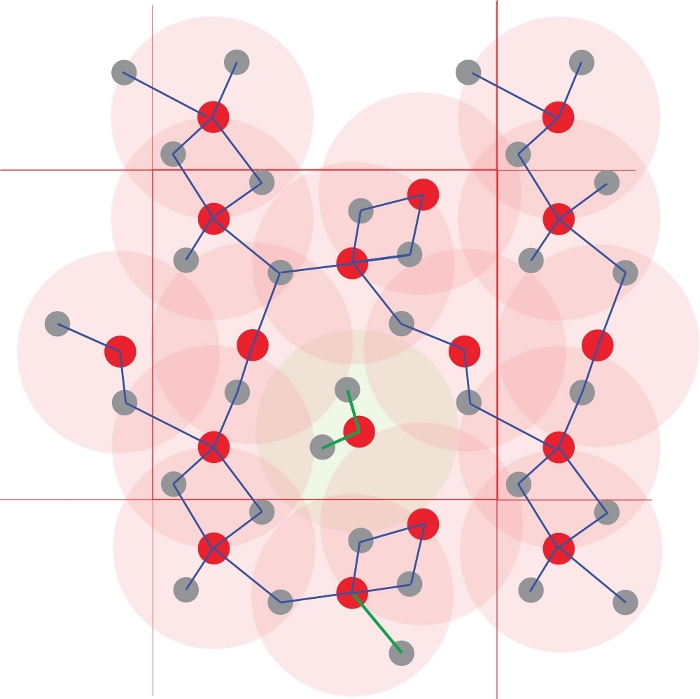
Figure 4 : Identification des amas atomiques.
Les polyèdres de coordination sont définis à l’aide de distances interatomiques. Tous les atomes à une distance inférieure à un rayon spécifié sont considérés comme liés. Ici, le seuil correspond à la première sphère de coordination (les cercles rouge clair), définie à la figure 1. La polymérisation et donc les espèces chimiques sont obtenues à partir des réseaux des atomes liés. Notez le cluster central Red1Grey2, qui est isolé des autres atomes, qui forment un polymère infini. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Exécutez le script de spéciation pour obtenir la matrice de connectivité et obtenir les polyèdres de coordination ou la polymérisation. Au niveau de la ligne de commande, tapez :
speciation_umd.py -f -s -i -l -c -a -m -r
où l’indicateur -i donne le fichier avec les distances de liaison interatomique, qui a été produit par exemple à l’étape précédente. Vous pouvez également exécuter le script avec une seule longueur pour toutes les liaisons définies par l’indicateur -l.
REMARQUE : L’indicateur -c spécifie les atomes centraux et l’indicateur -a les ligands. Les atomes centraux et les ligands peuvent être de différents types; dans ce cas, ils doivent être séparés par des virgules. Le drapeau -m donne le temps minimum de vie d’une espèce pour être prise en compte dans l’analyse. Par défaut, ce temps minimum est nul, toutes les occurrences étant comptées en dernière analyse.- Exécutez le script speciation_umd.py avec l’indicateur –r 0, qui échantillonne le graphique de connectivité au premier niveau pour identifier les polyèdres de coordination. Par exemple, un atome central, désigné comme un cation , peut être entouré d’un ou plusieurs anions (Figure 4). L’écriture de spéciation identifie chacun des polyèdres de coordination. La moyenne pondérée de tous les polyèdres de coordination donne le numéro de coordination, identique à celui obtenu à partir de l’intégration du PDF. Au niveau de la ligne de commande, tapez :
speciation_umd.py -f -i -c -a -r 0
REMARQUE: Les nombres moyens de coordination dans les fluides sont des nombres fractionnaires. Cette fractionnalité provient de la caractéristique moyenne de la coordination. La définition basée sur la spéciation donne une représentation plus intuitive et informative de la structure du fluide, où les proportions relatives des différentes espèces, c’est-à-dire les coordinations, sont quantifiées. - Exécutez le script speciation_umd.py avec l’indicateur –r 1, qui échantillonne le graphique de connectivité à tous les niveaux de profondeur pour obtenir la polymérisation. Le réseau à travers le graphe atomique a une certaine profondeur, car les atomes sont liés plus loin à d’autres liaisons (par exemple, dans des séquences de cations et d’anions alternatifs) (Figure 4).
- Exécutez le script speciation_umd.py avec l’indicateur –r 0, qui échantillonne le graphique de connectivité au premier niveau pour identifier les polyèdres de coordination. Par exemple, un atome central, désigné comme un cation , peut être entouré d’un ou plusieurs anions (Figure 4). L’écriture de spéciation identifie chacun des polyèdres de coordination. La moyenne pondérée de tous les polyèdres de coordination donne le numéro de coordination, identique à celui obtenu à partir de l’intégration du PDF. Au niveau de la ligne de commande, tapez :
- Ouvrez les deux fichiers . popul.dat et . stat.dat consécutivement; ceux-ci constituent la sortie du script de spéciation. Chaque amas est écrit sur une ligne, spécifiant sa formule chimique, l’heure à laquelle il s’est formé, l’heure à laquelle il est mort, sa durée de vie, une matrice avec la liste des atomes formant cet amas. Tracez la durée de vie de chaque amas atomique de toutes les espèces chimiques trouvées dans la simulation telles qu’elles se trouvent dans le fichier .popul.dat (Figure 5).
- Tracez l’analyse de la population avec l’abondance de chaque espèce, comme on le trouve dans le . stat.dat fichier. Cette analyse, tant absolue que relative, correspond aux statistiques réelles des polyèdres de coordination pour le cas -r 0 ; dans le cas de la polymérisation, avec -r 1, cela doit être traité avec soin, car une certaine normalisation sur le nombre relatif d’atomes pourrait devoir être appliquée. L’abondance correspond à l’intégrale au cours des vies. Le. stat.dat fichier répertorie également la taille de chaque cluster, c’est-à-dire le nombre d’atomes qui le forment.
4. Calculer les coefficients de diffusion
- Extraire les déplacements carrés moyens (MSD) des atomes en fonction du temps pour obtenir l’auto-diffusivité. La formule standard du MSD est la suivante :

où les préfacteurs sont des renormalisations. Avec l’outil MSD, il existe différentes façons d’analyser les aspects dynamiques des fluides.
REMARQUE: T est le temps total de la simulation et N α est le nombre d’atomes de type α. Le temps initial t0 est arbitraire et couvre la première moitié de la simulation. Ninit est le nombre de fois initiales. τ est la largeur de l’intervalle de temps sur lequel les TMS sont calculés; sa valeur maximale est la moitié de la durée de la simulation. Dans les implémentations MSD typiques, chaque fenêtre commence à la fin de la précédente. Mais un échantillonnage plus clairsemé peut accélérer le calcul du TMS, sans altérer la pente du TMS. Pour cela, la i-ème fenêtre commence à l’instant t0(i), mais la (i+1)-ème fenêtre commence à l’instant t0(i) + τ + v, où la valeur de v est définie par l’utilisateur. De même, la largeur de la fenêtre est augmentée en étapes discrètes définies par l’utilisateur, en tant que telles : τ(i) = τ(i-1) + z. Les valeurs de z (« pas horizontal ») et v (« pas vertical ») sont positives ou nulles ; la valeur par défaut pour les deux est 20. - Calculez la fiche MSD à l’aide de la série de scripts msd_umd . Leur sortie est imprimée dans un fichier . msd.dat fichier, où le MSD de chaque type atomique, atome ou cluster est imprimé sur une colonne en fonction du temps.
- Calculez le MSD moyen de chaque type atomique. Les MSD sont calculés pour chaque atome, puis moyennés pour chaque type atomique. Le fichier de sortie contient une colonne pour chaque type atomique. Au niveau de la ligne de commande, tapez :
msd_umd.py -f -z -v -b - Calculez le MSD de chaque atome. Les MSD sont calculés pour chaque atome, puis moyennés pour chaque type atomique. Le fichier de sortie contient une colonne pour chaque atome de la simulation, puis une colonne pour chaque type atomique. Cette fonctionnalité permet d’identifier les atomes qui diffusent dans deux environnements différents, comme le liquide et le gaz, ou deux liquides. Au niveau de la ligne de commande, tapez :
msd_all_umd.py -f -z -v -b - Calculer le TMS de l’espèce chimique. Utilisez la population de grappes identifiées avec l’écriture de spéciation et imprimées dans le fichier . popul.dat fichier. Les MSD sont calculés pour chaque cluster individuel. Le fichier de sortie contient une colonne pour chaque cluster. Pour éviter d’envisager des polymères à grande échelle, limitez la taille de la grappe; sa valeur par défaut est de 20 atomes. Au niveau de la ligne de commande, tapez :
msd_cluster_umd.py -f -p -s -b -c
REMARQUE : Les valeurs par défaut sont : –b 100 –s 1 –c 20.
- Calculez le MSD moyen de chaque type atomique. Les MSD sont calculés pour chaque atome, puis moyennés pour chaque type atomique. Le fichier de sortie contient une colonne pour chaque type atomique. Au niveau de la ligne de commande, tapez :
- Tracez le MSD à l’aide d’un tableur (Figure 6). Dans une représentation log-logarithmique du MSD par rapport au temps, identifiez le changement de pente. Séparer la première partie, généralement courte, qui représente le régime balistique , c’est-à-dire la conservation de la vitesse des atomes après les collisions. La deuxième partie plus longue représente le régime diffusif , c’est-à-dire la diffusion de la vitesse des atomes après des collisions.
- Calculer les coefficients de diffusion à partir de la pente du TMS comme suit:
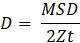
où Z est le nombre de degrés de liberté (Z = 2 pour la diffusion dans le plan, Z = 3 pour la diffusion dans l’espace), et t est le pas de temps.
5. Fonctions de corrélation temporelle
- Calculer les fonctions de corrélation temporelle comme mesure de l’inertie du système en utilisant la formule générale:

A peut être une variété de variables dépendantes du temps, telles que les positions atomiques, les vitesses atomiques, les contraintes, la polarisation, etc., chacune produisant – via les relations Green-Kubo12,13 – des propriétés physiques différentes, parfois après une transformation ultérieure. - Analyser les vitesses atomiques pour obtenir le spectre vibratoire du liquide et l’expression alternative des coefficients d’auto-diffusion atomique.
- Exécutez le script vibr_spectrum_umd.py pour calculer la fonction d’auto-corrélation vitesse-vitesse atomique (VAC) pour chaque type atomique et effectuer sa transformée de Fourier rapide. Au niveau de la ligne de commande, tapez :
vibr_spectrum_umd.py -f -t
où –t est la température qui doit être définie par l’utilisateur. Le script imprime deux fichiers : le fichier . vels.scf.dat fichier avec la fonction VAC pour chaque type atomique, et le fichier . vibr.dat fichier avec le spectre vibratoire décomposé sur chaque espèce atomique et la valeur totale. - Ouvrez et lisez le vels.scf.dat. Tracez la fonction VAC à partir du fichier vels.scf.dat à l’aide d’un logiciel de type feuille de calcul.
- Conservez la vraie partie du Fourier VAC. C’est ce qui donne le spectre vibratoire, en fonction de la fréquence:

où m sont les masses atomiques. - Tracez le spectre vibratoire à partir du fichier vibr.dat à l’aide d’un logiciel de type feuille de calcul (Figure 7). Identifier la valeur finie à ω=0 qui correspond au caractère diffusif du fluide et aux différents pics du spectre à fréquence finie. Identifier la participation de chaque type atomique au spectre vibratoire.
NOTE: La décomposition sur les types atomiques montre que différents atomes ont des contributions ω= 0 différentes, correspondant à leurs coefficients de diffusion. La forme générale du spectre est beaucoup plus lisse avec moins de caractéristiques que pour un solide correspondant. - Au niveau de la coquille, lisez l’intégrale sur le spectre vibratoire, ce qui donne les coefficients de diffusion pour chaque espèce atomique.
NOTE: Les propriétés thermodynamiques peuvent être obtenues par intégration à partir du spectre vibratoire, mais les résultats doivent être utilisés avec prudence en raison de deux approximations: l’intégration est valable dans l’approximation quasi-harmonique, qui ne tient pas nécessairement à des températures élevées; et la partie gazeuse du spectre correspondant à la diffusion doit être éliminée. L’intégration ne devrait alors se faire que sur la partie en treillis du spectre. Mais cette séparation nécessite généralement plusieurs étapes et calculs de post-traitement supplémentaires14, qui ne sont pas couverts par le package UMD actuel.
- Exécutez le script vibr_spectrum_umd.py pour calculer la fonction d’auto-corrélation vitesse-vitesse atomique (VAC) pour chaque type atomique et effectuer sa transformée de Fourier rapide. Au niveau de la ligne de commande, tapez :
- Exécutez le script viscosity_umd.py pour analyser l’auto-corrélation du tenseur de contrainte des composants afin d’estimer la viscosité de la fusion. Au niveau de la ligne de commande, tapez :
viscosity_umd.py -f -i < Étape initiale> -s -o -l
REMARQUE: Cette fonctionnalité est exploratoire et tous les résultats doivent être pris avec prudence. Tout d’abord, vérifiez soigneusement la convergence de la viscosité par rapport à la durée de la simulation.- Dériver la viscosité du fluide à partir de l’auto-corrélation du tenseur de contrainte15 comme suit :

où V et T sont respectivement le volume et la température, κB est la constante de Boltzmann et σ ij la composante hors diagonale ij du tenseur de contrainte, exprimée en coordonnées cartésiennes. - Utilisez un ajustement plus adéquat pour obtenir une estimation plus robuste de la viscosité15,16 et éviter le bruit de la fonction d’auto-corrélation contrainte-tenseur qui pourrait résulter de la taille finie et de la durée finie des simulations. Pour la fonction d’auto-corrélation du tenseur de contrainte, utilisez la forme fonctionnelle suivante15,16 qui donne de bons résultats :
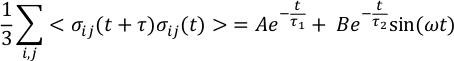
où A, B, τ1, τ2 et ω sont des paramètres d’ajustement. Après intégration, l’expression de la viscosité devient :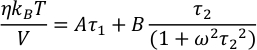
- Dériver la viscosité du fluide à partir de l’auto-corrélation du tenseur de contrainte15 comme suit :
6. Paramètres thermodynamiques issus des simulations.
- Exécutez averages.py pour extraire les valeurs moyennes et l’écart (en tant qu’écart type) pour la pression, la température, la densité et l’énergie interne des fichiers umd . Au niveau de la ligne de commande, tapez :
averages.py -f -s
avec –s 0 par défaut. - Calculez l’erreur statistique de la moyenne à l’aide de méthodes de blocage.
REMARQUE: Il existe différentes saveurs de cette méthode. Suite aux travaux d’Allen et Tildesley2, il est courant de faire la moyenne sur des séquences de blocs temporels, de plus en plus longues, et d’estimer l’écart-type par rapport à la moyenne arithmétique17. La convergence peut être atteinte à la limite de tailles de blocs multiples et suffisamment longues, lorsque l’échantillonnage n’est pas corrélé. Bien que la valeur seuil réelle de la convergence doive généralement être choisie manuellement.- Utilisez la méthode de réduction de moitié18 : en commençant par l’échantillon de données initial, à chaque étape κ, réduisez de moitié le nombre d’échantillons en faisant la moyenne sur deux échantillons consécutifs correspondants de l’étape précédente κ−1 :
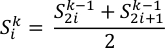
- Exécutez le script fullaverages.py pour effectuer l’analyse statistique complète, y compris l’erreur de la moyenne. Au niveau de la ligne de commande, tapez :
fullaverages.py -s -u
REMARQUE : Le script est automatisé au point de rechercher tous les fichiers .umd.dat dans le répertoire actif et d’effectuer l’analyse pour chacun d’eux. Les valeurs par défaut sont –s 0 –u 0. Pour -u 0, la sortie est minimale, et pour -u 1, la sortie est complète, avec plusieurs unités alternatives imprimées. Ce script nécessite un support graphique, car il crée une image graphique pour vérifier la convergence afin d’estimer l’erreur sur la moyenne.
- Utilisez la méthode de réduction de moitié18 : en commençant par l’échantillon de données initial, à chaque étape κ, réduisez de moitié le nombre d’échantillons en faisant la moyenne sur deux échantillons consécutifs correspondants de l’étape précédente κ−1 :
Résultats
La pyrolite est un modèle de silicate fondu multi-composants (0,5Na2O 2CaO 1,5Al2O3 4FeO 30MgO 24SiO2) qui se rapproche le mieux de la composition du silicate en vrac de la Terre – la moyenne géochimique ou notre planète entière, à l’exception de son noyau à base de fer19. La Terre primitive a été dominée par une série d’événements de fonte à grande échelle20, le dernier pourrait avoir englouti la planète entière, après sa condensation pour le disque protolunaire21. La pyrolite représente la meilleure approximation de la composition de ces océans magmatiques à l’échelle planétaire. Par conséquent, nous avons étudié de manière approfondie les propriétés physiques de la pyrolite fondue dans la plage de température de 3 000 à 000 K et la plage de pression de 0 0 052150 GPa à partir de simulations de dynamique moléculaire ab initio dans la mise en œuvre du VASP. Ces conditions thermodynamiques caractérisent entièrement les conditions océaniques magmatiques les plus extrêmes de la Terre. Notre étude est un excellent exemple d’utilisation réussie du package UMD pour l’ensemble de l’analyse approfondie des fondus22. Nous avons calculé la distribution et les longueurs moyennes des liaisons, nous avons tracé les changements dans la coordination cation-oxygène et comparé nos résultats à des études expérimentales et informatiques antérieures sur des silicates amorphes de diverses compositions. Notre analyse approfondie a permis de décomposer les nombres de coordination standard en leurs constituants de base, de décrire la présence de polyèdres de coordination exotiques dans la fonte et d’extraire les durées de vie de tous les polyèdres de coordination. Il a également souligné l’importance de l’échantillonnage dans les simulations en termes de longueur de la trajectoire et de nombre d’atomes présents dans le système modélisé. En ce qui concerne le post-traitement, l’analyse UMD est indépendante de ces facteurs, mais ils doivent être pris en compte lors de l’interprétation des résultats fournis par le package UMD. Ici, nous montrons quelques exemples de la façon dont le package UMD peut être utilisé pour extraire plusieurs caractéristiques des fondus, avec une application à la pyrolite fondue.
La fonction de distribution de paire Si-O obtenue à partir de l’écriture gofrs_umd.py montre que le rayon de la première sphère de coordination, qui est le premier minimum de la fonction g(r), se situe autour de 2,5 angströms à T = 3000 K et P = 4,6 GPa. Le maximum du g(r) se situe à 1,635 Å, c’est la meilleure approximation de la longueur de courbure. La longue queue est due à la température. En utilisant cette limite comme distance de liaison Si-O, l’analyse de spéciation montre que les unités SiO4, qui peuvent durer jusqu’à quelques picosecondes, dominent la fonte (Figure 5). Il y a une partie importante de la fonte qui montre une polymérisation partielle, reflétée par la présence de dimères comme Si2O7, et de trimères comme les unités Si3Ox. Leur durée de vie correspondante est de l’ordre de la picoseconde. Les polymères d’ordre supérieur ont tous une durée de vie considérablement plus courte.
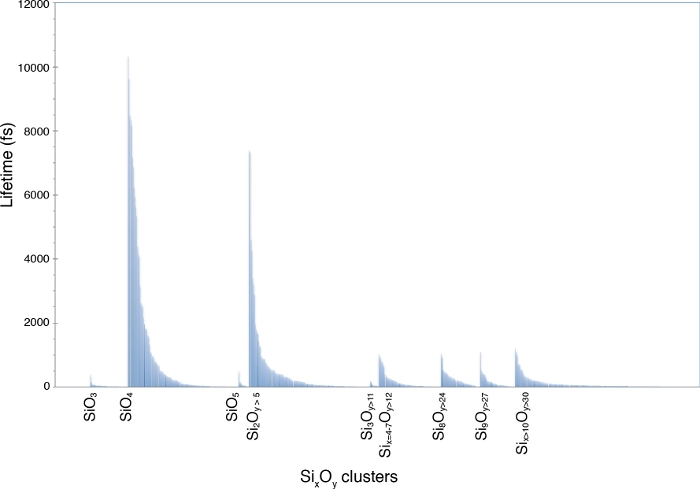
Figure 5 : Durée de vie de l’espèce chimique Si-O.
La spéciation est identifiée dans une fusion multi-composants à 4,6 GPa et 3000 K. Les étiquettes marquent les monomères SiO3, SiO4 et SiO5 et les différents polymères SixOy. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Les différentes valeurs des étapes verticales et horizontales, définies par les indicateurs –z et –v ci-dessus, donnent lieu à divers échantillonnages du TMS (figure 6). Même de grandes valeurs de z et v suffisent à définir les pentes et donc les coefficients de diffusion des différents atomes. Le gain de temps pour le post-traitement est remarquable lorsque l’on passe à de grandes valeurs de z et v. Le MSD offre un critère de validation très fort pour la qualité des simulations. Si la partie diffusion du MSD n’est pas suffisamment longue, c’est un signe que la simulation est trop courte et n’atteint pas l’état fluide au sens statistique. L’exigence minimale pour la partie diffusive du MSD dépend fortement du système. On peut exiger que tous les atomes changent de site au moins une fois dans la structure de la fonte pour qu’elle soit considérée comme un fluide10. Un excellent exemple d’applications en sciences planétaires est la fusion complexe de silicate à des pressions élevées proches ou même inférieures à leur ligne liquidus11. Les atomes de Si, les principaux cations formant des réseaux, changent de site après plus de deux douzaines de picosecondes. Des simulations plus courtes que ce seuil sous-échantillonneraient considérablement l’espace de configuration possible. Cependant, comme les anions coordonnateurs, à savoir les atomes O, se déplacent plus rapidement que les atomes Si centraux, ils peuvent compenser une partie de la lente mobilité de Si. En tant que tel, l’ensemble du système pourrait en effet couvrir un meilleur échantillonnage de l’espace de configuration que ce que l’on suppose uniquement à partir des déplacements Si.
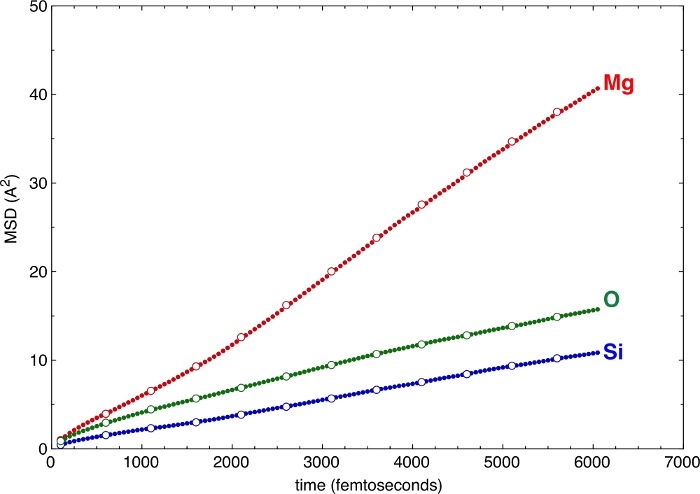
Figure 6 : Déplacements quadratiques moyens (TMS).
Les TMS sont illustrés pour quelques types atomiques d’un silicate fondu multi-composants. L’échantillonnage avec différentes étapes horizontales et verticales, z et v, donne des résultats cohérents. Cercles pleins : -z 50 –v 50. Cercles ouverts : -z 250 –v 500. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Enfin, les fonctions VAC atomiques produisent le spectre vibratoire de la fusion. La figure 7 montre le spectre dans les mêmes conditions de pression et de température que ci-dessus. Nous représentons les contributions des atomes Mg, Si et O, ainsi que la valeur totale. À fréquence nulle, il existe une valeur finie du spectre, qui correspond au caractère de diffusion de la fonte. L’extraction des propriétés thermodynamiques du spectre vibratoire doit éliminer ce caractère diffusif gazeux de zéro mais aussi prendre en compte correctement sa désintégration à des fréquences plus élevées.

Figure 7 : Spectre vibratoire de la pyrolite fondue.
La partie réelle de la transformée de Fourier de la fonction d’autocorraction vitesse-vitesse atomique donne le spectre vibratoire. Ici, le spectre est calculé pour une fusion de silicate multi-composants. Les fluides ont un caractère diffusif de type gaz non nul à fréquence nulle. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
Le package UMD a été conçu pour mieux fonctionner avec les simulations ab initio, où le nombre d’instantanés est généralement limité à des dizaines à des centaines de milliers d’instantanés, avec quelques centaines d’atomes par unité de cellule. Des simulations plus importantes sont également accessibles à condition que la machine sur laquelle le post-traitement s’exécute dispose de suffisamment de ressources de mémoire active. Le code se distingue par la variété des propriétés qu’il peut calculer et par sa licence open-source.
Les fichiers umd.dat sont adaptés aux ensembles qui préservent le nombre de particules inchangé tout au long de la simulation. Le package UMD peut lire des fichiers issus de calculs où la forme et le volume de la boîte de simulation varient. Ceux-ci couvrent les calculs les plus courants, comme NVT et NPT, où le nombre de particules, N, la température T, le volume, V et / ou la pression, P, sont maintenus constants.
Pour le moment où commencez, la fonction de distribution des paires ainsi que tous les scripts nécessaires pour estimer les distances interatomiques, comme les scripts de spéciation, ne fonctionnent que pour les cellules unitaires orthogonales, c’est-à-dire pour les cellules cubiques, tétragonales et orthorhombiques, où les angles entre les axes sont de 90 °.
Les principales lignes de développement de la version 2.0 sont la suppression de la restriction d’orthogonalité pour les distances et l’ajout de fonctionnalités supplémentaires pour les scripts de spéciation: analyser les liaisons chimiques individuelles, analyser les angles interatomiques et mettre en œuvre la deuxième sphère de coordination. Avec l’aide d’une collaboration externe, nous travaillons sur le portage du code sur un GPU pour une analyse plus rapide dans les systèmes plus grands.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Ces travaux ont été soutenus par le Conseil européen de la recherche (CER) dans le cadre du programme de recherche et d’innovation Horizon 2020 de l’Union européenne (numéro de convention de subvention 681818 IMPACT à RC), par la Direction de la physique et de la chimie extrêmes de l’Observatoire du carbone profond et par le Conseil norvégien de la recherche par le biais de son programme de financement des centres d’excellence, numéro de projet 223272. Nous reconnaissons l’accès aux supercalculateurs GENCI par le biais de la série stl2816 de subventions de calcul eDARI, au supercalculateur Irene AMD par le biais du projet PRACE RA4947 et au supercalculateur Fram par le biais de l’UNINETT Sigma2 NN9697K. FS a été soutenu par un projet Marie Skłodowska-Curie (convention de subvention ABISSE n° 750901).
matériels
| Name | Company | Catalog Number | Comments |
| getopt library | open-source | ||
| glob library | open-source | ||
| matplotlib library | open-source | ||
| numpy library | open-source | ||
| os library | open-source | ||
| Python software | The Python Software Foundation | Version 2 and 3 | open-source |
| random library | open-source | ||
| re library | open-source | ||
| scipy library | open-source | ||
| subprocess library | open-source | ||
| sys library | open-source |
Références
- Frenkel, D., Smit, B. . Understanding Molecular Simulation. From Algorithms to Applications. , (2001).
- Allen, M. P., Tildesley, D. J., Allen, T. . Computer Simulation of Liquids. , (1989).
- Zepeda-Ruiz, L. A., Stukowski, A., Oppelstrup, T., Bulatov, V. V. Probing the limits of metal plasticity with molecular-dynamics simulations. Nature Publishing Group. 550 (7677), 492-495 (2017).
- Humphrey, W., Dalke, A., Schulten, K. VMD: Visual molecular dynamics. Journal of Molecular Graphics & Modeling. 14 (1), 33-38 (1996).
- Momma, K., Izumi, F. VESTA3 for three-dimensional visualization of crystal, volumetric and morphology data. Journal of Applied Crystallography. 44 (6), 1272-1276 (2011).
- Brehm, M., Kirchner, B. TRAVIS - A free Analyzer and Visualizer for Monte Carlo and Molecular Dynamics Trajectories. Journal of Chemical Information and Modeling. 51 (8), 2007-2023 (2011).
- Stixrude, L. Visualization-based analysis of structural and dynamical properties of simulated hydrous silicate melt. Physics and Chemistry of Minerals. 37 (2), 103-117 (2009).
- Kresse, G., Hafner, J. Ab initio Molecular-Dynamics for Liquid-Metals. Physical Review B. 47 (1), 558-561 (1993).
- Gygi, F. Architecture of Qbox: A scalable first-principles molecular dynamics code. IBM Journal of Research and Development. 52 (1-2), 137-144 (2008).
- Harvey, J. P., Asimow, P. D. Current limitations of molecular dynamic simulations as probes of thermo-physical behavior of silicate melts. American Mineralogist. 100 (8-9), 1866-1882 (2015).
- Caracas, R., Hirose, K., Nomura, R., Ballmer, M. D. Melt-crystal density crossover in a deep magma ocean. Earth and Planetary Science Letters. 516, 202-211 (2019).
- Green, M. S. Markoff Random Processes and the Statistical Mechanics of Time-Dependent Phenomena. II. Irreversible Processes in Fluids. The Journal of Chemical Physics. 22 (3), 398-413 (1954).
- Kubo, R. Statistical-Mechanical Theory of Irreversible Processes. I. General Theory and Simple Applications to Magnetic and Conduction Problems. Journal of the Physical Society of Japan. 12 (6), 570-586 (1957).
- Lin, S. T., Blanco, M., Goddard, W. A. The two-phase model for calculating thermodynamic properties of liquids from molecular dynamics: Validation for the phase diagram of Lennard-Jones fluids. The Journal of Chemical Physics. 119 (22), 11792-11805 (2003).
- Meyer, E. R., Kress, J. D., Collins, L. A., Ticknor, C. Effect of correlation on viscosity and diffusion in molecular-dynamics simulations. Physical Review E. 90 (4), 1198-1212 (2014).
- Soubiran, F., Militzer, B., Driver, K. P., Zhang, S. Properties of hydrogen, helium, and silicon dioxide mixtures in giant planet interiors. Physics of Plasmas. 24 (4), 041401-041407 (2017).
- Flyvbjerg, H., Petersen, H. G. Error estimates on averages of correlated data. The Journal of Chemical Physics. 91 (1), 461-466 (1989).
- Tuckerman, M. E. . Statistical mechanics: theory and molecular simulation. , (2010).
- McDonough, W. F., Sun, S. S. The composition of the Earth. Chemical Geology. 120, 223-253 (1995).
- Elkins-Taton, L. T. Magma oceans in the inner solar system. Annual Review of Earth and Planetary Sciences. 40, 113-139 (2012).
- Lock, S. J., et al. The origin of the Moon within a terrestrial synestia. J. Geophysical Research: Planets. 123, 910-951 (2018).
- Solomatova, N. V., Caracas, R. Pressure-induced coordination changes in a pyrolitic silicate melt from ab initio molecular dynamics simulations. Journal of Geophysical Research: Solid Earth. 124, 11232-11250 (2019).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon