需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
无激光羟基基蛋白足迹,对蛋白质进行更高级的结构分析
摘要
该协议提出了一种使用内联基量程和等离子光源进行闪光氧化蛋白足迹的方法。该方法取代了危险的紫外激光器,简化和提高了蛋白质研究快速光化学氧化的可重复性。
摘要
羟基基蛋白足迹 (HRPF) 是一种新兴且很有前途的高阶结构分析技术,可提供有关蛋白质结构、蛋白质-蛋白质相互作用或蛋白质-脂蛋白相互作用变化的信息。HRPF 利用羟基基(▪OH) 不可逆转地标记蛋白质的溶剂可访问表面。执行HRPF固有的复杂性、成本和危险性大大限制了生物制药的广泛采用。这些因素包括:1) 使用复杂、危险和昂贵的激光,需要采取重大安全防范措施:和 2) 由于背景清理▪ OH而导致的 Hrpf 的不可复制性限制了比较研究。本出版物为无激光HRPF系统的运行提供了协议。这种无激光HRPF系统采用高能、高压等离子光源闪光氧化技术,采用内联基量程法。等离子光源比激光型HRPF系统更安全、更易于使用,在产生羟基基方面效率更高,并且在线基多厘米可增加研究的可重复性。无激光HRPF系统结合,解决并克服了上述激光技术的缺点和局限性。
引言
蛋白质构象和相关的高阶结构(HOS)是正常生物功能和异常行为的主要决定因素1。这同样适用于生物制药,其结构和功能活动取决于其生产和环境的各个方面。HOS的生物制药变化与不良药理学和患者免疫反应2、3的不良药物反应(ADR)有关。ADR的出现提醒生物制药行业注意蛋白质HOS在生物治疗剂的安全性和有效性中所起的关键作用,并且他们已确定需要新的和改进的HOS分析4。
羟基基蛋白足迹 (HRPF) 是跟踪蛋白质 HOS 变化的一项很有前途的技术。HRPF 涉及蛋白质外部不可逆转的标记,▪OH,然后是质谱 (MS) 分析,以确定蛋白质5、6、7的溶剂可访问表面。HRPF已成功地用于检测蛋白质HOS的缺陷及其功能8,9,特征为单克隆抗体(mAb)10,11,12,13的HOS,确定一个配体14的结合Kd,以及更多的15,16,17,18,19。产生HRPF的▪OH的常用方法是蛋白质的快速光化学氧化(FPOP),它使用高能、快速紫外线激光器从H2O2的光解中产生▪OH。在大多数情况下,FPOP使用昂贵的驱魔激光器使用有害气体(KrF),需要实质性的保障措施,以避免呼吸和眼睛受伤20。为了避免吸入危害,其他人使用频率翻倍的钛钛石榴石(Nd:YAG)激光器21,它消除了有毒气体的使用,但仍然昂贵,需要大量的操作专业知识,并要求广泛的杂散光控制,以保护用户免受眼睛损伤。
虽然可以利用HRPF获得充足的信息,但生物制药的广泛采用尚未得到满足。有限采用HRPF的两个障碍包括:1)使用危险和昂贵的激光,需要大量的安全防范措施20:和 2) 人力资源公积金的不可复制性造成的背景清理 ▪OH 限制比较研究22。为了取代激光的使用,开发了高速、高能量等离子闪光光解装置,以便于安全地执行FPOP。为了提高HRPF实验的不可生产性,实施实时激进量度。
由于背景清理了OH22,HRPF的实践受到不可生产性的限制,这归因于▪OH22的背景清理。虽然▪OH是蛋白质地形的优秀探针,但它们也与制剂中发现的许多成分发生反应,因此有必要测量可用于氧化靶蛋白的基蛋白的有效浓度。缓冲制备、过氧化氢浓度、配体特性或光解的变化可能导致控制组和实验组之间的氧化差异,这些差异在 HOS 差异研究中产生模糊性。增加实时基度量可以调整 OH 负载▪效果,从而在 HRPF 实验中增加置信力和可重复性。在FPOP中使用激进的学分法在其他地方被描述为23,24,25,并在最近出版的26号出版物中进一步详细讨论。在这里,我们描述了使用一种新的闪光光解系统和实时文量法来标记马的apo-肌红蛋白(aMb),比较了FPOP实验中肽氧化水平与使用兴奋激光时获得的水平。
研究方案
1. 安装毛细管
- 使用硅裂解石,将 250μm 内径 (ID) 二氧化硅毛细丝切成 27 英寸。检查毛细细毛的末端,获得干净、直切的切口。
- 通过燃烧聚酰胺涂层,创建两个长度约 15 毫米的窗户。从"低端"开始,使第一个光解窗口距离"低端"90毫米,第二个圆点窗口距离"低端"225毫米。
注意:一旦涂层被烧毁,毛细细毛是非常脆弱的。 - 拧开5号端口的螺母和铁杉,在铁杉圆锥端(图1A)之后插入毛细细纸的"下端"。
- 在光解模块上,通过直接拉出光解单元盖,然后取出磁性安装的金属面膜,将毛细金保持到位。
注意:在照片分析单元帽内是一面弯曲的镜子,不允许任何东西触摸镜子。 - 在圆分模块上,通过推上左边的标签打开圆分组,并将圆周细胞向右摆动。取出磁性安装的夹子,将毛细细毛放在原位。
- 将毛细细金石放入光解单元底部的凹槽通道中。将毛细细纸窗口与光解单元窗对中心。在用一只手将毛细毛保持原位时,添加磁性面膜以将毛细细毛保持到位。将照片分析盖放回原位。
- 将毛细管放入圆分细胞底部的凹槽通道中。将第二个毛细管窗口集中在多厘米细胞基基中心的小孔径上。一手握住毛细细毛,一手将两个磁性夹子放在原位,将毛细细毛放在原位。关闭多米细胞,直到它点击关闭。
- 将毛细金插入产品收集器毛细的提升上(图1B)上的螺母。将毛细延长到小瓶底部的正上方。
注意:毛细管到达小瓶底部非常重要,以确保毛细管在运行实验时被淹没在淬斗溶液中。
2. 安装注射回路
- 使用外径为 1/16" 和内径为 0.015" (381 μm) 的特氟隆管。按照方程1计算所需的体积所需的管长,使用方程1。
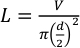
L 是毫米管的长度,V 是微升管中所需的体积,d 是毫米的管内径。对于 25 μL 的注塑量和 381μm 的内径,管长需要大约 219.3 mm。
注意:对于体积小于 20 μL 的卷,请使用内径为 0.01 的 PTFE 管。 - 使用管切割机将铁氟龙管切成必要的长度。检查管子的末端,以获得干净、直切的切口。
- 通过其中一个螺母插入新注射环的一端,并将新的铁杉放在管子的末端。将铁杉和螺母保持到位,将管插入注塑阀的端口 3,直到其底部进入阀门。牢牢地握住管子,紧紧螺母手指。使用扳手,拧紧螺母1×4进一步转动。拆下并检查装配。
注意:如果铁杉未固定到位,请进一步重新安装并拧紧 1/8 转。重复循环的另一端。 - 一旦两端有一个螺母和固定的铁杉,松散地拧一端到端口3和另一端端6(图1C)。一旦到位收紧双方手指紧,然后1/4转过去手指紧用扳手。
3. 初始化光解系统
- 按以下顺序打开光解模块:(1) 流体模块 (2) 透析模块 (3) 多西姆模块 (4) 产品收集器,最后 (5) 系统计算机并启动控制软件
注意:允许多西姆模块至少半小时从寒冷的开始热身。 - 从注射器泵上的"阀门"位置(图 1D)完全浸入 10 mL 缓冲器中。将管子从"废物"位置(图1D)和管子从注射器端口 2 端口(图 1C)放置到空容器中收集废物。
注意:使用缓冲器,您的蛋白质将被暂停。推荐的缓冲区为 10 m M 纳波4。 - 在产品收集器旋转木马上,将 1.5 mL 微中微管放置在标有 H 和 6 的位置。
- 如果使用 250μm ID 毛细,将负载流速率设置为 500 μL/min,处理流速率设置为 7.5 μL/min,废物流速将设置为 500 μL/min。
- 使用方程 2 计算使用半自动模式的闪存延迟、产品时间和浪费时间。
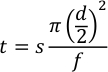
t 是分钟的时间,s 是毫米的毛细长距离,d 是毛细线的内径(毫米),f 是 μL/min 中的流速。- 对于闪光延迟,距离是从毛细细丝的"低端"到第一个窗口,这应该是大约90毫米。使用 250μm ID 毛细和 7.5μL/min 的处理流速,样品将在大约 35 秒内到达光解窗口。允许在样品到达之前在 1 Hz 时发生两次闪光,因此将闪光延迟设置为 33 秒。
- 对于产品时间,输入注射溶液的时间量将需要从注射阀流到毛细的末端。对于 27" 长 250μm ID 毛细,将产品时间设置为 4.5 分钟。
- 对于浪费时间,输入注射解决方案的总量需要收集的时间量。此时,产品收集器将从产品位置移动到废小瓶。对于 25μL 的注塑量和 7.5μL/min 的流速,将浪费时间设置为 7.8 分钟。
- 通过将 25 μL HPLC 级水注入注射端口,将注射阀设置为负载位置,将注射回路冲洗五次。
- 手动将注射阀调高至"喷射位置",以冲洗系统其余部分。选择控制软件上的 进程(出) 开始流动水。在流动时,通过在产品收集器下选择 向上 ,提高产品收集器毛细的提升,以便您可以看到毛细的末端。将水流过毛细骨,直到水滴形成。
注意:如果液滴未形成,请检查喷油器阀是否有泄漏。如果有泄漏,松开螺母,重新密封毛细管。
4. 确定实际 ▪OH 产量,以测试缓冲区的根本清除效果。
- 在控制软件中,通过选择 "过程(退出)"启动流程。在设置选项卡中,将闪光电压设置为 0 V。在多米数据部分 的手动控制 选项卡下,选择 "启动数据+ 自动零"。
- 选择产品小瓶 (H) 和废小瓶 (6) 的位置。
- 选择 就绪,手动将注射阀调低至负载位置,并将 25 μL 的 1 mM 腺苷与 100 mM H2O2 注入注射端口。注射后,手动将注射阀调至注射位置。
注意:这会自动触发系统开始流,打开等离子体源,并获取文档数据。 - 通过导航到设置选项卡并更改闪光电压来提升电压。使用 500 V、750 V、1000 V 和 1250 V 重复步骤 4.2 和 4.3。
- 选择计算选项卡,以确定每个样本的平均吸收度。首先,单击 "选择",然后手动选择峰值吸收的开始和结束。在可用空间中,写入示例的描述。对所有已获得的数据重复上述步骤。
注意:气泡可能会形成,导致文量读数激增。在选择数据以确定平均吸收度时,省略有气泡的区域。 - 在 Excel 中复制和粘贴数据,以计算每个电压的腺苷吸收量的平均变化,从而确定 OH 产量的有效▪。
- 如果使用多个样本条件(不同的缓冲器/添加剂)来正常化每个条件的有效 ▪OH 产量,则重复步骤 4.1-4.6。
5. 修改蛋白质以检测高阶结构的变化。
- 将 4 mM 腺苷与 20μM 蛋白质以一对一的比例混合,使溶液中含有 2 mM 腺苷和 10 μM 蛋白质。
- 使用 0.3 毫克/mL 催化物制作淬当溶液,分解多余的过氧化氢和 35 mM 甲硫酸,以淬灭任何剩余的基质。将 25 μL 淬斗溶液放入 200 μL 微管中,以便将同等量的淬便和标记产品混合在一起。
- 稀释 H2O2 到 200 mM 并保持冰上。
- 在设置选项卡中的控制软件上,以 0 V 启动闪光电压以确定任何背景氧化。
- 在手动控制选项卡中,选择 "启动数据+ 自动零",然后是 "过程"(Out),然后 "准备就绪",最后将注塑阀调低至负载位置。
- 在产品收集旋转木马上放置一个淬当解决方案。在系统控制软件上将产品小瓶更改为 1。
- 注射前,将腺苷和蛋白质混合物的12.5μL与12.5微L的H2O2 混合,最终浓度为1mM腺苷、5μM蛋白和100m H2O2。轻轻上下混合,快速向下旋转,并在混合后 10 秒内使用注射端口注射 25 μL。
- 将注射阀切换到注射位置,并在处理样品时等待。
- 重复采集 500 V、750 V、1000 V 和 1250 V. 以三分位执行每个电压测量。
- 计算步骤 4.5 中描述的平均吸收量。复制并粘贴所有数据到 Excel。
6. 关闭系统
- 收集完所有样品后,将注射阀设置为负载位置,然后注射 25 μL 的 HPLC 水五次,将注射器端口和样品循环冲出。
- 将注射阀调至喷射位置,用 HPLC 水冲洗系统其余部分。
- 停止流量,关闭系统控制软件,并关闭模块,从产品收集器、圆圆模块、光解模块开始,最后关闭流体模块。
7. 样品制备和液相色谱-质谱法
- 在50立方米和1立方米CaCl2的情况下,在80°C的孵化样品20分钟内分离蛋白质。将样品冷却至室温,并在蛋白质中加入1:20的特普辛。通过样品混合,在37°C下在一夜之间消化蛋白质。第二天早上,通过加热样品到95°C10分钟终止肌氨辛消化。
- 使用与 UPLC 系统相连的高分辨率 LC-MS/MS 系统检测肽。
- 先将样品加载到陷阱柱上(300 μm ID X 5 mm 100 + 孔径,5μm 颗粒大小),然后以 5.0 μL/mL 的速度洗涤 3 分钟,水中含有 2% 溶剂 B(乙二甲酸和 0.1% 的甲基酸)。
- 以300 nL/min的流速分离C18纳米胶质(0.75毫米 x 150毫米、2μm颗粒大小、100+孔径)上的肽,溶剂A(含0.1%的丙酸水)和溶剂B之间的梯度。肽弹性的梯度包括22分钟内从2%到35%B的线性增加,在5分钟内从坡度增加到95%B,并保持3分钟清洗柱,然后在3分钟内返回到2%B,并保持9分钟以重新平衡柱。
- 以正离子模式获取数据。将喷雾电压设置为 2400 V,将离子转移管的温度设置为 300 °C。
- 获得 250-2000 m/z 的完整 MS 扫描,然后对前八个最丰富的肽离子进行后续八次数据依赖 MS/MS 扫描。使用碰撞引起的分离在35%的正常化能量分裂肽。
- 使用可用的蛋白质分析软件,针对包含蛋白质序列和相关蛋白质酶的 FASTA 文件,识别从 MS/MS 分析中检测到的所有未修改肽。
- 使用HRPF数据处理软件搜索和量化修改后的肽。通过将修改肽的累计色谱峰值面积除以使用等式 3 修改和未修改的肽的总色谱峰值区域来计算每个已识别肽的氧化程度。
P = [I(单氧化) X 1 + I (加倍氧化) X 2 + I(三氧化) X 3 + ... /[I (单氧化) + I(双重氧化) + I(三氧化) ...
其中P表示每个肽分子的平均氧化事件数,我表示未氧化肽(无氧化)和具有 n氧化事件的肽的峰值区域。
8. 对于差异研究,在第二个条件下重复步骤 5-7。
注意:为了确认可重复性,建议除了针对每个条件的技术三位一体之外,还推荐两种生物复制品。
结果
高压等离子体源与实时量表相结合,能够更好地控制▪OH 产量,更准确地观察高阶蛋白质结构的变化。添加腺苷可以有效地实时激进量度。氧化后,腺苷在265纳米(图2A)时失去紫外线吸收性。腺苷吸收的变化与HRPF可用的激进分子的浓度直接相关,从而提供了一种手段,在缓冲器、辅料和配体等激进拾荒者在场的情况下有效监测激进浓度的变化(图2B...
讨论
在任何HRPF实验中,有几个关键步骤可以确保蛋白质的正确标记。首先,选择适当的流速和源闪速,以确保样品的每个玻色子都照射一次。这确保了蛋白质暴露在OH新形成 的▪的单一波卢斯中。一旦蛋白质被氧化,高阶的蛋白质结构就会改变。要确信原生蛋白质结构被探测到,每个蛋白质分子必须在一瞬间被修改。多西姆可用于测试原生蛋白质结构是否被探测。当羟基基的浓度增加氧化程?...
披露声明
E.E..C、J.S.S.和S.R.W.披露了GenNext技术公司的重大财务利益,该公司是一家寻求将高阶蛋白质结构分析技术商业化的早期公司。提供的代表数据已由S.K..M审查,他没有财务利益冲突。
致谢
这项工作由国家普通医学科学研究所(R43GM125420和R44GM125420)资助。
材料
| Name | Company | Catalog Number | Comments |
| 15 mL Conical Centrifuge Tubes | Fisher Scientific | 14-959-53A | any brand is sufficient |
| 50 µL SGE Gastight Syringes | Fisher Scientific | SG-00723 | |
| Acclaim PepMap 100 C18 nanocolumn (0.75 mm X 150 mm, 2 µm) | Thermo Scientific | ||
| Acetonitrile with 0.1% Formic Acid (v/v), LC/MS Grade | Fisher Scientific | LS120-500 | |
| Apomyoglobin | Sigma-Aldrich | ||
| Catalase | Sigma-Aldrich | C9322 | |
| Centrifuge | Eppendorf | 022625501 | |
| Delicate Task Wipers | Fisher Scientific | 06-666A | |
| Hydrogen Peroxide | Fisher Scientific | H325-100 | any 30% hydrogen peroxide is sufficient |
| Methionine amide | Chem-Impex | 03109 | |
| Microcentrifuge | Thermo Scientific | 75002436 | |
| Orbitrap Fusion Lumos Tribrid Mass Spectrometer | Thermo Scientific | Orbitrap Fusion Lumos Tribrid Mass Spectrometer | other high resolution instruments (e.g. Q exactive Orbitrap or Orbitrap Fusion) can be used |
| Pierce Trypsin Protease, MS Grade | Thermo Scientific | 90058 | |
| Polymicro Cleaving Stone, 1" x 1" x 1/32” | Molex | 1068680064 | any capillary tubing cutter is sufficient |
| UPLC | Thermo Scientific | ||
| Water with 0.1% Formic Acid (v/v), LC/MS Grade | Fisher Scientific | LS118-500 | |
| Water, LC/MS Grade | Fisher Scientific | W6-4 |
参考文献
- Nagarkar, R. P., Murphy, B. M., Yu, X., Manning, M. C., Al-Azzam, W. A. Characterization of protein higher order structure using vibrational circular dichroism spectroscopy. Current Pharmaceutical Biotechnology. 14 (2), 199-208 (2013).
- Giezen, T. J., Schneider, C. K. Safety assessment of biosimilars in Europe: a regulatory perspective. Generics and Biosimilars Initiative Journal. , 1-8 (2014).
- Giezen, T. J., Mantel-Teeuwisse, A. K., Strauss, S. Safety-related regulatory actions for biologicals approved in the United States and the Europena Union. Journal of the American Medical Society. 300 (16), 1887-1896 (2008).
- Gabrielson, J. P., Weiss, W. F. Technical decision-making with higher order structure data: starting a new dialogue. Journal of Pharmaceutical Sciences. 104 (1), 1240-1245 (2015).
- Brenowitz, M., Erie, D. A., Chance, M. R. Catching RNA polymerase in the act of binding: intermediates in transcription illuminated by synchrotron footprinting. Proceedings of the National Academy of Sciences U S A. 102 (13), 4659-4660 (2005).
- Guan, J. Q., Takamoto, K., Almo, S. C., Reisler, E., Chance, M. R. Structure and dynamics of the actin filament. Biochemistry. 44 (9), 3166-3175 (2005).
- Hambly, D. M., Gross, M. L. Laser flash photochemical oxidataion to locate heme binding and conformataional changes in myoglobin. International Journal of Mass Spectrometry. 259 (2007), 124-129 (2007).
- Li, K. S., Shi, L., Gross, M. L. Mass Spectrometry-Based Fast Photochemical Oxidation of Proteins (FPOP) for Higher Order Structure Characterization. Accounts of Chemical Research. 51 (3), 736-744 (2018).
- Watson, C., Sharp, J. S. Conformational Analysis of Therapeutic Proteins by Hydroxyl Radical Protein Footprinting. American Association of Pharmaceutical Scientists Journal. 14 (2), 206-217 (2012).
- Deperalta, G., et al. Structural analysis of a therapeutic monoclonal antibody dimer by hydroxyl radical footprinting. mAbs. 5 (1), 86-101 (2013).
- Jones, L. M., et al. Complementary MS methods assist conformational characterization of antibodies with altered S-S bonding networks. Journal of American Society of Mass Spectrometry. 24 (6), 835-845 (2013).
- Storek, K. M., et al. Monoclonal antibody targeting the β-barrel assembly machine of Escherichia coli is bactericidal. Proceedings of the National Academy of Sciences. , (2018).
- Vij, R., et al. A targeted boost-and-sort immunization strategy using Escherichia coli BamA identifies rare growth inhibitory antibodies. Scientific Reports. 8 (1), 7136 (2018).
- Liu, X. R., Zhang, M. M., Rempel, D. L., Gross, M. L. A Single Approach Reveals the Composite Conformational Changes, Order of Binding, and Affinities for Calcium Binding to Calmodulin. Analytical Chemistry. 91 (9), 5508-5512 (2019).
- Lu, Y., et al. Fast Photochemical Oxidation of Proteins Maps the Topology of Intrinsic Membrane Proteins: Light-Harvesting Complex 2 in a Nanodisc. Analytical Chemistry. 88 (17), 8827-8834 (2016).
- Marty, M., Zhang, H., Cui, W., Gross, M., Sligar, S. Interpretation and Deconvolution of Nanodisc Native Mass Spectra. Journal of American Society of Mass Spectrometry. 25, (2013).
- Johnson, D. T., Di Stefano, L. H., Jones, L. M. Fast photochemical oxidation of proteins(FPOP): A powerful mass spectrometry based structural proteomics tool. Journal of Biological Chemistry. , (2019).
- Chea, E. E., Jones, L. M. Analyzing the structure of macromolecules in their native cellular environment using hydroxyl radical footprinting. Analyst. 143 (4), 798-807 (2018).
- Aprahamian, M. L., Chea, E. E., Jones, L. M., Lindert, S. Rosetta Protein Structure Prediction from Hydroxyl Radical Protein Footprinting Mass Spectrometry Data. Analytical chemistry. 90 (12), 7721-7729 (2018).
- Linde. . Linde Specialty Gases of North America. , (2009).
- Aye, T. T., Low, T. Y., Sze, S. K. Nanosecond laser-induced photochemical oxidation method for protein surface mapping with mass spectrometry. Analytical Chemistry. 77 (18), 5814-5822 (2005).
- Niu, B., Zhang, H., Giblin, D., Rempel, D. L., Gross, M. L. Dosimetry determines the initial OH radical concentration in fast photochemical oxidation of proteins (FPOP). Journal of American Society of Mass Spectrometry. 26 (5), 843-846 (2015).
- Misra, S. K., Orlando, R., Weinberger, S. R., Sharp, J. S. Compensated Hydroxyl Radical Protein Footprinting Measures Buffer and Excipient Effects on Conformation and Aggregation in an Adalimumab Biosimilar. American Association of Pharmaceutical Scientists Journal. 21 (5), 87 (2019).
- Olson, L. J., Misra, S. K., Ishihara, M., Battaile, K. P., Grant, O. C., Sood, A., Woods, R. J., Kim, J. P., Tiemeyer, M., Ren, G., Sharp, J. S., Dahms, N. M. Allosteric regulation of lysosomal enzyme recognition by the cation-independent mannose 6-phosphate receptor. Communications Biology. 3 (1), 498 (2020).
- Sharp, J. S., Misra, S. K., Persoff, J. J., Egan, R. W., Weinberger, S. R. Real Time Normalization of Fast Photochemical Oxidation of Proteins Experiments by Inline Adenine Radical Dosimetry. Analytical Chemistry. 90 (21), 12625-12630 (2018).
- Misra, S. K., Sharp, J. S. Enabling Real-Time Compensation in Fast Photochemical Oxidations of Proteins for the Determination of Protein Topography Changes. Journal of Visualized Experiments. 163, (2020).
- Hambly, D. M., Gross, M. L. Laser flash photolysis of hydrogen peroxide to oxidize protein solvent-accessible residues on the microsecond timescale. Journal of American Society of Mass Spectrometry. 16 (12), 2057-2063 (2005).
- Liu, X. R., Zhang, M. M., Gross, M. L. Mass Spectrometry-Based Protein Footprinting for Higher-Order Structure Analysis: Fundamentals and Applications. Chemical Reviews. 120 (10), 4355 (2020).
- Jones, L. M., Sperry, B. J., Carroll, A. J., Gross, M. L. Fast photochemical oxidation of proteins for epitope mapping. Analytical chemistry. 83 (20), 7657-7661 (2011).
- Li, J., et al. Mapping the Energetic Epitope of an Antibody/Interleukin-23 Interaction with Hydrogen/Deuterium Exchange, Fast Photochemical Oxidation of Proteins Mass Spectrometry, and Alanine Shave Mutagenesis. Analytical chemistry. 89 (4), 2250-2258 (2017).
- Liu, X. R., Zhang, M. M., Rempel, D. L., Gross, M. L. A Single Approach Reveals the Composite Conformational Changes, Order of Binding, and Affinities for Calcium Binding to Calmodulin. Analytical Chemistry. 91 (9), 5508-5512 (2019).
- Kiselar, J. G., Janmey, P. A., Almo, S. C., Chance, M. R. Structural analysis of gelsolin using synchrotron protein footprinting. Molecular and Cellular Proteomics. 2 (10), 1120-1132 (2003).
- Chea, E. E., Deredge, D. J., Jones, L. M. Insights on the Conformational Ensemble of Cyt C Reveal a Compact State during Peroxidase Activity. Biophysical Journal. 118 (1), 128-137 (2020).
- Poor, T. A., et al. Probing the paramyxovirus fusion (F) protein-refolding event from pre- to postfusion by oxidative footprinting. Proceedings of the National Academy of Sciences U S A. 111 (25), 2596-2605 (2014).
- Roush, A. E., Riaz, M., Misra, S. K., Weinberger, S. R., Sharp, J. S. Intrinsic Buffer Hydroxyl Radical Dosimetry Using Tris(hydroxymethyl)aminomethane. Journal of American Society of Mass Spectrometry. 31 (2), 169-172 (2020).
- Everett, E. A., Falick, A. M., Reich, N. O. Identification of a critical cysteine in EcoRI DNA methyltransferase by mass spectrometry. Journal of Biological Chemistry. 265 (29), 17713-17719 (1990).
- Sanderson, R. J., Mosbaugh, D. W. Identification of specific carboxyl groups on uracil-DNA glycosylase inhibitor protein that are required for activity. Journal of Biological Chemistry. 271 (46), 29170-29181 (1996).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。