A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
טביעת רגל של חלבון הידרוקסיל רדיקלי ללא לייזר לביצוע ניתוח מבני מסדר גבוה יותר של חלבונים
In This Article
Summary
פרוטוקול זה מציג שיטה לשימוש בדוסימטריה רדיקלית מוטבעת ומקור אור פלזמה לביצוע טביעת רגל של חלבון חמצון פלאש. שיטה זו מחליפה את לייזר UV מסוכן כדי לפשט ולשפר את הרבייה של חמצון פוטוכימי מהיר של מחקרי חלבון.
Abstract
טביעת רגל של חלבון רדיקלי הידרוקסיל (HRPF) היא טכניקת ניתוח מבנית מתפתחת ומבטיחה מסדר גבוה יותר המספקת מידע על שינויים במבנה החלבון, אינטראקציות בין חלבונים לחלבון או אינטראקציות בין חלבונים. HRPF משתמש ברדיקלים הידרוקסיל(▪OH) כדי לתייג באופן בלתי הפיך משטח נגיש ממס של חלבון. המורכבות, העלות והאופי המסוכן הטמון בביצוע HRPF הגבילו באופן משמעותי את האימוץ הנרחב בביופארמה. גורמים אלה כוללים: 1) שימוש בלייזרים מורכבים, מסוכנים ויקרים הדורשים אמצעי זהירות משמעותיים; ו -2) חוסר ההתאמה של HRPF שנגרם על ידי ניקוי רקע של ▪OH המגבילים מחקרים השוואתיים. פרסום זה מספק פרוטוקול להפעלת מערכת HRPF נטולת לייזר. מערכת HRPF נטולת לייזר זו משתמשת בטכנולוגיית חמצון הבזק מקור פלזמה באנרגיה גבוהה ובלחץ גבוה עם דוזימטריה רדיקלית בשורה. מקור אור הפלזמה בטוח יותר, קל יותר לשימוש ויעיל יותר ביצירת רדיקלים הידרוקסיל מאשר מערכות HRPF מבוססות לייזר, והדוסימטר הרדיקלי בשורה מגביר את הרבייה של מחקרים. בשילוב, מערכת HRPF נטולת הלייזר מטפלת ומתגברת על החסרונות והמגבלות שהוזכרו בטכניקות מבוססות לייזר.
Introduction
קונפורמציה של חלבונים ומבנה מסדר גבוה יותר (HOS) הם הקובעים העיקריים של תפקוד ביולוגי תקין והתנהגות חריגה1. כנ"ל לגבי biopharmaceuticals, אשר מבנה ופעילות תפקודית תלויה בהיבטים שונים של הייצור והסביבה שלהם. שינוי Biopharmaceutical ב HOS קושרו תופעות לוואי (ADR) המיוחסים פרמקולוגיה לא רצויה תגובה אימונולוגית המטופל2,3. המראה של ADRs התריע על התעשייה הביופארמה לתפקיד הקריטי כי חלבון HOS ממלא הבטיחות והיעילות של biotherapeutics, והם הקימו את הצורך ניתוח HOS חדש ומשופר4.
טביעת רגל של חלבון הידרוקסיל רדיקלי (HRPF) היא טכניקה מבטיחה למעקב אחר השינוי בחלבון HOS. HRPF כרוך תיוג בלתי הפיך של החיצוני של חלבון עם ▪OH ואחריו ניתוח ספקטרומטריית מסה (MS) כדי לזהות את פני השטח נגיש ממס של החלבון5,6,7. HRPF שימש בהצלחה לגילוי פגמים בחלבון HOS ותפקודו8,9, לאפיין את HOS של נוגדנים חד שבטיים (mAb)10,11,12,13, לקבוע את Kd מחייב של ליגנד14, ועוד הרבה יותר15,16,17,18,19. שיטה נפוצה ליצירת ▪OH עבור HRPF היא חמצון פוטוכימי מהיר של חלבונים (FPOP), אשר משתמשת בלייזר UV מהיר ואנרגיה גבוהה כדי לייצר ▪OH מפוטוליזה של H2O2. על פי רוב, FPOP משתמש לייזרים excimer יקר העסקת גז מסוכן (KrF) הדורש אמצעי הגנה משמעותיים כדי למנוע פגיעה בדרכי הנשימה והעין20. כדי למנוע סכנות שאיפה, אחרים השתמשו בתדר מרובע ניאודימיום yttrium אלומיניום גארנט (Nd:YAG) לייזרים21, אשר מבטל את השימוש בגז רעיל אבל הם עדיין יקרים, דורשים מומחיות מבצעית משמעותית, ודורשים בקרות אור תועה נרחב כדי להגן על המשתמשים מפני פגיעה בעין.
למרות שניתן להשיג מידע רב באמצעות HRPF, אימוץ נרחב בביופארמה לא נענה. שני חסמים לאימוץ מוגבל HRPF כוללים: 1) שימוש בלייזרים מסוכנים ויקרים הדורשים אמצעי זהירות משמעותיים20; ו -2) חוסר ההתאמה של HRPF שנגרם על ידי ניקוי רקע של ▪OH המגבילים מחקרים השוואתיים22. כדי להחליף את השימוש בלייזר, במהירות גבוהה, אנרגיה גבוהה פלזמה פלאש פוטוליזה היחידה פותחה כדי לבצע בבטחה FPOP באופן קל. כדי לשפר את חוסר ההתאמה של ניסויי HRPF, דוסימטריה רדיקלית בזמן אמת מיושמת.
הנוהג של HRPF הוגבל על ידי חוסר תאימות המיוחס ניקוי רקע של ▪OH22. בעוד ▪OH הם בדיקות מצוינות של טופוגרפיה חלבון, הם גם מגיבים עם מרכיבים רבים שנמצאו בהכנות, מה שהופך אותו הכרחי כדי למדוד את הריכוז היעיל של רדיקלים זמינים כדי לחמצן חלבון היעד. וריאציות בהכנת חיץ, ריכוז מי חמצן, תכונות ליגנד, או פוטוליזה יכול לגרום הבדלי חמצון בין קבוצות בקרה וניסוי שיוצרים עמימות במחקרים דיפרנציאליים HOS. התוספת של דוזימטריה רדיקלית בזמן אמת מאפשרת התאמה של ההשפעה ▪עומס OH ולכן מגבירה את הביטחון והשחזור במהלך ניסוי HRPF. השימוש בדוזימטריה רדיקלית ב- FPOP תואר במקום אחר23,24,25, והוא נדון בפירוט בפרסום האחרון26. כאן, אנו מתארים את השימוש במערכת פוטוליזה פלאש רומן ודוזימטריה בזמן אמת כדי לתייג אפו-מיוגלובין סוסים (aMb), השוואת רמות של חמצון פפטיד בניסוי FPOP לזה של המתקבל בעת שימוש בלייזר excimer.
Protocol
1. התקנת הצינור נימי
- בעזרת אבן סחף סיליקה, cleave 250 מיקרומטר קוטר פנימי (ID) נימי סיליקה ל 27 אינץ '. בדוק את הקצוות נימי לחתוך נקי, ישר.
- צור שני חלונות בערך 15 מ"מ אורך על ידי שריפת ציפוי פולימיד. החל מ"הקצה התחתון" הופכים את חלון הפוצליזה הראשון ל-90 מ"מ הרחק מ"הקצה התחתון" וחלון הדוזימטר השני במרחק של 225 מ"מ מה"קצה התחתון".
הערה: ברגע שהציפיה נשרפת נימי הוא שביר מאוד. - פתחו את האגוז והפרול ביציאה 5 והכניסו את "הקצה התחתון" של הנימי ממש מעבר לקצה החרוטי של הפרול(איור 1A).
- על מודול photolysis, להסיר את כובע תא photolysis על ידי משיכת אותו ישר החוצה ולאחר מכן להסיר את מסכת מתכת רכוב מגנטי אשר יחזיק את נימי במקום.
התראה: בתוך מכסה התא הפוקוליזה יש מראה מעוגלת, אל תאפשר לשום דבר לגעת במראה. - במודול dosimeter, פתח את תא הדוסימטר על-ידי דחיפת הלשונית משמאל והנפת תא הדוזימטר פתוח לימין. הסר את הקליפים המותקנים מגנטית אשר יחזיקו את נימי במקום.
- מניחים את נימי לתוך הערוץ מחורצת בבסיס תא הפוטופליזה. מרכז את החלון הנימי באמצעות חלון תא הפוטיליזה. תוך החזקת נימי בעמדה ביד אחת, מוסיפים את המסכה המגנטית כדי להחזיק את נימי במקום. הנח את מכסה הפוטוליזה בחזרה בעמדה.
- מניחים את נימי לתוך הערוץ מחורצת בבסיס תא dosimeter. מרכז את החלון הנימי השני על הצמצם הקטן במרכז בסיס התא הדוזימטר. תוך החזקת נימי בעמדה ביד אחת, מניחים את שני הקליפים המגנטיים בעמדה להחזיק את נימי במקום. סגור את תא הדוזימטר עד שילחץ על סגור.
- הכנס את נימי דרך האגוז knurled על גבי המעלית נימי של אספן המוצר(איור 1B). מרחיבים את נימי רק מעל החלק התחתון של הבקבוקון.
הערה: חשוב עבור נימי להגיע לתחתית הבקבוקון כדי להבטיח כי נימי הוא שקוע בתמיסת להרוות בעת הפעלת ניסוי.
2. התקנת לולאת הזרקה
- השתמש צינורות טפלון עם קוטר חיצוני 1/16" וקוטר פנימי של 0.015" (381 מיקרומטר). בצע את המשוואה 1 כדי לחשב את אורך הצינור הדרוש לאמצעי האחסון הרצוי באמצעות משוואה 1.
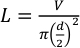
כאשר L הוא אורך הצינור במילימטרים, V הוא הנפח הרצוי במיקרוליטרים ו- d הוא הקוטר הפנימי של הצינור במילימטרים. עבור נפח הזרקה של 25 μL וקוטר פנימי של 381 מיקרומטר, אורך הצינור צריך להיות בערך 219.3 מ"מ.
הערה: עבור כמויות של פחות מ- 20 μL, השתמש בצינורות PTFE בקוטר פנימי של 0.01 אינץ'. - חותכים את צינורות טפלון לאורך הדרוש באמצעות חותך צינור. בדוק את הקצוות של הצינור לחתוך נקי, ישר.
- הכנס קצה אחד של לולאת ההזרקה החדשה דרך אחד האגוזים ומניחים פרול חדש על קצה הצינור. החזיקו את הפרול והאגוז במקום והכניסו את הצינור ליציאה 3 של שסתום ההזרקה עד שהוא יורד החוצה בשסתום. החזיקו את הצינור בחוזקה והדקו את אצבע האגוזים בחוזקה. באמצעות מפתח ברגים, להדק את האגוז 1/4 לפנות עוד יותר. הסר ובדוק את ההרכבה.
הערה: אם הפרול אינו קבוע במיקום, התקן מחדש והדק 1/8 פנה עוד יותר. חזור על הפעולה עם הקצה השני של הלולאה. - ברגע שלשני הקצוות יש אגוז ופרול קבוע, בורג רופף בקצה אחד ליציאה 3 והקצה השני ליציאה 6(איור 1C). פעם בעמדה להדק את שני הצדדים לאצבע חזק אז 1/4 להפוך את האצבע בעבר חזק עם מפתח ברגים.
3. אתחל את מערכת הפוטוליזה
- הפעל את מודולי הפוטוליזה בסדר הבא: (1) מודול Fluidics (2) מודול פוטוליזה (3) מודול דוסימטריה (4) אספן מוצר, ולבסוף (5) מחשב המערכת ולהפעיל את תוכנת הבקרה
הערה: אפשר למודול Dosimeter לפחות חצי שעה להתחמם מהתחלה קרה. - צינורות שקועים לחלוטין מעמדת "שסתום" במשאבת המזרק(איור 1D)ל-10 מ"ל של חיץ. הנח את הצינור מעמדת ה"פסולת"(איור 1D)ואת הצינור מיציאה 2 ביציאת המזרק(איור 1C)למכל ריק לאיסוף פסולת.
הערה: השתמש במאגר שהחלבון שלך יושעה בו. המאגר המומלץ הוא 10 mM NaPO4. - על קרוסלה אספן המוצר, מקום 1.5 צינורות microcentrifuge מ"ל במיקום מסומן H ו 6.
- אם אתה משתמש נימי מזהה μm 250, להגדיר את קצב זרימת העומס ל 500 μL / min, עיבוד קצב הזרימה ל 7.5 μL / min, ואת קצב זרימת הפסולת ל 500 μL / min.
- חשב את השהיית ההבזק, זמן המוצר וזמן הבזבוז כדי להשתמש במצב חצי אוטומטי באמצעות משוואה 2.
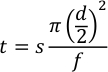
איפה זה הזמן בדקות, s הוא המרחק נימי במילימטרים, d הוא הקוטר הפנימי של נימי במילימטרים, ו f הוא קצב הזרימה ב μL / min.- עבור עיכוב פלאש, המרחק הוא מן "הקצה התחתון" של נימי לחלון הראשון, אשר צריך להיות בערך 90 מ"מ. באמצעות נימי מזהה μm 250 וקצב זרימת עיבוד של 7.5 μL / min המדגם יגיע לחלון הפוקוליזה בעוד כ -35 שניות. אפשר לשני הבזקים ב- 1 הרץ להתרחש לפני שהדגימות מגיעות, לכן הגדר את השהיית הפלאש ל- 33 שניות.
- עבור זמן המוצר, הזן את כמות הזמן פתרון מוזרק ייקח לזרום משסתום ההזרקה לסוף נימי. עבור נימי מזהה 27" באורך 250 מיקרומטר, הגדר את זמן המוצר ל- 4.5 דקות.
- עבור זמן הפסולת, הזן את משך הזמן הכולל של פתרון מוזרק ייקח לאסוף. בשלב זה, אספן המוצר יעבור מעמדת המוצר אל בקבוקון הפסולת. עבור נפח הזרקה של 25 μL וקצב זרימה של 7.5 μL / min, להגדיר את זמן הפסולת ל 7.8 דקות.
- יש לשטוף את לולאת ההזרקה חמש פעמים על ידי הזרקת 25 μL של מים ברמה של HPLC ליציאת ההזרקה כאשר שסתום ההזרקה מוגדר למיקום העומס.
- באופן ידני להפוך את שסתום ההזרקה עד 'להזריק מיקום' כדי לשטוף את שאר המערכת. בחר תהליך (החוצה) בתוכנת הבקרה כדי להתחיל לזרום מים. תוך כדי תנועה, להעלות את המוצר אספן נימי להרים על ידי בחירה למעלה תחת אספן המוצר, כך שאתה יכול לראות את סוף נימי. זרימת מים דרך נימי עד טיפה צורות.
הערה: אם טיפה אינה נוצרת, בדוק את שסתום המזרק עבור דליפות. אם יש דליפה, לשחרר את האגוז, reseat נימי, ו retighten.
4. לקבוע בפועל ▪OH תשואה כדי לבדוק השפעות ניקוי קיצוניות מן המאגר.
- בתוכנת הבקרה, הפעל את הזרימה על-ידי בחירה באפשרות תהליך (החוצה). בכרטיסייה 'הגדרות', הגדר את מתח ההבזק ל- 0 V. תחת הכרטיסיה שליטה ידנית במקטע נתוני dosimeter, בחר התחל נתונים + מצב אוטומטי.
- בחר את המיקום עבור בקבוקון המוצר (H) ובקבוקון פסולת (6).
- בחר מוכן, באופן ידני להפוך את שסתום ההזרקה עד למצב העומס, ולהזריק 25 μL של 1 mM אדנין עם 100 מ"מ H2O2 ליציאת ההזרקה. לאחר הזרקת, באופן ידני להפוך את שסתום ההזרקה עד למצב ההזרקה.
הערה: פעולה זו מפעילה באופן אוטומטי את המערכת כדי להתחיל את הזרימה, להפעיל את מקור הפלזמה, ולהשיג את נתוני הדוזימטריה. - הגבירו את המתח על-ידי ניווט לכרטיסיה 'הגדרות' ושינוי מתח ההבזק. חזור על שלבים 4.2 ו- 4.3 באמצעות 500 V, 750 V, 1000 V ו- 1250 V. בצע את הקריאה של ספיגת אדנין בכל מתח בטריפלים.
- בחר בכרטיסיה חישובים כדי לקבוע את הספיגה הממוצעת של כל דגימה. תחילה, לחץ על בחרולאחר מכן בחר באופן ידני את ההתחלה והסוף של ספיגת השיא. בשטח הפנוי, כתוב בתיאור של הדוגמה. חזור על הפעולה עבור כל הנתונים שנרכשו.
הערה: בועות עלולות להיווצר ולגרום לעלייה חדה בקריאת דוזימטר. בעת בחירת נתונים כדי לקבוע ספיגה ממוצעת, השמט אזורים עם בועות. - העתק והדבק נתונים ב- Excel כדי לחשב את השינוי הממוצע בספיגת אדנין עבור כל מתח, ובכך לקבוע את התשואה היעילה ▪OH.
- חזור על שלבים 4.1-4.6 אם נעשה שימוש בתנאי דגימה מרובים (מאגר/תוספים שונים) כדי לנרמל את ▪ התפוקה היעילה שלOH עבור כל תנאי.
5. שינוי של חלבון כדי לזהות שינויים במבנה בסדר גבוה יותר.
- יש לערבב 4 מ"מ אדנין עם 20 מיקרומטר חלבון ביחס של אחד לאחד כדי ליצור פתרון המכיל 2 מ"מ אדנין ו-10 מיקרומטר חלבון.
- הפוך את הפתרון להרוות באמצעות 0.3 מ"ג / מ"ל קטלאז כדי לשבור עודף מי חמצן ו 35 mM מתונין amide להרוות את כל הרדיקלים הנותרים. Aliquot 25 μL של פתרון מרווה לתוך microtube 200 μL כך נפח שווה של מרווה ואת המוצר שכותרתו מעורבב.
- לדלל H2O2 עד 200 מ"מ ולשמור על קרח.
- בכרטיסיה תוכנת בקרה בכרטיסייה הגדרות, הפעל את מתח הפלאש ב- 0 V כדי לקבוע כל חמצון רקע.
- בכרטיסיה בקרה ידנית, בחר התחל נתונים + מצב אוטומטי, ואחריו תהליך (החוצה), ולאחר מכן מוכן, ולבסוף להפוך את שסתום ההזרקה למטה למיקום העומס.
- מניחים פתרון מרווה בעמדה 1 על קרוסלת איסוף המוצר. בתוכנת בקרת המערכת שנה את בקבוקון המוצר ל- 1.
- מיד לפני ההזרקה, מערבבים 12.5 μL של תערובת אדנין וחלבון עם 12.5 μL של H2O2 כדי להפוך את הריכוז הסופי של 1 מ"מ אדנין, 5 μM חלבון, ו 100 מ"מ H2O2. בעדינות פיפטה למעלה ולמטה כדי לערבב, במהירות לסובב למטה, ולהזריק 25 μL באמצעות יציאת ההזרקה בתוך 10 שניות של ערבוב.
- לעבור שסתום ההזרקה למצב להזריק ולחכות בזמן המדגם מעובד.
- חזור על הרכישה עם 500 V, 750 V, 1000 V ו- 1250 V. בצע כל מדידת מתח משולשת.
- חשב את הספיגה הממוצעת כמתואר בשלב 4.5. העתק והדבק את כל הנתונים ל- Excel.
6. כיבוי המערכת
- לאחר כל הדגימות נאספו, לשטוף את יציאת המזרק ואת לולאת מדגם על ידי הגדרת שסתום ההזרקה עד למצב העומס ולהזריק 25 μL של מים HPLC חמש פעמים.
- להפוך את שסתום ההזרקה עד עמדת ההזרקה כדי לשטוף את שאר המערכת עם מים HPLC.
- לעצור את הזרימה, לסגור את תוכנת בקרת המערכת, ולכבות את המודולים החל אספן המוצר, מודול dosimeter, מודול פוטוליזה, ואז סוף סוף מודול fluidics.
7. הכנה לדוגמה וספקטרומטריית כרומטוגרפיה-מסה נוזלית
- Denature את החלבון על ידי דגירה דגימות ב 80 מעלות צלזיוס במשך 20 דקות בנוכחות 50 mM Tris ו 1 mM CaCl2. מצננים דגימות לטמפרטורת החדר ומוסיפים 1:20 טריפסין לחלבון. לעכל את החלבון לילה ב 37 מעלות צלזיוס עם ערבוב מדגם. למחרת בבוקר, לסיים את העיכול טריפסין על ידי חימום דגימות ל 95 °C (60 °F) במשך 10 דקות.
- זהה פפטידים באמצעות מערכת LC-MS/MS ברזולוציה גבוהה המחוברת למערכת UPLC.
- לטעון את המדגם הראשון על עמודת מלכודת (300 מיקרומטר מזהה X 5 מ"מ 100 Å גודל נקבובית, 5 מיקרומטר גודל חלקיקים) ולשטוף ב 5.0 μL / מ"ל במשך 3 דקות עם מים המכילים 2% ממס B (אצטוניטריל ו 0.1% חומצה פורמית).
- להפריד את הפפטידים על C18 nanocolumn (0.75 מ"מ x 150 מ"מ, 2 מיקרומטר גודל חלקיקים, 100 Å גודל נקבובית) בקצב זרימה של 300 nL / min עם שיפוע בין ממס A (מים המכילים 0.1% חומצה פורמית) ו ממס B. השיפוע עבור elution פפטיד מורכב עלייה ליניארית מ 2 ל 35% B על פני 22 דקות, השתולל ל 95% B מעל 5 דקות והחזיק במשך 3 דקות כדי לשטוף את הטור, ולאחר מכן חזר 2% B מעל 3 דקות והחזיק במשך 9 דקות כדי לאכוף מחדש את הטור.
- השג את הנתונים במצב יון חיובי. הגדר את מתח הריסוס ל 2400 V, ואת הטמפרטורה של צינור העברת היונים ל 300 מעלות צלזיוס.
- השג את סריקות MS המלאות מ- 250-2000 m/z ולאחר מכן שמונה סריקות MS/MS תלויות נתונים עוקבות בשמונת יוני הפפטיד הנפוצים ביותר. השתמש דיסוציאציה הנגרמת על ידי התנגשות ב 35% אנרגיה מנורמלת כדי לפצל את הפפטידים.
- זהה את כל הפפטידים שלא שונו שזוהו מניתוח MS/MS באמצעות תוכנת ניתוח חלבונים זמינה כנגד קובץ FASTA הדרוש המכיל את רצף החלבון ואת האנזים הפרוטאוליטי הרלוונטי.
- חפש וכומת פפטידים ששונו באמצעות תוכנת עיבוד נתונים של HRPF. מידת החמצון עבור כל פפטיד מזוהה מחושבת על ידי חלוקת אזור השיא הכרומטוגרפי המסוכם של פפטיד שונה על ידי אזור השיא הכרומטוגרפי הכולל של פפטיד זה שונה ולא השתנה באמצעות משוואה 3.
P = [אני(מחומצן באופן חמצנוני) X 1 + אני (מחומצן כפליים) X 2 + אני(מחומצן משולשת) X 3 + .../[ I לאמחומצן + אני(מחומצן כפליים)+ אני(מחומצן משולשת)...]
כאשר P מציין את המספר הממוצע של אירועי חמצון לכל מולקולת פפטיד, ואני מייצג את אזור השיא של פפטיד unoxidized (Iunoxidized) ואת פפטיד עם n אירועי חמצון.
8. למחקר דיפרנציאלי, לחזור על שלבים 5-7 על המצב השני.
הערה: כדי לאשר את הרבייה, מומלץ לבצע שני משכפלים ביולוגיים בנוסף לשלשים טכניים עבור כל תנאי.
תוצאות
מקור הפלזמה בלחץ גבוה בשילוב עם דוזימטריה בזמן אמת מאפשר שליטה טובה יותר של ▪OH תשואה להתבונן שינויים במבנה חלבון בסדר גבוה יותר בצורה מדויקת יותר. התוספת של אדנין מאפשרת דוזימטר רדיקלי יעיל בזמן אמת. עם החמצון, אדנין מאבד ספיגת UV ב 265 ננומטר (איור 2A). השינוי בספיגת אדנ?...
Discussion
ישנם מספר שלבים קריטיים כדי להבטיח תיוג נכון של חלבונים במהלך כל ניסוי HRPF. ראשית, קצב זרימה מתאים וקצב הבזק מקור נבחרים כדי לוודא שכל בולוס של המדגם מוקרן פעם אחת. זה מבטיח כי החלבון נחשף בולוס יחיד של ▪החדש שנוצר OH. לאחר חמצון החלבון, ניתן לשנות את מבנה החלבון מסדר גבוה יותר. כדי להיות...
Disclosures
E.E.C., J.S.S., ו S.R.W. לחשוף עניין פיננסי משמעותי GenNext טכנולוגיות, Inc., חברה בשלב מוקדם המבקשים למסחר טכנולוגיות לניתוח מבנה חלבון מסדר גבוה יותר. הנתונים הייצוגיים שסופקו נבדקו על ידי S.K.M., שאין לו ניגוד עניינים פיננסי.
Acknowledgements
עבודה זו מומנה על ידי המכון הלאומי למדעי הרפואה הכללית (R43GM125420 ו R44GM125420).
Materials
| Name | Company | Catalog Number | Comments |
| 15 mL Conical Centrifuge Tubes | Fisher Scientific | 14-959-53A | any brand is sufficient |
| 50 µL SGE Gastight Syringes | Fisher Scientific | SG-00723 | |
| Acclaim PepMap 100 C18 nanocolumn (0.75 mm X 150 mm, 2 µm) | Thermo Scientific | ||
| Acetonitrile with 0.1% Formic Acid (v/v), LC/MS Grade | Fisher Scientific | LS120-500 | |
| Apomyoglobin | Sigma-Aldrich | ||
| Catalase | Sigma-Aldrich | C9322 | |
| Centrifuge | Eppendorf | 022625501 | |
| Delicate Task Wipers | Fisher Scientific | 06-666A | |
| Hydrogen Peroxide | Fisher Scientific | H325-100 | any 30% hydrogen peroxide is sufficient |
| Methionine amide | Chem-Impex | 03109 | |
| Microcentrifuge | Thermo Scientific | 75002436 | |
| Orbitrap Fusion Lumos Tribrid Mass Spectrometer | Thermo Scientific | Orbitrap Fusion Lumos Tribrid Mass Spectrometer | other high resolution instruments (e.g. Q exactive Orbitrap or Orbitrap Fusion) can be used |
| Pierce Trypsin Protease, MS Grade | Thermo Scientific | 90058 | |
| Polymicro Cleaving Stone, 1" x 1" x 1/32” | Molex | 1068680064 | any capillary tubing cutter is sufficient |
| UPLC | Thermo Scientific | ||
| Water with 0.1% Formic Acid (v/v), LC/MS Grade | Fisher Scientific | LS118-500 | |
| Water, LC/MS Grade | Fisher Scientific | W6-4 |
References
- Nagarkar, R. P., Murphy, B. M., Yu, X., Manning, M. C., Al-Azzam, W. A. Characterization of protein higher order structure using vibrational circular dichroism spectroscopy. Current Pharmaceutical Biotechnology. 14 (2), 199-208 (2013).
- Giezen, T. J., Schneider, C. K. Safety assessment of biosimilars in Europe: a regulatory perspective. Generics and Biosimilars Initiative Journal. , 1-8 (2014).
- Giezen, T. J., Mantel-Teeuwisse, A. K., Strauss, S. Safety-related regulatory actions for biologicals approved in the United States and the Europena Union. Journal of the American Medical Society. 300 (16), 1887-1896 (2008).
- Gabrielson, J. P., Weiss, W. F. Technical decision-making with higher order structure data: starting a new dialogue. Journal of Pharmaceutical Sciences. 104 (1), 1240-1245 (2015).
- Brenowitz, M., Erie, D. A., Chance, M. R. Catching RNA polymerase in the act of binding: intermediates in transcription illuminated by synchrotron footprinting. Proceedings of the National Academy of Sciences U S A. 102 (13), 4659-4660 (2005).
- Guan, J. Q., Takamoto, K., Almo, S. C., Reisler, E., Chance, M. R. Structure and dynamics of the actin filament. Biochemistry. 44 (9), 3166-3175 (2005).
- Hambly, D. M., Gross, M. L. Laser flash photochemical oxidataion to locate heme binding and conformataional changes in myoglobin. International Journal of Mass Spectrometry. 259 (2007), 124-129 (2007).
- Li, K. S., Shi, L., Gross, M. L. Mass Spectrometry-Based Fast Photochemical Oxidation of Proteins (FPOP) for Higher Order Structure Characterization. Accounts of Chemical Research. 51 (3), 736-744 (2018).
- Watson, C., Sharp, J. S. Conformational Analysis of Therapeutic Proteins by Hydroxyl Radical Protein Footprinting. American Association of Pharmaceutical Scientists Journal. 14 (2), 206-217 (2012).
- Deperalta, G., et al. Structural analysis of a therapeutic monoclonal antibody dimer by hydroxyl radical footprinting. mAbs. 5 (1), 86-101 (2013).
- Jones, L. M., et al. Complementary MS methods assist conformational characterization of antibodies with altered S-S bonding networks. Journal of American Society of Mass Spectrometry. 24 (6), 835-845 (2013).
- Storek, K. M., et al. Monoclonal antibody targeting the β-barrel assembly machine of Escherichia coli is bactericidal. Proceedings of the National Academy of Sciences. , (2018).
- Vij, R., et al. A targeted boost-and-sort immunization strategy using Escherichia coli BamA identifies rare growth inhibitory antibodies. Scientific Reports. 8 (1), 7136 (2018).
- Liu, X. R., Zhang, M. M., Rempel, D. L., Gross, M. L. A Single Approach Reveals the Composite Conformational Changes, Order of Binding, and Affinities for Calcium Binding to Calmodulin. Analytical Chemistry. 91 (9), 5508-5512 (2019).
- Lu, Y., et al. Fast Photochemical Oxidation of Proteins Maps the Topology of Intrinsic Membrane Proteins: Light-Harvesting Complex 2 in a Nanodisc. Analytical Chemistry. 88 (17), 8827-8834 (2016).
- Marty, M., Zhang, H., Cui, W., Gross, M., Sligar, S. Interpretation and Deconvolution of Nanodisc Native Mass Spectra. Journal of American Society of Mass Spectrometry. 25, (2013).
- Johnson, D. T., Di Stefano, L. H., Jones, L. M. Fast photochemical oxidation of proteins(FPOP): A powerful mass spectrometry based structural proteomics tool. Journal of Biological Chemistry. , (2019).
- Chea, E. E., Jones, L. M. Analyzing the structure of macromolecules in their native cellular environment using hydroxyl radical footprinting. Analyst. 143 (4), 798-807 (2018).
- Aprahamian, M. L., Chea, E. E., Jones, L. M., Lindert, S. Rosetta Protein Structure Prediction from Hydroxyl Radical Protein Footprinting Mass Spectrometry Data. Analytical chemistry. 90 (12), 7721-7729 (2018).
- Linde. . Linde Specialty Gases of North America. , (2009).
- Aye, T. T., Low, T. Y., Sze, S. K. Nanosecond laser-induced photochemical oxidation method for protein surface mapping with mass spectrometry. Analytical Chemistry. 77 (18), 5814-5822 (2005).
- Niu, B., Zhang, H., Giblin, D., Rempel, D. L., Gross, M. L. Dosimetry determines the initial OH radical concentration in fast photochemical oxidation of proteins (FPOP). Journal of American Society of Mass Spectrometry. 26 (5), 843-846 (2015).
- Misra, S. K., Orlando, R., Weinberger, S. R., Sharp, J. S. Compensated Hydroxyl Radical Protein Footprinting Measures Buffer and Excipient Effects on Conformation and Aggregation in an Adalimumab Biosimilar. American Association of Pharmaceutical Scientists Journal. 21 (5), 87 (2019).
- Olson, L. J., Misra, S. K., Ishihara, M., Battaile, K. P., Grant, O. C., Sood, A., Woods, R. J., Kim, J. P., Tiemeyer, M., Ren, G., Sharp, J. S., Dahms, N. M. Allosteric regulation of lysosomal enzyme recognition by the cation-independent mannose 6-phosphate receptor. Communications Biology. 3 (1), 498 (2020).
- Sharp, J. S., Misra, S. K., Persoff, J. J., Egan, R. W., Weinberger, S. R. Real Time Normalization of Fast Photochemical Oxidation of Proteins Experiments by Inline Adenine Radical Dosimetry. Analytical Chemistry. 90 (21), 12625-12630 (2018).
- Misra, S. K., Sharp, J. S. Enabling Real-Time Compensation in Fast Photochemical Oxidations of Proteins for the Determination of Protein Topography Changes. Journal of Visualized Experiments. 163, (2020).
- Hambly, D. M., Gross, M. L. Laser flash photolysis of hydrogen peroxide to oxidize protein solvent-accessible residues on the microsecond timescale. Journal of American Society of Mass Spectrometry. 16 (12), 2057-2063 (2005).
- Liu, X. R., Zhang, M. M., Gross, M. L. Mass Spectrometry-Based Protein Footprinting for Higher-Order Structure Analysis: Fundamentals and Applications. Chemical Reviews. 120 (10), 4355 (2020).
- Jones, L. M., Sperry, B. J., Carroll, A. J., Gross, M. L. Fast photochemical oxidation of proteins for epitope mapping. Analytical chemistry. 83 (20), 7657-7661 (2011).
- Li, J., et al. Mapping the Energetic Epitope of an Antibody/Interleukin-23 Interaction with Hydrogen/Deuterium Exchange, Fast Photochemical Oxidation of Proteins Mass Spectrometry, and Alanine Shave Mutagenesis. Analytical chemistry. 89 (4), 2250-2258 (2017).
- Liu, X. R., Zhang, M. M., Rempel, D. L., Gross, M. L. A Single Approach Reveals the Composite Conformational Changes, Order of Binding, and Affinities for Calcium Binding to Calmodulin. Analytical Chemistry. 91 (9), 5508-5512 (2019).
- Kiselar, J. G., Janmey, P. A., Almo, S. C., Chance, M. R. Structural analysis of gelsolin using synchrotron protein footprinting. Molecular and Cellular Proteomics. 2 (10), 1120-1132 (2003).
- Chea, E. E., Deredge, D. J., Jones, L. M. Insights on the Conformational Ensemble of Cyt C Reveal a Compact State during Peroxidase Activity. Biophysical Journal. 118 (1), 128-137 (2020).
- Poor, T. A., et al. Probing the paramyxovirus fusion (F) protein-refolding event from pre- to postfusion by oxidative footprinting. Proceedings of the National Academy of Sciences U S A. 111 (25), 2596-2605 (2014).
- Roush, A. E., Riaz, M., Misra, S. K., Weinberger, S. R., Sharp, J. S. Intrinsic Buffer Hydroxyl Radical Dosimetry Using Tris(hydroxymethyl)aminomethane. Journal of American Society of Mass Spectrometry. 31 (2), 169-172 (2020).
- Everett, E. A., Falick, A. M., Reich, N. O. Identification of a critical cysteine in EcoRI DNA methyltransferase by mass spectrometry. Journal of Biological Chemistry. 265 (29), 17713-17719 (1990).
- Sanderson, R. J., Mosbaugh, D. W. Identification of specific carboxyl groups on uracil-DNA glycosylase inhibitor protein that are required for activity. Journal of Biological Chemistry. 271 (46), 29170-29181 (1996).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved