Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Huella De Proteína De Radicales Hidroxilo Libre De Láser Para Realizar Análisis Estructurales De Orden Superior De Proteínas
En este artículo
Resumen
Este protocolo presenta un método para utilizar dosimetría radical en línea y una fuente de luz plasmática para realizar la huella de proteínas de oxidación flash. Este método reemplaza el peligroso láser UV para simplificar y mejorar la reproducibilidad de la oxidación fotoquímica rápida de los estudios de proteínas.
Resumen
La huella de proteína radical hidroxilo (HRPF) es una técnica de análisis estructural de orden superior emergente y prometedora que proporciona información sobre los cambios en la estructura de la proteína, las interacciones proteína-proteína o las interacciones proteína-ligando. HRPF utiliza radicales hidroxilo(▪OH) para etiquetar irreversiblemente la superficie accesible del disolvente de una proteína. La complejidad inherente, el costo y la naturaleza peligrosa de la realización de HRPF han limitado sustancialmente la adopción de base amplia en la biofarmacia. Estos factores incluyen: 1) el uso de láseres complicados, peligrosos y costosos que exigen precauciones de seguridad sustanciales; y 2) la irreproducibilidad de HRPF causada por el barrido del fondo de ▪OH que limitan estudios comparativos. Esta publicación proporciona un protocolo para el funcionamiento de un sistema HRPF libre de láser. Este sistema HRPF libre de láser utiliza una tecnología de oxidación flash de fuente de luz de plasma de alta energía y alta presión con dosimetría radical en línea. La fuente de luz plasmática es más segura, más fácil de usar y más eficiente en la generación de radicales hidroxilo que los sistemas HRPF basados en láser, y el dosímetro radical en línea aumenta la reproducibilidad de los estudios. Combinado, el sistema HRPF sin láser aborda y supera las deficiencias y limitaciones mencionadas de las técnicas basadas en láser.
Introducción
La conformación de las proteínas y la estructura de orden superior (HOS) asociada son los principales determinantes de la función biológica adecuada y el comportamiento aberrante1. Lo mismo se aplica a los productos biofarmacéuticos, cuya estructura y actividad funcional depende de diversos aspectos de su producción y entorno. El cambio biofarmacéutico en el SCA se ha relacionado con reacciones adversas a fármacos (ADR) atribuidas a la farmacología indeseable y a la respuesta inmunológica del paciente2,3. La aparición de los ADR ha alertado a la industria biofarmacéutica sobre el papel crítico que juega la proteína HOS en la seguridad y eficacia de la bioterapéutica, y han establecido la necesidad de nuevos y mejorados análisis de HOS4.
La huella de proteína radical hidroxilo (HRPF) es una técnica prometedora para rastrear el cambio en la proteína HOS. Hrpf implica el etiquetado irreversible del exterior de una proteína con ▪OH seguido de espectrometría de masas (MS) análisis para identificar la superficie accesible solvente de la proteína5,6,7. Hrpf se ha utilizado con éxito para detectar defectos en la proteína HOS y su función8,9,caracterizar el HOS de anticuerpos monoclonales (mAb)10,11,12,13,determinar la unión Kd de un ligando14,y mucho más15,16,17,18,19. Un método común para generar el ▪OH para HRPF es la oxidación fotoquímica rápida de proteínas (FPOP), que emplea láseres UV rápidos y de alta energía para producir ▪OH a partir de la fotólisis deH2O2. En su mayor parte, FPOP utiliza costosos láseres excímeros que emplean gas peligroso (KrF) que exige protecciones sustanciales para evitar lesiones respiratorias y oculares20. Para evitar los peligros de inhalación, otros han utilizado láseres de granate de aluminio de neodimio cuadruplicadodefrecuencia (Nd: YAG), que elimina el uso de gases tóxicos pero siguen siendo costosos, requieren una experiencia operativa significativa y exigen amplios controles de luz extraviada para proteger a los usuarios de lesiones oculares.
Aunque se puede obtener amplia información utilizando HRPF, no se ha cumplido una amplia adopción en la biofarmacéutica. Dos barreras para la adopción limitada de HRPF incluyen: 1) el uso de láseres peligrosos y costosos que exigen precauciones de seguridad sustanciales20; y 2) la irreproducibilidad de la FCR causada por el barrido de fondo de ▪OH que limitan los estudios comparativos22. Para suplantar el uso del láser, se desarrolló una unidad de fotólisis de flash de plasma de alta velocidad y alta energía para realizar FPOP de forma segura de una manera fácil. Para mejorar la irreproducibilidad de los experimentos de HRPF, se implementa la dosimetría radical en tiempo real.
La práctica de HRPF ha sido limitada por la irreproducibilidad atribuida al barrido del fondo de ▪OH22. Mientras que ▪OH son sondas excelentes de la topografía de la proteína, también reaccionan con muchos componentes encontrados en preparaciones, haciéndole necesario medir la concentración eficaz de radical disponible para oxidar una proteína de la blanco. Las variaciones en la preparación del tampón, la concentración de peróxido de hidrógeno, las propiedades del ligando o la fotólisis pueden resultar en diferencias de oxidación entre los grupos de control y experimentales que crean ambigüedad en los estudios diferenciales de HOS. La adición de dosimetría radical en tiempo real permite el ajuste del efecto ▪carga de OH y, por lo tanto, aumenta la confianza y la reproducibilidad durante un experimento de HRPF. El uso de la dosimetría radical en FPOP se ha descrito en otra parte23,24,25,y se discute más detalladamente en una publicación reciente26. Aquí, se describe el uso de un nuevo sistema de fotólisis flash y dosimetría en tiempo real para etiquetar la apo-mioglobina equina (aMb), comparando los niveles de oxidación de péptidos en un experimento FPOP a la de obtenido cuando se utiliza un láser excímero.
Protocolo
1. Instalación del tubo capilar
- Usando una piedra de hendidura de sílice, escinda capilar de sílice de 250 μm de diámetro interior (ID) a 27 pulgadas. Revise los extremos capilares para un corte limpio y recto.
- Cree dos ventanas de aproximadamente 15 mm de longitud quemando el recubrimiento de poliimida. A partir del "extremo inferior" hacer la primera ventana de fotólisis 90 mm de distancia del "extremo inferior" y la segunda ventana del dosímetro 225 mm de distancia del "extremo inferior".
NOTA: Una vez que el recubrimiento se quema, el capilar es muy frágil. - Desenrosque la tuerca y la férula en el puerto 5 e inserte el "extremo inferior" del capilar justo más allá del extremo cónico de la férula (Figura 1A).
- En el módulo de fotólisis, retire la tapa de la celda de fotólisis tirando de ella hacia fuera y luego retire la máscara de metal montada magnéticamente que mantendrá el capilar en su lugar.
PRECAUCIÓN: Dentro de la tapa de la celda de fotólisis es un espejo curvo, no permita que nada toque el espejo. - En el módulo del dosímetro, abra la celda del dosímetro presionando la pestaña de la izquierda y gire la celda del dosímetro hacia la derecha. Retire los clips montados magnéticamente que mantendrán el capilar en su lugar.
- Coloque el capilar en el canal ranurado en la base de la célula de fotólisis. Centre la ventana capilar con la ventana de celda de fotólisis. Mientras mantiene el capilar en su posición con una mano, agregue la máscara magnética para mantener el capilar en su lugar. Vuelva a colocar la tapa de fotólisis en su posición.
- Coloque el capilar en el canal ranurado en la base de la célula del dosímetro. Centre la segunda ventana capilar en la pequeña abertura en el centro de la base de la celda del dosímetro. Mientras mantiene el capilar en posición con una mano, coloque los dos clips magnéticos en posición para mantener el capilar en su lugar. Cierre la celda del dosímetro hasta que haga clic en cerrar.
- Inserte el capilar a través de la tuerca atada sobre la elevación capilar del colector de producto (Figura 1B). Extienda el capilar justo por encima de la parte inferior del vial.
NOTA: Es importante que el capilar llegue a la parte inferior del vial para asegurarse de que el capilar esté sumergido en la solución de temple mientras se ejecuta un experimento.
2. Instalación de un bucle de inyección
- Utilice tubos de teflón con un diámetro exterior de 1/16" y un diámetro interior de 0,015" (381 μm). Siga la ecuación 1 para calcular la longitud de la tubería necesaria para el volumen deseado utilizando la ecuación 1.
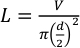
Donde L es la longitud de la tubería en milímetros, V es el volumen deseado en microlitros y d es el diámetro interior del tubo en milímetros. Para un volumen de inyección de 25 μL y un diámetro interior de 381 μm, la longitud del tubo debe ser de aproximadamente 219,3 mm.
NOTA: Para volúmenes inferiores a 20 μL, utilice tubos de PTFE con un diámetro interior de 0,01". - Corte el tubo de teflón a la longitud necesaria utilizando un cortador de tubos. Revise los extremos del tubo para un corte limpio y recto.
- Inserte un extremo del nuevo bucle de inyección a través de una de las tuercas y coloque una nueva férula en el extremo del tubo. Mantenga la férula y la tuerca en su lugar e inserte el tubo en el puerto 3 de la válvula de inyección hasta que suba fondo en la válvula. Sostenga el tubo firmemente y apriete el dedo de la tuerca apretado. Usando una llave inglesa, apriete la tuerca 1/4 de vuelta más. Retire e inspeccione el conjunto.
NOTA: Si la férula no está fija en su posición, vuelva a instalar y apriete 1/8 de vuelta más. Repita con el otro extremo del bucle. - Una vez que ambos extremos tienen una tuerca y una férula fija, atornille libremente un extremo al puerto 3 y el otro extremo al puerto 6 (Figura 1C). Una vez en posición apriete ambos lados para apretar el dedo y luego 1/4 gire más allá del dedo apretado con una llave inglesa.
3. Inicializar el sistema de fotólisis
- Encienda los módulos de fotólisis en el siguiente orden: (1) Módulo de Fluidics (2) Módulo de fotólisis (3) Módulo de dosimetría (4) Colector de productos y, por último,(5) el ordenador del sistema y ejecute el software de control
NOTA: Permita que el módulo dosímetro al menos media hora para calentarse desde un arranque en frío. - Sumergir completamente el tubo desde la posición "Válvula" en la bomba de la jeringa (Figura 1D) en 10 ml de tampón. Coloque el tubo desde la posición "Residuos"(Figura 1D)y el tubo desde el puerto 2 en el puerto de la jeringa(Figura 1C)a un recipiente vacío para recoger los residuos.
NOTA: Utilice el tampón en el que se suspenderá su proteína. Un buffer recomendado es 10 mM NaPO4. - En el carrusel del colector de productos, coloque tubos de microcentrífuga de 1,5 mL en la posición marcada H y 6.
- Si utiliza capilar de 250 μm id., establezca el caudal de carga en 500 μL/min, el caudal de procesamiento en 7,5 μL/min y el caudal de residuos en 500 μL/min.
- Calcule el retardo de flash, el tiempo del producto y la pérdida de tiempo para utilizar el modo semiautomático utilizando la ecuación 2.
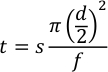
Donde t es el tiempo en minutos, s es la distancia capilar en milímetros, d es el diámetro interior del capilar en milímetros, y f es la velocidad de flujo en μL/min.- Para el retardo de flash, la distancia es desde el "extremo inferior" del capilar hasta la primera ventana, que debe ser de aproximadamente 90 mm. Utilizando un capilar de 250 μm id. y un caudal de procesamiento de 7,5 μL/min, la muestra llegará a la ventana de fotólisis en unos 35 segundos. Permita que se produzcan dos destellos a 1 Hz antes de que lleguen las muestras, así que establezca el retardo del flash en 33 segundos.
- Para el tiempo del producto, ingrese la cantidad de tiempo que la solución inyectada tardará en fluir desde la válvula de inyección hasta el extremo del capilar. Para un capilar de 27" de largo y 250 μm id., establezca el tiempo del producto en 4,5 minutos.
- Para el tiempo de pérdida, ingrese la cantidad de tiempo que el volumen total de solución inyectada tardará en ser recogido. En este momento, el colector de producto se moverá de la posición del producto al vial de residuos. Para un volumen de inyección de 25 μL y un caudal de 7,5 μL/min, establezca el tiempo de pérdida en 7,8 minutos.
- Enjuague el bucle de inyección cinco veces inyectando 25 μL de agua de grado HPLC en el puerto de inyección con la válvula de inyección configurada en la posición de carga.
- Gire manualmente la válvula de inyección hacia arriba hasta la "posición de inyección" para enjuagar el resto del sistema. Seleccione Proceso (salida) en el software de control para comenzar a fluir agua. Mientras fluye, eleve la elevación capilar del colector de producto seleccionando Arriba debajo del colector de producto para que pueda ver el final del capilar. Fluya agua a través del capilar hasta que se forme una gota.
NOTA: Si no se forma una gotita, revise la válvula del inyector para detectar fugas. Si hay una fuga, afloje la tuerca, vuelva a secar el capilar y vuelva a apretar.
4. Determinar el rendimiento real de ▪OH para probar los efectos de barrido radical del búfer.
- En el Software de control, inicie el flujo seleccionando Proceso (salida). En la pestaña de configuración, establezca el voltaje del flash en 0 V. En la ficha Control manual de la sección datos del dosímetro, seleccione Iniciar datos + AutoZero.
- Seleccione la posición para el vial del producto (H) y el vial de residuos (6).
- Seleccione Listo, gire manualmente la válvula de inyección hacia abajo a la posición de carga e inyecte 25 μL de Adenina de 1 mM con 100 mM H2O2 en el puerto de inyección. Una vez inyectado, gire manualmente la válvula de inyección hasta la posición de inyección.
NOTA: Esto activa automáticamente el sistema para iniciar el flujo, encender la fuente de plasma y adquirir los datos de dosimetría. - Aumente el voltaje navegando a la pestaña de configuración y cambiando el voltaje del flash. Repita los pasos 4.2 y 4.3 utilizando 500 V, 750 V, 1000 V y 1250 V. Realice la lectura de la absorbancia de adenina en cada voltaje en triplicados.
- Seleccione la pestaña de cálculos para determinar la absorbancia promedio de cada muestra. En primer lugar, haga clic en Seleccionary, a continuación, seleccione manualmente el principio y el final de la absorbancia de pico. En el espacio disponible, escriba una descripción del ejemplo. Repita el tiempo para todos los datos adquiridos.
NOTA: Las burbujas pueden formarse causando un pico en la lectura del dosímetro. Al seleccionar datos para determinar la absorbancia media, omita las áreas con burbujas. - Copie y pegue datos en Excel para calcular el cambio promedio en la absorbancia de adenina para cada voltaje, determinando así el rendimiento efectivo de ▪OH.
- Repita los pasos 4.1-4.6 si se utilizan múltiples condiciones de muestra (diferentes tampón/aditivos) para normalizar el rendimiento efectivo de ▪OH para cada condición.
5. Modificación de la proteína para detectar cambios en la estructura de orden superior.
- Mezcle 4 mM de adenina con 20 μM de proteína en una proporción uno a uno para hacer una solución que contenga 2 mM de adenina y 10 μM de proteína.
- Haga la solución de temple usando 0.3 mg/mL catalasa para descomponer el exceso de peróxido de hidrógeno y 35 mM de amida de metionina para apagar los radicales restantes. Alícuota 25 μL de solución de temple en un microtubo de 200 μL para que se mezcle un volumen igual de temple y el producto etiquetado.
- Diluir H2O2 a 200 mM y mantener en hielo.
- En el Software de control en la pestaña de configuración, inicie el voltaje del flash a 0 V para determinar cualquier oxidación de fondo.
- En la pestaña de control manual, seleccione Datos de inicio + Cero automático, seguido de Proceso (salida), luego Listoy, finalmente, baje la válvula de inyección a la posición de carga.
- Coloque una solución de temple en la posición 1 en el carrusel de recogida del producto. En el software de control del sistema cambie el vial del producto a 1.
- Inmediatamente antes de la inyección, mezcle 12,5 μL de la mezcla de adenina y proteína con 12,5 μL deH2O2 para hacer una concentración final de 1 mM de adenina, 5 μM de proteína y 100 mM deH2O2. Pipetee suavemente hacia arriba y hacia abajo para mezclar, gire rápidamente hacia abajo e inyecte 25 μL usando el puerto de inyección dentro de los 10 segundos de la mezcla.
- Cambie la válvula de inyección a la posición de inyección y espere mientras se procesa la muestra.
- Repita la adquisición con 500 V, 750 V, 1000 V y 1250 V. Realice cada medición de voltaje por triplicado.
- Calcule la absorbancia media como se describe en el paso 4.5. Copie y pegue todos los datos en Excel.
6. Apague el sistema
- Después de que se hayan recogido todas las muestras, enjuague el puerto de la jeringa y el bucle de la muestra fijando la válvula de inyección hasta la posición de carga e inyecte 25 μL de agua de HPLC cinco veces.
- Gire la válvula de inyección hasta la posición de inyección para enjuagar el resto del sistema con agua hplc.
- Detenga el flujo, cierre el software de control del sistema y apague los módulos comenzando con el colector de producto, el módulo dosímetro, el módulo de fotólisis y, finalmente, el módulo de fluídica.
7. Preparación de muestras y cromatografía líquida-espectrometría de masas
- Desnaturalizar la proteína incubando muestras a 80 °C durante 20 min en presencia de 50 mM Tris y 1 mM CaCl2. Enfríe las muestras a temperatura ambiente y agregue 1:20 tripsina a la proteína. Digiera la proteína durante la noche a 37 °C con la mezcla de muestras. A la mañana siguiente, termine la digestión de la tripsina calentando las muestras a 95 °C durante 10 minutos.
- Detecte péptidos utilizando un sistema LC-MS/MS de alta resolución conectado con un sistema UPLC.
- Cargue la muestra primero en la columna de trampa (300 μm ID X 5 mm 100 Å tamaño de poro, tamaño de partícula de 5 μm) y lave a 5,0 μL/mL durante 3 min con agua que contenga 2% de disolvente B (acetonitrilo y ácido fórmico al 0,1%).
- Separe los péptidos en una nanocolumna C18 (0,75 mm x 150 mm, tamaño de partícula de 2 μm, tamaño de poro de 100 Å) a una velocidad de flujo de 300 nL/min con un gradiente entre el disolvente A (agua que contiene ácido fórmico al 0,1%) y el disolvente B. El gradiente para la elución del péptido consiste en un aumento linear a partir del 2 al 35% B durante 22 minutos, rampado al 95% B durante 5 minutos y sostenido durante 3 minutos para lavar la columna, y después vuelto al 2% B durante 3 minutos y sostenido por 9 minutos para reequilibrar la columna.
- Adquirir los datos en modo de iones positivos. Ajuste el voltaje de pulverización a 2400 V y la temperatura del tubo de transferencia de iones a 300 °C.
- Adquiera las exploraciones completas del MS a partir de 250-2000 m/z seguidas por ocho exploraciones datos-dependientes subsecuentes del MS/MS en los ocho iones más abundantes del péptido. Utilice la disociación inducida por colisión al 35% de energía normalizada para fragmentar los péptidos.
- Identifique todos los péptidos no modificados detectados en el análisis de MS/MS utilizando un software de análisis de proteínas disponible contra el archivo FASTA necesario que contiene la secuencia de proteínas y la enzima proteolítica relevante.
- Busque y cuantifique péptidos modificados utilizando un software de procesamiento de datos HRPF. El grado de oxidación para cada péptido identificado se calcula dividiendo el área de pico cromatográfico sumado de un péptido modificado por el área de pico cromatográfico total de ese péptido modificado y sin modificar utilizando la ecuación 3.
P = [I(oxidado por separado) X 1 + I (doblemente oxidado) X 2 + I(oxidado triplicado) X 3 + .../[Iunoxidizado + I(oxidado por separado) + I(doblemente oxidado) + I(triplicado oxidado) ...]
donde P denota el número promedio de eventos de oxidación por molécula peptídica, e I representa el área pico del péptido no oxidado(Iunoxidized)y el péptido con n eventos de oxidación.
8. Para un estudio diferencial, repita los pasos 5-7 en la segunda condición.
NOTA: Para confirmar la reproducibilidad, se recomiendan dos réplicas biológicas además de triplicados técnicos para cada condición.
Resultados
La fuente de plasma de alta presión junto con la dosimetría en tiempo real permite un mejor control de ▪rendimiento de OH para observar los cambios en la estructura de proteínas de orden superior con mayor precisión. La adición de adenina permite un dosímetro radical eficaz en tiempo real. Tras la oxidación, la adenina pierde absorbancia UV a 265 nm(Figura 2A). El cambio en la absorbancia de la adenina está directamente relacionado con la concentración de radicales disp...
Discusión
Hay varios pasos críticos para asegurar el etiquetado adecuado de las proteínas durante cualquier experimento hrpf. En primer lugar, se selecciona un caudal adecuado y un tipo de inflamación de la fuente para asegurarse de que cada bolo de la muestra se irradia una vez. Esto asegura que la proteína se expone a un solo bolo de ▪oh recién formado. Una vez que una proteína se oxida, la estructura de la proteína de orden superior puede ser alterada. Para estar seguro de que la estructura de la proteína n...
Divulgaciones
E.E.C., J.S.S., y S.R.W. revelan un interés financiero significativo en GenNext Technologies, Inc., una compañía en etapa temprana que busca comercializar tecnologías para el análisis de la estructura de proteínas de orden superior. Los datos representativos proporcionados han sido revisados por S.K.M., que no tiene ningún conflicto de intereses financieros.
Agradecimientos
Este trabajo fue financiado por el Instituto Nacional de Ciencias Médicas Generales (R43GM125420 y R44GM125420).
Materiales
| Name | Company | Catalog Number | Comments |
| 15 mL Conical Centrifuge Tubes | Fisher Scientific | 14-959-53A | any brand is sufficient |
| 50 µL SGE Gastight Syringes | Fisher Scientific | SG-00723 | |
| Acclaim PepMap 100 C18 nanocolumn (0.75 mm X 150 mm, 2 µm) | Thermo Scientific | ||
| Acetonitrile with 0.1% Formic Acid (v/v), LC/MS Grade | Fisher Scientific | LS120-500 | |
| Apomyoglobin | Sigma-Aldrich | ||
| Catalase | Sigma-Aldrich | C9322 | |
| Centrifuge | Eppendorf | 022625501 | |
| Delicate Task Wipers | Fisher Scientific | 06-666A | |
| Hydrogen Peroxide | Fisher Scientific | H325-100 | any 30% hydrogen peroxide is sufficient |
| Methionine amide | Chem-Impex | 03109 | |
| Microcentrifuge | Thermo Scientific | 75002436 | |
| Orbitrap Fusion Lumos Tribrid Mass Spectrometer | Thermo Scientific | Orbitrap Fusion Lumos Tribrid Mass Spectrometer | other high resolution instruments (e.g. Q exactive Orbitrap or Orbitrap Fusion) can be used |
| Pierce Trypsin Protease, MS Grade | Thermo Scientific | 90058 | |
| Polymicro Cleaving Stone, 1" x 1" x 1/32” | Molex | 1068680064 | any capillary tubing cutter is sufficient |
| UPLC | Thermo Scientific | ||
| Water with 0.1% Formic Acid (v/v), LC/MS Grade | Fisher Scientific | LS118-500 | |
| Water, LC/MS Grade | Fisher Scientific | W6-4 |
Referencias
- Nagarkar, R. P., Murphy, B. M., Yu, X., Manning, M. C., Al-Azzam, W. A. Characterization of protein higher order structure using vibrational circular dichroism spectroscopy. Current Pharmaceutical Biotechnology. 14 (2), 199-208 (2013).
- Giezen, T. J., Schneider, C. K. Safety assessment of biosimilars in Europe: a regulatory perspective. Generics and Biosimilars Initiative Journal. , 1-8 (2014).
- Giezen, T. J., Mantel-Teeuwisse, A. K., Strauss, S. Safety-related regulatory actions for biologicals approved in the United States and the Europena Union. Journal of the American Medical Society. 300 (16), 1887-1896 (2008).
- Gabrielson, J. P., Weiss, W. F. Technical decision-making with higher order structure data: starting a new dialogue. Journal of Pharmaceutical Sciences. 104 (1), 1240-1245 (2015).
- Brenowitz, M., Erie, D. A., Chance, M. R. Catching RNA polymerase in the act of binding: intermediates in transcription illuminated by synchrotron footprinting. Proceedings of the National Academy of Sciences U S A. 102 (13), 4659-4660 (2005).
- Guan, J. Q., Takamoto, K., Almo, S. C., Reisler, E., Chance, M. R. Structure and dynamics of the actin filament. Biochemistry. 44 (9), 3166-3175 (2005).
- Hambly, D. M., Gross, M. L. Laser flash photochemical oxidataion to locate heme binding and conformataional changes in myoglobin. International Journal of Mass Spectrometry. 259 (2007), 124-129 (2007).
- Li, K. S., Shi, L., Gross, M. L. Mass Spectrometry-Based Fast Photochemical Oxidation of Proteins (FPOP) for Higher Order Structure Characterization. Accounts of Chemical Research. 51 (3), 736-744 (2018).
- Watson, C., Sharp, J. S. Conformational Analysis of Therapeutic Proteins by Hydroxyl Radical Protein Footprinting. American Association of Pharmaceutical Scientists Journal. 14 (2), 206-217 (2012).
- Deperalta, G., et al. Structural analysis of a therapeutic monoclonal antibody dimer by hydroxyl radical footprinting. mAbs. 5 (1), 86-101 (2013).
- Jones, L. M., et al. Complementary MS methods assist conformational characterization of antibodies with altered S-S bonding networks. Journal of American Society of Mass Spectrometry. 24 (6), 835-845 (2013).
- Storek, K. M., et al. Monoclonal antibody targeting the β-barrel assembly machine of Escherichia coli is bactericidal. Proceedings of the National Academy of Sciences. , (2018).
- Vij, R., et al. A targeted boost-and-sort immunization strategy using Escherichia coli BamA identifies rare growth inhibitory antibodies. Scientific Reports. 8 (1), 7136 (2018).
- Liu, X. R., Zhang, M. M., Rempel, D. L., Gross, M. L. A Single Approach Reveals the Composite Conformational Changes, Order of Binding, and Affinities for Calcium Binding to Calmodulin. Analytical Chemistry. 91 (9), 5508-5512 (2019).
- Lu, Y., et al. Fast Photochemical Oxidation of Proteins Maps the Topology of Intrinsic Membrane Proteins: Light-Harvesting Complex 2 in a Nanodisc. Analytical Chemistry. 88 (17), 8827-8834 (2016).
- Marty, M., Zhang, H., Cui, W., Gross, M., Sligar, S. Interpretation and Deconvolution of Nanodisc Native Mass Spectra. Journal of American Society of Mass Spectrometry. 25, (2013).
- Johnson, D. T., Di Stefano, L. H., Jones, L. M. Fast photochemical oxidation of proteins(FPOP): A powerful mass spectrometry based structural proteomics tool. Journal of Biological Chemistry. , (2019).
- Chea, E. E., Jones, L. M. Analyzing the structure of macromolecules in their native cellular environment using hydroxyl radical footprinting. Analyst. 143 (4), 798-807 (2018).
- Aprahamian, M. L., Chea, E. E., Jones, L. M., Lindert, S. Rosetta Protein Structure Prediction from Hydroxyl Radical Protein Footprinting Mass Spectrometry Data. Analytical chemistry. 90 (12), 7721-7729 (2018).
- Linde. . Linde Specialty Gases of North America. , (2009).
- Aye, T. T., Low, T. Y., Sze, S. K. Nanosecond laser-induced photochemical oxidation method for protein surface mapping with mass spectrometry. Analytical Chemistry. 77 (18), 5814-5822 (2005).
- Niu, B., Zhang, H., Giblin, D., Rempel, D. L., Gross, M. L. Dosimetry determines the initial OH radical concentration in fast photochemical oxidation of proteins (FPOP). Journal of American Society of Mass Spectrometry. 26 (5), 843-846 (2015).
- Misra, S. K., Orlando, R., Weinberger, S. R., Sharp, J. S. Compensated Hydroxyl Radical Protein Footprinting Measures Buffer and Excipient Effects on Conformation and Aggregation in an Adalimumab Biosimilar. American Association of Pharmaceutical Scientists Journal. 21 (5), 87 (2019).
- Olson, L. J., Misra, S. K., Ishihara, M., Battaile, K. P., Grant, O. C., Sood, A., Woods, R. J., Kim, J. P., Tiemeyer, M., Ren, G., Sharp, J. S., Dahms, N. M. Allosteric regulation of lysosomal enzyme recognition by the cation-independent mannose 6-phosphate receptor. Communications Biology. 3 (1), 498 (2020).
- Sharp, J. S., Misra, S. K., Persoff, J. J., Egan, R. W., Weinberger, S. R. Real Time Normalization of Fast Photochemical Oxidation of Proteins Experiments by Inline Adenine Radical Dosimetry. Analytical Chemistry. 90 (21), 12625-12630 (2018).
- Misra, S. K., Sharp, J. S. Enabling Real-Time Compensation in Fast Photochemical Oxidations of Proteins for the Determination of Protein Topography Changes. Journal of Visualized Experiments. 163, (2020).
- Hambly, D. M., Gross, M. L. Laser flash photolysis of hydrogen peroxide to oxidize protein solvent-accessible residues on the microsecond timescale. Journal of American Society of Mass Spectrometry. 16 (12), 2057-2063 (2005).
- Liu, X. R., Zhang, M. M., Gross, M. L. Mass Spectrometry-Based Protein Footprinting for Higher-Order Structure Analysis: Fundamentals and Applications. Chemical Reviews. 120 (10), 4355 (2020).
- Jones, L. M., Sperry, B. J., Carroll, A. J., Gross, M. L. Fast photochemical oxidation of proteins for epitope mapping. Analytical chemistry. 83 (20), 7657-7661 (2011).
- Li, J., et al. Mapping the Energetic Epitope of an Antibody/Interleukin-23 Interaction with Hydrogen/Deuterium Exchange, Fast Photochemical Oxidation of Proteins Mass Spectrometry, and Alanine Shave Mutagenesis. Analytical chemistry. 89 (4), 2250-2258 (2017).
- Liu, X. R., Zhang, M. M., Rempel, D. L., Gross, M. L. A Single Approach Reveals the Composite Conformational Changes, Order of Binding, and Affinities for Calcium Binding to Calmodulin. Analytical Chemistry. 91 (9), 5508-5512 (2019).
- Kiselar, J. G., Janmey, P. A., Almo, S. C., Chance, M. R. Structural analysis of gelsolin using synchrotron protein footprinting. Molecular and Cellular Proteomics. 2 (10), 1120-1132 (2003).
- Chea, E. E., Deredge, D. J., Jones, L. M. Insights on the Conformational Ensemble of Cyt C Reveal a Compact State during Peroxidase Activity. Biophysical Journal. 118 (1), 128-137 (2020).
- Poor, T. A., et al. Probing the paramyxovirus fusion (F) protein-refolding event from pre- to postfusion by oxidative footprinting. Proceedings of the National Academy of Sciences U S A. 111 (25), 2596-2605 (2014).
- Roush, A. E., Riaz, M., Misra, S. K., Weinberger, S. R., Sharp, J. S. Intrinsic Buffer Hydroxyl Radical Dosimetry Using Tris(hydroxymethyl)aminomethane. Journal of American Society of Mass Spectrometry. 31 (2), 169-172 (2020).
- Everett, E. A., Falick, A. M., Reich, N. O. Identification of a critical cysteine in EcoRI DNA methyltransferase by mass spectrometry. Journal of Biological Chemistry. 265 (29), 17713-17719 (1990).
- Sanderson, R. J., Mosbaugh, D. W. Identification of specific carboxyl groups on uracil-DNA glycosylase inhibitor protein that are required for activity. Journal of Biological Chemistry. 271 (46), 29170-29181 (1996).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados