Method Article
跨波森插入测序作为工具,以阐明细菌殖民因素在拉克霍尔德里亚角膜甲虫
摘要
这是一种适应方法,用于识别 伯克霍尔德里亚 有益共生的候选昆虫殖民因子。甲虫宿主感染了通过转波突变产生的随机突变库,殖民化后的库复杂性被比作 体外生长的对照。
摘要
通过操纵基因的活动来推断基因的功能是理解大多数生物过程的遗传基础的重要工具。分子微生物学的进步见证了各种突变技术的出现,用于基因的操纵。其中,转波子插入测序(Tn-seq)是一个有价值的工具,可以同时以非目标的方式评估许多候选基因的功能。该技术是确定几个致病微生物和几个有益共生体中真核宿主殖民化的分子机制的关键。
在这里,Tn-seq被确定为一种方法,以确定殖民因素在共同的伯克霍尔德里亚角斗士共生甲虫拉格里亚维洛萨。通过结合,Tn5转波介导插入抗生素耐药盒是在B.格拉迪奥利的随机基因组位置进行的。为了确定基因破坏对细菌殖民甲虫宿主的能力的影响,生成的B.角质变异库在甲虫卵上接种,而控制在液体培养介质中体外生长。在留出足够的时间进行殖民化后,DNA从体内和体外生长的库中提取。根据DNA库准备协议,DNA样本为转波子插入测序做好准备。选择包含转波插入边缘和侧翼细菌DNA的DNA片段,突变位点通过从转波插入边缘进行测序确定。最后,通过分析和比较体内和体外库之间每个突变体的频率,可以预测甲虫殖民化期间特定共生基因的重要性。
引言
伯克霍尔德里亚角斗士可以与拉格里亚维洛萨甲虫进行共生关联,在防御昆虫宿主4、5、6的微生物拮抗剂方面发挥重要作用。雌性甲虫在生殖系统的专门腺体中容纳了几株B.角质。产卵后,雌性在蛋表面涂抹B.角膜细胞,其中B.角蛋白产生的抗菌化合物通过致病真菌4、6抑制感染。在胚胎发育后期或幼虫孵化后的早期,细菌在幼虫的腹腔表面殖民性脑腔入侵。尽管有这种专门的本地化和垂直传输路线的共生,L.维洛萨大概也可以从环境4水平获得B.格拉迪奥利。此外,至少发现了三株B.格拉迪奥利与L.维洛萨4,6。其中,B.格拉迪奥利Lv-StA是唯一一个是适应体外培养。
B. 格拉迪奥利Lv-StA的基因组大小为8.56Mb6,包含7,468个基因。这些基因中哪些对B.角质细菌殖民甲虫宿主很重要?为了回答这个问题,我们使用转波插入测序(Tn-seq),一种探索性的方法来识别有条件的基本微生物基因1,2,3。使用 Tn5 转子创建了B. 格拉迪奥利Lv - sta 的突变库。通过从大肠杆菌捐献细胞到B.格拉迪奥利Lv-StA的结合,转移了携带Tn5转波子的pRL27质粒和一个抗生素耐药盒,旁边是倒置重复(图1)。由此产生了一组单独携带3,736个共生基因中断的突变体(图2)。
突变池被感染到甲虫卵上,以确定殖民化因素,并且,作为一种控制,也生长 在国王 的B(KB)介质体外。在留出足够的时间进行殖民化后,将孵化的幼虫收集并汇集起来进行DNA提取。使用经过修改的DNA库准备程序进行测序,选取了包含转波子插入物的DNA片段和 B.格拉迪奥利 Lv-StA侧翼基因组区域。阅读质量处理,然后与DESeq2进行分析,以确定特定的基因,关键 B.格拉迪奥利 Lv-STA殖民 L.维洛萨 幼虫时,通过蛋表面传播。
研究方案
1. 媒体和缓冲准备
- 准备 KB 和 LB 介质和 agar 板, 如表 1所示,并在 121 °C、15 psi、20 分钟时自动飞盘。
- 在培养 大肠杆 菌 WM3064 + pRL27 之前,将 50 μg/mL 过滤灭菌的卡纳霉素和 300 μM 过滤灭菌 2,6-氨基甲酸 (DAP) 添加到自动克隆的 LB 介质中。
- 加入 50 μg/mL 过滤消毒的卡纳霉素到自动结裂的 KB 琼脂倒板所需的选择成功的 B. 格拉迪奥利 Lv-StA 转糖剂。
- 通过混合以下组件,准备 1 倍磷酸盐缓冲盐水 (PBS): NaCl 8 g/L, KCl 0.201 g/L, Na2HPO4 1.42 g/L, 和 KH2PO4 0.272 g/L. 溶解蒸馏水中的盐,并在使用前 121 °C、15 psi、20 分钟自动将混合物解压。在室温下存放。
- 通过溶解以下组件,准备 2 倍绑定和洗涤缓冲:10 mM Tris-HCl(pH 7.5)、1 mM 乙烯胺四乙酸 (EDTA) 和蒸馏水中的 2 M NaCl。使用前过滤消毒混合物。在室温下存放。
- 在双蒸馏水中溶解 10 mM Tris-HCl (pH 8.0) 和 0.1 m EDTA,准备 1x 低 TE。在 121 °C、15 psi、20 分钟自动包装消毒。在室温下存放。
2. 结合生成转波变异库
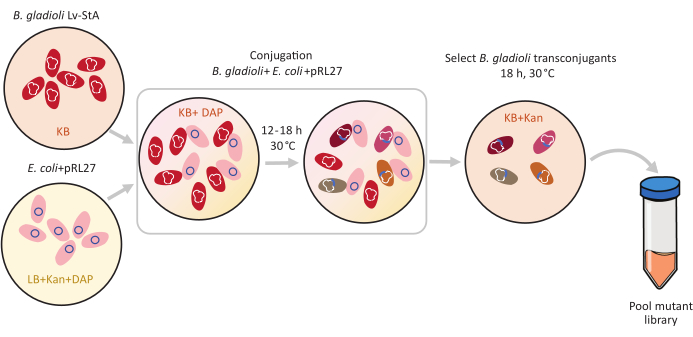
图1:结合协议步骤。 结合接受者 伯克霍尔德里亚角斗士 Lv-StA(红色)和捐赠者 Escherichia大肠杆菌 含有pRL27质粒(粉红色)分别生长在KB琼脂和LB,辅以卡纳霉素和DAB。在30°C下将质粒共生转移12-18小时后,在含有卡纳霉素的KB上选择转相融合 B.角质 细胞并聚集在一起。缩写: DAP = 2,6-亚米诺司他酸;坎 = 卡纳米辛。 请单击此处查看此图的较大版本。
- 在无菌罩下,接种新鲜的雌 性大肠杆菌 WM3064 + pRL27的捐赠培养物,在10 mL的LB介质中补充卡纳霉素和DAB。在 5 mL 的 KB 介质中接种 伯克霍尔德里亚角斗士 Lv - sta 接收细胞。在 250 rpm 的摇床上,在 30 °C 的夜间孵育文化。
- 隔夜生长后,离心机4ml的每个培养物在9,600× 克 6分钟颗粒细胞。丢弃超高纳特。
- 在无菌罩下,清洗含有 DAP 的 KB 介质中的颗粒细胞培养物,最后在 4 mL 的 KB + DAP 介质中分别重新使用培养物。
- 在新鲜的 15 mL 管中,将 250 微升洗过 的大肠杆菌 供体细胞与 1 mL 的洗涤 B. 格拉迪奥利 Lv-StA 接收细胞混合。
- 在含有 DAP 的 KB 琼脂板上发现 10μL 的这种结合细胞混合物。让盘子在室温下在无菌罩中不受干扰地休息1小时。然后,在 30 °C 的 12-18 小时下,将板块与结合点孵育。
注:结合期可根据目标物种进行调整。然而,长时间的结合会增加双重插入或质粒整合到基因组的风险。对于生长缓慢的细菌,允许更长的结合期。 - 孵化后,在无菌罩下的板中加入2-4mL的1x PBS,并使用细胞刮刀从琼脂中释放生长的细菌结合点。将结合细胞混合成 2 mL 微融合管。
- 通过离心在9,600×g的细胞中弹丸2分钟。 丢弃超高纳特,通过上下管道在 1 mL 的 1x PBS 中清洗颗粒两次。以 1200 微升 1x PBS 中重新使用最终颗粒。如果混合物中的细胞数量超过 1 × 104,则在电镀前进行稀释。
- 混合好,将200微升的细胞混合物撒在大KB阿加板(如果需要的话,6或更多)上,辅以卡纳霉素,并在30°C过夜孵育。
注:目标突变菌落出现在选择性阿加板的30小时内。由于抗生素耐药性标记,只有变异的菌落出现在选择性阿加板上。因此,所有殖民地都有望成为成功的转制体。 - 计算三个板块上的跨相聚落总数,并推断以计算所有板块中获得的突变体的大致数量。为了增加获得代表性库的机会,确保菌落总数比基因组中的基因总数高几倍。为了确认结合的成功,使用 10-20 个样本菌落执行针对插入盒式磁带的 PCR,如第 3 节所述。
注:目的是确保菌落的数量至少是整个基因组中基因数量的10倍,在这种情况下,>75,000突变体。然而,准确估计与完全具有代表性的图书馆相对应的殖民地数目通常具有挑战性。目前,由于未捕获基本基因的破坏,因此突变的独特基因数量并不明显,同一基因通常存在多个不同的突变位点,而Tn5转子产生的突变并非完全随机。 - 在无菌罩下,通过在油上添加 1-2 mL 的 1x PBS 来刮掉盘子中的菌落。将电池混合物从板中刮成 50 mL 管。漩涡库彻底混合,然后将 4 mL 的池式突变库拆分为几个低温管。在管子中加入 1 mL 的 70% 甘油,并储存在 -80 °C 下。
3. PCR 和凝胶电泳,以确认成功插入 B. 格拉迪奥利 Lv-StA
- 为了确认插入的存在,从第 2.9 步中的选择板中挑选单个突变菌落,并使用 表 2中列出的引物执行针对插入盒的 PCR。根据 表 3 准备 PCR 主组合,并在表 4中描述的热循环器中设置条件。
- 通过电泳(250 V,40 分钟)在 1.6% 的玫瑰凝胶上运行 PCR 产品,以检查放大的 DNA 片段是否预期长度为 1580 bp。
4. 甲虫卵突变池感染
- 库洗涤步骤
- 在冰上解冻准备好的突变库的一个小木板。离心机在 2,683 × g 10 分钟,并删除超高。在无菌罩下,用 4 mL 的 1x PBS 清洗细胞,从细胞中取出剩余的介质。将电池重新悬回 1x PBS 的 4 mL 中。
- 使用细胞计数室计算库中细胞的数量。将库的一部分稀释到 2 × 106 个单元格/μL,以 1x PBS 进行。
- 漩涡图书馆完全混合整个图书馆同质之前,采取所需的卷。
- 鸡蛋离合器绝育和 体内 感染
- 选择 L. 维洛萨 鸡蛋离合器。计算鸡蛋的数量,如果离合器包含超过100个鸡蛋,继续。
- 消毒整个鸡蛋离合器。
- 加入200微升70%乙醇,轻轻清洗鸡蛋5分钟。取出乙醇,用自动洗净水清洗鸡蛋两次。
- 加入 200 μL 的 12% 漂白剂 (NaOCl), 轻轻清洗鸡蛋 30s。立即取出漂白剂,用200微升的自动洗涤水再次清洗鸡蛋三次。
- 感染 2 × 106 细胞/μL 的洗过突变库消毒鸡蛋离合器 (2.5 μL 每个鸡蛋).
- 受感染的甲虫幼虫孵化两天后,每1.5mL微灌注管收集1002只 星形幼虫,储存在-80°C。
-
体外 突变库控制
- 在无菌罩下,在含有卡纳霉素的 KB 介质的 10 mL 中接种 250 μL 的 2 ×10 6 细胞/μL 的洗涤突变库。
- 在 30 °C 下以 20 小时孵育 体外 突变培养。
注:计算孵化时间,以匹配殖民期间体内WT B. 格拉迪奥利Lv-StA的近代数量。 - 在 20 h 孵化后, 在体外 突变培养中加入等量的 70% 甘油,并将其存储在 -80 °C 下。
5. 感染甲虫和 体外 突变库DNA提取
注:DNA提取使用DNA和RNA纯化套件进行,根据制造商的协议简要概述如下。
- 通过添加 1-2 mL 液氮和用害虫粉碎,将集合幼虫(每根微灌注管最多 4 毫克)同质化。
- 从冰上的甘油库存中解冻 体外 生长的突变培养物。在细胞裂解前10分钟,在9,600× 克 的离心下离心,对细胞进行颗粒。
- 在体外和体内样品中加入 300 μL 组织和细胞裂解溶液。加入 5 μL 的 10 毫克/mL 蛋白酶 K,在 60 °C 下孵育混合物 15 分钟,然后放在冰上 3-5 分钟。
- 在赖出物和漩涡中加入150微升的蛋白质沉淀试剂。通过离心将蛋白质碎片以 9,600 × 克 离心,10 分钟。
- 将超导管转移到 1.5 mL 微模糊管中。将 500 μL 的同位素添加到超高分子中,并轻轻倒置管子至少 40 次,然后在 -20 °C 下孵育 1 小时或过夜。
- 通过离心在9,600×g下离心10分钟,颗粒沉淀DNA。 丢弃超高氨酸,在DNA颗粒中加入冰冷的70%乙醇。
- 离心机在≥10,000× 克 5分钟。丢弃超高超剂,将样品留空至少 1 小时。
- 在 100 微升低 TE 缓冲器 中从体外 和 体内样本中 重新提取 DNA。
- 将样品存储在 -20 °C 下。
6. 测序库准备
注:DNA库制备的协议和试剂是根据DNA库制备套件制造商提供的说明进行调整和修改的。
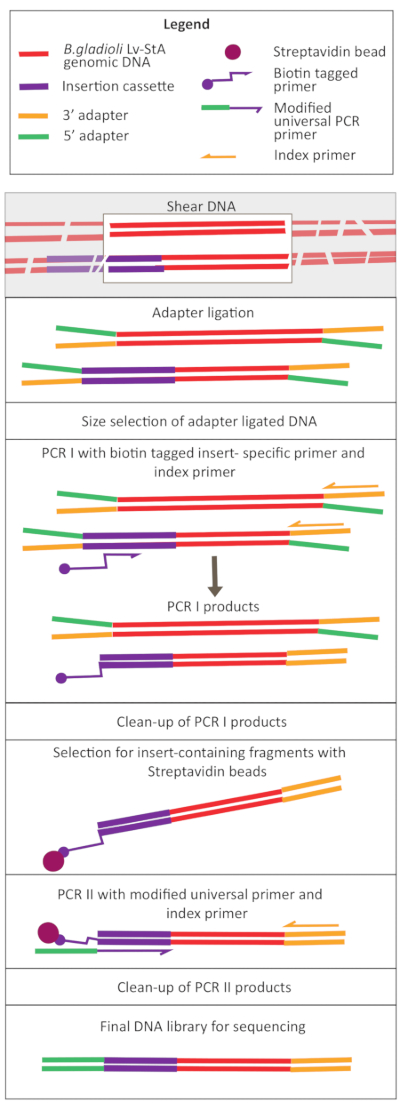
图2:DNA库准备步骤的示意图。 剪切和适配器连合后,修改后的协议包括链球菌珠选择步骤,以丰富包含插入盒式磁带的DNA片段。 请单击此处查看此图的较大版本。
- 将样品稀释至 20 ng/μL 浓度和 100 μL 的体积,并将其保留在冰上。
- 使用超声波仪 在体内 和 体外 采样DNA。将超声波器的功率设定为 70%。将样品短暂地切切至1分30秒。
注:超声波仪的设置因仪器而异。在这种情况下,碎片大小为 200-400 bp,适合这种 150 bp 的测序方法,配对端(见步骤 9.1)。剪切参数可以根据实验者的要求进行调整。 - 检查DNA是否剪切到所需的大小范围(在这种情况下,200-400桶/日)。装载5微升的未剪切和剪切DNA后,与凝胶加载染料混合在1:1的比例1.6%的玫瑰凝胶运行在250V 40分钟(图3A,B)。
- 为适配器结结所需的片段端准备
- 要将剪切的DNA的 50 μL 添加库制备套件中提供的末端制剂试剂:3 μL 的酶混合物和 7 μL 的反应缓冲器,并通过管道很好地混合。将加热盖的热循环器设置为 75 °C ≥,并在 20 °C 时孵育样品 30 分钟,在 65 °C 下孵育 30 分钟。 保持在 4 °C 下。
- 适配器连合
- 对于适配器结扎,在端准备步骤的产品中添加以下试剂:30 μL 配合主混合、1 μL 配结增强器和 2.5 μL 稀释适配器。通过管道彻底混合,在加热循环器中加热盖子,在 20 °C 下将样品孵育 15 分钟。
- 15分钟后,加入3微升酶(尿素DNA糖酶 + DNA糖酶-酶-酶内脏酶VIII)(见 材料表)。通过管道混合,在热循环器中将样品在 37 °C 下孵育 15 分钟,盖子在 ≥47 °C 下加热。
注:在此步骤中可以暂停协议,样品可以存储在 -20 °C 下。
- 适配器-利盖特DNA的尺寸选择,目标片段为250个基点
- 涡流磁珠溶液(参见 材料表),并在室温下放置30分钟后再使用。
- 将 0.3 倍的珠子添加到 96.5 μL 的液化 DNA 混合物中,并通过透彻的管道混合。孵化珠子混合物5分钟。
注:珠子混合物中盐和聚乙二醇的存在有助于珠子上DNA片段的沉淀。珠子与DNA分子的低比率导致只有较大的DNA片段与珠子结合。在这种情况下,长度超过250个基点的DNA片段与珠子结合。 - 将管子放在磁架上,拉下珠子,去除不需要大小的DNA片段。让珠子沉淀5分钟,然后将透明超强管转移到一个新的微富管(保持超强管)。
- 将0.15倍的新鲜珠子加入超高音,通过管道进行混合。将珠子混合物孵育5分钟,然后将管子放在磁架上,将绑在目标DNA上的珠子拉下来。等待 5 分钟, 然后丢弃超那特 (保持珠子) 。
注:珠子与DNA的这种比例导致所需的250个基点大小的碎片结合。 - 在磁架上放上珠子时,加入 200 微升 80% 乙醇(新鲜制备),等待 30s。派佩特出来,并丢弃乙醇洗仔细,而不会打扰磁性支架上的珠子。重复此步骤。
- 最后一次洗涤后,从珠子中取出乙醇的痕迹,然后用空气干燥珠子2分钟,直到它们看起来有光泽但尚未完全干涸。不要使珠子过干。
- 从磁架上取下管子,加入 17 微升 10 mM Tris-HCl 或 0.1x TE (低 TE)。通过管道混合 +10 次,并在室温下孵育混合物 2 分钟。
- 将管子放回磁架上,等待 5 分钟。一旦珠子安定下来,将DNA超高分子转移到一个新的管子上。
- PCR I 将生物素标签添加到包含插入盒式磁带的 DNA 片段中
- 使用转波素特异性生物素底漆(表5)和索引引线引物,在包含Tn5插入盒式磁带的DNA片段中加入生物染色底漆标签。根据 表6 准备PCR主组合,并遵循 表7中列出的热循环器的PCR条件。
- 无尺寸选择的 PCR I 的清理
- 漩涡 0.9 倍珠子,在清理前在室温下放置至少 30 分钟。
- 在 PCR 产品中加入 0.9 倍珠子,并彻底混合。
- 将珠子放在磁架上拉下珠子。
- 取出透明超高酸剂,用200微升新制备的80%乙醇清洗珠子DNA两次。
- 洗涤步骤后取出乙醇,将珠子晾干,直到它们看起来有光泽但不太干燥。
- 加入 32 微升 10 m M Tris-HCl 或 0.1X TE (低 TE), 孵育珠子 5 分钟。将混合物放回磁架上,将超高纳特转移到新鲜的微灌管中。
- 将生物素化DNA片段绑定到链球菌珠上
- 在 1x 绑定和洗涤缓冲器中重新使用 32 μL 的链球菌珠。将珠子放在磁性支架上时,用缓冲器清洗三次。
- 加入 32 μL 的 2x 绑定和洗涤缓冲器,并重新悬浮珠子。为此,添加 32 μL 的清理 PCR 1 产品。在室温下彻底混合并孵育30分钟。
- 将珠-DNA混合物放在磁架上2分钟。派佩特把超高纳特作为生物素标记的DNA, 含有插入边缘与珠子上的链球菌素结合。
- 用 500 μL 的 1x 绑定和洗涤缓冲器清洗珠子,然后用 200 μL 的低 TE 洗珠子。在 17 微升低 TE 中重新使用 DNA 绑定珠子。
- PCR II 将适配器添加到包含插入盒式磁带边缘的片段中
- 使用表5中列出的索引引引和修改的通用 PCR 底漆(如表 8所示)准备主组合。将前一步中 DNA 绑定链球菌珠的 15 μL 添加到 PCR 混合物中。有关热循环器条件,请参阅表 7。
- 清理 PCR 产品,而不会按照本协议第 6.8 步给出的尺寸选择。在30微升分子级水中提取最终DNA产品。
- 将样品存储在 -20 °C 下,并将其用于测序。
7. 测序和分析
- 使用高通量测序技术对库进行排序。根据转波子库大小调整测序深度,如下所示。使用 FastQC7评估读取质量。选择包含阅读 5'末端的 Tn5 插入边缘的读取,并使用Cutadapt8 和/或修剪9删除插入边缘序列。
注:在这里,使用配对端测序方法,以每读 150 个基点为目标,总共针对 8 Mio 读取。要获得具有代表性的数据集,确保序列读取总数超过库中可能的最大突变体数量,即从第 2.9 步开始的菌落总数。作为参考,此协议旨在将最大库大小的 40 倍。其他使用Tn-seq用于类似目的的成功研究,使阅读总数在相应的突变库22,23中实际插入次数的近25倍。 - 考虑到基因末端的突变在功能上没有破坏性,请将参考基因组GFF文件的基因注释两端削减5%。使用 Bowtie210将修剪后的读数映射到参考基因组。
- 计算对齐 BAM 文件中唯一 5' 位置的插入次数。
- 使用功能计数11,获取每个复制样本的命中基因数。
- 使用 RStudio 中的 DESeq212 包计算不同条件之间的突变丰度差异。
结果
宿主相关细菌可以利用几个因素来建立关联,包括那些中介粘附、活动性、化疗、应激反应或特定的运输工具。虽然对病原体-宿主相互作用很重要的因素已经报告为几个细菌13,14,15,16,17,18,包括伯克霍尔德里亚属19,20的成员,较少的研究已经探索了有益共生体用于殖民化的分子机制21,22,23.使用转波子插入测序,目的是确定分子因素,使B.格拉迪奥利殖民L.维洛萨甲虫。
跨波森介质变异是使用pRL27质粒进行的,它携带Tn5转子和卡纳霉素抵抗盒,两侧是倒置重复点。质粒通过与质粒捐赠者大肠杆菌WM3064菌株(如图1所示)结合,被引入目标B.格拉迪奥利Lv-StA细胞中。结合后,含有B.角质素接受者和大肠杆菌供体细胞的结合混合物被镀在含有卡纳霉素的选择性加板上。板块上缺乏DAB消除了供体大肠杆菌细胞,以及为成功的B.格拉迪奥利Lv-StA转种物选择的卡纳霉素的存在。从收获10万个跨聚变菌落中获得的B.格拉迪奥利Lv-StA突变库是准备使用经过修改的DNA库制备套件和自定义引物进行测序的。图2突出了DNA库的准备步骤。测序产生 4 Mio 配对读取;在B.格拉迪奥利Lv-StA的7,468个基因中,有3,736个基因被破坏。
为了识别宿主中具有殖民缺陷的突变体 ,B. gladioli Lv-StA 突变库在甲虫卵上被感染,并在 KB 介质 体外 生长作为对照。 体内 殖民化瓶颈大小是在实验前计算的。已知数量的 B.gladioli Lv-StA细胞在甲虫卵上被感染,通过从每个幼虫身上电镀悬浮和计算每个个体的群落形成单位,在新鲜孵化的第一个星内幼虫中获得了殖民细胞的数量。这些计算是为了确保殖民细胞的数量足以评估库中所有或高百分比的突变体,以评估它们对宿主进行殖民的能力。此外,体 外 和 体内 条件之间的生长时间根据细菌代数正常化,使这些样本具有可比性。
卵子孵化后,在13个池中收集了1,296只幼虫。相应的体外突变培养物被作为甘油储存。在超声波仪中提取和碎片提取体内和体外生长突变库的DNA。图3显示了剪切DNA的大小分布,其中大多数片段的跨度在100到400桶之间,如预期的那样。在此步骤之后,修改了测序DNA库准备协议。在协议的每一步,检查剩余DNA的浓度,以确保步骤正确执行,并跟踪DNA的损失。测序前的一次质量检查(见材料表)显示,DNA库中意外地含有大(>800个基点)DNA片段,这在体内库中更为明显。鉴于在测序通道中难以优化片段的聚类,有必要将测序深度增加到 10 Mio 配对读取在体内库中,以达到所需的读数。对测序结果的分析表明,在体内图书馆中平均有4个Mio读取,在体外库中平均有3.1个Mio读取,在Read-1(表9)的5'末端包含了Transposon边缘,这对于这个实验是令人满意的。图4显示,在原始库中,B.gladioli基因组中24,224个独特的插入的分布。 使用DESeq2进行的分析表明,271个突变体的丰度在体内和体外条件下有显著差异。
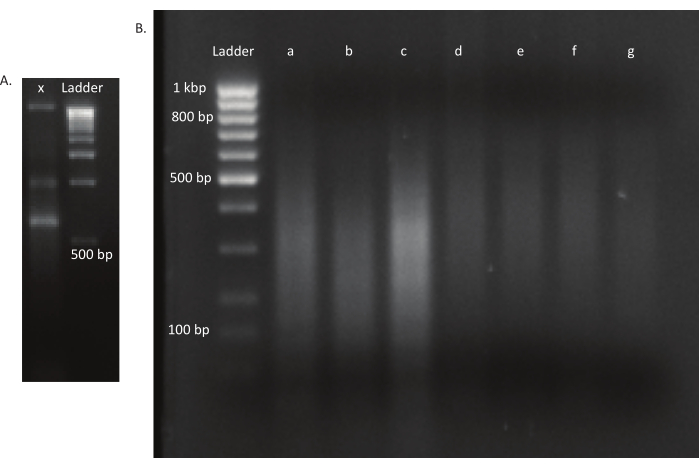
图3:突变体和DNA库的阿加罗斯凝胶。 (A) 阿加罗斯凝胶,带有车道 x 中突变体的未剪切 DNA 和 1 kbp 梯子。(B) 凝胶与剪切DNA库。第一车道梯子的带大小在左侧指示。前三条车道 a、b 和 c 包含 体内库中的 剪切 DNA 片段。 车道 d、e、f 和 g 包含 体外 库的剪切 DNA 片段。 请单击此处查看此图的较大版本。
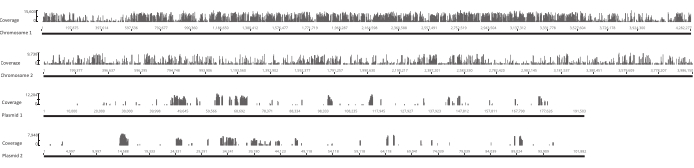
图4:在 伯克霍尔德里亚角斗士 Lv-StA基因组的四个复制物中,在原始库中独特的插入位点的位置。 沿 x 轴的每个条都位于插入点。沿 y 轴的条形高度对应于与该站点相关的读取次数。请注意,两条染色体和两个质粒显示全长,因此 x 轴上具有不同的刻度。 请单击此处查看此图的较大版本。
| 国王的 B 中等/阿加 | |
| 佩普顿(大豆) | 20克/升 |
| K2HPO4 | 1.5 克/升 |
| Mgso4。7H2O | 1.5 克/升 |
| 琼脂 | 15 克/升 |
| 溶解在蒸馏水中 | |
| LB 介质/阿加 | |
| 特里普通 | 10克/升 |
| 酵母提取物 | 5 克/升 |
| 纳克 | 10克/升 |
| 溶解在蒸馏水中 | |
表1:媒体组件。
| 不。 | 引 | 序列 | PCR 退火温度。 (+C) | |
| 1 | tpnRL17-1RC | 5 '- 克塔卡特切克特 - 3' | 58.2 | |
| 2 | tpnrl13-2RC | 5 '- 特格特加加格特格特克特格 - 3' | ||
表2:底漆,以确认结合的成功。
| 元件 | 卷 (μL) |
| HPLC 净化水 | 4.92 |
| 10 倍缓冲 S(高特异性) | 1 |
| MgCl2 (25mM) | 0.2 |
| dNTP (2 mM) | 1.2 |
| 引数 1 (10 pmol/μL) | 0.8 |
| 引数 2 (10 pmol/μL) | 0.8 |
| 塔克 (5 U/μL) | 0.08 |
| 主混音总计 | 9 |
| 模板 | 1 |
表3:PCR主组合,以确认结合的成功。 缩写:HPLC = 高性能液相色谱;dntps = 脱氧核苷三磷酸盐。
| 步骤 | 温度 +C | 时间 | 周期 |
| 初始变性 | 95 | 3 分钟 | 1 |
| 变性 | 95 | 40s | |
| 退火 | 58.2 | 40s | 30 至 35 |
| 外延 | 72 | 1-2 分钟 | |
| 最终扩展 | 72 | 4 分钟 | 1 |
| 拿 | 4 | ∞ | |
表4:PCR条件,以确认结合的成功。
| 引 | 序列 | Tm =C | 用 | 源 | |
| 跨波森特异性生物染色引线 | 5'-比奥汀-阿加卡塔克特加卡特 -3' | 63.5 | 6.7.1. PCR I | 习惯 | |
| 修改后的通用 PCR 引数 | 5'- 阿塔克塔克加恰恰加特克 塔克特克切克塔卡加克克 特克格加特加特卡特卡特加特 格特加特格特 - 3' | 62 | 6.10.1. PCR II | 习惯 | |
| 索引引点 | 请参阅制造商手册 | 6.7.1. PCR I = 6.10.1.PCR II | 伊卢米纳的 Nebnext 多路复用奥利戈斯 (索引器集 1) | ||
| 适配器 | 请参阅制造商手册 | 6.5. 适配器连合 | 尼布内斯特超 II DNA 库准备套件为伊卢米纳 | ||
表5:DNA库准备过程中PCR I和II的引点和适配器。
| PCR 组合 | (μL) |
| 适配器配体DNA片段 | 15 |
| NEBNext 超 II Q5 主组合 | 25 |
| 索引引引(10pmol/μL) | 5 |
| 跨波森特异性生物素化引数 (10 pmol/ μL) | 5 |
| 总成交量 | 50 |
表6:DNA库准备-PCR I主组合。
| 步骤 | 温度 | 时间 | 周期 |
| 初始变性 | 98 °C | 30s | 1 |
| 变性 | 98 °C | 10s | 6 至 12 |
| 退火 | 65 °C | 30s | |
| 外延 | 72 °C | 30s | |
| 最终扩展 | 72 °C | 2 分钟 | 1 |
| 拿 | 16 °C | ∞ | |
表7:DNA库制备-PCR I和II条件。
| PCR 组合 | (μL) |
| 珠子选择的DNA | 15 |
| NEBNext 超 II Q5 主组合 | 25 |
| 索引引点 | 5 |
| 修改后的通用 PCR 引数 | 5 |
| 总成交量 | 50 |
表8:DNA库制备-PCR II主组合。
| 图书馆 | 伊维沃-1 | 伊维沃-2 | 伊维沃-3 | 英维特罗-1 | 英维特罗-2 | 英维特罗-3 | 原始库 | |
| 不。读数 (PE) | 56,57,710 | 39,19,051 | 30,65,849 | 35,73,494 | 28,83,440 | 36,61,956 | 46,09,410 | |
| 不。包含 Tn 的阅读 - 阅读 1 末尾的 5' 边缘 | 54,15,880 | 37,31,169 | 29,36,247 | 33,00,499 | 27,35,705 | 33,50,402 | 41,53,270 | |
| 弓形 2 整体对齐率 (%) (仅限阅读-1) | 95.53% | 83.71% | 89.87% | 80.79% | 78.00% | 73.06% | 74.92% | |
| 唯一插入次数 | 8,539 | 4,134 | 7,183 | 18,930 | 18,421 | 20,438 | 24,224 | |
| 命中的基因数量 | 1575 | 993 | 1450 | 2793 | 2597 | 3037 | 3736 | |
表9:每个库的测序输出和转波插入频率摘要。 缩写:PE = 配对端。
讨论
生成了 B.角膜 突变库,以识别 L.维洛萨 甲虫和 B.角膜 菌共生相互作用中重要的宿主殖民因子。协议中的主要步骤是结合、宿主感染、DNA库准备和测序。
由于许多菌株的伯克霍尔德里亚是适应基因改造的结合24,25,质粒携带转波子和抗生素插入盒成功地结合到目标B.格拉迪奥利Lv-StA菌株从大肠杆菌。以前通过电电转换的尝试产生非常低到几乎没有B.角斗士变压剂。建议优化目标生物体的转化技术,以有效产生大量的转化剂。
一轮结合和40个结合点打乱了 B.格拉迪奥利 Lv-StA的3,736个基因。事后看来,要破坏7,468个基因中的大多数,并获得饱和库,需要多轮结合。值得注意的是,在结合期间的孵化时间不允许超过12-18小时,这是 B.gladioli指数增长阶段的结束。允许在细菌细胞的指数生长阶段之外进行结合,可以降低成功获得转相体26的机会。因此,结合期应根据细菌物种的生长情况进行调整。
要成功地进行一项涉及宿主中突变库感染的实验,重要的是要评估殖民化期间的细菌种群瓶颈大小和感染前库中突变体的多样性。在准备实验时,我们估计了必须感染的甲虫的最小数量,以便对库中的每个突变体进行采样并允许殖民的可能性很高。还计算了活体细菌生成时间的近似值和实验期间的代数。体外文化通过调整孵化时间,成长为可比的几代人。对于其他非模型宿主的类似感染实验,维持实验室培养和宿主生物源的能力是可取的。
随着变异库 在体内 、 体外 和样本采集中的发展,进行了变异体插入测序的DNA库准备方案的修改。协议中的修改包括设计自定义 PCR 引物并添加 PCR 步骤,以选择包含插入盒式磁带的 DNA 片段。由于协议是定制的,协议中额外的 PCR 周期增加了过度放大的风险,并在最终库中获取杂交适配器适配器片段。因此,建议在两个 PCR 之后进行最后的清理步骤(无尺寸选择),因为它有助于删除这些片段。DNA库的规模分布仍然比预期的要广。然而,增加测序深度提供了在生物信息学分析过程中过滤的足够数据,取得了令人满意的结果。
由于转波介导突变在单个实验中产生数千个随机插入,因此有可能生成一个饱和突变体库,其中包含除那些对细菌生长至关重要的基因被破坏的突变体以外的所有突变体。我们很可能没有与饱和突变库合作,因为在关于伯克霍尔德里亚sp的其他研究中,我们估计了基本基因。28,29.然而,一个非饱和图书馆有助于探索各种候选基因,以便使用靶向突变进行进一步研究。在实验之前,还必须记住,一些转位子有特定的插入目标位点,在基因组30的某些位点增加突变体的丰度。水手转位器已知瞄准AT站点插入31,和Tn5转子有GC偏置32,33。包括生物信息学分析期间识别转电体插入热点的步骤,将有助于评估任何分布偏差。
虽然容易受挫,但精心设计的转波子插入测序实验可以成为在单个实验中识别细菌中许多具有条件重要基因的有力工具。例如,通过结合转波子突变和基因组学34,在伯克霍尔德里亚半纳利斯的十几个基因对抑制兰叶坏死非常重要。除了伯克霍尔德里亚,一些粘附和动感基因和运输器已被确定为重要的殖民因素在斯诺德格拉塞拉阿尔维共生阿皮斯梅利费拉(蜜蜂)22,并在维布里奥菲舍里共生欧普林纳牛皮(夏威夷波尾鱿鱼)23使用跨波森插入突变方法。
作为另一种方法,转体突变后,可以使用选择性介质而不是测序来筛查单个突变体。表型筛查或生物测定,以识别缺陷,如运动性,生产生物活性二次代谢物,或特定的辅助物,是可行的。例如,筛选一个 伯克霍尔德里亚昆虫 库(重新分配给卡 巴列罗尼亚属35)变异库是确定共生体使用动能基因殖民 里普托图斯小虫的关键,他们的昆虫宿主36。此外,利用转波突变和表型筛查,在 伯克霍尔德里亚卡约菲利37中发现了生物活性二次代谢性肌蛋白的生物合成基因簇。在转体突变和筛查后,发现了 一种伯克霍尔德里亚伪多马雷 的辅助营养变异体,并且是一种可能衰减的疫苗候选者,可以预防甲状腺病,这是一种在人类和动物中的危险疾病。因此,转波子突变和测序是研究细菌分子特征的宝贵方法,这些分子特征对于在致病性或相互关联中与各自宿主的相互作用非常重要。
披露声明
作者宣称他们与这项研究没有利益冲突。
致谢
我们感谢李俊波在手术过程中提供 大肠杆菌 WM3064+pRL27菌株的结合和指导,感谢凯瑟琳·霍夫迈尔在突变图书馆生成过程中帮助故障排除,感谢安德烈·罗德里格斯教授支持昆虫收集和许可获取。我们还感谢雷贝卡·扬克和达格玛·克莱布施在昆虫的收集和饲养方面给予的支持。我们感谢巴西当局为昆虫标本的获取、收集和出口颁发下列许可证:SISBIO 授权 Nr. 45742-1、45742-7 和 45742-10, CNPq 流程 no 01300.004320/2014-21 和 01300.0013848/2017-33,IBAMA Nr. 14BR016151DF 和 20BR035212/DF)。这项研究得到了德国科学基金会(DFG)研究补助金FL1051/1-1和KA2846/6-1资助。
材料
| Name | Company | Catalog Number | Comments |
| 2,6- Diaminopimelic Acid | Alfa Aesar | B22391 | For E.coli WM3064+ pRL27 |
| Agar - Agar | Roth | 5210 | |
| Agarose | Biozym | 840004 | |
| AMPure beads XP (magentic beads + polyethylene glycol + salts) | Beckman Coulter | A63880 | Size selection in step 6.6 |
| Bleach (NaOCl) 12% | Roth | 9062 | |
| Bowtie2 v.2.4.2 | Bioinfromatic tool for read mapping. Reference 10 in main manuscript. | ||
| Buffer-S | Peqlab | PEQL01-1020 | For PCRs |
| Cell scraper | Sarstedt | 83.1830 | |
| Cutadapt v.2.10 | Bioinformatic tool for removing specific adapter sequences from the reads. Reference 8 in main manuscript. | ||
| DESeq2 | RStudio package for assessing differential mutant abundance. Usually used for RNAseq analysis. Reference 12 in main manuscript. | ||
| DNA ladder 100 bp | Roth | T834.1 | |
| dNTPs | Life Technology | R0182 | PCR for confirming success of conjugation |
| EDTA, Di-Sodium salt | Roth | 8043 | |
| Epicentre MasterPure Complete DNA and RNA Purification Kit | Lucigen | MC85200 | |
| Ethidium bromide | Roth | 2218.1 | |
| FastQC v.0.11.8 | Bioinformatic tool for assessing the quality of sequencing data. Reference 7 in main manuscript. | ||
| FeatureCounts v.2.0.1 | Bioinformatic tool to obtain read counts per genomic feature. Reference 11 in main manuscript. | ||
| Glycerol | Roth | 7530 | |
| K2HPO4 | Roth | P749 | |
| Kanamycin sulfate | Serva | 26899 | |
| KCl | Merck | 4936 | |
| KH2PO4 | Roth | 3904 | |
| MgSO4.7H2O | Roth | PO27 | |
| Na2HPO4 | Roth | P030 | |
| NaCl | Merck | 6404 | |
| NEBNext Multiplex Oligos for Illumina (Index primers set 1) | New England Biolabs | E7335S | |
| NEBNext Ultra II DNA library prep kit for Illumina | New England Biolabs | E7645S | |
| Peptone (soybean) | Roth | 2365 | For Burkholderia gladioli Lv-StA KB-medium |
| peqGOLD 'Hot' Taq- DNA Polymerase | VWR | PEQL01-1020 | PCR for confirming success of conjugation |
| Petri plates - 145 x 20 mm | Roth | XH90.1 | For selecting transconjugants |
| Petri plates - 90 x 16 mm | Roth | N221.2 | |
| Qiaxcel (StarSEQ GmbH, Germany) | Quality check after DNA library preparation | ||
| Streptavidin beads | Roth | HP57.1 | |
| Taq DNA polymerase | VWR | 01-1020 | |
| Trimmomatic v.0.36 | Bioinformatic tool for trimming low quality reads and also adapter sequences. Reference 9 in main manuscript. | ||
| Tris -HCl | Roth | 9090.1 | |
| Tryptone | Roth | 2366 | For Escherichia coli WM3064+pRL27 LB medium |
| Ultrasonicator | Bandelin | GM 70 HD | For shearing |
| USER enzyme (uracil DNA glycosylase + DNA glycosylase- lyase Endonuclease VIII) | New England Biolabs | E7645S | Ligation step 6.5.2 |
| Yeast extract | Roth | 2363 |
参考文献
- Cain, A. K., et al. A decade of advances in transposon-insertion sequencing. Nature Reviews Genetics. 21 (9), 526-540 (2020).
- Chao, M. C., Abel, S., Davis, B. M., Waldor, M. K. The design and analysis of transposon insertion sequencing experiments. Nature Reviews Microbiology. 14 (2), 119-128 (2016).
- Barquist, L., Boinett, C. J., Cain, A. K. Approaches to querying bacterial genomes with transposon-insertion sequencing. RNA Biology. 10 (7), 1161-1169 (2013).
- Flórez, L. V., et al. Antibiotic-producing symbionts dynamically transition between plant pathogenicity and insect-defensive mutualism. Nature Communications. 8 (1), 15172 (2017).
- Flórez, L. V., Kaltenpoth, M. Symbiont dynamics and strain diversity in the defensive mutualism between Lagria beetles and Burkholderia. Environmental Microbiology. 19 (9), 3674-3688 (2017).
- Flórez, L. V., et al. An antifungal polyketide associated with horizontally acquired genes supports symbiont-mediated defense in Lagria villosa beetles. Nature Communications. 9 (1), 2478 (2018).
- . FastQC A quality control tool for high throughput sequence data Available from: https://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (2012)
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet.journal. 17 (1), 10-12 (2011).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: A flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9 (4), 357-359 (2012).
- Liao, Y., Smyth, G. K., Shi, W. FeatureCounts: An efficient general purpose program for assigning sequence reads to genomic features. Bioinformatics. 30 (7), 923-930 (2014).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (12), 550 (2014).
- Gaytán, M. O., Martínez-Santos, V. I., Soto, E., González-Pedrajo, B. Type three secretion system in attaching and effacing pathogens. Frontiers in Cellular and Infection Microbiology. 6, 129 (2016).
- Hachani, A., Wood, T. E., Filloux, A. Type VI secretion and anti-host effectors. Current Opinion in Microbiology. 29, 81-93 (2016).
- Deep, A., Chaudhary, U., Gupta, V. Quorum sensing and bacterial pathogenicity: From molecules to disease. Journal of Laboratory Physicians. 3 (1), 4-11 (2011).
- Silva, A. J., Benitez, J. A. Vibrio cholerae biofilms and cholera pathogenesis. PLOS Neglected Tropical Diseases. 10 (2), 0004330 (2016).
- Navarro-Garcia, F., Ruiz-Perez, F., Cataldi, &. #. 1. 9. 3. ;., Larzábal, M. Type VI secretion system in pathogenic Escherichia coli: structure, role in virulence, and acquisition. Frontiers in Microbiology. 10, 1965 (2019).
- Ribet, D., Cossart, P. How bacterial pathogens colonize their hosts and invade deeper tissues. Microbes and Infection. 17 (3), 173-183 (2015).
- Schwarz, S., et al. Burkholderia Type VI secretion systems have distinct roles in eukaryotic and bacterial cell interactions. PLoS Pathogens. 6 (8), 1001068 (2010).
- Jones, C., et al. Kill and cure: genomic phylogeny and bioactivity of Burkholderia gladioli bacteria capable of pathogenic and beneficial lifestyles. Microbial Genomics. 7 (1), 000515 (2021).
- Takeshita, K., Kikuchi, Y. Riptortuspedestris and Burkholderia symbiont: an ideal model system for insect-microbe symbiotic associations. Research in Microbiology. 168 (3), 175-187 (2017).
- Powell, J. E., et al. Genome-wide screen identifies host colonization determinants in a bacterial gut symbiont. Proceedings of the National Academy of Sciences of the United States of America. 113 (48), 13887-13892 (2016).
- Brooks, J. F., et al. Global discovery of colonization determinants in the squid symbiont Vibrio fischeri. Proceedings of the National Academy of Sciences of the United States of America. 111 (48), 17284-17289 (2014).
- Somprasong, N., McMillan, I., Karkhoff-Schweizer, R. R., Mongkolsuk, S., Schweizer, H. P. Methods for genetic manipulation of Burkholderia gladioli pathovar cocovenenans. BMC Research Notes. 3 (308), (2010).
- Garcia, E. C. Burkholderia thailandensis: Genetic manipulation. Current Protocols in Microbiology. 45, 1-15 (2017).
- Headd, B., Bradford, S. A. The conjugation window in an Escherichia coli K-12 strain with an IncFII plasmid. Applied and Environmental Microbiology. 86 (17), 00948 (2020).
- Van Opijnen, T., Camilli, A. Transposon insertion sequencing: A new tool for systems-level analysis of microorganisms. Nature Reviews Microbiology. 11 (7), 435-442 (2013).
- Gallagher, L. A., Ramage, E., Patrapuvich, R., Weiss, E., Brittnacher, M., Manoil, C. Sequence-defined transposon mutant library of Burkholderia thailandensis. mBio. 4 (6), 00604-00613 (2013).
- Wong, Y. -. C., et al. Candidate essential genes in Burkholderia cenocepacia J2315 identified by genome-wide TraDIS. Frontiers in Microbiology. 7, 1288 (2016).
- Moule, M. G., et al. Genome-wide saturation mutagenesis of Burkholderia pseudomallei K96243 predicts essential genes and novel targets for antimicrobial development. mBio. 5 (1), 00926 (2014).
- Ding, Q., Tan, K. S. Himar1 transposon for efficient random mutagenesis in Aggregatibacter actinomycetemcomitans. Frontiers in Microbiology. 8, 1842 (2017).
- Green, B., Bouchier, C., Fairhead, C., Craig, N. L., Cormack, B. P. Insertion site preference of Mu, Tn5, and Tn7 transposons. Mobile DNA. 3, 3 (2012).
- Lodge, J. K., Weston-Hafer, K., Berg, D. E. Transposon Tn5 target specificity: Preference for insertion at G/C pairs. Genetics. 120 (3), 645-650 (1988).
- Ará Ujo, W. L., et al. Genome sequencing and transposon mutagenesis of Burkholderia seminalis TC3.4.2R3 identify genes contributing to suppression of orchid necrosis caused by B. gladioli. Molecular Plant-microbe Interactions: MPMI. 29 (6), 435-446 (2016).
- Dobritsa, A. P., Samadpour, M. Reclassification of Burkholderiainsecticola as Caballeroniainsecticola comb. nov. and reliability of conserved signature indels as molecular synapomorphies. International Journal of Systematic and Evolutionary Microbiology. 69 (7), 2057-2063 (2019).
- Ohbayashi, T., et al. Insect's intestinal organ for symbiont sorting. Proceedings of the National Academy of Sciences of the United States of America. 112 (37), 5179-5188 (2015).
- Ross, C., Scherlach, K., Kloss, F., Hertweck, C. The molecular basis of conjugated polyyne biosynthesis in phytopathogenic bacteria. Angewandte Chemie International Edition. 53 (30), 7794-7798 (2014).
- Atkins, T., et al. A mutant of Burkholderia pseudomallei, auxotrophic in the branched chain amino acid biosynthetic pathway, is attenuated and protective in a murine model of melioidosis. Infection and Immunity. 70 (9), 5290-5294 (2002).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。