Method Article
ラグリア・ビロサ・カブトムシのバークホルデリア・グラディオリ・シンビエントにおける細菌コロニー形成因子を解明するためのツールとしてのトランスポゾン挿入シーケンシング
要約
これは、 バークホルデリア 有益な共生者における候補昆虫コロニー形成因子を同定するための適応的方法である。このカブトムシ宿主はトランスポゾン変異生成を介して生成されたランダム変異体ライブラリーに感染し、コロニー形成後のライブラリーの複雑さは 、インビトロで増殖した対照と比較される。
要約
遺伝子の活性を操作することによって遺伝子の機能を推測することは、ほとんどの生物学的プロセスの遺伝的基盤を理解するための不可欠なツールです。分子微生物学の進歩により、遺伝子の操作に関する多様な変異発生技術が出現しています。その中でもトランスポゾン挿入シーケンシング(Tn-seq)は、多くの候補遺伝子の機能性を非標的化方法で同時に評価する貴重なツールです。この技術は、いくつかの病原性微生物およびいくつかの有益な共生生物における真核生物宿主の植民地化のための分子メカニズムを同定するための鍵となっている。
ここで、Tn-seqは、カブトムシラグリアビロサの相互主義的なバークホルデリア・グラディオリ共生者における植民地化因子を同定する方法として確立される。結合によって、Tn5トランスポゾン媒介性抗生物質耐性カセットの挿入は、B.グラディオリのランダムゲノム位置で行われる。カブトムシ宿主を植民地化する細菌の能力に対する遺伝子破壊の影響を同定するために、生成されたB.グラディオリトランスポゾン変異ライブラリーはカブトムシの卵に接種され、一方で液体培養培地で体外でコントロールが成長する。コロニー形成に十分な時間を与えた後、DNAはin vivoおよびin vitro成長ライブラリーから抽出される。DNAライブラリー調製プロトコルに従って、DNAサンプルはトランスポゾン挿入シーケンシング用に調製される。トランスポゾン挿入エッジと横向きの細菌DNAを含むDNA断片が選択され、突然変異部位はトランスポゾン挿入エッジから離れてシーケンシングすることによって決定されます。最後に、in vivoとin vitroライブラリの間の各変異体の頻度を分析および比較することによって、カブトムシのコロニー形成中の特異的共生遺伝子の重要性を予測することができる。
概要
バークホルデリア・グラディオリは、ラグリア・ビロサカブトムシと共生関連を持ち、昆虫宿主4、5、6の微生物アンタゴニストに対する防御において重要な役割を果たすことができる。女性のカブトムシは、生殖器系に特殊な腺アクセサリーでB.グラディオリのいくつかの株を収容します。産卵時に、雌はB.グラディオリによって産生される抗菌化合物がエントモ病原性真菌4、6によって感染を阻害する卵表面にB.グラディオリ細胞を塗りつぶす。後期胚発生時または幼虫孵化後の早期に、細菌は幼虫の後部表面に切開部の膣を植民地化する。この特殊な局在化と共生者の垂直伝送経路にもかかわらず、L.絨毛は環境4から水平にB.グラディオリを取得することもできます。さらに、少なくとも3株のB.グラディオリがL.絨毛4,6と関連して発見されている。これらの中で、B.グラディオリLv-StAは、インビトロで栽培するのに適している唯一のものです.
B. グラディオリLv-StAのゲノムサイズは8.56 Mb6で、7,468個の遺伝子が含まれています。B.グラディオリ菌がカブトムシ宿主を植民地化するために重要な遺伝子はどれですか?この質問に答えるために、条件付きで必須の微生物遺伝子1、2、3を同定するための探索的方法であるトランスポゾン挿入シーケンシング(Tn-seq)を用いた。B.グラディオリLv-StAの変異ライブラリーは、Tn5トランスポゾンを用いて作成されました。エシェリヒア・コリドナー細胞からB.グラディオリLv-StAへの結合を通じて、Tn5トランスポゾンを運ぶpRL27プラスミドと、逆繰り返しで横たわる抗生物質耐性カセットを移管した(図1)。それにより、3,736個の共生遺伝子の破壊を個別に運ぶ変異体のセットが生成された(図2)。
変異プールは、コロニー形成因子を同定するためにカブトムシ卵に感染し、対照として、キングスB(KB)培地 でインビトロで 成長した。コロニー形成に十分な時間を確保した後、孵化した幼虫を採取し、DNA抽出のためにプールした。トランスポゾン挿入物を含むDNAの断片と B.グラディオリ Lv-StAの横向きゲノム領域を、シーケンシング用の改変DNAライブラリー調製プロトコルを用いて選択した。読み取り品質処理に続いてDESeq2による分析を行い、B. グラディオリ Lv-StAが卵表面を介して透過したときに L.ビロサ 幼虫を植民地化するために重要な特定の遺伝子を同定するために行われました。
プロトコル
1. メディアおよびバッファの準備
- 表1に示すようにKBおよびLB培地と寒天プレートを準備し、オートクレーブを121°C、15 psi、20分で用意します。
- 50 μg/mL フィルター滅菌カナマイシンと 300 μM フィルター滅菌 2,6-ジアミノピム酸 (DAP) をオートクレーブ LB 培地に加えてから、 大腸菌 WM3064 + pRL27 を培養します。
- 50 μg/mL フィルター滅菌カナマイシンをオートクレーブされたKB寒天に加え、成功した B.グラディオリ Lv-StAトランスコンジュガントの選択に必要なプレートを注ぎます。
- 以下の成分を混合して1xリン酸緩衝生理食塩水(PBS)を調製する:NaCl 8 g/L、KCl 0.201 g/L、Na2HPO4 1.42 g/L、KH2 PO4 0.272 g/L.蒸留水に塩を溶解し、混合物を121°C、15 psi、20 minでオートクレーブします。室温で保管してください。
- 2x の結合洗浄バッファーを次のコンポーネントを溶解して準備します: 10 mM Tris-HCl (pH 7.5)、1 mM エチレンジアミンテトラ酢酸 (EDTA)、および蒸留水に 2 M NaCl.使用前に混合物をフィルター殺菌する。室温で保管してください。
- 10 mM Tris-HCl (pH 8.0) と 0.1 mM EDTA を二重蒸留水に溶解して、1x Low-TE を調製します。121°C、15 psi、20分でオートクレーブすることにより滅菌します。室温で保管してください。
2. トランスポゾン変異ライブラリを生成するコンジュゲーション
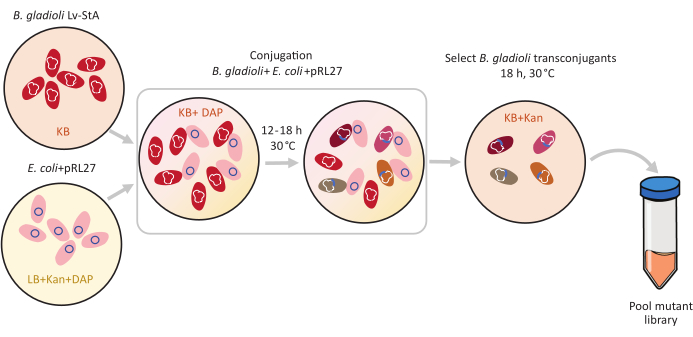
図1: 活用プロトコルの手順 共役の バークホルデリア・グラディオリ ・Lv-StA(赤)とpRL27プラスミド(ピンク)を含むドナー のエシェリヒア・コリ は、それぞれKB寒天およびLBで栽培され、カナマイシンおよびDAPを補う。プラスミドを30°Cで12〜18時間結合した後、トランスコンジュガント B.グラディオリ 細胞をカナマイシンを含むKB上で選択し、一緒にプールする。略語: DAP = 2,6-ジアミノピム酸;カン=カナマイシン。 この図の大きなバージョンを表示するには、ここをクリックしてください。
- 無菌フードの下で、カナマイシンおよびDAPを添加したLB培地の10mLで 、大腸菌 WM3064+pRL27の新鮮なドナー培養を接種する。5 mLのKB培地で バークホルデリア・グラディオリ Lv-StA レシピエント細胞を接種する。250 rpmでシェーカーで一晩30°Cで培養をインキュベートします。
- 一晩増殖した後、各 ×培養 物の4mLを6分間6分間培養した遠心分離機を6分間ペレットする。上清を捨てます。
- 無菌フードの下で、ペレット化された細胞培養物をDAPを含むKB培地で洗浄し、最後に4mLのKB+DAP培地で別々に再懸濁した。
- 新鮮な15 mLチューブに、洗浄された 大腸菌 ドナー細胞の250 μLを、洗浄 されたB.グラディオリ Lv-StAレシピエント細胞の1 mLと混合します。
- DAPを含むKB寒天プレート上のこの共役細胞混合物のスポット10 μL。プレートを室温で1時間放置します。次に、30°Cのコンジュゲーションスポットでプレートを12〜18時間インキュベートする。
注:結合期間は、ターゲット種に応じて調整することができます。しかし、長い結合期間は、ゲノムへの二重挿入またはプラスミドの統合のリスクを高める。増殖が遅い細菌の場合、より長い共役期間を可能にする。 - インキュベーション後、滅菌フードの下のプレートに2〜4mLのPBSを加え、細胞スクレーパーを使用して、成長した細菌結合スポットを寒天から放出します。コンジュゲートセルミックスを2 mLマイクロフュージチューブにピペットします。
- ペレット細胞は、9,600×gで2分間遠心分離して細胞をペレット化した。上清を捨て、1xPBSの1mLでペレットを上下にピペットで2回洗います。最終ペレットを1x PBSの1200 μLで再懸濁します。混合物中の細胞数が1×104を超える場合は、めっき前に希釈を行う。
- 大きなKB寒天プレート(必要に応じて6個以上)に200μLの細胞混合物をよく混ぜて広げ、カナマイシンを補充し、一晩で30°Cでインキュベートします。
注:標的変異コロニーは、選択的寒天プレート上で30時間以内に現れる。抗生物質耐性マーカーのために、変異したコロニーのみが選択的寒天プレートに現れる。したがって、すべてのコロニーは、トランスコンジュガントが成功することが期待されます。 - 3つのプレート上のトランスコンジュガントコロニーの総数を数え、全てのプレートで得られた変異体の概数を計算するために推定する。代表的なライブラリーを得る可能性を高めるには、コロニーの総数がゲノム内の遺伝子の総数よりも数倍高いことを確認してください。結合の成功を確認するために、セクション3に記載されているように、10〜20個のサンプルコロニーを用いて挿入カセットを標的とするPCRを行う。
注:目的は、コロニーの数がゲノム全体(この場合は75,000変異体 >)の遺伝子の数の少なくとも10倍であることを保証することです。しかし、完全に代表的な図書館に対応するコロニーの数を正確に推定することは一般的に困難です。変異したユニークな遺伝子の数は、本質的な遺伝子の破壊が捕捉されないことを考えると、同じ遺伝子に対して複数の異なる突然変異部位が存在することが多く、Tn5トランスポゾンで生成される突然変異は完全にランダムではないことを考えると、現時点では明らかではありません。 - 無菌フードの下で、寒天に1-2 mLの1x PBSを加えることによってプレートからコロニーを削ります。プレートから削り取った細胞混合物を50 mLチューブにプールします。ライブラリを完全に混合し、プールされた変異型ライブラリの4 mLをいくつかのクライオチューブに分割するボルテックス。70%のグリセロールを1mL加え、-80°Cで保存します。
3. B. グラディオリLv-StA での挿入成功を確認する PCR とゲル電気泳動
- 挿入の有無を確認するために、ステップ2.9の選択プレートから個々の変異コロニーを選び、 表2に記載されたプライマーを用いて挿入カセットを標的とするPCRを行う。 表3 に従ってPCRマスターミックスを準備し、サーマルサイクラーに条件を設定します( 表4に記載)。
- PCR産物を電気泳動(250 V、40分)で1.6%アガロースゲルで実行し、増幅されたDNA断片が予想される長さ1580bpであるかどうかを確認します。
4. カブトムシ卵の変異プール感染
- ライブラリの洗浄手順
- 氷上で調製された変異体ライブラリーのアリコートを解凍する。2,683×gで10分間遠心分離機を使用し、上清を取り除く。無菌フードの下で、細胞から残りの培地を取り除くために、4 mLの1x PBSで細胞を洗浄します。細胞を1x PBSの4 mLで再懸濁する。
- 細胞計数チャンバーを使用して、ライブラリのアリコート内のセル数をカウントします。ライブラリの一部を1x PBSで2×10個の6 セル/μLに希釈します。
- 必要なボリュームを取る前にライブラリ全体を均一に混合するためにライブラリアリコートを完全に渦巻く。
- 卵クラッチ滅菌と 生体内 感染
- L.ビロサエッグクラッチを選択します。卵の数を数え、クラッチに100個以上の卵が含まれている場合は続行します。
- 卵クラッチ全体を殺菌します。
- 200 μLの70%エタノールを加え、卵を5分間静かに洗います。エタノールを取り出し、卵をオートクレーブ水で2回洗います。
- 200 μLの 12%漂白剤(NaOCl)を加え、卵を30秒軽く洗います。すぐに漂白剤を取り出し、200μLのオートクレーブ水で卵を再び3回洗います。
- 滅菌された卵クラッチ(卵1個あたり2.5μL)で洗浄された変異体ライブラリの2×10 6細胞/μLに感染します。
- 感染したカブトムシの幼虫の孵化の2日後、1.5 mLマイクロフュージチューブあたり1002nd インスター幼虫を集め、-80°Cで保管します。
-
インビトロ 変異体ライブラリー制御
- 無菌フードの下で、20×の250μLを10個の6 細胞/μLの洗浄された変異体ライブラリーの10mLのKM培地にカナマイシンを含む。
- インビトロ変異培養を30°Cで20時間インキュベートする。
注:植え込み中にインキュベーションの持続時間を計算し、VIVOでWT B.グラディオリLv-StAの世代のおおよその数に一致させます。 - 20時間インキュベーション後、 インビトロ 変異培養液に70%グリセロールの等量を加え、-80°Cで保存する。
5. 感染したカブトムシと インビトロ 変異体ライブラリーDNA抽出
注:DNA抽出は、下記のメーカーのプロトコルに従ってDNAおよびRNA精製キットを使用して行われました。
- 1-2 mLの液体窒素を加え、害虫で粉砕することにより、プールされた幼虫(マイクロフュージチューブあたり最大4mg)を均質化します。
- 氷上のグリセロールストックからインビトロ成長変異培養物を解凍します。細胞リシスの前に9,600×gで遠心分離して10分間ペレットをペレット化した。
- イン ビトロ および インビボ サンプルに300 μLの組織および細胞溶解溶液を加えます。10 mg/mL プロテイナーゼ K 5 μL を加え、60 °C で 15 分間インキュベートし、氷の上に 3~5 分間置きます。
- 150 μLのタンパク質沈殿試薬を、ライゼトと渦に十分に加えます。9,600×gで遠心分離して10分間、タンパク質デブリをペレットにする。
- 上清を1.5 mLマイクロフュージチューブに移します。上清にイソプロパノールの500 μLを加え、1時間または一晩-20°Cでインキュベートする前に、チューブを少なくとも40回穏やかに反転させます。
- 9,600×gで遠心分離により沈殿したDNAを10分間ペレットにした。上清を捨て、氷冷70%エタノールをDNAペレットに加えます。
- 5分間≥10,000×gで遠心分離機。上清を捨て、サンプルを少なくとも1時間空気乾燥させたままにしておきます。
- 100 μLの低TEバッファーで インビトロ および インビボ サンプルからDNAを再懸濁します。
- サンプルは-20°Cで保管してください。
6. シーケンシングライブラリの準備
注:DNAライブラリ調製用のプロトコルおよび試薬は、DNAライブラリ調製キットの製造業者が提供する指示に基いて適合および改変されます。
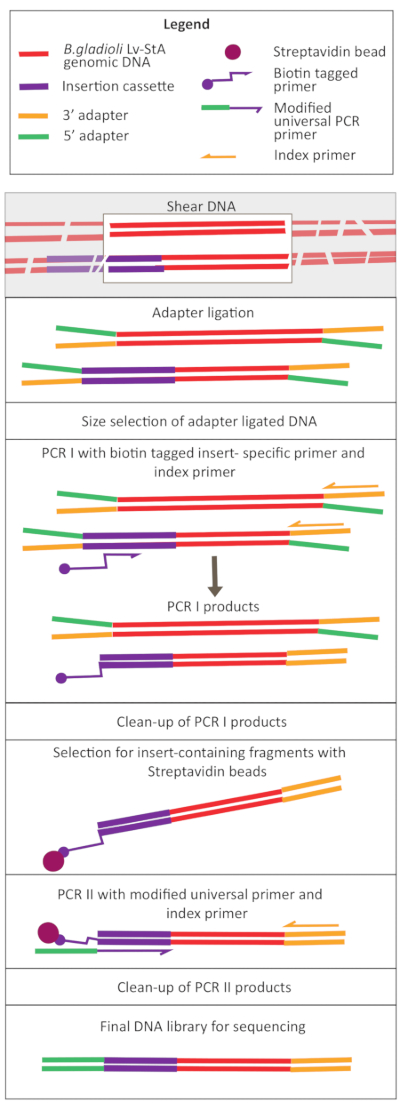
図2:DNAライブラリー調製工程の概略図 剪断およびアダプターのライゲーションの後、修飾されたプロトコルは、挿入カセットを含むDNA断片を濃縮するストレプトアビジンビーズ選択ステップを含む。 この図の大きなバージョンを表示するには、ここをクリックしてください。
- サンプルを20 ng/μL濃度、体積100μLに希釈し、氷の上に保管します。
- 超音波処理器を用いた 生体内 および インビトロ サンプルDNAの剪断。超音波装置を70%の電力に設定します。サンプルを短時間で渦を出し、1分30分間剪断する。
注:超音波装置の設定は、機器によって異なります。この場合、フラグメントサイズは200〜400bpであり、これは150 bpのこのシーケンシングアプローチ、ペアエンド(ステップ9.1を参照)に適している。せん断パラメータは、実験者の要件に応じて調整できます。 - DNAが所望のサイズ範囲(この場合は200-400 bp)までシア化されているかどうかを確認してください。250 Vで40分間、1.6%のアガロースゲルで1:1の比率でゲルローディング色素と混合した後、未シェレおよびシアドDNAの5μLをロードする(図3A、B)。
- アダプターの結紮に必要なフラグメント末端の調製
- 50 μL のせん綿 DNA に、ライブラリー調製キットに記載されているエンド調製用試薬を追加します: 酵素ミックスの 3 μL と反応バッファー 7 μL とピペットによる混合をよく行います。75°C ≥で加熱蓋をしたサーマルサイクラーをセットし、20°Cで30分間、65°Cで30分間インキュベートします。 4 °Cで保持します。
- アダプターライゲーション
- アダプターライゲーションの場合、エンド調製工程の製品に次の試薬を追加します: 30 μL ライゲーションマスターミックス、1 μL ライゲーションエンハンサー、および 2.5 μL 希釈アダプター。ピペットで十分に混合し、加熱された蓋をオフにしてサーマルサイクラーで20°Cで15分間インキュベートします。
- 15分後、酵素(ウラシルDNAグリコシラーゼ+DNAグリコシラーゼ-エンドヌクレアーゼVIII)を3μL添加する( 材料表参照)。ピペットでよく混ぜ、37°Cで15分間、蓋を≥47°Cで加熱したサーマルサイクラーでインキュベートします。
メモ:この手順ではプロトコルを一時停止することができ、サンプルは-20°Cで保存できます。
- 250 bpのアダプター連結DNA標的フラグメントのサイズ選択
- 磁気ビーズ溶液をボルテックスし( 材料表を参照)、室温で30分間使用します。
- 0.3倍のビーズを合体したDNA混合物の96.5 μLに加え、十分にピペットで混ぜます。ビーズ混合物を5分間インキュベートします。
注:ビーズ混合物中の塩およびポリエチレングリコールの存在は、ビーズ上のDNA断片の沈殿を容易にする。DNA分子に対するビーズの比率が低いと、より大きなDNA断片とビーズへの結合のみが可能になります。この場合、長さが250bpを超えるDNA断片はビーズに結合する。 - チューブを磁気スタンドに置いてビーズを引き下げ、不要なサイズのDNA断片を取り除きます。ビーズを5分間落ち着かせ、透明な上清を新しいマイクロフュージチューブに移します(上清を保ちます)。
- 上清に0.15倍の新鮮なビーズを加え、よくピペットで混ぜます。ビード混合物を5分間インキュベートし、チューブを磁気スタンドに置き、標的DNAに結合したビーズを引き下げます。5分間待ってから上清を捨てます(ビーズを保管してください)。
注:DNAに対するビーズのこの比率は、所望の250 bpサイズの断片の結合につながります。 - 磁気スタンドにビーズを付けて、200 μLの80%エタノール(作りたての)を加え、30秒待ちます。ピペットアウトし、磁気スタンドのビーズを邪魔することなく、慎重にエタノール洗浄を廃棄します。この手順を繰り返します。
- 最後の洗浄後、ビーズからエタノールの痕跡を取り除き、光沢があるように見えるが完全に乾燥しないまで2分間ビーズを空気乾燥させます。ビーズを過度に乾燥させないでください。
- 磁気スタンドからチューブを取り外し、10 mM Tris-HClまたは0.1x TE(ローテア)の17 μLを追加します。~10回ピペットで混ぜ、室温で2分間インキュベートします。
- チューブを磁気スタンドに戻し、5分間待ちます。ビーズが落ち着いたら、DNA上清を新しいチューブに移します。
- PCR I は、挿入カセットを含む DNA フラグメントにビオチン タグを追加します。
- トランスポゾン特異的ビオチン化プライマー(表5)とインデックスプライマーを使用して、Tn5挿入カセットを含むDNA断片にビオチン化プライマータグを追加します。 表 6 に従って PCR マスター ミックスを準備し、 表 7に示すサーマル サイクラーの PCR 条件に従ってください。
- サイズ選択なし PCR I のクリーンアップ
- 渦0.9xビーズを室温で30分以上置いてからクリーンアップします。
- PCR製品に0.9xビーズを加え、十分に混ぜます。
- ビーズを磁気スタンドに置き、ビーズを引き下げます。
- 透明な上清を取り除き、作りたての80%エタノールを2回2回200μLでビーズ結合DNAを洗います。
- 洗浄段階の後にエタノールを取り出し、光沢があるように見えるが乾燥しすぎないまでビーズを空気乾燥させます。
- 32 μLの 10 mM Tris-HCl または 0.1X TE (Low-TE) を加え、5 分間インキュベートします。マグネットスタンドに戻って混合物を置き、上清を新鮮なミクロフュージチューブに移します。
- ビオチン化されたDNA断片をストレプトアビジンビーズに結合する
- ストレプトアビジンビーズの32 μLを1xのバインド・アンド・ウォッシュバッファーに再懸濁します。磁気スタンドに置いた状態で、ビーズをバッファーで3回洗います。
- 2x バインド アンド ウォッシュ バッファの 32 μL を追加し、ビーズを再中断します。これに、クリーンアップ PCR 1 製品の 32 μL を追加します。完全に混ぜ合わせ、室温で30分間インキュベートします。
- ビーズとDNAの混合物を磁気スタンドに2分間置きます。ピペットは、挿入エッジを含むビオチンタグ付きDNAとして上清を出し、ビーズ上のストレプトアビジンに結合する。
- 500 μLの 1x バインド アンド ウォッシュ バッファでビーズを洗浄し、200 μL の Low-TE でビーズを洗います。DNA結合ビーズを17μLの低TEで再懸濁します。
- 挿入カセットエッジを含むフラグメントにアダプターを追加するPCR II
- 表 8に示すように、表5に示すインデックスプライマーおよび修飾ユニバーサル PCR プライマーを使用してマスター ミックスを準備します。前のステップから15μLのDNA結合ストレプトアビジンビーズをPCRミックスに添加します。サーマルサイクラーの状態については、表7を参照してください。
- このプロトコルのステップ6.8で示されているように、サイズを選択せずにPCR製品をクリーンアップします。最終的なDNA製品を分子グレードの水の30 μLで溶出します。
- サンプルを-20°Cに保存し、シーケンシングに使用してください。
7. シーケンシングと分析
- ハイスループットシーケンス技術を使用してライブラリをシーケンスします。以下に示すように、トランスポゾン ライブラリのサイズに応じてシーケンスの深さを調整します。FastQC7で読み取り品質を評価します。読み取りの 5' の終わりに Tn5 挿入エッジを含む読み取りを選択し、Cutadapt8 および/または Trimmomatic9を使用して挿入エッジシーケンスを削除します。
注: ここでは、ペアエンドのシーケンシングアプローチを使用して、読み取りあたり 150 bp をターゲットにし、合計で 8 Mio 読み取りが行われます。代表的なデータセットを取得するには、シーケンス読み取りの総数がライブラリ内の突然変異体の最大数(すなわち、ステップ2.9からのコロニーの合計推定数)を超えることを確認する。参考として、このプロトコルは、可能な最大ライブラリサイズの40倍を目指しました。同様の目的のためにTn-seqを用いた他の成功した研究は、対応する変異ライブラリー22,23における実際のユニーク挿入数の25倍近くの読み取りの総数を配列した。 - 遺伝子の末端の突然変異が機能的に破壊的ではないことを考慮して、参照ゲノムGFFファイルの遺伝子注釈の両端から5%をトリミングする。Bowtie210を使用して、トリミングされた読み取りを参照ゲノムにマッピングします。
- 配置 BAM ファイル内の一意の 5' 位置の数から挿入の数を計算します。
- FeatureCounts11を使用して、各反復サンプルのヒット遺伝子の数を取得します。
- RStudio の DESeq212 パッケージを使用して、異なる条件間の変異量の差を計算します。
結果
宿主関連細菌は、接着性、運動性、化学軸、ストレス応答、または特定のトランスポーターを媒介するものを含む、関連を確立するためにいくつかの要因を採用することができる。いくつかの細菌13、14、14、15、16、17、18に重要な因子が報告されているが、バークホルデリア属のメンバーを含む19、20、より少ない研究は、植民地化のための有益な共生者によって使用される分子メカニズムを探求している21、22、23 .トランスポゾン挿入シーケンシングを使用して、B.グラディオリがL.ビロサカブトムシを植民地化することを可能にする分子因子を同定することが目的であった。
トランスポゾン媒介性変異誘発は、転写反復部位に隣接するTn5トランスポゾンおよびカナマイシン耐性カセットを運ぶpRL27プラスミドを用いて行った。プラスミドを、プラスミドドナー大腸菌WM3064株との共役によって標的B.グラディオリLv-StA細胞に導入した(図1に示すように)。共役後、B.グラディオリレシピエントおよび大腸菌ドナー細胞を含む結合体混合物を、カナマイシンを含む選択的寒天プレート上にメッキした。プレート上のDAPの不在は、ドナー大腸菌細胞を排除し、成功したB.グラディオリLv-StAトランスコンジュガントのために選択されたカナマイシンの存在。100,000個のトランスコンジュガントコロニーを収穫した結果得られたプールされたB.グラディオリLv-StA変異体ライブラリーを、改変DNAライブラリー調製キットおよびカスタムプライマーを用いてシーケンシング用に調製した。図2は、DNAライブラリの作成手順を示しています。シーケンスは、4 Mio ペアの読み取りを生成しました。B.グラディオリLv-StAの7,468遺伝子のうち3,736個の遺伝子が破壊された。
宿主におけるコロニー形成欠陥があった変異体を同定するために 、B.グラディオリ Lv-StA変異体ライブラリーをカブトムシの卵に感染させ、コントロールとしてKB培地 でインビトロで 増殖した。イン ビボ コロニー形成ボトルネックサイズは実験前に計算した。既知の数の B.グラディオリ Lv-StA細胞がカブトムシの卵に感染し、各幼虫から懸濁液をメッキし、個体ごとにコロニー形成ユニットを数えることによって、最初に孵化した新しいインスター幼虫中のコロニー形成細胞の数を得た。これらの計算は、コロニー形成細胞の数が、宿主を植民地化する能力についてライブラリー内の変異体のすべてまたは高い割合を評価するのに十分であることを保証するために行われた。さらに 、in vitro と in vivo 条件の間の成長時間は、これらのサンプルを比較可能にするために細菌世代数に基づいて正規化された。
卵が孵化した後、1,296頭の幼虫が13のプールに集められました。対応する インビトロ 変異培養物を成長させ、グリセロールストックとして保存した。 in vivo およびin vitro 成長変異体ライブラリーのDNAを超音波処理器で抽出し断片化した。 図3 は、断片の大部分が予想通り100~400bpの範囲に及ぶ、せせられたDNAのサイズ分布を示しています。このステップの後に、シーケンスのための修飾されたDNAライブラリー調製プロトコルが続いた。プロトコルの各ステップで、残りのDNAの濃度をチェックして、ステップが正しく実行されたことを確認し、DNAの損失を追跡しました。シーケンシング前の品質チェック( 材料表を参照)により、DNAライブラリに予期せず大きな(>800 bp)DNA断片が含まれていることが明らかになっており、in vivoライブラリでは より顕著であった。シーケンスレーン内のフラグメントのクラスタリングを最適化するのが難しいため、読み取りの所望の数を達成するために 、in vivo ライブラリで10 Mioペアの読み取りにシーケンス深度を大きくする必要がありました。シーケンシング結果の分析結果から 、in vivo ライブラリ内の平均4 Mio読み取りと、in vitro ライブラリでの3.1 Mioの読み取りには、この実験に満足できるRead-1の5'末端(表9)にトランスポゾンエッジが含まれていることが明らかになった。元のライブラリーにおける B.グラディオリ ゲノム全体の24,224個のユニークな挿入の分布を 図4に示します。DESeq2を用いて行った分析により、271個の変異体の存在量が 生体内 と インビトロ の条件の間で有意に異なっていたことが明らかになった。
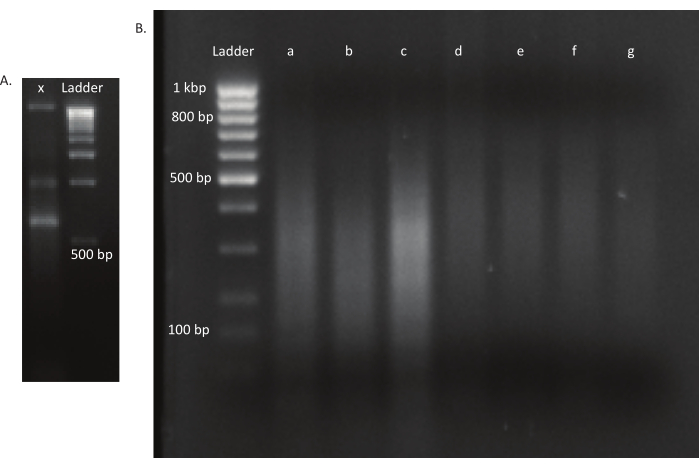
図3:変異体およびDNAライブラリーのアガロースゲル(A)車線xの突然変異体の未シェアDNAとスケール用1kbpのラダーを有するアガロースゲル。(B) シーアド DNA ライブラリーを用いたゲル化。最初の車線のラダーのバンドサイズは左側に示されています。最初の3レーンa、b、およびcには、in vivoライブラリのシアーDNA断片が含まれています。lanes d,e,f,gは、インビトロライブラリーのシアDNA断片を含む。この図の大きなバージョンを表示するには、ここをクリックしてください。
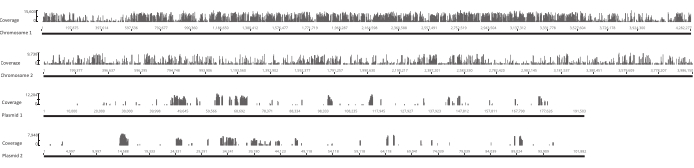
図4:バークホルデリア・グラディオリLv-StAゲノム内の4つのレパコンの元のライブラリ内のユニークな挿入部位の位置X 軸に沿った各バーは、挿入のサイトにあります。Y 軸に沿ったバーの高さは、そのサイトに関連付けられた読み取りの数に対応します。なお、2つの染色体と2つのプラスミドは全長で示され、X軸上で異なるスケールを有していることに注意してください。この図の大きなバージョンを表示するには、ここをクリックしてください。
| キングスBミディアム/寒天 | |
| ペプトン(大豆) | 20 g/L |
| K2HPO4 | 1.5 g/L |
| MgSO4.7H2O | 1.5 g/L |
| 寒天 | 15 g/L |
| 蒸留水に溶解 | |
| LBミディアム/寒天 | |
| トリプトン | 10 g/L |
| 酵母エキス | 5 g/L |
| ナクル | 10 g/L |
| 蒸留水に溶解 | |
表1:メディアコンポーネント
| いいえ。 | プライマー | 順序 | PCR アニール温度(°C) | |
| 1 | tpnRL17-1RC | 5'-CGTTACATCCCTGGCTGTT-3' | 58.2 | |
| 2 | tpnRL13-2RC | 5'-TCGTGAGAAGGTGtTG-3' | ||
表2:結合の成功を確認するプライマー。
| コンポーネント | ボリューム(μL) |
| HPLC精製水 | 4.92 |
| 10x バッファ S (特異性が高い) | 1 |
| MgCl2 (25 mM) | 0.2 |
| dMP (2 mM) | 1.2 |
| プライマー 1 (10 pmol/μL) | 0.8 |
| プライマー2(10 pmol/μL) | 0.8 |
| タク (5 U/μL) | 0.08 |
| マスターミックス合計 | 9 |
| テンプレート | 1 |
表3:結合の成功を確認するPCRマスターミックス。 略語: HPLC = 高速液体クロマトグラフィー;dNTPs = デオキシヌクレオシド三リン酸.
| ステップス | 温度 °C | 時間 | サイクル |
| 初期変性 | 95 | 3分 | 1 |
| 変性 | 95 | 40 s | |
| アニーリング | 58.2 | 40 s | 30~35 |
| 延長 | 72 | 1-2分 | |
| 最終拡張 | 72 | 4分 | 1 |
| 持つ | 4 | ∞ | |
表4:結合の成功を確認するPCR条件。
| プライマー | 順序 | Tm °C | 使う | 源 | |
| トランスポゾン特異的ビオチン化プライマー | 5'-ビオチン-アカガアカッチャッハックガキャット -3' | 63.5 | 6.7.1. PCR I | 習慣 | |
| 改変ユニバーサルPCRプライマー | 5'- アトガタックグッカッカッカガガタック タクトクトクトタッハタチャッカクガクト TTCCGATCTGAATTCATCGATATATAT GGTTガガットGT – 3 ' | 62 | 6.10.1. PCR II | 習慣 | |
| インデックスプライマー | 製造元のマニュアルを参照してください。 | 6.7.1. PCR I および 6.10.1.PCR II | NEBNext イルミナ用マルチプレックスオリゴス(インデックスプライマーセット1) | ||
| アダプタ | 製造元のマニュアルを参照してください。 | 6.5. アダプターライゲーション | NEB次のウルトラII DNAライブラリのイルミナ用準備キット | ||
表5:DNAライブラリー調製中のPCR IおよびII用プライマーおよびアダプター。
| PCR ミックス | (μL) |
| アダプター・リガイテッド DNA フラグメント | 15 |
| ネブネクスト ウルトラII Q5 マスターミックス | 25 |
| インデックスプライマー(10 pmol/μL) | 5 |
| トランスポゾン特異的ビオチン化プライマー(10 pmol/μL) | 5 |
| 総量 | 50 |
表 6: DNAライブラリー調製-PCR I マスターミックス
| ステップス | 温度 | 時間 | サイクル |
| 初期変性 | 98°C | 30 s | 1 |
| 変性 | 98°C | 10 s | 6~12 |
| アニーリング | 65°C | 30 s | |
| 延長 | 72°C | 30 s | |
| 最終拡張 | 72°C | 2分 | 1 |
| 持つ | 16°C | ∞ | |
表7:DNAライブラリー調製-PCRIおよびII条件
| PCR ミックス | (μL) |
| ビーズ選択DNA | 15 |
| ネブネクスト ウルトラII Q5 マスターミックス | 25 |
| インデックスプライマー | 5 |
| 改変ユニバーサル PCR プライマー | 5 |
| 総量 | 50 |
表 8: DNA ライブラリー調製 -PCR II マスター ミックス
| ライブラリ | イヴィーボ-1 | イヴィーボ-2 | インビボ-3 | インビトロ-1 | インビトロ-2 | インビトロ-3 | 元のライブラリ | |
| いいえ。読み取りの (PE) | 56,57,710 | 39,19,051 | 30,65,849 | 35,73,494 | 28,83,440 | 36,61,956 | 46,09,410 | |
| いいえ。読み取り-1の5'の終わりのTn - エッジを含む読み取りの | 54,15,880 | 37,31,169 | 29,36,247 | 33,00,499 | 27,35,705 | 33,50,402 | 41,53,270 | |
| ボウタイ2全体のアライメント率(%)(読み取り-1のみ) | 95.53% | 83.71% | 89.87% | 80.79% | 78.00% | 73.06% | 74.92% | |
| 一意の挿入の数 | 8,539 | 4,134 | 7,183 | 18,930 | 18,421 | 20,438 | 24,224 | |
| ヒットした遺伝子の数 | 1575 | 993 | 1450 | 2793 | 2597 | 3037 | 3736 | |
表9:ライブラリごとのシーケンス出力とトランスポゾン挿入周波数の概要 略語: PE = ペアエンド。
ディスカッション
B.グラディオリトランスポゾン変異ライブラリーを生成し、L.ビロサカブトムシとB.グラディオリ菌との共生相互作用における重要な宿主コロニー形成因子を同定した。プロトコルの主なステップは、結合、宿主感染、DNAライブラリ調製、およびシーケンシングであった。
バークホルデリアの多くの株が共役24、25によって遺伝子改変に適しているように、トランスポゾンおよび抗生物質挿入カセットを運ぶプラスミドは、大腸菌から標的B.グラディオリLv-StA株に正常に共役した。エレクトロポレーションによる変換の以前の試みは、ほぼないB.グラディオリ形質転換体に非常に低いをもたらしました。多数の形質転換体を効率よく生成するために、ターゲット生物の変換技術を最適化することをお勧めします。
1ラウンドの結合と40の結合点は 、B.グラディオリ Lv-StAの3,736個の遺伝子を破壊した。後から考えると、7,468個の遺伝子の大部分を破壊し、飽和ライブラリを得るためには、複数の結合が必要です。特に、結合時のインキュベーション時間は 、B.グラディオリの指数成長期の終わりである12-18時間を超えることは許されなかった。細菌細胞の指数関数的増殖期を超えて結合を可能にすることは、トランスコンジュガント26を得る成功の可能性を減少させる。したがって、結合期間は、細菌種の増殖に応じて調整されるべきである。
宿主における変異体ライブラリーの感染を含む実験を成功させるためには、コロニー形成中の細菌集団のボトルネックサイズおよび感染前のライブラリー内の変異体の多様性を評価することが重要である1、2、27。実験に備えて、感染しなければならないカブトムシの最小数は、ライブラリ内の各変異体がサンプリングされ、植民地化される可能性が高いと推定した。また、実験期間中の生体内細菌発生時間および世代数の概算も計算した。その後、インキュベーション時間を調整することで、インビトロ培養は同等の数の世代に成長しました。他の非モデル宿主における同様の感染実験では、宿主生物の実験室培養および一定の供給源を維持する能力が望ましい。
in vivoおよびin vitroおよびサンプル収集における変異ライブラリーの成長に続いて、トランスポゾン挿入シーケンシングのための改変DNAライブラリー調製プロトコルが実施された。このプロトコルの変更には、カスタム PCR プライマーの設計と、挿入カセットを含む DNA フラグメントを選択する PCR ステップの追加が含まれます。プロトコルがカスタマイズされたため、プロトコルの追加の PCR サイクルにより、オーバーアンプのリスクが高まり、エンド ライブラリ内のハイブリッド化されたアダプター アダプター フラグメントが取得されます。したがって、これらのフラグメントの除去に役立つため、2 つの PCB の後に(サイズを選択せずに)最終的なクリーンアップ手順を推奨します。DNAライブラリのサイズ分布は、予想以上に広かった。しかし、シーケンシング深度を大きくすると、バイオインフォマティクス解析中にフィルタリングされた十分なデータが得られ、満足のいく結果が得られた。
トランスポゾン媒介性変異誘発は、1回の実験で何千ものランダム挿入を生成するので、細菌の増殖に不可欠な遺伝子が破壊された変異体を除く全ての変異体を含む、飽和した変異体のライブラリーを生成することができる。バークホルデリアspに関する他の研究における必須遺伝子の推定を考えると、飽和変異体ライブラリでは機能しなかった可能性が最も高い。28、29.それにもかかわらず、非飽和ライブラリーは、標的突然変異誘発を用いたさらなる研究のための様々な候補遺伝子の探索に役立つ。実験の前に、いくつかのトランスポソンは、ゲノム30の特定の場所で変異体の存在量を増加させる特異的挿入標的部位を有することを覚えておくことも重要である。マリナートランスポゾンは、挿入用のAT部位を標的とすることが知られており、Tn5トランスポソンはGCバイアス32,33を有する。トランスポゾン挿入のホットスポットを認識するバイオインフォマティクス分析中のステップを含めると、分布バイアスの評価に役立ちます。
挫折を起こしやすいが、適切に設計されたトランスポゾン挿入シーケンシング実験は、単一の実験で細菌の多くの条件付きで重要な遺伝子を同定するための強力なツールであり得る。例えば、蘭葉壊死の抑制に重要なバークホルデリアの十数個の遺伝子をトランスポゾン変異誘発とゲノミクス34と組み合わせることにより同定した。バークホルデリアを超えて、いくつかの接着および運動性遺伝子およびトランスポーターは、アピス・メリケラ(ハニービー)22のスノドグラセラ・アルヴィ共生生物と、エウプリムナ・スコロペス(ハワイ・ボブ・スクイド)23の重要なコロニー形成因子として同定されている。
別のアプローチとして、トランスポゾン変異誘発は、シーケンシングの代わりに選択的培地を用いる個々の変異体のスクリーニングを続けてもよい。運動性、生理活性二次代謝物の産生、または特定のオーソトロフィーなどの欠乏を特定するための表現型スクリーニングまたはバイオアッセイは、実現可能である。例えば、 バークホルデリア昆虫菌 ( カバレロニア属に再割り当てされた)トランスポゾン変異ライブラリーのスクリーニングは、共生者が リプトルトゥス・ペデストリスを植民地化するための運動性遺伝子を採用していることを同定する上で重要であった。さらに、トランスポゾン変異誘発および表皮スクリーニングを用いて、生物活性二次代謝産物カルヨイネンシンの生合成遺伝子クラスターを 、バークホルデリア・カリョフィリ37において同定した。プセポゾン変異誘発およびスクリーニングに続いて 、バークホルデリア・シュードマレイ の栄養変異体が同定され、ヒトおよび動物38における危険な疾患であるメリオイドーシスに対して可能な減衰ワクチン候補である。したがって、トランスポゾン変異生成とシーケンシングは、病原性または相互的な関連においてそれぞれの宿主との相互作用にとって重要な細菌の分子形質を研究する上で貴重なアプローチである。
開示事項
著者らは、研究に関する利益相反はないと宣言している。
謝辞
手順の共役と指導のための 大腸菌 WM3064 +pRL27株を提供してくれたJunbeom Lee、ミュータントライブラリ生成中のトラブルシューティングを支援してくれたキャスリン・ヒュフマイヤー、昆虫の収集と許可の取得を支援してくれたアンドレ・ロドリゲス教授に感謝しています。また、レベッカ・ジャンケとダグマール・クレブシュが昆虫の収集と飼育を支援してくれたことに感謝します。我々は、ブラジル当局が、昆虫標本のアクセス、収集及び輸出に関する以下の許可を与えることを認める: SISBIO認可Nr。 45742-1、45742-7および45742-10、CNPqプロセスnº 01300.004320/2014-21および01300.0013848/2017-33、IBAMA Nr. 14BR016151および2033DF)この研究は、ドイツ科学財団(DFG)研究助成金FL1051/1-1およびKA2846/6-1からの資金提供によって支えられました。
資料
| Name | Company | Catalog Number | Comments |
| 2,6- Diaminopimelic Acid | Alfa Aesar | B22391 | For E.coli WM3064+ pRL27 |
| Agar - Agar | Roth | 5210 | |
| Agarose | Biozym | 840004 | |
| AMPure beads XP (magentic beads + polyethylene glycol + salts) | Beckman Coulter | A63880 | Size selection in step 6.6 |
| Bleach (NaOCl) 12% | Roth | 9062 | |
| Bowtie2 v.2.4.2 | Bioinfromatic tool for read mapping. Reference 10 in main manuscript. | ||
| Buffer-S | Peqlab | PEQL01-1020 | For PCRs |
| Cell scraper | Sarstedt | 83.1830 | |
| Cutadapt v.2.10 | Bioinformatic tool for removing specific adapter sequences from the reads. Reference 8 in main manuscript. | ||
| DESeq2 | RStudio package for assessing differential mutant abundance. Usually used for RNAseq analysis. Reference 12 in main manuscript. | ||
| DNA ladder 100 bp | Roth | T834.1 | |
| dNTPs | Life Technology | R0182 | PCR for confirming success of conjugation |
| EDTA, Di-Sodium salt | Roth | 8043 | |
| Epicentre MasterPure Complete DNA and RNA Purification Kit | Lucigen | MC85200 | |
| Ethidium bromide | Roth | 2218.1 | |
| FastQC v.0.11.8 | Bioinformatic tool for assessing the quality of sequencing data. Reference 7 in main manuscript. | ||
| FeatureCounts v.2.0.1 | Bioinformatic tool to obtain read counts per genomic feature. Reference 11 in main manuscript. | ||
| Glycerol | Roth | 7530 | |
| K2HPO4 | Roth | P749 | |
| Kanamycin sulfate | Serva | 26899 | |
| KCl | Merck | 4936 | |
| KH2PO4 | Roth | 3904 | |
| MgSO4.7H2O | Roth | PO27 | |
| Na2HPO4 | Roth | P030 | |
| NaCl | Merck | 6404 | |
| NEBNext Multiplex Oligos for Illumina (Index primers set 1) | New England Biolabs | E7335S | |
| NEBNext Ultra II DNA library prep kit for Illumina | New England Biolabs | E7645S | |
| Peptone (soybean) | Roth | 2365 | For Burkholderia gladioli Lv-StA KB-medium |
| peqGOLD 'Hot' Taq- DNA Polymerase | VWR | PEQL01-1020 | PCR for confirming success of conjugation |
| Petri plates - 145 x 20 mm | Roth | XH90.1 | For selecting transconjugants |
| Petri plates - 90 x 16 mm | Roth | N221.2 | |
| Qiaxcel (StarSEQ GmbH, Germany) | Quality check after DNA library preparation | ||
| Streptavidin beads | Roth | HP57.1 | |
| Taq DNA polymerase | VWR | 01-1020 | |
| Trimmomatic v.0.36 | Bioinformatic tool for trimming low quality reads and also adapter sequences. Reference 9 in main manuscript. | ||
| Tris -HCl | Roth | 9090.1 | |
| Tryptone | Roth | 2366 | For Escherichia coli WM3064+pRL27 LB medium |
| Ultrasonicator | Bandelin | GM 70 HD | For shearing |
| USER enzyme (uracil DNA glycosylase + DNA glycosylase- lyase Endonuclease VIII) | New England Biolabs | E7645S | Ligation step 6.5.2 |
| Yeast extract | Roth | 2363 |
参考文献
- Cain, A. K., et al. A decade of advances in transposon-insertion sequencing. Nature Reviews Genetics. 21 (9), 526-540 (2020).
- Chao, M. C., Abel, S., Davis, B. M., Waldor, M. K. The design and analysis of transposon insertion sequencing experiments. Nature Reviews Microbiology. 14 (2), 119-128 (2016).
- Barquist, L., Boinett, C. J., Cain, A. K. Approaches to querying bacterial genomes with transposon-insertion sequencing. RNA Biology. 10 (7), 1161-1169 (2013).
- Flórez, L. V., et al. Antibiotic-producing symbionts dynamically transition between plant pathogenicity and insect-defensive mutualism. Nature Communications. 8 (1), 15172(2017).
- Flórez, L. V., Kaltenpoth, M. Symbiont dynamics and strain diversity in the defensive mutualism between Lagria beetles and Burkholderia. Environmental Microbiology. 19 (9), 3674-3688 (2017).
- Flórez, L. V., et al. An antifungal polyketide associated with horizontally acquired genes supports symbiont-mediated defense in Lagria villosa beetles. Nature Communications. 9 (1), 2478(2018).
- Andrews, S. FastQC A quality control tool for high throughput sequence data. , Available from: https://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (2012).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet.journal. 17 (1), 10-12 (2011).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: A flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9 (4), 357-359 (2012).
- Liao, Y., Smyth, G. K., Shi, W. FeatureCounts: An efficient general purpose program for assigning sequence reads to genomic features. Bioinformatics. 30 (7), 923-930 (2014).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (12), 550(2014).
- Gaytán, M. O., Martínez-Santos, V. I., Soto, E., González-Pedrajo, B. Type three secretion system in attaching and effacing pathogens. Frontiers in Cellular and Infection Microbiology. 6, 129(2016).
- Hachani, A., Wood, T. E., Filloux, A. Type VI secretion and anti-host effectors. Current Opinion in Microbiology. 29, 81-93 (2016).
- Deep, A., Chaudhary, U., Gupta, V. Quorum sensing and bacterial pathogenicity: From molecules to disease. Journal of Laboratory Physicians. 3 (1), 4-11 (2011).
- Silva, A. J., Benitez, J. A. Vibrio cholerae biofilms and cholera pathogenesis. PLOS Neglected Tropical Diseases. 10 (2), 0004330(2016).
- Navarro-Garcia, F., Ruiz-Perez, F., Cataldi, Á, Larzábal, M. Type VI secretion system in pathogenic Escherichia coli: structure, role in virulence, and acquisition. Frontiers in Microbiology. 10, 1965(2019).
- Ribet, D., Cossart, P. How bacterial pathogens colonize their hosts and invade deeper tissues. Microbes and Infection. 17 (3), 173-183 (2015).
- Schwarz, S., et al. Burkholderia Type VI secretion systems have distinct roles in eukaryotic and bacterial cell interactions. PLoS Pathogens. 6 (8), 1001068(2010).
- Jones, C., et al. Kill and cure: genomic phylogeny and bioactivity of Burkholderia gladioli bacteria capable of pathogenic and beneficial lifestyles. Microbial Genomics. 7 (1), 000515(2021).
- Takeshita, K., Kikuchi, Y. Riptortuspedestris and Burkholderia symbiont: an ideal model system for insect-microbe symbiotic associations. Research in Microbiology. 168 (3), 175-187 (2017).
- Powell, J. E., et al. Genome-wide screen identifies host colonization determinants in a bacterial gut symbiont. Proceedings of the National Academy of Sciences of the United States of America. 113 (48), 13887-13892 (2016).
- Brooks, J. F., et al. Global discovery of colonization determinants in the squid symbiont Vibrio fischeri. Proceedings of the National Academy of Sciences of the United States of America. 111 (48), 17284-17289 (2014).
- Somprasong, N., McMillan, I., Karkhoff-Schweizer, R. R., Mongkolsuk, S., Schweizer, H. P. Methods for genetic manipulation of Burkholderia gladioli pathovar cocovenenans. BMC Research Notes. 3 (308), (2010).
- Garcia, E. C. Burkholderia thailandensis: Genetic manipulation. Current Protocols in Microbiology. 45, 1-15 (2017).
- Headd, B., Bradford, S. A. The conjugation window in an Escherichia coli K-12 strain with an IncFII plasmid. Applied and Environmental Microbiology. 86 (17), 00948(2020).
- Van Opijnen, T., Camilli, A. Transposon insertion sequencing: A new tool for systems-level analysis of microorganisms. Nature Reviews Microbiology. 11 (7), 435-442 (2013).
- Gallagher, L. A., Ramage, E., Patrapuvich, R., Weiss, E., Brittnacher, M., Manoil, C. Sequence-defined transposon mutant library of Burkholderia thailandensis. mBio. 4 (6), 00604-00613 (2013).
- Wong, Y. -C., et al. Candidate essential genes in Burkholderia cenocepacia J2315 identified by genome-wide TraDIS. Frontiers in Microbiology. 7, 1288(2016).
- Moule, M. G., et al. Genome-wide saturation mutagenesis of Burkholderia pseudomallei K96243 predicts essential genes and novel targets for antimicrobial development. mBio. 5 (1), 00926(2014).
- Ding, Q., Tan, K. S. Himar1 transposon for efficient random mutagenesis in Aggregatibacter actinomycetemcomitans. Frontiers in Microbiology. 8, 1842(2017).
- Green, B., Bouchier, C., Fairhead, C., Craig, N. L., Cormack, B. P. Insertion site preference of Mu, Tn5, and Tn7 transposons. Mobile DNA. 3, 3(2012).
- Lodge, J. K., Weston-Hafer, K., Berg, D. E. Transposon Tn5 target specificity: Preference for insertion at G/C pairs. Genetics. 120 (3), 645-650 (1988).
- Ará Ujo, W. L., et al. Genome sequencing and transposon mutagenesis of Burkholderia seminalis TC3.4.2R3 identify genes contributing to suppression of orchid necrosis caused by B. gladioli. Molecular Plant-microbe Interactions: MPMI. 29 (6), 435-446 (2016).
- Dobritsa, A. P., Samadpour, M. Reclassification of Burkholderiainsecticola as Caballeroniainsecticola comb. nov. and reliability of conserved signature indels as molecular synapomorphies. International Journal of Systematic and Evolutionary Microbiology. 69 (7), 2057-2063 (2019).
- Ohbayashi, T., et al. Insect's intestinal organ for symbiont sorting. Proceedings of the National Academy of Sciences of the United States of America. 112 (37), 5179-5188 (2015).
- Ross, C., Scherlach, K., Kloss, F., Hertweck, C. The molecular basis of conjugated polyyne biosynthesis in phytopathogenic bacteria. Angewandte Chemie International Edition. 53 (30), 7794-7798 (2014).
- Atkins, T., et al. A mutant of Burkholderia pseudomallei, auxotrophic in the branched chain amino acid biosynthetic pathway, is attenuated and protective in a murine model of melioidosis. Infection and Immunity. 70 (9), 5290-5294 (2002).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved