需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
基于结构的转录因子蛋白沿DNA从原子级步进到粗晶扩散运动的模拟和采样
* 这些作者具有相同的贡献
摘要
该协议的目标是揭示蛋白质沿DNA的一维扩散的结构动力学,使用植物转录因子WRKY结构域蛋白作为示例系统。为此,已经实施了原子和粗粒度分子动力学模拟以及广泛的计算采样。
摘要
转录因子(TF)蛋白沿DNA的一维(1-D)滑动对于促进TF的扩散以定位遗传调控的靶DNA位点至关重要。检测TF滑动或踩踏DNA的碱基对(bp)分辨率在实验上仍然具有挑战性。我们最近进行了全原子分子动力学(MD)模拟,捕获了小的WRKY结构域TF蛋白沿DNA的自发1-bp步进。基于从此类仿真中获得的10 μs WRKY步进路径,此处的协议显示了如何通过构建用于1-bp蛋白质步进的马尔可夫状态模型(MSM)对TF-DNA系统进行更广泛的构象采样,并测试各种数量的微观和宏观状态用于MSM构建。为了检查TF蛋白与结构基础的DNA的加工一维扩散搜索,该方案进一步展示了如何进行粗粒度(CG)MD模拟以采样系统的长期尺度动力学。与全原子模拟揭示的亚微秒到微秒的蛋白质步进运动相比,这种CG建模和模拟对于揭示蛋白质-DNA静电对TF蛋白质在几十微秒以上的过程扩散运动的影响特别有用。
引言
转录因子(TF)寻找靶DNA以结合和调节基因转录及相关活性1.除了三维(3D)扩散之外,TF的促进扩散被认为是靶DNA搜索所必需的,其中蛋白质还可以沿着一维(1D)DNA滑动或跳跃,或者在DNA2,3,4,5,6,7上进行节间转移。
在最近的一项研究中,我们对植物TF进行了数十微秒(μs)全原子平衡分子动力学(MD)模拟 - DNA8上的WRKY结构域蛋白。在微秒内捕获了WRKY在多晶A DNA上的完整1-bp步进。已经观察到蛋白质沿DNA槽的运动和氢键(HBs)断裂重整动力学。虽然这样的轨迹代表了一条采样路径,但整体蛋白质步进景观仍然缺乏。在这里,我们展示了如何使用构建的马尔可夫状态模型(MSM)围绕最初捕获的蛋白质步进路径扩展计算采样,该模型已广泛实施,用于模拟涉及实质性构象变化和时间尺度分离的各种生物分子系统9,10,11,12,13,14,15,16,17,18,19.目的是揭示TF蛋白沿DNA扩散的构象集合和亚稳态,用于一个循环步骤。
虽然上述MD模拟揭示了DNA上1 bp的蛋白质运动的原子分辨率,但TF以相同的高分辨率沿DNA的长期过程扩散的结构动力学几乎不容易获得。然而,在残留物水平上进行粗粒度(CG)MD仿真在技术上是可行的。CG模拟时间尺度可以有效地扩展到比原子模拟长20、21、22、23、24、25、26、27、28、29的几十倍或几百倍。在这里,我们展示了通过实施Takada lab30开发的CafeMol软件进行的CG模拟。
在目前的方案中,我们首先介绍了沿多A DNA和MSM构建的WRKY结构域蛋白的原子模拟,其重点是对沿DNA仅1 bp的蛋白质步进运动进行采样。然后,我们提出了同一蛋白质 - DNA系统的CG建模和模拟,该系统将计算采样扩展到蛋白质沿着DNA的数十个bps上的过程扩散。
在这里,我们使用GROMACS31,32,33 软件进行MD模拟,并使用MSMbuilder34 构建用于采样构象快照的MSM,以及使用VMD35 可视化生物分子。该协议要求用户能够安装和实现上述软件。然后,CafeMol30 软件的安装和实施对于进行CG MD模拟是必要的。在VMD中还对轨迹和可视化进行了进一步的分析。
研究方案
1. 从原子MD模拟构建马尔可夫状态模型(MSM)
- 自发蛋白质步进途径和初始结构收集
- 使用先前获得的10μs全原子MD轨迹8 从"向前"的1-bp步进路径(即,每纳秒一帧)中均匀地提取10000帧。帧的总数需要足够大,以包括所有代表性构象。
- 在 VMD 中准备具有 10000 帧的过渡路径,方法是单击 "文件">"保存坐标",在"所选原子"框中键入蛋白质或细胞核,然后在"帧"框中选择帧,单击" 保存 "以获取所需的帧。
注意:先前获得的10 μs全原子MD模拟轨迹(此处称为"正向步进轨迹"),用于在34 bp均相多晶A DNA8上进行WRKY步进1-bp距离,用作启动进一步构象采样的初始路径。然而,请注意,在大多数实践中,通过执行定向或有针对性的MD模拟,或实现一般的路径生成方法等,可以构造初始路径。 - 将参考DNA的长轴(从晶体结构)对齐到x轴,并将完整的34-bp DNA的初始质心(COM)设置在坐标空间的原点,以方便进一步数据分析。为此,请单击 VMD 中 Tk 控制台>扩展 ,然后在 Tk 控制台命令窗口中键入:
源旋转.tcl
tcl 脚本可以在 补充文件 3 中找到。 - 然后通过将中心10 bp DNA(A 14至23和T 14'至23')与晶体结构40的DNA对齐来计算蛋白质主链的均方根距离(RMSD),RMSD表示系统的几何测量值(见 图1A)。为此,请单击 "VMD >扩展>分析> RMSD 轨迹工具 ,然后在原子选择框中键入核和残基 14 到 23 和 46 到 55,单击 对齐 ,然后单击 RMSD 框以计算 RMSD 值。
- 在 MATLAB 中,通过键入命令,计算蛋白质围绕 y-z 平面上 DNA Θ(t) 的旋转度
rad2deg(atan(z/y))
初始角度定位定义为 Θ(0)=0,如之前执行的8 所示。 - 在 MATLAB41 中键入以下命令以使用 K-means 方法42,43,44 ,并通过键入将 10000 个结构分类为 25 个聚类:
[idx, C]=kmeans( X, 25)
这里的 X 是RMSD的2D矩阵和WRKY在DNA上的旋转角度。收集这 25 个集群中心的结构,以进行进一步的 MD 模拟。
注意:由于相对于DNA采样的蛋白质RMSD覆盖约25 Å的范围,因此我们选择25个簇以每埃具有一个簇。
- 进行第一 轮MD模拟和模拟设置
- 在 parmbsc1 力场45 下使用 GROMACS 5.1.2 软件32 并使用 shell 中补充文件 2 中的 buildsystem.sh 文件,为 25 个结构构建原子系统。
- 通过在shell中键入以下命令,在NPT集成下对这25个系统进行60-ns MD仿真,时间步长为2 fs:
gmx_mpi grompp -f md.mdp -c npt.gro -p topol.top -o md.tpr
gmx_mpi mdrun -deffnm md
- 聚类 1圣 圆形 MD 轨迹
- 通过在 shell 中键入来删除每个仿真轨迹的前 10 ns:
gmx_mpi trjcat -f md.xtc -b 10000 -e 600000 -o newtraj.xtc
并从 25 × 50 ns 轨迹中收集构象以进行聚类,以便为后续更广泛的采样(第 2 轮 MD 模拟)准备输入结构。
注意:为了减少初始路径的影响并允许局部平衡,删除了10-ns的初始仿真周期。 - 选择蛋白质和DNA之间的距离对作为时间无关成分分析(tICA)46,47,48 投影的输入参数。使用GROMACS中的 make_ndx 命令来执行此操作:
gmx_mpi make_ndx -f 输入.pdb -o index.ndx
注意:这里,选择了残基Y119,K122,K125,R131,Y133,Q146,K144,R135,W116,R117,Y134,K118,Q121的蛋白质CA原子和重原子(NH1,NH2,OH,NZ,NE2,ND2),它们可以与DNA核苷酸形成氢键(HBs),它们与DNA核苷酸的O1P O2P和N6原子配对(A14-20, T19-23)。选定的氨基酸可以形成稳定的HBs或与DNA的盐桥。 - 将上述选定的原子索引从 index.ndx 文件复制到新的文本文件(索引.dat)。通过 python 脚本从 补充文件 1 中获取这些原子之间的对信息,generate_atom_indices.py并键入:
python2.6 generate_atom_indices.py索引.dat >原子索引.txt
这在蛋白质和DNA之间生成415个距离对。 - 通过在 MSMbuilder 命令窗口中键入以下命令,计算每个轨迹的 415 个距离对:
msmb AtomPairsFeaturizer -out pair_features --pair_indices AtomIndices.txt --top references.pdb --trjs "trajectories/*.xtc" --transformed pair_features --stride 5 - 执行 tICA,通过键入以下内容,将数据维度减小到前 2 个与时间无关的分量 (tIC) 或向量上:
msmb tICA -i ../tica_rc_a/tmp/ -o tica_results --n_components 2 --lag_time 10 --伽马 0.05 -t tica_results.h5
注意:tICA是一种降维方法,它计算时间滞后相关矩阵 的特征值,通过以下公式确定仿真系统最慢的松弛自由度:
的特征值,通过以下公式确定仿真系统最慢的松弛自由度: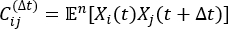
其中 Xi(t) 是时间 t 处第 i 个反应坐标的值,Xj(t+Δt) 是时间 t+Δt 时第 j 个反应坐标的值。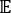 是 Xi(t) 和 X j(t + Δ t) 整体仿真轨迹的乘积的期望值。沿最慢松弛自由度的方向对应于上述时间滞后相关矩阵
是 Xi(t) 和 X j(t + Δ t) 整体仿真轨迹的乘积的期望值。沿最慢松弛自由度的方向对应于上述时间滞后相关矩阵 的最大特征值。在这里,2个tIC似乎是在我们的MSM构造上区分三个宏观状态的最小集合(稍后讨论)。例如,还可以计算广义矩阵瑞利商(GMRQ)得分49,以探索要使用的最佳分量集。
的最大特征值。在这里,2个tIC似乎是在我们的MSM构造上区分三个宏观状态的最小集合(稍后讨论)。例如,还可以计算广义矩阵瑞利商(GMRQ)得分49,以探索要使用的最佳分量集。 - 使用 MSMbuilder 中的命令,通过 K-center43,44 方法将投影数据集聚类到 100 个聚类中(参见图 1B):
msmb KCenters -i ./tica_results.h5 -o kcenters_output -t kcenters_output --n_clusters 100.
选择每个聚类的中心结构作为第 二轮 MD 模拟的初始结构。维护模拟的100个结构的模拟信息,包括位置、温度、压力等,速度除外。
注意:在第一轮25次模拟之后,初始路径的内存已经减少,因此我们在第二轮中生成了更多的聚类,例如100个聚类,以大幅扩展构象采样。
- 通过在 shell 中键入来删除每个仿真轨迹的前 10 ns:
- 进行第 二轮广泛的MD模拟
- 从这100个初始结构开始,在所有原子上施加随机初始速度后,进行60-ns MD仿真。通过在 mdp 文件中打开速度生成来添加随机初始速度,即将 md.mdp 文件gen_vel = 否 更改为 gen_vel = yes。
- 按照步骤 1.3.1 中所述,删除每个仿真的前 10 个 ns,从 100 个× 50 ns 轨迹中平均收集 2,500,000 个快照以构建 MSM。
注意:请注意,在后来的宏观状态构造中,发现了少量具有特别低总体(X-Θ平面底部约0.2%)的偏离路径状态。当宏态的总数设置为 3 到 6 时,这些偏离路径状态被分类为一个宏状态(图 2B)。由于如此低的总体宏观状态仅包括3个轨迹,这些轨迹最终被移除,因此该协议中显示的结果确实是从97×50 ns轨迹中获得的,总共有2,425,000帧或快照。
- 聚类第 2 轮 MD 轨迹
- 像以前一样,对第 2轮轨迹进行tICA。在 MSMbuilder 中键入:
msmb tICA -i ../tica_rc_a/tmp/ -o tica_results --n_components 2 --lag_time 10 --伽马 0.05 -t tica_results.h5 - 计算隐含的时间尺度,以验证相关延迟时间Δt和微观状态数的参数(见 图1C),
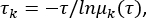
其中 τ 表示用于构建转移概率矩阵 (TPM) 的滞后时间;μk(τ) 表示滞后时间 τ 下 TPM 的第 k 个特征值。将 补充文件 1 中的 python 脚本用于此 python BuildMSMsAsVaryLagTime.py -d .。/ -f ../trajlist_num -i 50 -m 1000 -t 10 -n 20 -s 500. - 通过更改上面使用的参数来改变滞后时间 τ 和微状态数:
python BuildMSMsAsVaryLagTime.py -d ../ -f ../trajlist_num -i 50 -m 1000 -t 5 10 20 30 40 -n 20 -s 20 200 400 500 800 2000
注意:当隐含的时间尺度曲线开始随着时间尺度分离而趋于平稳时,系统被视为马尔可夫系统。然后,选择 Dt 作为相关延迟时间,选择 τ 作为隐含时间刻度开始趋于平稳以构建 MSM 的延迟时间。 - 因此,选择一个相对较大(但不太大)的状态数,N = 500,以及相对较短的相关延迟时间Δt = 10 ns。发现构建 MSM 的滞后时间为 τ =10 ns。
- 使用以下命令将构象分类为 500 个簇(参见 图 1D):
msmb KCenters -i ./tica_results.h5 -o kcenters_output -t kcenters_output --n_clusters 500
- 像以前一样,对第 2轮轨迹进行tICA。在 MSMbuilder 中键入:
- 中微管理建筑
- 将 500 个微观状态集中为 3–6 个宏观状态,以找出最适合根据 MSMbuilder 中的 PCCA+ 算法50 的宏观状态的数量,方法是使用补充文件 1 python msm_lumping_usingPCCAplus.py中的 python 脚本。通过构建少量的宏观状态,即在动力学上聚集数百个微观状态,如下所述,确定生物分子最基本的构象变化的模型的简化动力学网络。
- 如步骤1.1.3和1.1.4所述,将高维构象映射到X(蛋白质沿DNA长轴的运动)和蛋白质沿DNA的旋转角度(例如,没有状态具有过低的种群<1%;见 图2C)。然后找到最能代表系统的3个宏状态(图1E)。参见 图2D ,了解蛋白质沿DNA的运动和蛋白质围绕DNA的旋转角度的快照。
注意:在之前生成10 μs自发蛋白质正向步进路径的工作中,我们还进行了5 x 4 μs平衡MD模拟,以适度扩展采样。我们显示了原始正向路径的映射(见 图2A 左图)以及先前进行的正向路径上进一步的4μs采样轨迹(见 图2A 右)8。图中显示了本工作中使用的原始100×50 ns(见 图2B 左)8 和97×50 ns轨迹的映射(见 右图2B )。
- 计算平均首次通过时间 (MFPT)
- 根据 500 微状态 MSM 的 TPM 执行五个 10 ms 蒙特卡罗 (MC) 轨迹,并将 10 ns 的延迟时间设置为 MC 的时间步长。通过补充文件 1 python python mfpt_msm3.py中的 python 脚本计算每对宏状态之间的 MFPT52(图 3)。
- 使用 补充文件 2 中的 bash 文件计算 MFPT 的平均误差和标准误差,键入:
sh mfpt_analysis.bash
2. 进行粗粒度(CG)模拟,对长时间动力学进行采样
- 使用CafeMol 3.0软件30进行CG模拟。查看在扩展名为 .inp 的输入配置文件中指定的 CG 模拟设置,包括输入结构、模拟参数、输出文件等。在终端上键入以下命令以运行 CG 模拟:
咖啡酚 XXX.inp - 在输入文件中指定以下块,每个块都以标签 < and ending with >>>>开头。
- 设置文件名块(必需)以指定工作目录和输入/输出文件存储路径。为这些模拟的文件名块键入以下内容:
<<<<文件名
路径 = XXXXX(工作路径)
文件名 = wrky(输出文件名)
输出 psf pdb 电影 dcd rst
path_pdb = XXXXX(输入本机结构路径)
path_ini = XXXXX(输入初始结构路径)
path_natinfo = XXXXX(本机信息文件路径)
path_para = XXXXX(参数文件路径)
>>>>
注意:由于Go-model53 在CG建模中使用,即蛋白质将偏向于天然构象,因此需要将建模结构设置为天然构象。在这里,输入晶体结构被设置为原生构象。 - 设置作业控制块(必需)以定义模拟的运行模式。键入以下命令:
<<<< job_cntl
i_run_mode = 2(= 2 恒温模拟)
i_simulate_type = 1 (=1 朗格文动力学)
i_initial_state = 2(=2 表示初始配置为本机配置)
>>>>
选择恒温朗格文动力学仿真。 - 设置单位和状态块(必需)以定义输入结构的信息。键入以下命令:
<<<< unit_and_state
i_seq_read_style = 1(=1 表示从 PDB 文件读取序列)
i_go_native_read_style = 1(=1 表示本机结构来自 PDB 文件)
1蛋白质蛋白质.pdb(单位和状态molecular_type native_structure)
2-3 DNA DNA.pdb(单位和状态molecular_type native_structure)
>>>>
注意:需要初始输入结构文件(蛋白质.pdb和DNA.pdb此处)。这些结构以 pdb 格式编写。这里需要两个pdb文件:一个是包含WRKY重原子坐标(单元1)的蛋白质结构文件,另一个是200-bp双链(ds)DNA(单元2-3)的坐标。蛋白质最初放置在距离DNA15埃的位置。 - 设置energy_function块中定义的能量功能块(必需)。键入以下命令:
<<<< energy_function
本地(1) L_GO
本地(2-3) L_DNA2
NLOCAL(1/1) GO EXV ELE
NLOCAL(2-3/2-3) ELE DNA
NLOCAL(1/2-3) EXV ELE
i_use_atom_protein = 0
i_use_atom_dna = 0
i_para_from_ninfo = 1
i_triple_angle_term = 2
>>>>
注意:在CG模拟中,蛋白质由Go-model53 粗粒度表示,每个氨基酸由放置在其Cα位置的CG颗粒表示。蛋白质构象将在Go电位下偏向天然结构或晶体结构(图4A 左)。DNA由3SPN.2模型54描述,其中每个核苷酸由3个CG颗粒S,P,N表示,分别对应于糖,磷酸盐和含氮碱基(图4A 右)。考虑不同链之间的静电和vdW相互作用。在CG模拟中,蛋白质和DNA之间的静电相互作用近似于Debye-Hückel电位55。vdW斥力能量采用与围棋模型中相同的形式。 - 设置md_information块(必需)以定义仿真信息。键入以下命令:
<<<< md_information
n_step_sim = 1
n_tstep(1) = 500000000
tstep_size = 0.1
n_step_save = 1000
n_step_neighbor = 100
i_com_zeroing = 0
i_no_trans_rot = 0
温度 = 300.0
n_seed = -1
>>>>
n_tstep是模拟步骤。将tstep_size设置为每个MD步骤的时间长度,每个CG Cafemol时间步骤约为200 fs30,因此这里的每个MD步骤原则上为200×0.1 fs。每 100 MD 步更新一次邻居列表(n_step_neighbor = 100)。将模拟温度设置为300 K.通过使用Berendsen恒温器56更新蛋白质结构的速度型Verlet算法来控制温度。
注意:n_step_sim是基于Go模型势的盆地数,或能量曲线的局部最小数。多盆电位允许蛋白质构象偏向于不同的构象,以便蛋白质构象可以从一个局部最小值变化到另一个局部最小值。这里只使用单个盆地Go模型,这意味着在模拟中蛋白质只有一个偏置构象(晶体结构)。同时,由于在CG背景下没有蛋白质 - DNA氢键相互作用等,因此分子运动的采样速度甚至更快,即比原子模拟中的>10倍。 - 设置静电块(仅在使用静电相互作用时才需要)作为不同链之间的静电相互作用的考虑因素,因此使用此块通过键入来定义静电相互作用的参数:
<<<<静电
cutoff_ele = 10.0
ionic_strength = 0.15
>>>>
将静电相互作用中的 Debye 长度设置为 10 Å,对应于溶液条件。将离子强度设置为0.15 M,如生理状况一样。
- 设置文件名块(必需)以指定工作目录和输入/输出文件存储路径。为这些模拟的文件名块键入以下内容:
结果
旋转耦合滑动或 1 bp 步进的 WRKY 从 MSM 结构
DNA上的所有蛋白质构象都映射到蛋白质COM沿DNA的纵向运动X和旋转角度(见 图3A)。这两个度的线性偶联表示WRKY结构域蛋白在DNA上的旋转耦合步进。构象可以在 MSM 中进一步聚类为 3 个宏状态(S1、S2 和 S3)。然后,WRKY 的正向步进遵循宏观状态转换 S1->S2->S3。S1是指由建模结构(基于WRKY-DNA复合物40的?...
讨论
这项工作解决了如何进行基于结构的计算模拟和采样,以揭示转录因子或TF蛋白沿着DNA移动,不仅在步进的原子细节下,而且在过程扩散中,这对于TF在DNA靶标搜索中的促进扩散至关重要。为此,首先构建了小TF结构域蛋白WRKY沿着均匀的poly-A DNA步进1-bp的马尔可夫状态模型或MSM,以便可以揭示DNA上的蛋白质构象集合以及蛋白质 - DNA界面处的集体氢键或HB动力学。为了获得MSM,我们沿着自发蛋白质步?...
披露声明
作者没有利益冲突。
致谢
这项工作得到了NSFC Grant #11775016和#11635002的支持。JY得到了UCI的CMCF通过NSF DMS 1763272和Uci的西蒙斯基金会赠款#594598和启动基金的支持。LTD得到了上海市自然科学基金#20ZR1425400和#21JC1403100。我们也感谢北京计算科学研究中心(CSRC)的计算支持。
材料
| Name | Company | Catalog Number | Comments |
| CafeMol | Kyoto University | coarse-grained (CG) simulations | |
| GROMACS | University of Groningen Royal Institute of Technology Uppsala University | molecular dynamics simulations software | |
| Matlab | MathWorks | Numerical calculation software | |
| MSMbuilder | Stanford University | build MSM | |
| VMD | UNIVERSITY OF ILLINOIS AT URBANA-CHAMPAIGN | molecular visualization program |
参考文献
- Latchman, D. S. Transcription factors: an overview. The International Journal of Biochemistry & Cell Biology. 29 (12), 1305-1312 (1997).
- Berg, O. G., von Hippel, P. H. Selection of DNA binding sites by regulatory proteins. Statistical-mechanical theory and application to operators and promoters. Journal of Molecular Biology. 193 (4), 723-750 (1987).
- von Hippel, P. H., Berg, O. G. Facilitated target location in biological systems. The Journal of Biological Chemistry. 264 (2), 675-678 (1989).
- Halford, S. E., Marko, J. F. How do site-specific DNA-binding proteins find their targets. Nucleic Acids Research. 32 (10), 3040-3052 (2004).
- Slusky, M., Mirny, L. A. Kinetics of protein-DNA interaction: facilitated target location in sequence-dependent potential. Biophysical Journal. 87 (6), 4021-4035 (2004).
- Bauer, M., Metzler, R. Generalized facilitated diffusion model for DNA-binding proteins with search and recognition states. Biophysical Journal. 102 (10), 2321-2330 (2012).
- Shvets, A. A., Kochugaeva, M. P., Kolomeisky, A. B. Mechanisms of Protein Search for Targets on DNA: Theoretical Insights. Molecules. 23 (9), 2106 (2018).
- Dai, L., Xu, Y., Du, Z., Su, X. D., Yu, J. Revealing atomic-scale molecular diffusion of a plant-transcription factor WRKY domain protein along DNA. Proceedings of the National Academy of Sciences of the United States of America. 118 (23), 2102621118 (2021).
- Chodera, J. D., Singhal, N., Pande, V. S., Dill, K. A., Swope, W. C. Automatic discovery of metastable states for the construction of Markov models of macromolecular conformational dynamics. The Journal of Chemical Physics. 126 (15), 155101 (2007).
- Pan, A. C., Roux, B. Building Markov state models along pathways to determine free energies and rates of transitions. The Journal of Chemical Physics. 129 (6), 064107 (2008).
- Bowman, G. R., Huang, X., Pande, V. S. Using generalized ensemble simulations and Markov state models to identify conformational states. Methods. 49 (2), 197-201 (2009).
- Prinz, J. H., et al. Markov models of molecular kinetics: Generation and validation. The Journal of chemical physics. 134 (17), 174105 (2011).
- Chodera, J. D., Noé, F. Markov state models of biomolecular conformational dynamics. Current Opinion in Structural Biology. 25, 135-144 (2014).
- Malmstrom, R. D., Lee, C. T., Van Wart, A. T., Amaro, R. E. On the Application of Molecular-Dynamics Based Markov State Models to Functional Proteins. Journal of Chemical Theory and Computation. 10 (7), 2648-2657 (2014).
- Husic, B. E., Pande, V. S. Markov State Models: From an Art to a Science. Journal of the American Chemical Society. 140 (7), 2386-2396 (2018).
- Sittel, F., Stock, G. Perspective: Identification of collective variables and metastable states of protein dynamics. The Journal of chemical physics. 149 (15), 150901 (2018).
- Wang, W., Cao, S., Zhu, L., Huang, X. Constructing Markov State Models to elucidate the functional conformational changes of complex biomolecules. WIREs Computational Molecular Science. 8, 1343 (2018).
- Peng, S., et al. Target search and recognition mechanisms of glycosylase AlkD revealed by scanning FRET-FCS and Markov state models. Proceedings of the National Academy of Sciences of the United States of America. 117 (36), 21889-21895 (2020).
- Tian, J., Wang, L., Da, L. T. Atomic resolution of short-range sliding dynamics of thymine DNA glycosylase along DNA minor-groove for lesion recognition. Nucleic Acids Research. 49 (3), 1278-1293 (2021).
- Chu, J. -. W., Izveko, S., Voth, G. The multiscale challenge for biomolecular systems: coarse-grained modeling. Molecular Simulation. 32 (3-4), 211-218 (2006).
- Marrink, S. J., Risselada, H. J., Yefimov, S., Tieleman, D. P., De Vries, A. H. The MARTINI force field: coarse grained model for biomolecular simulations. The Journal of Physical Chemistry B. 111 (27), 7812-7824 (2007).
- Givaty, O., Levy, Y. Protein sliding along DNA: dynamics and structural characterization. Journal of Molecular Biology. 385 (4), 1087-1097 (2009).
- Khazanov, N., Levy, Y. Sliding of p53 along DNA can be modulated by its oligomeric state and by cross-talks between its constituent domains. Journal of Molecular Biology. 408 (2), 335-355 (2011).
- Riniker, S., Allison, J. R., van Gunsteren, W. F. On developing coarse-grained models for biomolecular simulation: a review. Physical Chemistry Chemical Physics : PCCP. 14 (36), 12423-12430 (2012).
- Kmiecik, S., et al. Coarse-Grained Protein Models and Their Applications. Chemical Reviews. 116 (14), 7898-7936 (2006).
- Bhattacherjee, A., Krepel, D., Levy, Y. Coarse-grained models for studying protein diffusion along DNA. WIREs Computational Molecular Science. 6, 515-531 (2016).
- Wang, J., et al. Machine Learning of Coarse-Grained Molecular Dynamics Force Fields. ACS Central Science. 5 (5), 755-767 (2019).
- Joshi, S. Y., Deshmukh, S. A. A review of advancements in coarse-grained molecular dynamics simulations. Molecular Simulation. 47 (10-11), 786-803 (2021).
- Bigman, L. S., Greenblatt, H. M., Levy, Y. What Are the Molecular Requirements for Protein Sliding along DNA. The Journal of Physical Chemistry B. 125 (12), 3119-3131 (2021).
- Kenzaki, H., et al. CafeMol: A Coarse-Grained Biomolecular Simulator for Simulating Proteins at Work. Journal of Chemical Theory and Computation. 7 (6), 1979-1989 (2011).
- Berendsen, H. J. C., vander Spoel, D., van Drunen, R. GROMACS: a message-passing parallel molecular dynamics implementation. Computer Physics Communications. 91 (1-3), 43-56 (1995).
- vander Spoel, D., et al. GROMACS: fast, flexible, and free. Journal of Computational Chemistry. 26 (16), 1701-1718 (2005).
- Abraham, M. J., et al. GROMACS: High performance molecular simulations through multi-level parallelism from laptops to supercomputers. SoftwareX. 1-2, 19-25 (2015).
- Harrigan, M. P., et al. MSMBuilder: Statistical Models for Biomolecular Dynamics. Biophysical journal. 112 (1), 10-15 (2017).
- Humphrey, W., Dalke, A., Schulten, K. VMD: visual molecular dynamics. Journal of Molecular Graphics. 14 (1), 33-38 (1996).
- Izrailev, S., et al. Steered Molecular Dynamics. Computational Molecular Dynamics: Challenges, Methods, Ideas. 4, 39-65 (1999).
- Schlitter, J., Engels, M., Krüger, P. Targeted molecular dynamics: a new approach for searching pathways of conformational transitions. Journal of Molecular Graphics. 12 (2), 84-89 (1994).
- Maragliano, L., Fischer, A., Vanden-Eijnden, E., Ciccotti, G. String method in collective variables: minimum free energy paths and isocommittor surfaces. The Journal of Chemical Physics. 125 (2), 24106 (2006).
- Weiss, D. R., Levitt, M. Can morphing methods predict intermediate structures. Journal of Molecular Biology. 385 (2), 665-674 (2009).
- Xu, Y. P., Xu, H., Wang, B., Su, X. D. Crystal structures of N-terminal WRKY transcription factors and DNA complexes. Protein. 11 (3), 208-213 (2020).
- Higham, D. J., Higham, N. J. MATLAB guide. Society for Industrial and Applied Mathematics. , (2016).
- Hartigan, J. A., Wong, M. A. Algorithm AS 136: A K-Means Clustering Algorithm. Journal of the Royal Statistical Society. Series C (Applied Statistics). 28 (1), 100-108 (1979).
- Gonzalez, T. F. Clustering to minimize the maximum intercluster distance. Theoretical Computer Science. 38, 293-306 (1985).
- Zhao, Y., Sheong, F. K., Sun, J., Sander, P., Huang, X. A fast parallel clustering algorithm for molecular simulation trajectories. Journal of Computational Chemistry. 34 (2), 95-104 (2013).
- Ivani, I., et al. Parmbsc1: a refined force field for DNA simulations. Nature Methods. 13 (1), 55-58 (2016).
- Naritomi, Y., Fuchigami, S. Slow dynamics of a protein backbone in molecular dynamics simulation revealed by time-structure based independent component analysis. The Journal of Chemical Physics. 139 (21), 215102 (2013).
- Naritomi, Y., Fuchigami, S. Slow dynamics in protein fluctuations revealed by time-structure based independent component analysis: the case of domain motions. The Journal of Chemical Physics. 134 (6), 065101 (2011).
- Pérez-Hernández, G., Paul, F., Giorgino, T., De Fabritiis, G., Noé, F. Identification of slow molecular order parameters for Markov model construction. The Journal of Chemical Physics. 139 (1), 015102 (2013).
- McGibbon, R. T., Pande, V. S. Variational cross-validation of slow dynamical modes in molecular kinetics. The Journal of Chemical Physics. 142 (12), 124105 (2015).
- Deuflhard, P., Weber, M. Robust Perron cluster analysis in conformation dynamics. Linear Algebra and its Applications. 398, 161-184 (2005).
- Silva, D. A., et al. Millisecond dynamics of RNA polymerase II translocation at atomic resolution. Proceedings of the National Academy of Sciences of the United States of America. 111 (21), 7665-7670 (2014).
- Swope, W. C., Pitera, J. W., Suits, F. Describing Protein Folding Kinetics by Molecular Dynamics Simulations. 1. Theory. The Journal of Physical Chemistry B. 108 (21), 6571-6581 (2004).
- Clementi, C., Nymeyer, H., Onuchic, J. N. Topological and energetic factors: what determines the structural details of the transition state ensemble and "en-route" intermediates for protein folding? An investigation for small globular proteins. Journal of molecular biology. 298 (5), 937-953 (2000).
- Hinckley, D. M., Freeman, G. S., Whitmer, J. K., De Pablo, J. J. An experimentally-informed coarse-grained 3-Site-Per-Nucleotide model of DNA: structure, thermodynamics, and dynamics of hybridization. The Journal of chemical physics. 139 (14), 144903 (2013).
- Debye, P., Huckel, E. The theory of the electrolyte II-The border law for electrical conductivity. Physikalische Zeitschrift. 24, 305-325 (1923).
- Berendsen, H. J., Postma, J. V., van Gunsteren, W. F., DiNola, A., Haak, J. R. Molecular dynamics with coupling to an external bath. The Journal of Chemical Physics. 81, 3684-3690 (1984).
- Bowman, G. R. Improved coarse-graining of Markov state models via explicit consideration of statistical uncertainty. The Journal of Chemical Physics. 137 (13), 134111 (2012).
- Jain, A., Stock, G. Identifying metastable states of folding proteins. Journal of Chemical Theory and Computation. 8 (10), 3810-3819 (2012).
- Röblitz, S., Weber, M. Fuzzy spectral clustering by PCCA+: application to Markov state models and data classification. Advances in Data Analysis and Classification. 7, 147-179 (2013).
- Mardt, A., Pasquali, L., Wu, H., Noé, F. VAMPnets for deep learning of molecular kinetics. Nature Communications. 9 (1), 5 (2018).
- Wang, W., Liang, T., Sheong, F. K., Fan, X., Huang, X. An efficient Bayesian kinetic lumping algorithm to identify metastable conformational states via Gibbs sampling. The Journal of Chemical Physics. 149 (7), 072337 (2018).
- Chen, W., Sidky, H., Ferguson, A. L. Nonlinear discovery of slow molecular modes using state-free reversible VAMPnets. The Journal of Chemical Physics. 150 (21), 214114 (2019).
- Gu, H., et al. RPnet: a reverse-projection-based neural network for coarse-graining metastable conformational states for protein dynamics. Physical Chemistry Chemical Physics :PCCP. 24 (3), 1462-1474 (2022).
- Lane, T. J., Bowman, G. R., Beauchamp, K., Voelz, V. A., Pande, V. S. Markov state model reveals folding and functional dynamics in ultra-long MD trajectories. Journal of the American Chemical Society. 133 (45), 18413-18419 (2011).
- Konovalov, K. A., Unarta, I. C., Cao, S., Goonetilleke, E. C., Huang, X. Markov State Models to Study the Functional Dynamics of Proteins in the Wake of Machine Learning. JACS Au. 1 (9), 1330-1341 (2021).
- Cao, S., Montoya-Castillo, A., Wang, W., Markland, T. E., Huang, X. On the advantages of exploiting memory in Markov state models for biomolecular dynamics. The Journal of Chemical Physics. 153 (1), 014105 (2020).
- Brandani, G. B., Takada, S. Chromatin remodelers couple inchworm motion with twist-defect formation to slide nucleosomal DNA. PLoS Computational Biology. 14 (11), 1006512 (2018).
- Tan, C., Terakawa, T., Takada, S. Dynamic Coupling among Protein Binding, Sliding, and DNA Bending Revealed by Molecular Dynamics. Journal of the American Chemical Society. 138 (27), 8512-8522 (2016).
- Terakawa, T., Takada, S. p53 dynamics upon response element recognition explored by molecular simulations. Scientific reports. 5, 17107 (2015).
- Brandani, G. B., Niina, T., Tan, C., Takada, S. DNA sliding in nucleosomes via twist defect propagation revealed by molecular simulations. Nucleic Acids Research. 46 (6), 2788-2801 (2018).
- Knotts, T. A., Rathore, N., Schwartz, D. C., de Pablo, J. J. A coarse grain model for DNA. The Journal of Chemical Physics. 126 (8), 084901 (2007).
- Freeman, G. S., Hinckley, D. M., Lequieu, J. P., Whitmer, J. K., de Pablo, J. J. Coarse-grained modeling of DNA curvature. The Journal of Chemical Physics. 141 (16), 165103 (2014).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。