需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
使用慢病毒介导的CRISPR / Cas9基因编辑开发敲除肌肉细胞系
摘要
该协议描述了如何使用CRISPR / Cas9生成敲除肌母细胞,从引导RNA的设计到细胞克隆和敲除克隆的表征。
摘要
簇状调节间隔短回文重复序列(CRISPR)/Cas 9的一个重要应用是敲除细胞系的开发,专门用于研究在基因诊断期间鉴定的与疾病相关的新基因/蛋白质的功能。对于这种细胞系的开发,必须解开两个主要问题:将CRISPR工具(Cas9和引导RNA)高效插入所选细胞中,以及将Cas9活性限制在所选基因的特定缺失上。这里描述的方案专门用于将CRISPR工具插入难以转染的细胞中,例如肌肉细胞。该协议基于使用慢病毒,用公开可用的质粒生产,其所有克隆步骤都被描述为靶向目的基因。Cas9活性的控制是使用先前描述的称为KamiCas9的系统进行的,其中用编码靶向Cas9的引导RNA的慢病毒转导细胞允许逐步消除Cas9表达。该方案已应用于 RYR1-敲除人肌肉细胞系的开发,该细胞系已在蛋白质和功能水平上进一步表征,以确认参与肌肉细胞内钙释放和激发 - 收缩耦合的这种重要钙通道的敲除。这里描述的过程可以很容易地应用于肌肉细胞或其他难以转染的细胞中的其他基因,并产生有价值的工具来研究人类细胞中的这些基因。
引言
随着基因测序的进展和特定组织中未知功能基因突变的鉴定,开发相关细胞模型以了解新靶基因的功能并确认其参与相关病理生理学机制构成了必不可少的工具。此外,这些模型对未来的治疗发展1,2具有重要意义,并且构成了与国际上关于减少在实验中使用动物的建议直线开发敲除动物模型的有趣替代方案。使用CRISPR / Cas9进行基因编辑是目前最强大的工具之一,它允许开发许多敲除/敲入模型,并且使用CRISPR / Cas9进行靶向基因验证是CRISPR / Cas93使用最广泛的应用之一。基因编辑的成功依赖于在靶细胞模型中引入CRISPR工具(引导RNA和核酸酶Cas9)的能力,这在许多难以转染的细胞(如肌肉细胞4)中可能是一个挑战。这种挑战可以通过使用病毒(通常是慢病毒)来克服,其具有有效转译许多细胞类型并递送其转基因的巨大优势。但它的主要缺点是转基因在宿主细胞基因组中的整合,导致定位在整合位点的基因的潜在改变和转基因的永久表达,这在核酸酶Cas9的情况下会导致破坏性后果5。Merienne及其同事6提出了一种智能解决方案,其中包括将靶向Cas9基因本身的引导RNA引入细胞,导致Cas9失活。本文将这种策略的适应性作为用户友好和多功能的方案提出,允许敲除难以转染的细胞中的几乎任何基因。
这里提出的方案的目标是诱导永生化肌肉细胞中目的基因的失活。它可用于敲除不同类型的永生化细胞中的任何目的基因。这里描述的方案包含设计指导RNA并将其克隆成慢病毒质粒的步骤,在慢病毒载体中生产CRISPR工具,用不同的慢病毒转导细胞,以及克隆细胞以产生均匀的编辑细胞系的步骤。
使用该协议,已经开发了永生化的人骨骼肌细胞,并删除了1型ryanodine受体(RyR1),这是一种参与细胞内钙释放和肌肉收缩的必需钙通道7。该基因的敲除(KO)已使用蛋白质印迹在蛋白质水平上得到证实,并且在功能水平上使用钙成像得到证实。
研究方案
肌肉活检是根据欧洲建议和法国立法从研究组织银行(Myobank,欧盟网络EuroBioBank的合作伙伴,法国巴黎)获得的。已获得所有人的书面知情同意。永生化成肌母细胞由V. Mouly博士(法国巴黎Myology Institute)友好地制作,并且协议由Myology Institute伦理委员会(MESRI,n AC-2019-3502)批准。
1. 防鲜膜导轨设计
- 确定要删除的基因区域。使用基因组浏览器工具(如 ensembl.org 或 genome.ucsc.edu)搜索其基因组序列,并确定两个区域的染色体坐标,以查找要删除的区域两侧的引导RNA(gRNA)。
- 对于这里使用的基因,获取 RYR1 基因的FASTA序列和外显子101的序列,如下所示。在ensembl中,在人类基因组的最新版本中搜索 RYR1 ,选择第一个条目,然后单击蛋白质编码序列的转录本。然后,单击 外显子 以重定向到该基因的外显子列表。
- 单击 "下载序列 "并选择" 仅基因组序列" 以下载整个基因的完整共识序列。向下滚动基因的外显子和内含子列表,然后选择靶向的外显子和内含子。
- 在基因中找到相应的核苷酸序列。选择要删除的外显子的上游和下游的内含子的核苷酸序列,这些序列将用于搜索gRNA。
- 使用在线工具(如 Crispor.tefor.net 8)在步骤1.1中确定的区域(要删除的区域上游和下游的内含子)中设计两个gRNA,此处称为引导1和引导2。选择由几百个碱基对(bp)分隔的两个gRNA,每个gRNA的序列正好是20个核苷酸长,没有原间隔相邻基序(PAM)。选择可用的最佳指南以限制脱靶。有关导轨设计的示例,请参见 图 1 。
- 预计两个导引物之间的序列将被删除,并导致目的基因的敲除,因此选择两个导联体的位置,使其删除目的基因中的必需序列或必需外显子。确保删除的序列/外显子不是仅存在于基因的替代转录本中和/或它编码蛋白质的重要部分,因此其缺失将导致功能性敲除。
注意:尽管Cas9裂解位点预计发生在原间隔相邻基序(PAM)上游3 bp处,但也可能发生较大距离的裂解,因此一个好的解决方案是不依赖于裂解位点的精确定位,例如内含子中的裂解。
- 预计两个导引物之间的序列将被删除,并导致目的基因的敲除,因此选择两个导联体的位置,使其删除目的基因中的必需序列或必需外显子。确保删除的序列/外显子不是仅存在于基因的替代转录本中和/或它编码蛋白质的重要部分,因此其缺失将导致功能性敲除。
- 在没有PAM的情况下确定每个gRNA的反向补体(RC)序列,以便具有以下序列:指南1和指南1-RC,指南2和指南2-RC。
- 订购 表1 中介绍的引物进行质粒的克隆。在整个方案中,在无菌H2O中使用浓度为10 nM的引物。
注意:添加到这些引物中的gRNA的序列(粗体和下划线)分别对应于引导蛋白,启动子和反式激活 - Crispr RNA(tracrRNA)之前和之后的质粒序列,并且不应进行修改以确保引物和质粒之间的良好重叠。
2. 质粒克隆
注意:在此步骤中,gRNA将入质粒骨架中以产生慢病毒。编码两种gRNA的盒首先通过连续聚合酶链反应(PCR)使用重叠的引物产生。然后将新盒插入慢病毒骨架质粒#87919中。
- 获得以下质粒:质粒 #87919,编码慢病毒载体中的 CRISPR 引导 RNA,以及编码慢病毒载体中 SpCas9 序列的质粒 #87904。
- 盒式结构
注:克隆协议摘要如下: 图2.- 运行PCR(A)反应,使用2μL质粒#87919,2μL primer_XmaIF,2μL primer_Guide1R,25μL聚合酶混合物和19μLH2O.运行以下PCR程序(程序1):在98°C下初始变性5分钟,然后在98°C下30次循环:30 s, 在60°C下30秒,在72°C下1分钟45秒,在72°C下最终伸长7分钟。 步骤1.4中描述的引物的Tm为60°C。
注意:虽然程序1中的伸长时间看起来很长,但选择这个伸长时间是为了确保生产足够多的合适尺寸的材料。事实上,由于质粒中的重复序列(RNA导体后tracrRNA的序列在质粒#87919中重复三次),预期DNA的PCR扩增很困难,并且在连续的PCR中产生额外的更小的条带。因此,由于不同PCR产物之间的竞争,要么延长了伸长时间(有利于最长的一个并在末端有足够的纯化材料),要么对长片段使用了接触式PCR(程序2)(例如步骤2.2.6中描述的PCR(最终)。 - 在Tris-硼酸盐-EDTA缓冲液(TBE)中的1%琼脂糖凝胶上分离PCR产物,切除并纯化300 bp片段。按照制造商的说明使用专用试剂盒进行纯化,洗脱最终体积为20μL。直接使用纯化的片段或储存在-20°C进行步骤2.2.5。
- 使用PCR程序1,使用2μL质粒#87919,2μL primer_Guide1F,2μL primer_Guide2R,25μL聚合酶混合物和19μLH2O进行PCR(B)反应。在TBE中的1%琼脂糖凝胶上分离PCR产物,在20μL洗脱缓冲液中切除并纯化400 bp片段。直接使用纯化的片段或储存在-20°C进行步骤2.2.5。
- 使用PCR程序1,使用2μL质粒#87919,2μL primer_Guide2F,2μL primer_BlpIR,25μL聚合酶混合物和19μLH2O进行PCR(C)反应。在TBE缓冲液中的1%琼脂糖凝胶上分离PCR产物。在20μL洗脱缓冲液中切除并纯化600 bp片段。直接使用纯化的片段或储存在-20°C进行步骤2.2.6。
注意:由于引物在质粒中重复区域上的引物杂交,900 bp处的另一个片段可能是可见的,如步骤2.2.1的注释中所述。如果存在,应丢弃此条带。 - 运行PCR(D)反应,用2μL洗脱PCR A(从步骤2.2.2开始),2μL洗脱PCR B(从步骤2.2.3开始),2μL primer_XmaIF,2μL primer_Guide2R,25μL聚合酶混合物和19μLH2O,使用PCR程序1。在TBE缓冲液中的1%琼脂糖凝胶上分离PCR产物;在20μL洗脱缓冲液中切除并纯化700 bp片段。直接使用纯化的片段或储存在-20°C进行步骤2.2.6。
注意:由于引物在重复区域的杂交,其他条带可能在>1,000 bp,400 bp和300 bp处可见,应丢弃。 - 运行PCR(最终)反应,用4μL洗脱PCR C(从步骤2.2.4开始),4μL洗脱PCR D(从步骤2.2.5开始),4μL primer_XmaIF,4μL primer_BlpIR,50μL聚合酶混合物和34μLH2O。使用的程序如下(PCR程序2):在98°C下初始变性5分钟;六个循环:98°C下30秒,66°C下30秒(每个循环杂交温度降低1°C),72°C下1分钟45;35个循环:98°C下30 s,60°C下30 s,72°C下1分钟45,72°C下最终伸长5分钟。
注意:这种最终扩增的PCR程序是一种向下的PCR,与前一个不同,因为最终扩增的尺寸很大,在每个导引物之后包含两个重复的tracrRNA序列。 - 在TBE缓冲液中的1%琼脂糖凝胶上分离PCR产物;切除并纯化最终的盒,其在20μL洗脱缓冲液中以约1,300 bp的速度迁移。量化洗脱产物。直接使用纯化的片段或储存在步骤2.3.2.1的-20°C。这是将插入慢病毒质粒中的最终产物。
注意:其他片段在>1000 bp,400 bp和300 bp时可见,对应于应丢弃的不完全PCR片段。
- 运行PCR(A)反应,使用2μL质粒#87919,2μL primer_XmaIF,2μL primer_Guide1R,25μL聚合酶混合物和19μLH2O.运行以下PCR程序(程序1):在98°C下初始变性5分钟,然后在98°C下30次循环:30 s, 在60°C下30秒,在72°C下1分钟45秒,在72°C下最终伸长7分钟。 步骤1.4中描述的引物的Tm为60°C。
- 将gRNA盒插入慢病毒质粒骨架中。
- 通过用限制性内切酶XmaI和BlpI对质粒#87919进行双重消化来线性化质粒。
- 用15μL推荐缓冲液,15μL质粒(1μg/ μL),7.5μLBlbI酶(10 U / μL),7.5μLXmaI酶(10 U / μL)和112.5μL H2O制备反应。 将总量上样于1%琼脂糖凝胶上,切出并用适当的试剂盒纯化〜10 kb质粒。在20μL缓冲液中进行洗脱,并使用光密度测量定量洗脱产物。
注意:对大型DNA片段使用适当的纯化方案,例如Sun和coll 9描述的方案。
- 用15μL推荐缓冲液,15μL质粒(1μg/ μL),7.5μLBlbI酶(10 U / μL),7.5μLXmaI酶(10 U / μL)和112.5μL H2O制备反应。 将总量上样于1%琼脂糖凝胶上,切出并用适当的试剂盒纯化〜10 kb质粒。在20μL缓冲液中进行洗脱,并使用光密度测量定量洗脱产物。
- 连接gRNA盒和质粒。用步骤2.2.7中的gRNA盒和步骤2.3.1.1中的线性化质粒制备反应混合物,加入2μL酶和H 2 O以具有10μL的最终体积 p_guides。
注意:DNA盒量应在50-100ng之间,质粒量应在100-200ng之间,摩尔比为2:1。
- 通过用限制性内切酶XmaI和BlpI对质粒#87919进行双重消化来线性化质粒。
- 使用2μL新鲜制备的质粒转化具有化学性质 的大肠杆菌 ,例如Stbl3(50μL)或XL10-Gold,并在37°C下生长1小时后,在没有抗生素的情况下用100μg/ mL氨苄西林铺展在LB琼脂平板上。在37°C孵育过夜。选择几个菌落,并按照制造商的说明使用商业套件进行小型准备。
- 使用PCR程序1对具有Primer_XmaI的微量prep DNA进行PCR扩增,并使用PCR程序1 Primer_BlpR(见 图2)。在TBE缓冲液中的1%琼脂糖凝胶上分离PCR产物。选择几个菌落(~5),其条带的预期大小约为1300 bp。
- 使用Primer_XmaI或Primer_BlpR对选定的菌落进行DNA测序,以确认gRNA盒的正确插入。
注意:其中一个序列验证的菌落在研究中进一步使用,质粒称为p_guides。 - 从步骤2.2(盒式结构)用引物 - 杀手F和R以及引物 - mCherry F和R重复。质粒称为p_Killer。
3. 慢病毒的产生
- 按照制造商的说明,使用不含内毒素的 maxi-prep 试剂盒生产和纯化大量所需的所有质粒。以2μg/ μL制备等分试样,在-20°C下储存
- 细胞制备(第1天)
- 准备18块145厘米的平板,每块板上接种1×106 HEK293细胞,在16 mL由Dulbecco的改良鹰培养基(DMEM)高葡萄糖丙酮酸盐组成的培养基中,补充10%胎牛血清(FBS)和1%青霉素/链霉素。在37°C下扩增细胞,在5%CO2 培养箱中培养3天。
注意:慢病毒的产生必须在生物安全2级实验室中谨慎进行,使用经过调整的保护装备,包括一次性防护服,防护帽和手套。所有实验必须在带过滤尖端的层流罩(BSLII安全柜)下进行。所有含有慢病毒的溶液和所有使用的塑料/玻璃废物必须用70%乙醇或其他病毒灭活剂灭活。
- 准备18块145厘米的平板,每块板上接种1×106 HEK293细胞,在16 mL由Dulbecco的改良鹰培养基(DMEM)高葡萄糖丙酮酸盐组成的培养基中,补充10%胎牛血清(FBS)和1%青霉素/链霉素。在37°C下扩增细胞,在5%CO2 培养箱中培养3天。
- 细胞转染(第4天)
- 检查细胞的汇合度,确保它们已达到60%-65%汇合。
- 制备含有每个板的转染溶液:20.8μg目标质粒(p-导体,或p-Killer,或pCas9 #87904),4.8μg编码包膜的质粒(VSV-G,#8454),20.8μg质粒psPAX2(#12260)用于慢病毒包装,136μL磷酸钙并用H2O调节至最终体积1,000μL。滴加该溶液并在搅拌下加入1mL2x HEPES缓冲盐水(HBS)。
注意:不要同时为所有板准备混合物,以确保试剂的最佳制备;同时为六个板准备混合物,例如,对于18个板,为六个板准备三次混合。 - 在室温(RT)下孵育至少10分钟,并向细胞中滴加2mL溶液。将转染试剂匀浆,轻轻搅拌板后,向前,向上和向下搅拌,在37°C,5%CO2 下孵育至少5小时。
注意:从现在开始,直到慢病毒生产结束,请佩戴额外的保护装备,包括第二副手套,保护套和一次性塑料。 - 转染后五小时,从平板上取出培养基,并用PBS冲洗以除去转染试剂。加入12mL新鲜培养基,并在37°C,5%CO2下孵育48小时。
- 收集病毒颗粒(第6天)
- 从所有盘子中收集并池化培养基。在4°C下以800× g 离心5分钟,以沉淀细胞碎片。使用0.45μm过滤器过滤上清液(需要多个过滤器)。
- 在摆动的桶形转子中以68,300× g 在4°C下离心2小时。除去上清液,让试管在纸上的安全柜下倒置5-10分钟以除去尽可能多的液体,然后每个沉淀加入100μLHEK增殖培养基。在4°C下至少2小时后,通过上下移液重悬沉淀。将所有重悬的颗粒池化。
注意:在等分之前,颗粒可以在4°C下在培养基中放置过夜。 - 在10μL或25μL样品量(取决于用途)中等分慢病毒,并用液氮速冻。储存在-80°C。 不要冷冻已经解冻的等分试样。
- 对其他感兴趣的质粒重复步骤3.2至3.4,以产生LV-导体,LV-Killer和LV-Cas9。
注意:作为替代方案,慢病毒可以从公司或病毒设施购买。
4. 慢病毒滴定
注意:病毒滴定在HEK293细胞上进行。滴定在后续步骤中为每个细胞(无论慢病毒的批次如何)加入精确数量的慢病毒对于感兴趣的细胞非常重要。有效转导细胞的病毒颗粒的数量称为感染的多重性(MOI):因此MOI 10对应于每个细胞引入10个病毒颗粒。由于冷冻/解冻循环影响慢病毒的存活率,因此使用冷冻的慢病毒等分试样进行滴定,并且每个后续实验将用同一池的新等分试样进行。这里描述了一种滴定方法,但也可以使用其他方法。
- 在第1天,将每块板1 x 105 个细胞接种在五个35 mm的平板中,底部有玻璃盖玻片,两个35mm的板没有盖玻片。
- 在第2天,从没有盖玻片的两个平板中,收集并计数胰蛋白酶消化后的细胞数量,并确定每块板(N)的平均细胞量。
- 在增殖培养基中制备100μL以1/10稀释的慢病毒。用不同体积的稀释病毒转导五个培养板,从1到50μL稀释的病毒。对于该方案,使用以下体积转导五个板:1μL,5μL,10μL,20μL,50μL。在37°C下在5%CO2中孵育48小时。
- 在第4天,通过将盖玻片在室温下孵育20至30分钟在4%多聚甲醛中来固定细胞。对于LV-Cas9,在室温下用0.1%Triton X100在磷酸盐缓冲盐水(PBS)中透化细胞10分钟,在PBS-0.1%Triton X100-2%山羊血清 - 0.5%牛血清白蛋白(BSA)中饱和20分钟,在RT下用一抗抗V5(稀释1/400)标记45分钟,然后在室室用荧光二抗孵育30分钟。用Hoescht(PBS中的10μg/ mL)在室温下标记细胞核10分钟。
- 将盖玻片安装在载玻片上,并使用配备20倍物镜的荧光显微镜进行观察。对于左心室导引和左心室杀手,固定后直接进入细胞核标记并安装盖玻片。对于每个盖玻片,计算视野中的细胞核总数(细胞总数)和标记的细胞数(用V5或mCherry),并确定为每个盖玻片标记的细胞比例。
- 选择盖玻片,其中转导细胞的比例至少为10%且不超过50%。确定该盖玻片的转导效率(X),并注意用于获得该转导效率的稀释病毒(V,以μL为单位)的体积。
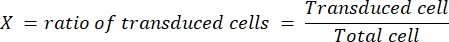
- 根据以下公式确定病毒的滴度(在感染性颗粒中,表示为ip / mL):

稀释因子是在步骤4.3中执行的慢病毒的稀释。我们常规获得LV-Cas9的最终滴度为1 x 109 ip / mL,LV-guide的最终滴度为1 x 10 10 ip / mL,在HEK细胞中测定。
5. 肌母细胞转导
注意:永生化成肌母细胞与先前产生的三种慢病毒连续转导。它们在由补充了20%FBS,2%青霉素/链霉素,2%Ultroser G的Ham's F10组成的增殖培养基中保持在低于50%的密度,并在37°C,5%CO2下培养。
- 根据以下公式,确定用LV-Cas9的MOI 10和LV-guides以及LV-killer的MOI 20治疗所选细胞数量的慢病毒体积:
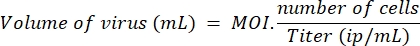
注意:在一项平行实验中,已经使用对照慢病毒(lenti-GFP)比较了成肌细胞和HEK的转导效率,并且我们已经确定,与HEK细胞相比,需要五倍的慢病毒来有效地转导成肌母细胞。因此,在HEK上测量的MOI 10对应于每个成肌细胞的两个病毒颗粒。此处使用的 MOI 是在 HEK 单元上计算的。 - 在第1天,在每孔100μL增殖培养基中用10,000个细胞接种96孔板。在第2天,通过添加步骤5.1中计算的适当体积的LV-导轨和LV-Cas9来转导安全柜下的细胞。将细胞放回培养箱,直到第7天。
注意:使用高于25的MOI,特别是对于LV-Cas9,可能不会改善编辑细胞的数量,因为在高浓度的慢病毒下细胞死亡率较高。 - 在第7天,进行胰蛋白酶消化并计数细胞。将细胞以40%至50%的汇合度接种在新板中,并将细胞返回到培养箱中。五小时后,以20的MOI(在步骤5.1中计算的体积)用左心室杀伤细胞转导细胞。转染后将细胞扩增5至10天,至少两次传代,并始终将它们保持在<50%的低汇合度。当细胞的生长恢复正常(由分裂时间估计)时,细胞已准备好进行下一步,即细胞克隆。
注意:转导后的增殖可能会慢一些。在此实验程序之前,可以通过估计细胞的分裂时间来确定正常的细胞生长。
6. 细胞克隆
注意:由于肌母细胞转导很困难,并且永远不会达到100%的效率,即使使用慢病毒,也需要细胞克隆才能获得完全校正的细胞系。这仅适用于永生化细胞,或可以在几周/几个月内培养和扩增的细胞。
- 胰蛋白酶消化并计数细胞。在10个细胞/ mL的增殖培养基中稀释细胞,并将细胞以1个细胞/孔接种在含有100μL/孔培养基的96孔板中。
注意:要播种的板的数量取决于预期基因编辑的概率,常规使用2至10个板。 - 监测细胞的生长,并在较大的板中逐渐扩增每个孔,直到达到至少35mm板,同时将成肌细胞的汇合度保持在50%以下。该步骤可持续2-6周,具体取决于所使用的细胞及其在孔中分离后生长的能力。
7. 克隆选择
注:执行此步骤是为了确定哪些不断增长的克隆已得到适当修改。
- 设计一组引物,由位于第一导引物(Primer_BeforeGuide1F)之前的引物和位于第二导联线(Primer_AfterGuide2R)之后的另一引物组成,以扩增包含假定修饰序列的区域。参见 表1 ,了解此处使用的引物。
- 从每个克隆中收集细胞,保留至少300,000个细胞以供将来扩增,并使用其余细胞上的任何标准方案提取基因组DNA。
- 如果大量克隆正在生长,要丢弃未编辑的克隆,请通过在同一管中汇集来自五个克隆的细胞来执行快速测试,以从该池中提取DNA并用PCR进行测试。根据需要重复尽可能多的克隆。然后,进一步分离包含编辑单元格的池以执行单独的分析。
- 按如下方式控制PCR的编辑。用1μL Primer_BeforeGuide1F,1μL Primer_AfterGuide2R,12.5μL聚合酶混合物,3μL基因组DNA和7.5μL H2O制备PCR反应。在1%琼脂糖凝胶上电泳以鉴定编辑的克隆。
- 通过排序控制编辑,如下所示。对所选克隆执行 Sanger 测序,以确认删除并确定在每个克隆中如何执行编辑。保留多个经过编辑的克隆,以确保只有靶基因被修饰,并负责观察到的生理效应,并保留一个未编辑的克隆,该克隆将在随后的实验中用作对照克隆(CTRL)。
- 展开选定的克隆。一旦每个克隆的汇合度达到约50%,胰蛋白酶消化细胞并将细胞接种在更大的培养皿中,直到产生足够的细胞来执行生化和功能表征(通常每个克隆超过1 x 106 ),并储存每个克隆的冷冻等分试样以备将来使用。
8. 编辑克隆的表征
注意:一旦通过DNA测序挑选并确认了几个克隆,就可以使用蛋白质印迹在蛋白质水平上确认靶向基因的缺失,如果该基因有功能性细胞测定,则可以在功能水平上确认靶基因的缺失。在 RYR1-KO的情况下,由于RyR1是钙通道,因此已在培养的细胞上使用钙成像进行功能表征。
- 编辑克隆中的蛋白质表达
注意:RyR1仅以分化的肌管10表示。已使用蛋白质印迹在肌管中评估其表达,以确认RyR1蛋白水平的缺失以及Cas9蛋白的缺失。- 在约1.76厘米2的表面上,在涂有层粘连蛋白的35毫米板上(表面对应于200μL层粘连蛋白滴在PBS中以10mg / mL的钙)的增殖培养基(如上所述,步骤5)中的板200 ,000个细胞。一旦细胞在37°C,5%CO 2下孵育2-3小时后粘附在板上,将培养基转移到由DMEM低葡萄糖+ 10%马血清+ 1%青霉素/链霉素组成的分化培养基上,并将细胞返回到培养箱中6天。
- 分化6天后,收集并用200μL补充蛋白酶抑制剂的RIPA裂解细胞。使用Folin Lowry方法测定蛋白质浓度11。
- 在Laemmli变性缓冲液中室室变性30分钟后,在5%-15%梯度丙烯酰胺凝胶上加载15μg蛋白质。电泳分离后,将蛋白质在0.8 V下转移到Immobilon P上4小时11。
- 在含有0.1%吐温20和5%脱脂奶粉的PBS中在RT下将膜饱和30分钟后,将膜与在同一缓冲液中稀释的一抗一抗一起在室温下孵育2小时或在4°C下过夜,用PBS-0.1%吐温20洗涤膜5分钟,并在室温下用二抗孵育膜1小时。使用的一抗是:针对V5标签(稀释度:1/5000)检测Cas9的抗体,抗GAPDH(稀释度:1/1000)作为负载对照,抗RyR1抗体12,13 (稀释:1/10.000),针对DHPR的α1亚基的抗体(稀释:1/1000)和针对肌球蛋白重链MF20的抗体(稀释:1/1000)。
- 用PBS-0.1%吐温20洗涤膜5分钟,干燥多余的液体并加入化学发光底物。按照底物提供者的建议进行检测化学发光信号。
- 编辑克隆的功能表征
注意:使用钙成像评估RyR1在由CTRL或KO克隆14产生的分化肌管中的功能。- 在涂有层粘连蛋白的35mm培养皿中心的0.2cm2 表面上板50,000个细胞(表面由50μL层粘连蛋白滴覆盖,在PBS中用钙以10mg / mL覆盖),并诱导分化6天,如步骤8.1.1所述。每次刺激准备三块板,进行生物一式三份。
- 用50μL氟4-直接,在分化培养基中稀释1:1稀释并在37°C下孵育30分钟。 用KREBS缓冲液以1mg / mL的速度用葡萄糖冲洗细胞两次。
- 使用倒置荧光显微镜或使用10倍物镜的共聚焦显微镜测量荧光变化。将板安装在显微镜的载物台上,并以每秒1帧的速度开始采集,持续90秒。
- 通过加入2mL KCl进行膜去极化(140mM终浓度)或2mL 4 CmC(500μM终浓度)用于RyR1直接刺激,除去剩余的KREB并在第25帧处刺激细胞。确保记录的现场至少存在10个肌管。
- 使用专用软件量化每个肌管中的荧光变化。选择每道菜至少10个肌管(理想情况下每个培养皿20-30个肌管)进行分析,在每个肌管的长轴上绘制一条线(或感兴趣区域(ROI)),并沿着这条线收集所有帧的荧光F。
- 确定初始荧光值 F0,对应于第 1 帧到第 24 帧。绘制荧光变化 (F-F0)/F0 作为 0 到 90 秒时间的函数。重复实验三次,以获得来自三种不同培养物的至少90个肌管的荧光变异。汇总90个肌管的所有结果,并计算每个时间范围内(F-F0)/F0的平均±SEM。量化每个刺激和每个克隆的钙释放峰值幅度。
结果
该协议应用于来自健康受试者15 的永生化成肌母细胞(所谓的HM细胞,用于人类成肌细胞),其中RyR1先前已被表征为16,以敲除编码 RyR1 蛋白的RYR1基因。指南RNA的设计是为了删除包含基因外显子101和内含子101部分的序列。预计删除部分外显子101将导致阅读框的中断。此外,外显子101编码蛋白质的孔,因此需要产生功能性钙通道。许多患有严重RyR1相关肌病...
讨论
在表征涉及病理学的未知功能基因的道路上,一个重要的步骤是开发相关的细胞模型来研究这些基因的功能。使用CRISPR / Cas9进行基因编辑是一个呈指数级增长的研究领域,这里介绍的敲除模型的开发是其使用最广泛的应用之一。在这种情况下,我们在这里提出了一种多功能方案,可以在任何感兴趣的基因中开发一种人类细胞系敲除,从而允许在相关的人类细胞模型中表征该基因。这里介绍的方案...
披露声明
作者没有利益冲突需要披露。
致谢
这项工作由法国肌病协会(AFM-Téléthon)和奥弗涅-罗纳阿尔卑斯大区(AURA)的赠款资助。
材料
| Name | Company | Catalog Number | Comments |
| Anti-CACNA1S antibody | Sigma-Aldrich | HPA048892 | Primary antibody |
| Blp I | NE BioLabs | R0585S | Restriction enzyme |
| CalPhos Mammalian Transfection Kit | Takara | 631312 | Transfection kit |
| Easy blot anti Mouse IgG | GeneTex | GTX221667-01 | HRP secondary antibody |
| Easy blot anti Rabbit IgG | GeneTex | GTX221666 | HRP secondary antibody |
| Fluo-4 direct | Molecular Probes | F10472 | Calcium imaging |
| GAPDH(14C10) Rabbit mAb | Cell Signaling Technology | #2118 | Primary antibody |
| HindIII | Fermentas | ER0501 | Restriction enzyme |
| InFusion HD Precision Plus | Takara | 638920 | Ligation kit |
| MasterMix Phusion High Fidelity with GC | ThermoFisher Scientific | F532L | Mix for PCR reaction with High fidelity Taq polymerase and dNTPs |
| Myosin Heavy Chain antibody | DHSB | MF20 | Primary antibody |
| NucleoBond Xtra Maxi EF | Macherey-Nagel | REF 740424 | Maxipreparation kit for purification of plasmids |
| NucleoSpin Gel and PCR Clean-up | Macherey-Nagel | 740609 | DNA purification |
| NucleoSpin Tissue | Macherey-Nagel | 740952 | Kit for DNA extraction from cell |
| One Shot Stbl3 Chemically Competent E. coli | ThermoFisher Scientific | C737303 | Chemically competent cells |
| Plasmid #87904 | Addgene | 87904 | Lentiviral plasmid encoding the SpCas9 (for LV-Cas9) |
| Plasmid #87919 | Addgene | 87919 | Lentiviral backbone for insertion of cassette with guides (for LV-guide-target) |
| Plasmid #12260 | Addgene | 12260 | Lentiviral plasmid encoding lentiviral packaging GAG POL |
| Plasmid #8454 | Addgene | 8454 | Lentiviral plasmid encoding envelope protein for producing lentiviral and MuLV retroviral particles |
| V5 Tag Monoclonal Antibody | Invitrogene | R96025 | Primary antibody |
| XL10-Gold Ultracompetent Cells | Agilent | 200317 | Chemically competent cells |
| Xma I | NE BioLabs | R0180S | Restriction enzyme |
参考文献
- Claussnitzer, M., Susztak, K. Gaining insight into metabolic diseases from human genetic discoveries. Trends in Genetics. 37 (12), 1081-1094 (2021).
- Fuster-García, C., García-Bohórquez, B., Rodríguez-Muñoz, A., Millán, J. M., García-García, G. Application of CRISPR tools for variant interpretation and disease modeling in inherited retinal dystrophies. Genes. 11 (5), 473 (2020).
- Modell, A. E., Lim, D., Nguyen, T. M., Sreekanth, V., Choudhary, A. CRISPR-based therapeutics: current challenges and future applications. Trends in Pharmacological Sciences. 43 (2), 151-161 (2022).
- Olson, E. N. Toward the correction of muscular dystrophy by gene editing. Proceedings of the National Academy of Sciences of the United States of America. 118 (22), (2021).
- Wu, X., Kriz, A. J., Sharp, P. A. Target specificity of the CRISPR-Cas9 system. Quantitative Biology. 2 (2), 59-70 (2014).
- Merienne, N., et al. The self-inactivating KamiCas9 system for the editing of CNS disease genes. Cell Reports. 20 (12), 2980-2991 (2017).
- Marty, I., Fauré, J. Excitation-contraction coupling alterations in myopathies. Journal of Neuromuscular Diseases. 3 (4), 443-453 (2016).
- Concordet, J. P., Haeussler, M. CRISPOR: intuitive guide selection for CRISPR/Cas9 genome editing experiments and screens. Nucleic Acids Research. 46, 242-245 (2018).
- Sun, Y., Sriramajayam, K., Luo, D., Liao, D. J. A quick, cost-free method of purification of DNA fragments from agarose gel. Journal of Cancer. 3, 93-95 (2012).
- Flucher, B. E., Conti, A., Takeshima, H., Sorrentino, V. Type 3 and type 1 ryanodine receptors are localized in triads of the same mammalian skeletal muscle fibers. The Journal of Cell Biology. 146 (3), 621-630 (1999).
- Hess, H. H., Lees, M. B., Derr, J. E. A linear Lowry--Folin assay for both water-soluble and sodium dodecyl sulfate-solubilized proteins. Analytical Biochemistry. 85 (1), 295-300 (1978).
- Garibaldi, M., et al. Dusty core disease' (DuCD): expanding morphological spectrum of RYR1 recessive myopathies. Acta Neuropathologica Communications. 7 (1), 3 (2019).
- Marty, I., et al. Biochemical evidence for a complex involving Dihydropyridine receptor and Ryanodine receptor in triad junctions of skeletal muscle. Proceedings of the National Academy of Sciences of the United States of America. 91 (6), 2270-2274 (1994).
- Oddoux, S., et al. Triadin deletion induces impaired skeletal muscle function. Journal of Biological Chemistry. 284 (50), 34918-34929 (2009).
- Mamchaoui, K., et al. Immortalized pathological human myoblasts: towards a universal tool for the study of neuromuscular disorders. Skeletal Muscle. 1, 34 (2011).
- Cacheux, M., et al. Functional characterization of a central core disease RyR1 mutation (p.Y4864H) associated with quantitative defect in RyR1 protein. Journal of Neuromuscular Diseases. 2 (4), 421-432 (2015).
- Luis, A. The old and the new: Prospects for non-integrating lentiviral vector technology. Viruses. 12 (10), 1103 (2020).
- Leenay, R. T., Beisel, C. L. Deciphering, communicating, and engineering the CRISPR PAM. Journal of Molecular Biology. 429 (2), 177-191 (2017).
- Salmon, P., Trono, D. Production and titration of lentiviral vectors. Current Protocols in Neurosciences. , (2006).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。