Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Разработка нокаутирующих мышечных клеточных линий с использованием лентивирусно-опосредованного редактирования генов CRISPR/Cas9
В этой статье
Резюме
Протокол описывает, как генерировать нокаутирующие миобласты с помощью CRISPR/Cas9, начиная от проектирования направляющих РНК до клеточного клонирования и характеристики нокаутирующих клонов.
Аннотация
Одним из важных применений кластерных регуляторных межпространственных коротких палиндромных повторов (CRISPR)/Cas 9 является разработка нокаутирующих клеточных линий, в частности, для изучения функции новых генов/белков, связанных с заболеванием, выявленных в ходе генетической диагностики. Для развития таких клеточных линий необходимо распутать две основные проблемы: вставка инструментов CRISPR (Cas9 и направляющая РНК) с высокой эффективностью в выбранные клетки и ограничение активности Cas9 специфической делецией выбранного гена. Протокол, описанный здесь, посвящен вставке инструментов CRISPR в трудно трансфектируемые клетки, такие как мышечные клетки. Этот протокол основан на использовании лентивирусов, продуцируемых с плазмидами, общедоступными, для которых все этапы клонирования описаны для нацеливания на интересующий ген. Контроль активности Cas9 был выполнен с использованием адаптации ранее описанной системы под названием KamiCas9, в которой трансдукция клеток лентивирусом, кодирующим направляющую РНК, нацеленную на Cas9, позволяет прогрессировать отмену экспрессии Cas9. Этот протокол был применен к разработке RYR1-нокаутирующей линии мышечных клеток человека, которая была дополнительно охарактеризована на белковом и функциональном уровне, чтобы подтвердить нокаут этого важного кальциевого канала, участвующего в внутриклеточном высвобождении кальция мышц и в связи возбуждение-сокращение. Процедура, описанная здесь, может быть легко применена к другим генам в мышечных клетках или в других трудно трансфектируемых клетках и производить ценные инструменты для изучения этих генов в клетках человека.
Введение
С прогрессом секвенирования генов и идентификацией мутаций в генах неизвестных функций в конкретной ткани разработка соответствующих клеточных моделей для понимания функции нового гена-мишени и подтверждения его участия в связанных патофизиологических механизмах представляет собой важный инструмент. Кроме того, эти модели имеют большое значение для будущих терапевтических разработок 1,2 и представляют собой интересную альтернативу разработке нокаутированных моделей животных в прямой линии с международными рекомендациями по сокращению использования животных в экспериментах. Редактирование генов с использованием CRISPR / Cas9 является одним из самых мощных инструментов, доступных в настоящее время, что позволило разработать множество нокаут-моделей, а целевая проверка генов с использованием CRISPR / Cas9 является одним из наиболее широко используемых применений CRISPR / Cas93. Успех редактирования генов зависит от способности вводить инструменты CRISPR (направляющие РНК и нуклеазу Cas9) в модель клеток-мишеней, что может быть проблемой во многих труднопереводимых клетках, таких как мышечные клетки4. Эта проблема может быть преодолена с помощью вируса, обычно лентивируса, который имеет большое преимущество для эффективного преобразования многих типов клеток и доставки их трансгена. Но его основным недостатком является интеграция трансгена в геном клетки-хозяина, что приводит к потенциальному изменению генов, локализованных в месте интеграции, и к постоянной экспрессии трансгена, что в случае нуклеазы Cas9 приведет к разрушительным последствиям5. Умный раствор был предложен Мерьенн и его коллегами6, который состоит из введения в клетки направляющей РНК, нацеленной на сам ген Cas9, что приводит к инактивации Cas9. Адаптация этой стратегии представлена здесь в виде удобного и универсального протокола, позволяющего нокаутировать практически любой ген в труднотрансфектируемых клетках.
Целью представленного здесь протокола является индуцирование инактивации гена, представляющего интерес, в увековеченных мышечных клетках. Его можно использовать для нокаутирования любого гена, представляющего интерес, в различных типах увековеченных клеток. Протокол, описанный здесь, содержит шаги по разработке направляющих РНК и их клонированию в лентивирусные плазмиды, для производства инструментов CRISPR в лентивирусных векторах, для трансдукции клеток с различными лентивирусами и для клонирования клеток для получения однородной отредактированной клеточной линии.
Используя этот протокол, были разработаны увековеченные клетки скелетных мышц человека с делецией рецептора рианодина типа 1 (RyR1), важного кальциевого канала, участвующего во внутриклеточном высвобождении кальция и сокращении мышц7. Нокаут (KO) гена был подтвержден на уровне белка с использованием вестерн-блоттинга и на функциональном уровне с использованием кальциевой визуализации.
протокол
Биопсии мышц были получены из Банка тканей для исследований (Myobank, партнер в сети ЕС EuroBioBank, Париж, Франция) в соответствии с европейскими рекомендациями и французским законодательством. Письменное информированное согласие было получено от всех лиц. Увековеченные миобласты были любезно изготовлены доктором В. Мули (Институт миологии, Париж, Франция), а протоколы были одобрены этическим комитетом Института миологии (MESRI, n AC-2019-3502).
1. Дизайн руководства CRISPR
- Определите область гена, подлежащего удалению. Найдите его геномную последовательность с помощью инструментов браузера генома, таких как ensembl.org или genome.ucsc.edu и определите хромосомные координаты двух областей, чтобы найти направляющую РНК (гРНК) по обе стороны области, подлежащую удалению.
- Для гена, используемого здесь, получите последовательность FASTA гена RYR1 и последовательность экзона 101 следующим образом. В ensembl выполните поиск RYR1 в самой последней версии генома человека, выберите первую запись и нажмите на расшифровку кодирующей последовательности белка. Затем нажмите на Exons , чтобы перенаправить на список экзонов гена.
- Нажмите «Загрузить последовательность» и выберите «Только геномная последовательность », чтобы загрузить полную консенсусную последовательность всего гена. Прокрутите вниз список экзонов и интронов гена и выберите целевой (ы).
- Найдите соответствующую нуклеотидную последовательность в гене. Выберите нуклеотидные последовательности интронов непосредственно вверх и вниз по течению экзона, которые будут удалены, которые будут использоваться для поиска гРНК.
- Разработайте две гРНК, называемые здесь руководством 1 и руководством 2, в регионах, определенных на этапе 1.1 (интроны вверх и вниз по течению региона, подлежащего удалению), используя онлайн-инструменты, такие как Crispor.tefor.net8. Выберите две гРНК, разделенные несколькими сотнями пар оснований (bp), причем последовательность каждой гРНК составляет ровно 20 нуклеотидов в длину без смежного мотива протоспейсера (PAM). Выберите лучшие доступные направляющие, чтобы ограничить выход за пределы цели. Пример проектирования направляющих приведен на рисунке 1 .
- Ожидается, что последовательность между двумя направляющими будет удалена и, что приведет к выбиванию интересующего гена, таким образом, выберите положение двух направляющих таким образом, чтобы оно удалило существенную последовательность или существенный экзон в интересующем гене. Убедитесь, что удаленная последовательность/экзон не присутствует исключительно в альтернативном транскрипте гена и/или что он кодирует важную часть белка, таким образом, его делеция приведет к функциональному нокауту.
ПРИМЕЧАНИЕ: Хотя предполагается, что участок расщепления Cas9 произойдет в 3 bp выше по течению от смежного мотива протоспейсера (PAM), расщепление на большем расстоянии также может произойти, поэтому хорошее решение не должно зависеть от точной локализации места расщепления, такого как расщепление в интроне.
- Ожидается, что последовательность между двумя направляющими будет удалена и, что приведет к выбиванию интересующего гена, таким образом, выберите положение двух направляющих таким образом, чтобы оно удалило существенную последовательность или существенный экзон в интересующем гене. Убедитесь, что удаленная последовательность/экзон не присутствует исключительно в альтернативном транскрипте гена и/или что он кодирует важную часть белка, таким образом, его делеция приведет к функциональному нокауту.
- Определите последовательность обратного комплемента (RC) для каждой гРНК без PAM, чтобы иметь следующие последовательности: Guide 1 и Guide 1-RC, Guide 2 и Guide 2-RC.
- Закажите праймеры, представленные в таблице 1 , для выполнения клонирования плазмид. На протяжении всего протокола применяют праймеры в концентрации 10 нМ в стерильномН2О.
ПРИМЕЧАНИЕ: Последовательности, добавленные к гРНК в этих праймерах (выделенные жирным шрифтом и подчеркнутые), соответствуют последовательности плазмиды до и после направляющей, промоторной и трансактивирующей CRISPR РНК (тракрРНК) соответственно и не должны модифицироваться для обеспечения хорошего перекрытия между праймерами и плазмидой.
2. Плазмидное клонирование
ПРИМЕЧАНИЕ: На этом этапе гРНК будут вставлены в плазмидную основу для производства лентивируса. Кассета, кодирующая две гРНК, сначала производится путем последовательных полимеразных цепных реакций (ПЦР) с использованием перекрывающихся праймеров. Затем новая кассета вставляется в лентивирусную основную плазмиду #87919.
- Получите следующие плазмиды: плазмиду #87919, кодирующую направляющие РНК CRISPR в лентивирусном векторе и плазмиду #87904, кодирующую последовательность SpCas9 в лентивирусном векторе.
- Кассетная конструкция
ПРИМЕЧАНИЕ: Протокол о клонировании кратко изложен в Рисунок 2.- Запустите реакцию ПЦР (А) с 2 мкл плазмиды #87919, 2 мкл primer_XmaIF, 2 мкл primer_Guide1R, 25 мкл полимеразной смеси и 19 мклН2О. Запустите следующую программу ПЦР (программа 1): начальная денатурация 5 мин при 98 °C, за которой следуют 30 циклов: 30 с при 98 °C, 30 с при 60 °C, 1 мин 45 с при 72 °C и конечное удлинение 7 мин при 72 °C. Tm грунтовок, описанных на шаге 1.4, составляет 60 °C.
ПРИМЕЧАНИЕ: Хотя время удлинения в программе 1 кажется довольно длительным, это время удлинения было выбрано для обеспечения производства достаточного количества материала нужного размера. Действительно, из-за повторяющихся последовательностей в плазмиде (последовательность тракрРНК после проводников РНК повторяется три раза в плазмиде #87919) ПЦР-амплификация ожидаемой ДНК затруднена, и в последовательных ПЦР образуются дополнительные более мелкие полосы. Таким образом, из-за конкуренции между различными продуктами ПЦР для длинного фрагмента было увеличено либо время удлинения (в пользу самого длинного и достаточного количества очищенного материала в конце), либо для длинного фрагмента была использована ПЦР (программа 2), как описано для ПЦР (заключительная) на этапе 2.2.6). - Отделите продукты ПЦР на 1% агарозном геле в буфере Tris-Borate-EDTA (TBE), рассейте и очистите фрагмент 300 bp. Выполните очистку с помощью специального комплекта в соответствии с инструкцией производителя, с элюированием в конечном объеме 20 мкл. Либо используйте очищенный фрагмент напрямую, либо храните при -20 °C для шага 2.2.5.
- Запустите реакцию ПЦР (B) с 2 мкл плазмиды #87919, 2 мкл primer_Guide1F, 2 мкл primer_Guide2R, 25 мкл полимеразной смеси и 19 мклH2Oс использованием программы ПЦР 1. Отделите продукты ПЦР на 1% агарозном геле в ТБЭ, рассейте и очистите фрагмент 400 bp в 20 мкл буфера элюирования. Либо используйте очищенный фрагмент непосредственно, либо храните при -20 °C для шага 2.2.5.
- Запустите реакцию ПЦР (С) с 2 мкл плазмиды #87919, 2 мкл primer_Guide2F, 2 мкл primer_BlpIR, 25 мкл полимеразной смеси и 19 мклH2Oс использованием программы ПЦР 1. Отделите продукты ПЦР на 1% агарозном геле в буфере TBE. Акцизируйте и очистите фрагмент 600 bp в 20 мкл буфера элюирования. Либо используйте очищенный фрагмент непосредственно, либо храните при -20 °C для шага 2.2.6.
ПРИМЕЧАНИЕ: Может быть виден еще один фрагмент при 900 bp из-за гибридизации праймера на повторяющихся областях в плазмиде, как описано в ПРИМЕЧАНИИ к шагу 2.2.1. Если она присутствует, эта полоса должна быть отброшена. - Запустите реакцию ПЦР (D) с 2 мкл элюирования ПЦР А (со стадии 2.2.2), 2 мкл элюирования ПЦР В (со стадии 2.2.3), 2 мкл primer_XmaIF, 2 мкл primer_Guide2R, 25 мкл полимеразной смеси и 19 мклH2Oс программой ПЦР 1. Отделить продукты ПЦР на 1% агарозном геле в буфере TBE; иссечь и очистить фрагмент 700 bp в 20 мкл буфера элюирования. Либо используйте очищенный фрагмент непосредственно, либо храните при -20 °C для шага 2.2.6.
ПРИМЕЧАНИЕ: Другие полосы могут быть видны при >1000 bp, 400 bp и 300 bp из-за гибридизации праймеров на повторяющихся областях и должны быть отброшены. - Запустить ПЦР (конечную) реакцию с 4 мкл элюирования ПЦР С (со стадии 2.2.4), 4 мкл элюирования ПЦР D (со стадии 2.2.5), 4 мкл primer_XmaIF, 4 мкл primer_BlpIR, 50 мкл полимеразной смеси и 34 мклН2О. Используется следующая программа (программа ПЦР 2): начальная денатурация в течение 5 мин при 98 °C; шесть циклов: 30 с при 98 °C, 30 с при 66 °C (снижение температуры гибридизации на 1 °C за цикл), 1 мин 45 при 72 °C; 35 циклов: 30 с при 98 °C, 30 с при 60 °C, 1 мин 45 при 72 °C и конечное удлинение 5 мин при 72 °C.
ПРИМЕЧАНИЕ: Программа ПЦР для этой окончательной амплификации представляет собой ПЦР с касанием, отличающуюся от предыдущей, из-за большого размера конечной амплификации, которая содержит две повторяющиеся последовательности тракрРНК сразу после каждого проводника. - Отделить продукты ПЦР на 1% агарозном геле в буфере TBE; иссечь и очистить конечную кассету, которая мигрирует примерно на 1 300 bp в 20 мкл буфера элюирования. Количественно оцените элюированный продукт. Либо используйте очищенный фрагмент непосредственно, либо храните при температуре -20 °C для этапа 2.3.2.1. Это конечный продукт, который будет вставлен в лентивирусную плазмиду.
ПРИМЕЧАНИЕ: Другие фрагменты могут быть видны при >1000 bp, 400 bp и 300 bp, что соответствует неполным фрагментам ПЦР, которые должны быть отброшены.
- Запустите реакцию ПЦР (А) с 2 мкл плазмиды #87919, 2 мкл primer_XmaIF, 2 мкл primer_Guide1R, 25 мкл полимеразной смеси и 19 мклН2О. Запустите следующую программу ПЦР (программа 1): начальная денатурация 5 мин при 98 °C, за которой следуют 30 циклов: 30 с при 98 °C, 30 с при 60 °C, 1 мин 45 с при 72 °C и конечное удлинение 7 мин при 72 °C. Tm грунтовок, описанных на шаге 1.4, составляет 60 °C.
- Вставка кассеты гРНК в лентивирусную плазмидную магистраль.
- Линеаризация плазмиды путем двойного переваривания плазмиды No87919 с ферментами рестрикции XmaI и BlpI.
- Готовят реакцию с 15 мкл рекомендуемого буфера, 15 мкл плазмиды (1 мкг/мкл), 7,5 мкл фермента BlpI (при 10 Ед/мкл), 7,5 мкл фермента XmaI (при 10 Ед/мкл) и 112,5 мклН2О. Инкубата в течение 1 ч при 37 °С, а затем в течение 20 мин при 65 °С. Загрузите общее количество на 1% агарозный гель, вырежьте и очистите плазмиду ~10 кб с помощью соответствующего набора. Элюирование выполняют в 20 мкл буфера, а элюированный продукт количественно определяют с помощью измерения оптической плотности.
ПРИМЕЧАНИЕ: Используйте соответствующий протокол очистки для больших фрагментов ДНК, такой как протокол, описанный Sun и coll9.
- Готовят реакцию с 15 мкл рекомендуемого буфера, 15 мкл плазмиды (1 мкг/мкл), 7,5 мкл фермента BlpI (при 10 Ед/мкл), 7,5 мкл фермента XmaI (при 10 Ед/мкл) и 112,5 мклН2О. Инкубата в течение 1 ч при 37 °С, а затем в течение 20 мин при 65 °С. Загрузите общее количество на 1% агарозный гель, вырежьте и очистите плазмиду ~10 кб с помощью соответствующего набора. Элюирование выполняют в 20 мкл буфера, а элюированный продукт количественно определяют с помощью измерения оптической плотности.
- Лигат кассеты гРНК и плазмиды. Готовят реакционную смесь с кассетой гРНК со стадии 2.2.7 и линеаризированной плазмидой со стадии 2.3.1.1, добавляют 2 мкл фермента иН2О, чтобы иметь конечный объем 10 мкл. Инкубат в течение 15 мин при 50 °С для получения конечной плазмиды, называемой p_guides.
ПРИМЕЧАНИЕ: Количество кассеты ДНК должно составлять от 50 до 100 нг, а количество плазмиды - от 100 до 200 нг с молярным соотношением 2:1.
- Линеаризация плазмиды путем двойного переваривания плазмиды No87919 с ферментами рестрикции XmaI и BlpI.
- Используйте 2 мкл свежеприготовленной плазмиды для преобразования химически компетентной кишечной палочки , такой как Stbl3 (50 мкл) или XL10-Gold, и наносите на агаровую пластину LB со 100 мкг/мл ампициллина после 1 ч роста при 37 °C без антибиотика. Инкубировать при 37 °C в течение ночи. Выберите несколько колоний и выполните мини-подготовку, используя коммерческий комплект, следуя инструкциям производителя.
- Выполните амплификацию ПЦР на днк минипрепа с Primer_XmaI и Primer_BlpR с помощью программы ПЦР 1 (см. Рисунок 2). Отделите продукты ПЦР на 1% агарозном геле в буфере TBE. Выберите несколько колоний (~5) с полосой ожидаемого размера около 1300 bp.
- Выполняют секвенирование ДНК выбранных колоний, используя Primer_XmaI или Primer_BlpR, для подтверждения правильной вставки кассеты гРНК.
ПРИМЕЧАНИЕ: Одна из проверенных последовательностей колоний дополнительно используется в исследовании, а плазмида называется p_guides. - Повторите шаг 2.2 (конструкция кассеты) с Primers-Killer F и R, а также Primers-mCherry F и R. Используйте одну колонию, проверенную последовательностью, для дальнейшего анализа. Плазмида называется p_Killer.
3. Лентивирусная продукция
- Производят и очищают большое количество всех необходимых плазмид с помощью комплекта макси-подготовки без эндотоксинов в соответствии с инструкциями производителя. Приготовьте аликвоты при 2 мкг/мкл. Хранить при -20 °C
- Подготовка клеток (День 1)
- Приготовьте 18 пластин по 145 см, посеянных 1 х 106 клетками HEK293 на пластину в 16 мл среды, состоящей из модифицированной орлиной среды Дульбекко (DMEM) с высоким содержанием пирувата глюкозы, дополненного 10% фетальной бычьей сывороткой (FBS) и 1% пенициллином / стрептомицином. Усиливают клетки при 37 °C, в 5% CO2 инкубаторе в течение 3 дней.
ПРИМЕЧАНИЕ: Производство лентивируса должно осуществляться с осторожностью в лаборатории уровня биобезопасности 2 с использованием адаптированных средств защиты, включая одноразовый защитный костюм, защитный колпачок и перчатки. Все эксперименты должны проводиться под ламинарной проточной вытяжкой (шкаф безопасности BSLII) с наконечниками фильтров. Весь раствор, содержащий лентивирусы, и все использованные пластиковые/стеклянные отходы должны быть инактивированы этанолом 70% или другим инактиватором вируса.
- Приготовьте 18 пластин по 145 см, посеянных 1 х 106 клетками HEK293 на пластину в 16 мл среды, состоящей из модифицированной орлиной среды Дульбекко (DMEM) с высоким содержанием пирувата глюкозы, дополненного 10% фетальной бычьей сывороткой (FBS) и 1% пенициллином / стрептомицином. Усиливают клетки при 37 °C, в 5% CO2 инкубаторе в течение 3 дней.
- Трансфекция клеток (День 4)
- Проверьте слияние ячеек, чтобы убедиться, что они достигли 60-65% слияния.
- Приготовьте трансфекционный раствор, содержащий для каждой пластины: 20,8 мкг интересующей плазмиды (p-гиды, или p-киллер, или pCas9 #87904), 4,8 мкг плазмиды, кодирующей оболочку (VSV-G, #8454), 20,8 мкг плазмиды psPAX2 (#12260) для лентивирусной упаковки, 136 мкл фосфата кальция и отрегулируйтеh2Oдо конечного объема 1000 мкл. Добавьте этот раствор по каплям и под перемешиванием до 1 мл 2x HEPES-буферизованного физиологического раствора (HBS).
ПРИМЕЧАНИЕ: Не готовьте смесь для всех пластин одновременно, чтобы обеспечить оптимальную подготовку реагентов; приготовьте смесь для шести тарелок одновременно, например, для 18 тарелок, приготовьте три раза смесь для шести тарелок. - Инкубировать при комнатной температуре (RT) в течение не менее 10 мин и добавлять 2 мл раствора по каплям в клетки. Гомогенизируют трансфекционный реагент с мягким перемешиванием пластины назад, вперед, вверх и вниз, инкубируют при 37 °C, 5% CO2 в течение не менее 5 ч.
ПРИМЕЧАНИЕ: С этого момента и до конца производства лентивируса носите дополнительное защитное снаряжение, включая вторую пару перчаток, защитные рукава и одноразовый пластрон. - Через пять часов после трансфекции удалите среду с пластин и промойте PBS, чтобы избавиться от трансфекционных реагентов. Добавить 12 мл свежей среды и инкубировать 48 ч при 37 °C, 5% CO2.
- Сбор вирусных частиц (День 6)
- Соберите и объедините среду со всех тарелок. Центрифуга при 800 х г в течение 5 мин при 4 °С, до гранулированного клеточного мусора. Фильтруйте супернатант с помощью фильтра 0,45 мкм (требуется несколько фильтров).
- Центрифуга при 68 300 х г в течение 2 ч при 4 °C в качающемся роторе ковша. Удалите супернатант и дайте трубкам перевернуться под шкафом безопасности на бумаге в течение 5-10 минут, чтобы удалить как можно больше жидкости, а затем добавьте 100 мкл пролиферационной среды HEK на гранулу. По крайней мере через 2 ч при 4 °C повторно суспендируйте гранулы путем пипетки вверх и вниз. Объедините все повторно суспендированные гранулы.
ПРИМЕЧАНИЕ: Гранулы можно оставить в среде на ночь при температуре 4 °C перед аликотированием. - Лентивирус Аликвот в размере образца 10 мкл или 25 мкл (в зависимости от применения) и мгновенно замораживает жидким азотом. Хранить при температуре -80 °C. Не замораживайте аликвоту, которая была разморожена.
- Повторите шаги с 3.2 по 3.4 с другими интересующими плазмидами, чтобы получить LV-направляющие, LV-Killer и LV-Cas9.
ПРИМЕЧАНИЕ: В качестве альтернативы лентивирусы можно приобрести у компании или вирусного учреждения.
4. Титрование лентивируса
ПРИМЕЧАНИЕ: Титрование вируса проводится на клетках HEK293. Титрование важно включить в последующие этапы точное количество лентивирусов на клетку (независимо от партии лентивируса) для интересующих клеток. Количество вирусных частиц, которые эффективно трансдуцируют клетку, называется множественностью инфекции (MOI): MOI 10, таким образом, соответствуют введению 10 вирусных частиц на клетку. Поскольку цикл замораживания/оттаивания влияет на жизнеспособность лентивируса, титрование проводят с замороженной лентивирусной аликвотой, и каждый последующий эксперимент будет проводиться с новой аликвотой того же пула. Один метод титрования описан здесь, но могут быть использованы и другие методы.
- На 1-й день высевают 1 х 105 ячеек на пластину в пяти 35-мм пластинах со стеклянными крышками на дне и двух 35-мм пластинах без чехлов.
- На 2-й день из двух пластин без обшивки соберите и подсчитайте количество клеток после трипсинизации и определите среднее количество клеток на пластину (N).
- Готовят 100 мкл лентивируса, разведенного на 1/10 в пролиферационной среде. Трансдуцируют пять культивируемых пластинок с различными объемами разбавленного вируса, от 1 до 50 мкл разбавленного вируса. Для этого протокола используют следующие объемы для преобразования пяти пластин: 1 мкл, 5 мкл, 10 мкл, 20 мкл, 50 мкл. Инкубировать в течение 48 ч при 37 °C, в 5% CO2.
- На 4-й день фиксируют клетки путем инкубации покровных пластинок при RT в течение 20-30 мин в 4% параформальдегиде. Для LV-Cas9 пропитывают клетки 0,1% тритона X100 в фосфатном буферном физиологическом растворе (PBS) в течение 10 мин при RT, насыщают в PBS-0,1% Triton X100-2% Козьей сыворотки - 0,5% бычьего сывороточного альбумина (BSA) в течение 20 мин при RT и этикетку в течение 45 мин при RT с первичным антителом анти-V5 (разведение 1/400) с последующей 30-мин инкубацией в RT с флуоресцентным вторичным антителом. Маркируйте ядра Хештом (10 мкг/мл в PBS) в течение 10 мин при RT.
- Установите крышки на слайд и наблюдайте с помощью флуоресцентного микроскопа, оснащенного 20-кратным объективом. Для LV-направляющих и LV-Killer после фиксации переходят непосредственно к маркировке ядер и монтируют крышки. Для каждого облицовочного листа подсчитайте общее количество ядер (общее количество клеток) в поле зрения и количество помеченных клеток (либо V5, либо mCherry) и определите соотношение клеток, помеченных для каждого облицовочного листа.
- Выберите крышку, в которой соотношение трансдуцированных клеток составляет не менее 10% и не превышает 50%. Определите эффективность трансдукции (X) для этого покровного листа и обратите внимание на объем разбавленного вируса (V, в мкл), используемого для получения этой эффективности трансдукции.
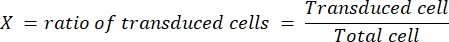
- Определяют титр (в инфекционных частицах, представленных в виде ip/mL) вируса по следующей формуле:

Коэффициент разбавления представляет собой разбавление лентивируса, выполненное на стадии 4.3. Мы регулярно получаем окончательный титр 1 x 109 ip/mL для LV-Cas9 и 1 x 1010 ip/mL для LV-направляющих, определяемый в ячейках HEK.
5. Миобластная трансдукция
ПРИМЕЧАНИЕ: Увековеченные миобласты последовательно трансдуцируются тремя ранее продуцированными лентивирусами. Они поддерживаются при плотности ниже 50% в пролиферационной среде, состоящей из F10 Хама, дополненной 20% FBS, 2% пенициллина / стрептомицина, 2% Ultroser G и культивируемой при 37 ° C, 5% CO2.
- Определить объем лентивируса необходимо для лечения выбранного количества клеток с помощью MOI 10 для LV-Cas9 и LV-гидов и MOI 20 для LV-киллера, по следующей формуле:
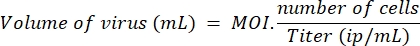
ПРИМЕЧАНИЕ: В параллельном эксперименте эффективность трансдукции сравнивалась на миобластах и HEK с использованием контрольного лентивируса (lenti-GFP), и мы определили, что для эффективной трансдукции миобласта требуется в пять раз больше лентивирусов по сравнению с клеткой HEK. Таким образом, MOI 10, измеренный на HEK, соответствует двум вирусным частицам на миобласт. Используемые здесь MOI рассчитываются на клетках HEK. - На 1-й день высевают 96-луночные пластины с 10 000 клеток в 100 мкл пролиферационной среды на лунку. На 2-й день переведите ячейки под шкаф безопасности, добавив соответствующий объем LV-направляющих и LV-Cas9, рассчитанных на шаге 5.1. Верните клетки в инкубатор до 7 дня.
ПРИМЕЧАНИЕ: Использование MOI выше 25, особенно для LV-Cas9, может не улучшить количество отредактированных клеток из-за более высокой гибели клеток при высокой концентрации лентивируса. - На 7 день выполняют трипсинизацию и подсчитывают клетки. Засейте клетки при слиянии от 40% до 50% в новую пластину и верните клетки в инкубатор. Через пять часов трансдуцируют клетки с помощью LV-киллера при MOI 20 (объем, рассчитанный на шаге 5.1). Амплируйте клетки в течение 5-10 дней после трансфекции, по крайней мере, в течение двух проходов, и всегда поддерживайте их при низкой конфюминации <50%. Клетки готовы к следующему этапу, клеточному клонированию, когда их рост возвращается к норме (оценивается по времени деления).
ПРИМЕЧАНИЕ: Пролиферация может быть немного медленнее после трансдукции. Нормальный рост клеток может быть определен до этой экспериментальной процедуры путем оценки времени деления клеток.
6. Клеточное клонирование
ПРИМЕЧАНИЕ: Поскольку трансдукция миобластов затруднена и никогда не достигает 100% эффективности, даже при использовании лентивируса требуется клеточное клонирование, чтобы получить полностью скорректированную клеточную линию. Это возможно только с увековеченными клетками или клетками, которые могут быть культивированы и усилены в течение нескольких недель / месяцев.
- Трипсинизируют и подсчитывают клетки. Разбавьте клетки в пролиферационной среде при 10 клетках/мл и засейте клетки в 1 клетку/лунку в 96-луночных пластинах, содержащих 100 мкл/лунку среды.
ПРИМЕЧАНИЕ: Количество пластин, подлежащих посеву, зависит от вероятности ожидаемого редактирования генов, обычно используется от 2 до 10 пластин. - Следите за ростом клеток и постепенно усиливайте каждую лунку в более крупной пластине, пока не достигнете по крайней мере 35 мм пластины, сохраняя при этом слияние миобластов ниже 50%. Этот шаг может длиться 2-6 недель, в зависимости от используемых клеток и их способности расти после выделения в колодце.
7. Выбор клонирования
ПРИМЕЧАНИЕ: Этот шаг выполняется для определения того, какие из растущих клонов были соответствующим образом изменены.
- Спроектируйте набор грунтовок, состоящих из грунтовки, расположенной перед первой направляющей (Primer_BeforeGuide1F), и другой грунтовки, расположенной после второй направляющей (Primer_AfterGuide2R), чтобы усилить область, заключающую предположительно измененную последовательность. Грунтовки, используемые здесь, см. в таблице 1 .
- Соберите клетки из каждого клона, сохраните не менее 300 000 клеток для будущей амплификации и извлеките геномную ДНК, используя любой стандартный протокол на оставшихся клетках.
- Если растет большое количество клонов, чтобы отбросить неотредактированные, выполните экспресс-тест, объединив клетки из пяти клонов в одной трубке, чтобы извлечь ДНК из этого пула и протестировать с помощью ПЦР. Повторите с таким количеством клонов, сколько необходимо. Затем отделите пулы, которые содержали отредактированные клетки, для выполнения индивидуального анализа.
- Контролируйте редактирование с помощью ПЦР следующим образом. Готовят реакцию ПЦР с 1 мкл Primer_BeforeGuide1F, 1 мкл Primer_AfterGuide2R, 12,5 мкл полимеразной смеси, 3 мкл геномной ДНК и 7,5 мклН2О. Амплифицируют в термоциклере в соответствии с инструкциями производителя и параметрами праймеров. Используйте 1% агарозный гель для идентификации отредактированных клонов.
- Управляйте редактированием с помощью последовательности следующим образом. Выполните секвенирование выбранных клонов Сэнгером, чтобы подтвердить удаление и определить, как редактирование было выполнено в каждом клоне. Сохраните более одного отредактированного клона, чтобы убедиться, что только целевой ген был модифицирован и отвечает за наблюдаемый физиологический эффект, и сохраните неотредактированный клон, который будет использоваться в качестве контрольного клона (CTRL) в последующих экспериментах.
- Разверните выбранные клоны. Как только слияние каждого клона достигнет около 50%, трипсинизируют клетки и помещают клетки в большую чашку, пока не будет произведено достаточное количество клеток для выполнения биохимических и функциональных характеристик (обычно более 1 х 106 на клон), и храните замороженные аликвоты каждого клона для будущего использования.
8. Характеристика отредактированных клонов
ПРИМЕЧАНИЕ: После того, как несколько клонов были выбраны и подтверждены секвенированием ДНК, делеция целевого гена может быть подтверждена на уровне белка с использованием вестерн-блот и на функциональном уровне, если функциональный клеточный анализ доступен для этого гена. В случае RYR1-KO, поскольку RyR1 является кальциевым каналом, функциональная характеристика была выполнена с использованием кальциевой визуализации на культивируемых клетках.
- Экспрессия белка в отредактированных клонах
ПРИМЕЧАНИЕ: RyR1 экспрессируется только в дифференцированных миотубах10. Его экспрессия была оценена в миотрубах с использованием вестерн-блоттинга, чтобы подтвердить делецию на уровне белка RyR1, а также делецию белка Cas9.- Пластина 200 000 клеток в пролиферационной среде (описанная выше, стадия 5) на поверхности около 1,76см2 в пластине размером 35 мм, покрытой ламинином (поверхность, соответствующая капле ламинина 200 мкл при 10 мг/мл в PBS с кальцием). Как только клетки прилипнут к пластине после инкубации в течение 2-3 ч при 37 °C, 5% CO2, переложите культуральную среду на среду дифференцировки, состоящую из DMEM с низким содержанием глюкозы + 10% сыворотки лошади + 1% пенициллина / стрептомицина, и верните клетки в инкубатор в течение 6 дней.
- После 6 дней дифференцировки собирают и лизируют клетки 200 мкл RIPA, дополненного ингибиторами протеазы. Определяют концентрацию белка с помощью метода11 Фолина Лоури.
- Загрузить 15 мкг белка, после денатурации в течение 30 мин при РТ в денатурационном буфере Леммли, на 5%-15% градиентный акриламидный гель. После электрофоретического разделения переносят белки на Иммобилон Р при 0,8 В в течение 4 ч11.
- После насыщения мембраны в течение 30 мин при RT в PBS, содержащей 0,1% Tween 20 и 5% обезжиренного сухого молока, инкубируют мембрану с первичными антителами, разведенными в том же буфере в течение 2 ч при RT или на ночь при 4 °C, промыть мембрану 5x в течение 5 мин PBS-0,1% Tween 20 и инкубировать мембрану с вторичными антителами в течение 1 ч при RT. Основными используемыми антителами являются: антитела против V5-метки (разведение: 1/5000) для обнаружения Cas9, анти-GAPDH (разведение: 1/1000) в качестве контроля нагрузки, анти-RyR1 антитело 12,13 (разведение: 1/10.000), антитело против альфа-1 субъединицы DHPR (разведение: 1/1000) и антитело против тяжелой цепи миозина MF20 (разведение: 1/1000).
- Промыть мембрану 5х в течение 5 мин PBS-0,1% Tween 20, высушить избыток жидкости и добавить хемилюминесцентную субстрату. Действуйте в соответствии с рекомендациями поставщика субстрата для обнаружения хемилюминесцентного сигнала.
- Функциональная характеристика отредактированных клонов
ПРИМЕЧАНИЕ: Функцию RyR1 оценивали с помощью кальциевой визуализации в дифференцированных миотрубах, полученных из клонов CTRL или KO14.- Пластина 50 000 клеток на поверхности 0,2см2 в центре 35 мм посуды покрыта ламинином (поверхность, покрытая каплей ламинина 50 мкл, при 10 мг/мл в PBS кальцием) и индуцируют дифференцировку в течение 6 дней, как описано на этапе 8.1.1. Подготовьте три тарелки для каждой стимуляции, чтобы иметь биологический трипликат.
- Нагружают миотрубы 50 мкл флуо-4-прямого, разбавляют 1:1 в дифференцировочной среде и инкубируют в течение 30 мин при 37 °С. Промыть клетки дважды буфером KREBS, дополненным глюкозой в 1 мг/мл.
- Измерьте вариации флуоресценции с помощью перевернутого флуоресцентного микроскопа или конфокального микроскопа с помощью 10-кратного объектива. Установите пластину на ступень микроскопа и начните съемку со скоростью 1 кадр в секунду в течение 90 с.
- Удалить оставшиеся KREBS и стимулировать клетки в рамке 25 путем добавления 2 мл KCl для мембранной деполяризации (конечная концентрация 140 мМ) или 2 мл 4 CmC (конечная концентрация 500 мкМ) для прямой стимуляции RyR1. Убедитесь, что в зарегистрированном поле присутствует не менее 10 миотубов.
- Количественно оцените вариации флуоресценции в каждой миотубе, используя специальное программное обеспечение. Выберите для анализа не менее 10 миотуб на блюдо (в идеале 20-30 миотуб на блюдо), нарисуйте линию (или область интереса (ROI)) на длинной оси каждой миотрубы и соберите флуоресценцию F вдоль этой линии для всех кадров.
- Определите начальное значение флуоресцента F0, соответствующее кадрам от 1 до 24. График флуоресцентного изменения (F-F0)/F0 в зависимости от времени от 0 до 90 с. Повторите эксперимент три раза, чтобы получить вариацию флуоресценции по меньшей мере из 90 миотубок из трех разных культур. Объедините все результаты для 90 миотуб и рассчитайте средний ± SEM (F-F0)/F0 на каждом временном интервале. Количественно оцените пиковую амплитуду высвобождения кальция для каждой стимуляции и каждого клона.
Результаты
Этот протокол был применен к увековеченным миобластам от здорового субъекта15 (так называемые клетки HM, для миобластов человека), в которых RyR1 ранее был охарактеризован16, чтобы выбить ген RYR1 , кодирующий белок RyR1. Конструкция направляющей РНК была сделана д...
Обсуждение
Важным шагом на пути к характеристике генов неизвестной функции, участвующих в патологиях, является разработка соответствующих клеточных моделей для изучения функции этих генов. Использование редактирования генов с использованием CRISPR / Cas9 является экспоненциально растущей областью ...
Раскрытие информации
У авторов нет конфликта интересов для раскрытия.
Благодарности
Эта работа финансировалась за счет грантов Французской ассоциации по борьбе с миопатиями (AFM-Téléthon) и Овернь-Роны Альпы Режион (AURA).
Материалы
| Name | Company | Catalog Number | Comments |
| Anti-CACNA1S antibody | Sigma-Aldrich | HPA048892 | Primary antibody |
| Blp I | NE BioLabs | R0585S | Restriction enzyme |
| CalPhos Mammalian Transfection Kit | Takara | 631312 | Transfection kit |
| Easy blot anti Mouse IgG | GeneTex | GTX221667-01 | HRP secondary antibody |
| Easy blot anti Rabbit IgG | GeneTex | GTX221666 | HRP secondary antibody |
| Fluo-4 direct | Molecular Probes | F10472 | Calcium imaging |
| GAPDH(14C10) Rabbit mAb | Cell Signaling Technology | #2118 | Primary antibody |
| HindIII | Fermentas | ER0501 | Restriction enzyme |
| InFusion HD Precision Plus | Takara | 638920 | Ligation kit |
| MasterMix Phusion High Fidelity with GC | ThermoFisher Scientific | F532L | Mix for PCR reaction with High fidelity Taq polymerase and dNTPs |
| Myosin Heavy Chain antibody | DHSB | MF20 | Primary antibody |
| NucleoBond Xtra Maxi EF | Macherey-Nagel | REF 740424 | Maxipreparation kit for purification of plasmids |
| NucleoSpin Gel and PCR Clean-up | Macherey-Nagel | 740609 | DNA purification |
| NucleoSpin Tissue | Macherey-Nagel | 740952 | Kit for DNA extraction from cell |
| One Shot Stbl3 Chemically Competent E. coli | ThermoFisher Scientific | C737303 | Chemically competent cells |
| Plasmid #87904 | Addgene | 87904 | Lentiviral plasmid encoding the SpCas9 (for LV-Cas9) |
| Plasmid #87919 | Addgene | 87919 | Lentiviral backbone for insertion of cassette with guides (for LV-guide-target) |
| Plasmid #12260 | Addgene | 12260 | Lentiviral plasmid encoding lentiviral packaging GAG POL |
| Plasmid #8454 | Addgene | 8454 | Lentiviral plasmid encoding envelope protein for producing lentiviral and MuLV retroviral particles |
| V5 Tag Monoclonal Antibody | Invitrogene | R96025 | Primary antibody |
| XL10-Gold Ultracompetent Cells | Agilent | 200317 | Chemically competent cells |
| Xma I | NE BioLabs | R0180S | Restriction enzyme |
Ссылки
- Claussnitzer, M., Susztak, K. Gaining insight into metabolic diseases from human genetic discoveries. Trends in Genetics. 37 (12), 1081-1094 (2021).
- Fuster-García, C., García-Bohórquez, B., Rodríguez-Muñoz, A., Millán, J. M., García-García, G. Application of CRISPR tools for variant interpretation and disease modeling in inherited retinal dystrophies. Genes. 11 (5), 473 (2020).
- Modell, A. E., Lim, D., Nguyen, T. M., Sreekanth, V., Choudhary, A. CRISPR-based therapeutics: current challenges and future applications. Trends in Pharmacological Sciences. 43 (2), 151-161 (2022).
- Olson, E. N. Toward the correction of muscular dystrophy by gene editing. Proceedings of the National Academy of Sciences of the United States of America. 118 (22), (2021).
- Wu, X., Kriz, A. J., Sharp, P. A. Target specificity of the CRISPR-Cas9 system. Quantitative Biology. 2 (2), 59-70 (2014).
- Merienne, N., et al. The self-inactivating KamiCas9 system for the editing of CNS disease genes. Cell Reports. 20 (12), 2980-2991 (2017).
- Marty, I., Fauré, J. Excitation-contraction coupling alterations in myopathies. Journal of Neuromuscular Diseases. 3 (4), 443-453 (2016).
- Concordet, J. P., Haeussler, M. CRISPOR: intuitive guide selection for CRISPR/Cas9 genome editing experiments and screens. Nucleic Acids Research. 46, 242-245 (2018).
- Sun, Y., Sriramajayam, K., Luo, D., Liao, D. J. A quick, cost-free method of purification of DNA fragments from agarose gel. Journal of Cancer. 3, 93-95 (2012).
- Flucher, B. E., Conti, A., Takeshima, H., Sorrentino, V. Type 3 and type 1 ryanodine receptors are localized in triads of the same mammalian skeletal muscle fibers. The Journal of Cell Biology. 146 (3), 621-630 (1999).
- Hess, H. H., Lees, M. B., Derr, J. E. A linear Lowry--Folin assay for both water-soluble and sodium dodecyl sulfate-solubilized proteins. Analytical Biochemistry. 85 (1), 295-300 (1978).
- Garibaldi, M., et al. Dusty core disease' (DuCD): expanding morphological spectrum of RYR1 recessive myopathies. Acta Neuropathologica Communications. 7 (1), 3 (2019).
- Marty, I., et al. Biochemical evidence for a complex involving Dihydropyridine receptor and Ryanodine receptor in triad junctions of skeletal muscle. Proceedings of the National Academy of Sciences of the United States of America. 91 (6), 2270-2274 (1994).
- Oddoux, S., et al. Triadin deletion induces impaired skeletal muscle function. Journal of Biological Chemistry. 284 (50), 34918-34929 (2009).
- Mamchaoui, K., et al. Immortalized pathological human myoblasts: towards a universal tool for the study of neuromuscular disorders. Skeletal Muscle. 1, 34 (2011).
- Cacheux, M., et al. Functional characterization of a central core disease RyR1 mutation (p.Y4864H) associated with quantitative defect in RyR1 protein. Journal of Neuromuscular Diseases. 2 (4), 421-432 (2015).
- Luis, A. The old and the new: Prospects for non-integrating lentiviral vector technology. Viruses. 12 (10), 1103 (2020).
- Leenay, R. T., Beisel, C. L. Deciphering, communicating, and engineering the CRISPR PAM. Journal of Molecular Biology. 429 (2), 177-191 (2017).
- Salmon, P., Trono, D. Production and titration of lentiviral vectors. Current Protocols in Neurosciences. , (2006).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены