Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Entwicklung von Knock-out-Muskelzelllinien mittels Lentivirus-vermittelter CRISPR/Cas9-Gen-Editing
In diesem Artikel
Zusammenfassung
Das Protokoll beschreibt, wie Knock-out-Myoblasten mit CRISPR/Cas9 erzeugt werden können, beginnend mit dem Design von Guide-RNAs bis hin zur zellulären Klonierung und Charakterisierung der Knock-out-Klone.
Zusammenfassung
Eine wichtige Anwendung von Clustered Regulatory Interspaced Short Palindromic Repeats (CRISPR)/Cas 9 ist die Entwicklung von Knock-out-Zelllinien, insbesondere zur Untersuchung der Funktion neuer Gene / Proteine, die mit einer Krankheit assoziiert sind und während der genetischen Diagnose identifiziert wurden. Für die Entwicklung solcher Zelllinien müssen zwei Hauptprobleme entwirrt werden: das Einfügen der CRISPR-Werkzeuge (die Cas9- und die Guide-RNA) mit hoher Effizienz in die ausgewählten Zellen und die Beschränkung der Cas9-Aktivität auf die spezifische Deletion des ausgewählten Gens. Das hier beschriebene Protokoll widmet sich dem Einsetzen der CRISPR-Werkzeuge in schwer transfizierende Zellen, wie z.B. Muskelzellen. Dieses Protokoll basiert auf der Verwendung von Lentiviren, die mit öffentlich zugänglichen Plasmiden hergestellt werden und für die alle Klonierungsschritte beschrieben werden, um auf ein Gen von Interesse abzuzielen. Die Kontrolle der Cas9-Aktivität wurde unter Verwendung einer Adaption eines zuvor beschriebenen Systems namens KamiCas9 durchgeführt, bei dem die Transduktion der Zellen mit einem Lentivirus, das eine auf das Cas9 abzielende Leit-RNA kodiert, die schrittweise Abschaffung der Cas9-Expression ermöglicht. Dieses Protokoll wurde auf die Entwicklung einer RYR1-Knockout-Zelllinie für menschliche Muskeln angewendet, die auf Protein- und Funktionsebene weiter charakterisiert wurde, um den Knockout dieses wichtigen Kalziumkanals zu bestätigen, der an der intrazellulären Kalziumfreisetzung des Muskels und an der Erregungs-Kontraktionskopplung beteiligt ist. Das hier beschriebene Verfahren kann leicht auf andere Gene in Muskelzellen oder in anderen schwer zu transfektierenden Zellen angewendet werden und liefert wertvolle Werkzeuge, um diese Gene in menschlichen Zellen zu untersuchen.
Einleitung
Mit dem Fortschritt der Gensequenzierung und der Identifizierung von Mutationen in Genen unbekannter Funktionen in einem bestimmten Gewebe stellt die Entwicklung relevanter zellulärer Modelle zum Verständnis der Funktion eines neuen Zielgens und zur Bestätigung seiner Beteiligung an den damit verbundenen pathophysiologischen Mechanismen ein wesentliches Werkzeug dar. Darüber hinaus sind diese Modelle von großer Bedeutung für zukünftige therapeutische Entwicklungen 1,2 und stellen eine interessante Alternative zur Entwicklung von Knock-out-Tiermodellen in direkter Linie mit den internationalen Empfehlungen zur Reduzierung des Einsatzes von Tieren in Experimenten dar. Die Genbearbeitung mit CRISPR/Cas9 gehört zu den leistungsfähigsten derzeit verfügbaren Werkzeugen, was die Entwicklung vieler Knock-out/Knock-in-Modelle ermöglicht hat, und die gezielte Genvalidierung mit CRISPR/Cas9 gehört zu den am weitesten verbreiteten Anwendungen von CRISPR/Cas93. Der Erfolg der Genbearbeitung hängt von der Fähigkeit ab, die CRISPR-Werkzeuge (die Leit-RNAs und die Nuklease Cas9) in das Zielzellmodell einzuführen, was in vielen schwer zu transfizierenden Zellen wie Muskelzellen eine Herausforderung darstellen kann4. Diese Herausforderung kann mit der Verwendung von Viren, in der Regel Lentivirus, überwunden werden, die den großen Vorteil haben, viele Zelltypen effizient zu transduzieren und ihr Transgen zu liefern. Sein Hauptnachteil ist jedoch die Integration des Transgens in das Genom der Wirtszelle, was zu einer möglichen Veränderung der an der Integrationsstelle lokalisierten Gene und zur dauerhaften Expression des Transgens führt, was im Falle der Nuklease Cas9 zu schädlichen Folgen führenwürde 5. Merienne und Kollegen6 haben eine intelligente Lösung vorgeschlagen, die darin besteht, eine Guide-RNA in die Zellen einzuführen, die auf das Cas9-Gen selbst abzielt und zu einer Inaktivierung von Cas9 führt. Eine Adaption dieser Strategie wird hier als benutzerfreundliches und vielseitiges Protokoll vorgestellt, das es ermöglicht, praktisch jedes Gen in schwer zu transfizierenden Zellen auszuschalten.
Ziel des hier vorgestellten Protokolls ist es, die Inaktivierung eines interessierenden Gens in immortalisierten Muskelzellen zu induzieren. Es kann verwendet werden, um jedes Gen von Interesse in verschiedenen Arten von unsterblichen Zellen auszuschalten. Das hier beschriebene Protokoll enthält Schritte zum Design der Leit-RNAs und ihrer Klonierung in lentivirale Plasmide, zur Herstellung der CRISPR-Werkzeuge in lentiviralen Vektoren, zur Transduzierung der Zellen mit den verschiedenen Lentiviren und zum Klonen der Zellen, um eine homogene editierte Zelllinie zu erzeugen.
Unter Verwendung dieses Protokolls wurden verewigte menschliche Skelettmuskelzellen mit Deletion des Typ-1-Ryanodinrezeptors (RyR1) entwickelt, einem essentiellen Kalziumkanal, der an der intrazellulären Kalziumfreisetzung und Muskelkontraktion beteiligt ist7. Der Knock-out (KO) des Gens wurde auf Proteinebene mit Western Blot und auf funktioneller Ebene mit Kalziumbildgebung bestätigt.
Protokoll
Muskelbiopsien wurden von der Bank of Tissues for Research (Myobank, einem Partner im EU-Netzwerk EuroBioBank, Paris, Frankreich) in Übereinstimmung mit den europäischen Empfehlungen und der französischen Gesetzgebung bezogen. Von allen Personen wurde eine schriftliche Einwilligung nach Aufklärung eingeholt. Immortalisierte Myoblasten wurden freundlicherweise von Dr. V. Mouly (Myology Institute, Paris, Frankreich) hergestellt, und die Protokolle wurden vom Ethikkomitee des Myology Institute genehmigt (MESRI, n AC-2019-3502).
1. CRISPR-Leitfaden-Design
- Identifizieren Sie die Region des zu löschenden Gens. Suchen Sie nach seiner genomischen Sequenz mit Genom-Browser-Tools wie ensembl.org oder genome.ucsc.edu und bestimmen Sie die chromosomen Koordinaten der beiden Regionen, um nach der Leit-RNA (gRNA) auf beiden Seiten der zu löschenden Region zu suchen.
- Für das hier verwendete Gen erhalten Sie die FASTA-Sequenz des RYR1-Gens und die Sequenz des Exons 101 wie folgt. Suchen Sie in ensembl nach RYR1 in der neuesten Version des menschlichen Genoms, wählen Sie den ersten Eintrag aus und klicken Sie auf das Transkript der proteinkodierenden Sequenz. Klicken Sie dann auf Exons, um zur Liste der Exons des Gens umzuleiten.
- Klicken Sie auf Sequenz herunterladen und wählen Sie Nur genomische Sequenz, um die vollständige Konsensussequenz des gesamten Gens herunterzuladen. Scrollen Sie in der Liste der Exons und Introns des Gens nach unten und wählen Sie die Ziel-Exons aus.
- Finden Sie die entsprechende Nukleotidsequenz im Gen. Wählen Sie die Nukleotidsequenzen der Introns unmittelbar vor und nach dem zu löschenden Exon aus, die zur Suche nach den gRNAs verwendet werden.
- Entwerfen Sie die beiden gRNA, hier Guide 1 und Guide 2 genannt, in den in Schritt 1.1 identifizierten Regionen (Introns vor- und nachgelagert der zu löschenden Region) mit Online-Tools wie Crispor.tefor.net8. Wählen Sie die beiden gRNAs, die durch einige hundert Basenpaare (bp) getrennt sind, wobei die Sequenz jeder gRNA genau 20 Nukleotide lang ist, ohne das Protospacer Adjacent Motif (PAM). Wählen Sie die besten verfügbaren Leitfäden aus, um das Off-Target zu begrenzen. Ein Beispiel für den Entwurf von Hilfslinien finden Sie in Abbildung 1.
- Es wird erwartet, dass die Sequenz zwischen den beiden Führungen gelöscht wird und zum Knock-out des interessierenden Gens führt, also wählen Sie die Position der beiden Führungen so, dass sie eine essentielle Sequenz oder ein essentielles Exon im Gen von Interesse löscht. Stellen Sie sicher, dass die gelöschte Sequenz/das gelöschte Exon nicht ausschließlich in einem alternativen Transkript des Gens vorhanden ist und/oder dass sie einen wichtigen Teil des Proteins kodiert, so dass ihre Deletion zu einem funktionellen Knockout führt.
HINWEIS: Obwohl vorhergesagt wird, dass die Cas9-Spaltungsstelle 3 bp vor dem Protospacer Adjacent Motif (PAM) auftritt, kann auch eine Spaltung in größerer Entfernung auftreten, so dass eine gute Lösung nicht von der genauen Lokalisation der Spaltungsstelle abhängt, wie z.B. der Spaltung im Intron.
- Es wird erwartet, dass die Sequenz zwischen den beiden Führungen gelöscht wird und zum Knock-out des interessierenden Gens führt, also wählen Sie die Position der beiden Führungen so, dass sie eine essentielle Sequenz oder ein essentielles Exon im Gen von Interesse löscht. Stellen Sie sicher, dass die gelöschte Sequenz/das gelöschte Exon nicht ausschließlich in einem alternativen Transkript des Gens vorhanden ist und/oder dass sie einen wichtigen Teil des Proteins kodiert, so dass ihre Deletion zu einem funktionellen Knockout führt.
- Bestimmen Sie die umgekehrte Komplementsequenz (RC) für jede gRNA ohne PAM, um die folgenden Sequenzen zu erhalten: Guide 1 und Guide 1-RC, Guide 2 und Guide 2-RC.
- Bestellen Sie die in Tabelle 1 dargestellten Primer, um das Klonen der Plasmide durchzuführen. Verwenden Sie während des gesamten Protokolls Primer in einer Konzentration von 10 nM in sterilem H2O.
HINWEIS: Die den gRNAs in diesen Primern hinzugefügten Sequenzen (fett und unterstrichen) entsprechen der Sequenz des Plasmids vor und nach der Guide-, Promotor- bzw. transaktivierenden Crispr-RNA (tracrRNA) und sollten nicht modifiziert werden, um eine gute Überlappung zwischen den Primern und dem Plasmid zu gewährleisten.
2. Klonen von Plasmid
HINWEIS: In diesem Schritt werden die gRNAs in das Plasmid-Backbone für die Lentivirus-Produktion eingefügt. Eine Kassette, die für die beiden gRNAs kodiert, wird zunächst durch aufeinanderfolgende Polymerase-Kettenreaktionen (PCR) unter Verwendung der überlappenden Primer hergestellt. Die neue Kassette wird dann in das lentivirale Rückgratplasmid #87919 eingesetzt.
- Erhalten Sie die folgenden Plasmide: Plasmid #87919, kodiert für CRISPR-Leit-RNAs in einem lentiviralen Vektor und Plasmid #87904, das für die SpCas9-Sequenz in einem lentiviralen Vektor kodiert.
- Kassettenbau
HINWEIS: Das Klonprotokoll ist zusammengefasst in Abbildung 2.- Führen Sie eine PCR (A)-Reaktion mit 2 μL Plasmid #87919, 2 μL primer_XmaIF, 2 μL primer_Guide1R, 25 μL Polymerasemischung und 19 μLH2Odurch. Führen Sie das folgende PCR-Programm aus (Programm 1): anfängliche Denaturierung 5 min bei 98 °C, gefolgt von 30 Zyklen von: 30 s bei 98 °C, 30 s bei 60 °C, 1 min 45 s bei 72 °C und eine Enddehnung von 7 min bei 72 °C. Der Tm der in Schritt 1.4 beschriebenen Primer beträgt 60 °C.
HINWEIS: Obwohl die Dehnungszeit in Programm 1 ziemlich lang erscheint, wurde diese Dehnungszeit gewählt, um die Produktion von genügend Material in der richtigen Größe sicherzustellen. Tatsächlich ist aufgrund der wiederholten Sequenzen im Plasmid (die Sequenz von tracrRNA nach RNA-Leitern wird dreimal im Plasmid #87919 wiederholt) die PCR-Amplifikation der erwarteten DNA schwierig, und in den aufeinanderfolgenden PCRs werden zusätzliche kleinere Banden produziert. Aufgrund der Konkurrenz zwischen den verschiedenen PCR-Produkten wurde also entweder die Dehnungszeit erhöht (um das längste zu bevorzugen und am Ende genügend gereinigtes Material zu haben), oder es wurde eine Touchdown-PCR (Programm 2) für das lange Fragment verwendet (wie für die PCR (endgültig) in Schritt 2.2.6 beschrieben). - Trennen Sie die PCR-Produkte auf einem 1% igen Agarosegel in Tris-Borat-EDTA-Puffer (TBE), entfernen und reinigen Sie das 300 bp Fragment. Die Reinigung erfolgt mit einem speziellen Kit gemäß den Anweisungen des Herstellers mit einer Elution in einem Endvolumen von 20 μL. Verwenden Sie das gereinigte Fragment entweder direkt oder lagern Sie es bei -20 °C für Schritt 2.2.5.
- Führen Sie eine PCR (B)-Reaktion mit 2 μL Plasmid #87919, 2 μL primer_Guide1F, 2 μL primer_Guide2R, 25 μL Polymerasemischung und 19 μLH2Omit dem PCR-Programm 1 durch. Trennen Sie die PCR-Produkte auf einem 1% igen Agarosegel in FSME, entfernen und reinigen Sie das 400 bp Fragment in 20 μL Elutionspuffer. Verwenden Sie das gereinigte Fragment entweder direkt oder lagern Sie es bei -20 °C für Schritt 2.2.5.
- Führen Sie eine PCR (C)-Reaktion mit 2 μL Plasmid #87919, 2 μL primer_Guide2F, 2 μL primer_BlpIR, 25 μL Polymerasemischung und 19 μLH2Ounter Verwendung des PCR-Programms 1 durch. Trennen Sie die PCR-Produkte auf einem 1% igen Agarosegel im TBE-Puffer. Das 600 bp-Fragment wird in 20 μL Elutionspuffer entfernt und gereinigt. Verwenden Sie das gereinigte Fragment entweder direkt oder lagern Sie es bei -20 °C für Schritt 2.2.6.
HINWEIS: Ein weiteres Fragment bei 900 bp kann aufgrund der Hybridisierung des Primers auf den wiederholten Regionen im Plasmid sichtbar sein, wie in der ANMERKUNG von Schritt 2.2.1 beschrieben. Falls vorhanden, sollte dieses Band verworfen werden. - Führen Sie eine PCR (D)-Reaktion mit 2 μL Elution PCR A (aus Schritt 2.2.2), 2 μL Elution PCR B (aus Schritt 2.2.3), 2 μL primer_XmaIF, 2 μL primer_Guide2R, 25 μL Polymerasemischung und 19 μLH2Omit PCR-Programm 1 durch. Trennen Sie die PCR-Produkte auf einem 1% igen Agarosegel im TBE-Puffer; Entfernen und reinigen Sie das 700 bp Fragment in 20 μL Elutionspuffer. Verwenden Sie das gereinigte Fragment entweder direkt oder lagern Sie es bei -20 °C für Schritt 2.2.6.
HINWEIS: Andere Bänder können aufgrund der Hybridisierung der Primer in den wiederholten Regionen bei >1.000 bp, 400 bp und 300 bp sichtbar sein und sollten verworfen werden. - Führen Sie eine PCR-Reaktion (endgültig) mit 4 μL Elutions-PCR C (aus Schritt 2.2.4), 4 μL Elutions-PCR D (aus Schritt 2.2.5), 4 μL primer_XmaIF, 4 μL primer_BlpIR, 50 μL Polymerasemischung und 34 μLH2Odurch. Das verwendete Programm ist das folgende (PCR-Programm 2): anfängliche Denaturierung für 5 min bei 98 °C; sechs Zyklen von: 30 s bei 98 °C, 30 s bei 66 °C (Verringerung der Hybridisierungstemperatur um 1 °C pro Zyklus), 1 min 45 bei 72 °C; 35 Zyklen von: 30 s bei 98 °C, 30 s bei 60 °C, 1 min 45 bei 72 °C und eine Enddehnung von 5 min bei 72 °C.
HINWEIS: Das PCR-Programm für diese abschließende Amplifikation ist eine Touchdown-PCR, die sich von der vorherigen unterscheidet, da die endgültige Amplifikation groß ist und zwei wiederholte TracrRNA-Sequenzen direkt nach jedem Guide enthält. - Trennen Sie die PCR-Produkte auf einem 1% igen Agarosegel im TBE-Puffer; Entfernen und reinigen Sie die Endkassette, die mit etwa 1.300 bp in 20 μL Elutionspuffer migriert. Quantifizieren Sie das eluierte Produkt. Verwenden Sie das gereinigte Fragment entweder direkt oder lagern Sie es bei -20 °C für Schritt 2.3.2.1. Dies ist das Endprodukt, das in das lentivirale Plasmid eingefügt wird.
HINWEIS: Andere Fragmente können bei >1000 bp, 400 bp und 300 bp sichtbar sein, was unvollständigen PCR-Fragmenten entspricht, die verworfen werden sollten.
- Führen Sie eine PCR (A)-Reaktion mit 2 μL Plasmid #87919, 2 μL primer_XmaIF, 2 μL primer_Guide1R, 25 μL Polymerasemischung und 19 μLH2Odurch. Führen Sie das folgende PCR-Programm aus (Programm 1): anfängliche Denaturierung 5 min bei 98 °C, gefolgt von 30 Zyklen von: 30 s bei 98 °C, 30 s bei 60 °C, 1 min 45 s bei 72 °C und eine Enddehnung von 7 min bei 72 °C. Der Tm der in Schritt 1.4 beschriebenen Primer beträgt 60 °C.
- Einsetzen der gRNAs-Kassette in das lentivirale Plasmid-Rückgrat.
- Linearisieren Sie das Plasmid durch Doppelaufschluss des Plasmids #87919 mit den Restriktionsenzymen XmaI und BlpI.
- Bereiten Sie die Reaktion mit 15 μL empfohlenem Puffer, 15 μL Plasmid (1μg/μL), 7,5 μL BlpI-Enzym (bei 10 U/μL), 7,5 μL XmaI-Enzym (bei 10 U/μL) und 112,5 μLH2Ovor. Inkubieren Sie für 1 h bei 37 °C und dann für 20 min bei 65 °C. Laden Sie die Gesamtmenge auf ein 1% Agarosegel, schneiden Sie das ~10 kb Plasmid aus und reinigen Sie es mit einem geeigneten Kit. Die Elution wird in 20 μL Puffer durchgeführt, und das eluierte Produkt wird mittels optischer Dichtemessung quantifiziert.
HINWEIS: Verwenden Sie ein geeignetes Reinigungsprotokoll für große DNA-Fragmente, wie das von Sun und Coll9 beschriebene Protokoll.
- Bereiten Sie die Reaktion mit 15 μL empfohlenem Puffer, 15 μL Plasmid (1μg/μL), 7,5 μL BlpI-Enzym (bei 10 U/μL), 7,5 μL XmaI-Enzym (bei 10 U/μL) und 112,5 μLH2Ovor. Inkubieren Sie für 1 h bei 37 °C und dann für 20 min bei 65 °C. Laden Sie die Gesamtmenge auf ein 1% Agarosegel, schneiden Sie das ~10 kb Plasmid aus und reinigen Sie es mit einem geeigneten Kit. Die Elution wird in 20 μL Puffer durchgeführt, und das eluierte Produkt wird mittels optischer Dichtemessung quantifiziert.
- Ligate die gRNA-Kassette und das Plasmid. Bereiten Sie die Reaktionsmischung mit der gRNA-Kassette aus Schritt 2.2.7 und dem linearisierten Plasmid aus Schritt 2.3.1.1 vor, fügen Sie 2 μL Enzym und H2 O hinzu,um ein Endvolumen von 10 μL zu erhalten. 15 min bei 50 °C inkubieren, um das endgültige Plasmid namens p_guides herzustellen.
HINWEIS: Die DNA-Kassettenmenge sollte zwischen 50-100 ng und die Plasmidmenge zwischen 100-200 ng liegen, mit einem Molverhältnis von 2: 1.
- Linearisieren Sie das Plasmid durch Doppelaufschluss des Plasmids #87919 mit den Restriktionsenzymen XmaI und BlpI.
- Verwenden Sie 2 μL des frisch zubereiteten Plasmids, um chemisch kompetente E.Coli wie Stbl3 (50 μL) oder XL10-Gold umzuwandeln und nach 1 h Wachstum bei 37 °C ohne Antibiotikum mit 100 μg/ml Ampicillin auf LB-Agarplatte zu verteilen. Über Nacht bei 37 °C inkubieren. Wählen Sie ein paar Kolonien aus und führen Sie eine Mini-Vorbereitung mit einem handelsüblichen Kit gemäß den Anweisungen des Herstellers durch.
- Führen Sie eine PCR-Amplifikation der Miniprep-DNA mit Primer_XmaI und Primer_BlpR mit dem PCR-Programm 1 durch (siehe Abbildung 2). Trennen Sie die PCR-Produkte auf einem 1% igen Agarosegel im TBE-Puffer. Wählen Sie einige Kolonien (~5) mit einem Band bei der erwarteten Größe von etwa 1300 bp.
- Führen Sie eine DNA-Sequenzierung der ausgewählten Kolonien unter Verwendung der Primer_XmaI oder Primer_BlpR durch, um das korrekte Einsetzen der gRNA-Kassette zu bestätigen.
HINWEIS: Eine der sequenzverifizierten Kolonien wird in der Studie weiter verwendet, und das Plasmid wird als p_guides bezeichnet. - Wiederholen Sie Schritt 2.2 (Kassettenkonstruktion) mit dem Primers-Killer F und R und den Primers-mCherry F und R. Verwenden Sie eine sequenzverifizierte Kolonie für weitere Analysen. Das Plasmid wird p_Killer genannt.
3. Lentivirus-Produktion
- Produzieren und reinigen Sie eine große Menge aller erforderlichen Plasmide mit einem endotoxinfreien Maxi-Prep-Kit gemäß den Anweisungen des Herstellers. Aliquots bei 2 μg/μL vorbereiten. Bei -20 °C lagern
- Vorbereitung der Zellen (Tag 1)
- Bereiten Sie 18 Platten mit 145 cm Besaat mit 1 x 106 HEK293-Zellen pro Platte in 16 ml Medium vor, das aus Dulbeccos modifiziertem Adlermedium (DMEM) hohem Glucosepyruvat besteht, ergänzt mit 10% fetalem Rinderserum (FBS) und 1% Penicillin / Streptomycin. Amplifizieren Sie die Zellen bei 37 °C, in einem 5% CO2 Inkubator für 3 Tage.
HINWEIS: Die Herstellung von Lentiviren muss in einem Labor der Biosicherheitsstufe 2 mit Vorsicht durchgeführt werden, wobei angepasste Schutzausrüstungen, einschließlich Einwegschutzanzug, Schutzkappe und Handschuhe, verwendet werden. Alle Experimente müssen unter einer Laminar-Flow-Haube (BSLII-Sicherheitswerkbank) mit Filterspitzen durchgeführt werden. Die gesamte Lösung, die Lentiviren enthält, und alle verwendeten Kunststoff-/Glasabfälle müssen mit Ethanol 70% oder einem anderen Virusinaktivator inaktiviert werden.
- Bereiten Sie 18 Platten mit 145 cm Besaat mit 1 x 106 HEK293-Zellen pro Platte in 16 ml Medium vor, das aus Dulbeccos modifiziertem Adlermedium (DMEM) hohem Glucosepyruvat besteht, ergänzt mit 10% fetalem Rinderserum (FBS) und 1% Penicillin / Streptomycin. Amplifizieren Sie die Zellen bei 37 °C, in einem 5% CO2 Inkubator für 3 Tage.
- Transfektion von Zellen (Tag 4)
- Überprüfen Sie den Zusammenfluss der Zellen, um sicherzustellen, dass sie einen Zusammenfluss von 60% -65% erreicht haben.
- Es wird die Transfektionslösung hergestellt, die für jede Platte enthält: 20,8 μg des interessierenden Plasmids (p-Guides oder p-Killer oder pCas9 #87904), 4,8 μg des Plasmids, das die Hülle kodiert (VSV-G, #8454), 20,8 μg des Plasmids psPAX2 (#12260) für die lentivirale Verpackung, 136 μL Calciumphosphat und mitH2Oauf ein Endvolumen von 1.000 μL einstellen. Fügen Sie diese Lösung tropfenweise und unter Rühren zu 1 ml 2x HEPES-gepufferter Kochsalzlösung (HBS) hinzu.
HINWEIS: Bereiten Sie keine Mischung für alle Platten gleichzeitig zu, um eine optimale Zubereitung von Reagenzien zu gewährleisten. bereiten Sie eine Mischung für sechs Teller gleichzeitig zu, z. B. für 18 Teller, bereiten Sie dreimal eine Mischung für sechs Teller vor. - Inkubieren Sie bei Raumtemperatur (RT) für mindestens 10 Minuten und fügen Sie 2 ml Lösung tropfenweise zu den Zellen hinzu. Homogenisieren Sie das Transfektionsreagenz mit sanfter Bewegung der Platte rückwärts, vorwärts, oben und unten, inkubieren Sie bei 37 °C, 5% CO2 für mindestens 5 h.
HINWEIS: Tragen Sie ab diesem Zeitpunkt und bis zum Ende der Produktion von Lentivirus zusätzliche Schutzausrüstung, einschließlich eines zweiten Handschuhs, Schutzhüllen und eines Einwegplastrons. - Fünf Stunden nach der Transfektion das Medium von den Platten entfernen und mit PBS spülen, um Transfektionsreagenzien loszuwerden. 12 ml frisches Medium zugeben und 48 h bei 37 °C, 5% CO2 inkubieren.
- Sammlung der Viruspartikel (Tag 6)
- Sammeln und bündeln Sie das Medium von allen Platten. Zentrifüte bei 800 x g für 5 min bei 4 °C, um Zelltrümmer zu pelletieren. Filtern Sie den Überstand mit einem 0,45-μm-Filter (mehrere Filter erforderlich).
- Zentrifuge bei 68.300 x g für 2 h bei 4 °C in einem schwingenden Schaufelrotor. Entfernen Sie den Überstand und lassen Sie die Röhrchen für 5-10 min unter der Sicherheitswerkbank auf einem Papier auf den Kopf stellen, um so viel Flüssigkeit wie möglich zu entfernen, und fügen Sie dann 100 μL HEK-Proliferationsmedium pro Pellet hinzu. Nach mindestens 2 h bei 4 °C die Pellets durch Pipettieren auf und ab resuspendieren. Pool aller suspendierten Pellets.
HINWEIS: Pellets können vor der Aliquotierung über Nacht bei 4 °C im Medium belassen werden. - Aliquot-Lentivirus in 10 μL oder 25 μL Probengröße (je nach Verwendung) und Snap-Freeze mit flüssigem Stickstoff. Bei -80 °C lagern. Ein aufgetautes Aliquot nicht einfrieren.
- Wiederholen Sie die Schritte 3.2 bis 3.4 mit anderen interessanten Plasmiden, um LV-Guides, LV-Killer und LV-Cas9 herzustellen.
HINWEIS: Alternativ können die Lentiviren von einem Unternehmen oder einer Viruseinrichtung erworben werden.
4. Lentivirus-Titration
HINWEIS: Die Virustitration wird an HEK293-Zellen durchgeführt. Die Titration ist wichtig, um in den nachfolgenden Schritten eine genaue Anzahl von Lentiviren pro Zelle (unabhängig von der Charge des Lentivirus) für die interessierenden Zellen zu integrieren. Die Anzahl der Viruspartikel, die eine Zelle effizient transduzieren, wird als Multiplizität der Infektion (MOI) bezeichnet: MOI 10 entspricht somit der Einführung von 10 Viruspartikeln pro Zelle. Da der Gefrier-/Auftauzyklus die Lebensfähigkeit des Lentivirus beeinflusst, wird die Titration mit einem gefrorenen Lentivirus-Aliquot durchgeführt, und jedes nachfolgende Experiment wird mit einem neuen Aliquot desselben Pools durchgeführt. Eine Titrationsmethode wird hier beschrieben, aber es können auch andere Methoden verwendet werden.
- Am Tag 1 säen Sie 1 x 105 Zellen pro Platte in fünf 35 mm Platten mit Glasabdeckungen auf der Unterseite und zwei 35 mm Platten ohne Deckgläser.
- Sammeln und zählen Sie an Tag 2 von den beiden Platten ohne Deckgläser die Anzahl der Zellen nach der Trypsinisierung und bestimmen Sie die durchschnittliche Anzahl der Zellen pro Platte (N).
- Bereiten Sie 100 μL Lentivirus vor, das bei 1/10 in Proliferationsmedium verdünnt ist. Transduzieren Sie die fünf kultivierten Platten mit unterschiedlichen Volumina des verdünnten Virus, von 1 bis 50 μL verdünntem Virus. Verwenden Sie für dieses Protokoll die folgenden Volumina, um die fünf Platten zu transduzieren: 1 μL, 5 μL, 10 μL, 20 μL, 50 μL. Inkubation für 48 h bei 37 °C, in einem 5% CO2.
- An Tag 4 fixieren Sie die Zellen durch Inkubation der Deckgläser bei RT für 20 bis 30 min in 4% Paraformaldehyd. Für das LV-Cas9 permeabilisieren Sie die Zellen mit 0,1% Triton X100 in Phosphatpufferkochsalzlösung (PBS) für 10 min bei RT, gesättigt in PBS-0,1% Triton X100-2% Ziegenserum - 0,5% Rinderserumalbumin (BSA) für 20 min bei RT und markieren Sie für 45 min bei RT mit primärem Antikörper-Anti-V5 (Verdünnung 1/400), gefolgt von 30 min Inkubation bei RT mit fluoreszierendem sekundärem Antikörper. Beschriften Sie die Kerne mit Hoescht (10 μg/ml in PBS) für 10 min bei RT.
- Montieren Sie die Deckgläser auf einem Objektträger und beobachten Sie mit einem fluoreszierenden Mikroskop, das mit einem 20-fachen Objektiv ausgestattet ist. Für LV-Führungen und LV-Killer gehen Sie nach der Fixierung direkt zur Kernbeschriftung und montieren Sie die Deckgläser. Zählen Sie für jedes Deckglas die Gesamtzahl der Kerne (Gesamtzahl der Zellen) im Sichtfeld und die Anzahl der markierten Zellen (entweder mit V5 oder mCherry) und bestimmen Sie das Verhältnis der für jedes Deckglas gekennzeichneten Zellen.
- Wählen Sie ein Deckglas, in dem das Verhältnis der transduzierten Zellen mindestens 10% und nicht mehr als 50% beträgt. Bestimmen Sie die Transduktionseffizienz (X) für dieses Deckglas und notieren Sie das Volumen des verdünnten Virus (V, in μL), das verwendet wird, um diese Transduktionseffizienz zu erhalten.
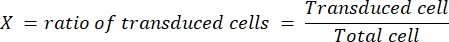
- Bestimmen Sie den Titer (in infektiösen Partikeln, dargestellt als ip/ml) des Virus nach folgender Formel:

Der Verdünnungsfaktor ist die Verdünnung des Lentivirus, die in Schritt 4.3 durchgeführt wird. Wir erhielten routinemäßig einen Endtiter von 1 x 109 ip/mL für LV-Cas9 und 1 x10 10 ip/mL für LV-Guide, bestimmt in HEK-Zellen.
5. Myoblast-Transduktion
HINWEIS: Die immortalisierten Myoblasten werden sukzessive mit den drei zuvor produzierten Lentiviren transduziert. Sie werden in einer Dichte unter 50% in einem Proliferationsmedium gehalten, das aus Ham's F10 besteht, ergänzt mit 20% FBS, 2% Penicillin/Streptomycin, 2% Ultroser G und kultiviert bei 37 °C, 5% CO2.
- Bestimmen Sie das Volumen des Lentivirus, das benötigt wird, um die gewählte Anzahl von Zellen mit MOI 10 für LV-Cas9 und LV-Guides und MOI 20 für LV-Killer gemäß der folgenden Formel zu behandeln:
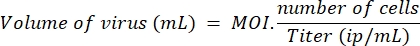
HINWEIS: In einem parallelen Experiment wurde die Transduktionseffizienz an Myoblasten und HEK unter Verwendung eines Kontroll-Lentivirus (Lenti-GFP) verglichen, und wir haben festgestellt, dass fünfmal mehr Lentiviren benötigt werden, um einen Myoblasten effizient zu transduzieren als eine HEK-Zelle. Somit entspricht MOI 10, gemessen an HEK, zwei viralen Partikeln pro Myoblast. Die hier verwendeten MOI werden auf HEK-Zellen berechnet. - An Tag 1 Samen Sie 96-Well-Platten mit 10.000 Zellen in 100 μL Proliferationsmedium pro Well. An Tag 2 werden die Zellen unter der Sicherheitswerkbank unter Zugabe des entsprechenden Volumens an LV-Führungen und LV-Cas9, berechnet in Schritt 5.1, transduziert. Bringen Sie die Zellen bis zum Tag 7 in den Inkubator zurück.
HINWEIS: Die Verwendung von MOI höher als 25, insbesondere für LV-Cas9, kann die Anzahl der editierten Zellen aufgrund des höheren Zelltods bei einer hohen Konzentration von Lentivirus nicht verbessern. - Führen Sie an Tag 7 eine Trypsinisierung durch und zählen Sie die Zellen. Samen Sie die Zellen mit einem Zusammenfluss von 40% bis 50% in einer neuen Platte und bringen Sie die Zellen in den Inkubator zurück. Fünf Stunden später transduzieren Sie die Zellen mit LV-Killer bei einem MOI von 20 (Volumen berechnet in Schritt 5.1). Amplifizieren Sie die Zellen für 5 bis 10 Tage nach der Transfektion, für mindestens zwei Passagen, und halten Sie sie immer bei einem niedrigen Zusammenfluss von <50%. Die Zellen sind bereit für den nächsten Schritt, das zelluläre Klonen, wenn ihr Wachstum wieder normal ist (geschätzt durch die Teilungszeit).
HINWEIS: Die Proliferation kann nach der Transduktion etwas langsamer sein. Das normale Zellwachstum kann vor diesem experimentellen Verfahren durch Abschätzung der Teilungszeit der Zellen bestimmt werden.
6. Klonen von Mobilfunk
HINWEIS: Da die Myoblastentransduktion schwierig ist und selbst bei Verwendung von Lentivirus nie eine 100% ige Effizienz erreicht, ist eine zelluläre Klonierung erforderlich, um eine vollständig korrigierte Zelllinie zu erhalten. Dies ist nur mit unsterblichen Zellen möglich oder Zellen, die während einiger Wochen / Monate kultiviert und verstärkt werden können.
- Trypsinisieren und zählen Sie die Zellen. Verdünnen Sie die Zellen in Proliferationsmedium bei 10 Zellen / ml und säen Sie die Zellen bei 1 Zelle / Well in 96-Well-Platten mit 100 μL / Well Medium.
HINWEIS: Die Anzahl der zu säenden Platten hängt von der Wahrscheinlichkeit der erwarteten Genbearbeitung ab, 2 bis 10 Platten werden routinemäßig verwendet. - Überwachen Sie die Zellen auf Wachstum und verstärken Sie nach und nach jede Vertiefung in einer größeren Platte, bis mindestens eine 35-mm-Platte erreicht ist, während die Konfluenz der Myoblasten unter 50% bleibt. Dieser Schritt kann 2-6 Wochen dauern, abhängig von den verwendeten Zellen und ihrer Fähigkeit, zu wachsen, sobald sie in einem Brunnen isoliert sind.
7. Auswahl klonen
HINWEIS: Dieser Schritt wird durchgeführt, um festzustellen, welche der wachsenden Klone entsprechend geändert wurden.
- Entwerfen Sie eine Reihe von Primern, die aus einem Primer vor der ersten Führung (Primer_BeforeGuide1F) und einem weiteren Primer nach der zweiten Führung (Primer_AfterGuide2R) bestehen, um den Bereich zu verstärken, der die vermeintlich modifizierte Sequenz umschließt. Die hier verwendeten Primer sind Tabelle 1 zu entnehmen.
- Sammeln Sie Zellen von jedem Klon, sparen Sie mindestens 300.000 Zellen für die zukünftige Amplifikation und extrahieren Sie genomische DNA mit einem Standardprotokoll für die verbleibenden Zellen.
- Wenn eine große Anzahl von Klonen wächst, um die nicht bearbeiteten zu verwerfen, führen Sie einen Schnelltest durch, indem Sie Zellen von fünf Klonen in derselben Röhre sammeln, um die DNA aus diesem Pool zu extrahieren und mit PCR zu testen. Wiederholen Sie den Vorgang mit so vielen Klonen wie nötig. Trennen Sie dann die Pools, die bearbeitete Zellen enthielten, weiter, um eine individuelle Analyse durchzuführen.
- Steuern Sie die Bearbeitung per PCR wie folgt. Bereiten Sie die PCR-Reaktion mit 1 μL Primer_BeforeGuide1F, 1 μL Primer_AfterGuide2R, 12,5 μL Polymerase-Mix, 3 μL genomischer DNA und 7,5 μLH2O vor. Amplifizieren Sie in einem Thermocycler gemäß den Anweisungen des Herstellers und den Primerparametern. Führen Sie ein 1% Agarosegel ein, um die bearbeiteten Klone zu identifizieren.
- Steuern Sie die Bearbeitung durch Sequenzierung wie folgt. Führen Sie eine Sanger-Sequenzierung der ausgewählten Klone durch, um den Löschvorgang zu bestätigen und festzustellen, wie die Bearbeitung in jedem Klon durchgeführt wurde. Bewahren Sie mehr als einen bearbeiteten Klon auf, um sicherzustellen, dass nur das Zielgen modifiziert wurde und für die beobachtete physiologische Wirkung verantwortlich ist, und behalten Sie einen nicht bearbeiteten Klon, der in den nachfolgenden Experimenten als Kontrollklon (CTRL) verwendet wird.
- Erweitern Sie die ausgewählten Klone. Sobald der Zusammenfluss jedes Klons etwa 50% erreicht hat, trypsinisieren Sie die Zellen und plattieren Sie die Zellen in einer größeren Schale, bis genügend Zellen produziert wurden, um die biochemischen und funktionellen Charakterisierungen durchzuführen (normalerweise mehr als 1 x 106 pro Klon), und speichern Sie gefrorene Aliquots jedes Klons für die zukünftige Verwendung.
8. Charakterisierung bearbeiteter Klone
HINWEIS: Sobald einige Klone entnommen und durch DNA-Sequenzierung bestätigt wurden, kann die Deletion des Zielgens auf Proteinebene mit Western Blot und auf funktioneller Ebene bestätigt werden, wenn ein funktioneller zellulärer Assay für dieses Gen verfügbar ist. Im Fall von RYR1-KO, da RyR1 ein Kalziumkanal ist, wurde die funktionelle Charakterisierung mittels Kalziumbildgebung an kultivierten Zellen durchgeführt.
- Proteinexpression in bearbeiteten Klonen
HINWEIS: RyR1 wird nur in differenzierten Myotubes10 exprimiert. Seine Expression wurde in Myotubes mit Western Blot untersucht, um die Deletion auf Proteinebene von RyR1 sowie die Deletion des Cas9-Proteins zu bestätigen.- Platte 200.000 Zellen in Proliferationsmedium (oben beschrieben, Schritt 5) auf einer Oberfläche von etwa 1,76 cm2 in einer mit Laminin beschichteten 35-mm-Platte (Oberfläche, die einem 200 μL-Tropfen Laminin bei 10 mg/ml in PBS mit Calcium entspricht). Sobald die Zellen nach der Inkubation für 2-3 h bei 37 ° C, 5% CO2 an der Platte haften, verschieben Sie das Kulturmedium auf ein Differenzierungsmedium, das aus DMEM-niedriger Glukose + 10% Pferdeserum + 1% Penicillin / Streptomycin besteht, und bringen Sie die Zellen für 6 Tage in den Inkubator zurück.
- Nach 6 Tagen Differenzierung sammeln und lysieren Sie die Zellen mit 200 μL RIPA, ergänzt mit Proteasehemmern. Bestimmen Sie die Proteinkonzentration mit der Folin Lowry-Methode11.
- Laden Sie 15 μg Protein, nach Denaturierung für 30 min bei RT in Laemmli Denaturierungspuffer, auf ein 5%-15% Gradient Acrylamid Gel. Nach der elektrophoretischen Trennung werden die Proteine auf Immobilon P bei 0,8 V für 4 h11 übertragen.
- Nach der Sättigung der Membran für 30 min bei RT in PBS mit 0,1% Tween 20 und 5% fettfreier Trockenmilch die Membran mit den im selben Puffer verdünnten primären Antikörpern für 2 h bei RT oder über Nacht bei 4 °C inkubieren, die Membran 5x für 5 min mit PBS-0,1% Tween 20 waschen und die Membran mit den sekundären Antikörpern für 1 h bei RT inkubieren. Die primär verwendeten Antikörper sind: Antikörper gegen V5-Tag (Verdünnung: 1/5000) zum Nachweis von Cas9, Anti-GAPDH (Verdünnung: 1/1000) als Belastungskontrolle, Anti-RyR1-Antikörper 12,13 (Verdünnung: 1/10.000), Antikörper gegen die alpha-1-Untereinheit von DHPR (Verdünnung: 1/1000) und Antikörper gegen die Myosin-Schwerkette MF20 (Verdünnung: 1/1000).
- Waschen Sie die Membran 5x für 5 min mit PBS-0,1% Tween 20, trocknen Sie den Flüssigkeitsüberschuss und fügen Sie das chemilumineszierende Substrat hinzu. Gehen Sie wie vom Substratanbieter empfohlen vor, um das Chemilumineszenzsignal zu erkennen.
- Funktionelle Charakterisierung von bearbeiteten Klonen
HINWEIS: Die Funktion von RyR1 wurde mittels Kalziumbildgebung in differenzierten Myotubes beurteilt, die aus CTRL- oder KO-Klonen14 hergestellt wurden.- Platte 50.000 Zellen auf einer Oberfläche von 0,2 cm2 in der Mitte von 35 mm Schalen, die mit Laminin beschichtet sind (Oberfläche bedeckt mit einem 50 μL Laminintropfen, bei 10 mg/ml in PBS mit Calcium) und induzieren eine Differenzierung für 6 Tage, wie in Schritt 8.1.1 beschrieben. Bereiten Sie drei Platten für jede Stimulation vor, um eine biologische Verdreifachung zu erhalten.
- Die Myotuben mit 50 μL Fluo 4-direkt, 1:1 im Differenzierungsmedium verdünnt und 30 min bei 37 °C inkubiert. Spülen Sie die Zellen zweimal mit KREBS-Puffer, ergänzt mit Glukose bei 1 mg / ml.
- Messen Sie die Fluoreszenzschwankungen mit einem inversen Fluoreszenzmikroskop oder einem konfokalen Mikroskop mit einem 10-fachen Objektiv. Installieren Sie die Platte auf dem Tisch des Mikroskops und starten Sie die Aufnahme mit 1 Bild pro Sekunde für 90 s.
- Entfernen Sie den verbleibenden KREBS und stimulieren Sie die Zellen im Rahmen 25 durch Zugabe von 2 ml KCl für die Membrandepolarisation (140 mM Endkonzentration) oder 2 ml 4 CmC (500 μM Endkonzentration) für die direkte RyR1-Stimulation. Stellen Sie sicher, dass mindestens 10 Myotubes im aufgezeichneten Feld vorhanden sind.
- Quantifizieren Sie die Fluoreszenzvariation in jeder Myotube mit einer speziellen Software. Wählen Sie für die Analyse mindestens 10 Myotubes pro Schale (idealerweise 20-30 Myotubes pro Dish), zeichnen Sie eine Linie (oder eine Region of Interest (ROI)) auf die Längsachse jeder Myotube und sammeln Sie die Fluoreszenz F entlang dieser Linie für alle Frames.
- Bestimmen Sie den anfänglichen Fluoreszenzwert F0, der den Frames 1 bis 24 entspricht. Zeichnen Sie die fluoreszierende Variation (F-F0)/F0 als Funktion der Zeit von 0 bis 90 s auf. Wiederholen Sie das Experiment dreimal, um die Fluoreszenzvariation von mindestens 90 Myotubes aus drei verschiedenen Kulturen zu erhalten. Fassen Sie alle Ergebnisse für die 90 Myotuben zusammen und berechnen Sie den Mittelwert ± SEM von (F-F0) / F0 in jedem Zeitrahmen. Quantifizieren Sie die Spitzenamplitude der Kalziumfreisetzung für jede Stimulation und jeden Klon.
Ergebnisse
Dieses Protokoll wurde auf immortalisierte Myoblasten eines gesunden Subjekts15 (sogenannte HM-Zellen, für menschliche Myoblasten) angewendet, in denen das RyR1 zuvor16 charakterisiert wurde, um das RYR1-Gen, das für das RyR1-Protein kodiert, auszuschalten. Das Design der Guides-RNA wurde vorgenommen, um die Sequenz zu löschen, die einen Teil des Exons 101 und das Intron 101 des Gens umfasst. Das Löschen eines Teils des Exon 101 wird voraussichtlich zu einer St...
Diskussion
Ein wichtiger Schritt auf dem Weg zur Charakterisierung von Genen unbekannter Funktion, die an Pathologien beteiligt sind, ist die Entwicklung relevanter zellulärer Modelle, um die Funktion dieser Gene zu untersuchen. Der Einsatz von Gen-Editing mit CRISPR/Cas9 ist ein exponentiell wachsendes Forschungsgebiet, und die Entwicklung von Knock-out-Modellen, wie sie hier vorgestellt werden, gehört zu den am weitesten verbreiteten Anwendungen. In diesem Zusammenhang schlagen wir hier ein vielseitiges Protokoll vor, um einen ...
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Diese Arbeit wurde durch Zuschüsse der Association Française contre les myopathies (AFM-Téléthon) und der Region Auvergne-Rhône Alpes (AURA) finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| Anti-CACNA1S antibody | Sigma-Aldrich | HPA048892 | Primary antibody |
| Blp I | NE BioLabs | R0585S | Restriction enzyme |
| CalPhos Mammalian Transfection Kit | Takara | 631312 | Transfection kit |
| Easy blot anti Mouse IgG | GeneTex | GTX221667-01 | HRP secondary antibody |
| Easy blot anti Rabbit IgG | GeneTex | GTX221666 | HRP secondary antibody |
| Fluo-4 direct | Molecular Probes | F10472 | Calcium imaging |
| GAPDH(14C10) Rabbit mAb | Cell Signaling Technology | #2118 | Primary antibody |
| HindIII | Fermentas | ER0501 | Restriction enzyme |
| InFusion HD Precision Plus | Takara | 638920 | Ligation kit |
| MasterMix Phusion High Fidelity with GC | ThermoFisher Scientific | F532L | Mix for PCR reaction with High fidelity Taq polymerase and dNTPs |
| Myosin Heavy Chain antibody | DHSB | MF20 | Primary antibody |
| NucleoBond Xtra Maxi EF | Macherey-Nagel | REF 740424 | Maxipreparation kit for purification of plasmids |
| NucleoSpin Gel and PCR Clean-up | Macherey-Nagel | 740609 | DNA purification |
| NucleoSpin Tissue | Macherey-Nagel | 740952 | Kit for DNA extraction from cell |
| One Shot Stbl3 Chemically Competent E. coli | ThermoFisher Scientific | C737303 | Chemically competent cells |
| Plasmid #87904 | Addgene | 87904 | Lentiviral plasmid encoding the SpCas9 (for LV-Cas9) |
| Plasmid #87919 | Addgene | 87919 | Lentiviral backbone for insertion of cassette with guides (for LV-guide-target) |
| Plasmid #12260 | Addgene | 12260 | Lentiviral plasmid encoding lentiviral packaging GAG POL |
| Plasmid #8454 | Addgene | 8454 | Lentiviral plasmid encoding envelope protein for producing lentiviral and MuLV retroviral particles |
| V5 Tag Monoclonal Antibody | Invitrogene | R96025 | Primary antibody |
| XL10-Gold Ultracompetent Cells | Agilent | 200317 | Chemically competent cells |
| Xma I | NE BioLabs | R0180S | Restriction enzyme |
Referenzen
- Claussnitzer, M., Susztak, K. Gaining insight into metabolic diseases from human genetic discoveries. Trends in Genetics. 37 (12), 1081-1094 (2021).
- Fuster-García, C., García-Bohórquez, B., Rodríguez-Muñoz, A., Millán, J. M., García-García, G. Application of CRISPR tools for variant interpretation and disease modeling in inherited retinal dystrophies. Genes. 11 (5), 473 (2020).
- Modell, A. E., Lim, D., Nguyen, T. M., Sreekanth, V., Choudhary, A. CRISPR-based therapeutics: current challenges and future applications. Trends in Pharmacological Sciences. 43 (2), 151-161 (2022).
- Olson, E. N. Toward the correction of muscular dystrophy by gene editing. Proceedings of the National Academy of Sciences of the United States of America. 118 (22), (2021).
- Wu, X., Kriz, A. J., Sharp, P. A. Target specificity of the CRISPR-Cas9 system. Quantitative Biology. 2 (2), 59-70 (2014).
- Merienne, N., et al. The self-inactivating KamiCas9 system for the editing of CNS disease genes. Cell Reports. 20 (12), 2980-2991 (2017).
- Marty, I., Fauré, J. Excitation-contraction coupling alterations in myopathies. Journal of Neuromuscular Diseases. 3 (4), 443-453 (2016).
- Concordet, J. P., Haeussler, M. CRISPOR: intuitive guide selection for CRISPR/Cas9 genome editing experiments and screens. Nucleic Acids Research. 46, 242-245 (2018).
- Sun, Y., Sriramajayam, K., Luo, D., Liao, D. J. A quick, cost-free method of purification of DNA fragments from agarose gel. Journal of Cancer. 3, 93-95 (2012).
- Flucher, B. E., Conti, A., Takeshima, H., Sorrentino, V. Type 3 and type 1 ryanodine receptors are localized in triads of the same mammalian skeletal muscle fibers. The Journal of Cell Biology. 146 (3), 621-630 (1999).
- Hess, H. H., Lees, M. B., Derr, J. E. A linear Lowry--Folin assay for both water-soluble and sodium dodecyl sulfate-solubilized proteins. Analytical Biochemistry. 85 (1), 295-300 (1978).
- Garibaldi, M., et al. Dusty core disease' (DuCD): expanding morphological spectrum of RYR1 recessive myopathies. Acta Neuropathologica Communications. 7 (1), 3 (2019).
- Marty, I., et al. Biochemical evidence for a complex involving Dihydropyridine receptor and Ryanodine receptor in triad junctions of skeletal muscle. Proceedings of the National Academy of Sciences of the United States of America. 91 (6), 2270-2274 (1994).
- Oddoux, S., et al. Triadin deletion induces impaired skeletal muscle function. Journal of Biological Chemistry. 284 (50), 34918-34929 (2009).
- Mamchaoui, K., et al. Immortalized pathological human myoblasts: towards a universal tool for the study of neuromuscular disorders. Skeletal Muscle. 1, 34 (2011).
- Cacheux, M., et al. Functional characterization of a central core disease RyR1 mutation (p.Y4864H) associated with quantitative defect in RyR1 protein. Journal of Neuromuscular Diseases. 2 (4), 421-432 (2015).
- Luis, A. The old and the new: Prospects for non-integrating lentiviral vector technology. Viruses. 12 (10), 1103 (2020).
- Leenay, R. T., Beisel, C. L. Deciphering, communicating, and engineering the CRISPR PAM. Journal of Molecular Biology. 429 (2), 177-191 (2017).
- Salmon, P., Trono, D. Production and titration of lentiviral vectors. Current Protocols in Neurosciences. , (2006).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten