A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
פיתוח קווי תאי שריר נוק-אאוט באמצעות עריכת גנים של קריספר/Cas9 בתיווך נגיף לנטי
In This Article
Summary
הפרוטוקול מתאר כיצד ליצור מיובלסטים נוק-אאוט באמצעות CRISPR/Cas9, החל מתכנון של רנ"א מנחים ועד לשיבוט תאי ואפיון של שיבוטים נוק-אאוט.
Abstract
יישום חשוב אחד של חזרות פלינדרומיות קצרות (CRISPR)/Cas 9 הוא פיתוח של קווי תאים נוק-אאוט, במיוחד כדי לחקור את תפקודם של גנים/חלבונים חדשים הקשורים למחלה, שזוהו במהלך האבחון הגנטי. לצורך פיתוח קווי תאים כאלה, יש להתיר שתי סוגיות עיקריות: החדרת כלי הקריספר (ה-Cas9 וה-RNA המנחה) ביעילות גבוהה לתאים שנבחרו, והגבלת פעילות ה-Cas9 למחיקה הספציפית של הגן הנבחר. הפרוטוקול המתואר כאן מוקדש להחדרת כלי הקריספר בתאים קשים להעברה, כגון תאי שריר. פרוטוקול זה מבוסס על שימוש ב- lentiviruses, המיוצרים עם פלסמידים הזמינים לציבור, שעבורם מתוארים כל שלבי השיבוט כדי להתמקד בגן בעל עניין. הבקרה על פעילות Cas9 בוצעה באמצעות התאמה של מערכת שתוארה בעבר בשם KamiCas9, שבה התמרת התאים עם נגיף לנטי המקודד RNA מנחה המכוון ל- Cas9 מאפשרת ביטול הדרגתי של ביטוי Cas9. פרוטוקול זה יושם על פיתוח קו תאי שריר אנושיים RYR1-knock out, אשר אופיין עוד יותר ברמת החלבון והתפקוד, כדי לאשר את הנוקאאוט של תעלת סידן חשובה זו המעורבת בשחרור סידן תוך תאי בשרירים ובצימוד עירור-התכווצות. ההליך המתואר כאן יכול בקלות להיות מיושם על גנים אחרים בתאי שריר או בתאים אחרים שקשה להעבירם ולייצר כלים חשובים לחקר גנים אלה בתאים אנושיים.
Introduction
עם התקדמות ריצוף הגנים וזיהוי מוטציות בגנים בעלי תפקודים לא ידועים ברקמה מסוימת, פיתוח מודלים תאיים רלוונטיים להבנת תפקודו של גן מטרה חדש ולאישור מעורבותו במנגנונים הפתופיזיולוגיים הקשורים מהווה כלי חיוני. בנוסף, מודלים אלה הם בעלי חשיבות עליונה לפיתוחים טיפוליים עתידיים 1,2, ומהווים חלופה מעניינת לפיתוח מודלים של בעלי חיים נוק-אאוט בקו ישר עם ההמלצות הבינלאומיות לצמצום השימוש בבעלי חיים בניסויים. עריכת גנים באמצעות CRISPR/Cas9 היא בין הכלים החזקים ביותר הקיימים כיום, מה שאיפשר פיתוח של מודלים רבים של נוק-אאוט/נוק-אין, ואימות גנים ממוקד באמצעות CRISPR/Cas9 הוא בין היישומים הנפוצים ביותר של CRISPR/Cas93. ההצלחה של עריכת גנים נשענת על היכולת להכניס את כלי הקריספר (הרנ"א המנחים והנוקלאז Cas9) במודל תאי המטרה, מה שיכול להוות אתגר בתאים רבים שקשה לבצע טרנספקט, כגון תאי שריר4. ניתן להתגבר על אתגר זה באמצעות שימוש בנגיף, בדרך כלל lentivirus, שיש לו את היתרון הגדול להתמר ביעילות סוגי תאים רבים ולספק את הטרנסג'ן שלו. אבל החיסרון העיקרי שלה הוא השילוב של הטרנסגן בגנום של התא המארח, מה שמוביל לשינוי פוטנציאלי של גנים הממוקמים באתר האינטגרציה ולביטוי הקבוע של הטרנסגן, שבמקרה של הנוקלאז Cas9 יגרום לתוצאות מזיקות5. פתרון חכם הוצע על ידי מריאן ועמיתיו6, המורכב מהכנסה לתאים של רנ"א מנחה המכוון לגן Cas9 עצמו, מה שמוביל להשבתת Cas9. התאמה של אסטרטגיה זו מוצגת כאן כפרוטוקול ידידותי למשתמש ורב-תכליתי המאפשר לדפוק כמעט כל גן בתאים קשים להעברה.
מטרת הפרוטוקול המוצג כאן היא לגרום להשבתה של גן בעל עניין בתאי שריר מונצחים. ניתן להשתמש בו כדי להפיל כל גן בעל עניין, בסוגים שונים של תאים מונצחים. הפרוטוקול המתואר כאן מכיל שלבים לתכנון הרנ"א המנחים ושיבוטם לפלסמידים לנטי-ויראליים, לייצור כלי הקריספר בווקטורים לנטי-ויראליים, להתמרת התאים עם נגיפי הלנטי-וירוסים השונים, ולשיבוט התאים כדי לייצר קו תאים ערוך הומוגני.
באמצעות פרוטוקול זה, פותחו תאי שרירי שלד אנושיים מונצחים עם מחיקה של קולטן ריאנודין מסוג 1 (RyR1), תעלת סידן חיונית המעורבת בשחרור סידן תוך תאי ובהתכווצות שרירים7. הנוק-אאוט (KO) של הגן אושר ברמת החלבון באמצעות כתם מערבי, וברמה התפקודית באמצעות הדמיית סידן.
Protocol
ביופסיות שרירים התקבלו מבנק הרקמות למחקר (Myobank, שותף ברשת EuroBioBank של האיחוד האירופי, פריז, צרפת) בהתאם להמלצות האירופיות ולחקיקה הצרפתית. הסכמה מדעת בכתב התקבלה מכל האנשים. המיובלסטים המונצחים הופקו בחביבות על ידי ד"ר ו' מולי (המכון למיולוגיה, פריז, צרפת), והפרוטוקולים אושרו על ידי ועדת האתיקה של המכון למיולוגיה (MESRI, n AC-2019-3502).
1. עיצוב מדריך קריספר
- זהה את האזור של הגן שיש למחוק. חפש את הרצף הגנומי שלו באמצעות כלי דפדפן גנומיים כגון ensembl.org או genome.ucsc.edu וקבע את הקואורדינטות הכרומוזומליות של שני האזורים כדי לחפש את הרנ"א המנחה (gRNA), משני צידי האזור שיש למחוק.
- עבור הגן המשמש כאן, קבל את רצף FASTA של הגן RYR1 ואת הרצף של exon 101 כדלקמן. ב- ensembl, חפש את RYR1 בגרסה העדכנית ביותר של הגנום האנושי, בחר את הערך הראשון ולחץ על התעתיק של רצף קידוד החלבונים. לאחר מכן, לחץ על Exons כדי להפנות לרשימת האקסונים של הגן.
- לחץ על הורד את רצף ובחר רק רצף גנומי כדי להוריד את רצף הקונצנזוס המלא של הגן כולו. גלול מטה את רשימת האקסונים והאינטונים של הגן ובחר את הממוקד(ים).
- מצא את רצף הנוקלאוטידים המתאים בגן. בחר את רצפי הנוקלאוטידים של האינטרונים מיד במעלה הזרם ובמורד הזרם של האקסון שיימחקו, אשר ישמשו לחיפוש ה- gRNA.
- תכנן את שני ה-gRNA, הנקראים מדריך 1 ומדריך 2 כאן, באזורים שזוהו בשלב 1.1 (אינטרונים במעלה הזרם ובמורד הזרם של האזור למחיקה) באמצעות כלים מקוונים כגון Crispor.tefor.net8. בחר את שני ה-gRNA המופרדים על ידי כמה מאות זוגות בסיסים (bp), כאשר הרצף של כל gRNA הוא בדיוק 20 נוקלאוטידים באורך ללא המוטיב הסמוך לפרוטוספייסר (PAM). בחר את המדריכים הטובים ביותר הזמינים כדי להגביל את היציאה מהיעד. ראו איור 1 דוגמה לעיצוב מדריך.
- הרצף בין שני המדריכים צפוי להימחק, ולגרום לנוק-אאוט של הגן המעניין, ובכך לבחור את המיקום של שני המדריכים כך שהוא מוחק רצף חיוני או אקסון חיוני בגן המעניין. יש לוודא שהרצף/אקסון שנמחק אינו קיים אך ורק בתעתיק חלופי של הגן ו/או שהוא מקודד חלק חשוב מהחלבון, ולכן מחיקתו תגרום לנוק-אאוט פונקציונלי.
הערה: למרות שאתר הבקיעה של Cas9 צפוי להתרחש 3 bp במעלה הזרם של המוטיב הסמוך לפרוטוספייסר (PAM), מחשוף במרחק גדול יותר יכול להתרחש גם הוא, ולכן פתרון טוב אינו תלוי בלוקליזציה המדויקת של אתר המחשוף, כגון מחשוף באינטרון.
- הרצף בין שני המדריכים צפוי להימחק, ולגרום לנוק-אאוט של הגן המעניין, ובכך לבחור את המיקום של שני המדריכים כך שהוא מוחק רצף חיוני או אקסון חיוני בגן המעניין. יש לוודא שהרצף/אקסון שנמחק אינו קיים אך ורק בתעתיק חלופי של הגן ו/או שהוא מקודד חלק חשוב מהחלבון, ולכן מחיקתו תגרום לנוק-אאוט פונקציונלי.
- קבע את רצף המשלים ההפוך (RC) עבור כל gRNA, ללא PAM, כדי שיהיו לו את הרצפים הבאים: מדריך 1 ומדריך 1-RC, מדריך 2 ומדריך 2-RC.
- הזמינו את הפריימרים המוצגים בטבלה 1 לבצע שיבוט של הפלסמידים. לאורך כל הפרוטוקול, יש להשתמש בפריימרים בריכוז של 10 ננומטר ב-H2O סטרילי.
הערה: הרצפים שנוספו ל-gRNA בפריימרים אלה (מודגשים ומסומנים בקו תחתון) תואמים את רצף הפלסמיד לפני ואחרי המדריך, המקדם וה-RNA של טרנס-מפעיל-קריספר (tracrRNA), בהתאמה ואין לשנותם על מנת להבטיח חפיפה טובה בין הפריימרים לפלסמיד.
2. שיבוט פלסמיד
הערה: בשלב זה, ה-gRNA יוכנסו לעמוד השדרה של הפלסמיד לייצור נגיף הלנטי. קלטת המקודדת את שני ה-gRNA מיוצרת לראשונה על ידי תגובות שרשרת פולימראז עוקבות (PCR), תוך שימוש בפריימרים החופפים. לאחר מכן מוכנסת הקלטת החדשה לתוך עמוד השדרה הלנטי-ויראלי פלסמיד #87919.
- השג את הפלסמידים הבאים: פלסמיד #87919, קידוד עבור רנ"א מדריך קריספר בווקטור לנטי-ויראלי ופלסמיד #87904 המקודד עבור רצף SpCas9 בווקטור לנטי-ויראלי.
- בניית קלטות
הערה: פרוטוקול השיבוט מסוכם ב תרשים 2.- הפעל תגובת PCR (A), עם 2 μL של פלסמיד #87919, 2 μL של primer_XmaIF, 2 μL של primer_Guide1R, 25 μL של תערובת פולימראז, ו- 19 μL של H2O. הפעל את תוכנית ה- PCR הבאה (תוכנית 1): דנטורציה ראשונית 5 דקות ב- 98 °C, ואחריה 30 מחזורים של: 30 שניות ב- 98 °C, 30 שניות ב-60 מעלות צלזיוס, דקה אחת 45 שניות ב-72 מעלות צלזיוס, והתארכות סופית של 7 דקות ב-72 מעלות צלזיוס. ה-Tm של הפריימרים המתוארים בשלב 1.4 הוא 60 מעלות צלזיוס.
הערה: למרות שזמן ההתארכות בתוכנית 1 נראה ארוך למדי, זמן התארכות זה נבחר כדי להבטיח ייצור של מספיק חומר בגודל הנכון. ואכן, בגלל הרצפים החוזרים על עצמם בפלסמיד (הרצף של tracrRNA לאחר מדריכי RNA חוזר על עצמו שלוש פעמים בפלסמיד #87919) הגברת ה-PCR של הדנ"א הצפוי היא קשה, ופסים קטנים נוספים מיוצרים ב-PCRs העוקבים. לכן, בגלל התחרות בין מוצרי ה-PCR השונים, או שזמן ההתארכות הוגדל (כדי להעדיף את הארוך ביותר וכדי שיהיה מספיק חומר מטוהר בסוף), או שנעשה שימוש ב-PCR (תוכנית 2) עבור השבר הארוך (כמו שתואר עבור ה-PCR (הסופי) בשלב 2.2.6). - הפרד את מוצרי ה-PCR על ג'ל אגרוז של 1% במאגר Tris-Borate-EDTA (TBE), הבלו וטהרו את השבר של 300 bp. בצע טיהור באמצעות ערכה ייעודית לאחר הוראת היצרן, עם elution בנפח סופי של 20 μL. או להשתמש בשבר המטוהר ישירות או לאחסן ב -20 °C עבור שלב 2.2.5.
- הפעל תגובת PCR (B), עם 2 μL של פלסמיד #87919, 2 μL של primer_Guide1F, 2 μL של primer_Guide2R, 25 μL של תערובת פולימראז, ו 19 μL של H2O באמצעות תוכנית PCR 1. הפרד את מוצרי ה- PCR על ג'ל אגרוז 1% ב- TBE, בלו וטהר את מקטע 400 bp ב- 20 μL של מאגר elution. או להשתמש בשבר המטוהר ישירות או לאחסן בטמפרטורה של -20 מעלות צלזיוס עבור שלב 2.2.5.
- הפעל תגובת PCR (C), עם 2 μL של פלסמיד #87919, 2 μL של primer_Guide2F, 2 μL של primer_BlpIR, 25 μL של תערובת פולימראז, ו-19 μL של H2O באמצעות תוכנית PCR 1. הפרד את מוצרי ה-PCR על ג'ל אגרוז 1% במאגר TBE. לבלות ולטהר את שבר 600 bp ב 20 μL של חיץ elution. או להשתמש בשבר המטוהר ישירות או לאחסן ב -20 °C עבור שלב 2.2.6.
הערה: קטע נוסף ב- 900 bp עשוי להיות גלוי, עקב הכלאה של הפריימר על האזורים החוזרים בפלסמיד, כמתואר בהערה של שלב 2.2.1. אם קיימת, יש להשליך את הלהקה הזו. - הפעל תגובת PCR (D), עם 2 μL של אלוטיון PCR A (משלב 2.2.2), 2 μL של elution PCR B (משלב 2.2.3), 2 μL של primer_XmaIF, 2 μL של primer_Guide2R, 25 μL של תערובת פולימראז, ו-19 μL של H2O, עם תוכנית PCR 1. להפריד את מוצרי ה-PCR על ג'ל אגרוז 1% ב-TBE buffer; לבלות ולטהר את שבר 700 bp ב 20 μL של חיץ elution. או להשתמש בשבר המטוהר ישירות או לאחסן ב -20 °C עבור שלב 2.2.6.
הערה: רצועות אחרות עשויות להיות גלויות ב->1,000 bp, 400 bp ו- 300 bp, עקב הכלאה של הפריימרים באזורים החוזרים ויש להשליך אותם. - הפעל תגובת PCR (סופית), עם 4 μL של אלוטיון PCR C (משלב 2.2.4), 4 μL של אלוטיון PCR D (משלב 2.2.5), 4 μL של primer_XmaIF, 4 μL של primer_BlpIR, 50 μL של תערובת פולימראז, ו 34 μL של H2O. התוכנית המשמשת היא כדלקמן (תוכנית PCR 2): denaturation ראשוני במשך 5 דקות ב 98 °C (84 °F); שישה מחזורים של: 30 שניות ב-98 מעלות צלזיוס, 30 שניות ב-66 מעלות צלזיוס (הפחתה בטמפרטורת ההכלאה של 1 מעלות צלזיוס לכל מחזור), דקה 45 ב-72 מעלות צלזיוס; 35 מחזורים של: 30 שניות ב-98 מעלות צלזיוס, 30 שניות ב-60 מעלות צלזיוס, דקה אחת 45 ב-72 מעלות צלזיוס, והתארכות סופית של 5 דקות ב-72 מעלות צלזיוס.
הערה: תוכנית ה-PCR להגברה סופית זו היא PCR מגע למטה, שונה מקודמתה, בגלל הגודל הגדול של ההגברה הסופית המכילה שני רצפי tracrRNA חוזרים רק אחרי כל מדריך. - להפריד את מוצרי ה-PCR על ג'ל אגרוז 1% ב-TBE buffer; לבלות ולטהר את הקלטת הסופית, אשר נודדת בערך 1,300 bp ב 20 μL של מאגר elution. לכמת את המוצר החמקמק. או להשתמש בשבר המטוהר ישירות או לאחסן ב -20 °C (74 °F) עבור שלב 2.3.2.1. זהו המוצר הסופי שיוכנס לפלסמיד הלנטיוויראלי.
הערה: מקטעים אחרים עשויים להיות גלויים ב- >1000 bp, 400 bp ו- 300 bp, המתאימים למקטעי PCR לא שלמים שיש להשליך.
- הפעל תגובת PCR (A), עם 2 μL של פלסמיד #87919, 2 μL של primer_XmaIF, 2 μL של primer_Guide1R, 25 μL של תערובת פולימראז, ו- 19 μL של H2O. הפעל את תוכנית ה- PCR הבאה (תוכנית 1): דנטורציה ראשונית 5 דקות ב- 98 °C, ואחריה 30 מחזורים של: 30 שניות ב- 98 °C, 30 שניות ב-60 מעלות צלזיוס, דקה אחת 45 שניות ב-72 מעלות צלזיוס, והתארכות סופית של 7 דקות ב-72 מעלות צלזיוס. ה-Tm של הפריימרים המתוארים בשלב 1.4 הוא 60 מעלות צלזיוס.
- החדרת קלטת ה-gRNA בעמוד השדרה של הפלסמיד הלנטי-ויראלי.
- ליניאריזציה של הפלסמיד על ידי עיכול כפול של הפלסמיד #87919 עם אנזימי ההגבלה XmaI ו- BlpI.
- הכן את התגובה עם 15 μL של מאגר מומלץ, 15 μL של פלסמיד (1μg / μL), 7.5 μL של אנזים BlpI (ב 10 U / μL), 7.5 μL של אנזים XmaI (ב 10 U / μL), ו 112.5 μL של H2O. אינקובאט במשך שעה אחת ב 37 ° C, ולאחר מכן במשך 20 דקות ב 65 ° C. טענו את הכמות הכוללת על ג'ל אגרוז 1%, גזרו וטהרו את הפלסמיד בנפח של כ-10 קילובייט עם ערכה מתאימה. Elution מבוצעת ב-20 μL של מאגר, והמכפלה הבוקעת מכמתת באמצעות מדידת צפיפות אופטית.
הערה: השתמש בפרוטוקול טיהור מתאים עבור מקטע דנ"א גדול, כגון הפרוטוקול שתואר על ידי Sun ו- coll9.
- הכן את התגובה עם 15 μL של מאגר מומלץ, 15 μL של פלסמיד (1μg / μL), 7.5 μL של אנזים BlpI (ב 10 U / μL), 7.5 μL של אנזים XmaI (ב 10 U / μL), ו 112.5 μL של H2O. אינקובאט במשך שעה אחת ב 37 ° C, ולאחר מכן במשך 20 דקות ב 65 ° C. טענו את הכמות הכוללת על ג'ל אגרוז 1%, גזרו וטהרו את הפלסמיד בנפח של כ-10 קילובייט עם ערכה מתאימה. Elution מבוצעת ב-20 μL של מאגר, והמכפלה הבוקעת מכמתת באמצעות מדידת צפיפות אופטית.
- ליגט את קלטת ה-gRNA ואת הפלסמיד. הכן את תערובת התגובה עם קלטת gRNA משלב 2.2.7 ואת הפלסמיד הליניארי משלב 2.3.1.1, הוסף 2 μL של אנזים ו- H2O כדי לקבל נפח סופי של 10 μL. אינקובציה למשך 15 דקות ב 50 °C כדי לייצר את הפלסמיד הסופי הנקרא p_guides.
הערה: כמות קלטת הדנ"א צריכה להיות בין 50-100 ננוגרם לכמות הפלסמיד בין 100-200 ננוגרם, עם יחס טוחנת של 2:1.
- ליניאריזציה של הפלסמיד על ידי עיכול כפול של הפלסמיד #87919 עם אנזימי ההגבלה XmaI ו- BlpI.
- השתמש ב-2 μL של הפלסמיד שזה עתה הוכן כדי להפוך E.Coli בעל יכולת כימית כגון Stbl3 (50 μL) או XL10-Gold והתפשט על צלחת אגר LB עם אמפיצילין של 100 מיקרוגרם/מ"ל לאחר צמיחה של שעה אחת ב-37 מעלות צלזיוס ללא אנטיביוטיקה. אינקובציה בטמפרטורה של 37 מעלות צלזיוס למשך הלילה. בחרו כמה מושבות ובצעו מיני הכנה באמצעות ערכה מסחרית בהתאם להוראות היצרן.
- בצע הגברה של PCR בדנ"א של miniprep עם Primer_XmaI Primer_BlpR באמצעות תוכנית PCR 1 (ראו איור 2). הפרד את מוצרי ה-PCR על ג'ל אגרוז 1% במאגר TBE. בחר כמה מושבות (~5) עם רצועה בגודל הצפוי של כ-1300 כ"ס.
- בצע ריצוף DNA של המושבות הנבחרות, באמצעות Primer_XmaI או Primer_BlpR, כדי לאשר את ההחדרה הנכונה של קלטת ה- gRNA.
הערה: אחת המושבות המאומתות ברצף משמשת במחקר נוסף, והפלסמיד נקרא p_guides. - חזרו על הפעולה משלב 2.2 (בניית קלטות) עם ה-Primers-Killer F ו-R, וה-Primers-mCherry F ו-R. השתמשו במושבה אחת המאומתת ברצף לניתוח נוסף. הפלסמיד נקרא p_Killer.
3. ייצור לנטי-וירוס
- לייצר ולטהר כמות גדולה של כל הפלסמידים הנדרשים באמצעות ערכת הכנה למקסי ללא אנדוטוקסין בהתאם להוראות היצרן. הכן aliquots ב 2 מיקרוגרם / μL. יש לאחסן ב -20 °C (75 °F)
- הכנת תאים (יום 1)
- הכינו 18 צלחות של 145 ס"מ שנזרעו עם 1 x 106 תאי HEK293 לצלחת ב-16 מ"ל של מדיום המורכב מפירובט גלוקוז גבוה (DMEM) של Dulbecco, בתוספת 10% סרום בקר עוברי (FBS) ו-1% פניצילין/סטרפטומיצין. הגבר את התאים ב 37 °C (84 °F), בחממה 5% CO2 במשך 3 ימים.
הערה: ייצור של lentivirus חייב להתבצע בזהירות במעבדה ברמת בטיחות ביולוגית 2, באמצעות גלגלי הגנה מותאמים, כולל חליפת מגן חד פעמית, כובע מגן וכפפות. כל הניסויים חייבים להיעשות תחת מכסה מנוע זרימה למינרי (ארון בטיחות BSLII) עם טיפים למסנן. כל התמיסה המכילה לנטי-וירוסים וכל פסולת הפלסטיק/זכוכית המשומשת חייבת להיות מושתקת עם אתנול 70% או עם גורם אחר להשבתת הנגיף.
- הכינו 18 צלחות של 145 ס"מ שנזרעו עם 1 x 106 תאי HEK293 לצלחת ב-16 מ"ל של מדיום המורכב מפירובט גלוקוז גבוה (DMEM) של Dulbecco, בתוספת 10% סרום בקר עוברי (FBS) ו-1% פניצילין/סטרפטומיצין. הגבר את התאים ב 37 °C (84 °F), בחממה 5% CO2 במשך 3 ימים.
- טרנספקציה של תאים (יום 4)
- בדוק את המפגש של התאים, כדי להיות בטוח שהם הגיעו למפגש של 60%-65%.
- הכן את תמיסת הטרנספקציה המכילה עבור כל צלחת: 20.8 מיקרוגרם של הפלסמיד בעל העניין (p-guides, או p-Killer, או pCas9 #87904), 4.8 מיקרוגרם של הפלסמיד המקודד את המעטפה (VSV-G, #8454), 20.8 מיקרוגרם של הפלסמיד psPAX2 (#12260) עבור אריזה לנטי-ויראלית, 136 μL של סידן פוספט והתאם עם H2O לנפח סופי של 1,000 μL. הוסף תמיסה זו בצורה טיפתית ותחת תסיסה ל-1 מ"ל של 2x מלח בעל חציית HEPES (HBS).
הערה: אין להכין תערובת לכל הצלחות בו זמנית כדי להבטיח הכנה אופטימלית של ריאגנטים; להכין תערובת עבור שש צלחות בו זמנית, למשל, עבור 18 צלחות, להכין שלוש פעמים תערובת עבור שש צלחות. - דגירה בטמפרטורת החדר (RT) למשך 10 דקות לפחות והוספת 2 מ"ל של תמיסה באופן טיפתי לתאים. הומוגניזציה של מגיב הטרנספקציה עם תסיסה עדינה של הצלחת אחורה, קדימה, למעלה ולמטה, דגירה בטמפרטורה של 37 מעלות צלזיוס, 5% CO2 למשך 5 שעות לפחות.
הערה: מנקודה זו ועד סוף הייצור של lentivirus, ללבוש ציוד הגנה נוסף, כולל זוג שני של כפפות, שרוולי הגנה, ו plastron חד פעמי. - חמש שעות לאחר הטרנספקציה, הסירו את המדיום מהצלחות ושטפו עם PBS כדי להיפטר מריאגנטים טרנספקטיביים. הוסף 12 מ"ל של בינוני טרי ודגירה 48 שעות ב 37 °C , 5% CO2.
- איסוף החלקיקים הנגיפיים (יום 6)
- לאסוף ולאסוף את המדיום מכל הצלחות. צנטריפוגה ב 800 x g במשך 5 דקות ב 4 ° C, כדי כדורית פסולת סלולרית. סנן את חומר העל באמצעות מסנן של 0.45 מיקרומטר (נדרשים מסננים מרובים).
- צנטריפוגה ב 68,300 x g במשך 2 שעות ב 4 °C ברוטור דלי מתנדנד. הסר את ה- supernatant ותן לצינורות להתהפך מתחת לארון הבטיחות על נייר במשך 5-10 דקות כדי להסיר כמה שיותר נוזלים, ולאחר מכן הוסף 100 μL של מדיום התפשטות HEK לכל גלולה. לאחר לפחות 2 שעות ב-4 מעלות צלזיוס, החיזרו את הכדורים על ידי צנרת למעלה ולמטה. נצלו את כל הכדורים המחודשים.
הערה: ניתן להשאיר את הכדורים באמצע הלילה בטמפרטורה של 4 מעלות צלזיוס לפני ההצבעה. - Aliquot lentivirus ב 10 μL או 25 μL גודל המדגם (בהתאם לשימוש) והקפאה הצמדה עם חנקן נוזלי. יש לאחסן בטמפרטורה של -80 מעלות צלזיוס. אל תקפיאו אליקוות שהופשרו.
- חזור על שלבים 3.2 עד 3.4 עם פלסמידים אחרים בעלי עניין על מנת לייצר מדריכי LV, LV-Killer ו- LV-Cas9.
הערה: כחלופה, ניתן לרכוש את ה- lentiviruses מחברה או ממתקן וירוסים.
4. טיטרציה של לנטיוירוס
הערה: טיטרציה של הנגיף מבוצעת על תאי HEK293. הטיטרציה חשובה כדי לשלב בשלבים הבאים מספר מדויק של לנטי-וירוסים לכל תא (תהיה אשר תהיה האצווה של נגיף הלנטי), עבור התאים המעניינים. מספר החלקיקים הנגיפיים שמתמרים ביעילות תא נקרא ריבוי הזיהום (MOI): MOI 10 מתאים אפוא להכנסת 10 חלקיקים נגיפיים לכל תא. מכיוון שמחזור ההקפאה/ההפשרה משפיע על הכדאיות של נגיף הלנטי, הטיטרציה מבוצעת עם נגיף לנטי קפוא aliquot, וכל ניסוי עוקב יבוצע עם אליקוט חדש של אותה בריכה. שיטת טיטרציה אחת מתוארת כאן, אך ניתן להשתמש בשיטות אחרות.
- ביום הראשון, זרע 1 x 105 תאים לכל צלחת בחמש צלחות 35 מ"מ עם כיסויי זכוכית בתחתית ושני לוחות 35 מ"מ ללא כיסויים.
- ביום השני, משתי הלוחות ללא כיסויים, אוספים וסופרים את מספר התאים לאחר טריפסיניזציה, וקובעים את כמות התאים הממוצעת לכל צלחת (N).
- מכינים 100 μL של נגיף לנטי מדולל ב-1/10 במדיום התפשטות. תעתיקו את חמשת הלוחות המתורבתים עם נפחים שונים של נגיף מדולל, מ-1 עד 50 מיקרול' של נגיף מדולל. עבור פרוטוקול זה, השתמש בנפחים הבאים כדי להתמר את חמשת הלוחות: 1 μL, 5 μL, 10 μL, 20 μL, 50 μL. אינקובציה למשך 48 שעות ב 37 °C, ב 5% CO2.
- ביום 4, לתקן את התאים על ידי דגירה של כיסויים ב- RT במשך 20 עד 30 דקות ב 4% paraformaldehyde. עבור LV-Cas9, חלחלו לתאים עם 0.1% Triton X100 ב-Phosphate Buffer Saline (PBS) למשך 10 דקות ב-RT, רוויים ב-PBS-0.1% Triton X100-2% סרום עיזים - 0.5% אלבומין סרום בקר (BSA) למשך 20 דקות ב-RT, ותווית למשך 45 דקות ב-RT עם נוגדן ראשוני נגד V5 (דילול 1/400) ואחריו דגירה של 30 דקות ב-RT עם נוגדן משני פלואורסצנטי. תייג את הגרעינים עם Hoescht (10 מיקרוגרם/מ"ל ב-PBS) למשך 10 דקות ב-RT.
- הרכיבו את הכיסויים על מגלשה והתבוננו באמצעות מיקרוסקופ פלואורסצנטי המצויד במטרה של פי 20. עבור מדריכי LV ו- LV-Killer, לאחר הקיבעון המשך ישירות לסימון הגרעינים והרכב את הכיסויים. עבור כל כיסוי, ספרו את המספר הכולל של גרעינים (מספר התאים הכולל) בשדה הראייה ואת מספר התאים המסומנים (עם V5 או mCherry), וקבעו את יחס התאים המסומנים עבור כל כיסוי.
- בחר כיסוי שבו היחס בין התאים המתומרים הוא לפחות 10% ולא יעלה על 50%. קבע את יעילות ההעתקה (X) עבור כיסוי זה, ושים לב לנפח של וירוס מדולל (V, ב- μL) המשמש להשגת יעילות התמרה זו.
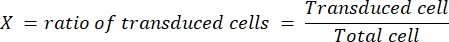
- קבע את הטיטר (בחלקיקים מזהמים, המיוצגים כ-ip/mL) של הנגיף על פי הנוסחה הבאה:

גורם הדילול הוא דילול נגיף הלנטי המבוצע בשלב 4.3. השגנו באופן שגרתי טיטר סופי של 1 x 109 ip/mL עבור LV-Cas9 ו-1 x 1010 10 ip/mL עבור מדריך LV, שנקבע בתאי HEK.
5. התמרת מיובלסט
הערה: המיובלסטים המונצחים מומרים ברצף עם שלושת הלנטי-וירוסים שיוצרו בעבר. הם נשמרים בצפיפות נמוכה מ-50% במדיום התפשטות המורכב מ-F10 של Ham עם תוספת של 20% FBS, 2% פניצילין/סטרפטומיצין, 2% אולטרוסר G, ותרבית ב-37 מעלות צלזיוס, 5% CO2.
- קבע את נפח נגיף הלנטי הדרוש לטיפול במספר התאים הנבחר עם MOI 10 עבור LV-Cas9 ו- LV-guides ו- MOI 20 עבור רוצח LV, על פי הנוסחה הבאה:
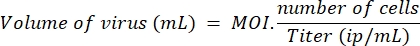
הערה: בניסוי מקביל, יעילות ההתמרה הושוותה על מיובלסטים ו- HEK, באמצעות נגיף לנטי בקרה (lenti-GFP), וקבענו כי יש צורך פי חמישה יותר לנטי-וירוסים כדי להתמר מיובלסט ביעילות בהשוואה לתא HEK. לפיכך, MOI 10 שנמדד על HEK מתאים לשני חלקיקים נגיפיים לכל מיובלסט. MOI המשמש כאן מחושבים על תאי HEK. - ביום הראשון, זרעו צלחות של 96 בארות עם 10,000 תאים ב-100 מיקרול' של מדיום התפשטות לבאר. ביום 2, תעתיק את התאים מתחת לארון הבטיחות על ידי הוספת הנפח המתאים של LV-guides ו- LV-Cas9 המחושב בשלב 5.1. מחזירים את התאים לחממה עד היום ה-7.
הערה: ייתכן שהשימוש ב-MOI גבוה מ-25, במיוחד עבור LV-Cas9, לא ישפר את מספר התאים הערוכים עקב מוות גבוה יותר של תאים בריכוז גבוה של נגיף הלנטי. - ביום 7, לבצע טריפסיניזציה ולספור את התאים. זרעו את התאים במפגש של 40% עד 50% בצלחת חדשה והחזירו את התאים לחממה. חמש שעות לאחר מכן, תמרו את התאים עם LV-Killer ב-MOI של 20 (נפח מחושב בשלב 5.1). הגבר את התאים במשך 5 עד 10 ימים לאחר ההעברה, עבור לפחות שני מעברים, ותמיד לשמור אותם במפגש נמוך של <50%. התאים מוכנים לשלב הבא, שיבוט תאים, כאשר הצמיחה שלהם תוחזר לקדמותה (מוערך על ידי זמן החלוקה).
הערה: ההתפשטות עשויה להיות מעט איטית יותר לאחר ההתמרה. ניתן לקבוע את צמיחת התאים הנורמלית לפני הליך ניסיוני זה על ידי הערכת זמן החלוקה של התאים.
6. שיבוט סלולרי
הערה: מכיוון שהתמרת מיובלסט היא קשה ולעולם אינה מגיעה ליעילות של 100%, גם בעת שימוש ב- lentivirus, נדרש שיבוט תאי על מנת לקבל קו תאים מתוקן במלואו. זה אפשרי רק עם תאים מונצחים, או תאים שניתן תרבית ולהגבירם במהלך כמה שבועות / חודשים.
- נסה לספור ולספור את התאים. מדללים את התאים בתווך התפשטות ב-10 תאים/מ"ל וזורעים את התאים ב-1 תאים/באר בלוחות של 96 באר המכילים 100 μL/באר של מדיום.
הערה: מספר הלוחות שיש לזרוע תלוי בהסתברות של עריכת הגנים הצפויה, 2 עד 10 לוחות נמצאים בשימוש שגרתי. - עקוב אחר התאים לצמיחה והגבר בהדרגה כל באר בצלחת גדולה יותר עד שתגיע לפחות לצלחת 35 מ"מ, תוך שמירה על המפגש של המיובלסטים מתחת ל -50%. שלב זה יכול להימשך 2-6 שבועות, תלוי בתאים המשמשים וביכולתם לגדול לאחר שהם מבודדים בבאר.
7. בחירת שיבוט
הערה: שלב זה מבוצע כדי לזהות אילו מהשיבוטים הגדלים שונו בהתאם.
- תכנן קבוצה של פריימרים המורכבים מפריימר הממוקם לפני המדריך הראשון (Primer_BeforeGuide1F) ופריימר נוסף הממוקם לאחר המדריך השני (Primer_AfterGuide2R) על מנת להגביר את האזור התוחם את הרצף שהשתנה באופן פוטטיבי. ראה טבלה 1 עבור הפריימרים המשמשים כאן.
- לאסוף תאים מכל שיבוט, לחסוך לפחות 300,000 תאים להגברה עתידית, ולחלץ דנ"א גנומי באמצעות כל פרוטוקול סטנדרטי על התאים הנותרים.
- אם מספר רב של שיבוטים גדלים, כדי להשליך את אלה שאינם ערוכים, בצע בדיקה מהירה על ידי איגום תאים מחמישה שיבוטים באותה שפופרת כדי לחלץ את הדנ"א ממאגר זה ולבדוק עם PCR. חזור עם כמה שיבוטים לפי הצורך. לאחר מכן, הפרד עוד יותר את הבריכות שהכילו תאים ערוכים כדי לבצע ניתוח פרטני.
- שלוט בעריכה על ידי PCR באופן הבא. הכן את תגובת ה- PCR עם 1 μL של Primer_BeforeGuide1F, 1 μL של Primer_AfterGuide2R, 12.5 μL של תערובת פולימראז, 3 μL של DNA גנומי, ו- 7.5 μL של H2O. הגבר את התרמוציפלר בתרמוציפלר על פי הוראות היצרן ופרמטרים פריימרים. הפעל על ג'ל אגרוז של 1% כדי לזהות את השיבוטים הערוכים.
- שלוט בעריכה על ידי רצף באופן הבא. בצע ריצוף סנגר של השיבוטים שנבחרו כדי לאשר את המחיקה ולזהות כיצד העריכה בוצעה בכל שיבוט. שמור יותר משיבוט ערוך אחד כדי לוודא שרק הגן הממוקד השתנה והוא אחראי להשפעה הפיזיולוגית שנצפתה ושמור על שיבוט לא ערוך שישמש כשיבוט בקרה (CTRL) בניסויים הבאים.
- הרחב את השיבוטים שנבחרו. לאחר שהמפגש של כל שיבוט הגיע לכ-50%, נסו את התאים וצלחו את התאים בצלחת גדולה יותר, עד שיוצרו מספיק תאים כדי לבצע את האפיונים הביוכימיים והתפקודיים (בדרך כלל יותר מ-1 x 106 לכל שיבוט), ואחסנו אליקוטים קפואים של כל שיבוט לשימוש עתידי.
8. אפיון שיבוטים ערוכים
הערה: לאחר שכמה שיבוטים נבחרו ואושרו על ידי ריצוף דנ"א, ניתן לאשר את מחיקת הגן הממוקד ברמת החלבון באמצעות כתם מערבי, וברמה התפקודית אם בדיקה תאית תפקודית זמינה עבור גן זה. במקרה של RYR1-KO, מכיוון ש-RyR1 היא תעלת סידן, האפיון הפונקציונלי בוצע באמצעות הדמיית סידן על תאים בתרבית.
- ביטוי חלבון בשיבוטים ערוכים
הערה: RyR1 מתבטא רק במיוטובות מובחנות10. הביטוי שלה הוערך במיוטובים באמצעות כתם מערבי, כדי לאשר את המחיקה ברמת החלבון של RyR1, כמו גם את המחיקה של חלבון Cas9.- צלחת 200,000 תאים במדיום התפשטות (מתואר לעיל, שלב 5) על משטח של כ-1.76 ס"מ2 בצלחת 35 מ"מ מצופה בלמינין (משטח המתאים לטיפה של 200 μL של למינין ב-10 מ"ג/מ"ל ב-PBS עם סידן). לאחר שהתאים נדבקים לצלחת לאחר הדגירה במשך 2-3 שעות בטמפרטורה של 37 מעלות צלזיוס, 5% CO2, העבירו את מדיום התרבית למדיום התמספית למדיום התמיינות המורכב מגלוקוז נמוך DMEM + 10% סרום סוסים + 1% פניצילין/סטרפטומיצין, והחזירו את התאים לאינקובטור למשך 6 ימים.
- לאחר 6 ימים של התמיינות, לאסוף וליזום את התאים עם 200 μL של RIPA בתוספת מעכבי פרוטאז. קבעו את ריכוז החלבון בשיטת Folin Lowry11.
- טענו 15 מיקרוגרם חלבון, לאחר דנטורציה למשך 30 דקות ב-RT במאגר הדנטורציה של לאמלי, על ג'ל אקרילאמיד שיפוע של 5%-15%. לאחר הפרדה אלקטרופורטית, העבירו את החלבונים באימובילון P ב-0.8 V למשך 4 שעותו-11.
- לאחר רוויה של הממברנה במשך 30 דקות ב- RT ב- PBS המכילה 0.1% Tween 20 ו- 5% חלב יבש שאינו שמן, דגירה של הממברנה עם הנוגדנים העיקריים המדוללים באותו חיץ במשך 2 שעות ב- RT או לילה ב- 4 °C, לשטוף את הממברנה 5x במשך 5 דקות עם PBS-0.1% Tween 20 ולדגום את הממברנה עם הנוגדנים המשניים במשך 1 שעה ב- RT. הנוגדנים העיקריים שבהם נעשה שימוש הם: נוגדנים נגד תג V5 (דילול: 1/5000) לזיהוי Cas9, אנטי-GAPDH (דילול: 1/1000) כבקרת העמסה, נוגדן נגד RyR112,13 (דילול: 1/10.000), נוגדן נגד תת-היחידה אלפא 1 של DHPR (דילול: 1/1000) ונוגדן נגד השרשרת הכבדה של מיוזין MF20 (דילול: 1/1000).
- לשטוף את הממברנה 5x במשך 5 דקות עם PBS-0.1% Tween 20, לייבש את עודף הנוזל ולהוסיף את המצע chemiluminescent. המשך כפי שהומלץ על ידי ספק המצע כדי לזהות את האות chemiluminescent.
- אפיון פונקציונלי של שיבוטים ערוכים
הערה: הפונקציה של RyR1 הוערכה באמצעות הדמיית סידן במיוטובים מובחנים, שהופקו משיבוטי CTRL או KO14.- צלחת 50,000 תאים על משטח 0.2 ס"מ2 במרכז כלים 35 מ"מ מצופה למינין (משטח מכוסה טיפת למינין של 50 μL, ב 10 מ"ג /מ"ל ב- PBS עם סידן) ולגרום התמיינות למשך 6 ימים כמתואר בשלב 8.1.1. הכינו שלוש צלחות לכל גירוי, כדי שיהיה טריפליקט ביולוגי.
- טען את המיוטובים עם 50 μL של פלואו 4-ישיר, מדולל 1:1 במדיום התמיינות ודגירה במשך 30 דקות ב 37 °C (64 °F). יש לשטוף את התאים פעמיים עם מאגר KREBS בתוספת גלוקוז במינון של 1 מ"ג/מ"ל.
- מדוד את הווריאציות הפלואורסצנטיות באמצעות מיקרוסקופ פלואורסצנטי הפוך או מיקרוסקופ קונפוקלי באמצעות מטרה של פי 10. התקינו את הצלחת על במת המיקרוסקופ והתחילו את הרכישה בפריים אחד לשנייה במשך 90 שניות.
- הסר את ה- KREBS הנותרים וגרה את התאים במסגרת 25 על ידי תוספת של 2 מ"ל של KCl לדה-פולריזציה של ממברנה (ריכוז סופי של 140 mM) או 2 מ"ל של 4 CmC (ריכוז סופי של 500 μM) עבור גירוי ישיר של RyR1. ודא שלפחות 10 myotubes נמצאים בשדה המוקלט.
- לכמת את השונות הפלואורסצנטית בכל מיוטוב, באמצעות תוכנה ייעודית. בחר לניתוח לפחות 10 מיו-טיובים למנה (באופן אידיאלי 20-30 מיו-טיובים למנה), צייר קו (או אזור עניין (ROI)) על הציר הארוך של כל מיוטו-טיוב ואסוף את הפלואורסצנציה F לאורך קו זה עבור כל המסגרות.
- קבע את הערך הפלואורסצנטי ההתחלתי, F0, המתאים למסגרות 1 עד 24. התווה את הווריאציה הפלואורסצנטית (F-F0)/F0 כפונקציה של זמן מ-0 עד 90 שניות. חזור על הניסוי שלוש פעמים כדי לקבל את הווריאציה הפלואורסצנטית מלפחות 90 מיו-טיובים משלוש תרבויות שונות. אסוף את כל התוצאות עבור 90 myotubes וחשב את הממוצע ± SEM של (F-F0)/F0 בכל מסגרת זמן. לכמת את משרעת השיא של שחרור סידן עבור כל גירוי וכל שיבוט.
תוצאות
פרוטוקול זה יושם על מיובלסטים מונצחים מנבדק בריא15 (מה שמכונה תאי HM, עבור מיובלסטים אנושיים), שבו RyR1 אופיין בעבר16, על מנת להפיל את הגן RYR1 המקודד את החלבון RyR1. התכנון של ה-RNA המנחה נעשה כדי למחוק את הרצף המקיף חלק מאקסון 101 ואינטרון 101 של הגן. מחיקה של חלק מאקסון 101 צ...
Discussion
צעד מרכזי בדרך לאפיון גנים בעלי תפקוד לא ידוע המעורבים בפתולוגיות הוא פיתוח מודלים תאיים רלוונטיים לחקר תפקודם של גנים אלה. השימוש בעריכת גנים באמצעות CRISPR/Cas9 הוא תחום מחקר הולך וגדל באופן אקספוננציאלי, ופיתוח מודלים של נוק-אאוט כפי שהוצגו כאן הוא בין היישומים הנפוצים ביותר שלו. בהקשר זה, א...
Disclosures
למחברים אין ניגודי עניינים לחשוף.
Acknowledgements
עבודה זו מומנה על ידי מענקים מטעם האגודה הצרפתית contre les myopathies (AFM-Téléthon) ומ-Auvergne-Rhône Alpes Région (AURA).
Materials
| Name | Company | Catalog Number | Comments |
| Anti-CACNA1S antibody | Sigma-Aldrich | HPA048892 | Primary antibody |
| Blp I | NE BioLabs | R0585S | Restriction enzyme |
| CalPhos Mammalian Transfection Kit | Takara | 631312 | Transfection kit |
| Easy blot anti Mouse IgG | GeneTex | GTX221667-01 | HRP secondary antibody |
| Easy blot anti Rabbit IgG | GeneTex | GTX221666 | HRP secondary antibody |
| Fluo-4 direct | Molecular Probes | F10472 | Calcium imaging |
| GAPDH(14C10) Rabbit mAb | Cell Signaling Technology | #2118 | Primary antibody |
| HindIII | Fermentas | ER0501 | Restriction enzyme |
| InFusion HD Precision Plus | Takara | 638920 | Ligation kit |
| MasterMix Phusion High Fidelity with GC | ThermoFisher Scientific | F532L | Mix for PCR reaction with High fidelity Taq polymerase and dNTPs |
| Myosin Heavy Chain antibody | DHSB | MF20 | Primary antibody |
| NucleoBond Xtra Maxi EF | Macherey-Nagel | REF 740424 | Maxipreparation kit for purification of plasmids |
| NucleoSpin Gel and PCR Clean-up | Macherey-Nagel | 740609 | DNA purification |
| NucleoSpin Tissue | Macherey-Nagel | 740952 | Kit for DNA extraction from cell |
| One Shot Stbl3 Chemically Competent E. coli | ThermoFisher Scientific | C737303 | Chemically competent cells |
| Plasmid #87904 | Addgene | 87904 | Lentiviral plasmid encoding the SpCas9 (for LV-Cas9) |
| Plasmid #87919 | Addgene | 87919 | Lentiviral backbone for insertion of cassette with guides (for LV-guide-target) |
| Plasmid #12260 | Addgene | 12260 | Lentiviral plasmid encoding lentiviral packaging GAG POL |
| Plasmid #8454 | Addgene | 8454 | Lentiviral plasmid encoding envelope protein for producing lentiviral and MuLV retroviral particles |
| V5 Tag Monoclonal Antibody | Invitrogene | R96025 | Primary antibody |
| XL10-Gold Ultracompetent Cells | Agilent | 200317 | Chemically competent cells |
| Xma I | NE BioLabs | R0180S | Restriction enzyme |
References
- Claussnitzer, M., Susztak, K. Gaining insight into metabolic diseases from human genetic discoveries. Trends in Genetics. 37 (12), 1081-1094 (2021).
- Fuster-García, C., García-Bohórquez, B., Rodríguez-Muñoz, A., Millán, J. M., García-García, G. Application of CRISPR tools for variant interpretation and disease modeling in inherited retinal dystrophies. Genes. 11 (5), 473 (2020).
- Modell, A. E., Lim, D., Nguyen, T. M., Sreekanth, V., Choudhary, A. CRISPR-based therapeutics: current challenges and future applications. Trends in Pharmacological Sciences. 43 (2), 151-161 (2022).
- Olson, E. N. Toward the correction of muscular dystrophy by gene editing. Proceedings of the National Academy of Sciences of the United States of America. 118 (22), (2021).
- Wu, X., Kriz, A. J., Sharp, P. A. Target specificity of the CRISPR-Cas9 system. Quantitative Biology. 2 (2), 59-70 (2014).
- Merienne, N., et al. The self-inactivating KamiCas9 system for the editing of CNS disease genes. Cell Reports. 20 (12), 2980-2991 (2017).
- Marty, I., Fauré, J. Excitation-contraction coupling alterations in myopathies. Journal of Neuromuscular Diseases. 3 (4), 443-453 (2016).
- Concordet, J. P., Haeussler, M. CRISPOR: intuitive guide selection for CRISPR/Cas9 genome editing experiments and screens. Nucleic Acids Research. 46, 242-245 (2018).
- Sun, Y., Sriramajayam, K., Luo, D., Liao, D. J. A quick, cost-free method of purification of DNA fragments from agarose gel. Journal of Cancer. 3, 93-95 (2012).
- Flucher, B. E., Conti, A., Takeshima, H., Sorrentino, V. Type 3 and type 1 ryanodine receptors are localized in triads of the same mammalian skeletal muscle fibers. The Journal of Cell Biology. 146 (3), 621-630 (1999).
- Hess, H. H., Lees, M. B., Derr, J. E. A linear Lowry--Folin assay for both water-soluble and sodium dodecyl sulfate-solubilized proteins. Analytical Biochemistry. 85 (1), 295-300 (1978).
- Garibaldi, M., et al. Dusty core disease' (DuCD): expanding morphological spectrum of RYR1 recessive myopathies. Acta Neuropathologica Communications. 7 (1), 3 (2019).
- Marty, I., et al. Biochemical evidence for a complex involving Dihydropyridine receptor and Ryanodine receptor in triad junctions of skeletal muscle. Proceedings of the National Academy of Sciences of the United States of America. 91 (6), 2270-2274 (1994).
- Oddoux, S., et al. Triadin deletion induces impaired skeletal muscle function. Journal of Biological Chemistry. 284 (50), 34918-34929 (2009).
- Mamchaoui, K., et al. Immortalized pathological human myoblasts: towards a universal tool for the study of neuromuscular disorders. Skeletal Muscle. 1, 34 (2011).
- Cacheux, M., et al. Functional characterization of a central core disease RyR1 mutation (p.Y4864H) associated with quantitative defect in RyR1 protein. Journal of Neuromuscular Diseases. 2 (4), 421-432 (2015).
- Luis, A. The old and the new: Prospects for non-integrating lentiviral vector technology. Viruses. 12 (10), 1103 (2020).
- Leenay, R. T., Beisel, C. L. Deciphering, communicating, and engineering the CRISPR PAM. Journal of Molecular Biology. 429 (2), 177-191 (2017).
- Salmon, P., Trono, D. Production and titration of lentiviral vectors. Current Protocols in Neurosciences. , (2006).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved