需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
酿酒酵母中单倍体交配效率的测定
摘要
在这项工作中,描述了一种定量酿 酒 酵母交配效率的稳健方法。该方法对于物种形成研究中合子前屏障的定量特别有用。
摘要
酿酒酵母 是遗传学、进化和分子生物学中广泛使用的模式生物。近年来,它也成为研究物种形成相关问题的流行模式生物。酵母的生命周期涉及无性和有性生殖阶段。易于进行进化实验和生物体的短生成时间允许研究生殖屏障的进化。两种配接类型(a 和 α)配合形成 a/α 二倍体的效率称为配接效率。单倍体之间交配效率的任何降低都表明存在合子前屏障。因此,为了量化两个单倍体之间的生殖隔离程度,需要一种可靠的方法来量化交配效率。为此,这里提出了一个简单且高度可重复的协议。该方案涉及四个主要步骤,包括修补YPD板上的单倍体,将等数量的单倍体混合,稀释和接种单个菌落,最后,根据脱落板上的菌落数量计算效率。使用营养标记来清楚地区分单倍体和二倍体。
引言
酿酒酵母,通常称为出芽酵母,是一种单细胞真核生物。它有两种交配类型, a 和 α, 并表现出无性和有性生殖周期。 a 和 α 交配类型是单倍体,可以在周围环境中没有其他交配类型的情况下进行有丝分裂,这代表了酵母的无性循环。当两种交配类型非常接近时,它们停止有丝分裂并融合形成二倍体细胞。二倍体酵母可以在营养物质存在时有丝分裂,也可以在存在不可发酵的不良碳源(例如乙酸盐1)的情况下在氮饥饿的条件下进行减数分裂。这导致孢子的形成,孢子保持休眠状态,直到有有利的生长条件。当这些孢子萌发并且两种单倍体类型被释放回单倍体池2,3 时,生命周期就完成了(图1)。
酵母细胞的交配包括几个步骤,例如凝集,形成交配投影或"shmoo",然后是细胞和核融合4,5。两种配接类型 a 和 α分别产生 a 因子和 α 因子以启动配接。这些因子是多肽信息素,与存在于相反交配类型5的细胞表面的受体(Ste2和Ste3)结合。信息素与受体的结合启动信息素反应途径,即丝裂原活化蛋白激酶(MAPK)信号转导途径6,7,8。这导致细胞周期在G1期停滞,导致代谢活跃的固定相9。然后细胞停止有丝分裂,合成交配所需的蛋白质。由于单倍体细胞不能相互移动,交配投影或"shmoo"指向交配伴侣。当细胞接触时,细胞壁降解,细胞质内容物融合,导致交配形成二倍体细胞10,11。单倍体之间的交配效率已被用作实验室进化菌株以及现存物种之间物种形成的量度12。
作为一种简单的真核生物,酵母是与复杂真核生物相关的大量研究问题的首选模型。其中一个问题与物种形成和生殖屏障的进化有关13,14。对于有性繁殖的生物,物种由Ernst Mayr15提出的生物物种概念(BSC)定义。根据这个概念,一个种群中的两个个体如果不能杂交并且繁殖隔离,则被称为属于两个不同的物种。有性生殖周期的破坏(包括配子融合形成受精卵,受精卵发育成后代,以及后代性成熟)导致生殖隔离。 如图1所示, 酿酒酵母 的生命周期与有性生殖周期相当:a)两种交配类型 a 和 α 的融合类似于有性繁殖生物中配子的融合;b)二倍体进行有丝分裂的能力相当于受精卵发育成后代;c)经历孢子形成的二倍体与配子发生过程相当14。
当观察到配种交配时,就会发生合子前分离。如果有机会与两种遗传上不同的 A 型交配, α 型优先与一种交配,反之亦然14。在单倍体在不同环境中进化的进化实验中,可以通过进行交配测定来确定交配前屏障的存在。与祖先相比,交配效率的降低表明交配前屏障的进化。由于二倍体无法进行有效的有丝分裂和/或孢子形成单倍体孢子,因此可能会出现合子分离后14。这些可以通过分别测量二倍体的生长速率和计算孢子形成效率来量化。因此,为了研究生殖屏障的进化,需要强大的方法来量化(a)交配效率,(b)二倍体的有丝分裂生长和(c)二倍体的孢子形成效率。在这项工作中,报告了一种量化酵母菌株交配效率的稳健方法。
在实验室实验中,检测交配发生的方法之一是使用补充营养需求的营养标志物。当两种交配类型对两种不同的氨基酸具有营养性时,只有两种交配类型融合形成的二倍体细胞才能在缺乏两种氨基酸的培养基上生长。因此,营养型标记物可用于定性和定量检测交配。定性测试足以确定减数分裂16 后菌株的交配类型。当一个人有兴趣在研究交配途径中涉及的基因时确定交配减少时,定量测试是必不可少的17,18.此外,随着酵母越来越多地用于物种形成研究,一种方便且可重复的交配测定是必要的,因为交配效率的量化是合子前屏障的衡量标准。
两种酵母交配类型之间的交配效率先前已量化为16,19,20。大多数以前使用的方法在设计上都是相似的,只有一些变化16,21,22,23,24,25。其中一些使用早期对数期培养物,而其他一些使用单倍体菌株的对数期中期培养物。两种交配类型混合的比例存在差异。几乎所有的方案都使用硝酸纤维素膜。从先前生长的培养物中取出的两种交配类型的悬浮液混合并过滤到放置在YPD板上的硝酸纤维素膜上。在协议的变体之一中,单倍体悬浮液直接贴在YPD板21上。在处理参与两种交配类型信息素产生的基因的实验中,在使两种交配类型的悬浮液24的同时向外部添加信息素。
混合单倍体后孵育几个小时(通常约5小时)后,将细胞从膜上洗掉,稀释并铺在选择性培养基上。在1973年报道的早期方法之一中,受精卵形成或交配的效率是通过使用血细胞计数器在显微镜下计数芽细胞,未出芽细胞和交配对的数量来计算的。然而,后来报道的大多数方法使用营养标记来区分单倍体和二倍体。交配效率计算为二倍体细胞相对于细胞池中二倍体和单倍体细胞数量的百分比16,21,23。
然而,尽管有许多报道使用酵母作为模型生物来研究物种形成,但到目前为止,文献中还没有报告用于计算交配效率的标准化方案。对数期的细胞可能不是量化交配效率的理想选择。在交配过程中,两个单倍体的细胞周期被阻止,因此,交配过程中的细胞没有分裂9。由于已知细胞周期在固定相27的细胞中被类似地阻滞,因此使用这种细胞可以使方案更具可重复性。固定相细胞可以混合并放置在YPD板上(即营养丰富的环境)上进行交配。传统程序还需要硝酸纤维素膜并冲洗掉细胞,使该过程繁琐且容易出现处理错误。此外,迄今为止使用的协议以一个单倍体来量化交配效率。然而,在测量生殖隔离时,对特定单倍体组合而不是单个单倍体的交配效率进行量化。
为了解决这些问题,我们在这里报告了一种用于定量酵母交配效率的稳健方法,该方法具有高度可重复性和易于使用性。此外,这种方法和这里采用的酵母菌株也可用于研究基因流对交配屏障进化的影响。
本研究使用了两种不同的酿酒酵母菌株。其中一个菌株来源于SK1背景;在我们的实验室中,通过在MAT位点附近添加营养不良标志物对此进行了修改。所得的单倍体基因型见表1 28、29、30。在SK1菌株中,单倍体在MAT位点附近插入TRP1基因,α单倍体在MAT位点附近插入LEU2基因。在ScAM菌株中,TRP1和URA3基因分别插入a和α单倍体中。插入的位置位于III号染色体的ARS区域(Chr III:197378..197609)。对于这里报告的协议,基因组上任何地方的营养标记就足够了。然而,在MAT位点附近具有营养性标记意味着这些菌株也可用于研究基因流对物种形成的影响31,32。标记物被添加到MAT位点附近,以防止由于重组而导致标记物的重新洗牌。因此,该协议可用于量化涉及物种形成的研究中的交配效率,以及在研究参与交配途径的蛋白质时确定交配效率的改变。
Access restricted. Please log in or start a trial to view this content.
研究方案
注意:该协议广泛涉及以下步骤:(1)在YPD板上修补交配效率网格中的单倍体,(2)孵育24小时后将等数量的单倍体混合,并给混合单倍体几个小时的交配时间(本研究中为7小时),(3)将混合细胞接种在YPD上,以便在30°C下7小时后分离单个菌落, 最后,(4)确定使用营养不足标记物形成的二倍体的数量。下面将详细讨论这些步骤(另请参阅 图 2)。
1. 拼接效率网格中的单倍体
- 通过在YPD琼脂平板(2%琼脂,2%葡萄糖,1%蛋白胨,0.5%酵母提取物)上划线,从冷冻储液中恢复单倍体 a 和α ,并让它们在30°C下生长48小时以获得分离的单菌落。
- 将YPD板中的单个菌落接种在5mL YPD培养基(2%葡萄糖,1%蛋白胨,0.5%酵母提取物)中,并在30°C下以250rpm振荡孵育48小时。在此孵育期之后,细胞处于生长的固定阶段。
- 在新鲜的YPD板上绘制配接效率网格。将网格绘制为一个 1 cm x 1.5 cm 的矩形,分为三个框,每个框的尺寸为 1 cm x 0.5 cm,如图 2A 所示。
- 最左侧和最右侧矩形上两种配对类型的 YPD 培养物的补丁 5 μL(图 2B)。该体积对应于每个部分中布置的大约 5 x 105 个 单倍体细胞。将板在30°C孵育24小时。
注意:网格的目的是使实验测量精确(例如实验中的细胞数)。网格尺寸足够小,可以在实验上处理,但又足够大,可以很容易地操纵(如从网格中提升细胞),并且不容易受到漂移或偶然事件的影响。
2. 单倍体的混合和交配
注意:24小时后(图2C),将相同数量的两种单倍体类型的细胞从两个网格上刮下来,混合并放置在中心矩形中(图2D)。
- 为了混合相同数量的细胞,使用无菌牙签去除放置在外盒中的约1/3贴片,并将每个单倍体重悬于无菌1.5mL小瓶中的20μL水中。
- 在 2 mL 水中稀释 5 μL 该悬浮液。使用分光光度计在600nm处测量该稀释悬浮液的OD。根据该特定菌株的 OD 值和 1 OD 中的细胞数/mL,混合来自两种菌株的相同数量的细胞。计算需要混合的体积,并从剩余的 15 μL 单个单倍体悬浮液中吸出。
注意:在中心矩形中修补的单元格数量使得单元格形成单层。考虑到酵母细胞是一个半径为2.58μm33的球体,一个1cm x 0.5cm的矩形盒子需要大约1.7 x 106个细胞才能形成单层。应注意确保修补用于交配的细胞与两个单倍体细胞网格之间没有物理接触。由于每种类型的单倍体细胞必须混合相同数量,因此从每种菌株中加入8.5 x 105个细胞。细胞数基于OD测量值计算,考虑600nm处的1 OD近似相当于1 x 107细胞34。例如,如果单倍体悬浮液的OD 600为0.17,α单倍体的OD600为0.11,则可以计算每个单倍体悬浮液的5μL中的细胞数。为确保每种单倍体类型有 8.5 x 105 个细胞,混合 1.25 μL 单倍体和 1.93 μL α 单倍体悬浮液。 - 将所需体积的两种单倍体加入新鲜无菌的 1.5 mL 小瓶中,并使用移液器充分混合。该悬浮液的最终体积通常在 6–8 μL 左右。 在中心网格中修补此悬浮液。将板在30°C孵育7小时,使单倍体有足够的时间交配(图2E)。
3. 在YPD琼脂上接种混合细胞
- 孵育期7小时后,使用牙签或移液器吸头从中心矩形刮下细胞,并在2mL无菌水中稀释。然后,将细胞悬液散布在YPD琼脂上以获得单个菌落。为了确定获得单个菌落所需的稀释因子,测量将刮片细胞加入的第一管的OD。需要为所使用的每种细胞类型/菌株确定特定的稀释因子。
注意:例如,OD 600 为 0.15 对应于 2 mL 悬浮液中的 3 x 106 个细胞(考虑 1 OD = 1 x 107 个细胞/mL)。为了在YPD平板上获得几百个菌落,将细胞悬液以1:20连续稀释两次,然后使用100μL的最终稀释液进行扩散。 - 铺板后,将YPD板在30°C孵育36-48小时,直到出现单个菌落。确保从每个交配实验中获得几百个单独的菌落进行筛选,以确保可以在数据中检测到统计学意义(图2F)。
4. 使用营养标志物筛选二倍体
- 确定获得的菌落中有多少部分是二倍体。为了鉴定平板上的二倍体菌落,通过将单个菌落单独划线到缺乏菌株营养的氨基酸的双脱落板(2%葡萄糖,0.66%氮碱,0.05%双脱落氨基酸混合物和2%琼脂)上转移单细胞菌落,如图 2G所示。将板在30°C孵育48小时。
注意:菌落也可以使用复制板转移到双脱落板上。本研究使用色氨酸和亮氨酸(trp− leu−)辍学培养基定量SK1AM菌株的交配效率,色氨酸和尿嘧啶(trp− ura−)脱落培养基用于ScAM菌株。只有二倍体菌落在双脱落板上生长,因为它们具有两种营养标记:SK1AM菌株中的TRP1和LEU2基因以及ScAM菌株中的TRP1和URA3基因。 - 此外,在单个脱落培养基(trp−或leu−或ura−)上划线或复制菌落,以量化种群中两种单倍体中每种的频率。
- η计算配接效率,如下所示:
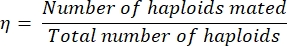 均衡器 (1)
均衡器 (1)
其中交配的单倍体数量仅等于双脱落板上确定的二倍体数量的两倍(因为每个二倍体都是由两个单倍体的交配产生的)。单倍体总数等于条纹单倍体数加上条纹二倍体数的两倍之和。
注意:例如,如果在双脱落培养基上划线/复制接种 100 个菌落后仅生长 60 个菌落,则交配效率可以量化为 75%(因为 60 x 2 个单倍体交配形成 60 个二倍体,40 个单倍体未交配)。
Access restricted. Please log in or start a trial to view this content.
结果
两种插拔类型配接效率的量化
此处描述的方案用于量化两种酵母菌株之间的交配效率 - SK1AM a 和 SK1AMα 之间以及 ScAMa 和 ScAMα 之间的交配效率(图 3A)。在这些实验中,两个单倍体之间的交配重复至少12次。在实验的每个重复中,至少有100个菌落在双脱落培养基上划线。该方案的稳健性使得SK1AM和ScAM两种菌株?...
Access restricted. Please log in or start a trial to view this content.
讨论
酿酒酵母交配效率的量化对于开展与交配途径相关基因相关的研究或研究外部环境对交配行为的影响至关重要。在过去的二十年中,酿酒酵母也已成为解决与物种形成14,36,37,38相关的问题的流行模型。两种交配类型的存在以及在实验室环境中易于进行遗传操作和维护,使其成为实时?...
Access restricted. Please log in or start a trial to view this content.
披露声明
作者声明他们在这项工作中没有竞争利益。作者很高兴分享SK1衍生的菌株,用于所有非营利用途。
致谢
这项工作由DBT/威康信托基金会(印度联盟)赠款(IA/S/19/2/504632)资助,由DBT/威康信托基金会(印度联盟)资助(IA/S/19/2/504632)资助。A.M.作为高级研究员(09/087(0873)/2017-EMR-I)得到了印度政府科学与工业研究委员会(CSIR)的支持。作者感谢Paike Jayadeva Bhat的讨论。
Access restricted. Please log in or start a trial to view this content.
材料
| Name | Company | Catalog Number | Comments |
| Adenine | Sigma Life Science | A8626 | |
| Agar Powder regular grade for bacteriology | SRL | 19661 (0140186) | |
| Ammonium Sulphate, Hi-AR | HiMedia | GRM1273 | |
| D-(+)-glucose | Sigma Life Science | G8270 | |
| Glass Petri plates | HiMedia | PW008 | 90 mm x 15 mm dimension |
| L-Arginine | Sigma Life Science | A8094 | |
| L-Aspartic acid | Sigma Life Science | A7219 | |
| L-Histidine monochloride monohydrate | Sigma Life Science | H5659 | |
| L-Isoleucine | Sigma Aldrich | I2752 | |
| L-Leucine | Sigma Life Science | L8912 | |
| L-Lysine | Aldrich | 62840 | |
| L-Methionine | Sigma Life Science | M5308 | |
| L-Phenylalanine | Sigma Life Science | P5482 | |
| L-Threonine | Sigma Aldrich | T8625 | |
| L-Tyrosine | Sigma Life Science | T8566 | |
| L-Valine | Sigma Life Science | V0513 | |
| Mating efficiency grid | 1 cm x 1.5 cm rectangular grid drawn on the Petri plate | ||
| Microcentrifuge tubes | Tarsons | 500010 | |
| Peptone | HiMedia | RM001 | |
| Uracil | Sigma Life Science | U0750 | |
| Yeast Extract Powder | HiMedia | RM027 | |
| Yeast Nitrogen Base w/o Amino acids and Ammonium Sulphate | BD Difco | 233520 |
参考文献
- Neiman, A. M. Sporulation in the budding yeast Saccharomyces cerevisiae. Genetics. 189 (3), 737-765 (2011).
- Duina, A. A., Miller, M. E., Keeney, J. B. Budding yeast for budding geneticists: A primer on the Saccharomyces cerevisiae model system. Genetics. 197 (1), 33-48 (2014).
- Herskowitz, I. Life cycle of the budding yeast Saccharomyces cerevisiae. Microbiological Reviews. 52 (4), 536-553 (1988).
- Erdman, S., Lin, L., Malczynski, M., Snyder, M. Pheromone-regulated genes required for yeast mating differentiation. Journal of Cell Biology. 140 (3), 461-483 (1998).
- Merlini, L., Dudin, O., Martin, S. G. Mate and fuse: How yeast cells do it. Open Biology. 3 (3), 130008(2013).
- Gustin, M. C., Albertyn, J., Alexander, M., Davenport, K. MAP kinase pathways in the yeast Saccharomyces cerevisiae. Microbiology and Molecular Biology Reviews. 62 (4), 1264-1300 (1998).
- Bardwell, L. A walk-through of the yeast mating pheromone response pathway. Peptides. 26 (2), 339-350 (2005).
- Reid, B. J., Hartwell, L. H. Regulation of mating in the cell cycle of Saccharomyces cerevisiae. Journal of Cell Biology. 75, 355-365 (1977).
- Williams, T. C., Peng, B., Vickers, C. E., Nielsen, L. K. The Saccharomyces cerevisiae pheromone-response is a metabolically active stationary phase for bio-production. Metabolic Engineering Communications. 3, 142-152 (2016).
- Bagnat, M., Simons, K. Cell surface polarization during yeast mating. Proceedings of the National Academy of Sciences of the United States of America. 99 (22), 14183-14188 (2002).
- Trueheart, J., Boeke, J. D., Fink, G. R. Two genes required for cell fusion during yeast conjugation: Evidence for a pheromone-induced surface protein. Molecular and Cellular Biology. 7 (7), 2316-2328 (1987).
- Sniegowski, P. D., Dombrowski, P. G., Fingerman, E. Saccharomyces cerevisiae and Saccharomyces paradoxus coexist in a natural woodland site in North America and display different levels of reproductive isolation from European conspecifics. FEMS Yeast Research. 1 (4), 299-306 (2002).
- Replansky, T., Koufopanou, V., Greig, D., Bell, G. Saccharomyces sensu stricto as a model system for evolution and ecology. Trends in Ecology and Evolution. 23 (9), 494-501 (2008).
- Greig, D. Reproductive isolation in Saccharomyces. Heredity. 102 (1), 39-44 (2009).
- Mayr, E. Systematics and the Origin of Species, from the Viewpoint of a Zoologist. , Harvard University Press. Cambridge, MA. (1999).
- Sprague, G. F. Assay of yeast mating reaction. Methods in Enzymology. 194, 77-93 (1991).
- McCaffrey, G., Clay, F. J., Kelsay, K., Sprague, G. F. Identification and regulation of a gene required for cell fusion during mating of the yeast Saccharomyces cerevisiae. Molecular and Cellular Biology. 7 (8), 2680-2690 (1987).
- Valtz, N., Peter, M., Herskowitz, I. FAR1 is required for oriented polarization of yeast cells in response to mating pheromones. Journal of Cell Biology. 131 (4), 863-873 (1995).
- Maclean, C. J., Greig, D. Prezygotic reproductive isolation between Saccharomyces cerevisiae and Saccharomyces paradoxus. BMC Evolutionary Biology. 8, 1(2008).
- Murphy, H. A., Kuehne, H. A., Francis, C. A., Sniegowski, P. D. Mate choice assays and mating propensity differences in natural yeast populations. Biology Letters. 2 (4), 553-556 (2006).
- Leu, J. Y., Murray, A. W. Experimental evolution of mating discrimination in budding yeast. Current Biology. 16 (3), 280-286 (2006).
- Kim, J., Hirsch, J. P. A nucleolar protein that affects mating efficiency in Saccharomyces cerevisiae by altering the morphological response to pheromone. Genetics. 149 (2), 795-805 (1998).
- Jin, M., et al. Yeast dynamically modify their environment to achieve better mating efficiency. Science Signaling. 4 (186), (2011).
- Rogers, D. W., Denton, J. A., McConnell, E., Greig, D. Experimental evolution of species recognition. Current Biology. 25 (13), 1753-1758 (2015).
- McClure, A. W., Jacobs, K. C., Zyla, T. R., Lew, D. J. Mating in wild yeast: Delayed interest in sex after spore germination. Molecular Biology of the Cell. 29 (26), 3119-3127 (2018).
- Sena, E. P., Radin, D. N., Fogel, S. Synchronous mating in yeast. Proceedings of the National Academy of Sciences of the United States of America. 70 (5), 1373-1377 (1973).
- Werner-Washburne, M., Braun, E., Johnston, G. C., Singer, R. A. Stationary phase in the yeast Saccharomyces cerevisiae. Microbiology Reviews. 57 (2), 383-401 (1993).
- Johnston, S. A., Hopper, J. E. Isolation of the yeast regulatory gene GAL4 and analysis of its dosage effects on the galactose/melibiose regulon. Proceedings of the National Academy of Sciences of the United States of America. 79 (22), 6971-6975 (1982).
- Mahilkar, A. Study of metabolic specialization leading to speciation, using yeast as a model system. , Indian Institute of Technology. Bombay, Mumbai. PhD thesis (2021).
- Blank, T. E., Woods, M. P., Lebo, C. M., Xin, P., Hopper, J. E. Novel Gal3 proteins showing altered Gal80p binding cause constitutive transcription of Gal4p-activated genes in Saccharomyces cerevisiae. Molecular and Cellular Biology. 17 (5), 2566-2575 (1997).
- Rice, W. R., Hostert, E. E. Laboratory experiments on speciation: What have we learned in 40 years. Evolution. 47 (6), 1637-1653 (1993).
- White, N. J., Snook, R. R., Eyres, I. The past and future of experimental speciation. Trends in Ecology and Evolution. 35 (1), 10-21 (2020).
- Milo, R., Jorgensen, P., Moran, U., Weber, G., Springer, M. BioNumbers--The database of key numbers in molecular and cell biology. Nucleic Acids Research. 38, 750-753 (2010).
- Domitrovic, T., et al. Structural and functional study of YER067W, a new protein involved in yeast metabolism control and drug resistance. PLoS One. 5 (6), 11163(2010).
- Lindegren, C. C., Spiegelman, S., Lindegren, G. Mendelian inheritance of adaptive enzymes. Proceedings of the National Academy of Sciences of the United States of America. 30 (11), 346-352 (1944).
- Dettman, J. R., Sirjusingh, C., Kohn, L. M., Anderson, J. B. Incipient speciation by divergent adaptation and antagonistic epistasis in yeast. Nature. 447 (7144), 585-588 (2007).
- Jhuang, H. Y., Lee, H. Y., Leu, J. Y. Mitochondrial-nuclear co-evolution leads to hybrid incompatibility through pentatricopeptide repeat proteins. EMBO Reports. 18 (1), 87-101 (2017).
- Lee, H. Y., et al. Incompatibility of nuclear and mitochondrial genomes causes hybrid sterility between two yeast species. Cell. 135 (6), 1065-1073 (2008).
- Pronk, J. T. Auxotrophic yeast strains in fundamental and applied research. Applied and Environmental Microbiology. 68 (5), 2095-2100 (2002).
- Madhani, H. D. From a to α: Yeast as a Model for Cellular Differentiation. , Cold Spring Harbor Laboratory Press. Long Island, New York. (2007).
- Huxley, C., Green, E. D., Dunham, I. Rapid assessment of S. cerevisiae mating type by PCR. Trends in Genetics. 6 (8), 236(1990).
- Rang, C., Galen, J. E., Kaper, J. B., Chao, L. Fitness cost of the green fluorescent protein in gastrointestinal bacteria. Canadian Journal of Microbiology. 49 (9), 531-537 (2003).
- Tusso, S., Nieuwenhuis, B. P. S., Weissensteiner, B., Immler, S., Wolf, J. B. W. Experimental evolution of adaptive divergence under varying degrees of gene flow. Nature Ecology and Evolution. 5 (3), 338-349 (2021).
Access restricted. Please log in or start a trial to view this content.
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。