Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Determinación de la eficiencia de apareamiento de haploides en Saccharomyces cerevisiae
En este artículo
Resumen
En este trabajo, se describe un método robusto para la cuantificación de la eficiencia de apareamiento en la levadura Saccharomyces cerevisiae . Este método es particularmente útil para la cuantificación de barreras precigóticas en estudios de especiación.
Resumen
Saccharomyces cerevisiae es un organismo modelo ampliamente utilizado en genética, evolución y biología molecular. En los últimos años, también se ha convertido en un organismo modelo popular para estudiar problemas relacionados con la especiación. El ciclo de vida de la levadura implica fases reproductivas asexuales y sexuales. La facilidad para realizar experimentos de evolución y el corto tiempo de generación del organismo permiten el estudio de la evolución de las barreras reproductivas. La eficiencia con la que los dos tipos de acoplamiento (a y α) se acoplan para formar el diploide a/α se conoce como eficiencia de acoplamiento. Cualquier disminución en la eficiencia de apareamiento entre haploides indica una barrera precigótica. Por lo tanto, para cuantificar el grado de aislamiento reproductivo entre dos haploides, se requiere un método robusto para cuantificar la eficiencia de apareamiento. Con este fin, se presenta aquí un protocolo simple y altamente reproducible. El protocolo implica cuatro pasos principales, que incluyen parchear los haploides en una placa YPD, mezclar los haploides en números iguales, diluir y chapar para colonias individuales y, finalmente, calcular la eficiencia basada en el número de colonias en una placa de abandono. Los marcadores auxotrofos se emplean para hacer claramente la distinción entre haploides y diploides.
Introducción
Saccharomyces cerevisiae, comúnmente llamada levadura en ciernes, es un eucariota unicelular. Tiene dos tipos de apareamiento, a y α, y exhibe ciclos reproductivos asexuales y sexuales. Los tipos de apareamiento a y α son haploides y pueden dividirse mitóticamente en ausencia del otro tipo de apareamiento en el entorno circundante, que representa el ciclo asexual de la levadura. Cuando los dos tipos de apareamiento están muy cerca, dejan de dividirse mitóticamente y se fusionan para formar una célula diploide. La levadura diploide puede dividirse mitóticamente cuando los nutrientes están presentes o sufrir meiosis en condiciones de falta de nitrógeno en presencia de una fuente de carbono pobre que no es fermentable, como el acetato1. Esto resulta en la formación de esporas, que permanecen latentes hasta que haya condiciones de crecimiento favorables. El ciclo de vida se completa cuando estas esporas germinan y los dos tipos haploides se liberan de nuevo a la piscina haploide 2,3 (Figura 1).
El apareamiento de las células de levadura incluye varios pasos, como la aglutinación, la formación de una proyección de apareamiento o "shmoo", seguida de la fusión celular y nuclear 4,5. Los dos tipos de apareamiento a y α producen factor a y factor α, respectivamente, para iniciar el apareamiento. Estos factores son feromonas polipeptídicas que se unen a los receptores (Ste2 y Ste3) presentes en la superficie celular del tipo de apareamiento opuesto5. La unión de las feromonas a los receptores inicia la vía de respuesta de feromonas, la vía de transducción de señales de la proteína quinasa activada por mitógenos (MAPK) 6,7,8. Esto resulta en la detención del ciclo celular en la fase G1, lo que lleva a una fase estacionaria metabólicamente activa9. Las células dejan de dividirse mitóticamente y se sintetizan las proteínas necesarias para el apareamiento. Como las células haploides no pueden moverse una hacia la otra, una proyección de apareamiento o "shmoo" se dirige hacia el compañero de apareamiento. Cuando las células entran en contacto, la pared celular se degrada y el contenido citoplasmático se fusiona, lo que resulta en el apareamiento para formar una célula diploide10,11. La eficiencia de apareamiento entre haploides se ha utilizado como medida de especiación en cepas evolucionadas en laboratorio, así como entre especies existentes12.
Al ser un organismo eucariota simple, la levadura es el modelo de elección para un gran número de preguntas de investigación asociadas con organismos eucariotas complejos. Una de esas preguntas está asociada a la especiación y a la evolución de las barreras reproductivas13,14. Para los organismos que se reproducen sexualmente, una especie se define por el concepto de especie biológica (BSC) propuesto por Ernst Mayr15. Según este concepto, se dice que dos individuos de una población pertenecen a dos especies diferentes si no pueden cruzarse y están aislados reproductivamente. La ruptura del ciclo reproductivo sexual (que implica la fusión de gametos para formar un cigoto, el desarrollo del cigoto en una progenie y el logro de la madurez sexual en la progenie) conduce al aislamiento reproductivo. Como se muestra en la Figura 1, el ciclo de vida de S. cerevisiae es comparable al ciclo reproductivo sexual: a) la fusión de los dos tipos de apareamiento a y α es similar a la fusión de gametos en organismos que se reproducen sexualmente; b) la capacidad del diploide para sufrir división mitótica es equivalente a que el cigoto se convierta en progenie; y c) el diploide sometido a esporulación es comparable al proceso de gametogénesis14.
El aislamiento precigótico ocurre cuando se observa apareamiento selectivo. Dada la misma oportunidad de aparearse con dos tipos a genéticamente diferentes, un tipo α se aparea preferentemente con uno sobre el otro o viceversa14. En el caso de experimentos de evolución en los que los haploides han evolucionado en diferentes entornos, la presencia de una barrera previa al apareamiento se puede determinar mediante la realización de un ensayo de apareamiento. Una disminución en la eficiencia de apareamiento en comparación con el ancestro indica la evolución de una barrera previa al apareamiento. El aislamiento postcigótico podría surgir debido a la incapacidad del diploide para sufrir una división mitótica efectiva y/o esporulación para formar esporas haploides14. Estos pueden cuantificarse midiendo la tasa de crecimiento de los diploides y calculando la eficiencia de esporulación, respectivamente. Por lo tanto, para estudiar la evolución de las barreras reproductivas, se requieren métodos robustos para cuantificar (a) la eficiencia de apareamiento, (b) el crecimiento mitótico del diploide y (c) la eficiencia de esporulación del diploide. En este trabajo, se informa un método robusto para cuantificar la eficiencia de apareamiento de las cepas de levadura.
En experimentos de laboratorio, una de las formas en que se puede detectar la ocurrencia de apareamiento es mediante el uso de marcadores auxotróficos que complementan los requisitos nutricionales. Cuando los dos tipos de apareamiento son auxotrofos para dos aminoácidos diferentes, solo la célula diploide formada por la fusión de los dos tipos de apareamiento puede crecer en un medio deficiente en ambos aminoácidos. Por lo tanto, los marcadores auxotrofos son útiles para detectar el apareamiento tanto cualitativa como cuantitativamente. Una prueba cualitativa será suficiente para identificar el tipo de apareamiento de una cepa después de la meiosis16. Las pruebas cuantitativas son esenciales cuando se está interesado en identificar una reducción en el apareamiento mientras se estudian los genes involucrados en la vía de apareamiento17,18. Además, dado que la levadura se utiliza cada vez más en estudios de especiación, es necesario un ensayo de apareamiento conveniente y reproducible, ya que la cuantificación de la eficiencia de apareamiento es una medida de la barrera precigótica.
La eficiencia de apareamiento entre los dos tipos de apareamiento de levadura se ha cuantificado previamente16,19,20. La mayoría de los métodos utilizados anteriormente son similares en su diseño con algunas variaciones 16,21,22,23,24,25. Algunos de ellos usan cultivos de fase logarítmica temprana, mientras que otros usan cultivos de fase logarítmica media de cepas haploides. Hay variaciones en las proporciones en las que se mezclan los dos tipos de apareamiento. Casi todos los protocolos utilizan una membrana de nitrocelulosa. Las suspensiones de ambos tipos de apareamiento tomadas de cultivos previamente cultivados se mezclan y se filtran sobre una membrana de nitrocelulosa colocada en una placa YPD. En una de las variaciones del protocolo, la suspensión haploide se parcha directamente en una placa YPD21. En experimentos relacionados con los genes involucrados en la producción de feromonas de los dos tipos de apareamiento, las feromonas se agregan externamente mientras se hacen las suspensiones de los dos tipos de apareamiento24.
Después de la incubación durante unas horas (típicamente alrededor de 5 h) después de mezclar los haploides, las células se lavan de la membrana, se diluyen y se colocan en medios selectivos. En uno de los métodos anteriores reportados en 1973, la eficiencia de la formación o apareamiento de cigotos se calculó contando el número de células brotadas, células sin brotes y pares de apareamiento bajo un microscopio utilizando un hemocitómetro26. Sin embargo, la mayoría de los métodos reportados más tarde usan marcadores auxotrofos para distinguir haploides y diploides. La eficiencia de apareamiento se calcula como el porcentaje de células diploides en relación con el número de células diploides y haploides en el conjunto celular 16,21,23.
Sin embargo, a pesar de una serie de informes que utilizan la levadura como organismo modelo para estudiar la especiación, no existe un protocolo estandarizado reportado en la literatura hasta ahora para calcular la eficiencia del apareamiento. Las células en la fase logarítmica pueden no ser ideales para la cuantificación de la eficiencia de apareamiento. Durante el apareamiento, el ciclo celular de los dos haploides se detiene, y por lo tanto, las células durante el apareamiento no se están dividiendo9. Como también se sabe que el ciclo celular se detiene de manera similar en las células en la fase estacionaria27, el uso de tales células puede hacer que el protocolo sea más reproducible. Las células de fase estacionaria se pueden mezclar y colocar en placas YPD (es decir, un ambiente nutricionalmente rico) para el apareamiento. Los procedimientos convencionales también requieren una membrana de nitrocelulosa y el lavado de las células, lo que hace que el proceso sea engorroso y susceptible de errores de manejo. Además, los protocolos utilizados hasta la fecha cuantifican la eficiencia de apareamiento en términos de un haploide. Sin embargo, cuando se mide el aislamiento reproductivo, la eficiencia de apareamiento se cuantifica para una combinación particular de haploides en lugar de un solo haploide.
Para abordar estos problemas, aquí, informamos un método robusto para la cuantificación de la eficiencia de apareamiento en levadura que es altamente reproducible y fácil de usar. Además, este método y las cepas de levadura empleadas aquí también se pueden utilizar en estudios que examinan el efecto del flujo génico en la evolución de las barreras de apareamiento.
En este estudio se utilizaron dos cepas diferentes de S. cerevisiae. Una de las cepas se deriva del fondo SK1; esto se modificó en nuestro laboratorio agregando los marcadores auxotrofos cerca del locus MAT. Los genotipos resultantes de los haploides se proporcionan en la Tabla 128,29,30. En la cepa SK1, el haploide tenía el gen TRP1 insertado cerca del locus MAT, y el haploide α tenía el gen LEU2 insertado cerca del locus MAT. En la cepa ScAM, los genes TRP1 y URA3 se insertaron en los haploides a y α, respectivamente. La localización de la inserción fue en la región ARS del cromosoma III (Chr III: 197378..197609). Para el protocolo reportado aquí, los marcadores auxotrofos en cualquier parte del genoma serían suficientes. Sin embargo, tener los marcadores auxotrofos cerca del locus MAT significa que estas cepas también pueden ser utilizadas para estudios que examinan el efecto del flujo génico sobre la especiación31,32. Los marcadores se agregaron cerca del locus MAT para evitar la reorganización de los marcadores debido a la recombinación. Por lo tanto, este protocolo se puede utilizar para cuantificar la eficiencia de apareamiento en estudios que involucran especiación y también para identificar la alteración de la eficiencia de apareamiento cuando se estudian las proteínas involucradas en la vía de apareamiento.
Protocolo
NOTA: El protocolo implica ampliamente los siguientes pasos: (1) parchear los haploides en las rejillas de eficiencia de apareamiento en una placa YPD, (2) mezclar los haploides en números iguales después de 24 h de incubación y dar a los haploides mixtos unas horas para aparearse (7 h en este estudio), (3) colocar las células mixtas en YPD para aislar colonias individuales después de 7 h a 30 ° C, y finalmente, (4) determinar el número de diploides formados utilizando los marcadores auxotróficos. Estos pasos se describen en detalle a continuación (consulte también la figura 2).
1. Parcheo de haploides en las rejillas de eficiencia de acoplamiento
- Revivir los haploides a y α de las existencias de congelación rayando en una placa de agar YPD (2% de agar, 2% de dextrosa, 1% de peptona, 0,5% de extracto de levadura), y dejar que crezcan durante 48 h a 30 ° C para obtener colonias individuales aisladas.
- Inocular colonias individuales de la placa YPD en 5 ml de medio YPD (2% dextrosa, 1% peptona, 0,5% extracto de levadura) e incubar a 30 °C durante 48 h con agitación de 250 rpm. Las células están en la fase estacionaria de crecimiento después de este período de incubación.
- Dibuje una cuadrícula de eficiencia de acoplamiento en una placa YPD nueva. Dibuje la cuadrícula como un rectángulo de 1 cm x 1,5 cm dividido en tres cajas, de modo que cada una tenga una dimensión de 1 cm x 0,5 cm, como se muestra en la figura 2A.
- Parche 5 μL del cultivo YPD de los dos tipos de apareamiento en los rectángulos más a la izquierda y más a la derecha (Figura 2B). Este volumen corresponde a aproximadamente 5 x 105 células haploides dispuestas en cada sección. Incubar las placas durante 24 h a 30 °C.
NOTA: El propósito de la cuadrícula es hacer que las medidas experimentales sean precisas (como el número de celdas en el experimento). El tamaño de la cuadrícula es lo suficientemente pequeño como para que sea experimentalmente tratable, pero lo suficientemente grande como para que pueda manipularse fácilmente (como levantar células de una rejilla) y no susceptible a la deriva o eventos casuales.
2. Mezcla de haploides y apareamiento
NOTA: Después de 24 h (Figura 2C), un número igual de celdas de los dos tipos haploides se raspan de las dos rejillas, se mezclan y se colocan en el rectángulo central (Figura 2D).
- Para mezclar un número igual de células, retire aproximadamente 1/3 del parche que se colocó en las cajas exteriores con un palillo de dientes estéril y vuelva a suspender en 20 μL de agua en un vial estéril de 1,5 ml para cada uno de los haploides.
- Diluir 5 μL de esta suspensión en 2 mL de agua. Mida el OD de esta suspensión diluida utilizando un espectrofotómetro a 600 nm. Mezcle un número igual de células de las dos cepas, según el valor de DO y el número de células/ml en 1 OD para esa cepa en particular. Calcule el volumen requerido para mezclar y aspire a partir de los 15 μL restantes de la suspensión haploide individual.
NOTA: El número de celdas parcheadas en el rectángulo central es tal que las celdas forman una monocapa. Considerando que la celda de levadura es una esfera con un radio de 2,58 μm33, una caja rectangular de 1 cm x 0,5 cm necesitaría aproximadamente 1,7 x 106 células para formar una monocapa. Se debe tener cuidado para asegurarse de que no haya contacto físico entre las células parcheadas para el apareamiento y las dos rejillas de células haploides. Dado que se deben mezclar números iguales de cada tipo de células haploides, se agregan 8.5 x 105 células de cada cepa. El número de células se calcula con base en las mediciones de OD, considerando que 1 OD a 600 nm es aproximadamente equivalente a 1 x 107 celdas34. Por ejemplo, si el OD600 de la suspensión haploide es 0,17 y el del haploide α es 0,11, se puede calcular el número de células en 5 μL de cada suspensión haploide. Para garantizar 8,5 x 105 células de cada tipo haploide, se mezclan 1,25 μL de las suspensiones haploide y 1,93 μL de las suspensiones haploides α. - Agregue los volúmenes requeridos de ambos haploides en un vial fresco y estéril de 1,5 ml, y mezcle bien con una pipeta. El volumen final de esta suspensión es generalmente de alrededor de 6-8 μL. Parche esta suspensión en la rejilla central. Incubar la placa a 30 °C durante 7 h, dejando que los haploides tengan tiempo suficiente para aparearse (Figura 2E).
3. Recubrimiento de células mixtas en agar YPD
- Después del período de incubación de 7 h, raspe las células del rectángulo central con un palillo de dientes o una punta de pipeta, y diluya en 2 ml de agua estéril. Luego, extienda la suspensión celular en agar YPD para obtener colonias individuales. Para determinar el factor de dilución necesario para obtener colonias individuales, mida el OD del primer tubo en el que se agregan las células raspadas. Es necesario determinar factores de dilución específicos para cada tipo de célula/cepa utilizada.
NOTA: Por ejemplo, un OD600 de 0,15 corresponde a 3 x 106 celdas en una suspensión de 2 ml (considerando 1 DO = 1 x 107 células/ml). Para obtener unos pocos cientos de colonias en la placa YPD, la suspensión celular se diluye en serie a 1:20 dos veces, y luego se utilizan 100 μL de la dilución final para esparcir. - Después del emplatado, incubar las placas YPD a 30 °C durante 36-48 h hasta que haya colonias individuales. Asegúrese de que se obtengan unos pocos cientos de colonias individuales de cada experimento de apareamiento para la detección a fin de garantizar que se pueda detectar la significación estadística en los datos (Figura 2F).
4. Cribado de diploides mediante marcadores auxotrofos
- Determinar qué fracción de las colonias obtenidas son diploides. Para identificar las colonias diploides en la placa, transfiera las colonias individuales rayándolas individualmente en una placa de doble gota (2% de glucosa, 0,66% de base nitrogenada, 0,05% de mezcla de aminoácidos de doble caída y 2% de agar) que carece de los aminoácidos para los que las cepas son auxotróficas, como se muestra en la Figura 2G. Incubar las placas a 30 °C durante 48 h.
NOTA: Las colonias también se pueden transferir a la placa de doble caída utilizando una réplica. En este estudio, se utilizó triptófano y leucina (trp− leu−) medio de abandono para cuantificar la eficiencia de apareamiento de las cepas SK1AM, y triptófano y uracilo (trp− ura−) medio de abandono para las cepas ScAM. Solo las colonias diploides crecen en la placa de doble abandono, ya que tienen ambos marcadores auxotróficos: genes TRP1 y LEU2 en las cepas SK1AM y genes TRP1 y URA3 en las cepas ScAM. - Además, rayar o replicar las placas de las colonias en medios de caída única (trp− o leu− o ura−) para cuantificar la frecuencia de cada uno de los dos tipos de haploides en la población.
- Calcule la eficiencia de apareamiento, η, de la siguiente manera:
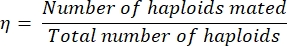 Eq (1)
Eq (1)
donde el número de haploides acoplados es simplemente igual al doble del número de diploides identificados en la placa de doble abandono (ya que cada diploide resultó del apareamiento de dos haploides). El número total de haploides es igual a la suma del número de haploides rayados más el doble del número de diploides rayados.
NOTA: Por ejemplo, si solo 60 colonias crecen después de rayar / replicar el recubrimiento de 100 colonias en un medio de doble abandono, la eficiencia de apareamiento se puede cuantificar como 75% (ya que 60 x 2 haploides se acoplaron para formar los 60 diploides, y 40 haploides no se aparearon).
Resultados
Cuantificación de la eficiencia de apareamiento de los dos tipos de apareamiento
El protocolo descrito aquí se utilizó para cuantificar la eficiencia de apareamiento entre dos cepas de levadura, entre SK1AM a y SK1AM α y entre ScAMa y ScAMα (Figura 3A). En estos experimentos, el apareamiento entre los dos haploides se repitió al menos 12 veces. En cada una de las repeticiones del experimento, al men...
Discusión
La cuantificación de la eficiencia de apareamiento en S. cerevisiae es esencial para llevar a cabo estudios relacionados con los genes implicados en las vías de apareamiento o estudiar la influencia del medio externo en el comportamiento de apareamiento. En las últimas dos décadas, S. cerevisiae también se ha convertido en un modelo popular para abordar cuestiones relacionadas con la especiación 14,36,37,38.
Divulgaciones
Los autores declaran que no tienen intereses contrapuestos en este trabajo. Los autores están felices de compartir las cepas derivadas de SK1 para todo uso sin fines de lucro.
Agradecimientos
Este trabajo fue financiado por una subvención DBT/Wellcome Trust (India Alliance) (IA/S/19/2/504632) a S.S. P.N. es un becario de investigación apoyado por una subvención DBT/Wellcome Trust (India Alliance) (IA/S/19/2/504632). A.M. cuenta con el apoyo del Consejo de Investigación Científica e Industrial (CSIR), Gobierno de la India, como investigador principal (09/087(0873)/2017-EMR-I). Los autores agradecen a Paike Jayadeva Bhat por las discusiones.
Materiales
| Name | Company | Catalog Number | Comments |
| Adenine | Sigma Life Science | A8626 | |
| Agar Powder regular grade for bacteriology | SRL | 19661 (0140186) | |
| Ammonium Sulphate, Hi-AR | HiMedia | GRM1273 | |
| D-(+)-glucose | Sigma Life Science | G8270 | |
| Glass Petri plates | HiMedia | PW008 | 90 mm x 15 mm dimension |
| L-Arginine | Sigma Life Science | A8094 | |
| L-Aspartic acid | Sigma Life Science | A7219 | |
| L-Histidine monochloride monohydrate | Sigma Life Science | H5659 | |
| L-Isoleucine | Sigma Aldrich | I2752 | |
| L-Leucine | Sigma Life Science | L8912 | |
| L-Lysine | Aldrich | 62840 | |
| L-Methionine | Sigma Life Science | M5308 | |
| L-Phenylalanine | Sigma Life Science | P5482 | |
| L-Threonine | Sigma Aldrich | T8625 | |
| L-Tyrosine | Sigma Life Science | T8566 | |
| L-Valine | Sigma Life Science | V0513 | |
| Mating efficiency grid | 1 cm x 1.5 cm rectangular grid drawn on the Petri plate | ||
| Microcentrifuge tubes | Tarsons | 500010 | |
| Peptone | HiMedia | RM001 | |
| Uracil | Sigma Life Science | U0750 | |
| Yeast Extract Powder | HiMedia | RM027 | |
| Yeast Nitrogen Base w/o Amino acids and Ammonium Sulphate | BD Difco | 233520 |
Referencias
- Neiman, A. M. Sporulation in the budding yeast Saccharomyces cerevisiae. Genetics. 189 (3), 737-765 (2011).
- Duina, A. A., Miller, M. E., Keeney, J. B. Budding yeast for budding geneticists: A primer on the Saccharomyces cerevisiae model system. Genetics. 197 (1), 33-48 (2014).
- Herskowitz, I. Life cycle of the budding yeast Saccharomyces cerevisiae. Microbiological Reviews. 52 (4), 536-553 (1988).
- Erdman, S., Lin, L., Malczynski, M., Snyder, M. Pheromone-regulated genes required for yeast mating differentiation. Journal of Cell Biology. 140 (3), 461-483 (1998).
- Merlini, L., Dudin, O., Martin, S. G. Mate and fuse: How yeast cells do it. Open Biology. 3 (3), 130008 (2013).
- Gustin, M. C., Albertyn, J., Alexander, M., Davenport, K. MAP kinase pathways in the yeast Saccharomyces cerevisiae. Microbiology and Molecular Biology Reviews. 62 (4), 1264-1300 (1998).
- Bardwell, L. A walk-through of the yeast mating pheromone response pathway. Peptides. 26 (2), 339-350 (2005).
- Reid, B. J., Hartwell, L. H. Regulation of mating in the cell cycle of Saccharomyces cerevisiae. Journal of Cell Biology. 75, 355-365 (1977).
- Williams, T. C., Peng, B., Vickers, C. E., Nielsen, L. K. The Saccharomyces cerevisiae pheromone-response is a metabolically active stationary phase for bio-production. Metabolic Engineering Communications. 3, 142-152 (2016).
- Bagnat, M., Simons, K. Cell surface polarization during yeast mating. Proceedings of the National Academy of Sciences of the United States of America. 99 (22), 14183-14188 (2002).
- Trueheart, J., Boeke, J. D., Fink, G. R. Two genes required for cell fusion during yeast conjugation: Evidence for a pheromone-induced surface protein. Molecular and Cellular Biology. 7 (7), 2316-2328 (1987).
- Sniegowski, P. D., Dombrowski, P. G., Fingerman, E. Saccharomyces cerevisiae and Saccharomyces paradoxus coexist in a natural woodland site in North America and display different levels of reproductive isolation from European conspecifics. FEMS Yeast Research. 1 (4), 299-306 (2002).
- Replansky, T., Koufopanou, V., Greig, D., Bell, G. Saccharomyces sensu stricto as a model system for evolution and ecology. Trends in Ecology and Evolution. 23 (9), 494-501 (2008).
- Greig, D. Reproductive isolation in Saccharomyces. Heredity. 102 (1), 39-44 (2009).
- Mayr, E. . Systematics and the Origin of Species, from the Viewpoint of a Zoologist. , (1999).
- Sprague, G. F. Assay of yeast mating reaction. Methods in Enzymology. 194, 77-93 (1991).
- McCaffrey, G., Clay, F. J., Kelsay, K., Sprague, G. F. Identification and regulation of a gene required for cell fusion during mating of the yeast Saccharomyces cerevisiae. Molecular and Cellular Biology. 7 (8), 2680-2690 (1987).
- Valtz, N., Peter, M., Herskowitz, I. FAR1 is required for oriented polarization of yeast cells in response to mating pheromones. Journal of Cell Biology. 131 (4), 863-873 (1995).
- Maclean, C. J., Greig, D. Prezygotic reproductive isolation between Saccharomyces cerevisiae and Saccharomyces paradoxus. BMC Evolutionary Biology. 8, 1 (2008).
- Murphy, H. A., Kuehne, H. A., Francis, C. A., Sniegowski, P. D. Mate choice assays and mating propensity differences in natural yeast populations. Biology Letters. 2 (4), 553-556 (2006).
- Leu, J. Y., Murray, A. W. Experimental evolution of mating discrimination in budding yeast. Current Biology. 16 (3), 280-286 (2006).
- Kim, J., Hirsch, J. P. A nucleolar protein that affects mating efficiency in Saccharomyces cerevisiae by altering the morphological response to pheromone. Genetics. 149 (2), 795-805 (1998).
- Jin, M., et al. Yeast dynamically modify their environment to achieve better mating efficiency. Science Signaling. 4 (186), (2011).
- Rogers, D. W., Denton, J. A., McConnell, E., Greig, D. Experimental evolution of species recognition. Current Biology. 25 (13), 1753-1758 (2015).
- McClure, A. W., Jacobs, K. C., Zyla, T. R., Lew, D. J. Mating in wild yeast: Delayed interest in sex after spore germination. Molecular Biology of the Cell. 29 (26), 3119-3127 (2018).
- Sena, E. P., Radin, D. N., Fogel, S. Synchronous mating in yeast. Proceedings of the National Academy of Sciences of the United States of America. 70 (5), 1373-1377 (1973).
- Werner-Washburne, M., Braun, E., Johnston, G. C., Singer, R. A. Stationary phase in the yeast Saccharomyces cerevisiae. Microbiology Reviews. 57 (2), 383-401 (1993).
- Johnston, S. A., Hopper, J. E. Isolation of the yeast regulatory gene GAL4 and analysis of its dosage effects on the galactose/melibiose regulon. Proceedings of the National Academy of Sciences of the United States of America. 79 (22), 6971-6975 (1982).
- Mahilkar, A. . Study of metabolic specialization leading to speciation, using yeast as a model system. , (2021).
- Blank, T. E., Woods, M. P., Lebo, C. M., Xin, P., Hopper, J. E. Novel Gal3 proteins showing altered Gal80p binding cause constitutive transcription of Gal4p-activated genes in Saccharomyces cerevisiae. Molecular and Cellular Biology. 17 (5), 2566-2575 (1997).
- Rice, W. R., Hostert, E. E. Laboratory experiments on speciation: What have we learned in 40 years. Evolution. 47 (6), 1637-1653 (1993).
- White, N. J., Snook, R. R., Eyres, I. The past and future of experimental speciation. Trends in Ecology and Evolution. 35 (1), 10-21 (2020).
- Milo, R., Jorgensen, P., Moran, U., Weber, G., Springer, M. BioNumbers--The database of key numbers in molecular and cell biology. Nucleic Acids Research. 38, 750-753 (2010).
- Domitrovic, T., et al. Structural and functional study of YER067W, a new protein involved in yeast metabolism control and drug resistance. PLoS One. 5 (6), 11163 (2010).
- Lindegren, C. C., Spiegelman, S., Lindegren, G. Mendelian inheritance of adaptive enzymes. Proceedings of the National Academy of Sciences of the United States of America. 30 (11), 346-352 (1944).
- Dettman, J. R., Sirjusingh, C., Kohn, L. M., Anderson, J. B. Incipient speciation by divergent adaptation and antagonistic epistasis in yeast. Nature. 447 (7144), 585-588 (2007).
- Jhuang, H. Y., Lee, H. Y., Leu, J. Y. Mitochondrial-nuclear co-evolution leads to hybrid incompatibility through pentatricopeptide repeat proteins. EMBO Reports. 18 (1), 87-101 (2017).
- Lee, H. Y., et al. Incompatibility of nuclear and mitochondrial genomes causes hybrid sterility between two yeast species. Cell. 135 (6), 1065-1073 (2008).
- Pronk, J. T. Auxotrophic yeast strains in fundamental and applied research. Applied and Environmental Microbiology. 68 (5), 2095-2100 (2002).
- Madhani, H. D. . From a to α: Yeast as a Model for Cellular Differentiation. , (2007).
- Huxley, C., Green, E. D., Dunham, I. Rapid assessment of S. cerevisiae mating type by PCR. Trends in Genetics. 6 (8), 236 (1990).
- Rang, C., Galen, J. E., Kaper, J. B., Chao, L. Fitness cost of the green fluorescent protein in gastrointestinal bacteria. Canadian Journal of Microbiology. 49 (9), 531-537 (2003).
- Tusso, S., Nieuwenhuis, B. P. S., Weissensteiner, B., Immler, S., Wolf, J. B. W. Experimental evolution of adaptive divergence under varying degrees of gene flow. Nature Ecology and Evolution. 5 (3), 338-349 (2021).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados