Method Article
获得 3C 的 A:本科生的染色体构象捕获
摘要
在这里,我们详细介绍了染色体构象捕获(3C)技术的改编,重点是本科生的参与和学习。
摘要
染色体构象捕获(3C)是一种强大的工具,催生了一系列类似的技术(例如,Hi-C,4C和5C,这里称为3C技术),这些技术提供了染色质三维组织的详细信息。3C技术已被广泛用于研究,从监测癌细胞染色质组织的变化到识别与基因启动子的增强子接触。虽然许多使用这些技术的研究都在通过复杂的样本类型(即单细胞分析)提出全基因组的大问题,但经常丢失的是,3C技术基于适用于广泛研究的基本分子生物学方法。通过解决紧密聚焦的染色质组织问题,这种尖端技术可用于增强本科生研究和教学实验室体验。本文提出了一个3C协议,并为本科生研究和教学经验的主要本科机构的实施提供了调整和重点。
引言
生物体的基因组不仅包含功能所需的所有基因,还包含有关如何以及何时使用它们的所有指令。这使得调节对基因组的访问成为细胞最重要的功能之一。有许多机制可以控制基因功能;然而,在其基础水平上,基因调控归结为调节转录因子(反式因子)与其特定DNA序列(顺式调节序列)结合的能力。这不是与生俱来的能力;相反,它由细胞核中基因组的组织/结构控制,该组织/结构控制顺式调节序列对反式因子1,2,3的可用性/暴露。如果反式因子找不到它们的顺式调节序列,那么反式因子就不能执行它们的调节任务。这使得理解基因组如何在细胞核中组织成为一个重要的研究来源。
人们普遍认为,在间期,细胞核中的真核染色体占据了锚定在核层和核基质上自己的域(图1),从而使染色体更像一片披萨,而不是一盘意大利面上的面条。染色体部分由蛋白质-DNA相互作用(染色质)凝聚,这些相互作用扭曲和循环染色体的部分。通过电子显微镜,三维DNA荧光原位杂交(FISH)和DNA标记技术(即荧光和人工DNA甲基化),已经发现染色质的非活性结构域沿着核外围紧密包装4,5,6,而在细胞核内部发现了部分活性的,不太浓缩的染色质7,8,9,10.这些实验提供了染色体动力学的广角视图,但对捕捉在DNase11,12和核小体13,14,15研究中观察到的基因促进子周围局部发生的变化几乎没有作用。
解锁更高分辨率染色质动力学的关键是3D染色体定位技术3C的制定。3C技术本身包括四个主要步骤:染色质交联,限制性内切酶消化染色质,染色质连接和DNA纯化(图2)。然后,可以表征该过程产生的新人工DNA片段,以揭示线性远距离DNA片段之间的密切物理联系16。3C技术成为创建多种衍生技术的基础,这些技术利用3C的初始步骤提出更广泛的全基因组问题(例如,Hi-C,4C,ChIP-C)。这一系列3C技术已经确定染色体被组织成多个离散单元,称为拓扑相关结构域(TAD)。TAD在基因组中编码,由染色质环定义,两侧是无环边界16,17,18,19。TAD边界由两个进化上保守且普遍存在的因子维持,包括CCCT结合因子(CTCF)和内聚力,它们阻止了单独TAD中的环相互作用16,20。环路由跨因子与其调节序列的相互作用以及CTCF和内聚力介导21。
尽管许多使用3C技术的研究提出了广泛的全基因组问题并采用了复杂的样本收集技术,但3C技术的配方是基于基本的分子生物学技术。这使得3C在本科研究和教学实验室中的部署都很有趣。3C技术可用于较小的焦点问题,并且根据所问问题的焦点和方向,可以灵活地扩大或缩小(单基因22,染色体16和/或基因组18)。该技术也已应用于广泛的模型系统7,16,19,23,并已被证明在其使用中具有多功能性。这使得3C成为本科生的绝佳技术,因为学生可以获得常见分子生物学技术的经验,同时也可以获得回答定向问题的宝贵经验。
这里介绍的是基于先前发布的方案24,25,26,27的3C文库制备的适应方案。该协议已针对大约 1 × 107 个细胞进行了优化,尽管它生成的 3C 文库只有 1 × 105 个细胞。该协议已被证明是通用的,并已用于从斑马鱼胚胎、斑马鱼细胞系和年轻成人 (YA) 秀丽隐杆线虫(蛔虫)生成 3C 文库。该方案还应适用于哺乳动物细胞系,并进一步适应酵母。
这些改编的目的是让本科生更容易接触到3C。已经注意使用类似于本科教学实验室中可以完成的技术。3C技术为本科生提供了许多学习基本分子生物学技术的机会,这将有利于他们在工作台上,课堂上和毕业后的努力中的发展。
研究方案
1. 底漆设计
注:3C引物设计工具可在线获取28.或者,学生可以设计定制的入门书(见下文)。
- 识别引物位置
- 打开UCSC基因组浏览器(http://genome.ucsc.edu/),选择研究的生物体,然后使用3C搜索要评估的基因组区域。
- 在下一个窗口中,通过单击Restr酶激活浏览器下方的“映射和测序”选项卡中的酶跟踪。
- 输入要使用的 限制性内切酶 ,并将 显示模式 设置为 包装。点击 提交。
- 在“ 变体”和“重复”中,确保 “重复掩码器 ”设置为 “密集”。
- 使用限制位点作为指南,识别不在屏蔽重复区域(黑条)内的感兴趣位点。通过单击位置( 最上面的轨道) 并拖动到所需的长度(~600 bp),突出显示限制性位点周围的上游和下游序列两侧的 300 个碱基对 (bp)。
- 松开鼠标,将出现一个弹出框。记下基因组位置,单击 放大,然后让浏览器重新调整到选择。
- 将鼠标悬停在浏览器功能区上方的 “视图 ”选项卡上,然后在下拉菜单中选择“ DNA”。
- 保留默认选项,注意屏蔽序列的名称(有一个选项可以使它们成为 N)。点击获取 DNA。
- 然后,突出显示的序列将显示在窗口中。将此序列复制并粘贴到Primer3(https://primer3.ut.ee/)中。
- 使用Primer3默认设置,通过单击 “选择引物”生成引物。
- 在 结果中,记下限制性内切酶的位置,并选择产生 200-500 bp PCR产物 的引物,限制 性位点 位于 中间。
- 按照这些步骤,从感兴趣的位点设计1千碱基(kb)、2 kb、5 kb、10 kb和20 kb的引物测试。
- 要设计输入对照引物,请对距离缺少限制性位点的感兴趣位点 1-2 kB 的位点重复这些步骤,这意味着它永远不会被切割。
- 功能引物验证
- 为了验证引物功能,请使用滴定的引物浓度和纯化的基因组DNA设置PCR反应。通过定量或半定量手段,确定引物是否产生预期的产物。
- 重新设计未通过验证的引物。
2. 第一天
注意:在染色质交联后和细胞核收集后,该方案可以暂停(在-20°C冷冻)。本科生的平均步骤需要 5-6 小时。
- 年轻成人(YA) 秀丽隐杆线虫 核集合(改编自Han等人29)
- 染色质交联
- 在 50 mL 锥形管中的 30 mL M9 培养基中收集 5,000 个 YA 蠕虫,并在室温 (RT) 下以 400 × g 离心 2 分钟。
- 用 M3 将蠕虫颗粒再清洗 9 倍以去除细菌。
- 除去上清液,将蠕虫沉淀重悬于含有2.7mL 37%甲醛(2%最终)的47.3mL M9中,并在室温下搅拌(摇动或章动)孵育30分钟。
注意:小心处理甲醛,并使用适当的个人防护设备(PPE 实验室外套、适当的手套和护目镜)在通风橱下工作。甲醛是一种刺激物,会影响眼睛、鼻子、喉咙和肺部。 - 在室温下以400× g 旋转蠕虫2分钟。
- 除去上清液,并将蠕虫沉淀重悬于50mL的1M甘氨酸中。在室温下以400× g 旋转蠕虫2分钟。
- 细胞核收集
- 将蠕虫沉淀重悬于6mL冷冻NP缓冲液中(pH 7.5下的50mM HEPES,40mM NaCl,90mM KCl,2mM EDTA,0.5mM EGTA,0.1%吐温20,0.2mM DTT,0.5mM亚精胺,0.25mM精胺,1x完全蛋白酶抑制剂)。
- 将蠕虫悬浮液转移到 7 mL 松散的冰上弹跳中。将样品弹跳 15 倍,并在冰上保持 5 分钟。
- 将蠕虫悬浮液转移到 7 mL 紧密贴合的冰上 Dounce 中。将样品弹跳 20 倍,并在冰上保持 5 分钟。
- 将蠕虫悬浮液转移到干净的 15 mL 锥形管中,并加入 NP 缓冲液至总共 10 mL(约 4 mL)。
- 将蜗杆悬浮液在高设置下涡旋30秒。将样品在冰上孵育5分钟。
- 重复上一步。
- 在4°C下以100× g 旋转蠕虫悬浮液5分钟。
- 将上清液转移到新鲜的 15 mL 锥形管中。
- 检查样品是否有蠕虫碎片。用光学显微镜观察 10 μL 样品。如果存在蠕虫碎片,则在4°C下以2,000× g 旋转样品5分钟,将沉淀重悬于新鲜的10mLNP缓冲液中,并在4°C下以100× g 再次旋转样品5分钟。 丢弃沉淀,检查上清液中的蠕虫碎片,然后重复直到样品没有蠕虫碎片。
注意:或者,也可以通过40μm细胞过滤器(6x)和20μm细胞过滤器(6x)过滤样品来去除蠕虫碎片。 - 如果样品中没有蠕虫碎片,则取 5 μL 样品,加入 5 μL 甲基绿派洛宁(细胞核为蓝色),并用血细胞计数器计数细胞核。
- 在4°C下以2,000× g 旋转样品5分钟
- 取出上清液,将样品放在冰上,然后继续;或者,快速冷冻细胞核,并储存在-80°C。
- 染色质交联
- 染色质消化
- 将细胞核重悬于 450 μL 清水中。将细胞核转移到干净的 1.5 mL 微量离心管中。
- 向交联样品中加入 60 μL 10x DpnII 限制性内切酶缓冲液,并充分混合。
注意:可以使用仍可切割交联DNA的其他限制性内切酶。 - 加入15μL10%十二烷基硫酸钠(SDS)以透化细胞核,并在37°C下搅拌(摇摆或章动)孵育1小时。
注意:SDS是一种刺激物,如果通过皮肤摄入或吸收,则有毒。小心处理,并使用适当的个人防护用品(实验室外套、适当的手套和护目镜)。 - 通过加入 75 μL 20% Triton X-100 并在 37 °C 下搅拌孵育 1 小时来淬灭 SDS。
- 从样品中取出 10 μL 作为未消化的对照。储存在4°C。
- 加入400U的DpnII,并在37°C下搅拌孵育过夜。
3. 第二天
注意:平均而言,本科生需要 5 小时才能完成这些步骤。
- 染色质消化
- 再加入200U的DpnII,并将样品在37°C下搅拌孵育4小时,以确保交联样品的完全消化。
- 从样品中取出 10 μL 等分试样作为消化对照。
- 通过在65°C(或根据制造商的说明)孵育样品20分钟来热灭活限制性内切酶。继续执行步骤 2.1.3。
- 如果酶不能通过加热灭活,则加入80μL的10%SDS,并将样品在65°C孵育30分钟。 然后,加入 375 μL 20% Triton X-100,并通过旋转混合。将样品在37°C孵育1小时,并继续步骤2.2。
- 染色质连接
- 将样品转移到干净的 50 mL 锥形管中,用分子级 H2O 将样品体积调节至 5.7 mL,然后旋转混合。
- 加入 700 μL 10x T4 连接酶缓冲液,旋转混合。
- 加入 60 U 的 T4 DNA 连接酶,并通过旋转混合。
- 在16°C孵育过夜。
4. 第三天
注意:平均而言,本科生需要 15-30 分钟才能完成这些步骤。孵育过夜后,样品可以冷冻。
- 蛋白质消化和反向交联
- 向 3C 样品中加入 30 μL 蛋白酶 K (10 mg/mL),并在 65 °C 下搅拌孵育过夜。向未消化和消化对照中加入 5 μL 蛋白酶 K (10 mg/mL),并在 65 °C 下搅拌孵育过夜。
5. 第四天
注意:平均而言,本科生需要 4-5 小时才能完成这些步骤。
- 3C文库的纯化
- 向 3C 样品中加入 30 μL RNase A (10 mg/mL),然后旋转混合。将样品在37°C孵育45分钟。
- 向样品中加入 7 mL 苯酚-氯仿,摇匀混合。
注意:苯酚-氯仿是一种皮肤和眼睛刺激物,如果不治疗接触,可能会导致灼伤。苯酚 - 氯仿应在通风橱中使用,并带有适当的眼睛保护和手套(丁腈)。 - 在室温下以 3,270 × g 离心样品 15 分钟。 收集水相,并转移到干净的 50 mL 锥形管中。加入等体积的氯仿,摇匀混合样品。
- 在室温下以 3,270 × g 离心样品 15 分钟。 收集水相,并转移到干净的 50 mL 锥形管中。加入 7.5 mL 分子级 H2O、35 mL 100% 乙醇和(可选)7 μL 糖原 (1 mg/mL)。通过振荡混合,并在-80°C下孵育直至样品冷冻。
注意:样品可能需要 1 小时或更长时间才能冻结。协议可以在此处暂停。- 当3C样品冷冻时,纯化对照样品。向对照样品中,使用分子级水将体积调节至 500 μL,并通过轻弹试管加入 2 μL RNase A (10 mg/mL) 混合物。
- 短暂旋转以收集管底部的样品。将样品在37°C孵育45分钟。
- 向对照样品中加入1 mL苯酚-氯仿,并通过摇动试管进行混合。在室温下以 3,270 × g 离心样品 15 分钟。 收集对照的水相,并放入干净的 1.5 mL 微量离心管中。
- 加入等体积的氯仿,摇匀混合样品。在室温下以 3,270 × g 离心样品 15 分钟。 收集对照的水相,并放入干净的 1.5 mL 微量离心管中。
- 加入 1 mL 100% 乙醇和 2 μL 糖原 (1 mg/mL)。摇动混合,并在-80°C下孵育30分钟。
- 将样品在4°C下以3,270 ×g 离心15分钟。 除去上清液,加入 750 μL 冷冻的 70% 乙醇。
- 将样品在4°C下以3,270× g 离心10分钟。 除去上清液,风干样品。将沉淀重悬于50μL分子级H2O.冷冻中,或继续步骤6。
- 将样品在4°C下以3,270× g 离心60分钟。 除去上清液,加入10mL冷冻的70%乙醇。通过摇动样品进行混合来破坏和分解DNA沉淀。
- 将样品在4°C下以3,270× g 离心30分钟。 除去上清液,让样品在室温下部分风干。 通过上下移液将沉淀重悬于 150 μL 10 mM Tris-HCl (pH 7.5) 中。这就是“3C图书馆”。
注意:冷冻样品,或继续分析。
6. 第五天
注意:平均而言,本科生需要 1-2 小时才能完成这些步骤。
- 使用对照引物的标准曲线确定样品质量
- 定量 3C 样品以及所有对照的 DNA 浓度,并将样品调整为 30 μg/μL(如果浓度允许)。连续将稀释样品稀释四倍,每个样品稀释五次(1x,0.5x,0.25x,0.125x,0.0625x)。
注意:如果无法轻松完成定量,请如上所述连续稀释样品,然后继续。 - 使用对照引物为每个稀释样品的 3C、基因组对照和消化对照设置 PCR 反应:1 μL DNA、10 μL 5x 反应缓冲液、1 μL 10 mM dNTP、1 μL 10 mM 对照引物(正向和反向混合)、1 μL Taq 聚合酶和 36 μL 水。
- 按照特定于PCR机的软件的说明,使用循环条件设置标准曲线PCR程序,如下所示:98°C下30秒;98°C下30次循环5秒,60°C下5秒,72°C下10秒;在72°C下1分钟;4°C保持。
- 使用该软件生成样品的标准曲线。软件生成曲线后,记下PCR效率和R2 值。
- 定量 3C 样品以及所有对照的 DNA 浓度,并将样品调整为 30 μg/μL(如果浓度允许)。连续将稀释样品稀释四倍,每个样品稀释五次(1x,0.5x,0.25x,0.125x,0.0625x)。
- 测定脱氧核糖核酸浓度
- 要确定3C样品的DNA浓度,请将样品与已知浓度的基因组DNA样品进行比较。将样品稀释至 30 ng/μL,并分两步将基因组对照从 10 ng/μL 连续稀释至 0.01 ng/μL,以创建标准曲线。
注意:如果无法轻松完成定量,请按1:10稀释样品,然后继续。 - 使用对照引物为每个稀释样品设置3C、基因组对照和酶解对照的PCR反应:1 μL DNA、10 μL 5x 反应缓冲液、1 μL 10 mM dNTP、1 μL 10 mM 对照引物(正向和反向混合)、1 μL Taq 聚合酶和 36 μL 水。
- 按照PCR机专用软件的说明,使用循环条件设置标准曲线PCR程序,如下所示:98°C下30秒;98°C下30次循环5秒,60°C下5秒,72°C下10秒;在72°C下1分钟;4°C保持。
- 使用该软件生成样品的标准曲线,在曲线上绘制3C样品。将3C样品丰度的位置与已知浓度的基因组DNA反应产生的标准曲线进行比较,并将3C样品调整为30ng / μL。
- 要确定3C样品的DNA浓度,请将样品与已知浓度的基因组DNA样品进行比较。将样品稀释至 30 ng/μL,并分两步将基因组对照从 10 ng/μL 连续稀释至 0.01 ng/μL,以创建标准曲线。
- 确定染色质相互作用的存在与否
- 使用 30 ng 的 3C 样品、基因组对照样品和消化的对照样品建立 qPCR 反应:1 μL DNA、10 μL 5x 反应缓冲液、1 μL 10 mM dNTP、1 μL 10 mM 引物(正向和反向混合)、1 μL Taq 聚合酶和 36 μL 水。
注意:应使用对照引物,单个基因组位点的测试引物以及用于确定染色质相互作用的测试引物(位点“a”向前和位点“b”反向)的所需组合(图3)来建立反应。 - 按照PCR机软件的说明,设置PCR程序如下:98°C下30秒;98 °C 下 40 次循环 5 秒,60 °C 下 5 秒,72 °C 下 10 秒;在72°C下1分钟;4°C保持。
- 运行PCR后,检查扩增图。确保PCR反应显示指数扩增,每个循环加倍。
注意:不显示指数扩增的反应无法进一步分析。 - 按照PCR机软件的说明,定义qPCR实验的阈值。
- 导出所有样本的 Ct 值。
- 使用公式(1)确定未消解对照(UC)和消解对照(DC)样品的测试引物(TP)和对照引物(CP)Ct值的消解效率。
消化百分比 = 100 -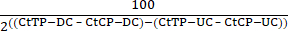 (1)
(1) - 这些值应该在80%-90%消化的范围内。记录这些值(图4A)。
- 使用公式(2)使用未消化对照(UC)和3C (3C)样品的测试引物组合(TPC)和对照引物(CP)Ct值确定相对染色质相互作用。
染色质相互作用 = 100 -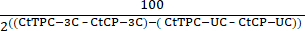 (2)
(2) - 在每个测试引物组合的条形图上绘制每个样品的值。
- 将3C样品的信号与对照样品进行比较,以确定3C样品是否在对照样品上富集了特定的染色质相互作用。显示富集超过对照的3C样品可以被认为是有条件的阳性,需要使用Sanger测序进行验证(图4B)。
注意:在分析图形数据时,重要的是要记住,除非使用“对照模板”(见讨论),否则无法比较反应之间的丰度(即,从一组引物到下一组引物)。
- 使用 30 ng 的 3C 样品、基因组对照样品和消化的对照样品建立 qPCR 反应:1 μL DNA、10 μL 5x 反应缓冲液、1 μL 10 mM dNTP、1 μL 10 mM 引物(正向和反向混合)、1 μL Taq 聚合酶和 36 μL 水。
- 3C产品的识别
- 在1.5%琼脂糖凝胶上运行PCR产物。
- 使用紫外灯箱或凝胶记录系统可视化凝胶以获得正确大小的PCR产物。
注意:构建良好的3C文库将包含多种DNA片段,尽管之前对引物的特异性进行了验证,但来自3C文库的PCR反应有可能包含许多条带(图5A)。这并不妨碍对3C库的分析。 - 切除对应于预期片段大小的产品条带,并按照商业试剂盒的说明或下面概述的自制方案对PCR产物进行凝胶提取。
注意:紫外线是一种已知的致癌物质,应非常小心地限制暴露在紫外线下的时间。应佩戴抗紫外线的护目镜、标本防护罩、手套和实验室外套。- 要构建自制纯化盒,请用针头在 0.5 mL 管底部戳一个孔。
- 将少量棉花从棉球中装入 0.5 mL 试管的底部,填充不超过试管的一半。
- 将 0.5 mL 管放入 1.5 mL 管中;确保较小的管子放在较大管子的边缘上,而不是在管子中。
- 小心地将凝胶片段切成小块,然后将其放入小柱的 0.5 mL 管中。
- 将组件置于-20°C冰箱中5分钟。在室温下以 13,000 × g 旋转组件 3 分钟。
- 将装有提取DNA的1.5 mL管保存在缓冲液中,并处理含有琼脂糖碎片的0.5 mL管。
- 纯化的DNA可以使用3C片段的正向和反向引物发送用于Sanger测序。
- 使用Blat识别染色质接触
- 测序完成后,确定样品是否符合测序报告中所示的质量控制标准。
- 对于通过QC的序列,在测序编辑程序(如另一个质粒编辑器(ApE))中打开.seq和.ab1(迹线)文件,检查.ab1文件是否有明确的碱基调用峰,并编辑.seq文件中错误调用的峰。
- 使用编辑后的 .seq 文件,搜索 DpnII 站点。根据序列结果,这应该在报告序列的一半。
- 要确定该序列是否是预期的靶序列,请突出显示DpnII位点和与3C测试基因组位点的前向引物相对应的上游序列的另外30-50 bp。针对目标物种执行此序列的 Blat 搜索。确保基因组位点与引物的基因组位点匹配。
- 对反向引物序列重复上述步骤,确保返回的基因组位点与反向引物的基因组位点匹配。
结果
该程序将产生一个实验性3C样品和两个对照样品(未消化和消化)。使用这三个样品进行qPCR。根据这些结果,计算消解效率(公式1)并记录(表1)。通过这些计算,确定3C样品在测试的七个基因组位点中具有约88%的消化效率(表1的平均值)。
接下来,使用位点特异性引物(表2)和qPCR的组合测试样品在不同基因组位点之间是否存在长距离染色质接触。利用这些结果,计算产物相对于对照引物组的丰度(公式 2)并绘制图表进行比较(图4)。这些数据表明,10个反应中有8个是长期相互作用的条件阳性。
然后在琼脂糖凝胶上运行PCR反应。对8个条件阳性反应和1个阴性反应的预期PCR产物进行凝胶纯化并送去Sanger测序。显示了代表性阳性(蓝色箭头,蓝色框)和阴性(红色箭头,红色框)反应的结果(图5)。
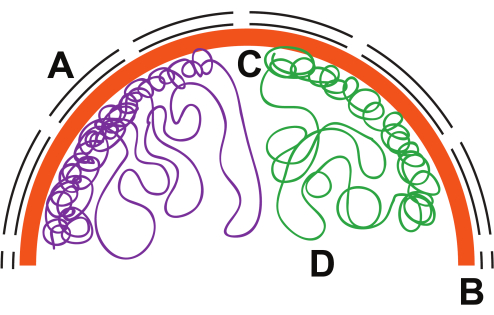
图1:细胞核中的染色体结构。 细胞核内的假设染色体组织。(一)核信封、黑线;(二)核叶片,橙色;(三)异染色质、压实线;(四)真染色质,松散的环。 请点击此处查看此图的大图。
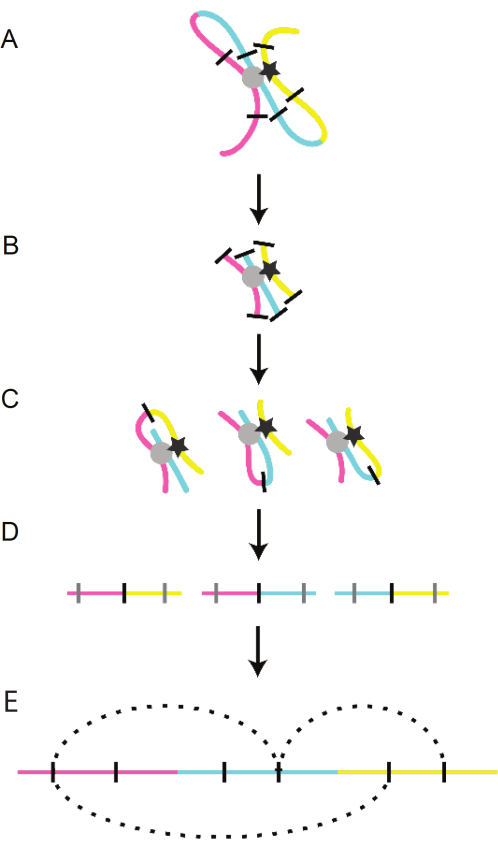
图 2:3C 协议的原理图。线性染色体的远处部分(蓝色、黄色和粉红色)通过调节环在细胞核内紧密相连。(A)环结构由转录因子(灰色圆圈和黑星)介导;这些相互作用通过化学交联得以保留。(B)环通过酶消化(黑线)被打破。(C)远处染色质片段通过消化产生的粘附末端连接在一起。(D)从蛋白质中纯化DNA。(E)片段内的序列被识别并映射回基因组。请点击此处查看此图的大图。
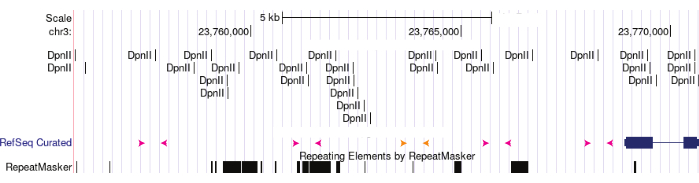
图 3:3C 引物方案。 3C引物围绕距离目标基因组位置不同距离的限制性位点设计。粉红色箭头代表围绕DpnII位点的实验引物组。实验引物可以混合和匹配,以评估该区域的染色质环。橙色引物代表没有DpnII位点的阴性对照。 请点击此处查看此图的大图。
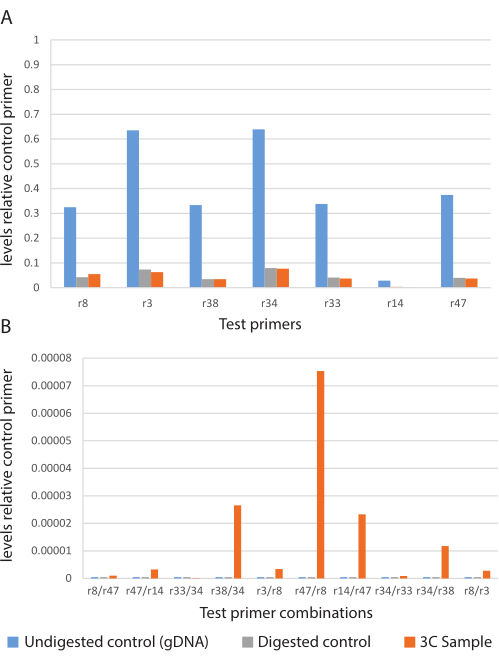
图 4:3C 实验的代表性 qPCR 数据。 3C测试引物组的相对丰度。(A)对照图显示未消化对照(蓝色)、消化对照(灰色)和3C样品(橙色)中产物的相对丰度。(二)3C实验;未消化对照(蓝色)、消化对照(灰色)和3C样品(橙色)中测试引物组合的产物相对丰度。 请点击此处查看此图的大图。
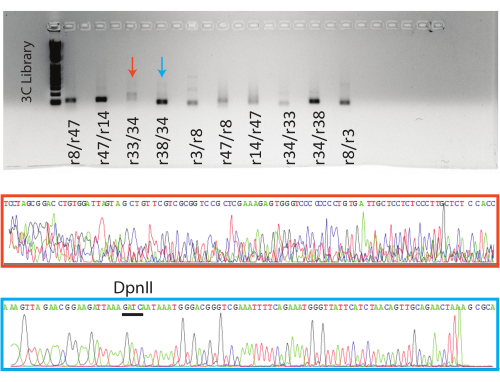
图 5:3C qPCR 产物的可视化。 顶部是凝胶,凝胶带有指示样品的qPCR终点产品。下图为指示反应的代表性Sanger序列示踪文件。橙色样品是假阳性的示例(见 图4, r38/34),蓝色样品是真阳性的示例,迹线上方指示了DpnII位点。 请点击此处查看此图的大图。
| 网站 | % 消化 |
| R8 | 86.98 |
| R3 | 88.44 |
| R38 | 89.64 |
| R34 | 87.55 |
| R33 | 87.85 |
| R14 | 86.97 |
| R47 | 89.45 |
表 1:计算的消解效率。
| 名字 | 序列 | 基因组位点 | ||
| r3 前驱 | ACGCAAGTAAAATTCTGGTTTTTGACC | chrX:11361475 | ||
| R3 房车 | TTTCCTGAGCTCTAACCATGTTTGC | chrX:11361561 | ||
| r38 前驱 | TTACTTCTGAAGTAATCTTTTCTTATCCCC | chrX:5859700 | ||
| R38 房车 | AGACGAGCTGATTAAAAGTAGTTGAGAG | chrX:5859775 | ||
| r34 前驱 | ATTTGTGGATTGCGTGGAGACG | chrX:5429702 | ||
| R34 房车 | AATAATCCTCTTAACAAACGTGGCC | chrX:5429777 | ||
| r33 前驱 | AAGAGTTGTCCAAAATAAATTGAGCTAAC | chrX:6296704 | ||
| R33 房车 | TTCAGAAAAGTAAACTTTGACTTGGAACG | chrX:6296807 | ||
| r14 前驱 | AATTATCGATTTCCATCGCGCAG | chrX:8036367 | ||
| R14 房车 | ATTTCAATGAAAATGTAAAAAATGTTCCTTCTC | chrX:8036427 | ||
| r47 前驱 | ATCTAGACTTGATAATTTGTGTGTCCTC | chrX:9464939 | ||
| R47 房车 | AAGTTCTGCAACTGTTAGATGAATGAATAACAC | chrX:9465064 | ||
| r8 前驱 | GAGAATGTTGTTCTGTTAACTGAAAACTTG | chrX:11094257 | ||
| R8 房车 | TTACGAAATTTGGTAGTTGGACC | chrX:11094362 | ||
| 对照底漆前驱 | CAATCGTCTCGCTCACTTGTC | chrX:7608049 | ||
| 对照底漆RVS | GATGTGAGCAACAAGGCACC | chrX:7608166 | ||
表2:代表性3C实验的引物。
讨论
3C是一种植根于基本分子技术的强大技术。正是这种基本工具的基础使3C成为一种与本科生一起使用的有趣技术。由于最近有如此多的研究在如此广泛的范围内观察染色质动力学,利用这些结果在单个基因或基因组区域设计一个狭隘的实验,有可能在本科研究中创造一个独特而有影响力的实验。通常,像这样的实验对于本科生来说被认为太先进了,但通过仔细的计划,它们很容易实现。需要注意的是,设计用于探测3C文库捕获的染色质连接的检测方法可以从半定量终点PCR到全基因组测序。事实上,来自第一篇3C论文16 的数据是由qPCR生成的。这种广泛的检测方法都可以使用,因为所有3C技术都产生相同的产品 - 代表细胞核中3D连接的DNA片段库。
这里介绍的是更灵活和更宽松的协议的改编,更适合本科研究人员。上面列出的暂停期意味着隔夜延误;然而,这些暂停可能会持续到周末,对于细胞和细胞核,可能会持续数周。最关键的考虑因素是工作何时完成。通常在协议中,当暂停不是一种选择时,存在时间敏感的步骤。除了几个点(第 1 天和第 2 天)之外,还有很多地方可以停止和冷冻样品。在与本科生合作时,这些至关重要,因为实验室工作的时间表和时间需要灵活。除了将这些站点设计到协议中外,还鼓励本科生成对甚至三到四人一组工作。小组适用于此协议,因为学生可以相互支持并创建一个伙伴系统,以便每个人都可以安全地工作。实验室工作也更有趣,其他人参与其中。通过小组,学生还可以处理各种问题,重点是染色质的组织,同时仍然执行相同的协议。因此,即使学生正在从事不同的项目,协议也将他们的努力联系起来,因此,他们可以相互支持。
其他改编旨在解决这样一个事实,即某些专业工具和设备不一定在所有本科院校中都能找到。这些设备包括但不限于qPCR热循环仪、凝胶记录系统和纳米体积光谱仪。事实上,这些设备很方便,但不是必需的。这里,引物设计部分也描述了3C的经典方法;这涉及确定感兴趣的基因组位点,并据此评估更远的染色质接触点的其他基因组位点。如果使用已发布的数据集,例如使用 Hi-C 的数据集,则此技术也非常有效,其中标识了已知的正(连接)和负(非连接)位点。使用这些已发布的数据集设计实验是教学实验室的另一个很好的适应,因为成功鉴定染色质连接的机会通常更大。此外,研究文章可以在课堂上讨论并用作参考。
该协议使用改进的qPCR方法来可视化3C产物的形成。控制对于3C技术的成功至关重要。每个实验都使用样品对照和引物对照来确定3C程序的完成情况。样品对照包括未消化的对照(基因组DNA)和消化的对照。未消化的对照确定引物组的基线信号,并与交联消化对照一起使用以确定消化效率预计任何针对限制性内切位点的引物的产物都会下降。将此值与未消化的对照进行比较,可以指示样品的消化情况。
PCR的引物包括对照引物和测试引物。对照引物是靠近被测定基因组区域的引物组,不包含限制性内切位点。这为确定测试引物PCR产物的丰度提供了基线。测试引物是正向和反向引物,位于特定感兴趣的基因组位点的限制性位点两侧(图3)。比较使用这些引物组的反应以确定消化效率,因为如果限制性位点被切割,产物丰度应该会下降。在确定染色质组织时,将来自一个位点的一个测试引物与来自不同基因组位点的另一个测试引物配对,以确定这两个位点在3D空间中是否靠近。在这种情况下,期望只能使用3C样品作为模板找到PCR产物。
需要注意的是,即使是经过验证的引物也有失败的倾向(图4:r14引物组)。此外,PCR产物经常在对照反应和未预测染色质连接的反应中鉴定(例如消化的对照,因为它没有连接)。这些实例经过排序,要么未通过 Sanger QC,要么在没有定义序列的情况下返回(图 5)。此外,传统的3C实验会产生一个“对照模板”,即一个未交联、消化和连接的DNA样品,它代表了用给定数量的DNA可以产生的所有可能的连接片段。“对照模板”在比较两个基因组位点之间的qPCR信号强度以确定信号是代表真正的相互作用还是只是随机关联方面起着重要作用。创建“对照模板”可能会有问题,因为大部分被测定的染色质必须以人工染色体的形式捕获并与3C样品一起处理。确保这样的结构可能不可行,并且创建一个可能超出了学期项目的范围。由于这些困难,我们建议使用对照引物。对照引物不会取代“对照模板”的所有功能,但仍提供分析数据以做出“存在”或“不存在”决定的机会。
进行qPCR时,使用等量的样品很重要。即使使用纳米分光光度计(如纳米滴),也应通过从已知浓度的基因组DNA生成标准曲线并将3C样品拟合到该线来确定。这些金额应记录在案,并在随后的PCR中使用。PCR反应的质量也很重要。当PCR在qPCR中运行时,使用荧光测量产物丰度并记录。该记录可在扩增图中访问。程序完成后,检查扩增图并确保反应(无模板对照除外)有三个阶段:基线、指数和平台/饱和阶段非常重要。重要的是要检查反应是否具有指数相,特别是对于设置阈值(见下文)。此外,对于连续稀释的样品,Ct值应与样品稀释度一致(最高浓度的Ct值最低,而最低浓度的Ct值最高)。在扩增图中没有反映这种变化的样品需要新的稀释或表明3C样品形成存在更大的问题。最后,在生成标准曲线的同时,PCR软件将计算PCR效率和R2 值。PCR效率应大于90%,R2 值应大于0.99。如果不满足这些条件中的任何一个,则可能是样品或PCR引物有问题。
qPCR后,可以使用qPCR Ct计算每个反应的消化百分比和3C相互作用的存在。要确定这些,首先必须设置PCR反应的阈值。这通常使用qPCR机附带的软件完成。设置阈值将定义将用于比较样品Ct值的PCR产物的浓度。阈值应将扩增指数阶段PCR反应的扩增曲线一分为二。只能比较具有指数扩增的PCR反应(在这种情况下,对照引物和测试引物反应),因为这是确保反应以相同速率扩增DNA并且可以忠实比较的唯一方法。在分析3C图时,条件阳性反应被确定为在对照样品,基因组对照和消化对照上具有更多产物的反应(图4B)。然而,在PCR产物的凝胶纯化之后,必须使用Sanger测序进一步验证这些样品。
桑格测序后,可以使用Blat分析通过QC的样品。该分析的目的是确定样品是否具有限制性位点两侧的两个靶基因组位点的序列(在本协议的情况下为 DpnII)。如果两个序列都被鉴定出来,那么3C片段可以被认为是经过验证的。如果Blat的结果没有返回预期的序列,这可能表明一个或两个引物不是最佳的,导致假阳性qPCR结果。假阳性样本的跟踪文件将具有未定义的基本峰,并且 Seq 报告将主要包含“n”个碱基调用。
Sanger验证至关重要,因为人工PCR产物形成的假阳性是可能的。当测序产物没有预期的靶序列或适当3C片段的DpnII位点特征时,可以识别这些假阳性(图5)。PCR片段的测序也为实验提供了另一个数据点,并让学生们明白,3C技术正在识别在细胞核内的3D空间中聚集在一起的远距离基因组位点。
3C技术以灵活,直接的程序为本科生提供了丰富的基础分子技术。这种3C技术也是其他结合下一代测序(NGS)的3C技术的起点。这些类型的实验可以使本科生了解生物信息学的重要方面,并植根于此处概述的基本原理。本科经验和参与是他们作为年轻科学家成功和发展的关键。通过提供这些机会,本科生可以加强对基本原则的理解,同时建立他们解决尖端技术和问题的信心。
披露声明
作者声明不存在利益冲突。
致谢
这项工作得到了美国国立卫生研究院国家普通医学科学研究所的罗德岛机构发展奖(IDeA)生物医学研究卓越网络(拨款号P20GM103430)和布莱恩特健康与行为科学中心的部分支持。
材料
| Name | Company | Catalog Number | Comments |
| 37% Formaldehyde | Millapore-Sigma | F8775 | |
| 100% Ethanol | Millapore-Sigma | E7023 | |
| CaCl2 | MP Biomedical | 215350280 | |
| chloroform | Millapore-Sigma | C0549 | |
| cOmplete, EDTA-free Protease Inhibitor Cocktail | Millapore-Sigma | COEDTAF-RO | mixed to 50x in water. Diluted to 1x in Sucrose buffer and GB buffer fresh |
| Dithiothreitol (DTT) | Millapore-Sigma | D0632 | 1 M stock diluted to 500 µM in Sucrose buffer and GB buffer fresh |
| DpnII | NEB | R0543M | |
| Glycerol | Millapore-Sigma | G9012 | |
| glycine | Millapore-Sigma | G8898 | |
| glycogen | Millapore-Sigma | 10901393001 | |
| HEPES | Millapore-Sigma | H3375 | |
| KCl | Millapore-Sigma | P3911 | |
| KH2PO4 | Millapore-Sigma | P5655 | |
| methyl green pyronin | Millapore-Sigma | HT70116 | |
| MgAc2 | Thermoscientific | 1222530 | |
| Na2HPO4 | Millapore-Sigma | S5136 | |
| NaCl | Millapore-Sigma | S9888 | |
| phenol-chloroform | Millapore-Sigma | P3803 | |
| Pronase | Millapore-Sigma | 11459643001 | |
| Proteinase K | IBI Scientific | IB05406 | |
| qPCR Ready mix (Phire Taq etc) | Millapore-Sigma | KCQS07 | |
| RNase A | Millapore-Sigma | R6148 | |
| Sodium Acetate | Millapore-Sigma | S2889 | |
| sodium dodecyl sulfate (SDS) | Millapore-Sigma | L3771 | |
| Sucrose | Millapore-Sigma | S0389 | |
| T4 DNA Ligase | Promega | M1804 | |
| Tris-HCl | Millapore-Sigma | 108319 | |
| Triton X-100 | Millapore-Sigma | T9284 | |
| Trypsin-EDTA | Millapore-Sigma | T4049 |
参考文献
- McBryant, S. J., Adams, V. H., Hansen, J. C. Chromatin architectural proteins. Chromosome Research. 14 (1), 39-51 (2006).
- Nalabothula, N., et al. The chromatin architectural proteins HMGD1 and H1 bind reciprocally and have opposite effects on chromatin structure and gene regulation. BMC Genomics. 15, 92 (2014).
- John, S., et al. Chromatin accessibility pre-determines glucocorticoid receptor binding patterns. Nature Genetics. 43 (3), 264-268 (2011).
- Fawcett, D. W. On the occurrence of a fibrous lamina on the inner aspect of the nuclear envelope in certain cells of vertebrates. The American Journal of Anatomy. 119 (1), 129-145 (1966).
- Reddy, K. L., Zullo, J. M., Bertolino, E., Singh, H. Transcriptional repression mediated by repositioning of genes to the nuclear lamina. Nature. 452 (7184), 243-247 (2008).
- Finlan, L. E., et al. Recruitment to the nuclear periphery can alter expression of genes in human cells. PLoS Genetics. 4 (3), e1000039 (2008).
- Chambeyron, S., Da Silva, N. R., Lawson, K. A., Bickmore, W. A. Nuclear re-organisation of the Hoxb complex during mouse embryonic development. Development. 132 (9), 2215-2223 (2005).
- Chambeyron, S., Bickmore, W. A. Chromatin decondensation and nuclear reorganization of the HoxB locus upon induction of transcription. Genes and Development. 18 (10), 1119-1130 (2004).
- Mahy, N. L., Perry, P. E., Gilchrist, S., Baldock, R. A., Bickmore, W. A. Spatial organization of active and inactive genes and noncoding DNA within chromosome territories. The Journal of Cell Biology. 157 (4), 579-589 (2002).
- Mahy, N. L., Perry, P. E., Bickmore, W. A. Gene density and transcription influence the localization of chromatin outside of chromosome territories detectable by FISH. The Journal of Cell Biology. 159 (5), 753-763 (2002).
- Li, B., Carey, M., Workman, J. L. The role of chromatin during transcription. Cell. 128 (4), 707-719 (2007).
- Chen, A., Chen, D., Chen, Y. Advances of DNase-seq for mapping active gene regulatory elements across the genome in animals. Gene. 667, 83-94 (2018).
- Hughes, A. L., Rando, O. J. Mechanisms underlying nucleosome positioning in vivo. Annual Review of Biophysics. 43, 41-63 (2014).
- Weiner, A., Hughes, A., Yassour, M., Rando, O. J., Friedman, N. High-resolution nucleosome mapping reveals transcription-dependent promoter packaging. Genome Research. 20 (1), 90-100 (2010).
- Hughes, A. L., Jin, Y., Rando, O. J., Struhl, K. A functional evolutionary approach to identify determinants of nucleosome positioning: A unifying model for establishing the genome-wide pattern. Molecular Cell. 48 (1), 5-15 (2012).
- Dekker, J., Rippe, K., Dekker, M., Kleckner, N. Capturing chromosome conformation. Science. 295 (5558), 1306-1311 (2002).
- van Berkum, N. L., Dekker, J. Determining spatial chromatin organization of large genomic regions using 5C technology. Methods in Molecular Biology. 567, 189-213 (2009).
- Smith, E. M., Lajoie, B. R., Jain, G., Dekker, J. Invariant TAD boundaries constrain cell-type-specific looping interactions between promoters and distal elements around the CFTR locus. American Journal of Human Genetics. 98 (1), 185-201 (2016).
- Crane, E. E. Two inputs into C. elegans dosage compensation: Chromosome conformation and the miRNA-specific argonaute ALG-2. University of California, Berkeley. , (2012).
- Bell, A. C., West, A. G., Felsenfeld, G. The protein CTCF is required for the enhancer blocking activity of vertebrate insulators. Cell. 98 (3), 387-396 (1999).
- Cuadrado, A., et al. Specific contributions of cohesin-SA1 and cohesin-SA2 to TADs and polycomb domains in embryonic stem cells. Cell Reports. 27 (12), 3500-3510 (2019).
- Sarro, R., et al. Disrupting the three-dimensional regulatory topology of the Pitx1 locus results in overtly normal development. Development. 145 (7), (2018).
- Tolhuis, B., et al. Interactions among polycomb domains are guided by chromosome architecture. PLoS Genetics. 7 (3), e1001343 (2011).
- Splinter, E., de Wit, E., van de Werken, H. J. G., Klous, P., de Laat, W. Determining long-range chromatin interactions for selected genomic sites using 4C-seq technology: From fixation to computation. Methods. 58 (3), 221-230 (2012).
- Fernández-Miñán, A., Bessa, J., Tena, J. J., Gómez-Skarmeta, J. L. Chapter 21 - Assay for transposase-accessible chromatin and circularized chromosome conformation capture, two methods to explore the regulatory landscapes of genes in zebrafish. Methods in Cell Biology. 135, 413-430 (2016).
- Dekker, J. The three "C" s of chromosome conformation capture: controls, controls, controls. Nature Methods. 3 (1), 17-21 (2006).
- Hagège, H., et al. Quantitative analysis of chromosome conformation capture assays (3C-qPCR). Nature Protocols. 2 (7), 1722-1733 (2007).
- Lajoie, B. R., van Berkum, N. L., Sanyal, A., Dekker, J. My5C: Webtools for chromosome conformation capture studies. Nature Methods. 6 (10), 690-691 (2009).
- Han, M., Wei, G., McManus, C. E., Hillier, L. W., Reinke, V. Isolated C. elegans germ nuclei exhibit distinct genomic profiles of histone modification and gene expression. BMC Genomics. 20 (1), 500 (2019).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。