Method Article
3C로 A 받기: 학부생을 위한 염색체 형태 캡처
요약
여기에서는 학부 참여 및 학습에 중점을 두고 염색체 형태 캡처(3C) 기술의 적응을 자세히 제시합니다.
초록
염색체 형태 포획(3C)은 염색질의 3차원 조직에 대한 자세한 정보를 제공하는 유사한 기술 제품군(예: Hi-C, 4C 및 5C, 여기서는 3C 기술이라고 함)을 생성한 강력한 도구입니다. 3C 기술은 암세포의 염색질 조직 변화를 모니터링하는 것부터 유전자 프로모터와의 인핸서 접촉 식별에 이르기까지 광범위한 연구에 사용되었습니다. 이러한 기술을 사용하는 많은 연구가 복잡한 샘플 유형 (즉, 단일 세포 분석)으로 게놈 전반에 걸친 큰 질문을하고 있지만, 종종 손실되는 것은 3C 기술이 광범위한 연구에 적용 할 수있는 기본 분자 생물학 방법에 기반을두고 있다는 것입니다. 염색질 조직에 대한 긴밀한 초점을 맞춘 문제를 해결함으로써 이 최첨단 기술을 사용하여 학부 연구 및 교육 실험실 경험을 향상시킬 수 있습니다. 이 논문은 3C 프로토콜을 제시하고 학부 연구 및 교육 경험에서 주로 학부 기관에서 구현하기 위한 적응 및 강조점을 제공합니다.
서문
유기체의 게놈은 기능에 필요한 모든 유전자뿐만 아니라 언제 어떻게 사용해야 하는지에 대한 모든 지침을 담고 있습니다. 이것은 게놈에 대한 접근을 조절하는 것을 세포의 가장 중요한 기능 중 하나로 만듭니다. 유전자 기능을 제어하는 많은 메커니즘이 있습니다. 그러나 기본 수준에서 유전자 조절은 특정 DNA 서열(시스 조절 서열)에 결합하는 조절 전사 인자(트랜스 인자)의 능력으로 귀결됩니다. 이것은 타고난 능력이 아닙니다. 대신, 그것은 핵에서 게놈의 조직/구조에 의해 지배되며, 이는 트랜스인자 1,2,3에 대한 시스-조절 서열의 가용성/노출을 제어합니다. 트랜스 인자가 시스 조절 서열을 찾을 수 없으면 트랜스 인자가 규제 작업을 수행 할 수 없습니다. 이것은 게놈이 핵에서 어떻게 구성되어 있는지 이해하는 것을 중요한 탐구의 원천으로 만들었습니다.
간기 동안 핵의 진핵 염색체는 핵층과 핵 매트릭스에 고정된 자체 영역을 차지하므로 염색체를 스파게티 접시의 국수가 아니라 피자 조각처럼 만든다는 것이 널리 받아들여지고 있습니다. 염색체는 염색체의 일부를 비틀고 반복하는 단백질-DNA 상호 작용(염색질)에 의해 부분적으로 응축됩니다. 전자 현미경, 3차원 DNA 형광 제자리 혼성화(FISH) 및 DNA 태깅 기술(즉, 형광 및 인공 DNA 메틸화)을 통해 염색질의 비활성 도메인은 핵 주변을 따라 단단히 채워져 있는 것으로 밝혀졌습니다 4,5,6, 활성, 덜 응축된 염색질의 일부는 핵 내부에서 발견됩니다 7,8,9, 10입니다. 이 실험은 염색체 역학에 대한 광각 보기를 제공하지만 DNase 11,12 및 뉴클레오솜13,14,15 연구에서 관찰된 유전자 프로모터 주변에서 국부적으로 발생하는 변화를 포착하는 데는 거의 도움이 되지 않습니다.
고분해능 염색질 역학을 여는 열쇠는 3D 염색체 매핑 기술인 3C의 공식화였습니다. 3C 기법 자체는 염색질의 가교, 제한 효소에 의한 염색질 분해, 염색질 결찰 및 DNA 정제의 네 가지 주요 단계로 구성됩니다(그림 2). 이 과정에 의해 생성된 새로운 인공 DNA 단편은 선형적으로 멀리 떨어져 있는 DNA 조각들 사이의 밀접한 물리적 연관성을 나타내도록 특성화될 수 있다(16). 3C 기술은 3C의 초기 단계를 활용하여 더 광범위한 게놈 전체 질문(예: Hi-C, 4C, ChIP-C)을 하는 여러 스핀오프 기술을 만드는 기초가 되었습니다. 이 3C 기술 제품군은 염색체가 위상 관련 도메인(TAD)이라고 하는 여러 개별 단위로 구성되어 있음을 확인했습니다. TAD는 게놈에 암호화되어 있으며 고리가 없는 경계 16,17,18,19 옆에 있는 염색질 루프에 의해 정의됩니다. TAD 경계는 CCCT 결합 인자(CTCF) 및 응집력을 포함하여 진화적으로 보존되고 유비쿼터스한 두 가지 요인에 의해 유지되며, 이는 개별 TAD 내의 루프가 상호 작용하는 것을 방지합니다(16,20). 루프는 트랜스 인자와 조절 서열의 상호 작용, CTCF 및 응집력에 의해 매개됩니다21.
3C 기술을 사용하는 많은 연구가 광범위한 게놈 전반에 걸친 질문을 하고 복잡한 샘플 수집 기술을 사용하지만 3C 기술의 공식화는 기본 분자 생물학 기술을 기반으로 합니다. 따라서 3C는 학부 연구 및 교육 실험실 모두에 배포하기에 흥미로울 수 있습니다. 3C 기술은 더 작은 집중 질문에 사용할 수 있으며 질문의 초점과 방향에 따라 확장 또는 축소(단일 유전자22, 염색체16 및/또는 게놈18)에 본질적으로 유연합니다. 이 기술은 또한 광범위한 모델 시스템 7,16,19,23에 적용되었으며 그 용도에 있어 다재다능한 것으로 입증되었습니다. 따라서 3C는 학생들이 일반적인 분자 생물학 기술에 대한 경험을 쌓을 수 있는 동시에 지시된 질문에 답하는 데 귀중한 경험을 얻을 수 있다는 점에서 학부생에게 훌륭한 기술입니다.
여기에 제시된 것은 이전에 발표된 프로토콜24,25,26,27에 기초한 3C 라이브러리 준비를 위한 적응된 프로토콜이다. 이 프로토콜은 약 1 × 107 셀에 최적화되어 있지만 1 × 105 셀로 3C 라이브러리를 생성했습니다. 이 프로토콜은 다재다능한 것으로 입증되었으며 제브라피쉬 배아, 제브라피쉬 세포주 및 젊은 성인(YA) Caenorhabditis elegans(회충)에서 3C 라이브러리를 생성하는 데 사용되었습니다. 이 프로토콜은 또한 포유류 세포주와 추가 적응을 통해 효모에 적합해야 합니다.
이러한 적응의 목표는 학부생이 3C에 더 쉽게 접근할 수 있도록 하는 것입니다. 학부 교수 실험실에서 수행할 수 있는 것과 유사한 기술을 사용하도록 주의를 기울였습니다. 3C 기술은 학부생이 벤치, 교실 및 졸업 후 노력에서 발전에 도움이 될 기본 분자 생물학 기술을 배울 수 있는 많은 학습 기회를 제공합니다.
프로토콜
1. 프라이머 디자인
참고: 3C 프라이머 설계 도구는 온라인에서 사용할 수 있습니다28. 또는 학생들이 맞춤형 프라이머를 디자인할 수 있습니다(아래 참조).
- 프라이머 위치 식별
- UCSC 게놈 브라우저(http://genome.ucsc.edu/)를 열고 연구 유기체를 선택한 후 3C를 사용하여 평가할 게놈 영역을 검색합니다.
- 다음 창에서 Restr Enzymes를 클릭하여 브라우저 아래의 Mapping and Sequencing 탭에서 효소 트랙을 활성화합니다.
- 사용할 제한효소 를 입력하고 표시 모드를 pack으로 설정합니다. 제출을 클릭합니다.
- 변형(Variation) 및 반복(Repeats)에서 리피트마스커(RepeatMasker)가 밀도(dense)로 설정되어 있는지 확인합니다.
- 제한 부위를 가이드로 사용하여 마스킹된 반복 영역(검은색 막대) 내에 있지 않은 관심 부위를 식별합니다. 위치(맨 위 트랙 )를 클릭하고 원하는 길이(~600bp)로 드래그하여 제한 사이트를 둘러싼 업스트림 및 다운스트림 시퀀스 옆에 있는 300개의 염기쌍(bp)을 강조 표시합니다.
- 마우스를 놓으면 팝업 상자가 나타납니다. 게놈 위치를 기록하고 확대를 클릭한 다음 브라우저가 선택 항목에 맞게 다시 조정하도록 합니다.
- 위쪽 브라우저 리본의 보기 탭 위로 마우스를 가져간 다음 드롭다운 메뉴에서 DNA를 선택합니다.
- 기본 옵션을 그대로 두고 마스킹된 시퀀스의 지정을 확인합니다( N으로 만드는 옵션이 있음). DNA 가져오기를 클릭합니다.
- 그러면 강조 표시된 시퀀스가 창에 표시됩니다. 이 시퀀스를 복사하여 Primer3(https://primer3.ut.ee/)에 붙여넣습니다.
- Primer3 기본 설정을 사용하여 프라이머 선택을 클릭하여 프라이머를 생성합니다.
- 결과에서 제한효소의 위치를 기록하고 중간에 제한효소 부위가 위치하는 200-500 bp PCR 산물을 만드는 프라이머를 선택합니다.
- 이 단계에 따라 관심 사이트에서 1킬로베이스(kb), 2kb, 5kb, 10kb 및 20kb의 테스트 프라이머를 설계합니다.
- 입력 제어 프라이머를 설계하려면 제한 부위가 없는 관심 부위에서 1-2kB 떨어진 부위에 대해 이 단계를 반복합니다.
- 기능성 프라이머 검증
- 프라이머 기능을 검증하려면 적정된 프라이머 농도와 정제된 게놈 DNA를 사용하여 PCR 반응을 설정합니다. 정량적 또는 반정량적 수단을 통해 프라이머가 예상 제품을 생성하는지 확인합니다.
- 검증에 실패한 프라이머를 재설계합니다.
2. 1일차
참고: 프로토콜은 염색질 가교 후 및 핵 수집 후 일시 중지(-20°C에서 동결)할 수 있습니다. 이 단계는 학부생의 경우 평균 5-6시간이 걸립니다.
- Young-adult (YA) C. elegans 핵 수집 (Han et al.29에서 발췌)
- 염색질 가교
- 50mL 원뿔형 튜브의 M9 배지 30mL에 5,000개의 YA 웜을 수집하고 실온(RT)에서 2분 동안 400× g 으로 회전합니다.
- 벌레 펠릿을 M3로 9배 더 씻어 박테리아를 제거합니다.
- 상층액을 제거하고 2.7mL의 37% 포름알데히드(2% 최종)를 함유하는 47.3mL의 M9에 웜 펠릿을 재현탁하고 RT에서 30분 동안 교반(흔들기 또는 너트화)과 함께 배양합니다.
주의 : 포름알데히드를 주의해서 다루고 적절한 개인 보호 장비(PPE 실험실 코트, 적절한 장갑 및 보안경)를 사용하여 흄 후드 아래에서 작업하십시오. 포름알데히드는 눈, 코, 목, 폐에 영향을 미치는 자극제입니다. - RT에서 400 × g 에서 2 분 동안 웜을 회전시킵니다.
- 상층액을 제거하고, 웜 펠릿을 1 M 글리신 50 mL에 재현탁시킨다. RT에서 400 × g 에서 2 분 동안 웜을 회전시킵니다.
- 핵 수집
- 웜 펠릿을 6mL의 냉장 NP 완충액(pH 7.5에서 50mM HEPES, 40mM NaCl, 90mM KCl, 2mM EDTA, 0.5mM EGTA, 0.1% 트윈 20, 0.2mM DTT, 0.5mM 스페르미딘, 0.25mM 스페르민, 1x 완전 프로테아제 억제제)에 재현탁합니다.
- 웜 현탁액을 7mL의 느슨한 Dounce on ice로 옮깁니다. 샘플을 15회 돌리고 얼음을 5분 동안 유지합니다.
- 웜 현탁액을 7mL의 꽉 끼는 Dounce on ice로 옮깁니다. 샘플을 20회 돌리고 얼음을 5분 동안 유지합니다.
- 웜 현탁액을 깨끗한 15mL 원뿔형 튜브로 옮기고 NP 완충액을 총 10mL(약 4mL)에 추가합니다.
- 웜 서스펜션을 30초 동안 높은 설정으로 소용돌이칩니다. 샘플을 얼음에서 5분 동안 배양합니다.
- 이전 단계를 반복합니다.
- 웜 현탁액을 100 × g 에서 4°C에서 5분 동안 회전시킨다.
- 상층액을 새로운 15mL 원뿔형 튜브로 옮깁니다.
- 샘플에 벌레 파편이 있는지 확인하십시오. 광학 현미경으로 시료 10μL를 시각화합니다. 웜 파편이 있는 경우 4°C에서 5분 동안 2,000×g에서 샘플을 회전시키고, 펠릿을 새로운 10mL의 NP 완충액에 재현탁하고, 4°C에서 5 분 동안 100× g 에서 샘플을 다시 회전시킵니다. 펠릿을 버리고 상청액에 벌레 파편이 있는지 확인한 다음 샘플에 벌레 파편이 없을 때까지 반복합니다.
참고: 또는 40μm 셀 스트레이너(6x)와 20μm 셀 스트레이너(6x)를 통해 샘플을 변형시켜 웜 파편을 제거할 수도 있습니다. - 샘플에 벌레 파편이 없으면 샘플 5μL를 채취하고 메틸 그린 피로닌 5μL(핵은 파란색)를 추가하고 혈구계로 핵을 계산합니다.
- 시료를 2,000 × g 에서 4°C에서 5분 동안 회전시킵니다.
- 상청액을 제거하고 샘플을 얼음 위에 놓고 계속 진행하십시오. 또는 핵을 스냅 동결하고 -80 ° C에서 보관합니다.
- 염색질 가교
- 염색질 소화
- 450 μL의 깨끗한 물에 핵을 재현탁합니다. 핵을 깨끗한 1.5mL 미세 원심분리기 튜브로 옮깁니다.
- 가교 결합된 샘플에 60μL의 10x DpnII 제한 효소 완충액을 넣고 잘 혼합합니다.
참고: 여전히 가교된 DNA를 절단하는 다른 제한 효소를 사용할 수 있습니다. - 15 μL의 10 % 나트륨 도데 실 설페이트 (SDS)를 첨가하여 핵을 투과시키고 37 ° C에서 1 시간 동안 교반 (흔들기 또는 너트 화)과 함께 배양합니다.
주의: SDS는 자극제이며 피부를 통해 섭취하거나 흡수하면 독성이 있습니다. 조심스럽게 다루고 적절한 PPE(실험복, 적절한 장갑 및 보안경)를 사용하십시오. - 75 μL의 20% 트리톤 X-100을 첨가하고 37°C에서 1시간 동안 교반하면서 배양하여 SDS를 켄칭합니다.
- 샘플에서 10 μL를 소화되지 않은 대조군으로 채취합니다. 4 °C에서 보관하십시오.
- 400 U의 DpnII를 첨가하고, 교반하면서 37°C에서 밤새 인큐베이션한다.
3. 2일차
참고: 평균적으로 학부생이 이 단계를 완료하는 데 5시간이 걸립니다.
- 염색질 소화
- 추가로 200 U의 DpnII를 첨가하고, 가교결합된 샘플의 완전한 소화를 보장하기 위해 교반과 함께 37°C에서 4시간 동안 샘플을 인큐베이션한다.
- 분해 대조군으로 샘플에서 10 μL 분취량을 취합니다.
- 제한효소를 65°C에서 20분 동안 인큐베이션하여 제한효소를 열-불활성화시킨다(또는 제조자의 지시에 따름). 2.1.3단계를 계속합니다.
- 효소가 열에 의해 불활성화될 수 없는 경우에, 10% SDS의 80 μL를 추가하고, 65 °C에 30 분 동안 검체를 배양하십시오. 그런 다음 375 μL의 20 % Triton X-100을 추가하고 소용돌이 치면서 혼합합니다. 샘플을 37°C에서 1시간 동안 인큐베이션하고, 단계 2.2를 계속한다.
- 염색질 결찰
- 샘플을 깨끗한 50mL 원뿔형 튜브로 옮기고 샘플 부피를 분자 등급 H2O로 5.7mL로 조정하고 소용돌이치며 혼합합니다.
- 700μL의 10x T4 Ligase 버퍼를 추가하고 소용돌이치면서 혼합합니다.
- 60U의 T4 DNA Ligase를 추가하고 소용돌이치면서 혼합합니다.
- 16°C에서 하룻밤 동안 배양합니다.
4. 3일차
참고: 평균적으로 학부생이 이 단계를 완료하는 데 15-30분이 걸립니다. 하룻밤 배양 후, 샘플을 동결시킬 수 있다.
- 단백질 분해 및 역가교
- 3C 샘플에 30μL의 프로테이나제 K(10mg/mL)를 추가하고 교반하면서 65°C에서 밤새 배양합니다. 5 μL의 proteinase K(10 mg/mL)를 소화되지 않은 소화 대조군에 첨가하고 65°C에서 하룻밤 동안 교반하면서 배양합니다.
5. 4일차
알림: 평균적으로 학부생이 이 단계를 완료하는 데 4-5시간이 걸립니다.
- 3C 라이브러리의 정제
- 3C 샘플에 30μL의 RNase A(10mg/mL)를 추가하고 소용돌이쳐 혼합합니다. 샘플을 37°C에서 45분 동안 인큐베이션한다.
- 7mL의 페놀-클로로포름을 시료에 넣고 흔들어 섞는다.
주의: 페놀-클로로포름은 피부와 눈을 자극하며 접촉을 치료하지 않으면 화상을 입을 수 있습니다. 페놀-클로로포름은 적절한 보안경과 장갑(니트릴)이 있는 흄 후드에 사용해야 합니다. - RT에서 3,270 × g 에서 15분 동안 샘플을 원심분리하고, 수성상을 수집하고, 깨끗한 50 mL 원뿔형 튜브로 옮긴다. 같은 부피의 클로로포름을 첨가하고 흔들어서 샘플을 혼합합니다.
- RT에서 3,270 × g 에서 15분 동안 샘플을 원심분리하고, 수성상을 수집하고, 깨끗한 50 mL 원뿔형 튜브로 옮긴다. 분자 등급 H2O7.5mL, 100% 에탄올 35mL 및 (선택 사항) 글리코겐 7μL(1mg/mL)를 추가합니다. 흔들어서 혼합하고 샘플이 얼 때까지 -80°C에서 배양합니다.
알림: 샘플이 얼기까지 1시간 이상이 소요될 수 있습니다. 여기에서 프로토콜을 일시 중지할 수 있습니다.- 3C 샘플이 동결되는 동안 대조군 샘플을 정제합니다. 대조군 샘플에 분자 등급의 물을 사용하여 부피를 500μL로 조정하고 튜브를 튕겨 RNase A(10mg/mL) 혼합물 2μL를 추가합니다.
- 튜브 바닥에서 샘플을 수집하기 위해 짧게 회전합니다. 샘플을 37°C에서 45분 동안 인큐베이션한다.
- 대조 샘플에 페놀-클로로포름 1mL를 넣고 튜브를 흔들어 혼합합니다. RT에서 3,270 × g 에서 15분 동안 샘플을 원심분리합니다. 대조군의 수성상을 수집하고 깨끗한 1.5mL 미세 원심분리기 튜브에 넣습니다.
- 같은 부피의 클로로포름을 첨가하고 흔들어서 샘플을 혼합합니다. RT에서 3,270 × g 에서 15분 동안 샘플을 원심분리합니다. 대조군의 수성상을 수집하고 깨끗한 1.5mL 미세 원심분리기 튜브에 넣습니다.
- 100% 에탄올 1mL와 글리코겐 2μL(1mg/mL)를 추가합니다. 진탕하여 혼합하고 -80°C에서 30분 동안 배양합니다.
- 샘플을 4°C에서 3,270 × g 에서 15분 동안 원심분리합니다. 상청액을 제거하고, 750 μL의 냉장 70% 에탄올을 첨가한다.
- 4°C에서 3,270 × g 에서 10분 동안 샘플을 원심분리합니다. 상층액을 제거하고 샘플을 자연 건조합니다. 펠릿을 50 μL의 분자 등급H2O에 재현탁하거나 동결시키거나 단계 6을 계속한다.
- 시료를 4°C에서 3,270 × g 에서 60분 동안 원심분리한다. 상층액을 제거하고 식힌 70% 에탄올 10mL를 첨가합니다. 샘플을 흔들어 혼합하여 DNA 펠릿을 파괴하고 분해합니다.
- 시료를 4°C에서 3,270 × g 에서 30분 동안 원심분리한다. 상청액을 제거하고, 샘플을 RT에서 부분적으로 공기 건조시킨다. 펠릿을 위아래로 피펫팅하여 10mM Tris-HCl(pH 7.5)의 150μL에 재현탁한다. 이것이 바로 "3C 라이브러리"입니다.
참고: 샘플을 얼리거나 분석을 계속합니다.
6. 5일차
참고: 평균적으로 학부생이 이 단계를 완료하는 데 1-2시간이 걸립니다.
- 대조 프라이머의 표준 곡선을 사용하여 샘플 품질 결정
- 3C 시료 및 모든 대조군에 대한 DNA 농도를 정량화하고 시료를 30μg/μL로 조정합니다(농도가 허용하는 경우). 연속적으로 희석된 샘플을 4배 2배로 희석하여 각 샘플에 대해 5개의 희석(1x, 0.5x, 0.25x, 0.125x, 0.0625x)을 생성합니다.
알림: 정량화를 쉽게 수행할 수 없는 경우 s를 연속적으로 희석amp위에 표시된 대로 s를 진행하고 진행합니다. - 대조군 프라이머를 사용하여 희석된 각 샘플에 대해 3C, 게놈 대조군 및 분해 대조군에 대한 PCR 반응을 설정합니다: DNA 1μL, 5x 반응 완충액 10μL, 10mM dNTP 1μL, 10mM 대조군 프라이머(정방향 및 역방향 혼합) 1μL, Taq 중합효소 1μL 및 물 36μL.
- PCR 기계에 특정한 소프트웨어에 대한 지침에 따라, 다음과 같이 사이클링 조건을 사용하여 표준 곡선 PCR 프로그램을 설정한다: 98°C에서 30초; 98°C에서 5초, 60°C에서 5초, 72°C에서 10초의 30주기; 72°C에서 1분; 4 °C 유지.
- 소프트웨어를 사용하여 샘플에 대한 표준 곡선을 생성합니다. 소프트웨어가 곡선을 생성한 후 PCR 효율과R2 값을 기록해 둡니다.
- 3C 시료 및 모든 대조군에 대한 DNA 농도를 정량화하고 시료를 30μg/μL로 조정합니다(농도가 허용하는 경우). 연속적으로 희석된 샘플을 4배 2배로 희석하여 각 샘플에 대해 5개의 희석(1x, 0.5x, 0.25x, 0.125x, 0.0625x)을 생성합니다.
- DNA 농도 결정
- 3C 샘플의 DNA 농도를 결정하려면 샘플을 알려진 농도의 게놈 DNA 샘플과 비교합니다. 샘플을 30 ng/μL로 희석하고 게놈 대조군을 10 ng/μL에서 0.01 ng/μL까지 2단계로 연속적으로 희석하여 표준 곡선을 만듭니다.
알림: 정량화를 쉽게 할 수 없는 경우 s를 희석하십시오.ample 1:10, 그리고 진행하십시오. - 대조군 프라이머를 사용하여 희석된 각 샘플에 대해 3C, 게놈 대조군 및 분해 대조군에 대한 PCR 반응을 설정합니다: DNA 1μL, 5x 반응 완충액 10μL, 10mM dNTP 1μL, 10mM 대조군 프라이머(정방향 및 역방향 혼합) 1μL, Taq 중합효소 1μL 및 물 36μL.
- PCR 기계에 특정한 소프트웨어에 대한 지시에 따라, 다음과 같이 사이클링 조건을 사용하여 표준 곡선 PCR 프로그램을 설정한다: 98°C에서 30초; 98°C에서 5초, 60°C에서 5초, 72°C에서 10초의 30주기; 72°C에서 1분; 4 °C 유지.
- 소프트웨어를 사용하여 샘플에 대한 표준 곡선을 생성하고 곡선에 3C 샘플을 플로팅합니다. 3C 샘플 풍부도의 위치를 알려진 농도의 게놈 DNA 반응에 의해 생성된 표준 곡선과 비교하고 3C 샘플을 30ng/μL로 조정합니다.
- 3C 샘플의 DNA 농도를 결정하려면 샘플을 알려진 농도의 게놈 DNA 샘플과 비교합니다. 샘플을 30 ng/μL로 희석하고 게놈 대조군을 10 ng/μL에서 0.01 ng/μL까지 2단계로 연속적으로 희석하여 표준 곡선을 만듭니다.
- 염색질 상호 작용의 존재 또는 부재 결정
- 30ng의 3C 샘플, 게놈 대조군 샘플 및 분해된 대조군 샘플(DNA 1μL, 5x 반응 완충액 10μL, 10mM dNTP 1μL, 10mM 프라이머(정방향 및 역방향 혼합) 1μL, Taq 중합효소 1μL 및 물 36μL로 qPCR 반응을 설정합니다.
참고: 반응은 대조군 프라이머, 개별 게놈 유전자좌에 대한 테스트 프라이머 및 염색질 상호작용을 결정하는 데 사용되는 테스트 프라이머의 원하는 조합(부위 "a" 순방향 및 부위 "b" 역방향)을 사용하여 설정해야 합니다(그림 3). - PCR 기계의 소프트웨어에 대한 지침에 따라 PCR 프로그램을 다음과 같이 설정합니다: 98°C에서 30초; 98°C에서 5초, 60°C에서 5초, 72°C에서 10초의 40주기; 72°C에서 1분; 4 °C 유지.
- PCR을 실행한 후 증폭 플롯을 검사합니다. PCR 반응이 기하급수적으로 증폭되어 매 주기마다 두 배로 증가하는지 확인합니다.
참고: 지수 증폭을 나타내지 않는 반응은 더 이상 분석할 수 없습니다. - PCR 기계의 소프트웨어에 대한 지침에 따라 qPCR 실험의 임계값을 정의합니다.
- 모든 샘플에 대한 Ct 값을 내보냅니다.
- 수학식 1을 사용하여 분해되지 않은 대조군(UC) 및 분해된 대조군(DC) 샘플에서 테스트 프라이머(TP) 및 대조군 프라이머(CP) Ct 값을 사용하여 소화 효율을 결정합니다.
다이제스트 백분율 = 100 -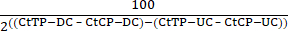 (1)
(1) - 이 값은 80%-90% 소화 범위에 있어야 합니다. 이 값을 기록합니다(그림 4A).
- 방정식 2를 사용하여 분해되지 않은 대조군(UC) 및 3C(3C) 샘플에서 테스트 프라이머 조합(TPC) 및 대조군 프라이머(CP) Ct 값을 사용하여 상대적인 염색질 상호작용을 결정합니다.
염색질 상호 작용 = 100 -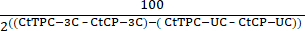 (2)
(2) - 각 검정 프라이머 조합에 대한 막대 그래프에 각 샘플의 값을 플로팅합니다.
- 3C 샘플의 신호를 대조군 샘플과 비교하여 3C 샘플이 대조군 샘플에 비해 특정 염색질 상호작용이 풍부한지 확인합니다. 대조군에 비해 농축을 보여주는 3C 샘플은 조건부 양성으로 간주될 수 있으며 Sanger 염기서열 분석을 사용한 검증이 필요합니다(그림 4B).
참고: 그래프 데이터를 분석할 때 "제어 템플릿"을 사용하지 않는 한(토론 참조) 반응 간의 풍부도(즉, 한 프라이머 세트에서 다음 프라이머 세트로)를 비교할 수 없다는 점을 기억하는 것이 중요합니다.
- 30ng의 3C 샘플, 게놈 대조군 샘플 및 분해된 대조군 샘플(DNA 1μL, 5x 반응 완충액 10μL, 10mM dNTP 1μL, 10mM 프라이머(정방향 및 역방향 혼합) 1μL, Taq 중합효소 1μL 및 물 36μL로 qPCR 반응을 설정합니다.
- 3C 제품 식별
- PCR 산물을 1.5% 아가로스 겔 상에서 실행한다.
- UV 라이트 박스 또는 겔 문서화 시스템을 사용하여 PCR 산물의 정확한 크기에 대해 겔을 시각화합니다.
참고: 잘 구성된 3C 라이브러리에는 다양한 범위의 DNA 단편이 포함되며, 특이성에 대한 프라이머의 이전 검증에도 불구하고 3C 라이브러리의 PCR 반응은 많은 밴드를 포함할 가능성이 있습니다(그림 5A). 이것은 3C 라이브러리의 분석을 방해하지 않습니다. - 예상 단편 크기에 해당하는 생성물 밴드를 절제하고 아래에 설명된 상용 키트 또는 수제 프로토콜의 지침에 따라 PCR 산물의 겔 추출을 수행합니다.
주의 : 자외선은 알려진 발암 물질이므로 자외선에 노출되는 시간을 제한하기 위해 각별한 주의를 기울여야 합니다. 자외선 차단 보안경, 검체 보호대, 장갑 및 실험복을 착용해야 합니다.- 수제 정화 카트리지를 만들려면 바늘로 0.5mL 튜브 바닥에 구멍을 뚫습니다.
- 면봉에서 소량의 면봉을 0.5mL 튜브 바닥에 넣고 튜브의 절반 이상을 채우지 마십시오.
- 0.5 mL 튜브를 1.5 mL 튜브에 넣고; 더 작은 튜브가 튜브가 아닌 더 큰 튜브의 립에 놓이도록 하십시오.
- 젤 조각을 조심스럽게 작은 조각으로 자르고 카트리지의 0.5mL 튜브에 넣습니다.
- 어셈블리를 -20°C 냉동고에 5분 동안 놓습니다. RT에서 13,000 ×g 에서 3분 동안 어셈블리를 회전시킵니다.
- 추출된 DNA가 담긴 1.5 mL 튜브를 완충액에 보관하고, 아가로스 파편이 들어 있는 0.5 mL 튜브를 폐기한다.
- 정제된 DNA는 3C 단편에 대한 정방향 및 역방향 프라이머를 사용하여 Sanger 시퀀싱을 위해 보낼 수 있습니다.
- Blat를 사용한 염색질 접촉 식별
- 시퀀싱이 완료되면 샘플이 시퀀싱 보고서에 표시된 품질 관리 표준을 충족하는지 확인합니다.
- QC를 통과하는 염기서열의 경우, ApE(Another Plasmid Editor)와 같은 염기서열 분석 편집 프로그램에서 .seq 및 .ab1(trace) 파일을 열고, .ab1 파일에서 명확한 염기 호출 피크를 검사하고, .seq 파일에서 잘못 호출된 피크를 편집합니다.
- 편집된 .seq 파일을 사용하여 DpnII 사이트를 검색합니다. 시퀀스 결과에 따라 보고된 시퀀스의 중간에 있어야 합니다.
- 서열이 예상되는 표적 서열인지 확인하기 위해, DpnII 부위 및 3C에 대해 시험된 게놈 유전자좌의 전방 프라이머에 상응하는 업스트림 서열의 추가 30-50 bp를 강조 표시한다. 대상 종에 대해 이 시퀀스의 Blat 검색을 수행합니다. 게놈 유전자좌가 프라이머의 게놈 유전자좌와 일치하는지 확인하십시오.
- 역방향 프라이머 서열에 대해 위의 단계를 반복하여 반환된 게놈 유전자좌가 역방향 프라이머의 게놈 유전자좌와 일치하는지 확인합니다.
결과
이 절차는 하나의 실험 3C 샘플과 두 개의 대조군 샘플(소화되지 않은 및 소화됨)을 생성합니다. 이들 3개의 샘플을 이용하여, qPCR을 수행하였다. 이러한 결과로부터, 소화 효율을 계산하고(수학식 1) 기록하였다(표 1). 이러한 계산으로부터, 3C 샘플은 시험된 7개의 게놈 유전자좌에 걸쳐 대략 88%의 소화 효율(표 1의 평균)을 갖는 것으로 결정되었다.
다음으로, 샘플은 유전자좌 특이적 프라이머(표 2)와 qPCR의 조합을 사용하여 서로 다른 게놈 유전자좌 사이의 장거리 염색질 접촉의 존재에 대해 테스트되었습니다. 이러한 결과를 사용하여 대조군 프라이머 세트에 대한 제품 존재비를 계산하고(방정식 2) 비교를 위해 그래프로 표시했습니다(그림 4). 이 데이터는 10 개의 반응 중 8 개가 장거리 상호 작용에 대한 조건부 양성임을 나타냅니다.
이어서, PCR 반응을 아가로스 겔 상에서 실행하였다. 8개의 조건부 양성 반응과 1개의 음성 반응에 대해 예상되는 PCR 산물을 겔 정제하고 Sanger 시퀀싱을 위해 보냈습니다. 대표적인 양성(파란색 화살표, 파란색 상자) 및 음성(빨간색 화살표, 빨간색 상자) 반응에 대한 결과가 표시됩니다(그림 5).
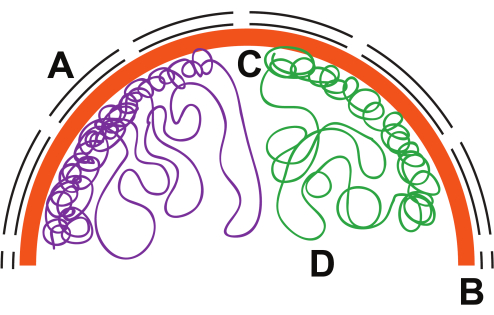
그림 1: 핵의 염색체 구조. 핵 내부의 가상의 염색체 조직. (A) 핵 외피, 검은 선; (b) 핵층, 오렌지색; (c) 헤테로크로마틴, 압축선; (D) 유크로마틴, 느슨한 루프. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
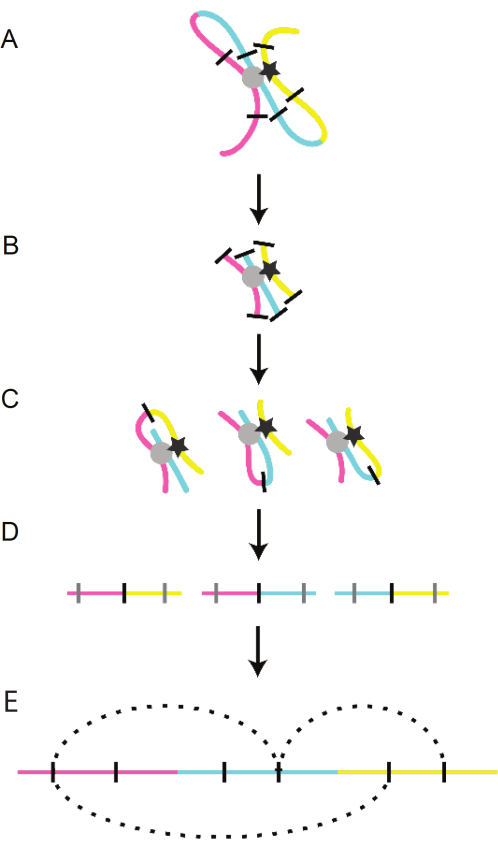
그림 2: 3C 프로토콜의 개략도. 선형 염색체의 먼 부분(파란색, 노란색, 분홍색)은 조절 루프를 통해 핵 내에서 서로 가깝게 연결됩니다. (A) 루프 구조는 전사 인자(회색 원 및 검은색 별)에 의해 매개됩니다. 이러한 상호 작용은 화학적 가교를 통해 보존됩니다. (B) 루프는 효소 소화를 통해 끊어집니다(검은색 선). (C) 멀리 있는 염색질 조각은 소화에 의해 생성된 접착 말단을 통해 함께 결찰됩니다. (D) DNA는 단백질로부터 정제된다. (E) 단편 내의 서열이 식별되고 게놈에 다시 매핑됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
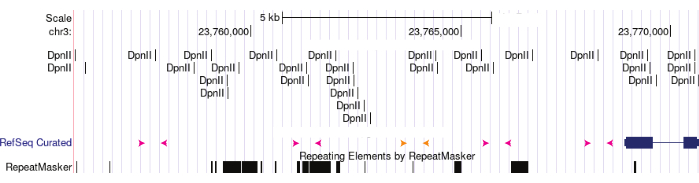
그림 3: 3C 프라이머 구성표. 3C 프라이머는 관심 게놈 위치에서 다양한 거리에 있는 제한 부위를 중심으로 설계되었습니다. 분홍색 화살표는 DpnII 부위를 둘러싼 실험용 프라이머 세트를 나타냅니다. 실험 프라이머를 혼합하고 일치시켜 해당 영역의 염색질 루핑을 평가할 수 있습니다. 주황색 프라이머는 DpnII 부위가 없는 음성 대조군을 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
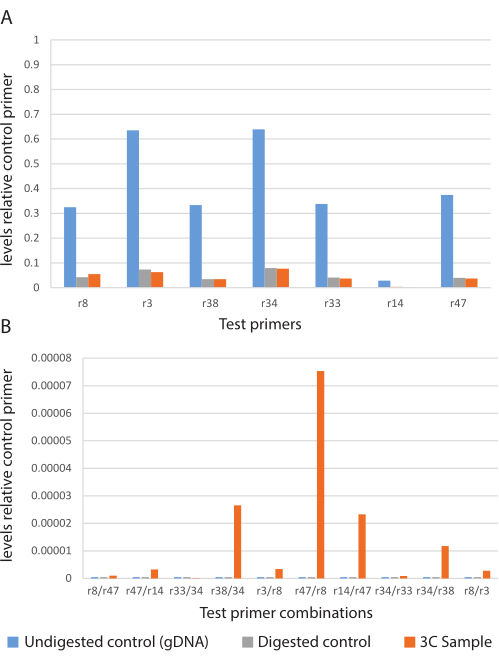
그림 4: 3C 실험에 대한 대표적인 qPCR 데이터. 3C 테스트 프라이머 세트의 상대적 풍부도. (A) 소화되지 않은 대조군(파란색), 소화된 대조군(회색) 및 3C 샘플(주황색)에서 생성물의 상대적 풍부도를 보여주는 대조군 그래프. (b) 3C 실험; 소화되지 않은 대조군(파란색), 분해된 대조군(회색) 및 3C 샘플(주황색)에서 테스트 프라이머 조합으로 인한 제품의 상대적 풍부도. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
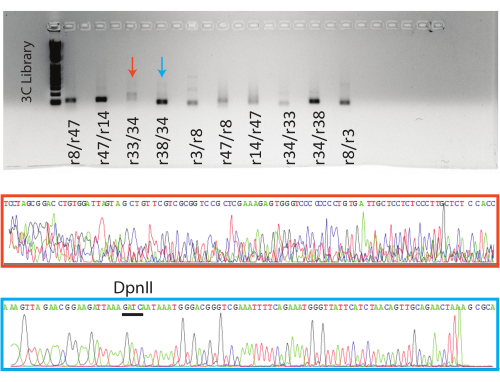
그림 5: 3C qPCR 산물의 시각화. 상단, qPCR 종말점 제품이 표시된 젤amples와 함께. 하단, 표시된 반응에 대한 대표적인 Sanger 서열 추적 파일. 주황색 샘플은 거짓 양성의 예이며( 그림 4, r38/34 참조), 파란색 샘플은 추적 위에 표시된 DpnII 부위가 있는 참 양성의 예입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| 사이트 | % 소화 |
| R8 | 86.98 |
| R3 (알3) | 88.44 |
| R38 (영어) | 89.64 |
| R34 (영어) | 87.55 |
| R33 (알33) | 87.85 |
| R14 (영어) | 86.97 |
| R47 (영어) | 89.45 |
표 1: 계산된 소화 효율.
| 이름 | 순서 | 게놈 유전자좌 | ||
| r3 FWD | ACGCAAGTAAAATTCTGGTTTTTGACC | chrX:11361475 | ||
| r3 RVS | TTTCCTGAGCTCTCTAACCATGTTTGC | chrX:11361561 | ||
| r38 FWD | TTACTTCTGAAGTAATCTTTTCTTATCCCC | chrX:5859700 | ||
| r38 RVS | AGACGAGCTGATTAAAAGTAGTTGAGAG | chrX:5859775 | ||
| r34 FWD (전륜) | ATTTGTGGATTGCGTGGAGACG | chrX:5429702 | ||
| r34 RVS | AATAATCCTCTTAACAAACGTGGCC | chrX:5429777 | ||
| r33 FWD | 아가그트그ccaaaataaattgagctaac | chrX:6296704 | ||
| r33 RVS | TTCAGAAAAGTAAACTTTGACTTGGAACG | chrX:6296807 | ||
| r14 FWD | AATTATCGATTTTTCCATCGCGCAG | chrX:8036367 | ||
| r14 RVS | ATTTCAATGAAAATGTAAAAATGTTCCTTC | chrX:8036427 | ||
| r47 FWD | ATCTAGACTTGATAATATTTGTGTGTCCTC | chrX:9464939 | ||
| r47 RVS | AAGTTCTGCAACTGTTAGATGAATAACAC | chrX:9465064 | ||
| r8 FWD | GAGAATGTTGTTCTGTAACTGAAAACTTG | chrX:11094257 | ||
| r8 RVS | TTACGAAATTTGGTAGTTTTGGACC | chrX:11094362 | ||
| 제어 프라이머 FWD | CAATCGTCTCGCTCACTTGTC | chrX:7608049 | ||
| 제어 프라이머 RVS | 갓트가그카아카그카크캉 | chrX:7608166 | ||
표 2: 대표적인 3C 실험을 위한 프라이머.
토론
3C는 기본 분자 기술에 뿌리를 둔 강력한 기술입니다. 3C를 학부생과 함께 사용할 수 있는 흥미로운 기술로 만드는 것은 이러한 기본 도구의 기초입니다. 광범위한 규모로 염색질 역학을 관찰하는 최근 연구가 너무 많기 때문에 이러한 결과를 사용하여 단일 유전자 또는 게놈 영역에 대한 좁은 초점을 맞춘 실험을 고안하면 학부 연구에서 독특하고 영향력 있는 실험을 만들 수 있는 잠재력이 있습니다. 종종 이와 같은 실험은 학부생에게 너무 진보된 것으로 간주되지만 신중한 계획을 통해 쉽게 달성할 수 있습니다. 3C 라이브러리에 의해 캡처된 염색질 연결을 조사하도록 설계된 분석은 반정량적 종말점 PCR에서 전체 게놈 시퀀싱에 이르기까지 다양할 수 있다는 점에 유의하는 것이 중요합니다. 실제로, 제1 3C 논문(16 )으로부터의 데이터는 qPCR로부터 생성되었다. 이 광범위한 분석은 모든 3C 기술이 동일한 제품, 즉 핵에서 3D 연결을 나타내는 DNA 단편 라이브러리를 생성하기 때문에 모두 사용할 수 있습니다.
여기에 제시된 것은 학부 연구원에게 더 적합한 보다 유연하고 수용적인 프로토콜의 적응입니다. 위에 나열된 일시 중지 기간은 야간 지연을 의미합니다. 그러나 이러한 일시 중지는 주말에 걸쳐 연장 될 수 있으며 세포와 핵의 경우 몇 주 동안 연장 될 수 있습니다. 가장 중요한 고려 사항은 작업이 완료되는 시기입니다. 프로토콜에는 일시 중지가 옵션이 아닌 경우 시간에 민감한 단계가 있는 경우가 많습니다. 몇 가지 지점(1일차와 2일차) 외에도 샘플을 멈추고 동결할 수 있는 곳이 많이 있습니다. 이는 실험실 작업의 일정과 타이밍이 유연해야 하는 학부생과 함께 작업할 때 매우 중요합니다. 이러한 중지를 프로토콜에 엔지니어링하는 것 외에도 학부생은 쌍 또는 3-4명의 소그룹으로 작업하는 것이 좋습니다. 학생들은 서로를 지원하고 모든 사람이 안전하게 작업할 수 있도록 친구 시스템을 만들 수 있기 때문에 그룹은 이 프로토콜에 적합합니다. 실험실 작업도 다른 사람들과 함께 하면 더 재미있습니다. 그룹을 통해 학생들은 동일한 프로토콜을 수행하면서 염색질 조직에 초점을 맞춘 다양한 질문에 대해 작업할 수도 있습니다. 따라서 학생들이 별도의 프로젝트를 진행하는 동안에도 프로토콜은 그들의 노력을 연결하고, 이로 인해 서로를 지원할 수 있습니다.
다른 적응은 특정 전문 도구와 장비가 모든 학부 기관에서 반드시 발견되는 것은 아니라는 사실을 해결하기 위한 것입니다. 이러한 장비에는 qPCR 열순환기, 겔 문서화 시스템 및 나노 부피 분광계가 포함되지만 이에 국한되지는 않습니다. 실제로 이러한 장비는 편리하지만 필수 사항은 아닙니다. 여기서, 3C의 고전적인 방법은 프라이머 디자인 부분에서도 설명됩니다. 여기에는 관심 있는 게놈 유전자좌를 식별하고 그로부터 더 멀리 떨어진 염색질 접촉점에 대한 다른 게놈 유전자좌를 평가하는 것이 포함됩니다. 이 기술은 알려진 양수(연결) 및 음수(비연결) 유전자좌가 식별되는 Hi-C를 사용하는 데이터 세트와 같이 게시된 데이터 세트를 사용하는 경우에도 잘 작동합니다. 이러한 공개된 데이터 세트를 사용하여 실험을 설계하는 것은 염색질 연결을 성공적으로 식별할 수 있는 기회가 일반적으로 더 크기 때문에 교육 실험실에 대한 또 다른 훌륭한 적응입니다. 또한 연구 논문은 수업 시간에 토론하고 참고 자료로 사용할 수 있습니다.
이 프로토콜은 수정된 qPCR 접근 방식을 사용하여 3C 생성물 형성을 시각화합니다. 제어는 3C 기술의 성공에 필수적입니다. 각 실험은 샘플 대조군과 프라이머 대조군을 모두 사용하여 3C 절차의 완료를 결정합니다. 샘플 대조군에는 소화되지 않은 대조군(게놈 DNA)과 소화된 대조군이 포함됩니다. 소화되지 않은 대조군은 프라이머 세트에 대한 기준선 신호를 결정하고, 분해 효율을 결정하기 위해 가교 분해 대조군과 함께 사용되며, 제한 부위를 가로질러 지시된 임의의 프라이머에 대해 생성물의 감소가 있을 것으로 예상된다. 이 값을 소화되지 않은 대조군과 비교하면 샘플이 얼마나 잘 소화되었는지를 알 수 있습니다.
상기 PCR용 프라이머는 대조군 프라이머 및 테스트 프라이머를 포함한다. 대조군 프라이머는 분석되는 게놈 영역 근처에 있고 제한 부위를 포함하지 않는 프라이머 세트입니다. 이는 테스트 프라이머 PCR 산물의 풍부도를 결정하기 위한 기준선을 제공합니다. 테스트 프라이머는 관심 있는 특정 게놈 유전자좌에 대한 제한 부위 옆에 있는 정방향 및 역방향 프라이머입니다(그림 3). 이러한 프라이머 세트를 사용하는 반응은 소화 효율을 결정하기 위해 비교되는데, 이는 제한 부위가 절단된 경우 제품 풍부도가 떨어져야 하기 때문이다. 염색질 조직을 결정할 때 한 유전자좌의 한 테스트 프라이머를 다른 게놈 유전자좌의 다른 테스트 프라이머와 쌍을 이루어 이 두 유전자좌가 3D 공간에서 서로 가까이 있는지 확인합니다. 이 경우 PCR 산물은 3C 샘플을 템플릿으로 사용하는 경우에만 발견될 것으로 예상됩니다.
검증된 프라이머도 실패하는 경향이 있다는 점에 유의해야 합니다(그림 4: r14 프라이머 세트). 또한, PCR 산물은 대조 반응 및 염색질 연결이 예측되지 않는 반응(예: 결찰되지 않기 때문에 분해된 대조군)에서 자주 식별됩니다. 이러한 인스턴스는 시퀀싱되어 Sanger QC에 실패하거나 정의된 시퀀스 없이 반환됩니다(그림 5). 또한 전통적인 3C 실험은 주어진 양의 DNA로 생성할 수 있는 모든 가능한 결찰 단편을 나타내는 가교되지 않은, 소화 및 결찰된 DNA 샘플인 "대조 주형"을 생성합니다. "제어 템플릿"은 두 게놈 유전자좌 사이의 qPCR 신호 강도를 비교하여 신호가 실제 상호 작용을 나타내는지 아니면 무작위 연관을 나타내는지 결정하는 데 중요한 역할을 합니다. "제어 템플릿"을 만드는 것은 분석되는 염색질의 상당 부분을 인공 염색체의 형태로 포획하고 3C 샘플과 함께 처리해야 하기 때문에 문제가 될 수 있습니다. 이러한 구조를 확보하는 것은 실현 가능하지 않을 수 있으며, 하나를 만드는 것은 학기 프로젝트의 범위를 벗어날 수 있습니다. 이러한 어려움으로 인해 대조군 프라이머를 사용하는 것이 좋습니다. 제어 입문서는 "제어 템플릿"의 모든 기능을 대체하지는 않지만 데이터를 분석하여 "존재" 또는 "부재" 결정을 내릴 수 있는 기회를 제공합니다.
qPCR을 수행할 때 동일한 양의 샘플을 사용하는 것이 중요합니다. 이는 나노드롭과 같은 나노분광광도계를 사용하더라도 알려진 농도의 게놈 DNA로부터 표준 곡선을 생성하고 3C 샘플을 해당 선에 피팅하여 결정해야 합니다. 이 양은 기록되어 후속 PCR에 사용되어야 합니다. PCR 반응의 품질 또한 중요합니다. PCR이 qPCR에서 실행됨에 따라 형광을 사용하여 제품 존재비를 측정하고 기록합니다. 이 기록은 증폭 플롯에서 액세스할 수 있습니다. 프로그램이 완료되면 증폭 플롯을 확인하고 반응(템플릿 제어 없음 제외)에 기준선, 지수 및 고원/포화 단계의 세 단계가 있는지 확인하는 것이 중요합니다. 특히 임계값을 설정하기 위해 반응에 지수 위상이 있는지 확인하는 것이 중요합니다(아래 참조). 또한 연속 희석된 샘플의 경우 샘플의 희석과 일치하는 Ct 값의 이동이 있어야 합니다(가장 높은 농도는 가장 낮은 Ct 값을 가지며 가장 낮은 농도는 가장 높은 Ct 값을 갖습니다). 증폭 플롯에서 이러한 변화를 반영하지 않는 샘플은 새로운 희석이 필요하거나 3C 샘플 형성에 더 큰 문제가 있음을 나타냅니다. 마지막으로, 표준 곡선을 생성하는 동안, PCR 소프트웨어는 PCR 효율 및R2 값을 계산할 것이다. PCR 효율은 90% 이상이어야 하고,R2 값은 0.99보다 커야 한다. 이러한 조건 중 하나라도 충족되지 않으면 샘플 또는 PCR 프라이머에 문제가 있을 수 있습니다.
qPCR 후, 소화 백분율 및 3C 상호작용의 존재는 각 반응에 대한 qPCR Ct를 사용하여 계산할 수 있습니다. 이를 결정하기 위해서는 먼저 PCR 반응에 대한 임계 값을 설정해야합니다. 이것은 일반적으로 qPCR 기계와 함께 제공되는 소프트웨어를 사용하여 수행됩니다. 임계값을 설정하면 샘플 Ct 값을 비교하는 데 사용할 PCR 산물의 농도가 정의됩니다. 임계값은 증폭의 지수 단계에서 PCR 반응의 증폭 곡선을 이등분해야 합니다. 지수 증폭이 있는 PCR 반응(이 경우 대조군 프라이머 및 테스트 프라이머 반응)만 비교할 수 있는데, 이는 반응이 동일한 속도로 DNA를 증폭하고 충실하게 비교할 수 있는 유일한 방법이기 때문입니다. 3C 그래프를 분석할 때 조건부 양성 반응은 대조군 샘플, 게놈 대조군 및 소화 대조군보다 더 많은 생성물을 가진 반응으로 식별됩니다(그림 4B). 그러나 이러한 샘플은 PCR 산물의 겔 정제 후 Sanger 염기서열 분석을 사용하여 추가로 검증해야 합니다.
Sanger 염기서열 분석 후 QC를 통과한 시료는 Blat를 사용하여 분석할 수 있습니다. 이 분석의 목표는 샘플이 제한 부위 옆에 있는 두 표적 게놈 유전자좌의 서열을 가지고 있는지 확인하는 것입니다(이 프로토콜의 경우 DpnII). 두 서열이 모두 확인되면 3C 단편이 검증된 것으로 간주될 수 있습니다. Blat의 결과가 예상된 서열을 반환하지 않는 경우, 이는 하나 또는 두 프라이머가 최적이 아님을 나타낼 수 있으며, 그 결과 위양성 qPCR 결과가 발생할 수 있습니다. 거짓 양성 샘플에 대한 추적 파일에는 정의되지 않은 기본 피크가 있으며 Seq 보고서에는 대부분 "n" 기본 호출이 포함됩니다.
Sanger 검증은 인공성 PCR 산물 형성으로 인한 위양성이 가능하기 때문에 필수적입니다. 이러한 위양성은 염기서열 분석 산물이 예상되는 표적 서열 또는 적절한 3C 단편의 DpnII 부위 특성을 가지고 있지 않을 때 식별할 수 있습니다(그림 5). PCR 단편의 시퀀싱은 또한 실험을 위한 또 다른 데이터 포인트를 제공하고 학생들에게 3C 기술이 핵 내의 3D 공간에서 함께 모이는 먼 게놈 유전자좌를 식별하고 있다는 사실을 알려줍니다.
3C 기술은 유연하고 간단한 절차로 학부생을 위한 풍부한 기초 분자 기술을 제공합니다. 이 3C 기술은 차세대 시퀀싱(NGS)을 통합하는 다른 3C 기술의 출발점이기도 합니다. 이러한 유형의 실험은 학부생을 생물 정보학의 중요한 측면에 노출시킬 수 있으며 여기에 설명 된 기본 원칙에 뿌리를두고 있습니다. 학부 경험과 참여는 젊은 과학자로서의 성공과 발전의 열쇠입니다. 이러한 기회를 제공함으로써 학부생은 기본 원리에 대한 이해를 강화하는 동시에 최첨단 기술과 질문에 대처할 수 있는 자신감을 키울 수 있습니다.
공개
저자는 이해 상충을 선언하지 않습니다.
감사의 말
이 작업은 보조금 번호 P20GM103430에 따라 국립 보건원 (National Institutes of Health)의 국립 일반 의학 연구소 (National Institute of General Medical Sciences)와 브라이언트 보건 및 행동 과학 센터 (Bryant Center of Health and Behavioral Sciences)의 로드 아일랜드 기관 개발 상 (IDeA) 생물 의학 연구 우수 네트워크에 의해 부분적으로 지원되었습니다.
자료
| Name | Company | Catalog Number | Comments |
| 37% Formaldehyde | Millapore-Sigma | F8775 | |
| 100% Ethanol | Millapore-Sigma | E7023 | |
| CaCl2 | MP Biomedical | 215350280 | |
| chloroform | Millapore-Sigma | C0549 | |
| cOmplete, EDTA-free Protease Inhibitor Cocktail | Millapore-Sigma | COEDTAF-RO | mixed to 50x in water. Diluted to 1x in Sucrose buffer and GB buffer fresh |
| Dithiothreitol (DTT) | Millapore-Sigma | D0632 | 1 M stock diluted to 500 µM in Sucrose buffer and GB buffer fresh |
| DpnII | NEB | R0543M | |
| Glycerol | Millapore-Sigma | G9012 | |
| glycine | Millapore-Sigma | G8898 | |
| glycogen | Millapore-Sigma | 10901393001 | |
| HEPES | Millapore-Sigma | H3375 | |
| KCl | Millapore-Sigma | P3911 | |
| KH2PO4 | Millapore-Sigma | P5655 | |
| methyl green pyronin | Millapore-Sigma | HT70116 | |
| MgAc2 | Thermoscientific | 1222530 | |
| Na2HPO4 | Millapore-Sigma | S5136 | |
| NaCl | Millapore-Sigma | S9888 | |
| phenol-chloroform | Millapore-Sigma | P3803 | |
| Pronase | Millapore-Sigma | 11459643001 | |
| Proteinase K | IBI Scientific | IB05406 | |
| qPCR Ready mix (Phire Taq etc) | Millapore-Sigma | KCQS07 | |
| RNase A | Millapore-Sigma | R6148 | |
| Sodium Acetate | Millapore-Sigma | S2889 | |
| sodium dodecyl sulfate (SDS) | Millapore-Sigma | L3771 | |
| Sucrose | Millapore-Sigma | S0389 | |
| T4 DNA Ligase | Promega | M1804 | |
| Tris-HCl | Millapore-Sigma | 108319 | |
| Triton X-100 | Millapore-Sigma | T9284 | |
| Trypsin-EDTA | Millapore-Sigma | T4049 |
참고문헌
- McBryant, S. J., Adams, V. H., Hansen, J. C. Chromatin architectural proteins. Chromosome Research. 14 (1), 39-51 (2006).
- Nalabothula, N., et al. The chromatin architectural proteins HMGD1 and H1 bind reciprocally and have opposite effects on chromatin structure and gene regulation. BMC Genomics. 15, 92 (2014).
- John, S., et al. Chromatin accessibility pre-determines glucocorticoid receptor binding patterns. Nature Genetics. 43 (3), 264-268 (2011).
- Fawcett, D. W. On the occurrence of a fibrous lamina on the inner aspect of the nuclear envelope in certain cells of vertebrates. The American Journal of Anatomy. 119 (1), 129-145 (1966).
- Reddy, K. L., Zullo, J. M., Bertolino, E., Singh, H. Transcriptional repression mediated by repositioning of genes to the nuclear lamina. Nature. 452 (7184), 243-247 (2008).
- Finlan, L. E., et al. Recruitment to the nuclear periphery can alter expression of genes in human cells. PLoS Genetics. 4 (3), e1000039 (2008).
- Chambeyron, S., Da Silva, N. R., Lawson, K. A., Bickmore, W. A. Nuclear re-organisation of the Hoxb complex during mouse embryonic development. Development. 132 (9), 2215-2223 (2005).
- Chambeyron, S., Bickmore, W. A. Chromatin decondensation and nuclear reorganization of the HoxB locus upon induction of transcription. Genes and Development. 18 (10), 1119-1130 (2004).
- Mahy, N. L., Perry, P. E., Gilchrist, S., Baldock, R. A., Bickmore, W. A. Spatial organization of active and inactive genes and noncoding DNA within chromosome territories. The Journal of Cell Biology. 157 (4), 579-589 (2002).
- Mahy, N. L., Perry, P. E., Bickmore, W. A. Gene density and transcription influence the localization of chromatin outside of chromosome territories detectable by FISH. The Journal of Cell Biology. 159 (5), 753-763 (2002).
- Li, B., Carey, M., Workman, J. L. The role of chromatin during transcription. Cell. 128 (4), 707-719 (2007).
- Chen, A., Chen, D., Chen, Y. Advances of DNase-seq for mapping active gene regulatory elements across the genome in animals. Gene. 667, 83-94 (2018).
- Hughes, A. L., Rando, O. J. Mechanisms underlying nucleosome positioning in vivo. Annual Review of Biophysics. 43, 41-63 (2014).
- Weiner, A., Hughes, A., Yassour, M., Rando, O. J., Friedman, N. High-resolution nucleosome mapping reveals transcription-dependent promoter packaging. Genome Research. 20 (1), 90-100 (2010).
- Hughes, A. L., Jin, Y., Rando, O. J., Struhl, K. A functional evolutionary approach to identify determinants of nucleosome positioning: A unifying model for establishing the genome-wide pattern. Molecular Cell. 48 (1), 5-15 (2012).
- Dekker, J., Rippe, K., Dekker, M., Kleckner, N. Capturing chromosome conformation. Science. 295 (5558), 1306-1311 (2002).
- van Berkum, N. L., Dekker, J. Determining spatial chromatin organization of large genomic regions using 5C technology. Methods in Molecular Biology. 567, 189-213 (2009).
- Smith, E. M., Lajoie, B. R., Jain, G., Dekker, J. Invariant TAD boundaries constrain cell-type-specific looping interactions between promoters and distal elements around the CFTR locus. American Journal of Human Genetics. 98 (1), 185-201 (2016).
- Crane, E. E. Two inputs into C. elegans dosage compensation: Chromosome conformation and the miRNA-specific argonaute ALG-2. University of California, Berkeley. , (2012).
- Bell, A. C., West, A. G., Felsenfeld, G. The protein CTCF is required for the enhancer blocking activity of vertebrate insulators. Cell. 98 (3), 387-396 (1999).
- Cuadrado, A., et al. Specific contributions of cohesin-SA1 and cohesin-SA2 to TADs and polycomb domains in embryonic stem cells. Cell Reports. 27 (12), 3500-3510 (2019).
- Sarro, R., et al. Disrupting the three-dimensional regulatory topology of the Pitx1 locus results in overtly normal development. Development. 145 (7), (2018).
- Tolhuis, B., et al. Interactions among polycomb domains are guided by chromosome architecture. PLoS Genetics. 7 (3), e1001343 (2011).
- Splinter, E., de Wit, E., van de Werken, H. J. G., Klous, P., de Laat, W. Determining long-range chromatin interactions for selected genomic sites using 4C-seq technology: From fixation to computation. Methods. 58 (3), 221-230 (2012).
- Fernández-Miñán, A., Bessa, J., Tena, J. J., Gómez-Skarmeta, J. L. Chapter 21 - Assay for transposase-accessible chromatin and circularized chromosome conformation capture, two methods to explore the regulatory landscapes of genes in zebrafish. Methods in Cell Biology. 135, 413-430 (2016).
- Dekker, J. The three "C" s of chromosome conformation capture: controls, controls, controls. Nature Methods. 3 (1), 17-21 (2006).
- Hagège, H., et al. Quantitative analysis of chromosome conformation capture assays (3C-qPCR). Nature Protocols. 2 (7), 1722-1733 (2007).
- Lajoie, B. R., van Berkum, N. L., Sanyal, A., Dekker, J. My5C: Webtools for chromosome conformation capture studies. Nature Methods. 6 (10), 690-691 (2009).
- Han, M., Wei, G., McManus, C. E., Hillier, L. W., Reinke, V. Isolated C. elegans germ nuclei exhibit distinct genomic profiles of histone modification and gene expression. BMC Genomics. 20 (1), 500 (2019).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유