Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Magnetic Resonance Spectroscopy von Live- Drosophila melanogaster Verwendung von Magic Angle Spinning
In diesem Artikel
Zusammenfassung
Diese Technik ermöglicht den Einsatz von hochauflösenden magic angle spinning Protonen MR-Spektroskopie (HR-MAS-1H-MRS) für die molekulare Charakterisierung von Live- Drosophila melanogaster Mit einem herkömmlichen 14,1-Tesla-Spektrometer mit einer HR-MAS-Sonde ausgestattet.
Zusammenfassung
High-Resolution Magic Angle Spinning (HR-MAS-) Protonen-Magnetresonanz-Spektroskopie (
Protokoll
Teil 1: Vorbereiten Drosophila für HR-MAS-Messungen
- Verwendung von Standard flylab Verfahren 1, sammeln neu schlüpfende Fliegen für 3 Tage und sie auf Flaschen mit frischen Lebensmitteln zu fliegen fliegen. Inkubieren Sie die gesammelten Fliegen für 5 Tage, so dass die Fliegen wird 5-8 Tage alt just vor dem Experiment. Nur ein Geschlecht (meist Männer) ist für Experimente verwendet.
- Verwenden Sie gesunden, fliegt intakten Wildtyp mit behandelten zB traumatisierte oder genetisch veränderte fliegt 1 zu vergleichen.
- Legen Sie einzelne fliegt in 2ml Röhrchen mit einem Stück (~ 0,2 ml) von essen fliegen 24 Stunden vor dem Experiment. Diese Rohre tragen ein Loch in das Rohr Tasse erfolgt über eine Flamme erwärmt Insulin Nadel.
- Wiegen Sie die Rohre unmittelbar vor und nach der Fliege Einsetzen mit einer hohen Präzision Balance definieren und jeder fliegen s Gewicht durch Subtraktion der erste Wert aus der zweiten. Ein einzelnes Männchen fliegen wiegt in der Regel 0,7-1 mg.
Teil 2: HR-MAS-Rotor Vorbereitung.
- Legen Sie eine einzelne Röhre auf Eis weniger als einer Minute auf die Fliege im Inneren zu betäuben.
- Lay the fly auf einer Schicht aus Aluminiumfolie auf Eis gelegt, und drücken Sie die Fliege sanft in die halbkugelförmigen Hohlraum des NMR Rotoreinsatz mit einer weichen Bürste in Anspruch zu fliegen Einstecken zu gewährleisten bei gleichzeitiger Vermeidung fliegen Verletzungen.
- Platzieren Sie den Einsatz in der Zirkonoxid (ZrO 2) Rotor Rohr (4 mm Durchmesser, 50 ul), um die Fliegen zwischen den Rotor einlegen und den Boden des Rotors (siehe Abbildung 1) zu finden.
- Schließen Sie den Einsatz mit einer Schraube und bedecken Sie es mit Parafilm (siehe Abbildung 1), um den Kontakt zwischen dem Fliegen und der TSP-Standardlösung (TSP zu verhindern: trimethylsilyl-Propionsäure-2 ,2,3,3-d4-säure, Mw = 172, δ = 0,00 ppm, 50 mM in deuteriertem Wasser-D 2 O, diese Funktion als Referenz für beide Resonanz chemische Verschiebung und Quantifizierung).
- Add 8 ul TSP-Standardlösung auf der Oberseite des Rotors einzufügen. (Siehe Abbildung 1).
- Sichere und ziehen das ganze bis in die Rotoren mit einer Kappe (siehe Abbildung 1).
Teil 3: HR-MAS-Datenerfassung
- Einführung des Rotors in die HR-MAS-Sonde und positionieren Sie es im magischen Winkel 54,7 ° (siehe Abbildung 1).
- Stellen Sie die Temperatur um 4 ° C durch einen BTO-2000-Einheit in Kombination mit dem MAS Pneumatikeinheit. Fliegen sind bei 4 ° C, während in das Spektrometer Anästhesie beizubehalten gehalten.
- Stellen Sie die HR-MAS-1 H MRS Spinngeschwindigkeit bei 2 kHz MAS.
- Stabilisieren Sie die MAS Drehfrequenz bei 2,0 ± 0,001 kHz mit einem MAS Drehzahlregler (Drehfrequenz).
- Für Magnet Zubereitung: Melodie und passen die Spule für optimale Leistung und Shim der Magnet für die optimale Qualität der Spektren.
- Erwerben Sie eindimensionalen 1 H-Spektren mit Rotor synchronisiert Carr-Purcell-Meiboom-Gill (CPMG) Spin-Echo-Pulssequenz 2, [90 ° - (τ-180 ° - τ) n-Übernahme], die als T 2 Filter werden Entfernen Sie die spektrale Verbreiterung 1 H-NMR-Spektren single-fly zu erwerbenden eindimensionale Daten zu allen Proben. Synchronisieren Sie die inter-Puls-Verzögerung (τ = 500μs), um die MAS Drehfrequenz (2 kHz). Legen Sie die Anzahl von Transienten bei 256 mit 32.768 (32k) Datenpunkten. Acquisition Zeit 9 min.
- Erwerben Sie zweidimensionale (2D) 1 H-1 H HR-MAS-NMR-Spektren single-fly bei allen Proben mit einem TOBSY Sequenz mit adiabatischer Impulse 3. Acquisition Parameter sind: 2k Datenpunkte direkten Dimension (11 ppm spektrale Breite), 1 s Wasser pre-Sättigung während des Relaxationsdelay, 8 Scans pro Inkrement, 128 Stufen, 2 s insgesamt Wiederholzeit, 45 ms Mischzeit und insgesamt Übernahme Zeit von 29 min.
Teil 4: Data Processing / Analyse
- Analysieren Sie die MRspectra von Proben mit MestReC Software (Mestrelab Research, www.mestrec.com)
- Verwenden Sie eine Linienverbreiterung Apodizierungsfunktion von 0,5 Hz und gelten für alle HR-MAS-1 H FIDs vor der Fourier-Transformation.
- Verweisen Sie auf die MR-Spektren in Bezug auf δ = 0,0 ppm (externer Standard) TSP.
- Phase-Spektren manuell und wenden Sie einen Whittaker Baseline Schätzer der breiten Komponenten der Ausgangswert vor den Peakfläche Berechnungen subtrahieren.
- Estimate Peakflächen mit MestReC Software. Scale-Peakhöhen in Bezug auf für jede erworbene Spektrum (TSP peak height = 1) TSP. Verwenden Sie t-Tests (zweiseitig, p <0,05), um innerhalb der Gruppe, zwischen Gruppen-Kennzahlen zu vergleichen.
- 2D TOBSY Prozessparameter sind: QSINE = 2 Fenster-Funktion in beiden Dimensionen, FT mit 2k Punkte in der direkten Dimension und Null-Füllung auf 1k in der zweiten Dimension, Phasenkorrektur in beiden Dimensionen und Baseline-Korrektur in der zweiten Dimension.
- Produzieren Sie das 2D-Spektren mit Sparky Programm (TD Goddard und DG Kneller, SPARKY 3, USCF, http://www.cgl.ucsf.edu/home / sparky /)
Teil 5: Repräsentative Spectra von Live Drosophila melanogaster Flies
Die oben beschriebenen Verfahren erlauben eine reproduzierbare Spektren von lebenden Drosophila melanogaster Fliegen zu sammeln. Abbildungen 2 und 3 zeigen repräsentative MR-Spektren in Wildtyp-(wt) Oregon-R erworben Fliegen. Abbildung 2 zeigt 1D 1 H HR-MAS-Spektren CPMG. Auftraggeber Lipidkomponenten [C H 3 (0,89 ppm), (C H 2) n (1,33 ppm), C H 2 C-CO (1.58ppm), C H 2 C = C (2,02 ppm), C H 2 C = O (2,24 ppm), C H = C H (5,33 ppm)], Glycerin (1,3-C H 4,10 ppm und 4,30 ppm; 2-CH 2 5,24 ppm), und kleine Metaboliten: β-Alanin (β-Ala wurden 2,57 ppm), Acetat (Ac, 1,97 ppm), phosphocholin (PC, 3,22 ppm) und phophoetanolamine (PE, 3.23ppm) erkannt und zugeordnet in Übereinstimmung mit früheren Berichten 4, 5. Signale bei 2,02 ppm wurden Methylenprotonen der C H2-CH = CH-Einheit von einfach ungesättigten Fettsäuren (zB Palmitoleinsäure) zugeordnet. Die ungesättigten Säuren wurden durch ein Signal am 5,33 ppm durch Protonen der-CH = CH-Einheit produziert identifiziert. Kleine Metaboliten, die nicht zugeordnet werden konnten oder nicht sichtbar waren mit dem 1D-Spektrum wurden unter Verwendung von 2D-1 H-1 H TOBSY HR-MAS (siehe Abbildung 3).

Abbildung 1. Versuchsaufbau aus in vivo HR-MAS-1H MRS für die Untersuchung von lebenden Drosophila mit 14,1 T. Externer Standard trimethylsilyl-Propionsäure-2 ,2,3,3-d4 Säure in deuteriertem Wasser (TSP / D 2 O). Auf dem Platz: Rotorlage um den magischen Winkel in HR-MAS-Sonde.
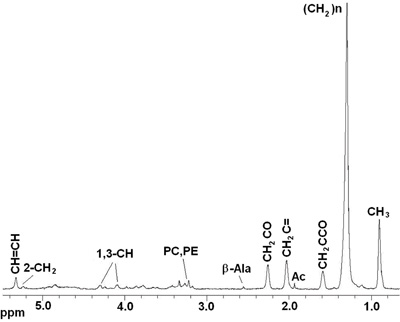
Abbildung 2. In-vivo-1D HR-MAS-1 H CPMG Spektren von einer Live-Drosophila melanogaster wt fliegen. Lipid-Komponenten: C H 3 (0,89 ppm), (C H 2) n (1,33 ppm), C H 2 C-CO (1.58ppm), Acetat (Ac), C H 2 C = C (2,02 ppm), C H 2 C = O (2,24 ppm), β-Alanin (β-Ala), phosphocholin (PC) und phophoetanolamine (PE), Glycerin (1,3-C H 4,10, 4,30 ppm; 2-C H 2 5,22 ppm) , C H = C H (5,33 ppm).
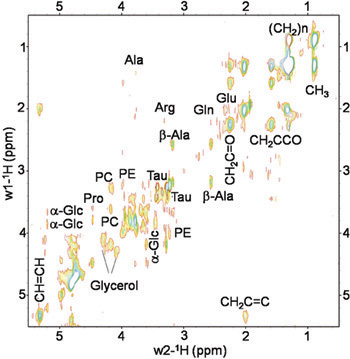
Abbildung 3. In vivo 2D-1H-1H TOBSY HR-MAS-Spektrum von einer Live-Drosophila melanogaster wt fliegen mit 14,1 T. Klein Metaboliten und Lipid-Komponenten wurden identifiziert. Metaboliten: Alanin (Ala), β-Alanin (β-Ala), Arginin (Arg), Glutamin (Gln), Glutamat (Glu), PC phosphocholin (PC), phophoetanolamine (PE), Taurin (Tau), α-Glucose (α-Glc) und Glycerin. Lipids Komponenten: C H 3 (0,89 ppm), (C H 2) n (1,33 ppm), C H 2 C-CO (1,58 ppm), C H 2 C = C (2,02 ppm), C H 2 C = O (2,24 ppm), C H = C H (5,33 ppm).
Access restricted. Please log in or start a trial to view this content.
Diskussion
Mit Ausnahme des jüngsten Berichts über die Durchführbarkeit der in-vivo-MRT in Fruchtfliegen 6 haben in vivo MRS-Studien in Drosophila noch nicht berichtet worden. In der vorliegenden Protokoll, beschreiben wir die Implementierung eines neuartigen In-vivo-HR-MAS-1 H-MRS-Ansatz zum Nachweis von biologisch wichtigen Molekülen. Insbesondere wiesen wir Lipiden und kleinen Metaboliten in lebenden Drosophila-Fliegen mit 14,1 T in etwa 45 min, die eine ang...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Danksagungen
Diese Arbeit wurde teilweise durch einen National Institutes of Health (NIH) gewähren AI063433 zu Laurence G. Rahme, ein National Institute Institutes of Health (NIH) Center Grant (P50GM021700) Ronald G. Tompkins (A. Aria Tzika, Direktor der unterstützten der NMR-Kern) und eine Shriner Hospital for Children Forschungsstipendium (# 8893) an A. Aria Tzika. Wir danken Dionyssios Mintzopoulos Ph.D. für die Unterstützung in den ersten Phasen der Entwicklung dieses Protokolls und Ovidiu C. Andronesi Ph.D. für die Unterstützung bei der TOBSY Pulssequenz.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments | |
| Deuterium oxide | Reagent | Sigma-Aldrich | 7789-20-0 | |
| 3-(trimethylsilyl)propionic-2,2,3,3-d4 acid | Reagent | Sigma-Aldrich | 24493-21-8 | |
| agar, sucrose, yeast, cornmeal | Food | Genesee Scientific | http://www.flystuff.com/ | |
| Oregon RS or Canton-S flies | Adult fly lines | Bloomington Stock center | http://flystocks.bio.indiana.edu/ | |
| Paintbrush | Equipment | (size 0) | ||
| 2ml tubes | Equipment | Fisher Scientific | K749521-1590 | |
| Fly incubators | Equipment | high humidity capacity (60-75%), adjustable temperature, and a 12 h:12 h light: dark cycle. | ||
| Bruker Bio-Spin Avance NMR spectrometer (600.13 MHz) 4mm triple resonance (1H, 13C, 2H) HRMAS probe | Equipment | Bruker Corporation | ||
| BTO-2000 unit in combination with a MAS pneumatic unit | Equipment | Bruker Corporation | ||
| 4mm zirconium oxide rotor (capacity 50 ul) | Equipment | Bruker Corporation | B3829 (Bruker store) | |
| MestReC (Mestrelab Research) | Software | 1D NMR spectra analysis http://mestrelab.com/ | ||
| SPARKY 3, USCF | Software | 2D NMR spectraanalysis http://www.cgl.ucsf.edu/home/sparky/ |
Referenzen
- Apidianakis, Y., Rahme, L. G. Drosophila melanogaster as a model host for studying Pseudomonas aeruginosa infection. Nat Protoc. 4, 1285-1294 (2009).
- Meiboom, S., Gill, D. Modified spin-echo method for measuring nuclear relaxation time. Rev Sci Instrum. 29, 688-691 (1958).
- Andronesi, O. C., Mintzopoulos, D., Struppe, J., Black, P. M., Tzika, A. A. Solid-state NMR adiabatic TOBSY sequences provide enhanced sensitivity for multidimensional high-resolution magic-angle-spinning 1H MR spectroscopy. J Magn Reson. 193, 251-258 (2008).
- Astrakas, L. G. Proton NMR spectroscopy shows lipids accumulate in skeletal muscle in response to burn trauma-induced apoptosis. Faseb J. 19, 1431-1440 (2005).
- Fan, T. W. M. Metabolite profiling by one- and two dimensional NMR analysis of complex mixtures. Prog Nuc Magn Reson Spec. 28, 161-219 (1996).
- Null, B., Liu, C. W., Hedehus, M., Conolly, S., Davis, R. W. High-resolution, in vivo magnetic resonance imaging of Drosophila at 18.8 Tesla. PLoS One. 3, e2817-e2817 (2008).
- Zektzer, A. S. Improved signal to noise in high-resolution magic angle spinning total correlation spectroscopy studies of prostate tissues using rotor-synchronized adiabatic pulses. Magn Reson Med. 53, 41-48 (2005).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten