Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Spectroscopie par résonance magnétique du direct Drosophila melanogaster En utilisant Magic Angle Spinning
Dans cet article
Résumé
Cette technique permet l'utilisation de l'angle magique à haute résolution filer spectroscopie par résonance magnétique protonique (HRMAS 1H-MRS) pour la caractérisation moléculaire de vivre Drosophila melanogaster Avec un spectromètre de 14,1 teslas conventionnelle équipée d'une sonde HRMAS.
Résumé
High-Resolution Magic Angle Spinning (HRMAS) protons spectroscopie par résonance magnétique (
Protocole
Partie 1: Préparation de la drosophile pour les mesures de HRMAS
- Utilisation de la norme flylab procédures 1, collecter nouvellement eclosing mouches pendant 3 jours et de les transférer à voler la nourriture volée flacons contenant frais. Incuber les mouches recueillies pendant 5 jours afin que les mouches deviennent 5-8 jours anciens, juste avant l'expérience. Un seul sexe (généralement des hommes) est utilisé pour des expériences.
- Utilisez saine, intacte mouches de type sauvage à comparer avec traitées par exemple traumatisés ou mouches génétiquement modifiées 1.
- Placez mouches simples dans des tubes 2ml contenant un morceau (~ 0,2 ml) de voler la nourriture 24 heures avant l'expérience. Ces tubes portent un trou dans la coupe tube en utilisant une aiguille d'insuline flamme chauffé.
- Peser les tubes immédiatement avant et après l'insertion volée en utilisant une balance de haute précision et à définir le poids de chaque volée s en soustrayant la première valeur de ce dernier. Une seule mouche mâle pèse généralement de 0,7 à 1 mg.
Partie 2: Préparation du rotor HRMAS.
- Placez un seul tube sur la glace pendant moins d'une minute pour anesthésier l'intérieur voler.
- Lay à la volée sur une couche de papier d'aluminium placé sur la glace et pousser à la volée en douceur dans l'espace semi-sphérique creux de l'insert du rotor RMN en utilisant une brosse douce pour assurer une insertion complète volée tout en évitant les blessures voler.
- Placer l'insert dans l'oxyde de zirconium (ZrO2) tube de rotor (4 mm de diamètre, 50 pi) pour localiser les mouches entre l'insert rotor et le fond du rotor (voir figure 1).
- Fermer l'insert avec une vis et le couvrir avec du parafilm (voir figure 1) pour empêcher le contact entre la mouche et la solution de TSP standard (TSP: triméthylsilyl-propionique-2 ,2,3,3-d4 acide, Mw = 172, δ = 0,00 ppm, 50 mM dans l'eau deutérée-D 2 O, qui fonctionnent comme une référence tant pour le déplacement chimique de résonance et de quantification).
- Ajouter 8 pl de TSP solution standard sur le dessus de l'insert rotor. (Voir figure 1).
- Sécurisé et serrer le tout mis en place dans les rotors avec un plafond haut (voir figure 1).
Partie 3: Acquisition de données HRMAS
- Introduire le rotor dans la sonde HRMAS et le positionner à l'angle magique 54,7 ° (voir figure 1).
- Réglez la température à 4 ° C par une unité de BTO-2000 en combinaison avec l'unité pneumatique MAS. Les mouches sont conservés à 4 ° C alors que dans le spectromètre pour maintenir l'anesthésie.
- Réglez le HRMAS 1 H MRS vitesse de rotation à 2 kHz MAS.
- Stabiliser la fréquence de rotation MAS à 2,0 ± 0,001 kHz par un régulateur de vitesse MAS (fréquence de rotation).
- Pour la préparation de l'aimant: hauteur et faire correspondre la bobine pour des performances optimales et la cale de l'aimant pour une qualité optimale des spectres.
- Acquérir une spectres unidimensionnels H en utilisant du rotor synchronisée Carr-Purcell-Meiboom-Gill (CPMG) en écho de spin séquence d'impulsions 2, [90 ° - (τ-180 ° - τ) n-acquisition], qui fonctionne comme un filtre pour T 2 retirez l'élargissement spectral RMN 1H seule volée spectres acquis une dimension de données sur tous les échantillons. Synchroniser le retard entre les impulsions (τ = 500μs) à la fréquence de rotation MAS (2 kHz). Réglez le nombre de transitoires à 256 avec 32 768 (32k) points de données. Le temps d'acquisition 9 min.
- Acquérir en deux dimensions (2D) 1 H-1 H RMN HRMAS seule volée spectres sur tous les échantillons en utilisant une séquence d'impulsions adiabatiques avec TOBSY 3. Les paramètres d'acquisition sont les suivants: la dimension 2k points de données directes (11 ppm largeur spectrale), 1 s d'eau pré-saturation pendant le délai de relaxation, 8 balayages par incrément de 128 incréments, le temps de 2 s répétition total, 45 ms temps de mélange, et un total d'acquisition temps de 29 min.
Partie 4: Traitement des données / Analyse
- Analyser les MRspectra de spécimens à l'aide du logiciel MestReC (Mestrelab recherche, www.mestrec.com)
- Utilisez une ligne d'élargissement de la fonction d'apodisation de 0,5 Hz et s'appliquent à tous une HRMAS FID H avant la transformation de Fourier.
- Référence des spectres MR à l'égard de TSP à δ = 0,0 ppm (standard externe).
- Phase spectres manuellement et appliquer un estimateur de base Whittaker pour soustraire les composantes de l'éventail de base des calculs de superficie avant de pointe.
- Estimation des domaines de pointe utilisant des logiciels MestReC. Échelle hauteurs de pics par rapport au TSP pour chaque spectre acquis (TSP pic de hauteur = 1). Utilisez les tests t (bilatéral, p <0,05) pour comparer l'intérieur des groupes d'entre-groupes de ratios.
- Paramètres 2D processus TOBSY sont: Qsine = fonction fenêtre 2 dans les deux dimensions, avec des points de FT 2k dans la dimension directe et zéro de remplissage pour 1k dans la deuxième dimension, la correction de phase dans les deux dimensions et la correction de base dans la deuxième dimension.
- Produire les spectres 2D à l'aide du programme Sparky (TD Goddard et DG Kneller, Sparky 3, USCF, http://www.cgl.ucsf.edu/Accueil / sparky /)
Partie 5: spectres représentatifs de Live mouches Drosophila melanogaster
Les procédures décrites ci-dessus permettent de collecter des spectres reproductibles à partir de mouches vivent Drosophila melanogaster. Les figures 2 et 3 montrent représentant de M. spectres acquis au type sauvage (WT) Oregon-R mouches. La figure 2 présente 1D 1 H HRMAS CPMG spectres. Principaux composants lipidiques [C 3 H (0,89 ppm), (C H 2) n (ppm 1,33), C H 2 C-CO (1.58ppm), C H 2 C = C (2,02 ppm), C H 2 C = O (2,24 ppm), C = C H H (5,33 ppm)], du glycérol (1,3-C H 4,10 ppm et 4,30 ppm; 2-CH 2 5,24 ppm), et des métabolites de petite taille: β-alanine (β-Ala , 2,57 ppm), acétate (Ac, 1,97 ppm), phosphocholine (PC, 3,22 ppm) et phophoetanolamine (PE, 3.23ppm) ont été détectées et attribuées conformément aux rapports antérieurs 4, 5. Signaux à 2,02 ppm ont été assignés à protons du méthylène du C H2-CH = CH fraction de mono-acides gras insaturés (ie palmitoléique). Les acides insaturés ont été identifiés par un signal à 5,33 ppm produites par les protons de l'-CH = CH-fraction. Métabolites petits qui ne pouvaient pas être cédés ou n'étaient pas visibles en utilisant le spectre 1D ont été détectés en utilisant 2D 1 H-1 H TOBSY HRMAS (voir figure 3).

Figure 1. Expérimental mis en place in vivo HRMAS 1H SRM pour l'enquête sur la drosophile vivons à 14,1 T. externe standard de triméthylsilyl-2-propionique ,2,3,3-d4 acide dans l'eau deutérée (TSP / D 2 O). Dans le carré: la position du rotor à l'angle magique en HRMAS sonde.
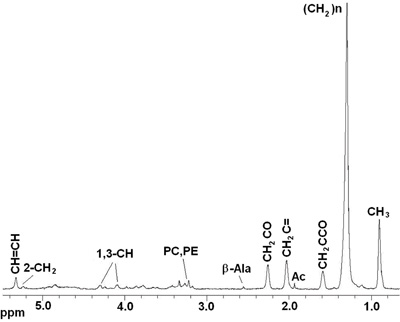
Figure 2. En 1D HRMAS spectres 1 H CPMG vivo d'une mouche Drosophila melanogaster vivre en poids. Composants lipidiques: C H 3 (0,89 ppm), (C H 2) n (ppm 1,33), C H 2 C-CO (1.58ppm), acétate (Ac), C H 2 C = C (2,02 ppm), C H 2 C = O (2,24 ppm), β-alanine (β-Ala), phosphocholine (PC) et phophoetanolamine (PE), Glycérol (1,3-C H 4,10, 4,30 ppm; 2-C H 2 5,22 ppm) , C = C H H (5,33 ppm).
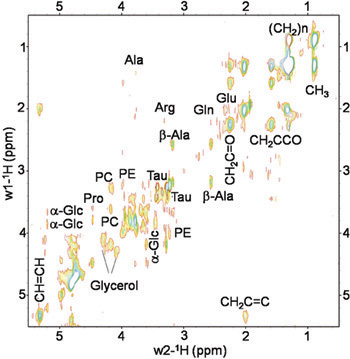
Figure 3. In vivo 2D 1H-1H TOBSY HRMAS spectre d'une mouche Drosophila melanogaster direct au poids métabolites T. 14.1 Petites et composants lipidiques ont été identifiés. Métabolites: alanine (Ala), β-alanine (β-Ala), l'arginine (Arg), de la glutamine (Gin), le glutamate (Glu), phosphocholine PC (PC), phophoetanolamine (PE), taurine (Tau), α-glucose (α-Glc) et du glycérol. Composants Lipides: C 3 H (0,89 ppm), (C H 2) n (ppm 1,33), C H 2 C-CO (1,58 ppm), C H 2 C = C (2,02 ppm), C H 2 C = O (2,24 ppm), C = C H H (5,33 ppm).
Access restricted. Please log in or start a trial to view this content.
Discussion
À l'exception du récent rapport de la faisabilité de l'IRM in vivo dans les mouches des fruits 6, études in vivo chez la drosophile MRS n'ont pas encore été signalés. Dans le présent protocole, nous décrivons la mise en œuvre d'un roman in vivo HRMAS 1 H-MRS approche pour la détection de molécules biologiquement importantes. Plus précisément, nous avons détecté des lipides et des métabolites de petites mouches drosoph...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Remerciements
Ce travail a été soutenu en partie par un National Institutes of Health (NIH) aux subventions AI063433 Laurence G. Rahme, un des Instituts Institut national de la santé (NIH) Centre Grant (P50GM021700) à Ronald G. Tompkins (A. Aria Tzika, directeur de le cœur RMN), et l'Hôpital des Shriners pour enfants de subvention de recherche (# 8893) à A. Aria Tzika. Nous remercions Dionyssios Mintzopoulos Ph.D. pour l'assistance dans les phases initiales du développement de ce protocole et Ovidiu C. Andronesi Ph.D. pour l'assistance à la séquence d'impulsions TOBSY.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments | |
| Deuterium oxide | Reagent | Sigma-Aldrich | 7789-20-0 | |
| 3-(trimethylsilyl)propionic-2,2,3,3-d4 acid | Reagent | Sigma-Aldrich | 24493-21-8 | |
| agar, sucrose, yeast, cornmeal | Food | Genesee Scientific | http://www.flystuff.com/ | |
| Oregon RS or Canton-S flies | Adult fly lines | Bloomington Stock center | http://flystocks.bio.indiana.edu/ | |
| Paintbrush | Equipment | (size 0) | ||
| 2ml tubes | Equipment | Fisher Scientific | K749521-1590 | |
| Fly incubators | Equipment | high humidity capacity (60-75%), adjustable temperature, and a 12 h:12 h light: dark cycle. | ||
| Bruker Bio-Spin Avance NMR spectrometer (600.13 MHz) 4mm triple resonance (1H, 13C, 2H) HRMAS probe | Equipment | Bruker Corporation | ||
| BTO-2000 unit in combination with a MAS pneumatic unit | Equipment | Bruker Corporation | ||
| 4mm zirconium oxide rotor (capacity 50 ul) | Equipment | Bruker Corporation | B3829 (Bruker store) | |
| MestReC (Mestrelab Research) | Software | 1D NMR spectra analysis http://mestrelab.com/ | ||
| SPARKY 3, USCF | Software | 2D NMR spectraanalysis http://www.cgl.ucsf.edu/home/sparky/ |
Références
- Apidianakis, Y., Rahme, L. G. Drosophila melanogaster as a model host for studying Pseudomonas aeruginosa infection. Nat Protoc. 4, 1285-1294 (2009).
- Meiboom, S., Gill, D. Modified spin-echo method for measuring nuclear relaxation time. Rev Sci Instrum. 29, 688-691 (1958).
- Andronesi, O. C., Mintzopoulos, D., Struppe, J., Black, P. M., Tzika, A. A. Solid-state NMR adiabatic TOBSY sequences provide enhanced sensitivity for multidimensional high-resolution magic-angle-spinning 1H MR spectroscopy. J Magn Reson. 193, 251-258 (2008).
- Astrakas, L. G. Proton NMR spectroscopy shows lipids accumulate in skeletal muscle in response to burn trauma-induced apoptosis. Faseb J. 19, 1431-1440 (2005).
- Fan, T. W. M. Metabolite profiling by one- and two dimensional NMR analysis of complex mixtures. Prog Nuc Magn Reson Spec. 28, 161-219 (1996).
- Null, B., Liu, C. W., Hedehus, M., Conolly, S., Davis, R. W. High-resolution, in vivo magnetic resonance imaging of Drosophila at 18.8 Tesla. PLoS One. 3, e2817-e2817 (2008).
- Zektzer, A. S. Improved signal to noise in high-resolution magic angle spinning total correlation spectroscopy studies of prostate tissues using rotor-synchronized adiabatic pulses. Magn Reson Med. 53, 41-48 (2005).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon