Method Article
Bestimmung von Molekülstrukturen von HIV Hüllglycoproteine mit Cryo-Elektronen-Tomographie und automatisierte Sub-Tomogramm Averaging
In diesem Artikel
Zusammenfassung
Das Protokoll beschreibt eine High-Throughput-Ansatz zur Bestimmung Strukturen von Membranproteinen mittels Kryo-Elektronen-Tomographie und 3D-Bildverarbeitung. Es deckt die Einzelheiten der Probenvorbereitung, Datenerfassung, Datenverarbeitung und-interpretation, und schließt mit der Produktion von einer repräsentativen Ziel für den Ansatz, die HIV-1 Envelope Glykoproteins. Diese Berechnungsverfahren sind in einer Weise, Forscher und Studenten ermöglicht, remote arbeiten und einen Beitrag zur Datenverarbeitung und Statik ausgelegt.
Zusammenfassung
Seit ihrer Entdeckung vor fast 30 Jahren haben mehr als 60 Millionen Menschen mit dem humanen Immundefizienz-Virus (HIV) infiziert worden (www.usaid.gov) . Das Virus infiziert und zerstört CD4 + T-Zellen so lähmend das Immunsystem und verursacht eine erworbene Immunschwäche-Syndrom (AIDS) 2. Die Infektion beginnt, wenn die HIV Envelope Glykoprotein "spike" Kontakt mit dem CD4-Rezeptor auf der Oberfläche der CD4 + T-Zellen. Diese Interaktion induziert eine Konformationsänderung in die Spitze, die Interaktion fördert mit einem zweiten Zelloberflächen-Co-Rezeptor 5,9. Die Bedeutung dieser Protein-Wechselwirkungen in der HIV-Infektion Weg macht sie von grundlegender Bedeutung in grundlegender HIV-Forschung und in der Verfolgung eines HIV-Impfstoffs.
Die Notwendigkeit zum besseren Verständnis der molekularen Maßstab Wechselwirkungen von HIV Zell-Kontakt und Neutralisation motiviert die Entwicklung einer Technik zur Bestimmung derStrukturen des HIV spike Interaktion mit Zelloberflächenrezeptor Proteine und Moleküle, die Block-Infektion. Mittels Kryo-Elektronen-Tomographie und 3D-Bildverarbeitung, haben wir vor kurzem gezeigt, die Fähigkeit, solche Strukturen auf der Oberfläche von nativen Virus, bei ~ 20 Å Auflösung 9,14 zu bestimmen. Dieser Ansatz ist nicht zu lösen HIV Envelope Strukturen beschränkt und kann zu anderen viralen Membranproteinen und Proteinen rekonstituiert auf einer Liposomen erweitert werden. In diesem Protokoll beschreiben wir, wie die Strukturen des HIV-Envelope-Glykoproteine ab gereinigt HIV-Virionen und Verfahren schrittweise durch die Vorbereitung verglaste Proben, Sammeln, Kryo-Elektronenmikroskopie Daten, Wiederherstellung und Bearbeitung von 3D-Datenvolumen, Mittelung und Klassifizierung von 3D-Protein Untervolumina zu erhalten, und Interpretation der Ergebnisse an ein Protein-Modell zu erzeugen. Die rechnerische Aspekte unseres Ansatzes wurden in Module, die abgerufen und remote über das Biowulf GNU / Linux parallel p ausgeführt werden angepasstrocessing Cluster an der NIH (http://biowulf.nih.gov) . Dieser Remote-Zugriff, mit Low-Cost-Computer Hard-und High-Speed-Netzwerk zugreifen kombiniert, möglich gemacht hat die Beteiligung von Forschern und Studenten aus der Schule oder zu Hause.
Protokoll
Der hier beschriebene Ansatz wurde mit einem spezifischen Satz von Instrumenten, Werkzeugen und Software. Da alle Labors werden nicht mit dem gleichen Versuchsaufbau wurde Anstrengungen unternommen, um den Ansatz zu verallgemeinern, wo möglich, und bieten Alternativen, wo dies nicht möglich war.
1. Vorbereiten verglast Virusproben
Im folgenden Abschnitt verglasten Netze werden unter Verwendung der FEI Vitrobot Mark III, ein Löschpapier und tauchen Einfrieren Roboter. Siehe Iancu, et al. Für ein ausführliches Protokoll über die Verwendung dieses System 7. Als Alternative zu einem Roboter-Kolben wird eine Guillotine-Stil oder die Schwerkraft Kolben vollkommen ausreichend 6.
In den Abschnitten 1.2. und 1,3. des Gatan Solarus 950 Glimmentladung dient zum Beispiel Gitter sauber und erhöhen ihre Hydrophilie, obwohl jede Glimmentladung Gerät für die Verwendung mit Elektronen-Mikroskopie Raster konzipiert ersetzt werden können.
content "> Obwohl die Menge des Probenvolumens aufgetragen, um das Gitter vor dem Löschpapier und tauchen Einfrieren sollte im Bereich von 2 ul werden, wird der Virus-und Protein-A Goldkolloid Volumen in der Probe Mischung variieren je nach Quelle. Als solche es ist wahrscheinlich, dass eine Reihe von Viren-und Gold-Verdünnungen oder Verhältnisse müssen geprüft werden und abgebildet in das Mikroskop und eine ideale Mischung empirisch ermittelt. Beachten Sie, dass, wenn Bilddaten in Abschnitt 3 erworben wird, zwischen 10 und 20 Gold Fiducial Marker sollten vorhanden sein für die richtige Neigung Reihenschaltung.Achtung: Dieser Abschnitt beschreibt die Verwendung von gasförmigem Ethan, Wasserstoff und Sauerstoff, die leicht entzündlich sind. Proper Vorsicht ist geboten, wenn die Verwendung dieser Gase sein. Darüber hinaus sollte darauf geachtet werden, Virus-Proben in Übereinstimmung mit Biosicherheit Empfehlungen zu behandeln. Schließlich tragen Sie immer eine Schutzbrille und Kleidung beim Umgang mit flüssigem Stickstoff (N 2) und Ethan.
- Bereiten FEI Vitrobot MarkIII durch Drehen auf der N 2-Gas liefern, die Installation der Luftbefeuchter Patrone und dann Füllen Sie die Patrone mit gefiltertem deionisiertem Wasser. Power on Vitrobot und legen Sie die Klimakammer Temperatur auf 22 ° C, Luftfeuchtigkeit Ziel zu 100%, Löschpapier auf 6 sec und Blot-Offset auf -2 mm.
- Schalten Sie den Gatan Solarus 950 Glimmentladung Einheit und offene H 2 und O 2 Kanister, dass das Gerät zu liefern. Pre-clean Probenkammer, indem Sie Glimmentladung für 1 min bei 50 Watt.
- Ordnen Sie die gewünschte Anzahl der löchrigen Kohlenstoff Gitter auf einem Deckglas, Kohlenstoff Seite nach oben, und im Inneren Glimmentladung Probenkammer durch die obere Ladeklappe. Saubere Grids, indem Sie Glimmentladung für 6-10 sec bei 25 Watt. (Beachten Sie, dass die Kohlenstoff-Seite des Quantifoil Marke Netze in diesem Protokoll verwendet matte Aussehen sind. Das wird nicht für alle kommerziellen Netzen wahr, so ist es am besten, diese Informationen vom Hersteller zu erhalten.)
- Legen Sie Gitterbox (en) innen coolant Behälter Schüssel und thermische Diffusor Spindel auf dem zentralen Tasse, die innerhalb des Kühlmittels Schüssel sitzt. Langsam pour flüssigem N 2 in die Schüssel, bis kräftig sprudelnden Subventionen, was darauf hinweist Gleichgewicht zwischen Schüssel und Flüssigkeit. Pflegen Sie flüssigem N 2-Niveau während der Rest des Verfahrens, wobei darauf geachtet, um es aus der zentralen Tasse.
- Befestigen Sie ein Ende eines Kunststoff-Schlauch in die Ethan-Gasflasche, und das andere Ende an einer Glaspipette. Halten Glas Pipettenspitze nach unten zentralen Topf, stellen Sie eine langsame, stetige Ethan fließen, und Ethan beginnt kondensierend. Weiter füllen, bis das zentrale Maß ist voll. Entfernen Spindel vom zentralen Tasse.
- Bereiten Probe durch Zugabe von 2 ul Protein-A Goldkolloid fiducial bis 10 ul AT-2 inaktiviert HIV-1 Virus-Suspension. Gently Pipette Mischung Fiducials in Lösung zu bringen. Keep on ice.
- Fassen Sie den äußeren Rand eines Rasters mit den spezialisierten Vitrobot Pinzette. Bewerben 2 ul der Probe Mischung zu Kohlenstoff side des Gitters. Unmittelbar anweisen Roboter Probe in befeuchtet Klimakammer, Blot und tauchen Freeze Belastung in flüssigem Ethan.
- Transfer verglasten Raster Gitterbox. Wiederholen Sie Schritt 1.7. um die gewünschte Anzahl von Grids. Wenn Sie fertig sind, ziehen Sie die Schrauben auf Gitterbox Deckel (s) dann schnell Verteilergetriebe (es) für kleine flüssigem Stickstoff Transport Dewar.
2. Loading Proben in Transmissions-Elektronenmikroskop
In diesem Abschnitt soll die Flüssigkeit N-2-Ebene in den Laderaum wachsam beobachtet werden, und Flüssigkeit nachgefüllt wie nötig, um sicherzustellen, dass Gitterboxen eingetaucht bleiben. Bevor bringt ein Werkzeug in Kontakt mit einem Raster, kühlen sie durch Eintauchen in flüssigen N 2, bis sie mit der Flüssigkeit im Gleichgewicht. Nach jedem Gebrauch sollten Instrumente aufgetaut mit einer Heißluftpistole werden.
In diesem Abschnitt der FEI Tecnai G2 Polara Transmissionselektronenmikroskop (TEM) wird in Verbindung mitmit einem Gatan Cryo Workstation. Während das Prinzip der Be-Proben unter flüssigem N 2 Bedingungen gelten für alle Kryo-Elektronenmikroskopie funktioniert, sind die Schritte in diesem Abschnitt speziell auf die Ausrüstung in dem Experiment verwendet. Die Schritte für die verschiedenen verwendeten Geräte werden von Hersteller zu Hersteller unterschiedlich.
ACHTUNG: In diesem Abschnitt immer eine Schutzbrille aufsetzen und Kleidung beim Umgang mit flüssigen N 2.
- Stellen Sie sicher, dass TEM Compustage enthält keine Probe Patrone, und dass die Probe Auswahl Stab und Stecken Stab sind unter 110 K.
- Cool und bereiten Cryo Workstation zum Laden Grids in Probenlagerung Block, welcher Teil der Stichprobe Stange ist.
- Übertragen Sie die gekühlt (<110 K) Auswahl Stab von Mikroskop zu Cryo Workstation.
- Slide Auswahl Stab nach vorn, um die Auswahl Block in Stickstoff-Bad Einsatz auf Cryo Workstation. Entfernen Sie alle Proben Patronen aus Blocklager mit spezialisierten cartridge-Handling Pinzette. Legen Sie Gitterbox (n) in Be-Workstation und lockern Deckel (s) mit einem Schraubendreher.
- Verwenden einer Pinzette Gitter aus Gitterbox und in Patrone zu entfernen. Verwenden Sie die spezielle C-Clip Placement-Tool zu einem C-Clip auf der Oberseite des Grid-Lagerstätte, die Sicherung Gitter in einer Patrone.
- Wiederholen Sie Schritt 2,5. bis die gewünschte Anzahl der Netze sind in Kartuschen. Transfer-Kassetten Probe Auswahl zu blockieren.
- Bereiten Cryo Workstation für die Entfernung der Proben Auswahl Stange. Slide Auswahl Stange zurück zur Auswahl Block von Stickstoff-Bad zu entfernen. Entfernen Auswahl Stab von Cryo Workstation und heften sich an TEM.
3. Acquiring Kryo-Elektronenmikroskopie Daten
Während dieses Abschnitts ist die FEI Batch-Tomographie-Software-Schnittstelle für die Einrichtung einer Batch-Datei, die die Koordinaten aller Startplätze von Interesse speichert. Wenn der Benutzer angewiesen, wird die Batch-Tomographie-Software erneut jede der Positionen und koordinierenKippserie Erwerb über Digital Micrograph. Wenn diese Software nicht verfügbar ist, ist die Leginon Softwarepaket lebensfähigen freien, Open-Source-Alternative 13. Imaging wurde bei 200 keV durchgeführt, unter Verwendung eines Gatan Imaging Filter (GIF) mit einer post-GIF 2K x 2K-CCD-Kamera (Gatan).
- Position Compustage so dass Elektronenstrahl geht durch Vakuum. Wechseln Sie zu hohe Vergrößerung (34kX) Belichtungssteuerung und führen Sie direkt Ausrichtungen.
- Richten Null Verlust Gipfel auf Energie-Filter. (Dieser liefert die Energie-Filter mit einer Referenz für Elektronen mit Null-Energie-Verlust.)
- Bewegen Sie den Compustage, um ein Raster-Bereich mit Kohlenstoff. In geringer Vergrößerung (4.5kX) Suchmodus, verwenden Sie den Wobbler-Funktion auf der Bühne hin und her kippen zwischen + / -15 °. In der Zwischenzeit stellen Sie die Z-Achse der Bühne Position bis minimal Bühne Verschiebung zu beobachten ist. (Diese Positionen der Bühne in etwa auf euzentrische Höhe). Disable Wobbler, wenn keine Bühne Verschiebung zu beobachten ist.
- Verwenden Sie die automatische Euzentrische Höhe f Salbung zu verfeinern euzentrische Höhe. (Diese Routine verwendet Kreuzkorrelation, um eine genauere euzentrische Höhe als in Schritt 3.3) zu erhalten. Euzentrische Höhe wird automatisch zum späteren Nachschlagen von Batch-Tomographie-Software gespeichert.
- Finden Sie einen Bereich von Interesse mit rund drei Minuten vor sechs Virionen und nicht weniger als zehn Fiducial Markers. Fügen Sie diese Startposition, um die Batch-Tomographie-Datei.
- Wiederholen Sie die Schritte 3.4. und 3,5. bis die gewünschte Anzahl von Positionen bezeichnet werden. Definieren Sie Batch-Parameter (± 60 ° mit 2 ° Neigung erhöht, 1-2 E-/ A 2 / tilt view) und die Batch.
4. Die Rekonstruktion Kryo-Elektronen-Tomographie
- Richten Sie Kippserie mit Hilfe der automatischen fiducial-basierten Ausrichtung in RAPTOR 1.
- Rekonstruieren Tomogramme mit R-gewichtete Rückprojektion in IMOD 8. Das Standard-Dateiformat für die daraus entstehenden Datenmengen ist das MRC-Datei mit der Dateiendung. Mrc.
In diesem Abschnitt wird eine individuelle Erweiterung des IMOD, dass der Benutzer Virion Untervolumina im Tomogramm bezeichnen kann. In Abschnitt 6 werden die Benutzer definiert Virion Grenzen als eine Fläche, auf denen Spikes automatisch ausgewählt werden.
- Öffnen IMOD und laden ein Tomogramm mit Virionen besiedelt.
- Navigieren entlang der Z-Achse des Tomogramm, bis der zentralen Schnitt durch eine der Virionen befindet. Überprüfen Sie, ob das Virion Schwerpunkt identifiziert worden ist. Nach tun, Hinterlegung einer Virion Marker. Dieser Marker wird durch die automatische Segmentierung Virion Dienstprogramm in Abschnitt 6 verwendet werden.
- Weiter Schwerpunkt Bezeichnung, bis alle Virion Bände identifiziert werden.
- Close Tomogramm und speichern Marker gesetzt Definition Virion Schwerpunkte. Wiederholen, bis Virionen in alle Tomogramme ausgewiesen worden sind.
- Mit IMOD, down-Probe Virion subtomograms um einen Faktor von vier.
- Denoise Virionen mit Rand-Verbesserung anisotrope Diffusion als in IMOD umgesetzt.
- Subject Virionen zu unbeaufsichtigt Membran-Segmentierung mit einer Energie-basierte dreidimensionale Ansatz (ausführliche in Bartesaghi et al. 2005) 3.
- Spikes sind an Standorten auf der segmentierten Virion Flächen entsprechend den lokalen Maxima der Kreuzkorrelation zwischen den jeweiligen Standort und einem synthetischen symmetrischen spike-like Volumen identifiziert. Jene Orte oberhalb einer bestimmten Schwelle sind, um eine Liste von Koordinaten für putative spike Untervolumina aufgenommen.
6. Klassifizierung und Mittelung der Partikel
- Rechnerisch extrahieren Sie die Tomogramm Untervolumina (100 x 100 x 100 Voxel) an jedem Standort, die in Schritt 5,8 identifiziert wurde. (Dies ist ohne Rauschunterdrückung oder Binning getan). Die daraus resultierende Ansammlung von Untervolumina enthalten die Spike-Proteine, die klassifiziert und werden im Durchschnitt in den folgenden Schritten wird.
- Determine die Orientierungen der langen Achse der Spitze mit der Normalen zu der automatisch segmentierte Membran an der Lage der einzelnen Spikes. Dieser Ansatz liefert erste Schätzungen für zwei der drei Euler-Winkel.
- Randomize die übrigen in-place Drehung um mögliche Verzerrungen in den folgenden Ausrichtungen zu verhindern.
- Übernehmen Euler-Winkel, um Untervolumina, dann translatorisch ausrichten Untervolumina ihre zylindrisch gemittelt globalen Durchschnitt um sicherzustellen, dass sie alle den gleichen Schwerpunkt.
- Entfernen Sie die 10% der Teilvolumina, dass die meisten schlecht korrelieren mit den aktualisierten globalen Durchschnitt. Dies geschieht, um Dichten das größte Risiko irrtümlich Spikes auszuräumen.
- Richten und zu klassifizieren spike Volumes ohne Verwendung von externen Referenzen und mit ordnungsgemäßer Buchführung des fehlenden Keil (ausführliche in Bartesaghi et al. 2008) 4. Schrittweise zu verfeinern Subvolume Ausrichtungen und klassifizieren spike Volumina bei jeder Iteration.
- Dreizählige sollte klar in den frühen Phasen der Klassifizierung beobachtet werden, und bei der vierten Iteration, 3-fach Symmetrie auferlegt wird. In jeder Runde sind die klar definierten Klassen gemittelt und als Referenz für die nächste Runde.
- Typischerweise ~ 4000 Spikes sollte für jeden Datensatz ausgewählt werden. Enddichte Karten sind nach ca. 5-12 Verfeinerung Runden gewonnen und wird Beiträge von ~ 50% der Teilvolumina in jedem Datensatz enthalten.
7. Coordinate Montage
- Die Dichte Karten aus Schritt 6,8 sind in UCSF Chimera 12 geöffnet.
- Erstellung eines simulierten 20 Å Karte des X-ray-Koordinaten mit Chimera oder EMAN 10.
- Dock-Koordinaten in zufälligen Orientierungen. Mit Chimera in steilsten Aufstieg lokale Optimierung gebaut, fit Koordinaten, indem Sie ein Vielfaches von 100 steilsten Anstiegs Schritte bis zur Konvergenz erreicht ist.
8. Repräsentative Ergebnisse
Mit 3D-Mittelung und koordinieren Montage, eine Vielzahl von HIV und SIV Env spike Strukturen wurden aufgelöst. Präsentiert in Abbildung 3A ist die Struktur des Envelope Spike von der HIV-1-Stamm, BaL (violett). Der Dorn hat dreizählige mit Abmessungen von ca. 120 Å als von der Membran auf die Spike-Spitze gemessen, und eine maximale Breite von ca. 150 Å, die sich verjüngt, um ~ 35 Å an der Basis des Stachels. Wie in Abbildung 3B zu sehen, durch die Kombination der Dichte Karte für trimere Env bestimmt mittels Kryo-Elektronen-Tomographie mit der kristallographisch bestimmten Struktur für monomeres gp120 (rot, abgeleitet von PDB ID, 2NY7), konnten wir eine Arbeitsgruppe molekulare Modell für die zu erhalten trimere Hüllglycoprotein Komplexes (PDB ID, 3DNN) 9.
Unser Ansatz ermöglicht auch die Strukturanalyse von Komplexen zwischen trimere Env und eine Vielzahl von gp120-spezifische Protein gebildetFragmente und Antikörper. Ein Beispiel dafür, in welche Env mit den breit neutralisierenden b12 Fab komplexiert ist in Abbildung 4A (der gesamte Komplex ist in lila dargestellt) gezeigt. In dieser Figur wird die zusätzliche Dichte entspricht, b12 gesehen nach außen ragt aus der Spitze, und parallel zur Membran. Strukturelle Interpretationen solcher Komplexe kann auf die molekulare Ebene, indem man die Dichte Karten mit Atomkoordinaten für Teile der Spitze, gebunden an den entsprechenden Liganden erweitert werden. Wie in Abbildung 4B, wenn diese Dichte Karte mit den Koordinaten des gp120 Spike-Protein (rot, von PDB ID, 2NY7 abgeleitet) durch b12 Fab (weiß), die relative Orientierung der drei gp120-Kerne gebunden ist ausgestattet gesehen werden kann erkennen (PDB ID, 3DNL). Diese Konformationsänderungen Beziehungen sind lehrreich für das Verständnis, wie solche Liganden mit dem Dorn zu interagieren und interferieren mit Viren-Aktivität. Einige andere Strukturen unliganded und mit Liganden Env, dass durch das Verfahren geklärt sind hier präsentierten inkl.ude denen von HIV-1 R3A, SIV CP-MAC, SIVmneE11S und SIVmac239, die ternären Komplexes zwischen HIV-1 Env Bal, sCD4 und 17b Fab und SIV CP-Mac Env im Komplex mit dem 7D3 Antikörper 9,14.

Abbildung 1 Von Virussuspension zu 3D-Struktur:. Konzeptionelle Schritte in Kryo-Elektronenmikroskopie und 3D-Rekonstruktion.
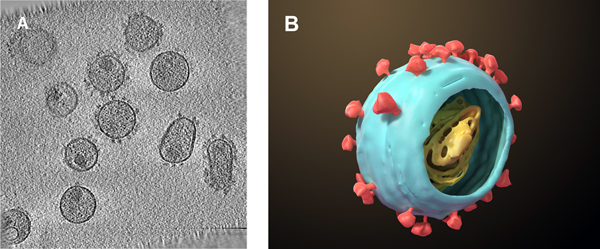
Abbildung 2. (A) Ein Vertreter Scheibe aus einem Tomogramm von HIV-1 Virionen. (B) Eine einzelne HIV-1 Virionen, mit dem Kern und Oberfläche Spikes schematisch dargestellt.
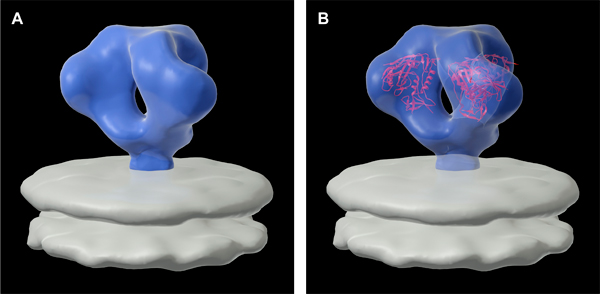
Abbildung 3. (A) Dreidimensionale Struktur des HIV-1 BaL Envelope Glykoprotein (Oberfläche Spikes), wie auf der Oberfläche der Virushülle angezeigt. (B) Molekulares Modell für die trimeric Spikes, indem er drei Exemplare der Strukturen für die monomeren gp120 (rot) durch Röntgenstrukturanalyse in die Dichte Karte 9 abgeleitet wird.
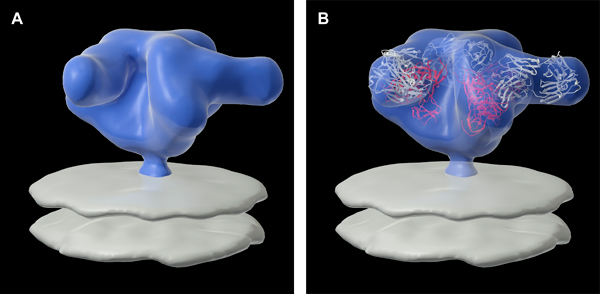
Abbildung 4. (A) Dreidimensionale Struktur von HIV-1 BaL Envelope Glykoprotein im Komplex mit dem Fab-Fragment eines breit neutralisierenden Antikörper b12. (B) Molekulares Modell für die komplexen, indem er drei Exemplare der Strukturen für die monomeren gp120-B12 Fab-Komplexes durch Röntgen-Kristallographie in der Dichte Karte 9 abgeleitet wird.
Diskussion
Die Kryo-Elektronenmikroskopie und dreidimensionale Klassifizierung und Mittelung hier vorgestellten Methoden erlauben die Bestimmung der dreidimensionalen Strukturen von Hüllglycoprotein Komplexe ~ 20 Å Auflösung. Einbau von X-ray-Koordinaten der monomeren Komponenten, um die Dichte-Karten ermöglicht strukturelle Interpretation auf molekularer Ebene. Weitere Verbesserungen in Low-Cost-Computing Equipment mit den Entwicklungen in Open-Source-wissenschaftliche Instrumente und die Verbreitung von Netzwerk-Infrastruktur kombiniert ermöglichen bisher unvorstellbaren wissenschaftlichen Zusammenarbeit bemüht. Wir nutzen diese Entwicklungen und zeigen, dass fortschrittliche wissenschaftliche Techniken innerhalb der Reichweite von Studierenden vieler Zeitalter gebracht werden können.
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Wir danken dem Biological Products Abteilung des AIDS und Krebs Virus Program, SAIC Frederick, Inc., für die Bereitstellung von gereinigtem, AT-2 behandelten HIV-1 Viren, Steven Fellini und Kollegen um Unterstützung bei der Nutzung des High-Performance-rechnerische Fähigkeiten der Biowulf Linux-Cluster am NIH, Bethesda, MD (http://biowulf.nih.gov) , FEI Company für die Unterstützung bei der Elektronenmikroskopie und Ethan Tyler für fachkundige Unterstützung mit Zahlen. Diese Arbeit wurde durch Mittel aus dem Center for Cancer Research am National Cancer Institute, National Institutes of Health, Bethesda, MD unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenz | Firma | Katalog-Nummer | Kommentare |
| Multi-A Holey Carbon-Grids | Quantifoil | - | 200 mesh |
| Protein A Gold-Kolloid | University of Utrechter - Niederlande | - | 10 nm Durchmesser |
| HIV-1 BaL | NIH, NCI, Frederick, MD | - | ~ 10 11 Virionen / ml |
| Cryo-EM-Aufbewahrungsbox | Pacific Grid Tech | GB-4R | 4-Loch, rund |
| Vitrobot Mark III | FEI | - | 6 Sekunden Blot Zeit, -2 mm Offset, 100% Luftfeuchtigkeit |
| Tecnai G2 Polara Transmissionselektronenmikroskop | FEI | - | 200 keV |
| Gatan Imaging Filter | Gatan | - | - |
| Gatan Post-GIF CCD | Gatan | - | 2K x 2K Pixel |
| Gatan Cryo Workstation | Gatan | - | - |
| Gatan Solarus 950 Plasmaspiegel Cleaning System | Gatan | - | Sauerstoff und Wasserstoff-Plasmen |
| C - Clips | FEI | ZIT0634 | - |
| Biowulf Computing-Cluster | National Institutes of Health | - | http://biowulf.nih.gov/ |
| UCSF Chimera | University of California - San Francisco | - | http://www.cgl.ucsf.edu/chimera/ |
| IMOD | University of Colorado - Boulder | - | / IMOD / "> http://bio3d.colorado.edu/imod/ |
| EMAN | Baylor College of Medicine | - | http://blake.bcm.tmc.edu/eman/ |
| RAPTOR | Stanford University | - | http://www-vlsi.stanford.edu/TEM/index.htm |
Referenzen
- Amat, F. Markov random field based automatic image alignment for electron tomography. J. Struct. Biol. 161, 260-275 (2008).
- Bowen, D. L., Lane, H. C., Fauci, A. S. Immunopathogenesis of the acquired immunodeficiency syndrome. Ann. Intern. Med. 103, 704-709 (1985).
- Bartesaghi, A., Sapiro, G., Subramaniam, S. An Energy-Based Three-Dimensional Segmentation Approach for the Quantitative Interpretation of Electron Tomograms. IEEE. T. Image. Process. 14, 1314-1323 (2005).
- Bartesaghi, A. Classification and 3D averaging with missing wedge correction in biological electron tomography. J. Struct. Biol. 162, 437-450 (2008).
- Harris, A. Trimeric HIV-1 gp140 immunogens and native HIV-1 envelope glycoproteins display the same closed and open quaternary molecular architectures. Proc. Natl. Acad. Sci. U.S.A. 108, 11440-11445 (2011).
- Harris, R. J., Adrian, M. Preparation of thin-film frozen-hydrated/vitrified biological specimens for cryoelectron microscopy. Methods Mol. Biol. 117, 31-48 (1999).
- Iancu, C. Electron cryotomography sample preparation using the Vitrobot. Nat. Protoc. 1, 2813-2819 (2006).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. J. Struct. Biol. Computer Visualization of Three-Dimensional Image Data using IMOD. 116, 71-76 (1996).
- Liu, J., Bartesaghi, A., Borgnia, M. J., Sapiro, G., Subramaniam, S. Molecular architecture of native HIV-1 gp120 trimers. Nature. 455, 109-113 (2008).
- Ludtke, S. J., Baldwin, P. R., Chiu, W. EMAN: Semiautomated Software for High-Resolution Single-Particle Reconstructions. J. Struct. Biol. 128, 82-97 (1999).
- Milne, J. L. S., Subramaniam, S. Cryo-electron tomography of bacteria: progress, challenges and future prospects. Nat. Rev. Microbiol. 7, 666-675 (2009).
- Pettersen, E. F. UCSF Chimera - A Visualization System for Exploratory Research and Analysis. J. Comput. Chem. 25, 1605-1612 (2004).
- Suloway, C. Automated molecular microscopy: the new Leginon system. J. Struct. Biol. 151, 41-60 (2005).
- White, T. Molecular architectures of trimeric SIV and HIV-1 envelope glycoproteins on intact viruses: strain-dependent variation in quaternary structure. PLoS. Pathog. 6, e1001249-e1001249 (2010).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten