Method Article
Determinação de estruturas moleculares de glicoproteínas do envelope HIV usando Cryo-Electron Tomografia e Média Sub-tomogram Automated
Neste Artigo
Resumo
O protocolo descreve uma abordagem de alto rendimento para determinação de estruturas de proteínas de membrana usando tomografia crio-eletrônica e processamento de imagens 3D. Abrange os detalhes da preparação da amostra, coleta de dados, processamento e interpretação dos dados, e conclui com a produção de um alvo representante para a abordagem, o HIV-1 glicoproteína do envelope. Estes procedimentos computacionais são projetados de uma forma que permite aos pesquisadores e estudantes para trabalhar remotamente e contribuir para processamento de dados e análise estrutural.
Resumo
Desde a sua descoberta há quase 30 anos, mais de 60 milhões de pessoas foram infectadas com o vírus da imunodeficiência humana (HIV) (www.usaid.gov) . O vírus infecta e destrói CD4 + células-T, assim, corroendo o sistema imunológico, e causando uma síndrome da imunodeficiência adquirida (AIDS) 2. Infecção começa quando a glicoproteína do envelope do HIV "pico" faz contato com o receptor CD4 na superfície das células T CD4 +. Essa interação induz uma mudança conformacional na espiga, o que promove a interação com uma superfície segunda célula co-receptor 5,9. A significância dessas interações proteína na via de infecção pelo HIV torna de profunda importância na pesquisa do HIV fundamental, e na busca de uma vacina contra o HIV.
A necessidade de melhor compreender as interações em escala molecular do HIV contato celular e neutralização motivou o desenvolvimento de uma técnica para determinar aestruturas da espiga HIV interagindo com proteínas de superfície celular receptores e moléculas que a infecção bloco. Usando tomografia crio-eletrônica e processamento de imagens 3D, recentemente demonstrou a capacidade de determinar tais estruturas na superfície do vírus da nativa, a ~ 20 Å de resolução 9,14. Esta abordagem não se limita às estruturas Envelope resolver HIV, e pode ser estendido a outras proteínas da membrana viral e proteínas reconstituído em um lipossoma. Neste protocolo, descrevemos como obter estruturas de glicoproteínas do invólucro do HIV a partir de partículas virais purificadas e HIV processo passo a passo através de preparação de amostras vitrificados, a coleta, os dados crio-microscopia eletrônica, reconstituindo e processamento de volumes 3D de dados, calculando a média e classificação subvolumes proteína 3D, e interpretação dos resultados para produzir um modelo de proteína. Os aspectos computacionais da nossa abordagem foram adaptados em módulos que podem ser acessados e executados remotamente usando o Biowulf GNU / Linux paralela paglomerado ROCESSAMENTO no NIH (http://biowulf.nih.gov) . Esse acesso remoto, combinado com hardware de baixo custo computador e acesso de alta velocidade da rede, tornou possível o envolvimento de pesquisadores e estudantes que trabalham na escola ou em casa.
Protocolo
A abordagem aqui descrita foi desenvolvida usando um conjunto específico de instrumentos, ferramentas e software. Porque todos os laboratórios não estarão usando esta mesma configuração experimental, o esforço foi feito para generalizar a abordagem, sempre que possível, e fornecer alternativas quando isso não era possível.
1. Preparação de amostras de vírus vitrificados
Na seção seguinte grades vitrificados são preparados com o FEI Vitrobot Mark III, um robô de congelamento mata-borrão e mergulho. Veja Iancu, et al. Para um protocolo detalhado sobre a utilização deste sistema 7. Como uma alternativa para um êmbolo de robótica, um êmbolo guilhotina estilo ou a gravidade é perfeitamente suficiente 6.
Nas Seções 1.2. e 1.3. um Gatan Solarus 950 unidade de descarga luminescente é usado para limpar grades amostra e aumentar sua hidrofilicidade, apesar de qualquer dispositivo de descarga luminescente desenvolvido para uso com redes de microscopia eletrônica pode ser substituído.
> content "Embora a quantidade de volume de amostra aplicado à rede antes de mata-borrão e congelamento de mergulho deve ser na faixa de 2 mL, o vírus ea proteína-A de ouro coloidal de volume na mistura amostra irá variar dependendo da fonte. Como tal, é provável que uma gama de vírus e de ouro diluições ou rácios terão de ser testados e fotografado no microscópio, e uma mistura ideal determinada empiricamente. Note que, quando dados de imagem é adquirida na Seção 3, entre 10 e 20 de ouro marcadores fiduciais devem estar presentes para um alinhamento adequado tilt série.Atenção: Esta seção descreve o uso de etano gasoso, hidrogênio e oxigênio que são altamente inflamáveis. A devida cautela deve ser tomada ao usar esses gases. Além disso, cuidados devem ser tomados para lidar com amostras de vírus, de acordo com as recomendações de biossegurança. Finalmente, use sempre óculos de proteção e roupas ao manusear nitrogênio líquido (N 2) e etano.
- Prepare FEI Vitrobot MarkIII ligando o seu fornecimento de gás N 2, a instalação do cartucho de umidificador e, em seguida, encher o cartucho com água filtrada deionizada. Poder em Vitrobot e definir sua temperatura câmara climática a 22 ° C, umidade alvo a 100%, o tempo blotting a 6 seg, e blot compensar a -2 mm.
- Alimentação na unidade de descarga Solarus Gatan 950 brilho e abrir H 2 e O 2 latas de que o abastecimento da unidade. Pré-limpeza da câmara de amostra através da execução de descarga luminescente para 1 min a 50 Watts.
- Organizar o número desejado de grades de carbono holey em uma lamela de vidro, de carbono lado para cima, e coloque dentro de brilho câmara de amostras de descarga através da porta de carga superior. Grades limpa executando descarga luminescente para 60-10 seg a 25 Watts. (Note que o lado de carbono das grades marca Quantifoil utilizado neste protocolo são matte na aparência. Isso não vai ser verdade para todas as redes comerciais por isso é melhor para obter essa informação do fabricante.)
- Coloque caixa de grade (es) dentro de coolant tigela container, e veio difusor térmico no copo central que fica dentro da tigela de refrigeração. Lentamente, deitar líquidos N 2 na bacia enquanto se mantiver a borbulhar vigoroso, indicando equilíbrio entre a bacia e líquido. Manter líquidos N 2 nível durante todo o restante do procedimento, tendo o cuidado de mantê-lo fora do copo central.
- Prenda uma extremidade de uma mangueira de plástico para o botijão de gás etano, ea outra extremidade a uma pipeta de vidro. Segure a ponta da pipeta de vidro de fundo do copo central, estabelecer um processo lento, o fluxo constante de etano, e etano começará condensação. Continue enchendo até a taça central é completo. Retire do eixo do copo central.
- Prepare a mistura da amostra por adição de 2 mL de proteína-A de ouro coloidal fiducial a 10 mL AT-2 HIV-1 inativado suspensão de vírus. Pipeta suavemente a mistura de trazer fiducials na solução. Manter no gelo.
- Segure a borda externa de uma grade com as pinças Vitrobot especializados. Aplicar 2 mL da mistura da amostra de carbono side da grade. Imediatamente instruir robô para carregar amostra em umidificado blot clima de câmara, e congelar mergulhar em etano líquidos.
- Transferência de grade vitrificados para a caixa de grade. Repita o passo 1.7. para produzir o número desejado de grades. Quando terminar, aperte os parafusos na tampa da grade caixa (s), em seguida, transferir rapidamente box (es) para pequenas nitrogênio líquido de transporte dewar.
2. Amostras de carregamento em microscópio eletrônico de transmissão
Em toda esta seção, o líquido N 2 nível na câmara de carga devem ser monitorados atentamente, e líquido repostos conforme a necessidade, para garantir que as caixas de rede continuam imersos. Antes de trazer uma ferramenta em contato com uma grade, resfriá-lo por submersão em líquido N 2 até que ela equilibra com o líquido. Após cada utilização, as ferramentas devem ser descongelados usando uma pistola de ar quente.
Ao longo desta secção da FEI Tecnai G2 Polara microscópio eletrônico de transmissão (TEM) é usado em conjuntocom uma Workstation Cryo Gatan. Embora o princípio de amostras de carga em N 2 líquido condições é aplicável a todo o trabalho de microscopia crio-eletrônica, os passos desta seção são específicas para os equipamentos utilizados no experimento. As etapas usadas para diferentes equipamentos variam de acordo com fabricante.
ATENÇÃO: Em toda esta seção, sempre use óculos de proteção e vestuário de manipulação de líquidos quando N 2.
- Garantir que Compustage TEM não contém um cartucho de amostra, e que a amostra de seleção haste e inserção da haste estão abaixo de 110 K.
- Fresco e preparar Workstation Cryo para carregar grades em bloco da amostra de armazenamento, que faz parte da haste de seleção da amostra.
- Transferência da vara seleção refrigerado (<110 K) de microscópio para Cryo Workstation.
- Rod seleção deslizar para a frente para inserir bloco de seleção em banho de nitrogênio em Workstation Cryo. Remova todos os cartuchos de amostra de bloco de armazenamento usando especializado cartridge de manuseio de fórceps. Coloque caixa de grade (es) em estação de trabalho de carga e afrouxar tampa (s) com uma chave de fenda.
- Use uma pinça para remover grade da caixa de grade, e colocados no cartucho. Use a ferramenta de colocação especializado C-clip para depositar um clip-C em cima da grade, garantindo grade em cartucho.
- Repita o passo 2.5. até o número desejado de grades são em cartuchos. Transferência de cartuchos para bloquear a seleção da amostra.
- Prepare Workstation Cryo para a remoção da haste de seleção da amostra. Deslize vara seleção de volta para remover blocos de seleção do banho de nitrogênio. Remover rod seleção Workstation Cryo e anexar a TEM.
3. Aquisição de dados crio-microscopia eletrônica
Durante esta seção, o FEI Batch interface de software Tomografia é usado para criar um arquivo de lote que armazena as coordenadas de todas as posições no grid de interesse. Quando solicitado pelo usuário, o software Tomografia Batch vai revisitar cada uma das posições e coordenarátilt aquisição série via Micrografia Digital. Se esse software não está disponível, o pacote de software Leginon é viável fonte livre, aberta alternativa 13. Imagem foi feita em 200 keV, utilizando uma imagem Gatan Filter (GIF), com uma pós-GIF 2K x 2K câmera CCD (Gatan).
- Posição tal que Compustage feixe de elétrons passa através de vácuo. Mude para o modo de exposição elevada ampliação (34kX) e executar alinhamentos direta.
- Alinhar de zero pico de perda de energia filtro. (Isto fornece o filtro de energia com uma referência para os elétrons com zero perda de energia.)
- Mova o Compustage a uma área com grade de carbono. Em baixa ampliação (4.5kX) Modo de Busca, use a função de Wobbler para inclinar o palco e para trás entre + / -15 °. Enquanto isso ajustar a posição estágio Z-eixo até mudança de estágio mínimo é observado. (Isto posiciona o palco aproximadamente na altura eucentric). Wobbler desativar quando não há mudança de fase é observada.
- Use o f Altura automatizado Eucentric unção para refinar a altura eucentric. (Essa rotina usa correlação cruzada para obter uma altura mais precisa do que em eucentric passo 3.3). Eucentric altura será automaticamente armazenada para posterior referência por lote software Tomografia.
- Encontrar uma área de interesse com cerca de 3-6 virions, e nada menos que dez marcadores fiduciais. Adicione esta posição no grid para o arquivo de tomografia lote.
- Repita os passos 3.4. e 3.5. até o número desejado de posições são designados. Definir parâmetros de lote (± 60 ° com incrementos de 2 ° de inclinação, 1-2 e-/ a 2 / view tilt) e executar o lote.
4. Reconstruir crio-eletrônica tomogramas
- Alinhar série tilt uso de alinhamento automático baseado fiducial em RAPTOR 1.
- Reconstruir tomogramas usando R-ponderada de projeção traseira em IMOD 8. O formato de arquivo padrão para os volumes de dados resultante é o arquivo de MRC, com extensão de arquivo. Mrc.
Esta seção usa uma extensão personalizada de IMOD que permite ao usuário designar subvolumes virion dentro da tomografia. Na seção 6, o usuário limites definidos virion são usados como uma superfície ao longo da qual picos são automaticamente selecionados.
- Abra IMOD e carregar um tomogram preenchida com virions.
- Navegar ao longo do eixo Z do tomogram até que a fatia central através de um dos virions está localizado. Verifique se o centróide virion foi identificado. Após fazer isso, depositar um marcador virion. Este marcador será usado pelo utilitário automatizado virion segmentação na Seção 6.
- Continue designação centróide até que todos os volumes virion são identificados.
- Fechar tomogram e salvar set marcador definir centróides virion. Repita até que virions tenham sido designados em todos os tomogramas.
- Usando IMOD, subtomograms baixo amostra virion por um fator de quatro.
- Virions Denoise usando borda de melhoria da difusão anisotrópica conforme implementado no IMOD.
- Virions sujeitos a segmentação não supervisionada membrana usando uma abordagem baseada em energia tridimensionais (detalhado no Bartesaghi et al. 2005) 3.
- Picos são identificados em locais nas superfícies virion segmentado correspondentes aos valores máximos locais de correlação cruzada entre o local em particular e um volume de pico sintéticos-como simétrica. Nesses locais acima de um limite especificado são adicionados a uma lista de coordenadas para subvolumes suposto pico.
6. Classificação e da média das partículas
- Computacionalmente extrair o subvolumes tomogram (100 x 100 x 100 voxels) em cada local que foi identificado no passo 5.8. (Isto é feito sem denoising ou binning). A coleção resultante de subvolumes contêm as proteínas pico que será classificado e média nas etapas seguintes.
- Determine as orientações do eixo longitudinal do pico usando o normal para a membrana automaticamente segmentada na localização de cada ponto. Esta abordagem fornece estimativas iniciais para dois dos três ângulos de Euler.
- Randomize os restantes rotação no local para evitar qualquer possível viés nos alinhamentos subseqüentes.
- Aplique ângulos de Euler para subvolumes, então traducionalmente alinhar o subvolumes a sua média cilindricamente média global para garantir que todos eles compartilham o mesmo centro de massa.
- Remova os 10% de subvolumes que se correlacionam mais mal com a média atualizada global. Isto é feito para remover densidades em maior risco de ser erroneamente identificados picos.
- Alinhar e classificar volumes pico sem o uso de referências externas e com contabilidade da cunha em falta (detalhado no Bartesaghi et al. 2008) 4. Progressivamente refinar alinhamentos subvolume e classificar volumes pico a cada iteração.
- Três vezes simetria devem ser claramente observado nas fases iniciais de classificação, e na quarta iteração, 3 vezes simetria é imposta. Em cada rodada, os mais bem definidas as classes média e são usados como referência para a próxima rodada.
- Tipicamente ~ 4000 pontos devem ser selecionados para cada conjunto de dados. Mapas de densidade final são obtidos após ~ 5-12 rodadas requinte e incluirá contribuições de ~ 50% de sub-volumes em cada conjunto de dados.
7. Coordenar montagem
- Os mapas de densidade resultante de 6,8 passo são abertos em 12 UCSF Chimera.
- Produzir um simulado 20 um mapa da X-ray usando coordenadas Chimera ou EMAN 10.
- Doca coordenadas em orientações aleatórias. Usando Chimera é construído em íngreme subida de otimização local, cabem coordenadas através da realização de múltiplos de 100 degraus íngremes de subida até a convergência é obtida.
8. Resultados representante
Usando uma média de 3D e coordenar montagem, uma variedade de HIV e SIV estruturas Env pico foram resolvidos. Apresentado na Figura 3A é a estrutura do pico Envelope do HIV-1 estirpe, Bal (roxo). O pico possui três vezes simetria com dimensões de ~ 120 Å, medida a partir da membrana para o ápice da espiga, e uma largura máxima de ~ 150 Å que se afila até a ~ 35 Å na base da espiga. Como visto na Figura 3B, combinando o mapa de densidade para trimérico Env determinada usando tomografia crio-eletrônica com a estrutura cristalograficamente determinado para gp120 monomérico (vermelho, derivado do PDB ID, 2NY7), fomos capazes de obter um modelo de trabalho molecular para o complexo trimérico da glicoproteína do envelope (PDB ID, 3DNN) 9.
Nossa abordagem também permite a análise estrutural dos complexos formados entre trimérico Env e uma variedade de proteínas gp120 específicasfragmentos e anticorpos. Um exemplo disto, na qual Env é complexado com o amplamente neutralizantes b12 Fab é mostrado na Figura 4A (todo o complexo é mostrado em roxo). Nesta figura, a densidade extra correspondente a b12 é visto projetando para fora do pico, e paralelos à membrana. Interpretações estruturais de tais complexos pode ser estendido para o nível molecular do ajuste dos mapas de densidade com coordenadas atômicas para partes do ponto, ligado ao ligante correspondente. Como visto na Figura 4B, quando este mapa de densidade é equipado com coordenadas da proteína gp120 pico (vermelho, derivado do PDB ID, 2NY7) vinculadas por b12 Fab (branco), as orientações relativas dos três núcleos gp120 pode ser discernido (PDB ID, 3DNL). Estas relações conformacional são instrutivas para entender como ligantes tais interagir com a espiga e interferir com a atividade de vírus. Algumas outras estruturas de Env unliganded liganded e que foram resolvidas pelo método aqui apresentado include os de HIV-1 R3A, SIV CP-MAC, SIVmneE11S e SIVmac239, o complexo ternário entre o HIV-1 Bal Env, sCD4 e Fab 17b, e SIV CP-Mac Env complexada ao anticorpo 7D3 9,14.

Figura 1 A partir da suspensão viral a estrutura 3D:. Passos conceituais em crio-microscopia eletrônica e reconstrução 3D.
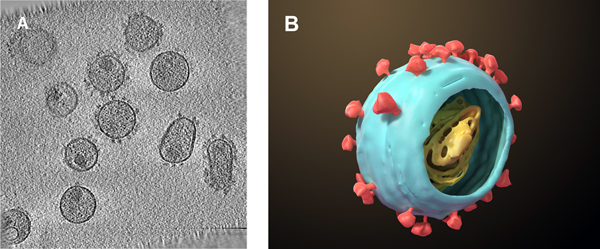
Figura 2. (A) Uma fatia representativa a partir de uma tomografia de HIV-1 virions. (B) Um vírion HIV-1 única, com os pontos principais e de superfície mostrado esquematicamente.
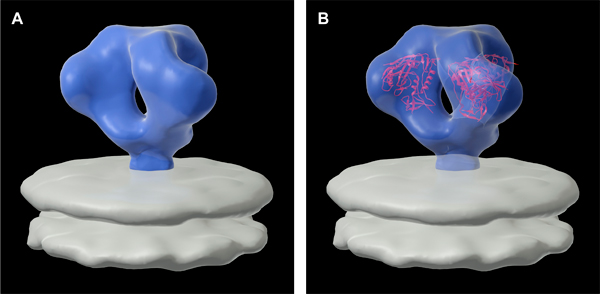
Figura 3. (A) a estrutura tridimensional da glicoproteína do envelope do HIV-1 BaL (picos de superfície), como exibido na superfície da membrana viral. (B) para o modelo Molecular TRIMEpicos ric determinado pela colocação de três cópias das estruturas de gp120 monomérico (vermelho) obtidas por cristalografia de raios X na densidade mapa 9.
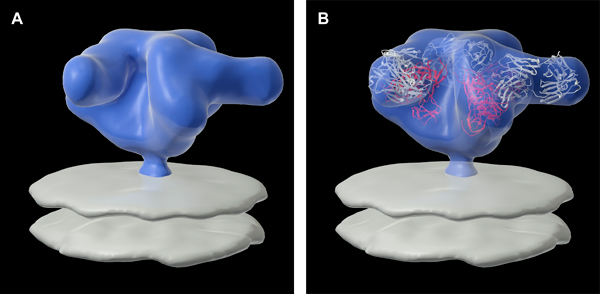
Figura 4. (A) a estrutura tridimensional do HIV-1 glicoproteína Envelope BaL no complexo com o fragmento Fab de um anticorpo amplamente neutralizantes, b12. (B) modelo molecular para o complexo determinado pela colocação de três cópias das estruturas de complexos monoméricos Fab gp120-B12 obtidas por cristalografia de raios X na densidade mapa 9.
Discussão
A microscopia eletrônica de crio-e tridimensional de classificação e métodos de média aqui apresentados permitem a determinação de estruturas tridimensionais de complexos envelope glicoproteína a ~ 20 Å de resolução. Montagem de X-ray coordenadas dos componentes monoméricos com os mapas de densidade permite uma interpretação estrutural ao nível molecular. Melhorias contínuas nos equipamentos de baixo custo de computação combinada com a evolução das ferramentas de código aberto científica ea proliferação de infra-estrutura de rede tornam possível antes inimagináveis esforços de colaboração científica. Aproveitamos estes desenvolvimentos e mostrar que avançadas técnicas científicas podem ser trazidos ao alcance de alunos de muitas idades.
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
Agradecemos à Seção de Produtos Biológicos da AIDS e do Programa de Virus Câncer, SAIC Frederick, Inc, para a prestação de purificada, AT-2 tratados vírus HIV-1, Steven Fellini e colegas de assistência com a utilização dos recursos de alto desempenho computacional do Biowulf Linux cluster em NIH, Bethesda, MD (http://biowulf.nih.gov) , FEI Company para obter ajuda com microscopia eletrônica, e Tyler Ethan para assistência especializada com figuras. Este trabalho foi financiado por fundos do Centro de Pesquisa do Câncer do National Cancer Institute, National Institutes of Health, Bethesda, MD.
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente | Companhia | Número de catálogo | Comentários |
| A Multi-Carbono Grids Holey | Quantifoil | - | 200 mesh |
| A proteína de ouro coloidal | Universidade de Utrech - Holanda | - | 10 nm de diâmetro |
| HIV-1 BaL | NIH, NCI, Frederick, MD | - | ~ 10 11 virions / mL |
| Cryo-EM caixa de armazenamento | Pacific Grade Tecnologia | GB-4R | 4 furos, redondo |
| Vitrobot Mark III | FEI | - | 6 Tempo blot segunda mm, -2 compensar umidade, 100% |
| Tecnai G2 Polara microscópio eletrônico de transmissão | FEI | - | 200 keV |
| Gatan imagem Filtro | Gatan | - | - |
| Gatan Post-GIF CCD | Gatan | - | 2K x 2K pixels |
| Gatan Cryo Workstation | Gatan | - | - |
| Gatan Solarus 950 Sistema de Limpeza Plasma | Gatan | - | Plasmas de oxigênio e hidrogênio |
| C - clips | FEI | ZIT0634 | - |
| Biowulf computação em cluster | National Institutes of Health | - | http://biowulf.nih.gov/ |
| UCSF Chimera | Universidade da Califórnia - San Francisco | - | http://www.cgl.ucsf.edu/chimera/ |
| IMOD | University of Colorado - Boulder | - | / Imod / "> http://bio3d.colorado.edu/imod/ |
| REMA | Baylor College of Medicine | - | http://blake.bcm.tmc.edu/eman/ |
| RAPTOR | Stanford University | - | http://www-vlsi.stanford.edu/TEM/index.htm |
Referências
- Amat, F. Markov random field based automatic image alignment for electron tomography. J. Struct. Biol. 161, 260-275 (2008).
- Bowen, D. L., Lane, H. C., Fauci, A. S. Immunopathogenesis of the acquired immunodeficiency syndrome. Ann. Intern. Med. 103, 704-709 (1985).
- Bartesaghi, A., Sapiro, G., Subramaniam, S. An Energy-Based Three-Dimensional Segmentation Approach for the Quantitative Interpretation of Electron Tomograms. IEEE. T. Image. Process. 14, 1314-1323 (2005).
- Bartesaghi, A. Classification and 3D averaging with missing wedge correction in biological electron tomography. J. Struct. Biol. 162, 437-450 (2008).
- Harris, A. Trimeric HIV-1 gp140 immunogens and native HIV-1 envelope glycoproteins display the same closed and open quaternary molecular architectures. Proc. Natl. Acad. Sci. U.S.A. 108, 11440-11445 (2011).
- Harris, R. J., Adrian, M. Preparation of thin-film frozen-hydrated/vitrified biological specimens for cryoelectron microscopy. Methods Mol. Biol. 117, 31-48 (1999).
- Iancu, C. Electron cryotomography sample preparation using the Vitrobot. Nat. Protoc. 1, 2813-2819 (2006).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. J. Struct. Biol. Computer Visualization of Three-Dimensional Image Data using IMOD. 116, 71-76 (1996).
- Liu, J., Bartesaghi, A., Borgnia, M. J., Sapiro, G., Subramaniam, S. Molecular architecture of native HIV-1 gp120 trimers. Nature. 455, 109-113 (2008).
- Ludtke, S. J., Baldwin, P. R., Chiu, W. EMAN: Semiautomated Software for High-Resolution Single-Particle Reconstructions. J. Struct. Biol. 128, 82-97 (1999).
- Milne, J. L. S., Subramaniam, S. Cryo-electron tomography of bacteria: progress, challenges and future prospects. Nat. Rev. Microbiol. 7, 666-675 (2009).
- Pettersen, E. F. UCSF Chimera - A Visualization System for Exploratory Research and Analysis. J. Comput. Chem. 25, 1605-1612 (2004).
- Suloway, C. Automated molecular microscopy: the new Leginon system. J. Struct. Biol. 151, 41-60 (2005).
- White, T. Molecular architectures of trimeric SIV and HIV-1 envelope glycoproteins on intact viruses: strain-dependent variation in quaternary structure. PLoS. Pathog. 6, e1001249-e1001249 (2010).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados