Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Ein hoher Durchsatz In situ Hybridisierung Methode zur mRNA Expression Patterns in der Fetal-Maus unteren Urogenitaltrakts Charakterisieren
In diesem Artikel
Zusammenfassung
Hier beschreiben wir eine effiziente Hochdurchsatz- In situ Hybridisierung (ISH)-Methode zur Visualisierung von Mustern der mRNA-Expression in der Entwicklung fetalen Maus Prostatagewebe Abschnitten. Die Methode kann leicht angepasst werden, um mRNA-Expressionsmuster in anderen Maus Gewebe oder in Gewebe von anderen Arten zu visualisieren.
Zusammenfassung
Entwicklung des unteren Urogenitaltraktes (LUT) ist ein komplizierter Vorgang. Diese Komplexität wird während der Bildung der Prostata aus dem fötalen männlichen Harnröhre, die auf androgene Signale und epithelial-mesenchymale Interaktionen 1,2 stützt belegt. Das Verständnis der molekularen Mechanismen, die für Prostata-Entwicklung kann sich herausstellen, Wachstum Mechanismen, die unangemessen wiedererweckt werden später im Leben zu steigen, um Prostataerkrankungen, wie gutartige Prostata-Hyperplasie und Prostatakrebs geben.
Die Entwicklung von LUT ist anatomisch komplexer. Zu der Zeit, Prostata angehende beginnt auf 16,5 Tage nach der Empfängnis (DPC), sind zahlreiche Zelltypen vor. Gefäßsystem, Nerven und glatten Muskelzellen in den mesenchymalen Stromazellen 3 befinden. Das Stroma umgibt ein mehrschichtiges Epithel und gibt Anlass zu der fetalen Prostata durch Androgen-Rezeptor-abhängige parakrine Signale 4. Die Identität der Stroma-Androgen-Rezeptor-responsive Gene für Prostata-Entwicklung und der Mechanismus, mit dem Prostata-duktalen Epithel bildet in Reaktion auf diese Gene nicht vollständig verstanden erforderlich. Die Fähigkeit, genau zu identifizieren Zelltypen und zu lokalisieren Expression spezifischer Faktoren in ihnen ist zwingend notwendig, um weiter zu verstehen Prostata Entwicklung. In-situ-Hybridisierung (ISH) ermöglicht die Lokalisierung von mRNAs in einem Gewebe. Somit kann diese Methode verwendet, um Muster und Timing der Expression von Signalmolekülen und ihren Rezeptoren zu identifizieren, damit die Aufklärung potenzieller Prostata Entwicklungsregulatoren werden.
Hier beschreiben wir einen hohen Durchsatz ISH Technik, um mRNA-Expressionsmuster in der fetalen Maus LUT mit vibrierenden Mikrotom-Zuschnitte zu identifizieren. Diese Methode bietet einige Vorteile gegenüber anderen ISH-Protokolle. Performing ISH am dünne Schnitte verklebt, um eine Folie ist technisch schwierig, Kryoschnitten haben häufig schlechte bauliche Qualität, während sowohl Gefrierschnitte und Paraffinschnitte oft in schwachen Signal Auflösung führen. Performing ISH auf whole mount Gewebe können in der Sonde Trapping führen. Im Gegensatz dazu nutzt unsere Hochdurchsatz-Technik dick geschnittenen Abschnitte, die detaillierte Gewebe Architektur offenbaren. Geändert Mikrozentrifugenröhrchen ermöglichen eine einfache Handhabung der Teile während der ISH Verfahren. Es können maximal 4 mRNA-Transkripte können von einem einzigen 17.5dpc LUT mit bis zu 24 mRNA-Transkripte in einem einzigen Durchlauf erkannt abgeschirmt werden, wodurch Kosten und Maximierung der Effizienz. Diese Methode ermöglicht es mehreren Behandlungsgruppen identisch und als eine Einheit verarbeitet werden, wodurch eine Tendenz für die Interpretation der Daten. Die meisten treffend für Prostata-Forscher, stellt diese Methode eine räumliche und zeitliche Lage der niedrigen und hohen Fülle mRNA-Transkripte in der fetalen Maus Harnröhre, die Anlass zu der Prostata duktalen Netzwerk.
Protokoll
1. Synthese eines Digoxigenin-11-UTP-markierten Riboprobe aus einer PCR-Generated-Vorlage
- Zur Synthese eines Gens spezifischen Ribosonde, verwenden Entrez Gene (http://www.ncbi.nlm.nih.gov/sites/entrez), um das Gen cDNA Referenzsequenz (RefSeq) zu erhalten. Verwenden Sie die Primer3 Programm (http://frodo.wi.mit.edu/primer3/) 5 bis genspezifischen PCR-Primer gegen die 3'-Region der cDNA-Sequenz-Design. Empfohlene Parameter für PCR-Primer Auswahl an anderer Stelle (http://www.gudmap.org/Research/Protocols/Vezina/Riboprobe_Syn.html) beschrieben.
- Verwenden Sie die Megablast Program (http://www.ncbi.nlm.nih.gov/genome/seq/BlastGen/BlastGen.cgi?taxid=10090) 6 bis Spezifität der ausgewählten DNA-Sequenz zu beurteilen. Die DNA-Sequenz betrachtet wird, wenn bestimmte, mit einem ERWARTEN Schwelle von 0,01, ist es nicht mit anderen Sequenzen in der Datenbank RefSeq auszurichten.
- PCR Verstärkung des Ribosonde Vorlage. PCR-Reaktion Komponenten und Thermocycling Bedingungen sollten für jedes Primer-Set optimiert werden. Eine typische 50 pl Reaktion enthält: 1X Puffer, 2 mM MgCl 2, 0,2 mM dNTPs, 1X Q-Lösung, 1 g cDNA, 2,5 U Taq DNA Polymerase, 0.25μm Primer und Nuklease-freiem H 2 O. Ein typisches Thermocycling Protokoll beinhaltet eine initiale Denaturierung bei 94 ° C für 2min von 40 Zyklen mit 94 ° C für 30 Sekunden gefolgt, 57 ° C für 30 Sekunden, 72 ° C für 1 Minute und einer abschließenden Extension bei 72 ° C für 10min. Die cDNA in den PCR-Reaktionen verwendet wird, aus Maus urogenitalen mRNA synthetisiert.
- Separate des PCR-Produkts durch Agarosegelelektrophorese, Reinigung mit dem Gel Extraction Kit, und Quantifizierung der gereinigten Produkte spektralphotometrisch. Die erwartete Rendite liegt bei 1,2 - 3.6μg.
- Transcribe des PCR-Produkts in ein beschriftetes Ribosonde. Die Transkriptions-Reaktion (40 ul) enthält: 400ng gereinigte PCR-Produkt, 1X Nukleotid Labeling Mix mit Digoxigenin 11-UTP, 1X Transkription Puffer, 5U RNase-Inhibitor, 80U T7-RNA-Polymerase und Nuklease-freiem H 2 O. Inkubieren 3-4 Std. bei 37 ° C gestellt und dabei die Proben alle 30min.
- Verwenden Sie die Qiagen RNeasy Mini Kit Ribosonden auf Anweisungen für die RNA-Aufreinigung mit on-column DNase-Verdau Basis zu reinigen. Quantifizieren Ribosonden durch Spektrophotometrie. Die erwartete Rendite ist 4-20 pg. Beurteilen Sonde Qualität durch die Trennung ein Aliquot durch Elektrophorese auf einem 1,5% nicht-denaturierenden Agarosegel. Hochwertige Sonden als distinkte Banden mit minimalem Verschmieren zu migrieren.
- Um die Ribosonde erkennt spezifisch das Ziel, eine positive Kontrolle Gewebe zum ersten ISH Experiment. Die Ribosonde die Ziel-mRNA-Muster sollten in dieser positive Kontroll-Gewebe bekannt sein.
2. Vorbereitung der Vibrating Mikrotomklinge (Basierend auf zuvor beschriebenen Protokoll) 7
- Bereiten Sie eine 4% Low-Melt Agarose-Lösung in Phosphat-gepufferter Kochsalzlösung (PBS). Mikrowelle Lösung Agarose aufzulösen und zu pflegen Lösung bei 62 ° C.
- Bereiten Sie eine Polystyrol Ringform, indem die Membran aus einem 12mm Durchmesser Millicell Kultur Platte gut einfügen und behalten Polystyrol Ring als Agarose Form zu verwenden. Weichen Sie die Ringe über Nacht in RNase-Inhibitor-Lösung vor jedem Gebrauch.
- Um eine glatte Schnittfläche, Rost-Inhibitor und anderen Zusätzen aus der Oberfläche des Wilkinson Klinge durch Spülen mit folgenden Lösungsmitteln, bei 100% Konzentration: Petrolether, Xylol, Chloroform, Methanol und MilliQ Wasser. Trennen Sie die Doppel-Blade-Länge nach in zwei einzelnen Blades.
- Halten die Wilkinson Klingen zu einem Mikrotom Klinge mit Loctite Kleber. Das Mikrotom Klinge ist nicht zum Schneiden verwendet, es fügt Steifigkeit der Wilkinson Klinge. 4mm von der Schneidkante der Klinge Wilkinson - Die Mikrotomklinge um die Länge der Klinge Wilkinson mit Blechscheren und einmal eingehalten geschnitten werden sollte, sollte um 3 ausgeglichen werden.
3. Dissection, Lagerung und Vorbereitung der Urogenital Gewebe zum Schneiden
- Bereiten PBSTw Lösung (PBS mit 0,1% Tween ™ 20 und 0,2 mM Natriumazid, durch die 0,22 &mgr; m gefiltert Stericup ® Filter-Einheit). Die Lösung kann im Voraus zubereitet und gelagert werden bei 25 ° C.
- Inkubieren einer frisch präparierten Maus LUT (Blase, Becken Harnröhre, und die damit verbundenen Wolffschen und Mϋllerian Kanal-derived Strukturen) über Nacht bei 4 ° C in PBS mit 4% Paraformaldehyd Fixativ.
- Entwässern Gewebe durch Waschen für 10 min bei 25 ° C in eine Reihe von abgestuften Methanol / PBSTw (1:3, 1:1, 3:1 v / v) Lösungen. Shop-Proben bei -20 ° C in 100% Methanol mindestens über Nacht. Archivierte Gewebe können mindestens 2 Jahre aufbewahrt werden.
- Bereiten Gewebe zum Schneiden von rückfettenden archiviert Gewebe. Wash für 10min bei 25 ° C in eine Reihe von abgestuften Methanol / PBSTw (3:1, 1:1, 1:3 v / v) Lösungen.
- Zerlegen und entsorgen Sie etwa zwei Drittel der Blase, so dass die meisten der Trigonum Region an der Harnröhre.
4. Embedding Urogenital Tissue in Agarose
- Legen Sie die Polystyrol Ringform, flache Oberfläche nach unten auf eine 25 ° C deutlich Glasobjektträger.
- Füllen Sie die Ringform mit 62 ° C erhitzten Agarose-Lösung und cool für ca. 2min.
- Entfernen LUT Gewebe aus PBSTw abspülen und auf einem saugfähigen Tuch trocken.
- Übertragung des Gewebes in Agarose-Lösung.
- Verwenden einer Pinzette zu orientieren LUT Gewebe in Agarose, so dass es auf halbem Weg zwischen dem oberen und unteren Rand des Ringform suspendiert und inkubieren des Gewebes bei 4 ° C, bis die Agarose erstarrt ist.
- Wenn das Gewebe sinkt vollständig während des Prozesses der Agarose Erstarrung, kann es aus der Agarose und re-embedded herausgeschnitten werden. Passen Sie die Kühlzeit der Agarose als während der re-Einbettung erforderlich.
5. Sectioning Urogenital Tissue mit einem vibrierenden Mikrotom (Basierend auf zuvor beschriebenen Protokoll) 7
- Montieren Sie die verstärkte Wilkinson Klinge in der vibrierenden Mikrotom und stellen Sie den Anstellwinkel bis 35 °. Füllen deluxe Probe Bad mit PBS und pack nassem Eis um die Probe Badewanne.
- Entfernen Sie die erstarrte Agarose Stecker aus der Ringform und blot die Bodenfläche mit einem saugfähigen Tuch. Stellen Sie sicher, dass das Gewebe richtig ausgerichtet ist. Gewebe Orientierung kann durch die Verwendung einer Rasierklinge zur Abschrägung der flachen Kante des Agarose-Stecker angepasst werden.
- Halten die Agarose Stecker auf einer vibrierenden Mikrotom Probe Tragscheibe mit Loctite Klebstoff, wie in Abb. gezeigt. 1A.
- Legen Sie die Probe Montage-Diskette in das Vibratom.
- Passen Sie das Mikrotom Schnittdicke bis 50 um, die Geschwindigkeit auf 2, und die Klinge Amplitude bis 4 und beginnen Schneiden von Gewebe Abschnitten.
- Verwenden Sie stumpfen Pinzette für jeden Gewebeschnitt (Abb. 1B) zu einer 24-well Platte gut, dass eiskalter 0,5 ml PBSTw enthält übertragen.
- Zur Vorbereitung Proben für in-situ-Hybridisierung, Verbrauchsteuern meisten der Agarose um jeden Gewebeschnitt (die restlichen Agarose wird während der ISH Verfahren Schmelze) und entfernen Sie alle damit verbundenen Schmutz. Shop Abschnitten bis zu 48 Stunden bei 4 ° C in PBSTw.
6. Beispiel Basket Vorbereitung zur in situ-Hybridisierung
- Schneiden Sie den Boden einer Mikrozentrifugenröhrchen auf 100 &mgr; markieren.
- Erhitzen Sie die Schnittkante des Rohres in einer Flamme, bis der Kunststoff weich und drücken Sie dann die Mikrozentrifugenröhrchen fest auf die Mitte eines 0.5in Polyester-Mesh-Platz.
- Schneiden Sie überschüssige Gewebe und mit einem beheizten 18-Gauge-Nadel zu durchstechen zwei Löcher in jedes Röhrchen Deckel in den Warenkorb Vorbereitung (Abb. 1C) abgeschlossen.
- Entfernen Sie den Deckel eines 24-Well-Platte und bohrt ein 12mm Loch über jedes gut zentriert. Verwenden Sie den Deckel auf Probe Körben zwischen wäscht der ISH-Protokoll (Abb. 1D) zu übertragen.
7. Embryo Pulveraufbereitung zur in situ-Hybridisierung (Basierend auf zuvor beschriebenen Protokoll) 8
- Sammeln Maus-Embryo Gewebe von Mäusen, die die gleiche Stufe wie die Gewebeschnitte, die zu beurteilenden sind und bei -80 ° C Legen gefrorenen Gewebe in einen Mörser aus Keramik, tauchen Gewebe in flüssigem Stickstoff, und verwenden Sie einen Mörser, um Gewebe zu einem feinen Pulver zermahlen.
- Kombinieren Embryo-Pulver mit 4 Volumen Aceton versetzt und mit mehreren Schlägen eines Dounce Homogenisator.
- Transfer-Homogenat zu einem 15mL Glas mit Schraubverschluss Fläschchen und extrahieren Nacht bei 4 ° C.
- Pellet Embryo-Pulver durch Zentrifugation bei 5000rpm für 10min bei 4 ° C. Entfernen und Entsorgen der Lipid-haltigen Überstand. Resuspendieren Gewebe Pellet in 4vol von frischem Aceton und Extrakt für 2 Std. bei 4 ° C.
- Pellet der Embryo-Pulver durch Zentrifugation bei 5000rpm für 10min bei 4 ° C. Entfernen und den Überstand verwerfen.
- An der Luft trocknen Pellets auf eine # 2 Whatman Filterpapier. Crush Pellet zu einem feinen Pulver und lagern in einem dicht verschlossenen Glasgefäß bei 4 ° C. Ausbeute Die ungefähre Ausbeute beträgt 50 mg Pulver pro 1 g Embryo Nassgewicht.
8. In-Situ-Hybridisierung Tag 1
- Vorheizen Prähybridisierungslösung (50% Formamid, 5x SSC, 1% Blocking Reagenz, 10μg/mL Hefe tRNA, 10μg/mL Heparin bei -20 ° C) auf 60,5 ° C. Diese Lösung kann im Voraus zubereitet werden und bei -20 ° C.
- Bereiten Sie eine befeuchtete Hybridisierung Kammer, indem eine kleine Kunststoff-Vorratsbehälter mit ca. 0,5 in Leitungswasser. Deckel Behälter und Vorwärmung auf 60,5 ° C.
- Add 2 ml PBSTw in die Vertiefungen einer 24-well Platten. Wägegut Körbe in die Löcher der 24-Well-Platte Deckel und Transfer Gewebeschnitten in die Körbe (bis zu 10 Abschnitten pro Korb verwendet wurden).
- Inkubieren Gewebeschnitte für 30 min bei 25 ° C in 6% H 2 O 2. Dieser und alle nachfolgenden Inkubationen sollte mit leichtem Schütteln auf einem Schüttler durchgeführt werden, sofern nicht anders angegeben. Alle Inkubationen eind Waschungen sind in 24-Well-Platten durchgeführt und mit einem Gesamtvolumen der Lösung von 2mL/well.
- Wash Gewebeschnitten 4 x 5 min bei 25 ° C in PBSTw.
- Inkubieren Gewebeschnitten für 12min bei 25 ° C in PBSTw mit 5μg/mL Proteinase K.
- Wash Gewebeschnitten 1 x 5 min bei 25 ° C in PBSTw.
- Post-fix Gewebeschnitten für 20min bei 25 ° C in PBS mit 4% Paraformaldehyd und 0,2% Glutaraldehyd.
- Wash Gewebeschnitten 2 x 5 Minuten bei 25 ° C in PBSTw.
- Add 2mL/well der vorgewärmten Prähybridisierungspuffer und inkubieren Gewebeschnitten innen befeuchtet Hybridisierung Kammer für mindestens 1 Stunde bei 60,5 ° C.
- Add 0.65μg gekennzeichnet Ribosonde die Prähybridisierungspuffer in jede Vertiefung und inkubieren Gewebeschnitte über Nacht in die befeuchtete Hybridisierung Kammer bei 60,5 ° C.
9. In-Situ-Hybridisierung Tag 2
- Bereiten Sie die folgenden Lösungen für die Post-Hybridisierung Waschschritte: Lösung 1 (50% Formamid, 5x SSC, 1% SDS), Lösung 2 (10 mM Tris-HCL pH 7,5, 0,5 M NaCl, 0,1% Tween ™ 20, 0,2 mm Natriumazid , 0,22 &mgr; m gefiltert) und Lösung 3 (2x SSC, 50% Formamid). Diese Lösungen können im Voraus vorbereitet werden. Die Lösungen 1 und 3 sind bei -20 ° C und Lösung 2 gespeichert ist, bei 25 ° C gelagert Die Haltbarkeit der Lösungen ist mindestens 3 Monate.
- Wash Gewebeschnitte 3 X 30 min bei 60,5 ° C mit vorgewärmten Lösung 1. Verwenden Sie die feuchten Kammer während wäscht.
- Wash Gewebeschnitten 1 X 10 min bei 60,5 ° C mit vorgewärmten Lösung 1/Solution 2 (1:1 v / v) Lösung. Verwenden Sie die feuchten Kammer während des Waschvorgangs.
- Wash Gewebeschnitten 4 X 10 Minuten bei 25 ° C mit Lösung 2.
- Inkubieren Gewebe Abschnitte für 15min bei 37 ° C in Lösung 2 enthält 0.25μg/mL RNase.
- Wash Gewebeschnitten 1 X 10 Minuten bei 25 ° C mit Lösung 2 (ohne RNase).
- Wash Gewebeschnitten 1 X 10 Minuten bei 25 ° C mit Lösung 3, um 2 X1hr gefolgt Wäschen bei 60,5 ° C mit Lösung 3. Verwenden Sie die feuchten Kammer bei der 60,5 ° C wäscht.
- Bereiten Sie die folgenden Lösungen für die immunhistochemische Detektion der DIG-markierten Ribosonden: Tissue Blocking Buffer (TB, 1X TBS, 10% Schafserum, 1% Blocking Reagenz, 1% BSA, 0,1% Tween ™ 20, 0,22 &mgr; m gefiltert), Antikörperverdünnung Buffer (AD, 1xTBS, 5% Schafserum, 1% Blocking Reagenz, 1% BSA, 0,1% Tween ™ 20, 0,2 mm Natriumazid, 0,22 ™ m gefiltert) Antikörper Absorption Buffer (AA, 1xTBS, 5% Schafserum, 1 % Blocking Reagenz, 1% BSA, 6mg/mL Embryo-Pulver) und TBSTw (1xTBS, 0,1% Tween ™ 20, 0,2 mm Natriumazid, gefiltert 0,22 &mgr; m). Diese Lösungen können im Voraus vorbereitet werden. TBSTw bei 25 ° C gelagert wird, sind alle anderen Lösungen bei -20 ° C gelagert
- Wash 3 X 10 min bei 25 ° C mit TBSTw.
- Inkubieren Sie die Gewebeschnitte mindestens 2 Std. bei 25 ° C in TB-Puffer.
- Während Gewebe in TB-Puffer inkubiert, fügen 1.1μL anti-DIG Antikörper pro 200 ul AA-Puffer für jeden gut. Inkubieren AA-Puffer + Antikörper mindestens 2 Std. bei 4 ° C, dann bei 10.000 rpm für 1min Zentrifuge. Entfernen Sie die AA-Puffer Überstand und fügen Sie diese 2 ml AD-Puffer.
- Entfernen Sie Gewebeschnitte von TB-Puffer und inkubieren sie über Nacht in einer feuchten Kammer bei 4 ° C in AD-Puffer mit Antikörper.
10. In-Situ-Hybridisierung Tag 3
- Bereiten Farbentwicklung Lösung NTMT (100 mM Tris-HCL pH 9,5, 100 mM NaCl, 50 mM MgCl 2, 0,2 mM Natriumazid, 0,22 &mgr; m gefiltert). Diese Lösung kann im Voraus zubereitet und gelagert werden bei 25 ° C. Unmittelbar vor der Verwendung, fügen 2mM Levamisol und 0,1% Tween ™ 20.
- Entfernen der Antikörper-Lösung (AD-Puffer + Antikörper) aus dem Brunnen und Shop-Lösung bei 4 ° C. Es kann bis zu zwei weitere Male wiederverwendet werden.
- Waschen Sie den Geweben 8 X 10 min bei 25 ° C mit TBSTw mit 2mM Levamisol.
- Sorgfältig Transfer Gewebe aus den Körben auf eine Petrischale mit TBSTw. Verwenden einer Pinzette, um sichtbare Verschmutzungen zu entfernen. Übertragen Sie die Gewebe in saubere Reaktionsgefäße.
- Wash Gewebe 1 X 10 min bei 25 ° C mit 1 ml NTMT.
- Entfernen Sie die NTMT und fügen 1mL/tube einer Mischung mit 50% NTMT (mit 2 mM Levamisol) und 50% BM Purple, statt die Röhrchen in einem Licht geschützt Feld und inkubieren bei 25 ° C. Farbe Entwicklungszeit im Bereich von mehreren Stunden bis mehrere Tage. Wenn Farbe nur langsam entwickeln, können 100% BM Lila verwendet werden.
- Monitor Farbe Entwicklungs-und Veränderungsprozessen NTMT / BM Lila Lösung, wenn es sich ansammelt ausgefallenen Kristalle oder wenn es erfährt Farbumschlag von gelb bis violett. Nach der vollständigen Farbentwicklung (4-250 Stunden), waschen Gewebe 2 X 5 min bei 25 ° C mit 1mL/tube von NTMT mit 2mM Levamisol.
- Inkubieren Gewebe über Nacht bei 4 ° C in 1mL/tube PBS mit 4% Paraformaldehyd post-Fixativ.
- Zum Bleichen der Gewebe, inkubieren Gewebe für 30 min bei 25 ° C in 1mL/tube von PBSTw mit 3% H 2 O 2 . Dann waschen Sie den Geweben 1 X 10 Minuten bei 25 ° C in 1mL/tube von PBSTw und lagern bei 4 ° C in 1mL/tube PBS mit 4% Paraformaldehyd post-Fixativ.
- Gewebeschnitte werden auf Glasträger montiert, eingedeckt und bebildert mit einem zusammengesetzten Mikroskop.
11. Repräsentative Ergebnisse:
Die räumliche Orientierung der LUT Gewebe in Agarose bestimmt die Ebene von Gewebeschnitten. Für Sagittalschnitten wird mindestens zwei Drittel der Blase entfernt und die verbleibenden LUT Gewebe wird in Agar, so dass die Harnröhre Mittellinie parallel zur flachen Oberfläche der Agarose-Stecker (Abbildung 1A) eingebettet ist. Kleinere Anpassungen in das Gewebe Ebene kann durch Abschrägung der flachen Kante des Agarose-Stecker hergestellt werden. Ein Vertreter Sagittalschnitt von einem 17.5dpc männlichen LUT Gewebe, das ausgerichtet ist auf dieser Ebene ist in Abbildung 1B gezeigt.
Beispiel Körbe schützen das empfindliche Gewebe Abschnitte vor Verlust und die Ansammlung von Staub und Feinstaub während des mehrtägigen ISH Verfahren. Beispiel Körbe werden durch Schmelzen Polyester-Mesh, um das abgeschnittene Ende eines 1,5 ml Mikrozentrifugenröhrchen (Abbildung 1C) vorbereitet. Ein kleines Loch in den Deckel von jeder Probe Korb Lösung fließen und aus den Körben erleichtern durchbohrt. Beispiel Körbe sind in ISH Lösungen, indem sie in 12mm Löcher in 24-Well-Platte Deckel (Abbildung 1D) gebohrt ausgesetzt. Die modifizierte Platte Deckel Unterstützung Körbe, wenn sie zwischen 24-Well-Platten werden während Lösung Veränderungen übertragen.
Es ist eine Herausforderung, um unspezifische Hintergrundfärbung während der langen Inkubationszeit für den Nachweis von geringen Mengen vorhandenen mRNAs erforderliche Maß zu beschränken. Der Zusatz von 0,2 mM Natriumazid zu Probenpuffer und deren anschließende Filtration durch 0,22 &mgr; m-Filter erschienen, um die Hintergrundfärbung (Abb. 2) zu begrenzen. Mit der hier beschriebenen Methode, scheint es nicht zu sichtbaren Unterschieden in Hintergrundfärbung werden, wenn die Proben in Farbe Lösung für die Entwicklung über einen längeren Zeiträumen (Abbildung 3) inkubiert werden.

Abbildung 1. Vorbereitung einer Maus urogenitalem (LUT) Trakt Gewebeschnitt und Mikrozentrifugenröhrchen ein Korb für die ISH. Eine LUT enthält Teil der Blase, Becken Harnröhre und der damit verbundenen Wolffschen und Mϋllerian Kanal-derived-Struktur) ist in einem zylindrischen Stopfen von 4% mit niedrigem Schmelzpunkt Agarose eingebettet. (A) Der Stecker wird auf eine Probe Tragscheibe verklebt und (B) in 50 um Abschnitte mit einem vibrierenden Mikrotom geschnitten. (C) Ein LUT Abschnitt ist in einem Mikrozentrifugenröhrchen Korb, durch Einstechen ein Loch in das Rohr Deckel und Absicherung Polyester-Mesh, um den Schnitt am unteren Ende des Rohres vorbereitet übertragen wird. (D) Die Mikrozentrifugenröhrchen in 12mm Löcher in eine 24-Well-Platte Deckel gebohrt, so dass Gewebeschnitte in Pufferlösung während der ISH-Protokoll ausgesetzt werden eingefügt. Pfeilspitzen zeigen die LUT Gewebe in der Agarose-Stecker.
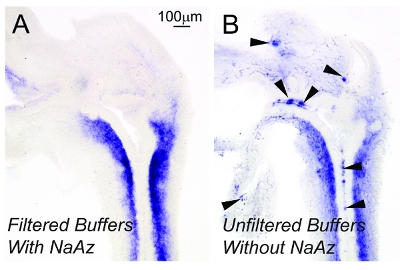
Abbildung 2. Incorporation von 0,2 mm Natriumazid in 0,22 &mgr; m gefiltert Lösungen verbessert Tissue-Qualität und senkt Hintergrundfärbung. 17.5dpc männliche Maus unteren Urogenitaltraktes (LUTs) wurden im Schnitt in einer Sagittalebene zu einer Dicke von 50 um. Gewebeschnitte wurden von ISH mit einer Sonde gegen Twist Homolog 1 gerichtet gefärbt. Puffer für ISH verwendet wurden entweder (A) 0,22 &mgr; m gefiltert und ergänzt mit 0,2 mM Natriumazid (Naaz) oder (B) ungefiltert und nicht mit Naaz ergänzt. Pfeilspitzen zeigen Hintergrundfärbung. Die Bilder wurden mit der gleichen Vergrößerung aufgenommen. Ergebnisse sind repräsentativ Färbungsmuster für n = 3 Wurf-unabhängige Mäusen.
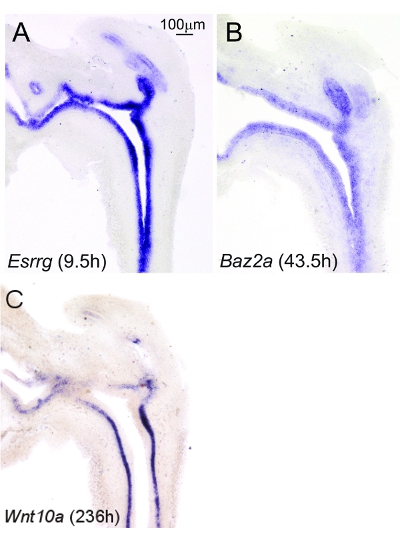
Abbildung 3. Hintergrundfärbung Intensität scheint nicht mit einer längeren Farbe Entwicklung zu erhöhen. 17.5dpc männliche Maus unteren Urogenitaltraktes (LUTs) wurden im Schnitt in einer Sagittalebene zu einer Dicke von 50 um. Die Schnitte wurden durch ISH durch Inkubation in Chromagen Färbelösung für (A) 9.5h mit einer Sonde, dass die hohe Fülle Transkript Östrogen-related receptor gamma (Esrrg) erkennt, (B) für 43.5h mit einer Sonde, die das Medium Fülle erkennt gebeizt Transkript Bromodomäne neben Zink-Finger-Domäne, 2A (Baz2a), oder (C) für 236H mit einer Sonde, dass die geringe Menge Transkript flügellosen-type MMTV Integration site Familie, 10a (Wnt10a) erkennt. Die Bilder wurden mit der gleichen Vergrößerung aufgenommen. Ergebnisse sind repräsentativ Färbungsmuster für n = 3 Wurf-unabhängige Mäusen.
Access restricted. Please log in or start a trial to view this content.
Diskussion
Mit dem hier beschriebenen Verfahren ist es möglich, mRNAs in allen wichtigen Zelltypen und Gewebe Fächer des fetalen männlichen und weiblichen Mäusen LUTs inklusive der mesenchymalen Pads, Urothel, glatte Muskelzellen, Prostata-Knospen, Ductus ejaculatorius und Vagina zu erkennen. Der 50 um Abschnitte in diesem Protokoll haben den Vorteil, dass dick genug, um Gewebe-Architektur (z. B. Blutgefäße) zu lösen, sind aber dünn genug, um Sonde Trapping, die ein methodisches Problem häufig während der gesamten ISH-mo...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Die Autoren bedanken sich bei Dr. Lan Yi, Cancer Institute of New Jersey, um technische Unterstützung zu danken, bei der Vorbereitung Gewebe Körbe. Diese Arbeit wurde vom National Institutes of Health Grants DK083425 und DK070219 finanziert.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenzes | Firma | Katalog-Nummer | |
|---|---|---|---|
| Anti-Digoxigenin-Antikörper, Fab-Fragmente | Roche Applied Science | 11214667001 | |
| Blockierungsreagenz | Roche Applied Science | 11096176001 | |
| BM Lila AP Substrat, Ausfällen | Roche Applied Science | 11442074001 | |
| Bovine Serum Albumin | Fisher Scientific | BP1600-100 | |
| Zellkulturplatte, 24 und | Corning | 3524 | |
| Digoxigenin 11-UTP | Roche Applied Science | 1277073910 | |
| dNTPs | Roche Applied Science | 11969064001 | |
| Zweischneidige Rasierklinge | Wilkinson Sword | Klassisches Modell | |
| Eliminase RNase-Entferner | Decon Laboratories | 1102 | |
| Formamid | Sigma | F5786-1L | |
| Gel Extraction Kit | Qiagen | 28704 | |
| Glutaraldehyd, 25% ige Lösung in H 2 O | Sigma | G6257-100ML | |
| Heparin, Natriumsalz | Sigma | H3393 | |
| Wasserstoffperoxid, 30% ige Lösung in H 2 O | Fisher Scientific | BP2633-500 | |
| Levamisol | Sigma | L9756 | |
| Loctite 404 schnelle Einrichtung Sekundenkleber | Henkel Corp | 46551 | |
| Magnesiumchlorid | Fisher Scientific | M33-500 | |
| Maleinsäure | Sigma | M0375-500G | |
| Reaktionsgefäße, 1,5 ml | Biologix Research Company | BP337-100 | |
| Millicell Kultur Platte einfügen | Millipore | PICM01250 | |
| Molecular Anreibeharz | G-Biosciences | 786-138PR | |
| Paraformaldehyd, 4% ige Lösung in Phosphat-gepufferter Kochsalzlösung | Affymetrix | 19943 | |
| Phosphat-gepufferte Salzlösung, ohne Ca und Mg | MP Biomedicals | ICN1760420 | |
| Polyester Mesh, 33 micron, 12 "x 24" | Kleinteile Inc | CMY-0033-D | |
| Proteinase K-Lösung 20mg/ml | Amresco | E195-5ML | |
| QIAshredder Columns | Qiagen | 79654 | |
| Q-Lösung | Qiagen | Ausgestattet mit Taq DNA Polymerase | |
| RNase | Sigma | R6513 | |
| RNase-Inhibitor | Roche Applied Science | 03335399001 | |
| RNeasy Mini Kit | Qiagen | 74104 | |
| RNeasy Mini Kit | Qiagen | 74104 | |
| RQ1 RNase-freie DNase | Promega | M6101 | |
| SeaPlaque Low-Melt Agarose | Lonza | 50101 | |
| Sheep Serum | Sigma | S2263-500ml | |
| Stericup Filtereinheit, 0,22 &mgr; m, Polyethersulfon, 500ml | Millipore | SCGPU05RE | |
| Natriumazid, körnige | Fisher Scientific | S227I-100 | |
| Kochsalz | Fisher Scientific | BP358-212 | |
| Natriumdodecylsulfat | Fisher Scientific | S529-500 | |
| SSC, 20X-Lösung | Forschung Products International | S24022-4000,0 | |
| SuperScript III Erststrangsynthese System | Invitrogen | 18080-051 | |
| T7-RNA-Polymerase | Roche Applied Science | 10881767001 | |
| Taq DNA Polymerase | Qiagen | 201203 | |
| Tris-HCl | Fisher Scientific | BP153-1 | |
| Tween 20 | Fisher Scientific | BP337-100 | |
| Vibrating Mikrotom mit Deluxe-Exemplar Bad | Leica Microsystems | VT1000A | |
| Hefe tRNA | Roche Applied Science | 109495 |
Referenzen
- Cunha, G. R. The possible influence of temporal factors in androgenic responsiveness of urogenital tissue recombinants from wild-type and androgen-insensitive (Tfm) mice. Journal of Experimental Zoology. 205, 181-181 (1978).
- Cunha, G. R. Hormonal, cellular, and molecular regulation of normal and neoplastic prostatic development. Journal of Steroid Biochemistry and Molecular Biology. 92, 221-221 (2004).
- Price, D. Comparative aspects of development and structure in the prostate. National Cancer Institute Monographs. 12, 1-1 (1963).
- Cunha, G. R. Stromal-epithelial interactions--I. Induction of prostatic phenotype in urothelium of testicular feminized (Tfm/y) mice. Journal of Steroid Biochemistry. 14, 1317-1317 (1981).
- Rozen, S. Primer3 on the WWW for general users and for biologist programmers. Methods in Molecular Biology. 132, 365-365 (2000).
- Zhang, Z. A greedy algorithm for aligning DNA sequences. Journal of Computational Biology. 7, 203-203 (2000).
- Vezina, C. M. Dioxin causes ventral prostate agenesis by disrupting dorsoventral patterning in developing mouse prostate. Toxicological Sciences. 106, 488-488 (2008).
- Wilkinson, D. G. Detection of messenger RNA by in situ hybridization to tissue sections and whole mounts. Methods in Enzymology. 225, 361-361 (1993).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten