É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
A High Throughput In situ Método de hibridização para caracterizar padrões de expressão de mRNA no Rato Fetal Trato urogenital inferior
Neste Artigo
Resumo
Aqui, descrevemos uma produção eficiente de alta In situ Hibridação método (ISH) para a visualização de padrões de expressão de mRNA no desenvolvimento fetal do rato secções de tecido da próstata. O método pode ser facilmente adaptado para visualizar padrões de expressão de mRNA nos tecidos dos camundongos outros ou em tecidos de outras espécies.
Resumo
Desenvolvimento do trato urogenital (LUT) é um processo complexo. Essa complexidade é evidenciada durante a formação da próstata a partir da uretra fetal masculino, que se baseia em sinais androgênicos e epitelial-mesenquimal 1,2 interações. Compreensão dos mecanismos moleculares responsáveis pelo desenvolvimento da próstata pode revelar os mecanismos de crescimento que são inadequadamente despertado mais tarde na vida para dar origem a doenças da próstata como a hiperplasia prostática benigna e câncer de próstata.
A LUT desenvolvimento é anatomicamente complexa. Pelo tempo de brotamento da próstata começa na concepção pós 16,5 dias (DPC), numerosos tipos de células estão presentes. Vasculatura, nervos e músculos lisos residir no estroma mesenquimal 3. Este estroma envolve um epitélio multicamada e dá origem à próstata fetal através de receptor de andrógeno-dependentes sinais parácrinos 4. A identidade do receptor de andrógeno estroma-responsive genes necessários para a próstata desenvolvimento eo mecanismo pelo qual as formas de próstata epitélio ductal em resposta a esses genes não é totalmente compreendido. A capacidade de identificar com precisão os tipos de células e localizar a expressão de factores específicos dentro deles é imperativo para entender melhor o desenvolvimento da próstata. A hibridização in situ (ISH) permite a localização de mRNAs dentro de um tecido. Assim, este método pode ser usado para identificar padrões e tempo de expressão de moléculas sinalizadoras e seus receptores, elucidando assim reguladores de próstata potencial de desenvolvimento.
Aqui, descrevemos uma técnica de ISH alta taxa de transferência para identificar padrões de expressão de mRNA na LUT rato fetal usando vibrando micrótomo de corte seções. Este método oferece diversas vantagens sobre outros protocolos de ISH. Realizar ISH em seções finas adere a um slide é tecnicamente difícil; criosecções freqüentemente têm qualidade estrutural pobres, enquanto ambos os criosecções e seções de parafina muitas vezes resultam em resolução do sinal fraco. Realizar ISH em tecidos todo suporte pode resultar em aprisionamento sonda. Em contraste, a nossa técnica de alto rendimento utiliza espessura de corte seções que revelam a arquitetura do tecido detalhadas. Tubos de microcentrífuga modificada permitir fácil manuseio das seções durante o procedimento ISH. Um máximo de 4 transcrições de mRNA podem ser rastreados a partir de um único LUT 17.5dpc com até 24 transcrições de mRNA detectados em uma única corrida, reduzindo custos e maximizando a eficiência. Este método permite que múltiplos grupos de tratamento para ser processado de forma idêntica e como uma única unidade, eliminando assim qualquer viés de interpretação dos dados. Mais pertinente para os pesquisadores de próstata, este método fornece uma localização espacial e temporal de transcritos mRNA baixa e alta abundância na uretra fetal do rato que dá origem à rede ductal da próstata.
Protocolo
1. Síntese de uma Riboprobe digoxigenina-11-UTP-identificadas a partir de um modelo de PCR-Generated
- Para sintetizar uma riboprobe gene específico, use Entrez Gene (http://www.ncbi.nlm.nih.gov/sites/entrez) para obter o gene seqüência de referência cDNA (RefSeq). Use o programa Primer3 (http://frodo.wi.mit.edu/primer3/) 5 para projetar primers gene-específicos PCR contra a região 3'-da seqüência de cDNA. Parâmetros recomendados para a seleção de primer PCR estão descritos em outra parte (http://www.gudmap.org/Research/Protocols/Vezina/Riboprobe_Syn.html).
- Utilizar o Programa Megablast (http://www.ncbi.nlm.nih.gov/genome/seq/BlastGen/BlastGen.cgi?taxid=10090) 6 para avaliar a especificidade da sequência de DNA selecionado. A seqüência de DNA é considerado específico quando, utilizando um limiar de 0,01 ESPERAR, não alinhar com outras seqüências no banco de dados RefSeq.
- PCR amplificar o modelo riboprobe. Componentes reação de PCR e as condições de termociclagem devem ser otimizados para cada conjunto de primers. Uma reação típica 50 mL contém: tampão 1X, 2mM MgCl 2, 0,2 mM dNTPs, 1X solução Q, 1μg cDNA, Taq DNA polimerase 2.5U, primers 0.25μm, e livre de nuclease H 2 O. Um protocolo de termociclagem típico inclui uma desnaturação inicial a 94 ° C por 2min, seguida de 40 ciclos de 94 ° C por 30s, 57 ° C por 30s, 72 ° C por 1min e uma extensão final a 72 ° C por 10 min. O cDNA utilizado nas reações de PCR é sintetizada a partir de mRNA do mouse urogenital.
- Separar o produto de PCR por eletroforese em gel de agarose, purificar utilizando o Kit de Extração de Gel, e quantificar os produtos purificados por espectrofotometria. O rendimento esperado é de 1,2 - 3.6μg.
- Transcrever o produto de PCR em um riboprobe rotulados. A reação de transcrição (40 mL) contém: 400ng purificada PCR produto, 1X mix rotulagem de nucleotídeos contendo digoxigenina 11-UTP, 1X tampão de transcrição, 5U RNase inibidor, 80U T7 RNA polimerase, e livre de nuclease H 2 O. Incubar 3 a 37 amostras 4hr ° C e agitar todos os 30min.
- Use o kit Qiagen RNEASY Mini para purificar riboprobes com base em instruções para limpeza com RNA na coluna digestão DNase. Quantificar riboprobes por espectrofotometria. O rendimento esperado é de 4-20 mg. Avaliar a qualidade da sonda, separando uma alíquota por eletroforese em gel de agarose 1,5% não desnaturante. Sondas de alta qualidade migrar como bandas distintas, com manchas mínimas.
- Para garantir a riboprobe reconhece especificamente o seu alvo, incluir um tecido de controle positivo para o primeiro experimento ISH. O padrão riboprobe do mRNA alvo deve ser conhecido neste tecido controle positivo.
2. Preparação de vibração da lâmina micrótomo (Baseado no protocolo descrito anteriormente) 7
- Prepare a 4% de baixa derreter solução de agarose em tampão fosfato salino (PBS). Microondas solução para dissolver agarose e manter solução a 62 ° C.
- Prepare um molde do anel de poliestireno, removendo a membrana de uma placa de cultura 12 milímetros de diâmetro Millicell bem inserir e reter poliestireno anel para usar como um molde de agarose. Mergulhe os anéis durante a noite em RNase solução inibidora antes de cada utilização.
- Para garantir uma superfície de corte mais suave, retire inibidor de ferrugem e outros aditivos a partir da superfície da lâmina Wilkinson por lavagem com os seguintes solventes, numa concentração de 100%: éter de petróleo, xileno, clorofórmio, metanol e água MilliQ. Separar a lâmina dupla longitudinalmente em duas lâminas único.
- Aderir as lâminas Wilkinson para uma lâmina de micrótomo com adesivo Loctite. A lâmina de micrótomo não é usado para o corte, que acrescenta rigidez à lâmina Wilkinson. A lâmina de micrótomo deve ser cortado ao comprimento da lâmina Wilkinson usando uma tesoura de metal e, uma vez aderido, devem ser compensados por 3 - 4 mm da ponta da lâmina Wilkinson.
3. Dissecção, Armazenamento, Preparação de tecidos Urogenital para Seccionamento
- Preparar a solução PBSTw (PBS contendo 0,1% Tween 20 e 0,2 mM ™ azida de sódio, filtrada através do Stericup 0.22μm ® unidade de filtro). Solução pode ser preparado com antecedência e armazenado a 25 ° C.
- Incubar uma LUT rato recém-dissecado (bexiga, uretra pélvica e associados Wolff e Mϋllerian duto-estruturas derivadas) durante a noite a 4 ° C em PBS contendo fixador paraformaldeído a 4%.
- Desidratar tecidos de lavagem para 10 minutos a 25 ° C em uma série de metanol classificados / PBSTw (1:3, 1:1, 3:1 v / v) soluções. Armazene as amostras a -20 ° C em metanol 100%, pelo menos, durante a noite. Tecidos arquivados podem ser mantidos pelo menos 2yr.
- Prepare tecidos para corte através da hidratação de tecidos arquivados. Lavagem para 10 minutos a 25 ° C em uma série de metanol classificados / PBSTw (3:1, 1:1, 1:3 v / v) soluções.
- Dissecar e descartar aproximadamente dois terços da bexiga, deixando a maioria da região do trígono ligado à uretra.
- Coloque o anel de molde de poliestireno superfície plana para baixo, em um 25 ° C lâmina de microscópio simples de vidro.
- Preencher o molde do anel com 62 ° C e solução de agarose esfriar por cerca de 2min.
- Remover o tecido LUT de PBSTw e secar sobre um absorvente wipe.
- Transferir o tecido em solução de agarose.
- Use uma pinça para orientar o tecido LUT em agarose para que seja suspensa a meio caminho entre a parte superior e inferior do molde do anel e incubar o tecido a 4 ° C até que a agarose solidificou.
- Se o tecido afunda completamente durante o processo de solidificação agarose, ele pode ser retirado a partir do agarose e re-incorporado. Ajustar o tempo de resfriamento do agarose, conforme necessário durante o processo de re-incorporação.
5. Seccionamento do tecido urogenital em micrótomo vibratório (Baseado no protocolo descrito anteriormente) 7
- Monte a lâmina Wilkinson reforçada no micrótomo vibrando e definir o ângulo da lâmina a 35 °. Preencha banho espécime de luxo com PBS e gelo molhado em torno do banho espécime.
- Remova o plugue agarose solidificada do molde anel e seque a superfície inferior com um absorvente wipe. Verifique se o tecido está correctamente orientado. Orientação tecidos podem ser ajustados usando uma lâmina de barbear chanfrar a borda lisa do plug agarose.
- Aderem a ficha de agarose em um espécime micrótomo vibrando de montagem do disco com adesivo Loctite como mostrado na figura. 1A.
- Insira o disco de amostra de montagem nos vibratome.
- Ajustar a espessura de corte micrótomo a 50 ìm, a velocidade a 2, ea amplitude da lâmina a 4 e começar a cortar secções de tecido.
- Use uma pinça blunt para transferir cada secção de tecido (Figura 1B) a uma placa de cultura de 24 poços que contém bem gelada PBSTw 0,5 ml.
- Para preparar amostras para hibridização in situ especiais de consumo, a maioria dos agarose em torno de cada secção de tecido (o restante agarose vai derreter durante o procedimento ISH) e remover todos os detritos associados. Seções armazenar até 48hr a 4 ° C em PBSTw.
6. Preparação da amostra Basket Por Hibridização In Situ
- Corte a parte inferior de um tubo de microcentrífuga na marca de 100μL.
- Aqueça a borda do corte do tubo em uma chama até que o plástico é amolecida, em seguida, pressione o tubo de microcentrífuga firmemente no centro de um quadrado de poliéster 0.5in malha.
- Caimento de malha em excesso e usar uma agulha de calibre 18 aquecida para perfurar dois furos em cada tampa do tubo para completar a preparação cesto (Fig. 1C).
- Remova a tampa de uma placa de 24 poços e um furo centrado sobre 12 milímetros cada poço. Use a tampa para transferência de cestas de amostra entre as lavagens do protocolo de ISH (Figura 1D).
7. Preparação embrião Pó Para Hibridização In Situ (Baseado no protocolo descrito anteriormente) 8
- Coletar tecido do embrião de rato de ratos que são mesmo palco que as secções de tecido que estão sendo avaliados e armazenar a -80 ° C. Coloque tecido congelado em uma cerâmica tecido submergir argamassa, em nitrogênio líquido, e usar um pilão para moer o tecido em um pó fino.
- Combine pó embrião com 4 volumes de acetona e homogeneizar com várias pinceladas de um homogeneizador Dounce.
- Transferência homogeneizado a um vidro 15mL com tampa do frasco e extrato de overnight a 4 ° C.
- Pellet pó embrião por centrifugação a 5000rpm por 10min a 4 ° C. Remova e descarte o sobrenadante contendo lipídios. Ressuspender o sedimento em tecidos 4vol de acetona fresco e extrato para 2hr a 4 ° C.
- Pellet o pó embrião por centrifugação a 5000rpm por 10min a 4 ° C. Remova e descarte o sobrenadante.
- O ar seco do sedimento em um papel Whatman n º 2 do filtro. Esmagamento de pelotas para produzir um pó fino e armazenar em um frasco de vidro hermeticamente fechado, a 4 ° C. O rendimento aproximado é de 50 mg de pó por um peso embrião g molhado.
8. No dia 1 Hibridização In Situ
- Pré-aqueça solução prehybridization (formamida 50%, SSC 5x, 1% de reagente de bloqueio, 10μg/mL levedura tRNA, 10μg/mL de heparina armazenar a -20 ° C) a 60,5 ° C. Esta solução pode ser preparado com antecedência e armazenado a -20 ° C.
- Preparar uma câmara de hibridação umidificado, preenchendo um pequeno recipiente de armazenamento de plástico com cerca de 0,5 em de água da torneira. Tampa do depósito e pré-aquecimento para 60,5 ° C.
- Adicionar 2mL PBSTw aos poços de uma placa de cultura de 24 poços. Coloque cestas amostra nos furos da tampa da placa de 24 poços e cortes de tecidos de transferência nos cestos (até 10 seções por cesta de ter sido utilizado).
- Incubar cortes de tecido por 30min a 25 ° C em 6% H 2 O 2. Esta e todas as incubações subseqüentes devem ser realizados com agitação suave em um agitador orbital, salvo indicação em contrário. Todas as incubações umd lavagens são realizadas em placas de 24 poços e usar um volume total de solução 2mL/well.
- Lavar cortes de tecidos 4 x 5min a 25 ° C em PBSTw.
- Incubar seções de tecido para 12min a 25 ° C em PBSTw contendo proteinase K. 5μg/mL
- Lavar cortes de tecido 1 x 5min a 25 ° C em PBSTw.
- Pós-fix cortes de tecido por 20min a 25 ° C em PBS contendo paraformaldeído 4% e glutaraldeído 0,2%.
- Lavar cortes de tecido 2 x 5min a 25 ° C em PBSTw.
- Adicionar 2mL/well do buffer prehybridization pré-aquecida e incubar secções de tecido no interior da câmara de hibridização umidificado por pelo menos 1h em 60,5 ° C.
- Adicionar riboprobe rotulados 0.65μg para o buffer prehybridization em cada secções de tecido bem e incubar durante a noite na câmara de hibridação umidificado em 60,5 ° C.
9. No dia 2 Hibridização In Situ
- Prepare as seguintes soluções para as etapas pós-hibridação lavagem: Solução 1 (formamida 50%, SSC 5x, SDS 1%), Solução 2 (10 mM Tris-HCL pH 7,5, NaCl 0,5 M, 0,1% Tween ™ 20, azida de sódio 0,2 mM , 0.22μm filtrada) e Solução 3 (2x SSC, 50% formamida). Essas soluções podem ser preparadas com antecedência. Soluções 1 e 3 são armazenadas a -20 ° C e Solução 2 é armazenado a 25 ° C. O tempo de armazenamento das soluções é de pelo menos 3 meses.
- Lavar cortes de tecido 3 X 30min em 60,5 ° C, com pré-aquecido a Solução 1. Use a câmara úmida durante a lavagem.
- Lavar cortes de tecido 1 X 10min em 60,5 ° C, com pré-aquecido 1/Solution Solução 2 (1:1 v / v). Use a câmara úmida durante a lavagem.
- Lavar cortes de tecidos 4 X 10 minutos a 25 ° C com a Solução 2.
- Incubar tecidos seções por 15min a 37 ° C em solução contendo 2 0.25μg/mL RNase.
- Lavar cortes de tecido 1 X 10min a 25 ° C com solução 2 (sem RNase).
- Lavar cortes de tecido 1 X 10min a 25 ° C com a solução 3, seguido por 2 X1hr lava em 60,5 ° C com a Solução 3. Use a câmara úmida durante a 60,5 ° C lavagens.
- Prepare as seguintes soluções para a detecção imunohistoquímica da riboprobes DIG marcado: Buffer Tissue bloqueio (TB, TBS 1X, 10% de soro de ovelha, 1% de reagente de bloqueio, BSA 1%, 0,1% Tween 20 ™, 0.22μm filtrada), diluição do anticorpo buffer (AD, 1xTBS, 5% de soro de ovelha, 1% de reagente de bloqueio, BSA 1%, 0,1% Tween ™ 20, azida de sódio 0,2 mM, 0,22 m ™ filtrada) Buffer de Absorção de anticorpos (AA, 1xTBS, 5% de soro de ovelha, uma reagente% de bloqueio, 1% BSA, pó embrião 6mg/mL), e TBSTw (1xTBS, 0,1% Tween 20 ™, 0,2 mM de azida de sódio, 0.22μm filtrada). Essas soluções podem ser preparadas com antecedência. TBSTw é armazenada a 25 ° C, todas as outras soluções são armazenadas a -20 ° C.
- Lavar 3 X 10 min a 25 ° C com TBSTw.
- Incubar as secções de tecido, pelo menos, 2hr a 25 ° C em tampão TB.
- Enquanto os tecidos são incubação em tampão TB, adicione anticorpo anti-DIG 1.1μL por 200μL tampão AA para cada poço. Incubar tampão AA + de anticorpos pelo menos 2hr a 4 ° C, em seguida, centrifugar a 10.000 rpm por 1 min. Retire o sobrenadante tampão AA e adicioná-lo 2mL de tampão AD.
- Remove seções de tecido de buffer de TB e incubar-los durante a noite em câmara úmida a 4 ° C em tampão contendo anticorpo AD.
10. No dia 3 Hibridização In Situ
- Prepare o desenvolvimento de soluções de cor NTMT (100mM Tris-HCL pH 9,5, 100mM NaCl, 50mM MgCl 2, 0,2 mM de azida de sódio, 0.22μm filtrada). Esta solução pode ser preparado com antecedência e armazenado a 25 ° C. Imediatamente antes de usar, adicione levamisole 2mM e 0,1% Tween 20 ™.
- Remover a solução de anticorpos (AD tampão anticorpo +) dos poços e solução de armazenamento a 4 ° C. Ele pode ser reutilizado até duas vezes adicionais.
- Lavar os tecidos 8 X 10 min a 25 ° C com TBSTw contendo 2mM levamisole.
- Transferir cuidadosamente tecidos dos cestos a uma placa de Petri contendo TBSTw. Use uma pinça para remover os restos visíveis. Transferir os tecidos em tubos de microcentrífuga limpo.
- Lavar tecidos 1 X 10 min a 25 ° C com NTMT 1mL.
- Remover o NTMT e adicione 1mL/tube de uma mistura contendo 50% NTMT (contendo levamisole 2mM) e 50% BM Purple, tubos lugar em uma caixa de luz protegida e incubar a 25 ° C. Tempo de desenvolvimento de cor varia de algumas horas a vários dias. Se a cor é lento para se desenvolver, 100% BM Purple pode ser usado.
- Acompanhar o desenvolvimento e mudança de cor NTMT / solução Roxo BM se acumula cristais precipitados ou se sofre mudança de cor do amarelo ao roxo. Após o desenvolvimento de cores (4-250 horas), lavagem de tecidos 2 X 5min a 25 ° C com 1mL/tube de NTMT contendo 2mM levamisole.
- Incubar tecidos durante a noite a 4 ° C em 1mL/tube de PBS contendo paraformaldeído a 4% pós-fixador.
- Para branquear os tecidos, tecidos incubar por 30min a 25 ° C em 1mL/tube de PBSTw contendo 3% H 2 O 2 . Em seguida, lave os tecidos 1 X 10min a 25 ° C em 1mL/tube de PBSTw e armazenar a 4 ° C em 1mL/tube de PBS contendo paraformaldeído a 4% pós-fixador.
- Secções de tecido são montados em lâminas de vidro, lamínulas e fotografada com um microscópio composto.
11. Resultados representativos:
A orientação espacial do tecido LUT em agarose determina o plano de cortes de tecido. Para cortes sagitais, pelo menos, dois terços da bexiga é extirpado e tecido LUT restante é incorporado em agar de tal forma que a linha média da uretra é paralela à superfície plana da ficha de agarose (Figura 1A). Pequenos ajustes no plano do tecido pode ser feita por chanfradura na borda plana do plug-agarose. A secção representativa sagital de um tecido LUT 17.5dpc macho que é orientado neste plano é mostrado na Figura 1B.
Cestas amostra proteger os cortes de tecidos delicados de perda e de acumular poeira e partículas de matéria durante o processo de multi-dia ISH. Cestas de amostra são preparados pela fusão de malha de poliéster para a extremidade cortada de um tubo de microcentrífuga 1,5 ml (Figura 1C). Um pequeno buraco é perfurado na tampa de cada cesta de exemplo para facilitar o fluxo de solução dentro e fora dos cestos. Cestas de amostra são suspensos em soluções ISH, colocando-os em buracos perfurados 12 milímetros em 24 tampas bem-placa (Figura 1D). As tampas placa modificada apoio cestas quando eles são transferidos entre placas de 24 poços durante as mudanças de solução.
É um desafio limite não-específica coloração de fundo durante os períodos de incubação longo necessário para a detecção de mRNAs baixa abundância. A adição de azida de sódio 0,2 mM para buffers de amostra e sua posterior filtração através de filtros 0.22μm apareceu para limitar coloração de fundo (Figura 2). Usando o método descrito aqui, ali não parece haver diferenças visíveis na coloração de fundo quando as amostras são incubadas em solução de desenvolvimento de cores para um período de tempo prolongado (Figura 3).

Figura 1. Preparação de um rato urogenital inferior (LUT) secção de tecido do trato e tubo de microcentrífuga de uma cesta para ISH. A LUT contendo parte da bexiga, uretra pélvica e Wolff associados e estrutura do duto derivados Mϋllerian) é incorporado em um plugue cilíndrico de 4% de baixa derreter agarose. (A) A ficha está colado um disco de amostra e de montagem (B) cortado em seções de 50 ìm com um micrótomo vibrando. (C) Uma seção LUT é transferido para um tubo de microcentrífuga cesta que é preparado pela perfuração de um buraco na tampa do tubo e tela de poliéster de fusão para a extremidade inferior de corte do tubo. (D) O tubo de microcentrífuga é inserido em furos de 12 milímetros perfurado em uma tampa de placa de 24 poços, de modo que as secções de tecido são suspensas em solução tampão durante o protocolo de ISH. As setas indicam o tecido LUT na ficha agarose.
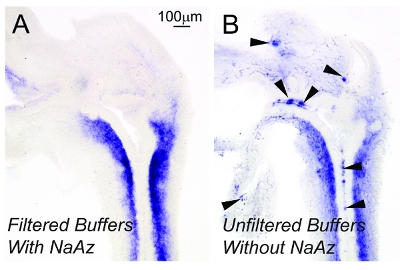
Figura 2. Incorporação de azida de sódio 0,2 mM em 0.22μm soluções filtradas melhora a qualidade do tecido e reduz a coloração de fundo. 17.5dpc trato urogenital de rato macho inferior (Luts) foram seccionados em um plano sagital a uma espessura de 50 micra. Secções de tecido foram corados pela ISH usando uma sonda contra homólogo torção 1. Buffers usados para ISH eram (A) 0.22μm filtrada e suplementado com 0,2 mM de azida de sódio (Naaz) ou (B) não filtrada e não suplementado com Naaz. As setas indicam coloração de fundo. Imagens foram capturadas com a mesma ampliação. Resultados são representativos dos padrões de coloração para n = 3 ninhada independente camundongos.
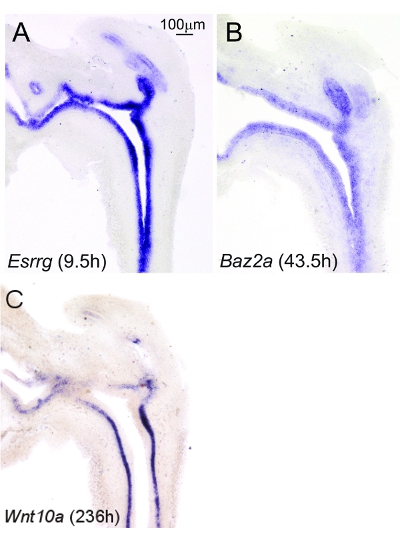
Figura 3. Intensidade de coloração de fundo não parece aumentar com o desenvolvimento da cor prolongada. 17.5dpc trato urogenital de rato macho inferior (Luts) foram seccionados em um plano sagital a uma espessura de 50 micra. Os cortes foram corados pela ISH, incubando-as em Chromagen solução de coloração para (A) 9.5h usando uma sonda que reconhece a transcrição alta abundância de estrogênio relacionadas receptor gama (Esrrg), (B) para 43.5h usando uma sonda que reconhece a abundância média bromodomain transcrição adjacente ao domínio dedo de zinco, 2A (Baz2a), ou (C) para 236H usando uma sonda que reconhece a baixa abundância transcrição wingless-type MMTV sítio de integração familiar, membro 10a (Wnt10a). Imagens foram capturadas com a mesma ampliação. Resultados são representativos dos padrões de coloração para n = 3 ninhada independente camundongos.
Access restricted. Please log in or start a trial to view this content.
Discussão
Usando o método descrito aqui, é possível detectar mRNAs em todos os principais tipos de células e dos compartimentos de tecido fetal do rato LUTs masculino e feminino, incluindo as almofadas mesenquimais, urotélio, o músculo liso, botões da próstata, ducto ejaculatório, e vagina. As seções 50 ìm utilizado neste protocolo tem a vantagem de ser grossa o suficiente para resolver arquitetura do tecido (como vasos sanguíneos), mas são finas o suficiente para evitar armadilhas sonda, que é um problema metodol?...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
Os autores gostariam de agradecer ao Dr. Lan Yi, Cancer Institute de Nova Jersey, para assistência técnica na preparação de cestas de tecido. Este trabalho foi financiado pelo National Institutes of Grants Saúde DK083425 e DK070219.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente | Companhia | Número de catálogo | |
|---|---|---|---|
| Anticorpo anti-digoxigenina, fragmentos Fab | Roche Applied Science | 11214667001 | |
| Bloqueio reagente | Roche Applied Science | 11096176001 | |
| BM Roxo substrato AP, precipitando | Roche Applied Science | 11442074001 | |
| Albumina de soro bovino | Fisher Scientific | BP1600-100 | |
| Placa de cultura de células, 24 bem | Corning | 3524 | |
| Digoxigenina 11 UTP | Roche Applied Science | 1277073910 | |
| dNTPs | Roche Applied Science | 11969064001 | |
| De dois gumes lâmina de barbear | Wilkinson Sword | Modelo clássico | |
| Eliminase removedor de RNase | Decon Laboratories | 1102 | |
| Formamida | Sigma | F5786-1L | |
| Gel kit de extração | Qiagen | 28704 | |
| Solução de glutaraldeído, 25% em H 2 O | Sigma | G6257-100ML | |
| Heparina, sal de sódio | Sigma | H3393 | |
| Solução de peróxido de hidrogênio, 30% em H 2 O | Fisher Scientific | BP2633-500 | |
| Levamisol | Sigma | L9756 | |
| Loctite 404 adesivo instantâneo rápida set | Henkel Corp | 46551 | |
| Cloreto de magnésio | Fisher Scientific | M33-500 | |
| Ácido maleico | Sigma | M0375-500G | |
| Tubos de microcentrífuga, 1,5 ml | Biologix Empresa de Pesquisa | BP337-100 | |
| Millicell inserir placa de cultura | Millipore | PICM01250 | |
| Resina de moagem Molecular | G-Biosciences | 786-138PR | |
| Solução de paraformaldeído, 4% em tampão fosfato salino | Affymetrix | 19943 | |
| Tampão fosfato salino, sem Ca e Mg | Biomedicals MP | ICN1760420 | |
| Polyester mesh, 33 micron, 12 "x 24" | Peças pequenas Inc | CMY-0033-D | |
| Proteinase K solução, 20mg/ml | Amresco | E195-5mL | |
| Colunas QIAshredder | Qiagen | 79654 | |
| Solução Q | Qiagen | Fornecido com Taq DNA polimerase | |
| RNase | Sigma | R6513 | |
| RNase inibidor | Roche Applied Science | 03335399001 | |
| RNeasy mini-kit | Qiagen | 74104 | |
| RNEASY mini-kit | Qiagen | 74104 | |
| RQ1 RNase DNase | Promega | M6101 | |
| SeaPlaque baixo-melt agarose | Lonza | 50101 | |
| Soro de ovelha | Sigma | S2263-500mL | |
| Stericup unidade de filtro, 0.22μm, polietersulfona, 500mL | Millipore | SCGPU05RE | |
| Azida de sódio, granular | Fisher Scientific | S227I-100 | |
| Cloreto de sódio | Fisher Scientific | BP358-212 | |
| Dodecilsulfato de sódio | Fisher Scientific | S529-500 | |
| SSC, 20X solução | Research Products International | S24022-4.000,0 | |
| SuperScript III sistema de síntese de primeira fita | Invitrogen | 18080-051 | |
| T7 RNA polimerase | Roche Applied Science | 10881767001 | |
| Taq DNA polimerase | Qiagen | 201203 | |
| Tris-HCl | Fisher Scientific | BP153-1 | |
| Tween 20 | Fisher Scientific | BP337-100 | |
| Vibrando com micrótomo deluxe banho espécime | Leica Microsystems | VT1000A | |
| Levedura tRNA | Roche Applied Science | 109495 |
Referências
- Cunha, G. R. The possible influence of temporal factors in androgenic responsiveness of urogenital tissue recombinants from wild-type and androgen-insensitive (Tfm) mice. Journal of Experimental Zoology. 205, 181-181 (1978).
- Cunha, G. R. Hormonal, cellular, and molecular regulation of normal and neoplastic prostatic development. Journal of Steroid Biochemistry and Molecular Biology. 92, 221-221 (2004).
- Price, D. Comparative aspects of development and structure in the prostate. National Cancer Institute Monographs. 12, 1-1 (1963).
- Cunha, G. R. Stromal-epithelial interactions--I. Induction of prostatic phenotype in urothelium of testicular feminized (Tfm/y) mice. Journal of Steroid Biochemistry. 14, 1317-1317 (1981).
- Rozen, S. Primer3 on the WWW for general users and for biologist programmers. Methods in Molecular Biology. 132, 365-365 (2000).
- Zhang, Z. A greedy algorithm for aligning DNA sequences. Journal of Computational Biology. 7, 203-203 (2000).
- Vezina, C. M. Dioxin causes ventral prostate agenesis by disrupting dorsoventral patterning in developing mouse prostate. Toxicological Sciences. 106, 488-488 (2008).
- Wilkinson, D. G. Detection of messenger RNA by in situ hybridization to tissue sections and whole mounts. Methods in Enzymology. 225, 361-361 (1993).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados