Method Article
RNA Isolierung von Pseudomonas aeruginosa Kolonisierung der Murine Gastrointestinaltrakt
In diesem Artikel
Zusammenfassung
Eine zuverlässige Methode für die RNA-Isolierung Pseudomonas aeruginosa Erholte sich von murinen cecums beschrieben. Die RNA zurückgewonnen wird in ausreichender Menge und Qualität für die anschließende qPCR, Transkription Profiling und RNA Seq Experimente. Diese Technik kann für die RNA-Isolierung von anderen Darmbakterien angepasst werden.
Zusammenfassung
Pseudomonas aeruginosa (PA) Infektionen führen zu einer signifikanten Morbidität und Mortalität in der Hosts mit geschwächtem Immunsystem, wie Patienten mit Leukämie, schwere Brandwunden oder Organtransplantationen 1. Bei Patienten mit hohem Risiko für die Entwicklung von PA Infektionen der Blutbahn, ist die gastrointestinale (GI-Trakt) der Haupt-Reservoir für die Besiedlung 2, aber die Mechanismen, durch die PA Übergänge von einer asymptomatischen Besiedlung Mikrobe zu einem invasiven und oft tödlich, sind Erreger unklar. Zuvor führten wir in vivo Transkription Profiling Experimenten durch die Rückgewinnung von PA mRNA aus Bakterienzellen mit Wohnsitz in den cecums der kolonisierten Mäusen 3 im Hinblick auf Veränderungen in der bakteriellen Genexpression während Änderungen an der Host-Immunstatus zu identifizieren.
Wie bei jedem Transkriptionsprofilierung Experiment wird der geschwindigkeitsbestimmende Schritt bei der Isolierung von ausreichenden Mengen an hochwertigen mRNA. Angesichts der Fülle von Enzymen, Schutt, Speisereste und Feinstaub in der GI-Trakt, ist die Herausforderung der RNA-Isolierung entmutigend. Hier präsentieren wir eine Methode für die zuverlässige und reproduzierbare Isolierung von bakteriellen RNA aus den murinen GI-Trakt erholt. Diese Methode nutzt ein gut etabliertes Mausmodell der PA GI Kolonisierung und Neutropenie-induzierte Verbreitung 4. Sobald GI Besiedlung mit PA bestätigt wird, werden die Mäuse getötet und cecal Inhalt zurückgewonnen werden und schockgefroren. RNA wird dann extrahiert mit einer Kombination von mechanischen Störungen, Kochen, Phenol / Chloroform Extraktionen, DNase Behandlung, und Affinitätschromatographie. Menge und Reinheit durch Spektrophotometrie (Nanodrop Technologies) und Bioanalyzer (Agilent Technologies) (Abb. 1) bestätigt. Diese Methode der GI mikrobielle RNA-Isolierung kann leicht auf andere Bakterien und Pilze sowie angepasst werden.
Protokoll
1. Mausmodell der P. aeruginosa GI Colonization und Verbreitung
- C3H/HeN-Mäusen (6-8 Wochen alt, weiblich, Harlan) mit oralen Antibiotika behandelt werden, um Kommensalflora führen und dann mono-kolonisierten mit PA wie zuvor 4 beschrieben.
2. Harvesting Murine Blinddarm Luminal Inhalt
- Tauchen Sie ein gereinigtes Edelstahl Mörtel in ein Bad aus flüssigem Stickstoff.
- Euthanize Mäusen durch Kohlendioxid Ersticken.
- Sichere Maus Karkasse auf einer Styropor-Platte und eine Dusche mit 95% Ethanol. Machen Sie eine Mittellinie Längsschnitt durch die Haut vom Brustbein zu den Damm. Zurückhalten der Haut und des Bauchfells in die Bauchhöhle aussetzen.
- Resektion des gesamten Blinddarm. Halten Sie den Blinddarm mit einer Pinzette auf dem Edelstahl-Mörtel. Snip beiden Enden des Blinddarms mit Dissektion Schere.
- Legen Sie eine P1000 Pipettenspitze mit 1 ml cecal flushate Puffer (10 mM Tris-HCl, 1 mM EDTA und 200 mM NaCl) 5 in das proximale Ende des Blinddarms gefüllt. Spülen Sie den Blinddarm flushate Puffer und cecal luminalen Inhalte in die Edelstahl-Mörtel. 1 ml der Blinddarm flushate Puffer ausreichen wird für die Spülung alle cecal Inhalte. Blinddarm flushate Inhalte umgehend einfrieren bei Kontakt mit dem Mörtel.
- Grind der Blinddarm flushate Inhalt mit einem sterilen Pistill.
- Legen Boden gefroren cecal luminalen Inhalt in ein 50 ml Polypropylen-konischen Rohr in einem Trockeneis / Ethanol getaucht.
- Wiederholen Sie die Schritte 2.2 bis 2.7 für jede weitere Maus.
- Lagerung bei -80 ° C.
3. Bakterielle RNA Isolation
- Warm 1 Volumen (auf Grundlage Endvolumen von zwei cecal luminalen Inhalte, etwa 3 ml) von saurem Phenol / Chloroform (5:1, v / v) in einer 50 ml Oak Ridge Zentrifugenröhrchen mit der Mütze in Parafilm in einer 65 gesichert enthaltenen ° C Wasserbad.
- Boil 0,5 Volumen Lysepuffer (2% SDS, 16 mM EDTA und 200 mM NaCl) 6 in 50 ml Polypropylen-konischen Rohr für 5 Minuten enthalten.
- Kochendem Lysepuffer cecal luminalen Inhalte. Homogenisieren. 5 Minuten lang kochen mit periodischen Vortexen.
- Fügen Sie die 100 ° C cecal Inhalte / Lyse Probe auf die 65 ° C Säure Phenol / Chloroform in den Oak Ridge Zentrifugenröhrchen. Seal cap mit Parafilm.
- Inkubation bei 65 ° C für 10 Minuten mit periodischen Vortexen (alle 2 Minuten).
- Centrifuge Probe bei 2.500 g bei 4 ° für 15 Minuten.
- Sorgfältig Transfer wässrigen Phase (Vermeidung eines der weißen Schnittstelle) in ein frisches 50 ml Oak Ridge Zentrifuge. Das Volumen der wässrigen Phase wird ca. 50% des Gesamtvolumens der Blinddarm Inhalte, Lysepuffer und saure Phenol / Chloroform werden. Fügen Sie dem gleichen Volumen an Säure Phenol / Chloroform.
- Seal cap mit Parafilm und mischen Sie gut durch Vortexen mit hoher Geschwindigkeit.
- Centrifuge Probe bei 2.500 g bei 4 ° für 15 Minuten.
- Wiederholen Sie die Schritte 3,7 bis 3,9, bis keine sichtbaren weißen Schnittstelle zwischen der wässrigen und der organischen Phase.
- Sorgfältig Transfer wässrige Phase in ein frisches 50 ml Oak Ridge Zentrifuge. Fügen Sie dem gleichen Volumen Chloroform / Isoamylalkohol (24:1, v / v).
- Seal cap und gut mischen durch Vortexen.
- Centrifuge Probe bei 2.500 g bei 4 ° für 15 Minuten. Entfernen wässrigen Phase (Volumen ca. 50% der gesamten Reaktionsvolumen werden) auf 15 ml Polypropylen-konischen Rohr und ein gleiches Volumen an Isopropanol.
- Inkubation bei -20 ° für mindestens 2 Stunden oder über Nacht.
- Centrifuge Probe bei 2.500 g bei 4 ° für 45 Minuten. Eine gelartige Pellet wird an der Unterseite des Rohres bilden. Überstand entfernen.
- Waschen des Pellets mit 1 ml eiskaltem 70% Ethanol. Vortex. Centrifuge Probe bei 10.000 g bei 4 ° C für 5 Minuten.
- Entfernen Sie den Überstand und das Röhrchen, das Pellet bei Raumtemperatur trocknen lassen für 10 Minuten.
- Die RNA Pellet in 200 ul RNase-freiem Wasser.
4. DNase-Behandlung (Turbo DNA-Free, Applied Biosystems)
- Add 20 ul 10x Turbo DNase-Puffer und 2 ul Turbo DNase und vorsichtig mischen.
- Bei 37 ° für 20 Minuten.
- Add 20 ul resuspendiert DNase Inaktivierung Reagenz und gut mischen.
- Inkubieren 5 Minuten bei Raumtemperatur, Mischen gelegentlich.
- Zentrifuge bei 10.000 g bei Raumtemperatur für 1,5 Minuten und den Überstand in ein neues Mikrozentrifugenröhrchen.
- Fügen Sie ein Volumen von kaltem (4 °) Säure Phenol / Chloroform und gründlich durch Vortexen für 1 Minute.
- Zentrifuge bei 12.500 g bei Raumtemperatur für 2 Minuten.
- Übertragen Sie die wässrige Phase in ein neues Röhrchen geben und 1 Volumen Chloroform / Isoamylalkohol (49:1 v / v), Vortex.
- Zentrifuge bei 12.500 g bei Raumtemperatur für 2 Minuten.
- Übertragen Sie die wässrige Phase (ca. 50% der gesamten Reaktionsvolumen) in ein neues Röhrchen.
- Add 0,1 Volumen 3 M Natriumacetat, pH 5,5 und2,5 Volumina eiskaltem 100% Ethanol. Vortex.
- Inkubation bei -20 ° für mindestens 2 Stunden oder bei -80 ° für 30 Minuten.
- Zentrifuge bei 12.000 g für 30 Minuten bei 4 °
- Überstand entfernen.
- Waschen des Pellets mit 1 ml eiskaltem 70% igem Ethanol durch Zentrifugation für 5 Minuten bei 10.000 g bei 4 °.
- Entfernen Sie den Überstand und an der Luft trocknen für 10 Minuten.
- Das Pellet in 100 ul RNase-freiem Wasser. Möglicherweise müssen Sie in 65 ° Wasserbad Ort zu resuspendieren und lösen das Pellet.
- Zur Überprüfung der Wirksamkeit der DNase Behandlung könnte eine Standard-RT-PCR-Reaktion zur Amplifikation eines ribosomalen Proteins (oder einer anderen Referenz-Gen) für behandelten und nicht behandelten Proben durchgeführt werden, wobei keine RT steuert.
5. RNA Cleanup Step (Qiagen, RNeasy Kit)
- Siehe Seiten 56-57 des Qiagen RNeasy Mini Handbook (4. Auflage, April 2006). Folgen Sie wie angegeben.
6. Repräsentative Ergebnisse
Die Menge der bakteriellen Gesamt-RNA mit Hilfe dieses Protokolls gewonnen wird etwa 2-3 pg von zwei cecums. Die RNA zurückgewonnen wird in ausreichender Menge und Qualität für die anschließende qPCR, Transkription Profiling und RNA Seq Experimente. RNA Reinheit wird routinemäßig durch die Messung der 260nm/280nm Verhältnis 7 der Probe untersucht, doch ist diese Methode liefert keine Informationen über RNA-Integrität. Die Agilent Bioanalyzer ist ein Mikrofluidik-basierte Plattform für die Dimensionierung, Quantifizierung und Qualitätskontrolle von DNA, RNA, Proteine und Zellen, und nutzt eine RNA Integrität metrischen als RNA Integrity Number (RIN) 8 bekannt. RNA extrahiert mit diesem Protokoll produziert 260/280 Verhältnissen zwischen 1,7 bis 2,0 und RIN-Werte ≥ 7,0. Ein Beispiel für eine Agilent Bioanalyzer Analyse der bakteriellen RNA durch dieses Protokoll zurückgewonnen wird in Abbildung 1 dargestellt.
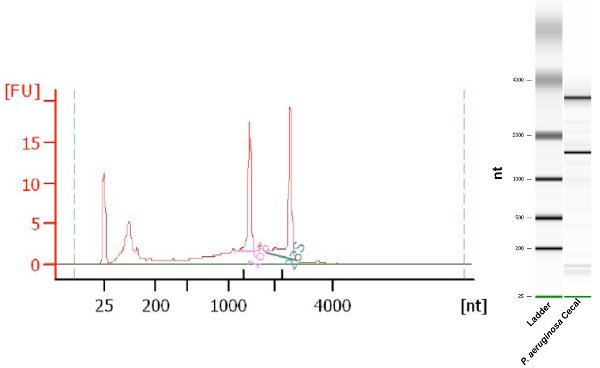
Abbildung 1. Agilent Bioanalyzer Elektropherogramm und gelartige Bild von Pseudomonas aeruginosa RNA-Probe isoliert und erholte sich von murinen cecal Inhalte. RIN 8,0.
Diskussion
Die RNA-Extraktion hier beschriebene Methode ermöglicht die Wiederherstellung von ausreichenden Mengen an hochwertigen Pseudomonas aeruginosa Gesamt-RNA aus den murinen GI-Trakt geerntet. Diese Methode ist nicht auf P. eingeschränkt aeruginosa und kann potenziell auf andere Bakterien eingesetzt werden. Die Erholung der ausreichend Mikroorganismen aus dem Darm wird erheblich variieren von Organismus zu Organismus. In unserem Mausmodell, P. aeruginosa in der Regel besiedelt das murine GI-Trakt auf einem Niveau zwischen 5 x 07 bis 5 Oktober x 10 8 KBE / g Kot 4. Da das gewonnene cecal Inhalte sind etwa 0,5 Gramm, die geschätzte Zahl der P. aeruginosa aus zwei cecums zurückgewonnen wird zwischen 5 x 07 bis 5 Oktober x 10 8 KBE. Wenn andere Mikroorganismen eingesetzt werden, wäre es klug, um GI Kolonisierung Ebenen zu überprüfen und dann die Berechnung der Anzahl der cecums benötigt, um die angestrebte Zahl von cfu erholen. Es ist auch wichtig zu beachten, dass bei der Verwendung dieser speziellen Maus-Modell, Antibiotika-behandelten Mäusen nicht mit PA infizierten keine quantifizierbare Mengen an RNA aus ihren Blinddarm Inhalte isoliert zu haben.
Unsere Mausmodell der Pseudomonas aeruginosa Magen-Darm-Kolonisation und Verbreitung versucht, die Pathogenese von P. emulieren aeruginosa Bakteriämie bei Krebs und Stammzellentransplantation Patienten. In dieser Patientengruppe ist Kommensalflora oft erschöpft sekundär zu Antibiotikum oder chemotherapeutischen Behandlung (zB das Antibiotikum Erschöpfung der GI Kommensalflora), was zu einem überschießenden Wachstum pathogener Keime (zB Mono-Verbindung mit P. aeruginosa) und dann anschließenden Verbreitung nach Immunsuppression. Die Vorteile und Grenzen dieses Mausmodell wurden bereits zuvor 4 behandelt. Der Zweck dieser Studie besteht darin, eine Methode zur Isolierung von mikrobiellen Gesamt-RNA aus dem Gastrointestinaltrakt stellen. Dieses Protokoll kann leicht auf andere Mausmodellen, dass andere Aspekte der mikrobiellen Pathogenese im GI-Trakt (zB Kommensalflora Interaktionen, bakterielle Wirkung auf entzündliche Darmerkrankungen, etc.) Studie angepasst werden.
Ein Vorteil dieser Methode ist der Einbau von mehreren Lyse Schritte einschließlich Frost / Tau, mechanische Störungen (Pulverisieren mit Mörtel / Stößel, Homogenisierung), Kochen und chemische Lyse (z. B. SDS). Trotz der Vielzahl der Lyse Schritte können einige Mikroorganismen (insbesondere Gram-positive Bakterien und Pilze / Hefen) erfordern zusätzliche mechanische Störungen. Nach dem heißen Lyse / Säure-Phenol-Chloroform-Inkubation (Schritt 3,5), die Zugabe von Glasperlen (0,1 mm für Bakterien und / oder 0,5-0,7 mm für Hefe) und einer anschließenden bead-schlagen Schritt sollte ausreichen, um diese Organismen 9 lysieren, 10.
Angesichts der komplexen Natur der Materialien aus der murinen Blinddarm, den wiederholten kalten acid-phenol/chloroform Extraktionen (Schritt 3,7 bis 3,9) gewonnen werden, unbedingt erforderlich, um akzeptable RNA Qualität und Integrität für nachgelagerte Reaktionen zu erzielen. Zwischen 3-5 kalten acid-phenol/chloroform Extraktionen können, bevor die weißen Schnittstelle zwischen der wässrigen und der organischen Phase erforderlich ist eliminiert. Schließlich sind die Kombination der beiden DNase-Behandlung (Schritt 4) und RNeasy Cleanup-Protokoll (Schritt 5) Voraussetzung für das Entfernen kontaminierenden DNA und kleinen Nicht-mRNA (5s und tRNA). Wie bereits erwähnt, ist die RNA durch die Nutzung dieses Protokoll wieder in ausreichender Menge und Qualität für die spätere Transkription Profilierung 3, qPCR (unveröffentlicht), und RNA Seq (unveröffentlicht) Experimente.
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Diese Arbeit wurde von den National Institutes of Health gewährt AI62983 (AYK), AI22535 (GPB) finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenzes | Firma | Katalog-Nummer | |
|---|---|---|---|
| Mörser und Stößel | Fischer | 12-947-1 | |
| Homogenisator | Omni | TM-125 | |
| Oak Ridge Zentrifugenröhrchen (50 ml) | Nalgene | 3119-0050 | |
| Saure Phenol / Chloroform | Ambion | AM9720 | |
| Chloroform | Sigma-Aldrich | C2432 | |
| Isoamylalkohol | Sigma-Aldrich | W205702 | |
| Isopropanol | Sigma-Aldrich | 190764 | |
| Diethyl (DEPC) | Sigma-Aldrich | 40718 | |
| DNase | Ambion | AM2238 | |
| RNeasy Kit | Qiagen | 74104 | |
| 3M Natriumacetat | Ambion | AM9740 | |
| 100% Ethanol | Sigma-Aldrich | E7023 |
Referenzen
- Bodey, G. P., Bolivar, R., Fainstein, V., Jadeja, L. Infections caused by Pseudomonas aeruginosa. Rev Infect Dis. 5, 279-279 (1983).
- Bertrand, X. Endemicity, molecular diversity and colonisation routes of Pseudomonas aeruginosa in intensive care units. Intensive Care Med. 27, 1263-1268 (2001).
- Koh, A. Y. Utility of in vivo transcription profiling for identifying Pseudomonas aeruginosa genes needed for gastrointestinal colonization and dissemination. PLoS One. 5, e15131-e15131 (2010).
- Koh, A. Y., Priebe, G. P., Pier, G. B. Virulence of Pseudomonas aeruginosa in a murine model of gastrointestinal colonization and dissemination in neutropenia. Infect Immun. 73, 2262-2272 (2005).
- Alexander, R. J., Raicht, R. F. Purification of total RNA from human stool samples. Dig Dis Sci. 43, 2652-2658 (1998).
- Fitzsimons, N. A., Akkermans, A. D., de Vos, W. M., Vaughan, E. E. Bacterial gene expression detected in human faeces by reverse transcription-PCR. J Microbiol Methods. 55, 133-140 (2003).
- Manchester, K. L. Use of UV methods for measurement of protein and nucleic acid concentrations. Biotechniques. 20, 968-970 (1996).
- Schroeder, A. The RIN: an RNA integrity number for assigning integrity values to RNA measurements. BMC Mol Biol. 7, 3-3 (2006).
- Turnbaugh, P. J. A core gut microbiome in obese and lean twins. Nature. 457, 480-484 (2009).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten