Method Article
Isolamento de RNA Pseudomonas aeruginosa Colonizar o trato gastrointestinal Murino
Neste Artigo
Resumo
Um método confiável para o isolamento do RNA Pseudomonas aeruginosa Recuperou de cecums murino é descrito. O RNA recuperado é de quantidade e qualidade suficientes para qPCR subseqüentes, perfis de transcrição, RNA e experimentos Seq. Esta técnica pode ser adaptada para o isolamento de RNA outros micróbios intestinais.
Resumo
Pseudomonas aeruginosa (PA) resultado infecções em significativa morbidade e mortalidade em hosts com sistemas imunológicos comprometidos, tais como doentes com leucemia, queimaduras graves, ou transplantes de órgãos 1. Em pacientes de alto risco para o desenvolvimento de infecções da corrente sanguínea PA, do trato gastrointestinal (TGI) é o principal reservatório para a colonização 2, mas os mecanismos pelos quais as transições de um micróbio PA colonização assintomática a uma invasivo, e muitas vezes mortal, patógeno não são claras. Anteriormente, foi realizada em experimentos in vivo perfis de transcrição, recuperando mRNA PA a partir de células bacterianas residentes no cecums de ratos colonizados 3, a fim de identificar mudanças na expressão gênica de bactérias durante a alterações do estado imunológico do hospedeiro.
Como acontece com qualquer experiência de perfis de transcrição, o passo limitante da velocidade está no isolamento de quantidades suficientes de mRNA de alta qualidade. Dada a abundância de enzimas, detritos, resíduos de alimentos, e as partículas no trato gastrintestinal, o desafio do isolamento do RNA é assustador. Aqui, apresentamos um método para o isolamento fiável e reprodutível de RNA bacterianos recuperados do trato gastrointestinal de camundongos. Este método utiliza um modelo bem estabelecido murino de PA colonização GI e neutropenia induzida por difusão 4. Colonização, uma vez GI com PA for confirmada, os ratos são sacrificados e conteúdo cecal são recuperados e flash congelado. RNA é então extraído usando uma combinação de perturbação mecânica, ferver, fenol / clorofórmio extrações, tratamento de DNase, e cromatografia de afinidade. Quantidade e pureza são confirmadas por espectrofotometria (Nanodrop Technologies) e Bioanalyzer (Agilent Technologies) (Fig. 1). Este método de GI isolamento microbiano RNA pode ser facilmente adaptado para outras bactérias e fungos.
Protocolo
1. Modelo murino de P. Colonização aeruginosa GI e Difusão
- Camundongos C3H/HeN (6-8 semanas de idade, do sexo feminino, Harlan) são tratadas com antibióticos orais para esgotar flora comensal e então mono-colonizados com PA como descrito anteriormente 4.
2. Colheita murino Cecal Conteúdo Luminal
- Mergulhe uma argamassa de aço inoxidável limpa em um banho de nitrogênio líquido.
- Euthanize ratos por asfixia dióxido de carbono.
- Carcaça de camundongo segura em uma placa de isopor e chuveiro com etanol a 95%. Faça uma incisão mediana longitudinal através da pele do esterno para o períneo. Refletir a pele e peritônio para expor a cavidade abdominal.
- Ressecção do ceco inteiro. Segure o ceco com uma pinça sobre a argamassa de aço inoxidável. Snip ambas as extremidades do ceco com tesouras de dissecação.
- Inserir uma ponteira P1000 cheia com 1 ml de tampão flushate cecal (10 mM TrisHCl, 1 mM EDTA e 200 mM NaCl) 5 na extremidade proximal do ceco. Liberar o buffer flushate cecal e conteúdo cecal luminal na argamassa de aço inoxidável. 1 ml de tampão flushate cecal será suficiente para a lavagem de todo o conteúdo cecal. Conteúdo cecal flushate irá congelar imediatamente após entrar em contato com a argamassa.
- Moer o conteúdo flushate cecal com um pilão estéril.
- Solo congelado lugar cecal conteúdo luminal em um tubo de polipropileno 50 ml submersa em um banho de gelo / etanol seco.
- Repita os passos 2.2 a 2.7 para cada rato adicionais.
- Armazenar a -80 ° C.
3. Isolamento de bactérias RNA
- Volume 1 quente (com base no volume final de dois cecal conteúdo luminal, cerca de 3 ml) de ácido fenol / clorofórmio (5:1, v / v) contido em um carvalho de 50 ml tubo de centrifugação cume com a tampa garantidos em parafilme em 65 · Banho de água C.
- Ferva 0,5 volume de tampão de lise (2% SDS, 16 mM EDTA e 200 mM NaCl) 6 contida em um tubo de polipropileno 50 ml por 5 minutos.
- Adicionar tampão de lise de ebulição para cecal conteúdo luminal. Homogeneizar. Ferva por 5 minutos com vortex periódica.
- Adicione a 100 ° C conteúdo cecal / amostra de lise para a 65 ° C ácido fenol / clorofórmio no tubo de ensaio de Oak Ridge. Seal tampa com parafilme.
- Incubar a 65 ° C por 10 minutos com vortex periódicas (a cada 2 minutos).
- Centrifugar a 2.500 g de amostra a 4 ° por 15 minutos.
- Transferir cuidadosamente fase aquosa (evitando qualquer da interface branco) para uma centrífuga de 50 ml fresco Oak Ridge. O volume da fase aquosa será de aproximadamente 50% do volume total do conteúdo cecal, tampão de lise e ácido fenol / clorofórmio. Adicionar um volume igual de ácido fenol / clorofórmio.
- Seal tampa com parafilme e misturar bem em vórtice em alta velocidade.
- Centrifugar a 2.500 g de amostra a 4 ° por 15 minutos.
- Repita as etapas 3,7-3,9 até que não haja interface visível em branco entre as fases aquosa e orgânica.
- Cuidado de transferência de fase aquosa a uma nova centrífuga 50 ml de Oak Ridge. Adicionar um volume igual de clorofórmio / álcool isoamílico (24:1, v / v).
- Seal tampa e misture bem em vórtice.
- Centrifugar a 2.500 g de amostra a 4 ° por 15 minutos. Remover fase aquosa (volume será de aproximadamente 50% do volume total da reacção) a 15 tubo de polipropileno ml cônico e adicionar um volume igual de isopropanol.
- Incubar a -20 ° por pelo menos duas horas ou durante a noite.
- Centrifugar a 2.500 g de amostra a 4 ° por 45 minutos. Um gel-like pellet formarão no fundo do tubo. Remover o sobrenadante.
- Lave o pellet com 1 ml de etanol gelado 70%. Vortex. Centrífuga de amostra em menos 10.000 g a 4 ° por 5 minutos.
- Remover o sobrenadante e inverter o tubo para deixar o pellet secar em temperatura ambiente por 10 minutos.
- Ressuspender o sedimento em 200 mL de RNA de RNase água.
4. DNase Tratamento (Turbo DNA-Livre, a Applied Biosystems)
- Adicionar 20 mL de tampão 10X DNase Turbo e 2 l Turbo DNase e misture delicadamente.
- Incubar a 37 ° por 20 minutos.
- Adicionar 20 mL de reagente ressuspenso inativação DNase e misture bem.
- Incubar 5 minutos à temperatura ambiente, mexendo ocasionalmente.
- Centrifugar a 10.000 g à temperatura ambiente durante 1,5 minutos e transferir o sobrenadante para um tubo de microcentrífuga novo.
- Adicionar um volume de ácido (4 º) frio de fenol / clorofórmio e misture bem em vortex por 1 minuto.
- Centrifugar a 12.500 g à temperatura ambiente por 2 minutos.
- Transferir a fase aquosa para um novo tubo e adicionar 1 volume de clorofórmio / álcool isoamílico (49:1 v / v), vórtice.
- Centrifugar a 12.500 g à temperatura ambiente por 2 minutos.
- Transferir a fase aquosa (aproximadamente 50% do volume total de reacção) para um novo tubo.
- Adicionar 0,1 volume de acetato de sódio 3M, pH 5,5 e2,5 volumes de etanol gelado de 100%. Vortex.
- Incubar a -20 ° por pelo menos 2 horas ou a -80 ° por 30 minutos.
- Centrifugar a 12.000 g por 30 minutos a 4 °
- Remover o sobrenadante.
- Lave o pellet com 1 ml de etanol gelado 70% por centrifugação por 5 minutos a 10.000 g a 4 °.
- Remover o sobrenadante e secar o ar por 10 minutos.
- Ressuspender o sedimento em 100 ml de RNase água. Pode precisar para colocar em banho-maria 65 ° a ressuspender e dissolver o sedimento.
- Para testar a eficácia do tratamento DNase, um padrão de reação RT-PCR para a amplificação de uma proteína ribossomal (ou gene outra referência) poderia ser realizado para as amostras tratadas e não tratadas, utilizando qualquer controles RT.
5. RNA etapa de limpeza (Qiagen, RNeasy Kit)
- Consulte as páginas 56-57 da Qiagen RNeasy Mini Manual (4 ª Edição, Abril de 2006). Siga o caminho.
6. Resultados representante
A quantidade de bactérias RNA total recuperado usando esse protocolo é de aproximadamente 2-3 mg de dois cecums. O RNA recuperado é de quantidade e qualidade suficientes para qPCR subseqüentes, perfis de transcrição, RNA e experimentos Seq. Pureza RNA é avaliada rotineiramente através da medição da relação de 260nm/280nm 7 da amostra, mas este método não fornece nenhuma informação sobre a integridade do RNA. O Bioanalyzer Agilent é uma plataforma microfluídica baseado para o dimensionamento, quantificação e controle de qualidade de DNA, RNA, proteínas e células, e utiliza uma métrica integridade RNA conhecido como RNA Número Integrity (RIN) 8. RNA extraído com este protocolo produz 260/280 proporções que variam 1,7-2,0 e valores RIN ≥ 7.0. Um exemplo de uma análise Bioanalyzer Agilent do RNA bacteriano recuperado por este protocolo é mostrado na Figura 1.
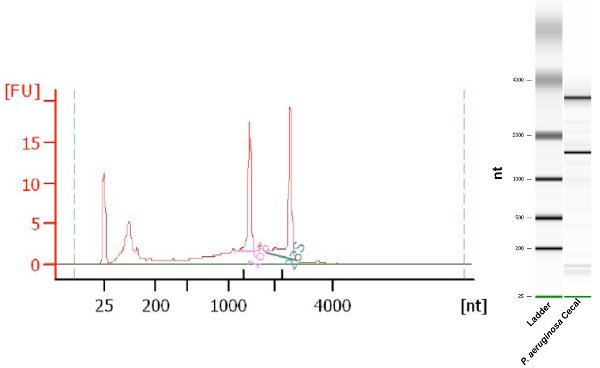
Figura 1. Agilent Bioanalyzer electroferograma e gel-como a imagem de Pseudomonas aeruginosa amostra de RNA total isolado e recuperado de murino conteúdo cecal. RIN 8.0.
Discussão
O método de extração de RNA descrita aqui permite a recuperação de quantidades suficientes de alta qualidade Pseudomonas aeruginosa RNA total colhido a partir do trato GI murino. Este método não é restrito a P. aeruginosa e pode potencialmente ser aplicada a outras bactérias. A recuperação de microrganismos suficientes a partir do intestino irá variar significativamente de organismo para organismo. Em nosso modelo murino, P. aeruginosa tipicamente coloniza o trato GI murino em níveis entre 5 x 10 7 a 5 x 10 8 ufc / g fezes 4. Uma vez que o conteúdo recuperado cecal são aproximadamente 0,5 grama, o número estimado de P. aeruginosa recuperou de duas cecums está entre 5 x 7-5 x 10 10 8 ufc. Se outros microorganismos são utilizados, seria prudente para verificar os níveis de colonização GI e depois calcular o número de cecums necessário para recuperar o número alvo de ufc. Também é importante notar que, ao usar este modelo em particular murino, tratado com antibióticos camundongos não infectados com o PA tem nenhum valor quantificável de RNA isolado de seu conteúdo cecal.
Nosso modelo murino de Pseudomonas aeruginosa gastrointestinal e as tentativas de divulgação para emular a patogênese de P. aeruginosa bacteremia em pacientes de câncer e transplante de células-tronco. Nesta população de pacientes, comensais da flora é muitas vezes esgotadas secundárias ao tratamento com antibióticos ou quimioterápicos (por exemplo, o esgotamento antibiótico de GI comensais flora), resultando em crescimento de micróbios patogênicos (por exemplo, mono-associação com P. aeruginosa) e posterior divulgação após a supressão imunológica. As vantagens e limitações deste modelo murino, já foram abordados anteriormente 4. O objetivo deste estudo é fornecer uma metodologia para isolar RNA total microbiana do trato GI. Este protocolo pode ser facilmente adaptado para outros modelos murinos que estudam outros aspectos da patogênese microbiana no trato GI (ou seja, interações comensais flora, efeitos bacteriana na doença inflamatória intestinal, etc.)
Uma vantagem deste método é a incorporação das etapas de lise múltiplos, incluindo congelamento / descongelamento, a perturbação mecânica (pulverização com argamassa / pilão homogeneização), fervendo, e lise química (por exemplo SDS). Apesar da multiplicidade de etapas de lise, alguns microorganismos (principalmente bactérias gram-positivas e fungos / leveduras) podem exigir a interrupção mecânica adicional. Após a lise quente / ácido fenol-clorofórmio de incubação (Passo 3.5), a adição de contas (0,1 mm para as bactérias e / ou 0,5-0,7 mm para levedura) e um passo-a bater talão subseqüentes deve ser suficiente para lisar estes 9 organismos, 10.
Dada a natureza complexa de materiais recuperados a partir do ceco murino, o frio acid-phenol/chloroform repetidas extrações (Passo 3,7-3,9) são absolutamente necessários para atingir a qualidade aceitável RNA e integridade para outras reações a jusante. Entre 3-5 frio acid-phenol/chloroform extrações podem ser necessários antes que a interface branco entre as fases aquosa e orgânica é eliminada. Finalmente, a combinação de ambos o tratamento DNase (Etapa 4) e Protocolo de Limpeza RNeasy (Passo 5) são essenciais para a remoção de DNA contaminante e pequenas não mRNA (5s e tRNA). Como dito anteriormente, o RNA recuperado através da utilização deste protocolo é de quantidade e qualidade suficientes para posterior transcrição perfil 3, qPCR (não publicado), e Seq RNA (não publicado) experimentos.
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
Este trabalho foi financiado pelos Institutos Nacionais de Saúde concede AI62983 (AYK), AI22535 (GPB).
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente | Companhia | Número de catálogo | |
|---|---|---|---|
| Almofariz e pilão | Pescador | 12-947-1 | |
| Homogeneizador | Omni | TM-125 | |
| Oak Ridge tubos de centrífuga (50 ml) | Nalgene | 3119-0050 | |
| Ácido fenol / clorofórmio | Ambion | AM9720 | |
| Clorofórmio | Sigma-Aldrich | C2432 | |
| Álcool isoamílico | Sigma-Aldrich | W205702 | |
| Isopropanol | Sigma-Aldrich | 190764 | |
| Dietil pyrocarbonate (DEPC) | Sigma-Aldrich | 40718 | |
| DNase | Ambion | AM2238 | |
| RNeasy Kit | Qiagen | 74104 | |
| 3M acetato de sódio | Ambion | AM9740 | |
| O etanol 100% | Sigma-Aldrich | E7023 |
Referências
- Bodey, G. P., Bolivar, R., Fainstein, V., Jadeja, L. Infections caused by Pseudomonas aeruginosa. Rev Infect Dis. 5, 279-279 (1983).
- Bertrand, X. Endemicity, molecular diversity and colonisation routes of Pseudomonas aeruginosa in intensive care units. Intensive Care Med. 27, 1263-1268 (2001).
- Koh, A. Y. Utility of in vivo transcription profiling for identifying Pseudomonas aeruginosa genes needed for gastrointestinal colonization and dissemination. PLoS One. 5, e15131-e15131 (2010).
- Koh, A. Y., Priebe, G. P., Pier, G. B. Virulence of Pseudomonas aeruginosa in a murine model of gastrointestinal colonization and dissemination in neutropenia. Infect Immun. 73, 2262-2272 (2005).
- Alexander, R. J., Raicht, R. F. Purification of total RNA from human stool samples. Dig Dis Sci. 43, 2652-2658 (1998).
- Fitzsimons, N. A., Akkermans, A. D., de Vos, W. M., Vaughan, E. E. Bacterial gene expression detected in human faeces by reverse transcription-PCR. J Microbiol Methods. 55, 133-140 (2003).
- Manchester, K. L. Use of UV methods for measurement of protein and nucleic acid concentrations. Biotechniques. 20, 968-970 (1996).
- Schroeder, A. The RIN: an RNA integrity number for assigning integrity values to RNA measurements. BMC Mol Biol. 7, 3-3 (2006).
- Turnbaugh, P. J. A core gut microbiome in obese and lean twins. Nature. 457, 480-484 (2009).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados