Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Probenvorbereitung von
In diesem Artikel
Zusammenfassung
Die metabolomische Profil Mycobacterium tuberculosis Wird nach Wachstum in Bouillon-Kulturen bestimmt. Bedingungen können variiert werden, um die Wirkungen von Nahrungsergänzungsmitteln, Oxidantien und Anti-Tuberkulose-Mittel auf das metabolische Profil von diesem Mikroorganismus getestet werden. Verfahren für Extraktzubereitung gilt sowohl für 1D 1 H und 2D 1 H- 13 C-NMR-Analysen.
Zusammenfassung
Mycobacterium tuberculosis ist eine der Hauptursachen der Sterblichkeit des Menschen auf einer globalen Skala. Die Entstehung der beiden Multi-(MDR) und extensiv-(XDR) resistente Stämme droht aktuellen Seuchenbekämpfung Bemühungen entgleisen. So gibt es ein dringender Bedarf an Medikamenten und Impfstoffen, die wirksamer als die gegenwärtig verfügbar sind zu entwickeln. Das Genom von M. Tuberkulose ist seit mehr als 10 Jahren bekannt, doch gibt es erhebliche Lücken in unserem Wissen von Genfunktionen und Wesentlichkeit. Viele Studien haben seit Genexpressionsanalyse an beiden transkriptomischen und Proteomik-Ebenen verwendet werden, um die Auswirkungen von Medikamenten, Oxidantien und Wachstumsbedingungen in globalen Muster der Genexpression zu bestimmen. Letztlich wird die endgültige Antwort dieser Veränderungen in der Zusammensetzung des metabolischen Bakterium einschließlich einiger tausend kleinem Molekulargewicht Chemikalien reflektiert. Vergleicht man die metabolische Profile von Wildtyp-und mutierten Stämmen, entweder unbehandelt oder treated mit einem bestimmten Wirkstoff, kann effektiv ermöglichen Zielidentifikation und kann zur Entwicklung von neuartigen Inhibitoren mit Anti-Tuberkulose-Aktivität führen. Ebenso können die Wirkungen von zwei oder mehreren Bedingungen auf der Metabolom ebenfalls bewertet werden. Kernspinresonanz (NMR) ist eine leistungsstarke Technologie, die zur Identifizierung und Quantifizierung von Stoffwechsel-Zwischenprodukte wird. Bei diesem Protokoll, Verfahren zur Herstellung von M. Tuberkulose Zellextrakten für NMR metabolomische Analyse beschrieben. Zellkulturen werden unter geeigneten Bedingungen und erforderlichen Biosicherheitsstufe 3 Containment, 1 geerntet gewachsen, und mechanischen Lyse unter Beibehaltung Kälte zur Konservierung von Metaboliten zu maximieren. Zelllysaten gewonnen werden, gefiltert sterilisiert, und bei extrem niedrigen Temperaturen. Aliquots von diesen Zellextrakte auf Middlebrook 7H9 Agar für koloniebildenden Einheiten der Abwesenheit von lebensfähigen Zellen zu überprüfen ausplattiert. Nach zwei Monaten Inkubation bei 37 ° C, wenn kein viLage Kolonien beobachtet werden, werden Proben aus dem Containment Anlage zur Weiterverarbeitung entfernt. Extrakte werden lyophilisiert, erneut in deuteriertem Puffer und injiziert in der NMR-Gerät, Erfassen spektroskopischer Daten, die anschließend einer statistischen Analyse unterzogen wird. Die beschriebenen Verfahren können sowohl für eindimensionale (1D) 1 H-NMR-und zweidimensionaler (2D) 1 H-13 C-NMR-Analysen angewandt werden. Diese Methode liefert zuverlässigere geringem Molekulargewicht Identifizierung von Metaboliten und mehr zuverlässige und empfindliche quantitative Analysen der Zellextrakt metabolische Zusammensetzungen als chromatographischen Methoden. Variationen der beschriebenen Verfahren nach der Zelllyse Schritt kann auch für parallele Proteomanalyse angepasst werden.
Protokoll
Ein. Protokoll Text
Dieses Protokoll zeigt die Anpassung der NMR-Methodik auf M. Tuberkulose (Klasse III-Agent). Daher müssen Biosafety Level 3 (BSL3) Praktiken zu beachten bei der Durchführung von M. Tuberkulose-Forschung in einem jährlich zertifizierten Labor. Exposition gegenüber Labor erzeugten Aerosole ist die wichtigste Gefahren durch Personal, das mit diesen Mikroorganismen auftreten. Die folgenden Verfahren werden an unserem Institut durchgeführt und Variationen auf der Institutional Biosafety Committee, basiert auf Empfehlungen existieren. Gemeinsame persönliche Schutzausrüstung wird aus einem Tyvek Anzug, bouffant Kappe, Booties, N95 Atemschutzmaske, Schutzbrille, Ärmeln und einem doppelten Paar Nitril-Handschuhe bestehen. Arbeiten, bei denen M. tuberculosis Kulturen und / oder Manipulationen mit offenen M. Tuberkulose Behälter in der Art A2 oder B2 biologischen Sicherheitswerkbank durchgeführt. Kunststoff-saugfähigen Papier ist auf der w platziertBetriebsstrom Oberfläche. Alle Materialien / supplies zu entsorgen oder entfernt werden aus der Anlage muss in zwei biohazard Taschen platziert und dekontaminiert werden durch Autoklavieren. Arbeitsflächen und Geräte im Schrank (FastPrep-24 Lyse Homogenisator, Spektralphotometer, Eimer mit Eis, etc.) verwendet muss nach jeder Arbeitssitzung mit 1% Amphyl (tuberkulozid, bakterizide, fungizide und viruzide Mittel) desinfiziert werden. M. tuberculosis Kulturen müssen unter doppelter Containment für den Transport zu größeren Anlagen außerhalb der biologischen Sicherheitswerkbank wie Gefriergeräte, Inkubatoren, Zentrifugen, Kühlschränke platziert. Zentrifugation mit beiliegenden Sicherheitshinweise Tassen und O-Ring Schraubverschluss Röhrchen durchgeführt. Zur weiteren Analyse außerhalb des BSL3 Labor werden zellfreie Extrakte durch einen 0,2 um Filter oder Mikroorganismen Wärme bei 95 ° C für 15 min getötet filtriert. 2 Proben werden ausplattiert, um die Abwesenheit von koloniebildenden Einheiten vor der Entfernung aus Containment verifizieren.
- Übertragen 110 ml Middlebrook 7H9 komplette Medien enthaltend Polysorbat 80 (Tween 80, um Verklumpen zu verhindern) Brühe (MADC-TW) oder einem geeigneten Medium in einem 250 ml Erlenmeyerkolben. Dreifachen Kulturen pro Bedingung werden routinemäßig gewachsen, wenn mittels 13 C-Metaboliten, und zehn identischen Kulturen werden für 1D 1 H NMR Profilierung gewachsen. Für zwei Bedingungen (z. B. mit und ohne Zusatz von Wirkstoff), doppelte Volumen Kultur pro Wiederholung können gezüchtet und aufgeteilt in zwei identische Kulturen. Alle Rezepturen Reagenz und Lösung befinden sich am Ende des Protokolls bereitgestellt.
- Beimpfen Brühe mit 0,150 ml M. Tuberkulose 50% Glycerin Lager (lassen auf Eis auftauen). Siehe Hinweis 1 unten.
- Lassen die Kultur bei 37 ° C wachsen Schütteln bei 100 Upm für ungefähr 6 Tage (OD 600 von 0,6 bis 0,8). Siehe Anmerkung 2 unten.
- Wenn keine Antibiotika, alternative Zugänge oder andere Behandlungen notwendig sind, sind die Kulturen bereit, geerntet werden. Wenn Behandlungen verwendet werden, weiterhindirekt zu Schritt 5. Verschmutzung Tests phänotypische Analyse, PCR-Tests: Entfernen einer 0,5-ml-Probe von jeder Flasche, in ein Mikrozentrifugenröhrchen und bei 4 ° C für Kultur Titration und Qualitätskontrolle zu übertragen. Siehe Anmerkung 3 unten. An diesem Punkt, fahren Sie mit Schritt 6 fort.
- Entfernen Kolben aus Shaker und führen Sie die gewünschte Behandlung (z. B. fügen Medikament oder Metaboliten). Ort flaschen wieder in den Shaker geben und inkubieren für einen zusätzlichen Zeitraum (zB 6-18 h). Am Ende dieser Zeit nehmen weitere 1,0 ml Aliquots und bestimmen OD 600. Entfernen einer 0,5-ml-Probe von jeder Flasche, in ein Mikrozentrifugenröhrchen und bei 4 ° C für Kultur Titration und Qualitätskontrolle zu übertragen.
- Ort Kulturen auf Eis für 5 min. Nach diesem Schritt verlassen Zellen auf Eis während des gesamten verbleibenden Protokoll. Ernte Kulturen durch Zentrifugation bei 2.000 × g und 4 ° C für 15 min in 50 ml Röhrchen mit einer Tischzentrifuge. Jede Kultur wird vier Röhren mit je 25 ml (insgesamt 1 erfordern00 ml erforderlich ist, um ein ausreichendes Signal-zu-Rausch für 2D 1 zu erhalten H-13 C-NMR-Experimente, in denen nur 50 ml pro Kultur für 1D 1 H-NMR-Experimente erforderlich ist).
- Waschen jedem Zellpellet zweimal mit eiskalter ddH 2 O (ca. 15 ml das erste Mal, und 10 ml zum zweiten Mal) durch die Zentrifugation oben beschriebenen Parameter. Für das zweite waschen, kombinieren die 10 ml Aliquots in das gleiche Rohr vor dem Spinnen. Zellpellet in einem Endvolumen von 1,0 ml ddH 2 O (müssen für Zellpellet Lautstärke). Wenn eine einzelne Zellsuspension frei von Verklumpungen bei diesem Schritt gewünscht wird, können die Zellen kurz beschallt werden und / oder durch einen 27-G-Nadel dreimal. Alternativ kann das Zellpellet bei -80 ° C eingefroren und gelagert werden bis zur weiteren Verarbeitung. Im letzteren Fall werden gefrorenen Pellets auf Eis vor Resuspendierung aufgetaut. Resuspendieren der Zellpellets in einem Endvolumen von 1,0 ml ddH 2 O wie oben angegeben.
- Transfer die 1,0 ml Zellsuspension auf eine 2,0 ml Schraubverschluss Röhrchen mit Lysing Matrix B (0,1 mm Silica-Kügelchen). Legen Sie die FastPrep-24 Lyse Homogenisator im Biosicherheitswerkbank. Setze die Proben in den Probenhalter, Befestigen der Retention sprach Platte oben auf dem Rohr. Verarbeiten der Proben für 60 sec in dem Homogenisator bei einer Geschwindigkeitseinstellung von 6 m / sec.
- Dreh die Proben in einer Mikrozentrifuge bei 15.000 × g und 4 ° C für 10 min bis Pellet Zelltrümmer und intakten Zellen.
- Überstand entfernen und Pass Probe durch einen Spritzenfilter (0,2 pm) in einem sterilen Röhrchen. Platte 0,1 ml Probe (oder einem repräsentativen Anteil der Probe, wie z. B. 10%) auf MADC Agar, um zu überprüfen, dass keine lebensfähigen Zellen. Die Ermittler können auch bei mehr als einem Filtrationsschritt und / oder Überprüfung der Anwesenheit von lebensfähigen Zellen in den Extrakten mittels Live-dead Färbeverfahren um so zu verhindern oder zu identifizieren, mögliche biologische Sicherheit betrifft. Einfrieren Proben in einem Ethanol-Trockeneisbad aufzubewahrenbei -80 ° C, bis sie bereit sind, lyophilisiert und in einem NMR-Anlage verarbeitet werden sollen.
- Nach 2 Monaten, überprüfen Platten, um die Abwesenheit von koloniebildenden Einheiten verifizieren. Wenn keine CFU gefunden werden, können Proben aus der BSL3 Labor gebracht werden. Die weitere Analyse erfolgt in einem Standard-NMR-Anlage, die in der Regel dient Multi-User und betreibt unter keinen spezifischen Einschließungsmaßnahmen Anforderungen durchgeführt.
- Lyophilisieren die Proben bis zur Trockenheit, dann in 0,7 ml-NMR-Puffer und Transfer in ein Mikrozentrifugenröhrchen resuspendieren. Zentrifuge für 3 min bei 13.000 x g. Entfernen Sie 0,6 ml und Transfer zu einem 5 mm-NMR-Röhrchen. Alternativ könnte lyophilisierten Proben zu einer externen Anlage als nicht-infektiöses regelmäßigen Proben verschickt werden.
- NMR-Daten werden sofort aufgefangen. Trotz aller Vorsichtsmaßnahmen kann aktive Enzyme noch vorhanden sein und die Probe ist in der Regel nicht für längere Zeit stabil. Veränderungen in der NMR-Spektrum sind bemerkbar, wenn die Proben durchgeführt werden, bei Raumtemperatur oder bei 4 ° C für m linksehr als 1 Woche. Außerdem wird NMR Datenerhebung zwischen unbehandelten und Drogen behandelten Kulturen, wo die Proben nach dem Zufallsprinzip aus jeder Kategorie ausgewählt werden abgewechselt. Dadurch werden unnötige Bias weil eine bestimmte Kategorie hatte eine längere Verzögerung, bevor die NMR-Spektren wurden gesammelt. Eine Vorspannung in den Daten auftreten würde, wenn die NMR-Spektren für alle unbehandelten Proben wurden zunächst gesammelt und dann gefolgt von den Wirkstoff behandelten Proben. Wenn die Proben nicht sofort durch NMR analysiert werden sollen, sollten die Proben in 1 ml Eppendorfröhrchen bei -80 ° C gelagert werden
- Nach dem Einlegen der Proben in einem BACS-120 Probenwechsler, Gerätekalibrierung, die ein paar Routine-Schritte wie Schloss, Shim, Abstimmung und Optimierung der 90 ° Impulslänge beinhaltet, ist erforderlich, um die Qualität der Ergebnisse zu maximieren. Eine einzelne Probe wird verwendet, um die 90 ° Pulslänge kalibrieren und stimmen das Instrument für die verbleibenden Proben. Sperren, Shim und NMR Datenerfassung für jede Probe ist eine automatisierte using ICONNMR und gradshim.
- Ein 1D 1 H-NMR-Spektrum wird gesammelt, indem die Bruker zgesgp Impulsfolge mit Wasser Suppression mittels Anregung Bildhauerei. Insgesamt 32k Datenpunkte mit einem Sweep Breite 5482.5Hz, 128 Scans, und 16 Dummy-Scans verwendet werden. Ein 2D-1 H-13 C-HSQC-Spektrum gesammelt mit dem Bruker hsqcetgp Impulsfolge. Insgesamt 2048 Datenpunkte mit einem Sweep Breite von 5000,0 Hz ist auf dem direkten 1 H-Maß gesammelt und 64 Datenpunkte mit einem Sweep Breite von 18864,9 Hz entlang der indirekten 13 C Dimension. Das Spektrum wird mit 128 Scans und 16 Dummy Scans gesammelt, um gute Signal-Rausch erhalten.
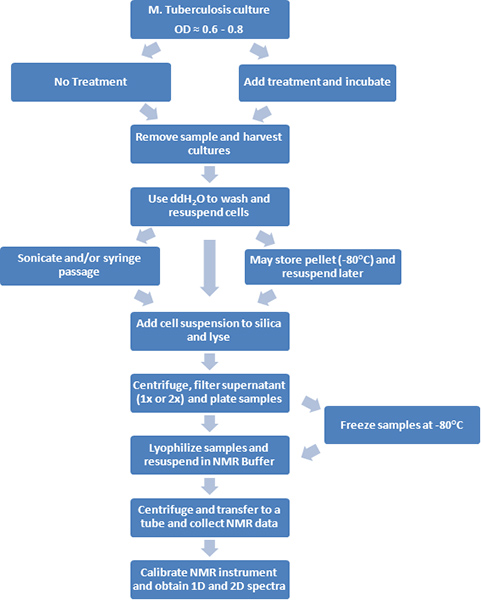
Abbildung 1. Ein Flussdiagramm der experimentellen Verfahren dargestellt.
NMR-Puffer
Stammlösung von 50 mM Kaliumphosphat Schwabbelscheibeer mit 50 uM TMSP:
- 2,17 g K 2 HPO 4 (Kaliumchlorid Dinatriumhydrogenphosphat)
- 1,70 g KH 2 PO 4 (Kaliumphosphat einbasischen)
- 7,86 mg Natrium-3-Trimethylsilylpropionate (TMSP-2 ,2,3,3-D 4 (D, 98%))
- Man löst in 500 ml von "100%" D 2 O
- Die endgültige Lösung sollte mit einer pH-Wert 7,2 (unkorrigiert) sein
MADC-TW oder MOADC-TW (1 L)
- 4,7 g Middlebrook 7H9 Bouillon Basis
- 900 ml ddH 2 O
- 2 ml Glycerin
- Mischen Sie die Komponenten und Autoklaven für 25 min. Kühle Flüssigkeit zu berühren und fügen Sie die folgenden Lösungen:
- 100 ml ADC bei der Vorbereitung MADC oder 100 ml OADC bei der Vorbereitung MOADC
- 2,5 ml 20% Tween 80 (alternativ 1 ml nicht metabolisierbaren 20% Tyloxapol)
- 10 ml 1% Cycloheximid
ADC (1 L)
- 20 g D (+)-Glucose
- 50 g BSA FractionV
- 8,5 g NaCl
- 800 ml ddH 2 O
- Lösen Sie zusammen, die Lautstärke auf 1 L und sterilisieren mit 0,2 um Filter. Lagerung bei 4 ° C.
Alternativ dazu OADC (1L) vorzubereiten, fügen Sie alle oben genannten Komponenten plus 50 ml 1% Ölsäure (Rezept unten macht eine 250 ml Charge) aufgeführt. Lösen Sie zusammen, die Lautstärke auf 1 L und sterilisieren mit 0,2 um Filter. Flasche muss in Alufolie und bei 4 ° C gewickelt werden
- 2,5 g Ölsäure
- 250 ml 0,2 M NaOH
- Auftau Ölsäure (verfestigt sich beim kälte) durch Erhitzen bei 55 ° C für 10 min und in den NaOH-Lösung und Wärme unter Rühren für 60 min. Store in sterile Glasflaschen, die in Aluminiumfolie eingewickelt werden. Seal Flaschen mit Parafilm und bei 4 ° C.
Falls gewünscht, können Sie kommerziellen BD BBL Middlebrook ADC oder OADC mit Katalase Enrichment erwerben.
1% Cycloheximid (100ml)
- 1 g Cycloheximid
- 100 ml ddH 2 O
Auflösen und sterilisieren mit 0,2 um Filter. Lagerung bei 4 ° C. Achtung: Cycloheximid ist giftig so mit äußerster Sorgfalt.
20% (v / v) Tween 80 (100ml)
- 20 ml Tween 80
- 80 ml ddH 2 O
Alternativ wiegen 20 g Tween 80 (etwa 18,9 ml; Dichte 1,06 g / ml), auf 20% w / v-Lösung herzustellen. Wärme-Lösung auf 55 ° C für 30 min auflösen und mischen vollständig. Sterilisieren Flüssigkeit mit einem 0,2 um Filter. Bewahren Sie die endgültige Lösung bei Raumtemperatur.
20% (v / v) Tyloxapol (100ml)
- 20 ml Tyloxapol
- 80 ml ddH 2 O
Alternativ wiegen 20 g Tyloxapol (ca. 18,2 ml; Dichte 1,1 g / ml), auf 20% w / v-Lösung herzustellen. Wärme-Lösung auf 55 ° C für 30 min bis dissolve und mischen vollständig. Sterilisieren Flüssigkeit mit einem 0,2 um Filter. Bewahren Sie die endgültige Lösung bei Raumtemperatur.
Hinweis 1: Glycerol Bestände M. tuberculosis werden unter Verwendung des folgenden Protokolls.
Stocking von M. Tuberkulose
- Wachsen M. tuberculosis in 50 ml MADC zur Sättigung (OD 600 = 1,5 bis 2,0) bei 37 ° C unter Schütteln Bedingungen (100 rpm). Je nach Beanspruchung, dafür wird er 7-14 Tage.
- Spin Proben bei 2.000 × g bei 4 ° C für 15 min bis Pellet Kultur. Überstand entfernen.
- Resuspendieren in 6 ml sterile 50% Glycerin.
- Aliquot 1,5 ml in 4 Corning Kryoröhrchen und Label angemessen.
- Unmittelbar Blitzeis Fläschchen in Ethanol / Trockeneis-Bad und bei -80 ° C.
Hinweis 2: Beimpfen Stamm H37Rv (-80 ° C Aktien bis zu 1,5 Jahre alt) in 70 ml MADC Medien ergibt sich eine OD 600 </ Sub> von etwa 0,6 nach 5 Tagen Wachstum bei 37 ° C unter Schütteln Bedingungen (100 rpm) in einem Innova 40 Shaker.
Hinweis 3: Um Kultur Verunreinigungen testen, Ermittler könnte eine Kultur Aliquot auf Standard-Vollmedium impfen und überprüfen Abwesenheit von Wachstum nach Inkubation über Nacht. Routinemäßig werden Kulturen auf MADC Agar zur Koloniemorphologie beobachten und untersuchen, indem Phasenkontrast-Mikroskopie. Wenn gewünscht, Kulturen können durch PCR unter Verwendung überprüft werden IS 6110 Primer wie beschrieben. 3
2. Repräsentative Ergebnisse
Eine Probe, die gut vorbereitet wird, wird ein NMR-Spektrum zu erhalten, ähnlich dem in 2 dargestellt. Dieses Spektrum ist eine Darstellung des M. tuberculosis Wildtyp metabolischen Pool. Die identifizierten Metaboliten sind direkt oder indirekt mit dem D-Alanin-pathway. Auch ist die Größe der Peakintensitäten proportional Metabolitkonzentrationen präsentierenim Zellextrakt. Deshalb Veränderungen in Peakintensitäten zwischen unbehandelten Kulturen, medikamentöse Behandlung und Mutanten können angeben, Störungen in Metaboliten und Stoffwechselwege. Der M. Tuberkulose 1D 1 H-NMR-Spektren wurden auf einem Bruker Avance 500 MHz-Spektrometer mit einer Dreifach-Resonanz, Z-Achsen-Gradienten Kryosonde ausgestattet gesammelt. Das Spektrum enthält ca. 400 Spitzen, von dem es möglich war, zu identifizieren und zu quantifizieren, 40-50 Metaboliten, einschließlich Aminosäuren, Nucleotidvorläufer und glykolytische und Zitronensäure Zwischenprodukte. Ein BACS-120 Probenwechsler mit Bruker Icon Software wurde verwendet, um die NMR Datenerfassung zu automatisieren. 1D 1 H-NMR-Spektren wurden unter Verwendung Anregung sculpting 4 bis effizient entfernen des Lösungsmittels und die Aufrechterhaltung eines flachen Basislinie, sodass keine Notwendigkeit Basislinie Auflistung, die Artefakte in den nachfolgenden Hauptkomponentenanalyse (PCA) oder Partial Least Squares orthogonalen Diskriminanzanalyse kann induzieren (OPLS- DA). A 1D 1 H-NMR-Spektrum wird bei 25 ° C mit einer spektralen Breite von 5482,5 Hz und 32K Datenpunkte gesammelt. Insgesamt 16 Dummy Scans und 128 Scans wurden verwendet, um das Spektrum zu erhalten. Der ACD Labs 1D-NMR-Prozessor-Software wurde auf semi-automatisch verarbeiten alle 1D 1 H-NMR-Spektren verwendet. Die Spektren wurden Fourier-Transformation, auslaufen, und bezogen auf den TMSP peak (0,0 ppm). Die NMRpipe Softwarepaket wurde verwendet, um einzeln verarbeiten 2D 1 H-13 C-NMR-Spektren und analysiert mit NMRDraw. Die Bruker FID Datendatei zuerst wurde in ein Dateiformat erkennbar NMRpipe umgewandelt und dann das Spektrum wurde fouriertransformierten, korrigierte Phase und Null gefüllt. Die beobachteten Spitzen in dem NMR-1D 1 H NMR-Spektrum und 2D 1 H-13 C-NMR-Spektrum sind bestimmten Metaboliten unter Verwendung von 1 H und 13 C-chemische Verschiebung Toleranzen von 0,05 und 0,50 ppm, und die Madison Metabolomics Consortium Datenbank (MMCD zugeordneten ), 5 die BioMagResBank, 6 und das menschliche Metabolomanalyse Database. 7 Insbesondere sind die 1D-und 2D-NMR-Spektren manuell Spitze gepflückt, wo die Peak-Liste der NMR chemischen Verschiebungen wird dann auf die menschliche Metabolomanalyse Database hochgeladen. Die Metaboliten von der Human Metabolomanalyse Datenbank identifiziert sind, um das NMR-Spektrum sowohl die Maximierung der Anzahl der übereinstimmenden NMR Resonanzen und die Zugehörigkeit zu einer metabolischen Netzwerks zugewiesen. Jede Verbindung oder Metaboliten in der Regel mehrere CH-Paare und entsprechend mehrere NMR-Resonanzen. Somit kann die mehrere dieser NMR-Signale, die in der experimentellen NMR-Spektrum beobachtet werden, desto wahrscheinlicher ist der Metabolit vorliegt. Auch die Ermittlung mehrerer Metaboliten assoziieren mit den gleichen Stoffwechselweg erhöht die Wahrscheinlichkeit der richtigen Zuordnungen. Das Vorhandensein von Metaboliten und Stoffwechselwege sind mit dem Kyoto Encyclopedia of Genes and Genome (KEGG) 8 und den MetaCyc Datenbanken überprüft. 9 Mycobacterium smegmatis ist ein nützliches Modell für M. Tuberkulose und anderen mykobakteriellen Erregern. Wie an anderer Stelle beschrieben, Proben von M. smegmatis könnte auch durch Ultraschall aufgebrochen werden. 10
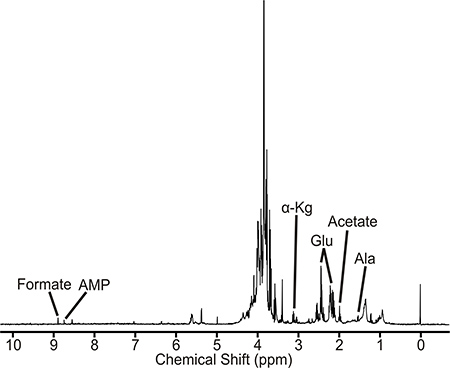
Abbildung 2. 1D 1 H NMR-Spektrum von Mycobacterium tuberculosis Zellextrakt. NMR-Peaks mit repräsentativen Metaboliten verbunden sind beschriftet. Abkürzungen sind: AMP, Adenosin Mono Phosphat; α-kg, α-Ketoglutarat; Glu, Glutamate, und Ala, Alanin.
Diskussion
Eine beträchtliche Anzahl von Studien haben die Transkriptom-und Proteom-Profile von M. analysiert Tuberkulose unter einer Vielzahl von in vitro und in vivo-Bedingungen. 11-16 Letztlich Veränderungen in der Genexpression und Enzymaktivität von Variationen in den Konzentrationen der niedermolekulare Moleküle führen. Die vollständige Beschreibung dieser Verbindungen bildet den Metabolom. So können die Auswirkungen von Drogen und unterschiedlichen Wachstumsbedingungen a...
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Die Autoren möchten sich bei allen Mitgliedern des Labors von Dr. Barletta und Dr. Powers für hilfreiche Kommentare bei der Entwicklung des Protokolls. Wir danken Wendy Austin für hilfreiche Diskussionen und Korrekturlesen des Manuskripts. Die Arbeit in diesem Manuskript beschrieben wurde durch Samen Pilotbeihilfen jeder Ermittler oben von der University of Nebraska-Lincoln Redox Biology Center (parent Grant # NCRR 2P20RR 017675, D. Becker, PI) aufgeführt finanziert. Darüber hinaus danken wir Dr. Ofelia Chacon für die Bereitstellung von Mitteln aus ihrem R21 Zuschuss (1R21AI087561-01A1) für Forschung Lieferungen und Mr. Halouska partielle Gehalts Unterstützung NMR-Techniken in dieser Veröffentlichung enthaltenen standardisieren.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenz / Equipment | Firma | Katalog-Nummer | Kommentare |
| ADC Enrichment | BD BBL Middlebrook | 212352 | |
| BACS-120 Sample Changer | Bruker | ||
| Bruker Avance NMR | Bruker | 500 MHz | |
| Rinderserumalbumin | Fisher Scientific | BP1600-100 | Fraktion V |
| Zentrifugieren | Beckman Coulter | Allegra X-15R | Benchtop |
| Zentrifugenröhrchen | Corning | 430291 | 50 ml sterile Polypropylen |
| Kryoröhrchen | Corning | 430488 | 2,0 ml sterile Polypropylen |
| Cyclohexanolximide | AG Scientific | C-1189 | Giftig |
| D (+) - Glucose | ACROS | 41095-0010 | |
| Deuteriumoxid | Sigma Aldrich | 617385 | |
| Erlenmeyerkolben | VWR | 89095-266 | Sterile, abgeflachter, Polycarbonat, 0,22 um PTFE-Membran belüftete Kappe |
| Blitzeis Flask | VWR | 82018-226 | 750 ml |
| Gefriertrocknungsanlage | VWR | 82019-038 | 4,5 L Benchtop |
| Glycerol | GibcoBRL | 15514-029 | |
| Inkubator | New Brunswick | Innova 40 | Benchtop Schüttler |
| Lysing Matrix B | MP Biomedicals | 6911-100 | |
| Lysis Maschine | MP Biomedicals | FastPrep-24 | |
| Microcentrifuge | Eppendorf | 5415D | Benchtop |
| Microcentrifuge | Beckman Coulter | Microfuge 22R | Benchtop |
| Middlebrook 7H9 Broth | Difco | 271310 | |
| NMR-Röhrchen | Norell | ST500-7 | 5mM |
| OADC Enrichment | BD BBL Middlebrook | 212351 | |
| Ölsäure | Sigma | O1008 | |
| Kaliumphosphat Dibasic | VWR | BDH0266 | |
| Kalium monobasisches | VWR | BDH0268 | |
| Rotor - Microfuge 22R | Beckman Coulter | F241.5P | Sealed und Polypropylen |
| Rotor - Allegra X-15R | Beckman Coulter | SX4750 | Mit bio-zertifizierten Abdeckungen |
| Kochsalz | Fisher Scientific | S271-3 | |
| Natrium-3-trimethylsilylpropionate-2 ,2,3,3-D4 | Cambridge Isotope | DLM-48 | |
| Spektrophotometer | Beckman Coulter | DU-530 | |
| Spektralphotometer Küvetten | Rettungsleine | LS-2410 | 1,5 ml Polystyrol, 2 klare Seiten |
| Spritze | Becton Dickinson | 309585 | Sterile, 3 ml Luer-Lok |
| Syringe Filter | Nalgene | 190-2520 | 0,2 um sterile Celluloseacetat |
| Tween 80 | Fisher Scientific | BP338-500 |
Referenzen
- Larsen, M. H., Biermann, K., Tandberg, S., Hsu, T., Jacobs, W. R. Genetic Manipulation of Mycobacterium tuberculosis. Curr. Protoc. Microbiol. Chapter 10, 2 (2007).
- Larsen, M. H., Biermann, K., Jacobs, W. R. Laboratory Maintenance of Mycobacterium tuberculosis. Curr. Protoc. Microbiol. Chapter 10, 1 (2007).
- Clarridge, J. E., Shawar, R. M., Shinnick, T. M., Plikaytis, B. B. Large-scale use of polymerase chain reaction for detection of Mycobacterium tuberculosis in a routine mycobacteriology laboratory. J. Clin. Microbiol. 31, 2049-2056 (1993).
- Nguyen, B. D., Meng, X., Donovan, K. J., Shaka, A. J. SOGGY: solvent-optimized double gradient spectroscopy for water suppression. A comparison with some existing techniques. J. Magn. Reson. 184, 263-274 (2007).
- Cui, Q. Metabolite identification via the Madison Metabolomics Consortium Database. Nat. Biotechnol. 26, 162-164 (2008).
- Ulrich, E. L. BioMagResBank. Nucleic Acids Res. 36, 402-408 (2008).
- Wishart, D. S. HMDB: the Human Metabolome Database. Nucleic Acids Res. 35, 521-526 (2007).
- Kanehisa, M. KEGG for linking genomes to life and the environment. Nucleic Acids Res. 36, 480-484 (2008).
- Karp, P. D. Expansion of the BioCyc collection of pathway/genome databases to 160 genomes. Nucleic Acids Res. 33, 6083-6089 (2005).
- Halouska, S. Use of NMR metabolomics to analyze the targets of D-cycloserine in mycobacteria: role of D-alanine racemase. J. Proteome. Res. 6, 4608-4614 (2007).
- Boshoff, H. I. The transcriptional responses of Mycobacterium tuberculosis to inhibitors of metabolism: novel insights into drug mechanisms of action. J. Biol. Chem. 279, 40174-40184 (2004).
- Mehaffy, C. Descriptive proteomic analysis shows protein variability between closely related clinical isolates of Mycobacterium tuberculosis. Proteomics. 10, 1966-1984 (2010).
- Schnappinger, D. Transcriptional Adaptation of Mycobacterium tuberculosis within Macrophages: Insights into the Phagosomal Environment. J. Exp. Med. 198, 693-704 (2003).
- Schnappinger, D., Schoolnik, G. K., Ehrt, S. Expression profiling of host pathogen interactions: how Mycobacterium tuberculosis and the macrophage adapt to one another. Microbes. Infect. 8, 1132-1140 (2006).
- Shui, W. Quantitative proteomic profiling of host-pathogen interactions: the macrophage response to Mycobacterium tuberculosis lipids. J. Proteome. Res. 8, 282-289 (2009).
- Talaat, A. M., Lyons, R., Howard, S. T., Johnston, S. A. The temporal expression profile of Mycobacterium tuberculosis infection in mice. Proc. Natl. Acad. Sci. U.S.A. 101, 4602-4607 (2004).
- Forgue, P. NMR metabolic profiling of Aspergillus nidulans to monitor drug and protein activity. J. Proteome Res. 5, 1916-1923 (2006).
- Goodacre, R., Vaidyanathan, S., Dunn, W. B., Harrigan, G. G., Kell, D. B. Metabolomics by numbers: acquiring and understanding global metabolite data. Trends Biotechnol. 22, 245-252 (2004).
- Shin, J. H. NMR-based Metabolomic Profiling in Mice Infected with Mycobacterium tuberculosis. J. Proteome Res. 10, 2238-2247 (2011).
- Jaki, B. U., Franzblau, S. G., Cho, S. H., Pauli, G. F. Development of an extraction method for mycobacterial metabolome analysis. J. Pharm. Biomed. Anal. 41, 196-200 (2006).
- de Carvalho, L. P. Metabolomics of Mycobacterium tuberculosis reveals compartmentalized co-catabolism of carbon substrates. Chem. Biol. 17, 1122-1131 (2010).
- de Carvalho, L. P. Activity-based metabolomic profiling of enzymatic function: identification of Rv1248c as a mycobacterial 2-hydroxy-3-oxoadipate synthase. Chem. Biol. 17, 323-332 (2010).
- Marrero, J., Rhee, K. Y., Schnappinger, D., Pethe, K., Ehrt, S. Gluconeogenic carbon flow of tricarboxylic acid cycle intermediates is critical for Mycobacterium tuberculosis to establish and maintain infection. Proc. Natl. Acad. Sci. U.S.A. 107, 9819-9824 (2010).
- Tang, Y. J. Central metabolism in Mycobacterium smegmatis during the transition from O2-rich to O2-poor conditions as studied by isotopomer-assisted metabolite analysis. Biotechnol. Lett. 31, 1233-1240 (2009).
- Kweon, O. A polyomic approach to elucidate the fluoranthene-degradative pathway in Mycobacterium vanbaalenii PYR-1. J. Bacteriol. 189, 4635-4647 (2007).
- Hasan, M. R., Rahman, M., Jaques, S., Purwantini, E., Daniels, L. Glucose 6-phosphate accumulation in mycobacteria: implications for a novel F420-dependent anti-oxidant defense system. J. Biol. Chem. 285, 19135-19144 (2010).
- Soga, T. Quantitative metabolome analysis using capillary electrophoresis mass spectrometry. J. Proteome Res. 2, 488-494 (2003).
- Metz, T. O. The future of liquid chromatography-mass spectrometry (LC-MS) in metabolic profiling and metabolomic studies for biomarker discovery. Biomark Med. 1, 159-185 (2007).
- Ludwig, C., Viant, M. R. Two-dimensional J-resolved NMR spectroscopy: review of a key methodology in the metabolomics toolbox. Phytochem. Anal. 21, 22-32 (2010).
- Simpson, R. J., Inglis, J. . Proteins and Proteomics: A Laboratory Manual. , 425-595 (2003).
- Beste, D. J., McFadden, J. System-level strategies for studying the metabolism of Mycobacterium tuberculosis. Mol. Biosyst. 6, 2363-2372 (2010).
- Rhee, K. Y. Central carbon metabolism in Mycobacterium tuberculosis: an unexpected frontier. Trends Microbiol. , (2011).
- Who. Health Organization. Anti-tuberculosis Drug Resistance in the World: Report No. 4. , (2008).
- Jassal, M., Bishai, W. R. Extensively drug-resistant tuberculosis. Lancet Infect Dis. 9, 19-30 (2009).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten