É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Preparação de amostras de
Neste Artigo
Resumo
O perfil de metabolômica Mycobacterium tuberculosis É determinada após crescimento em culturas em caldo. As condições podem ser variadas para testar os efeitos de suplementos nutricionais, oxidantes e agentes anti-tuberculose no perfil metabólico deste microrganismo. Procedimento para a preparação de extracto é aplicável tanto para 1D 1 H e 2D 1 H- 13 Análises de C NMR.
Resumo
Mycobacterium tuberculosis é uma das principais causas de mortalidade em seres humanos em escala global. O surgimento de ambos multi-(MDR) e-(XDR) resistentes à drogas-cepas ameaça descarrilar os esforços atuais de controle da doença. Assim, há uma necessidade urgente de desenvolver drogas e vacinas que são mais eficazes do que aqueles atualmente disponíveis. O genoma de M. tuberculose tem sido conhecida há mais de 10 anos, ainda existem lacunas importantes no conhecimento da função dos genes e essencialidade. Muitos estudos têm utilizado uma vez que a análise da expressão do gene, tanto a nível transcriptomic e proteômica para determinar os efeitos das drogas, oxidantes, e condições de crescimento sobre os padrões globais de expressão do gene. Em última análise, a resposta final destas alterações é reflectido na composição metabólica da bactéria incluindo alguns milhares de pequenos produtos químicos de peso molecular. Comparando os perfis metabólicos de tipo selvagem e estirpes mutantes, quer não tratadas ou treated com um fármaco particular, podem efectivamente permitir a identificação do alvo e pode levar ao desenvolvimento de novos inibidores com actividade anti-tuberculoso. Da mesma forma, os efeitos de duas ou mais condições do metaboloma pode também ser avaliada. Ressonância magnética nuclear (RMN) é uma tecnologia poderosa, que é utilizado para identificar e quantificar os intermediários metabólicos. Neste protocolo, os processos para a preparação de M. extractos de células de tuberculose para análise de RMN são descritos metabolômica. As culturas de células são cultivadas em condições adequadas e exigidas Biossegurança Nível 3 de confinamento, uma colhidas e submetidas a lise mecânica, mantendo a temperaturas frias para maximizar a preservação de metabolitos. Os lisados de células são recuperadas, filtrado esterilizado e armazenado a temperaturas ultra-baixas. Alíquotas destas extractos celulares foram semeadas em agar Middlebrook 7H9 para unidades formadoras de colónias para verificar a ausência de células viáveis. Após dois meses de incubação a 37 ° C, na ausência de vicolónias capazes são observadas, as amostras são removidas da unidade de contenção para o processamento a jusante. Os extratos são liofilizadas, ressuspensas em tampão de deuterado e injectada no instrumento de RMN, a captura de dados espectroscópicos que é então submetida a análise estatística. Os procedimentos descritos podem ser aplicados tanto para unidimensional (1D) 1 H NMR e de duas dimensões (2D) 1 H-13 C análises de RMN. Esta metodologia proporciona mais fiável de identificação de baixo peso molecular e do metabolito mais fiáveis e sensíveis análises quantitativas de composições de extracto de células metabólicas de métodos cromatográficos. Variações do processo descrito a seguir ao passo de lise de células pode também ser adaptado para análise proteômica paralelo.
Protocolo
1. Texto protocolo
Este protocolo destaca a adaptação da metodologia de RMN de M. tuberculose (Classe agente III). Portanto, as práticas do Nível de Biossegurança 3 (BSL3) precisam ser seguidas na condução M. pesquisa da tuberculose em um laboratório certificado anualmente. A exposição a aerossóis gerados laboratório é o risco mais importante encontrado por pessoas que trabalham com esses microrganismos. Os seguintes procedimentos são realizados na instituição, e variações podem existir com base em recomendações do Comitê Institucional de Biossegurança. Pessoal comum equipamento de proteção será composto de um terno Tyvek, gorro, botas, máscara N95, óculos de protecção, luvas e um par de luvas de borracha nitrílica dupla. Trabalhos que impliquem M. culturas de tuberculose e / ou manipulações com M. aberto tuberculose recipientes é realizado na A2 tipo ou cabine de segurança biológica B2. Plástico coberta de papel absorvente é colocado no wempo de superfície. Todos os materiais / materiais a serem eliminados ou removidos da instalação tem de ser colocadas em dois sacos de risco biológico e descontaminados em autoclave. As superfícies de trabalho e equipamentos utilizados dentro do armário (FastPrep-24 lise homogeneizador, espectrofotômetro, balde com gelo, etc) devem ser desinfetados após cada sessão de trabalho com Amphyl 1% (agente, tuberculocida bactericida, fungicida e viricida). M. culturas de tuberculose deve ser colocada sob contenção dupla para o transporte de equipamentos de maior porte localizados fora da cabine de segurança biológica, tais como freezers, incubadoras, centrífugas e refrigeradores. A centrifugação é conduzida com segurança fechados copos e tubos O-ring de enroscar. Para análise adicional fora do laboratório BSL3, extractos livres de células são filtradas através de um filtro de 0,2 | iM ou de calor microorganismos mortos, a 95 ° C durante 15 min. Duas amostras são plaqueadas a verificar a ausência de unidades formadoras de colónias antes da remoção de contenção.
- Transferir 110 ml de Middlebrook 7H9 meio completo contendo polissorbato 80 (Tween 80 para evitar aglomeração) caldo (MADC-TW) ou de um meio adequado para um frasco Erlenmeyer de 250 ml. Culturas em triplicado são rotineiramente cultivadas por condição quando se utiliza 13 C-metabolitos, e 10 culturas idênticas são cultivadas para 1D 1 H NMR profiling. Por duas condições (por exemplo, com e sem a adição da droga), o volume duplo de cultura de cada repetição podem ser cultivadas e dividida em duas culturas idênticas. Todos os reagentes e soluções receitas são fornecidos no final do protocolo.
- Inocular caldo com 0,150 ml de M. tuberculose estoque de glicerol 50% (deixar descongelar em gelo). Veja a Nota 1 abaixo.
- Deixe que a cultura crescer a 37 ° C com agitação a 100 rpm durante cerca de 6 dias (DO600 0,6-0,8). Nota 2 abaixo.
- Se não antibióticos, acréscimos ou tratamentos alternativos são necessárias outras, as culturas estão prontas para serem colhidas. Se os tratamentos forem utilizados, continuardiretamente para a Etapa 5. Remover uma amostra de 0,5 ml de cada frasco, transferir para um tubo de microcentrífuga e lugar a 4 ° C durante a titulação da cultura e o controlo de qualidade: testes de contaminação, a análise fenotípica, os testes de PCR. Veja a Nota 3 a seguir. Neste ponto, siga para a Etapa 6.
- Remover frascos de agitador e realizar o tratamento desejado (por exemplo, adicionar fármaco ou metabólito). Lugar flasks volta no agitador e incubar por um período adicional (por exemplo, 6-18 horas). No final deste tempo, tomar uma outra alíquota de 1,0 ml e determina OD 600. Remover uma amostra de 0,5 ml de cada frasco, transferir para um tubo de microcentrífuga e lugar a 4 ° C durante a titulação da cultura e do controlo de qualidade.
- Colocar as culturas em gelo durante 5 min. Após este passo, deixa as células em gelo durante todo o protocolo restante. Culturas de colheita por centrifugação a 2000 xg e 4 ° C durante 15 min em tubos de 50 ml, utilizando uma centrífuga de bancada. Cada cultura exigirá quatro tubos com 25 ml cada (um total de 100 ml é necessária para obter adequada de sinal-para-ruído de 2D 1 H-13 C experiências de RMN, onde apenas 50 ml por cultura é necessária para 1D experimentos 1 H RMN).
- Lava-se cada pelete de células duas vezes com gelado DDH 2 O (aproximadamente 15 ml da primeira vez e 10 ml na segunda vez) pelos parâmetros de centrifugação acima descritas. Para a segunda lavagem, combinar as alíquotas de 10 ml para dentro do tubo mesmo antes da fiação. Ressuspender o sedimento de células, num volume final de 1,0 ml DDH 2 O (necessidade de ajustar para o volume de sedimento de células). Se uma única suspensão de células livres de aglomeração é desejado nesta etapa, as células podem ser brevemente sonicada e / ou passada através de uma agulha de calibre 27, três vezes. Em alternativa, a pelete de células podem ser congeladas a -80 ° C e armazenados até posterior processamento. Neste último caso, as peletes congelados foram descongelados em gelo antes da ressuspensão. Ressuspender as peletes de células, num volume final de 1,0 ml DDH 2 O como indicado acima.
- Transfer a 1,0 ml de suspensão de células para um tubo de tampa de rosca contendo 2,0 ml de lise matriz B (esferas de sílica 0,1 mm). Coloque o homogeneizador FastPrep-24 lise dentro do gabinete de biossegurança. Coloque as amostras para o suporte de amostras, assegurando a retenção falou placa na parte superior do tubo. Processe as amostras para 60 seg no homogeneizador a uma regulação da velocidade de 6 m / seg.
- Girar as amostras numa microcentrífuga a 15.000 xg e 4 ° C durante 10 min para sedimentar os detritos celulares e células intactas.
- Remover o sobrenadante da amostra e passagem através de um filtro de seringa (0,2 um) em um tubo estéril. 0,1 ml da amostra de placa (ou de uma parte representativa da amostra, como por exemplo 10%) em ágar MADC para verificar que não existem células viáveis. Os investigadores também podem realizar mais do que um passo de filtração e / ou verificar a presença de células viáveis nos extractos utilizando procedimentos de coloração live-mortos, de modo a prevenir ou identificar quaisquer problemas potenciais de biossegurança. Congele as amostras num banho de gelo seco-etanol para armazenarà temperatura de -80 ° C até estarem prontas para ser liofilizada e processada numa instalação de RMN.
- Após 2 meses, verificar as placas a fim de verificar a ausência de unidades formadoras de colónias. Se o não CFU são encontrados, as amostras podem ser retiradas de laboratório BSL3. Uma análise adicional é levada a cabo numa instalação de RMN padrão, que normalmente serve de multi-utilizadores e opera sob nenhumas exigências específicas de confinamento.
- Liofilizar as amostras até à secura e, em seguida ressuspender em 0,7 ml de tampão de RMN e transferir para um tubo de microcentrífuga. Centrifugar durante 3 minutos a 13.000 x g. Remover 0,6 ml e transferir para um tubo de RMN de 5 mm. Alternativamente, amostras liofilizadas poderia ser enviado para uma instalação externa como não-biológicos perigosos amostras regulares.
- RMN dados são recolhidos imediatamente. Apesar de todas as precauções, enzimas activas podem estar presentes na amostra e não é geralmente estável durante longos períodos de tempo. As mudanças no espectro de RMN são perceptíveis quando as amostras são deixadas à temperatura ambiente ou a 4 ° C para mminério de 1 semana. Além disso, a coleta de dados de RMN é alternado entre tratados e não tratados droga culturas, onde as amostras são selecionados aleatoriamente de cada categoria. Isto evita viés desnecessário porque uma categoria particular, tinha um longo atraso antes de os espectros de RMN foram coletados. Uma tendência nos dados que ocorreria se os espectros de RMN para todas as amostras não tratadas foram recolhidas em primeiro lugar, e, em seguida, seguido pelas amostras de drogas tratados. Se as amostras não são capazes de ser imediatamente analisada por RMN, as amostras devem ser armazenadas em tubos Eppendorf de 1 ml a -80 ° C.
- Depois de inserir as amostras em um BACS-120 amostra de calibração de instrumentos changer, que envolve passos de rotina a tais como bloqueio, calço, ajustar e otimização do comprimento de pulso de 90 °, é necessário para maximizar a qualidade dos resultados. Um exemplo simples é utilizado para calibrar o comprimento de impulso 90 ° e afinar o instrumento para as amostras restantes. Bloqueio, calços, e RMN de coleta de dados para cada amostra é usin automatizadog ICONNMR e gradshim.
- A 1D 1 H espectro NMR é recolhida utilizando o Bruker zgesgp sequência de impulsos com supressão de água usando escultura de excitação. Um total de pontos de 32k de dados com uma largura de varredura de 5482.5Hz, 128 exames e 16 exames fictícios são usados. Um 2D 1 H-13 C espectro HSQC são coletados usando o Bruker hsqcetgp seqüência de pulsos. Um total de 2048 pontos de dados com uma largura de varredura de 5000,0 Hz é coletado ao longo da dimensão direta H 1, e 64 pontos de dados, com uma largura de varredura de 18864,9 Hz ao longo da dimensão indireta C 13. O espectro é coletada com 128 exames e 16 exames falsos para obter bom sinal ao ruído.
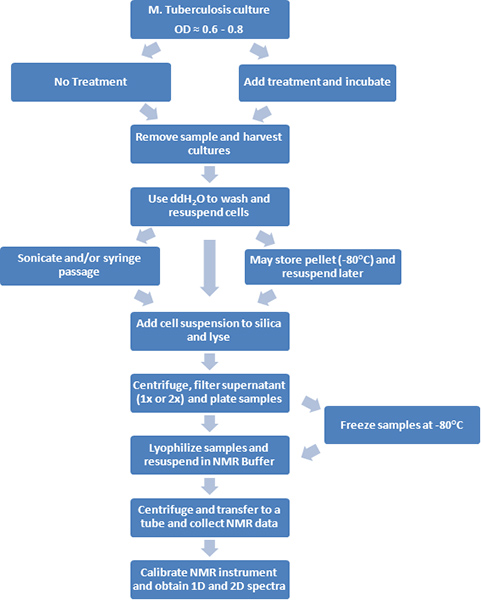
Figura 1. Um diagrama de fluxo dos procedimentos experimentais é apresentada.
RMN tampão
Solução estoque de 50 mM fosfato de potássio lustreer com 50 uM TMSP:
- 2,17 g de K 2 HPO 4 (fosfato de potássio dibásico)
- 1,70 g de KH 2 PO 4 (fosfato de potássio monobásico)
- 7,86 mg de sódio-3-trimetilsilil-(TMSP-2 ,2,3,3-D 4 (D, 98%))
- Dissolver em 500 ml de "100%" D 2 O
- A solução final deve ser a um pH 7,2 (não corrigido)
MADC-TW ou MOADC-TW (1 L)
- 4,7 g Middlebrook 7H9 caldo de base
- 900 ml DDH 2 O
- 2 ml de glicerol
- Misturar os componentes e autoclavagem durante 25 minutos. Líquido fria ao toque e adicionar as seguintes soluções:
- 100 ml ADC MADC na preparação ou 100 ml OADC ao preparar MOADC
- 2,5 ml de Tween 20 a 80% (em alternativa, 1 ml de não-metabolizável Tyloxapol 20%)
- 10 ml de cicloheximida 1%
ADC (1 L)
- 20 g de D (+)-Glucose
- 50 g de BSA FracçãoV
- 8,5 g de NaCl
- 800 ml DDH 2 O
- Dissolver em conjunto, ajustar o volume para 1 litro e esteriliza com filtro de 0,2 um. Armazenar a 4 ° C.
Como alternativa, para preparar OADC (1 L), adicionar todos os componentes listados acima, mais 50 ml de ácido oleico de 1% (receita a seguir faz a 250 ml de lote). Dissolver em conjunto, ajustar o volume para 1 litro e esteriliza com filtro de 0,2 um. Garrafa necessita de ser envolvido em folha de alumínio e armazenar a 4 ° C.
- 2,5 g Ácido oleico
- 250 ml de NaOH 0,2 M
- Ácido oleico degelo (solidifica após refrigeração) por aquecimento a 55 ° C durante 10 min e adicionar a uma solução de NaOH e de calor, com agitação durante 60 min. Conservar em frascos de vidro esterilizados que são enrolados em papel alumínio. Garrafas de vedação com parafilme e armazenar a 4 ° C.
Se preferir, você pode comprar comercial BD BBL Middlebrook ADC ou OADC com o enriquecimento de catalase.
Cicloheximida 1% (100 ml)
- 1 g cicloheximida
- 100 ml DDH 2 O
Dissolver e esterilizar com filtro de 0,2 ^ m. Armazenar a 4 ° C. Atenção: cicloheximida é tóxico para lidar com extremo cuidado.
20% (v / v) de Tween 80 (100 ml)
- 20 ml de Tween 80
- 80 ml DDH 2 O
Alternativamente, pesar 20 g de Tween 80 (aproximadamente 18,9 ml, densidade 1,06 g / ml), para preparar uma solução a 20% w / v. Solução de calor a 55 ° C durante 30 min para dissolver e misturar completamente. Esterilizar o líquido com um filtro de 0,2 um. Armazene a solução final à temperatura ambiente.
20% (v / v) de tiloxapol (100ml)
- 20 ml Tyloxapol
- 80 ml DDH 2 O
Alternativamente, pesar 20 g de tiloxapol (aproximadamente 18,2 ml, densidade 1,1 g / ml), para preparar uma solução a 20% w / v. Solução de calor a 55 ° C durante 30 min para dissolve, e misturar completamente. Esterilizar o líquido com um filtro de 0,2 um. Armazene a solução final à temperatura ambiente.
Nota 1: ações glicéricos de M. tuberculose são preparadas usando o seguinte protocolo.
Estocagem de M. tuberculose
- Crescer M. tuberculose em 50 ml de MADC saturação (OD 600 = 1,5 a 2,0) a 37 ° C sob condições de agitação (100 rpm). Dependendo da estirpe, isso vai levar 7-14 dias.
- Girar as amostras a 2000 xg, a 4 ° C durante 15 min para a cultura da pelota. Remover o sobrenadante.
- Ressuspender em 6 ml de glicerol a 50% estéril.
- Ml alíquota 1,5 em 4 frascos Corning criogênicas e etiqueta de forma adequada.
- Imediatamente de flash frascos de congelamento em etanol banho de gelo seco / e armazenar a -80 ° C.
Nota 2: A inoculação estirpe H37Rv (-80 ° C estoque até 1,5 anos de idade) em 70 ml de meios de comunicação MADC produz uma OD 600 </> Sub de cerca de 0,6 após 5 dias de crescimento a 37 ° C sob condições de agitação (100 rpm) num agitador Innova 40.
Nota 3: Para testar contaminação da cultura, os investigadores poderiam inocular uma alíquota de cultura em meio rico padrão e verificar a ausência de crescimento após incubação durante a noite. Rotineiramente, as culturas são semeadas em ágar MADC para observar a morfologia das colônias e examinar por contraste de fase microscopia. Se desejado, as culturas podem ser verificadas por PCR utilizando os iniciadores IS 6110, tal como descrito. 3
2. Resultados representativos
Um exemplo que é bem preparada irá produzir um espectro de RMN semelhante ao representado na Figura 2. Este espectro é uma representação do M. tuberculose piscina metabólica tipo selvagem. Os metabolitos identificados são directa ou indirectamente associada com a via de D-alanina. Além disso, a magnitude das intensidades de pico é proporcional à concentração de metabólitos apresentamno extracto celular. Portanto, as mudanças nas intensidades de pico entre culturas não tratadas, tratamento medicamentoso, e cepas mutantes podem indicar perturbações em metabolitos e vias metabólicas. O M. tuberculose 1D 1 H RMN foram obtidos num espectrómetro Bruker Avance 500 MHz equipado com uma tripla ressonância criossonda gradiente, Z-eixo. O espectro contém ca. 400 picos, a partir da qual foi possível identificar e quantificar 40-50 metabolitos, incluindo aminoácidos, precursores de nucleótidos, e intermediários de ácido cítrico e glicolíticas. Um trocador BACS-120 amostra com software Bruker Icon foi usado para automatizar a coleta de dados de RMN. 1D 1 H RMN foram obtidos utilizando escultura de excitação de 4 a remover eficientemente o solvente e manter uma linha de base plana, eliminando qualquer necessidade de recolha de linha de base, que pode induzir a artefactos em análise subsequente componente principal (PCA) ou ortogonal análise discriminante mínimos quadrados parciais (OPLS- DA). A 1D 1 H espectro de RMN é recolhida a 25 ° C, com uma largura de espectro de 5482,5 Hz e pontos de 32K de dados. Um total de 16 varrimentos do manequim e 128 scans foram utilizados para obter o espectro. A ACD Labs 1D software processador de RMN foi usado para semi-processar automaticamente todos os 1D 1 H RMN. Os espectros foram transformadas de Fourier, por fases, e referem-se ao pico TMSP (0,0 ppm). O pacote de software foi utilizado para NMRpipe individualmente processar a 2D 1 H-13 C Os espectros de RMN e analisados com NMRDraw. A Bruker FID ficheiro de dados foi primeiro convertido para um formato de arquivo reconhecível por NMRpipe e, em seguida, a análise do espectro foi transformada de Fourier, a fase corrigida, e zero preenchido. Os picos observados no RMN 1 H RMN 1D e espectro de 1 H-13 C espectro de RMN 2D são atribuídos aos metabolitos específicos utilizando 1 H e 13 C tolerâncias desvio químico de 0,05 e 0,50 ppm, respectivamente, e o Madison Metabolomics Consortium Database (MMCD ), 5 a BioMagResBank, 6 e a base de dados humana Metaboloma. 7 Especificamente, os espectros de RMN de 1D e 2D são manualmente pico-escolhido, em que a lista de pico de deslocamentos químicos de RMN é então enviado para a base de dados humana Metaboloma. Os metabolitos identificados pela base de dados Metaboloma humano são designados para o espectro de RMN de base em ambas a maximização do número de correspondência de ressonâncias de RMN e pertencente a uma rede metabólica. Cada composto ou metabolito tipicamente tem pares múltiplos CH e ressonâncias de RMN correspondentemente múltiplas. Assim, quanto mais destas ressonâncias de RMN que são observados no espectro de RMN experimental, o mais provável é o metabolito presente. Do mesmo modo, identificar metabolitos múltiplos associados com o mesmo percurso aumenta a probabilidade de as atribuições correctas. A presença de metabolitos e vias metabólicas são verificados com a Enciclopédia de Kyoto de genes e genomas (KEGG) 8 e as bases de dados MetaCyc. 9 Mycobacterium smegmatis é um sistema modelo útil para o M. tuberculosis e outras micobactérias patógenos. Como já descrito, amostras de M. smegmatis também poderia ser rompidas por sonicação 10.
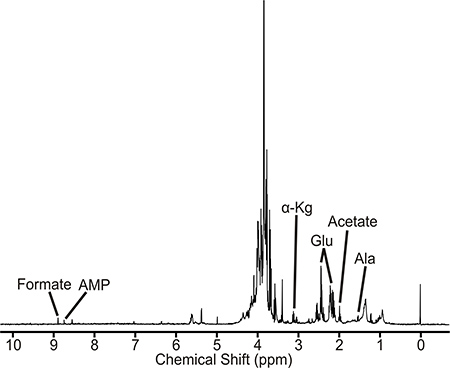
Figura 2. 1D 1 H espectro NMR de Mycobacterium tuberculosis extrato celular. Picos de RMN metabolitos associados representativos são rotulados. As abreviaturas são as seguintes: AMP, fosfato de mono adenosina; α-Kg, α-cetoglutarato; Glu, glutamato, e Ala, alanina.
Discussão
Um número significativo de estudos analisaram os perfis transcriptomic e proteômica de M. tuberculosis, sob uma variedade de ensaios in vitro e in vivo. 11-16 Em última análise, as alterações na expressão de genes e a actividade da enzima levar a variações nas concentrações de pequenas moléculas de peso molecular. A descrição completa destes compostos constitui o metaboloma. Assim, os efeitos da droga e as condições de crescimento diferentes sobre as vias me...
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
Os autores gostariam de agradecer a todos os membros dos laboratórios do Dr. Barletta e Poderes Dr. pelos comentários úteis durante o desenvolvimento do protocolo. Agradecemos Wendy Austin para discussões úteis e revisão do manuscrito. O trabalho descrito neste manuscrito foi financiado por doações piloto de sementes para cada investigador listados acima, da Universidade de Nebraska-Lincoln Center Biologia Redox (pai concessão # NCRR 2P20RR 017675, D. Becker, PI). Além disso, podemos agradecer ao Dr. Ofelia Chacon do fornecimento de fundos a partir de sua concessão R21 (1R21AI087561-01A1) para fontes de pesquisa e de apoio Sr. Halouska do salário parcial de padronizar as técnicas de RMN incluídos nesta publicação.
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente / Equipamentos | Companhia | Número de Catálogo | Comentários |
| Enriquecimento ADC | BD BBL Middlebrook | 212352 | |
| BACS-120 Changer Amostra | Bruker | ||
| RMN Bruker Avance | Bruker | 500 MHz | |
| Albumina de Soro Bovino | Fisher Scientific | BP1600-100 | Fração V |
| Centrifugador | Beckman Coulter | Allegra X-15R | Bancada |
| Tubos da centrífuga | Corning | 430291 | 50 ml de polipropileno estéril |
| Os frascos criogênicos | Corning | 430488 | 2,0 ml de polipropileno estéril |
| Cycloheximide | AG Científico | C-1189 | Tóxico |
| D (+) - Glucose | ACROS | 41095-0010 | |
| Óxido de deutério | Sigma Aldrich | 617385 | |
| Garrafa de Erlenmeyer | VWR | 89095-266 | Base, estéril plana, policarbonato, 0,22 um tampa da membrana PTFE ventilada |
| Flash Flask Congelar | VWR | 82018-226 | 750 ml |
| Liofilizador | VWR | 82019-038 | 4,5 L de bancada |
| Glicerina | GibcoBRL | 15514-029 | |
| Incubadora | New Brunswick | Innova 40 | Agitador de bancada |
| Lise Matrix B | Biomedicals MP | 6911-100 | |
| Máquina de lise | Biomedicals MP | FastPrep-24 | |
| Microcentrífuga | Eppendorf | 5415D | Bancada |
| Microcentrífuga | Beckman Coulter | Microcentrífuga 22R | Bancada |
| Middlebrook 7H9 Broth | Difco | 271310 | |
| RMN tubos | Norell | ST500-7 | 5mM |
| Enriquecimento OADC | BD BBL Middlebrook | 212351 | |
| Ácido oléico | Sigma | O1008 | |
| Fosfato de Potássio Dibásico | VWR | BDH0266 | |
| Fosfato de potássio monobásico | VWR | BDH0268 | |
| Rotor - Microcentrifugue 22R | Beckman Coulter | F241.5P | Selado e polipropileno |
| Rotor - Allegra X-15R | Beckman Coulter | SX4750 | Com bio-certificadas capas |
| Cloreto de Sódio | Fisher Scientific | S271-3 | |
| Sódio-3-trimetilsilil-2 ,2,3,3-D4 | Cambridge Isotope | DLM-48 | |
| Espectrofotômetro | Beckman Coulter | DU-530 | |
| Cubetas Espectrofotômetro | Corda salva-vidas | LS-2410 | 1,5 ml de poliestireno, 2 lados claros |
| Seringa | Becton Dickinson | 309585 | Estéril, 3 ml de Luer-Lok |
| Filtrar seringa | Nalgene | 190-2520 | 0,2 mM de acetato de celulose estéril |
| Tween 80 | Fisher Scientific | BP338-500 |
Referências
- Larsen, M. H., Biermann, K., Tandberg, S., Hsu, T., Jacobs, W. R. Genetic Manipulation of Mycobacterium tuberculosis. Curr. Protoc. Microbiol. Chapter 10, 2 (2007).
- Larsen, M. H., Biermann, K., Jacobs, W. R. Laboratory Maintenance of Mycobacterium tuberculosis. Curr. Protoc. Microbiol. Chapter 10, 1 (2007).
- Clarridge, J. E., Shawar, R. M., Shinnick, T. M., Plikaytis, B. B. Large-scale use of polymerase chain reaction for detection of Mycobacterium tuberculosis in a routine mycobacteriology laboratory. J. Clin. Microbiol. 31, 2049-2056 (1993).
- Nguyen, B. D., Meng, X., Donovan, K. J., Shaka, A. J. SOGGY: solvent-optimized double gradient spectroscopy for water suppression. A comparison with some existing techniques. J. Magn. Reson. 184, 263-274 (2007).
- Cui, Q. Metabolite identification via the Madison Metabolomics Consortium Database. Nat. Biotechnol. 26, 162-164 (2008).
- Ulrich, E. L. BioMagResBank. Nucleic Acids Res. 36, 402-408 (2008).
- Wishart, D. S. HMDB: the Human Metabolome Database. Nucleic Acids Res. 35, 521-526 (2007).
- Kanehisa, M. KEGG for linking genomes to life and the environment. Nucleic Acids Res. 36, 480-484 (2008).
- Karp, P. D. Expansion of the BioCyc collection of pathway/genome databases to 160 genomes. Nucleic Acids Res. 33, 6083-6089 (2005).
- Halouska, S. Use of NMR metabolomics to analyze the targets of D-cycloserine in mycobacteria: role of D-alanine racemase. J. Proteome. Res. 6, 4608-4614 (2007).
- Boshoff, H. I. The transcriptional responses of Mycobacterium tuberculosis to inhibitors of metabolism: novel insights into drug mechanisms of action. J. Biol. Chem. 279, 40174-40184 (2004).
- Mehaffy, C. Descriptive proteomic analysis shows protein variability between closely related clinical isolates of Mycobacterium tuberculosis. Proteomics. 10, 1966-1984 (2010).
- Schnappinger, D. Transcriptional Adaptation of Mycobacterium tuberculosis within Macrophages: Insights into the Phagosomal Environment. J. Exp. Med. 198, 693-704 (2003).
- Schnappinger, D., Schoolnik, G. K., Ehrt, S. Expression profiling of host pathogen interactions: how Mycobacterium tuberculosis and the macrophage adapt to one another. Microbes. Infect. 8, 1132-1140 (2006).
- Shui, W. Quantitative proteomic profiling of host-pathogen interactions: the macrophage response to Mycobacterium tuberculosis lipids. J. Proteome. Res. 8, 282-289 (2009).
- Talaat, A. M., Lyons, R., Howard, S. T., Johnston, S. A. The temporal expression profile of Mycobacterium tuberculosis infection in mice. Proc. Natl. Acad. Sci. U.S.A. 101, 4602-4607 (2004).
- Forgue, P. NMR metabolic profiling of Aspergillus nidulans to monitor drug and protein activity. J. Proteome Res. 5, 1916-1923 (2006).
- Goodacre, R., Vaidyanathan, S., Dunn, W. B., Harrigan, G. G., Kell, D. B. Metabolomics by numbers: acquiring and understanding global metabolite data. Trends Biotechnol. 22, 245-252 (2004).
- Shin, J. H. NMR-based Metabolomic Profiling in Mice Infected with Mycobacterium tuberculosis. J. Proteome Res. 10, 2238-2247 (2011).
- Jaki, B. U., Franzblau, S. G., Cho, S. H., Pauli, G. F. Development of an extraction method for mycobacterial metabolome analysis. J. Pharm. Biomed. Anal. 41, 196-200 (2006).
- de Carvalho, L. P. Metabolomics of Mycobacterium tuberculosis reveals compartmentalized co-catabolism of carbon substrates. Chem. Biol. 17, 1122-1131 (2010).
- de Carvalho, L. P. Activity-based metabolomic profiling of enzymatic function: identification of Rv1248c as a mycobacterial 2-hydroxy-3-oxoadipate synthase. Chem. Biol. 17, 323-332 (2010).
- Marrero, J., Rhee, K. Y., Schnappinger, D., Pethe, K., Ehrt, S. Gluconeogenic carbon flow of tricarboxylic acid cycle intermediates is critical for Mycobacterium tuberculosis to establish and maintain infection. Proc. Natl. Acad. Sci. U.S.A. 107, 9819-9824 (2010).
- Tang, Y. J. Central metabolism in Mycobacterium smegmatis during the transition from O2-rich to O2-poor conditions as studied by isotopomer-assisted metabolite analysis. Biotechnol. Lett. 31, 1233-1240 (2009).
- Kweon, O. A polyomic approach to elucidate the fluoranthene-degradative pathway in Mycobacterium vanbaalenii PYR-1. J. Bacteriol. 189, 4635-4647 (2007).
- Hasan, M. R., Rahman, M., Jaques, S., Purwantini, E., Daniels, L. Glucose 6-phosphate accumulation in mycobacteria: implications for a novel F420-dependent anti-oxidant defense system. J. Biol. Chem. 285, 19135-19144 (2010).
- Soga, T. Quantitative metabolome analysis using capillary electrophoresis mass spectrometry. J. Proteome Res. 2, 488-494 (2003).
- Metz, T. O. The future of liquid chromatography-mass spectrometry (LC-MS) in metabolic profiling and metabolomic studies for biomarker discovery. Biomark Med. 1, 159-185 (2007).
- Ludwig, C., Viant, M. R. Two-dimensional J-resolved NMR spectroscopy: review of a key methodology in the metabolomics toolbox. Phytochem. Anal. 21, 22-32 (2010).
- Simpson, R. J., Inglis, J. . Proteins and Proteomics: A Laboratory Manual. , 425-595 (2003).
- Beste, D. J., McFadden, J. System-level strategies for studying the metabolism of Mycobacterium tuberculosis. Mol. Biosyst. 6, 2363-2372 (2010).
- Rhee, K. Y. Central carbon metabolism in Mycobacterium tuberculosis: an unexpected frontier. Trends Microbiol. , (2011).
- Who. Health Organization. Anti-tuberculosis Drug Resistance in the World: Report No. 4. , (2008).
- Jassal, M., Bishai, W. R. Extensively drug-resistant tuberculosis. Lancet Infect Dis. 9, 19-30 (2009).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados