Method Article
Radioaktiv In-situ- Hybridisierung zum Nachweis Diverse Genexpressionsmuster in Tissue
In diesem Artikel
Zusammenfassung
Dieses Protokoll wird erfolgreich eingesetzt, um quantitativ zu detektieren Ebenen und räumliche Muster der mRNA-Expression in mehreren Gewebetypen über Wirbeltierarten. Das Verfahren lässt sich erkennen geringer Menge Transkripte und ermöglicht die Verarbeitung von Hunderten von Dias gleichzeitig. Wir präsentieren dieses Protokoll mit Expression Profiling der aviären embryonalen Gehirn Bildung als Beispiel.
Zusammenfassung
Die Kenntnis der Zeitpunkt, Stufe, zelluläre Lokalisation und Zelltyp, dass ein Gen, das exprimiert wird, trägt zum Verständnis der Funktion des Gens. Jedes dieser Merkmale kann mit in situ-Hybridisierung an mRNAs in den Zellen erreicht werden. Hier präsentieren wir eine radioaktive in situ Hybridisierung Methode von Clayton et al. (1988) 1, die gearbeitet hat erfolgreich in unserem Labor seit vielen Jahren, vor allem für erwachsene Wirbeltieren Gehirne 5.2 geändert. Die langen komplementärer RNA (cRNA) Sonden an die Zielsequenz erlaubt die Detektion von niedriger Häufigkeit Transkripte 6,7. Der Einbau von radioaktiven Nukleotiden in die cRNA-Sonden können zur weiteren Nachweisempfindlichkeit geringer Menge Transkripte und quantitativen Analysen, entweder durch lichtempfindlichen Röntgenfilm oder Emulsion beschichteten über das Gewebe. Diese Nachweisverfahren eine langfristige Aufzeichnung der Expression des Zielgens. Im Vergleich zu nicht-radioaktive Sonde Meth-Methoden, wie z. B. DIG-Etikettierung ist die radioaktive Sonde Hybridisierungsverfahren nicht mehrere Schritte Amplifikation unter Verwendung von HRP-Antikörper und / oder TSA-Kit geringer Menge Transkripte zu erfassen. Daher stellt dieses Verfahren eine lineare Beziehung zwischen der Signalintensität und gezielte mRNA-Mengen für die quantitative Analyse. Es erlaubt die Verarbeitung von 100 bis 200 Objektträger gleichzeitig. Es funktioniert gut für die verschiedenen Entwicklungsstadien der Embryonen. Die meisten Entwicklungsstudien der Genexpression nutzen ganzen Embryonen und nicht-radioaktive Ansätze 8,9, zum Teil weil embryonalem Gewebe ist zerbrechlicher als erwachsenem Gewebe, mit weniger Zusammenhalt zwischen den Zellen, was es schwierig macht Grenzen zwischen Zell-Populationen mit Gewebeschnitten zu sehen. Im Gegensatz dazu ist unsere radioaktive Ansatz, aufgrund der größeren Auswahl an Sensibilität, in der Lage, einen höheren Kontrast in der Auflösung der Genexpression zwischen Gewebe Regionen zu erhalten, wodurch es leichter, Grenzen zwischen den Populationen zu sehen. Mit dieser Methode konnten die Forscher zeigen, diemögliche Bedeutung eines neu identifizierte Gen, und ferner vorherzusagen die Funktion des Gens von Interesse.
Protokoll
1. Gewebepräparation
- Ernten Sie frische Gewebe. Für Vogelembryonen, Eier vorsichtig öffnen und reinigen Sie den Embryo in einer Schale mit 1XPBS, zwei mal. Für erwachsene Gehirne, schnell zu entfernen, die das Gehirn und vorsichtig mit 1 x PBS waschen.
- Einbetten Embryonen oder erwachsenen Gehirn in eine eingebettete Form voller Oktober Tissue Tek, die Orientierung des Gewebes, wie zum Schneiden benötigt wird, und schnell einfrieren den Block, indem Sie es in ein Ethanol und Trockeneis-Mischung, man aufpassen, nicht, um das Gemisch im Inneren des Blocks erhalten .
- Schneiden Sie die gefrorene Probe in einem Kryostaten in 10 bis 12 mu m dicke Schnitte. Für Gehirn und embryonalem Gewebe, ist der beste Schnitt gemäßigt im Bereich von -18 ° C bis -20 ° C.
- Montieren Sie Gefrierschnitten auf Super Plus Objektträgern.
- Lagern Sie die Abschnitte in einem Dia-Box bei -80 ° C
2. Erzeugung radioaktiver Ribosonden (Verwenden Strahlung Sicherheitsverfahren Ihrer Institution)
- Generieren Sie eine gereinigte lineare DNEine Vorlage des cDNA von Interesse entweder durch enzymatische Verdau eines eingeschränkten klonierten Fragments durch RNA-Polymerase-Promotor Gebieten aus einer Plasmid-DNA oder durch PCR des Einsatzes an den RNA-Polymerase-Promotor Bindungsstellen umgeben. Es ist auch möglich, PCR-Fragmente mit der RNA-Polymerase-Promotoren als Teil der 3 'und 5' PCR-Primer zu erzeugen. Gele reinigen Sie Ihre offenen oder den PCR erzeugten Fragmente mit dem GENECLEAN Gel Purification Kit.
- In einen 0,5 ml Eppendorf-Röhrchen, fügen 0,5-1 ug gereinigten linearen DNA-Matrize, die Transkription Puffer, DTT, RNasin, AGC Nukleotid-Mix-Lösung und RNase-freiem Wasser, um das Volumen zu bringen, um die gewünschte Menge. Dann fügen S 35-UTP und die entsprechende RNA-Polymerase, um entweder Antisense-oder Sense Ribosonden machen.
- Inkubieren der Mischung für 1 Stunde in einem 37 ° C Wasserbad. Fügen Sie ein weiteres Aliquot der RNA-Polymerase und inkubieren Sie für 1 weitere Stunde.
- Add 3M Natriumacetat-Lösung (0,1 fache des Gesamtvolumens) und 100% EtOH (2,5 FOld des Gesamtvolumens) in die Eppendorf-Röhrchen zur Ausfällung des synthetisierten RNA Ribosonde.
- Inkubieren in Trockeneis oder -80 ° C für 15 min oder mehr als 3 Stunden, um eine Ausfällung zu unterstützen.
- Pellet-RNA durch Zentrifugation bei 4 ° C und 15.000 UpM in einer Tisch-Eppendorf-Zentrifuge für 30 min.
- Entfernen Sie den Überstand und Waschen des Pellets mit 70% EtOH, mit dem Finger tippen, um das Pellet gut mischen.
- Pellet RNA wieder durch Zentrifugation bei 4 ° C und 15000 Upm für 30 Minuten.
- Entfernen Sie den Überstand vollständig durch Pipettieren und fügen Sie dann 40 ul Hybridisierungslösung. Gut mischen durch Pipettieren in-und auswendig.
- Stellen Sie 1 ul der Lösung in 3 ml Sicherheits-Solve-Lösung in der Szintillationsfläschchen, gut mischen, und messen die zählt in der Szintillationszähler.
- Setzen Sie den Eppendorf mit dem Ribosonde bei -20 ° C für den Einsatz innerhalb einer Woche.
3. Gewebebehandlung
- In einer Kapuze, bereiten Sie frisches PBS gepuffert 4% paraformaldehyde Lösung auf etwa 3-5 ° C über Raumtemperatur. Die Objektträger von -80 ° C Lagerung in einem Metallgestell auf Trockeneis, um Arbeitsraum unter der Haube tragen, und legen Sie dann das Gestell in die 4% Paraformaldehyd-Lösung und Inkubation für 5 min bei Raumtemperatur.
- 3 x waschen in 1 x PBS je ca. 15 Dips (ca. 1 Sekunde pro dip).
- In der Haube machen Acetylierung Puffer und schütteln Sie sie kräftig innerhalb von 10 Sekunden. Sofort in einen Behälter mit dem Rack von Dias gießen und 10 Min. inkubieren, um die Bindung von Ribosonde Hintergrund zu reduzieren.
- Spülen Sie 3-mal in 2 x SSPE für 15 Dips.
- Entwässern in 70%, 95% und 100% EtOH Seriennummer für 2 min in jedem Schritt (muss nicht in der Haube sein).
- Trocknen Sie die Folien unter der Haube für mindestens 10-15 min.
4. Hybridisierung
- Berechnen Menge Ribosonde (0,5-1x10 6 cpm pro Folie) und Hybridisierungslösung (100 ul pro Folie) für alle Folien benötigt werden. Prewarm die Ribosonde-Hybridisierungsmix bis 65 ° C für 5 min zur Denaturierung des Ribosonde.
- Je 100 ul Ribosonde-Hybridisierungslösung in einer Linie quer über die Folie, und verwenden Sie ein Deckglas, um es gleichmäßig über das Gewebe und Deckglas. Die Objektträger horizontal, mit Blick in eine aufrechte Metallgestell und legen Zahnstange langsam aufrecht in die 65 ° C Ölbad für ein Minimum von 4 Stunden und maximal 16 Stunden. Das Öl schafft eine luftdichte Abdichtung um die Deckgläser.
- Entfernen Sie Metallregale aus dem Ölbad und wischen Sie das überschüssige Öl rund Rack mit Seidenpapier.
- Abwaschen Öl aus den gedeckten Rutschen und Metall-Rack in Glas oder Metall Schalen mit Chloroform, zwei mal. Die 2. Chloroform Waschen kann wie die erste für den nächsten Versuch verwendet werden.
- Übertragen Sie das Rack mit gedeckten Rutschen in eine Schale mit in 0,1% β-Mercaptoethanol + 2 x SSPE-Lösung und nach oben und unten ein paar Dips auf Deckgläsern Lösen und entfernen Sie überschüssiges auflösen Hybridisierung Lösungention.
- Übertragen Sie das Rack mit gedeckten Rutschen der frischen Lösung von 0,1% β-Mercaptoethanol + 2 x SSPE, und entfernen Sie dann die Deckgläser mit einem RNase freien Pinzette in Lösung zur Vermeidung von Kratzern Gewebeschnitten. Übertragen Sie die ungedeckten Dias an die frische Rack in einem Fach Horizontalschlitten Rack-Halter, in einer Lösung von 0,1% β-Mercaptoethanol in 2 x SSPE.
- Inkubieren Sie die mit den Objektträgern in der frischen 0,1% β-Mercaptoethanol + 2 x SSPE-Lösung bei Raumtemperatur für 1 Stunde, um überschüssiges ungebundenes RNA-Sonde zu entfernen. Entsorgen Sie diese und die vorherigen wässrigen Waschlösungen, sowie Deckgläser, als radioaktiver Abfall.
- Übertragen der mit den Objektträgern zu einem vorgewärmten 2x SSPE-Lösung bei 65 ° C, fügen β-Mercaptoethanol zu einer endgültigen Konzentration von 0,1%, und Inkubieren bei 65 ° C für 1 Stunde. Entsorgen Sie als radioaktiver Abfall.
- Übertragen und inkubieren Sie die mit den Objektträgern zwei Mal in vorgewärmten 0,1 x SPPE bei 65 ° C für 30 min je. Die Menge der Radioaktivität entferntDieser Schritt ist sehr klein und nicht mehr als übermäßig radioaktiven Abfälle nach diesem Schritt.
- Entwässern des Racks und Folien in 70%, 95% und 100% EtOH für jeweils 2 Min.
- Trocknen Sie die Folien in der Haube für mindestens 30 min.
5. Visualisierung von radioaktives Signal
- Ort trocknen Folien in eine Filmkassette und in einem dunklen Raum, legen Sie den Röntgenfilm (Kodak Biomax MR Film) über die Rutschen, und schließen Sie die Kassette. Stellen Sie sicher, dass die Dias stehen vor der Schichtseite des Röntgenfilms. Setzen Sie die Dias für ~ 1-7 Tage, je nach erwarteten Fülle der Transkripte.
- Entwickeln Sie die x-ray Film in Standard-Entwickler und Fixierer. Das Hybridisierungssignal zeigt sich als schwarz (belichteten Silberkörner in der Emulsion) auf dem Film (1).
- (Optional) Um zellulärer Auflösung bestimmen und sehen Signal auf das Gewebe, müssen die Folien in fotografischen Emulsion getaucht und gegengefärbt.Wenn Sie werden schließlich mit Cresylviolett Gegenfärbung, dann delipidize Abschnitte durch Inkubation in Xylol für 5 min bei Raumtemperatur, zweimal, rehydrieren jeweils 1 min in 100%, 100%, 95%, 95%, 70% und 50% EtOH und dann in entionisiertem Wasser. Wenn Sie nicht vorhaben, die mit einem Farbstoff, der keine delipidization Fleck, dann delipidization ist nicht erforderlich. Trockene gleitet gut unter einer Haube für mindestens 2-3 Stunden.
- In dem dunklen Raum mit einem sicheren Licht, aushöhlen genug Kodak NTB-Emulsion, die Hälfte der Folie in Längsrichtung (dh Gewebe) in Glas-Container Tauchen abdecken, und dann schmelzen in einem 42 ° C warmen Wasserbad für 20-30 min. Dann verdünnt es mit destilliertem Wasser im Verhältnis 1:1 zu. Die Höhe der Emulsion sollte nun alle Abschnitte auf einer Folie, wenn die Folie darin eingetaucht wird. Wenn Sie eine Menge von Dias haben, müssen Sie eventuell zusätzliche Emulsion herzustellen.
- Die Objektträger in die verdünnte Emulsion im Bereich von 42 ° C Wasserbad getaucht und trocken Folien in einem geschlossenen Behältnis Licht overnight in der Dunkelkammer oder in einem Ofen bei 37 ° C für 2-3 Stunden, mit Licht aus.
- Die Objektträger in Gestelle Schlitze in schwarzen Kisten mit Exsikkatoren, wobei die Folien vorsichtig einander nicht zu berühren und schaffen damit Artefakte. Seal die Ränder der Felder mit schwarzem Isolierband langsam auf statische Licht-induzierte Funken zu verhindern und dann wickeln Sie die Kästchen in Alufolie. Lagern Sie die Boxen bei 4 ° C mehrere Tage bis Wochen (Signal von 1 Tag auf Röntgenfilm ist ähnlich zu 5 Tage unter Emulsion).
- Wärmen Sie die Dia-Boxen auf Raumtemperatur für 1 Stunde.
- In der Dunkelkammer, entfernen Sie mit den Objektträgern (oder dem Ort Folien in einer Metall-Rack bei Verwendung von Kisten mit Dia-Slots) aus den Boxen und entwickeln sie in der Kodak D-19 Entwickler bei 16 ° C für 3,5 min.
- Waschen Sie die entwickelten Dias in Leitungswasser bei Raumtemperatur für 1 min.
- Die Objektträger zweimal in Fixierer bei 19 ° C für 6 Min. jeweils. Leuchtet auf während der zweiten Fixierbad Inkubation gedreht werden.
- Waschen Sie die Folien in fließendem Wasser bei Raumtemperatur für mindestens 30 min und kratzen die Emulsion von der Rückseite des Schlittens, während "nassen" mit einer Rasierklinge um Kratzer zu vermeiden das Glas.
- Stain Gewebe mit 0,3% Kresylviolett in Leitungswasser für 5 min.
- Waschen Sie zusätzliche Kresylviolett-Lösung in frischem Leitungswasser für ~ 15 Dips.
- Entwässern die Folien für ~ 15 taucht in jeder Alkohol-Lösung: 50%, 70%, 95%, 95%, 100% und 100% EtOH.
- Die Objektträger in Xylol für 5 min bei Raumtemperatur, zweimal.
- Deckglas mit Permount Medium auf der Folie bedeckt und trocknen Sie die Folie in der Haube über Nacht (> 16 h), es wird mehrere Tage dauern, bevor der Leim ist robust genug, um die Folien weiter zu reinigen.
6. Generieren Dunkelfeld Farbbilder
- Falls erforderlich, weitere saubere überschüssige Emulsion auf der Rückseite der Folien (nicht-coversliped Seite ohne Gewebe) durch Benetzung mit Wasser und Schaben mit einer Rasierklinge.
- Die Objektträger mit 80% EtOH-Lösung und wischen 1-2 mal sanft, um loszuwerden, Schutt und Staub.
- Nehmen Sie Bilder unter Dunkelfeld oder Hellfeld-Beleuchtung. Schritte 6.2 und 6.3 kann wiederholt 2-3 mal werden, um eine gute Bilder ohne Staubpartikel, die leicht im Dunkelfeld unter einem Präpariermikroskop gesehen zu erhalten.
7. Repräsentative Ergebnisse
Es gibt zwei Hauptwege, die sich in situ Hybridisierung Ergebnisse auf der Gewebeschnitte mit S 35 radioaktiven Sonden hybridisiert: 1) x-ray Film gibt, der die Folien oder 2 zugeordnet wurde) Emulsion, die auf den Folien beschichtet wurde. Ein dritter Ansatz wird mit Hilfe eines Phosphorimagerschirm über den Schlitten gelegt, aber wir haben nicht mit der Auflösung dieses Ansatzes erfüllt sind. X-ray-Filme bieten einen schnellen Ergebnis und Analysen von den allgemeinen Zustand der Hybridisierung. Der Röntgenfilm zeigt auch Daten breite anatomische Auflösung und kann zur quantitativen Analys verwendet werdenist 10. Beispiele für Röntgenfilm Bilder der späten Vogelembryo Köpfe mit Antisense-Sonden für FOXP1 und CoupTF2 Genexpression hybridisiert sind in den 1A und B. Beide Gene sind in großen Mengen im Gehirn spezifische Unterteilungen. Eine gute Qualität Röntgenfilm Ergebnis sollte scharf sein (nicht verschwommen) und haben einen hohen Signal-zu-Hintergrund-Verhältnis. Ein unscharfes Bild kann auf dem unebenen Kontakt zwischen einem Röntgenfilm und der Glasobjektträger mit der hybridisierten Gewebe.
Für getaucht Folien enthält die Emulsion lichtempfindlichen Silbersalze auf das Gewebe, um als der Kunststoff des Röntgenfilms angelagert beschichtet. Bei der Entwicklung, die S 35-exponierten Silbersalze umgewandelt Silberkörner metallischem, ähnlich wie in der Röntgenfilm. Allerdings sind die Silber-Lagerstätten direkt sichtbar in den Zellen darstellt, die Gen-Expression beobachtet und gemessen werden kann, qualitativ unter dem Mikroskop. Die metallische Silber Körner blockieren direktes Licht durchund wie die schwarzen Punkte unter Hellfeld Ansicht angezeigt werden. Die Kresylviolett Gegenfärbung erscheint in der Farbe lila (Abb. 3A, 3C und Abb. 4). Im Dunkelfeld, spiegeln die Silberkörnchen Licht von der Seite und erscheinen als weiße Punkte (Abb. 1C, 1D, 3B und 3D). In dieser Situation erscheint der Kresylviolett Fleck in der Farbe rot. Im Hellfeld ist das Hybridisierungssignal leichter unter hoher Vergrößerung auf zellulärer Auflösung anzuzeigen, während in Dunkelfeld, zusätzlich das Hybridisierungssignal unter geringer Vergrößerung über die gesamte Gewebe angesehen werden kann. Das Dunkelfeld Sicht ist der Ansatz, wir gemeinhin verwenden, um die Gesamt-Genexpressionsmuster zeigen. Allerdings bezogen auf die schnelle Folge von x-ray-Filme erhalten, nimmt die Emulsion getaucht Dias längere Zeit (eine bis mehrere Wochen) und ist empfindlicher auf die Erlangung Hintergrund.
Es gibt vier gemeinsame Quellen der starken Hintergrund: 1) Hintergrund der ganzen Röntgenfilm wird in der Regel tun, umProbleme mit dem Entwickler oder Fixierer, oder teilweise belichteten Film, 2) Hintergrund auf den Glasträger ist in der Regel aufgrund von Problemen mit Wasch-oder Glasträger Vorbereitungen, wie zB unsachgemäße silination von Folien aus der kommerziellen Quelle oder selbst erstellte, 3) Emulsion Belichtung und Entwicklung Hintergrund, und 4) auf dem Abschnitt Hintergrund entweder mit fehlender sorgfältiger Post-Hybridisierung Waschschritten zu niedrig von einer Hybridisierungstemperatur, schlechte Qualität der Hybridisierungslösung, Paraformaldehyd Kontamination in Abwasch verursacht Sonden dauerhaft zu vernetzen auf das Gewebe, Ribosonde Abbau führt zu kleinen Molekülen Markierung des Gewebes nicht speziell die inaktiven DTT oder β-Mercaptoethanol wodurch Vernetzung des S 35-RNA-Sonden in di-Sulfid-Bindungen an das Gewebe und zu lange warten für die Acetylierung. Es ist entscheidend, um die Folien bei der Acetylierung Lösung innerhalb von Sekunden nach Vermischen der Essigsäureanhydrid und Triethanolamin haben. Wenn mehrere Minuten vergehen without Zugabe der Lösung zu den Folien, dann Acetyl-Gruppen nicht effizient entfernt werden und dann binden um nicht-spezifisch RNA. Andere Faktoren sind die Hybridisierung über 20 h, die Erzeugung zu stark von einem Signal können, und überschüssiges Öl-Tröpfchen auf den Folien, die Hybridisierungslösung sequestrieren auf den Folien während der wässrigen Reiniger, was zu einer radioaktiven Flecken auf der Gewebe-und gleitet geben dunklen Hintergrund Signale. Wenn die Arbeit mit vielen Dias (mehr als 100 Scheiben), fügen Sie ein 3 rd Chloroform waschen oder ändern Sie die Chloroform-Wäschen, um überschüssiges Öl aus den verbleibenden Teilchen auf den Folien zu verhindern. Careless Gewebebehandlung, also nicht frieren schnell genug (innerhalb von 5-10 Minuten nach der Sektion) oder Auftauen und erneute Gefrieren erhöht auch Hintergrund durch Abbau der mRNA. Achten Sie darauf, Hintergrund für die Überbelichtung zu verwechseln.
Für Hintergrundinformationen über die Emulsion getaucht Dias ist möglich, weil es sehr lichtempfindlich ist und erfordert lange Belichtung im Dunkeln. Com Mo Hintergrund Problemen gehören eine zu hohe Temperatur für die Entwickler und Fixierer. Wenn die Temperatur höher als 19 ° C, in der Nähe oder wärmer als die Raumtemperatur ist, ist mehr Silberkorns Hintergrund erhalten. Die Exposition gegenüber geringen Mengen von Licht in eine Dunkelkammer undicht wird dazu führen, Emulsion Hintergrund. Nicht Auswaschen Fixierer lange genug (mindestens 30 min in Wasser), verlässt die Fixiererzusammensetzung reagiert dann mit Cresylviolett eine bräunlicher Niederschlag in der Emulsion zu erzeugen. Wenn jedoch die Folien in Wasser mehr als 90 min nach der Fixierung vor Cresylviolett Färbung gewaschen werden, kann dies dazu führen, dass die Emulsion sich lösen und die Abschnitte schlecht zu färben. Wenn es nicht genügend Zeit, um die Folien in einer 30-90 min Fenster Fleck nach Fixierung und Waschen, nach dem 30 min waschen, trocknen Sie die Folien über Nacht und fahren Sie mit Kresylviolett Färbung am nächsten Tag. Im Allgemeinen haben die meisten mRNAs spezifischer Genexpressionsmuster, während Hintergrund-Signal ist einheitlicher.
e_content "> Gefaltete Gewebe könnte für die Genexpression Ergebnisse irreführend sein auf Röntgenfilm, was zu einer Region mit dunkler Signal. Um festzustellen, ob das Gewebe gefaltet, untersuchen nicht-gefärbten Schnitten unter Dunkelfeld oder Kresylviolett gefärbten Schnitten unter Hellfeld. Cresyl Violett Gegenfärbung bietet einen besseren Weg, um das Gewebe Zustand zu untersuchen.Für eine bessere Interpretation der Sonde Spezifität, sollte Regelsinn Sonden auf mehrere benachbarte Abschnitte angewendet werden. Die meisten Sinn Sonden zeigen nicht ein Signal, aber einige tun, und wenn sie es tun, finden wir, dass es oft anders als die Antisense-Signal. Wir glauben, dass diese in Verbindung stehen könnten, um die Synthese oder einem anderen Gen auf dem Antisense-Strang des Genoms Antisense werden. Wir ein Beispiel Pax6 Sense-und Antisense-Sonden (Abb. 2A und B). Der Antisense-Strang zeigt die Kennzeichnung entlang der ventrikulären Zone des Vorderhirn, Kleinhirn und Auge wie erwartet (Abb. 2A), aber der Sinn enthüllt laBeling in der Pigmentschicht der Netzhaut (Abb. 2B).
Für Sonde Größen verwenden wir cDNA-Sonden überall im Bereich von 300-5000 bps. Sonden weniger als 300 bps Arbeit, aber die Signale sind in der Regel schwächer. Wir haben nicht Sonden größer als 5000 bps versucht. Am besten ist es Sonden, die auf Gewebe aus der gleichen Spezies zu verwenden, wenn möglich. Wenn nicht, wir kreuzhybridisieren Sonden auf Abschnitten der anderen Arten und verringert die Hybridisierung und Waschen Temperaturen in 3-5 ° C-Schritten auf Sequenzidentität basieren, sofern bekannt. Wenn nicht bekannt ist, dann führen wir trial and error-Hybridisierung und Waschtemperaturen. Wenn die Temperatur zu weit abgesenkt wird, kann die cDNA-Sonde mit anderen mRNAs von ähnlichen Sequenzen in Art oder in der gleichen Spezies 11 kreuzhybridisieren. In der Praxis finden wir, dass cDNAs, die ~ 95% oder mehr identisch mit der mRNA-Ziel im Gewebe, die stringenten Hybridisierungs-und Waschtemperatur (65 ° C) Bedingungen sind gut funktioniert. Für Sequenzen, die in der klingelte sindes von ~ 85 bis 94% identisch ist, die Hybridisierung und Waschtemperatur müssen möglicherweise in dem Bereich von ~ 50 bis 60 ° C gesenkt werden
Der Grund für die Verwendung eines Metall-Gestell in den meisten der Schritte ist die Verwendung von Chloroform wäscht und Xylol. Beide organischen schmelzen viele Arten von Kunststoffen. Glas und einige Arten von Kunststoffen sind resistent gegen diese organischen Verbindungen. Doch Glas ist leichter zu brechen, und einige Kunststoffe, die zunächst sind resistent werden über lange Zeiträume-Exposition gegenüber den organischen schmelzen.
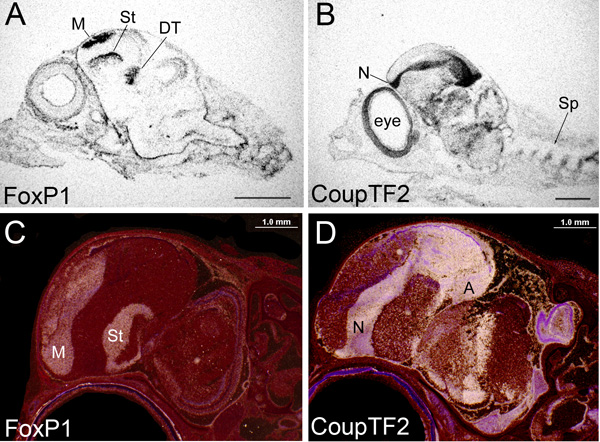
Abbildung 1. Autoradiographie von in-situ-Hybridisierung Bilder von x-ray Filme und Dias getaucht. (AB) Röntgenfilm Bilder der sagittalen ganzen Kopf Abschnitte des Zebrafinken Singvogel an embryonalen Tag 10, mit Antisense Ribosonden zu (A) FOXP1 oder (B) CoupTF2, mit Hellfeld-Beleuchtung unter einem Binokular genommen hybridisiert. Schwarz, ausgesetzt Körner im Film, der zeigt mRNA-ExpressIonen. Maßstab = 500 um. (CD) getaucht slide Bilder der sagittalen ganzen Kopf Abschnitte des Zebrafinken am Tag nach dem Schlüpfen 6, hybridisiert mit antisense Ribosonden bis (C) FOXP1 und (D) CoupTF2, mit Dunkelfeldbeleuchtung unter einer Dissektion Lupe genommen. Weiß-, Silber-Körner in der Emulsion oben Gewebe zeigen mRNA Expression ausgesetzt. Rot, Kresylviolett Fleck. Maßstab = 200 um. Für alle Bild, ist der Schnabel rostral nach links. Die Röntgenfilm Bilder wurden für einen Tag, getaucht Dias für 3 Tage ausgesetzt. Die Sonde ist FOXP1 178 bps auf die 1544-1711 bp Teil der mRNA; CoupTF2 ist 545 bps auf die 1-545 bp Teil der mRNA. Wie ersichtlich, werden in das Vorderhirn FOXP1 mRNA in der mesopallium (M), Striatum (St) und dorsalen Thalamus (DT) angereichert ist, während in der CoupTF2 nidopallium (N), arcopallium (A) angereichert ist, und ventralen Thalamus . Es besteht Übereinstimmung im Ausdruck zwischen der Exposition Typen (und Alter). Mit den getauchten Folien, jedoch eine höhere Auflösung Kennzeichnung gesehen, eined Gewebe Grenzen und die Untergliederung werden direkt identifiziert werden. Diese und alle anderen Bilder in der Zeitung gezeigt, sind aus den Abschnitten mit dem Standard-65 ° C mit hoher Stringenz Hybridisierung.
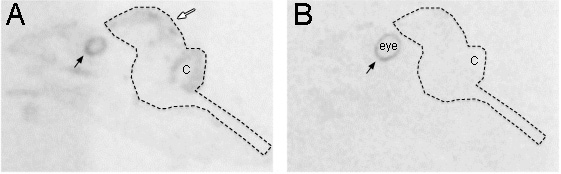
Abbildung 2. Vergleich von Antisense-und Sense Kennzeichnung, die unterschiedliche Muster zeigt. (A) Die Antisense-Strang von Pax6 wurde im Gehirn, insbesondere der ventrikulären Zone (weißer Pfeil) ausgedrückt. Gezeigt ist die Autoradiographie auf Röntgenfilmen der sagittalen ganzen Kopf Scheiben von Zebrafinken am embryonalen Tag 12 genommen. (B) benachbarten Abschnitt mit dem Sense-Strang von Pax6 hybridisiert zeigt keine Background-Expression in den Embryo Kopf, aber deutlich Expression in der Pigmentschicht der Netzhaut (schwarze Pfeile) als Antisense-Strang. Die gestrichelte Linie zeigt die Kontur des gesamten Gehirns. C: Kleinhirn.
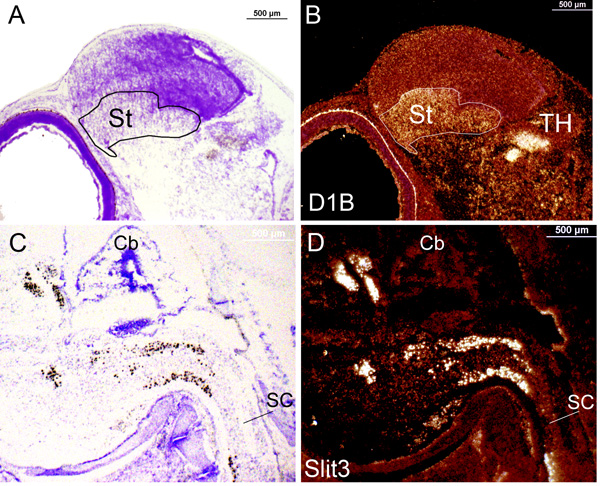
rong> Abbildung 3. In-situ-Signale der Genexpression in Emulsion getaucht Dias von Zebrafinken Gehirn während der späten embryonalen Stadien unter Hell-und Dunkelfeld Aussicht genommen. (A) Hellfeldaufnahme D1B Ausdruck an embryonalen Tag 10 von einer normalen Belichtung der Emulsion. Das Label (schwarz) kann kaum bei dieser Vergrößerung gesehen werden. Probe ist 625 bps auf die 1-625 bp Teil der mRNA. (B) Identische Abschnitt und Vergrößerung wie in (A) wechselte aber zum Dunkelfeld Ansicht Etikett (weiß) im Striatum (St) und Thalamus (TH). (C) Hellfeldaufnahme Slit3 Ausdruck an embryonalen Tag 12 von einer Überbelichtung zu der Emulsion. Label (schwarz) kann leicht gesehen werden. Probe ist 779 bps auf die von 1243 bis 2021 Teil der mRNA. (B) Identische Abschnitt und Vergrößerung wie in (A) eingeschaltet, um Dunkelfeld Ansicht Etikett (weiß) im Rückenmark (SC), die das Hellfeld Bild übereinstimmt. Rostra orientiert sich an der linken Seite. Cb: Kleinhirn.
/ 3764/3764fig4.jpg "alt =" Bild 4 "/>
Abbildung 4. Silber Korn Auflösung auf zellulärer Ebene. Dargestellt ist FOXP1 mRNA-Label in der Zebrafinken Vorderhirn mit Silberkörner (schwarze Punkte) oberhalb Zellen (Kresylviolett) in verschiedenen Hirnregionen und Alters verglichen mit niedrigerer Leistung Bildern der 1A und 1C. (A) Hohe Fülle Ausdruck über einzelne Zellen (schwarze Pfeile) in der erwachsenen Zebrafinken mesopallium. (B) geringer Menge Ausdruck über einzelne Zellen (schwarze Pfeile) in der benachbarten nidopallium der gleichen Rubrik. (C) Hohe FOXP1 mRNA-Expression in Zellen (schwarze Pfeile) in der embryonalen Tag 12 mesopallium. (D) geringer Häufigkeit der Expression über Zellen (schwarze Pfeile) in der benachbarten nidopallium den gleichen Querschnitt. Beispiel Zellen werden mit einer gelben Linie eingekreist. Embryonalen Zellen (C und D) sind kleiner und enger im Vergleich zu den adulten Zellen (A und B) verpackt. Die da einige Raum zwischen den Zellen und der Emulsion im Bereich der freigelegten Silver Körner aus dem S 35-Sonde sind etwas groß ist als die Fläche der Zellkörper. Balken = 10 um.
Diskussion
Radioaktive in situ-Hybridisierung der mRNA-Expression wird häufig für verschiedene Zwecke, einschließlich der für die Untersuchung regionalen Gewebeorganisation, Zelltypen und Gehirn funktionelle Aktivität 2-5,10,12-14 verwendet. Die spätere Verwendung auf Gene, deren mRNA-Expression im Gehirn ist abhängig von erhöhten neurale Aktivität, die oft als leistungsabhängige Gene oder unmittelbar frühen Gene. Mit diesen Anwendungen hat sich unser Verfahren auf mehrere Arten angewendet worden, darunter in Vögel, Säugetiere (zB menschliche), Fische und Amphibien; in mehreren Geweben, einschließlich Gehirn, Haut und Muskel, und mehrere Altersklassen, einschließlich Jungtiere / Neugeborenen, Jugendlichen , Erwachsene, und hier im ganzen Embryo Abschnitten 2,3,5,15-17. Die besonderen Merkmale unseres Protokolls sind: (1) Es erzeugt ein Gleichgewicht zwischen anatomischen Besonderheit und quantitative Spezifität. Um die Genexpression auf dem Röntgenfilm zu quantifizieren, wir digitale Bilder der Bilder (. Bsp.: Abb. 1A und 1B) nehmen, verwenden Sie die Photoshop (Adobe) Histogramm-Funktion, die Pixeldichte in den Regionen von Interesse zu messen und subtrahieren die Hintergrundwerte auf dem Film außerhalb des Gewebes, aber immer noch auf dem Objektträger 2,4. Um die Expression auf zellulärer Ebene zu quantifizieren, nehmen wir Bilder von Silberkörnern über Zellen bei starker Vergrößerung (40-100X; Abb. 4). Wir verwenden die Schwelle und Messfunktionen der Bild J von Wayne Rasband am NIH, die Anzahl der Silberkörner in Bildes zu zählen, subtrahieren den Hintergrund Anzahl in einem ähnlichen Bereich ohne Zellen auf dem Objektträger, durch die Anzahl der Zellen teilen, die a-Werte von Expression pro Zelle. 4,18 (2) relativ hohen Durchsatz sein kann, damit die Verarbeitung von 100-200 Folien gleichzeitig, durch die dichte Abdichtung durch das Deckglas Mineralölbad angelegt zu erhalten. Standard-in-situ Hybridisierung Methoden länger dauern, um die Folien mit Parafilm, Nagellack und andere Mittel, wo Dias nehmen viel Raum zu versiegeln, (3) Es ist höchstempfindlich für geringe Häufigkeit Transkripte durch Dextransulfat und Denhardt-Lösung im Hybridisierungspuffer 2,13, (4) Die Bildgebungsverfahren im Dunkelfeld liefert Bilder mit hohem Kontrast durch Fotografieren unter Dunkelfeldbeleuchtung auf einem Präpariermikroskop 4,5. Darüber hinaus ermöglicht es empfindliche Detektion von kleinen Veränderungen in der Genexpression, wie in Aktivitäts-abhängigen Genexpression auf bestimmte Hirnregionen während der Wahrnehmung und Produktion von spezifischen Verhaltensweisen 19 aktiviert zu identifizieren. Die Einschränkungen in Bezug auf nicht-radioaktiven Protokolle sind, dass diese deutlicher in zellulärer Auflösung und die Position der mRNA in der Zelle sind, und die Arbeit mit Emulsion ist sehr empfindlich gegenüber Manipulation und Licht. Es ist möglich, unsere Methode mit anderen Methoden, wie nicht-radioaktive in situ-Hybridisierung an mRNA-Expression von mehr als einem Gen in dem gleichen Gewebe 10,17,20 kennzeichnen kombinieren. Es kann mit Immunzytochemie kombiniert werden, beide RNA-und Protein-Expression mit der gleichen Probe zu kennzeichnen, um die Co-Lokalisation der mRNA mit bestimmten Zelltypen 10. Die Protokoll-Modifikationen notwendig für eine solche doppelte Kennzeichnung Experimente sind in den zitierten Referenzen beschrieben.
Zusammenfassend ermöglicht unser Ansatz Verständnis der Zeitpunkt und die zelluläre Lokalisation der Genexpression, um das Verständnis Region Organisation, Gewebe funktionelle Aktivität und Genfunktionen.
Offenlegungen
Wir haben nichts zu offenbaren.
Danksagungen
Die Autoren möchten allen Jarvis Labor Mitglieder, die das Protokoll über die Jahre verbessert danken.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenzes | Firma | Katalog-Nummer | Kommentare |
| Essigsäureanhydrid | VWR | MK242002 | |
| Chloroform | VWR | BDH1109 | |
| Kresylviolett Acetat | Sigma | C5042 | |
| Kryostat | Thermo Scientific | Microm HM550 | |
| Deionisiertem Formamid | Sigma | F9037 | |
| DTT | Promega | P1171 | 100 mM |
| EDTA | Sigma | ED | |
| Einbettform | VWR | 15160-215 | |
| Marke Fisher Superfrost plus Rutsche | Fisher Scientific | 22-034-979 | |
| Formaldehyd | VWR | BDH0506-4LP | |
| Formamid | Sigma | F7508 | |
| Kit GENECLEAN | Q-Bio-Gen | 1001-200 | |
| Kodak Biomax MR Film | Sigma | Z350370 | |
| Kodak NTB Emulsion | Carestream Health | 8895666 | |
| KODAK PROFESSIONAL Entwickler D19 | Kodak | 1462593 | |
| Kodak Professional Fixer | Kodak | 1971746 | |
| β-Mercaptoethanol | Calbiochem | 444203 | |
| Mineralöl | VWR | IC15169491 | |
| NaOH | VWR | SX0600-1 | |
| Paraformaldehyd | Sigma | 76240 | |
| Poly-A | Invitrogen | POLYA.GF | |
| rATP | Promega | P1132 | 10 mM |
| rCTP | Promega | P1142 | 10 mM |
| rGTP | Promega | P1152 | 10 mM |
| RNasin | Promega | N2111 | 40Units/μl |
| S 35 UTP | PerkinElmer | NEG039C001MC | |
| Sicherheits-Solve-Lösung | Sicherheit Solve Research Products International | 111177 | |
| Natriumacetat | Sigma | S7899 | 3M |
| Dinatriumhydrogenphosphat | Sigma | S3264 | |
| Mononatriumphosphat | Sigma | S3139 | |
| SP6RNA-Polymerase | Promega | P1085 | |
| Die Färbung Metallgestell | Electron Microscopy Sciences | 70312-54 | |
| T7-RNA-Polymerase | Promega | P2075 | |
| Tissue-Tek Oktober | Sakura | 4583 | |
| 5x Transkription Puffer | Promega | P1181 | |
| Triethanolamin | VWR | IC15216391 | |
| Tris-HCl (1 M, pH 8,0) | VWR | 101449-446 | |
| tRNA | Roche | 10109509001 |
Lösungen:
- Reagenzien für die Herstellung von Ribosonden: 1,5 ul DNA-Template (0,2-0,3 ug / ul), 2 ul 5x optimierte Transkription Puffer, 1 ul 100 mM DTT (kommt mit Polymerase von Promega, mixauch bei Raumtemperatur), 0,3 ul RNasin (40 Einheiten / ul), 1,5 ul AGC Ribonukleotid-Mix-Lösung, 3,5 ul der S 35 UTP, und 1 ul RNA-Polymerase. Bringt bis zu 10 ul mit Nuklease-freiem Wasser.
- AGC Ribonukleotid-Mix-Lösung: Mix gleiche Mengen von 10 mM ATP, GTP und CTP zusammen.
- Natriumacetat-Lösung für EtOH Niederschläge auf freie S 35 UTP entfernen: 40 ul RNase und DNase freies Wasser, 5 ul 3 M Natriumacetat und 125 ul 100% EtOH.
- Hybridisierungspuffer (10ml Lager): 5 ml 100% deionisiertes Formamid, 600 ul 5 M NaCl, 1 M Tris-HCl (pH = 8,0), 240 ul 0,5 M EDTA (pH = 8,0), 100 ul 100x Denharts Lösung, 100 ul 1 M DTT, 250 ul 20mg/ml tRNA, 125 ul 20 mg / ml Poly A, und 1 g Natriumdextransulfat. In DEPC-behandeltem Wasser auf ein Gesamtvolumen von 10 ml zu bringen. Kräftig schütteln und dann inkubieren bei 55 ° C, bis alle Natriumdextransulfat aufgelöst wird. Lagern Sie den Hybridisierungspuffer in-20 ° C, was gut für ~ 6 Monate ist.
- 4% gepuffertem Paraformaldehyd-Lösung: Fügen Sie 40 g Paraformaldehyd in 760 ml destilliertem Wasser in einen Kolben für Paraformaldehyd bezeichnet, Hitze bis 50 ° C auf einer Heizplatte unter Rühren zugeben. Fügen Sie 320 ul von 10N NaOH um das Lösen des Paraformaldehyd. Nach dem Auflösen (~ 10 min), 100 ml 10x PBS, und bringe das Volumen mit destilliertem Wasser bis 1 Liter. Umrühren und erhitzen Sie die Lösung bis Paraformaldehyd gelöst ist. Der pH-Wert sollte 7,4 sein.
- Acetylierung Puffer: 13,6 ml Triethanolamin und 2,52 ml Acetanhydrid in 1 Liter destilliertem Wasser.
- 20x SSPE-Lösung: 3M NaCl, 200 mM NaH 2 PO 4-H 2 O und 200 mM EDTA in destilliertem Wasser. Passen Lösung auf pH 7,4 mit 10 N NaOH.
- 10x PBS-Puffer: 80 g NaCl, 2,0 g KCl, 14,4 g Na 2 HPO 4 und 2,4 g KH 2 PO 4 in destilliertem Wasser. Passen Lösung auf pH 7,0 und bringen Gesamtvolumen auf 1 liter.
- Zweiten Waschlösung: 0,1% β-Mercaptoethanol und 50% Formamid in 2x SSPE
- Kresylviolett-Acetat-Lösung: 3% Kresylviolett Acetat in Leitungswasser (destilliertes Wasser verhindert, dass eine gute Färbung), über Nacht unter Rühren in einem Kolben bei Raumtemperatur gelöst. Filter mit Vakuumabsaugung durch ein 1mm Whatman-Filterpapier und Büchner-Trichter.
Referenzen
- Clayton, D. F., Huecas, M. E., Sinclair-Thompson, E. Y., Nastiuk, K. L., Nottebohm, F. Probes for rare mRNAs reveal distributed cell subsets in canary brain. Neuron. 1, 249-261 (1988).
- Wada, K., Sakaguchi, H., Jarvis, E. D., Hagiwara, M. Differential expression of glutamate receptors in avian neural pathways for learned vocalization. J. Comp. Neurol. 476, 44-64 (2004).
- Haesler, S. FoxP2 expression in avian vocal learners and non-learners. J. Neurosci. 24, 3164-3175 (2004).
- Jarvis, E. D., Nottebohm, F. Motor-driven gene expression. Proc. Natl. Acad. Sci. U.S.A. 94, 4097-4102 (1997).
- Holzenberger, M. Selective expression of insulin-like growth factor II in the songbird brain. J. Neurosci. 17, 6974-6987 (1997).
- Mahmood, R., Mason, I. In-situ hybridization of radioactive riboprobes to RNA in tissue sections. Methods Mol. Biol. 461, 675-686 (2008).
- Wilkinson, D. G., Nieto, M. A. Detection of messenger RNA by in situ hybridization to tissue sections and whole mounts. Methods Enzymol. 225, 361-373 (1993).
- Acloque, H., Wilkinson, D. G., Nieto, M. A. In situ hybridization analysis of chick embryos in whole-mount and tissue sections. Methods Cell Biol. 87, 169-185 (2008).
- Moorman, A. F., Houweling, A. C., de Boer, P. A., Christoffels, V. M. Sensitive nonradioactive detection of mRNA in tissue sections: novel application of the whole-mount in situ hybridization protocol. J. Histochem. Cytochem. 49, 1-8 (2001).
- Horita, H., Wada, K., Rivas, M. V., Hara, E., Jarvis, E. D. The dusp1 immediate early gene is regulated by natural stimuli predominantly in sensory input neurons. J. Comp. Neurol. 518, 2873-2901 (2010).
- Braissant, O., Wahli, W. A simplified in situ hybridization protocol using non-radioactively labelled probes to detect abundant and rare mRNAs on tissue sections. Biochemica. 1, 10-16 (1998).
- Kubikova, L., Wada, K., Jarvis, E. D. Dopamine receptors in a songbird brain. J. Comp. Neurol. 518, 741-769 (2010).
- Wada, K. A molecular neuroethological approach for identifying and characterizing a cascade of behaviorally regulated genes. Proc. Natl. Acad. Sci. U.S.A. 103, 15212-15217 (2006).
- Jarvis, E. D. Avian brains and a new understanding of vertebrate brain evolution. Nat. Rev. Neurosci. 6, 151-159 (2005).
- Hoke, K. L., Ryan, M. J., Wilczynski, W. Social cues shift functional connectivity in the hypothalamus. Proc. Natl. Acad. Sci. U.S.A. 102, 10712-10717 (2005).
- Burmeister, S. S., Jarvis, E. D., Fernald, R. D. Rapid behavioral and genomic responses to social opportunity. PLoS. Biol. 3, e363 (2005).
- Jarvis, E. D., Schwabl, H., Ribeiro, S., Mello, C. V. Brain gene regulation by territorial singing behavior in freely ranging songbirds. Neuroreport. 8, 2073-2077 (1997).
- Jarvis, E. D., Scharff, C., Grossman, M. R., Ramos, J. A., Nottebohm, F. For whom the bird sings: context-dependent gene expression. Neuron. 21, 775-788 (1998).
- Feenders, G. Molecular mapping of movement-associated areas in the avian brain: a motor theory for vocal learning origin. PLoS One. 3, e1768 (2008).
- Chen, C. C., Fernald, R. D. Distributions of two gonadotropin-releasing hormone receptor types in a cichlid fish suggest functional specialization. J. Comp. Neurol. 495, 314-323 (2006).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten