Method Article
Radioativo In situ Hibridação para detecção de padrões de expressão dos genes nos tecidos diversos
Neste Artigo
Resumo
Este protocolo é usado com sucesso para detectar quantitativamente os níveis e padrões espaciais de expressão de mRNA em vários tipos de tecido em todas as espécies de vertebrados. O método pode detectar transcritos baixa abundância e permite o tratamento de centenas de lâminas simultaneamente. Apresentamos este protocolo com perfil de expressão de formação cérebro das aves embrionárias como um exemplo.
Resumo
Sabendo o tempo, o nível, a localização celular, e tipo de célula que um gene é expresso em contribui para a nossa compreensão da função do gene. Cada uma destas características pode ser realizado com a hibridização in situ para mRNAs dentro das células. Aqui apresentamos uma radioativo no método de hibridização in situ modificado de Clayton et al. (1988) 1 que tem trabalhado com sucesso em nosso laboratório por muitos anos, especialmente para adultos cérebros vertebrados 2-5. Os longos complementares de RNA (cRNA) sondas com a sequência alvo permite a detecção de transcritos baixa abundância 6,7. Incorporação de nucleótidos radioactivos para as sondas crna permite ainda mais a sensibilidade de detecção de transcritos baixa abundância e análises quantitativas, quer pela luz filme de raios-X sensível ou emulsão revestido sobre o tecido. Estes métodos de detecção de fornecer um registo de longo prazo da expressão do gene alvo. Em comparação com os não-radioativo sonda methODS, tais como DIG-rotulagem, o método de hibridação da sonda radioactiva não requer passos de amplificação múltiplas usando HRP-anticorpos e / ou TSA kit para detectar transcritos baixa abundância. Portanto, este método proporciona uma relação linear entre a intensidade do sinal e quantidades de mRNA-alvo para a análise quantitativa. Ele permite o processamento simultâneo 100-200 lâminas. Ele funciona bem para diferentes estágios de desenvolvimento de embriões. A maioria dos estudos de desenvolvimento de expressão do gene utilizar embriões inteiros e não radioactivos abordagens 8,9, em parte porque o tecido embrionário é mais frágil do tecido adulto, com menos de coesão entre as células, tornando-a difícil de ver os limites entre as populações de células com secções de tecido. Em contraste, a nossa abordagem radioactivos, devido à maior gama de sensibilidade, é capaz de obter maior contraste na resolução da expressão do gene entre as regiões de tecido, tornando-o mais fácil de ver os limites entre as populações. Usando este método, os pesquisadores poderiam revelar apossível significado de um gene recentemente identificado, e ainda prever a função do gene de interesse.
Protocolo
1. Preparação de Tecidos
- Colher tecidos frescos. Para os embriões de aves, ovos cuidadosamente abrir e limpar o embrião em um prato com 1xPBS, duas vezes. Para cérebros adultos, remover rapidamente o cérebro e lavar cuidadosamente com 1 x PBS.
- Incorpore embriões ou cérebro adulto em um molde incorporado cheia de outubro tecido tek, orientando o tecido conforme necessário para cortar e congelar rapidamente o bloco, colocando-o em uma mistura de etanol e gelo seco, tomando cuidado para não começar a mistura dentro do bloco .
- Corte a amostra congelada em um criostato em 10-12 mM de espessura. Para o tecido cerebral e embrionário, a melhor temperado de corte está dentro do intervalo de -18 ° C a -20 ° C.
- Monte cortes congelados em slides Super Plus vidro.
- Guarde as seções de uma caixa de lâminas a -80 ° C.
2. Geração de Riboprobes radioativos (Utilize procedimentos de segurança de radiação da sua instituição)
- Gerar um DN purificada linearUm modelo da sua ADNc de interesse, quer por digestão enzimática restrito de um fragmento clonado rodeado por locais de RNA polimerase promotor de um DNA de plasmídeo, ou por PCR do inserto ligado aos locais de polimerase de RNA de promotor de ligação. Também é possível gerar fragmentos de PCR com os promotores da polimerase de RNA, como parte da extremidade 3 'e 5' iniciadores de PCR. Géis purificar suas restritas ou PCR fragmentos gerados com o kit GENECLEAN gel purificação.
- Para um tubo Eppendorf 0,5 ml, adicionar 0,5-1 ug de molde de ADN purificado linear, tampão de transcrição, DTT, RNasin, AGC solução mistura de nucleótidos, e RNase água livre para levar o volume até a quantidade desejada. Em seguida, adicione S 35-UTP e da RNA polimerase adequada para fazer riboprobes quer antisense ou sentido.
- Incubar a mistura durante 1 hora num banho de água 37 ° C. Adicionar outra aliquota de RNA polimerase e incubar durante outra 1 h.
- Adicionar 3M solução de acetato de sódio (0,1 vezes do volume total) e 100% de EtOH (2,5 fold do volume total) dentro do tubo de Eppendorf para precipitar o sintetizado riboprobe RNA.
- Incubar em gelo seco ou -80 ° C durante 15 min ou mais de 3 horas para auxiliar a precipitação.
- Pelete de RNA por centrifugação a 4 ° C e 15.000 rpm numa centrífuga de bancada Eppendorf durante 30 min.
- Remover o sobrenadante e lava-se o sedimento com EtOH a 70%, toque com o dedo para misturar o sedimento bem.
- Pelete de RNA de novo por centrifugação a 4 ° C e 15.000 rpm durante 30 min.
- Remover o sobrenadante completamente por pipetagem e depois adicionar 40 uL solução de hibridação. Misture bem por pipetagem dentro e para fora.
- Coloque 1 ul da solução em 3 ml de solução de Segurança-Solve na mistura de cintilação frasco, bem, e medir as contagens no contador de cintilação.
- Coloque a Eppendorf com o riboprobe a -20 ° C para utilização dentro de uma semana.
3. Tratamento tecido
- Em um capuz, preparar uma nova solução tampão PBS paraformaldehy 4%de solução a cerca de 3-5 ° C acima da temperatura ambiente. Colocar as lâminas de -80 ° C de armazenamento em uma prateleira de metal em gelo seco, para transportar espaço de trabalho sob a capa, e depois colocar o suporte para a solução de paraformaldeído a 4%, e incubar durante 5 min à temperatura ambiente.
- Lavar 3 vezes em 1 x PBS a cerca de 15 depressões cada (cerca de 1 segundo por mergulho).
- Na capa, fazer tampão acetilação e agite-a vigorosamente dentro de 10 segundos. Imediatamente verter em uma bandeja que contém o bastidor de lâminas e incubar durante 10 min, para reduzir a ligação de fundo de riboprobe.
- Lavar 3 vezes em 2 x SSPE, durante 15 depressões.
- Desidratar em 70%, 95% e 100% de série EtOH durante 2 min em cada passo (não tem de estar na capa).
- Secam-se as lâminas sob a capa, pelo menos, 10-15 min.
4. Hibridização
- Calcular quantidade de riboprobe (0,5-1x10 6 cpm por slide) e solução de hibridação (100 ul por slide), necessário para todos os slides. Prewaa mistura rm riboprobe-hibridação a 65 ° C durante 5 min para desnaturar o riboprobe.
- Pipete 100 de riboprobe hibridização solução em uma linha que atravessa o slide, e usar uma lamela de vidro para espalhar uniformemente sobre o tecido e lamela. Lugar desliza horizontalmente, de frente para vertical num bastidor de metal e cremalheira lugar lentamente na posição vertical dentro do banho de óleo 65 ° C durante um mínimo de 4 horas e um máximo de 16 horas. O óleo cria uma vedação hermética em torno das lamelas.
- Remover prateleiras de metal do banho de óleo e limpe o excesso de óleo em torno de rack com papel de seda.
- Lavar óleo das lâminas cobertas e cremalheira de metal em vidro ou metal bandejas contendo clorofórmio, duas vezes. A 2 ª clorofórmio de lavagem pode ser utilizado como o primeiro para a experiência seguinte.
- Transfira o rack com lâminas cobertas em uma bandeja com em 0,1% + 2 x β-mercaptoetanol solução SSPE e mover para cima e para baixo por alguns poucos mergulhos para afrouxar e remover lamínulas dissolver o excesso de hibridização soluçãoção.
- Transferir a cremalheira com lâminas abrangidos em solução fresca de 0,1% β-mercaptoetanol + 2 x SSPE, e depois remover lamelas com uma pinça de RNase livre em solução para evitar arranhões secções de tecido. Transferir as lâminas descoberto em cremalheira fresco em um suporte de prateleira corrediça horizontal bandeja, em uma solução de 0,1% β-mercaptoetanol em 2 x SSPE.
- Incubar a cremalheira com lâminas na fresco 0,1% β-mercaptoetanol + 2 x SSPE solução à temperatura ambiente durante 1 h para remover o excesso de RNA não acoplado sonda. Descartar este e os anteriores soluções de lavagem aquosas, bem como lamelas, como resíduos radioactivos.
- Transferir a cremalheira com lâminas para um pré-aquecido solução SSPE 2x a 65 ° C, adicionar β-mercaptoetanol para uma concentração final de 0,1%, e incubar a 65 ° C durante 1 hora. Descartar como resíduos radioactivos.
- Transferir e incubar a cremalheira com lâminas duas vezes em pré-aquecido 0,1 x SPPE a 65 ° C durante 30 min cada. A quantidade de radioactividade removido ineste passo é muito pequeno e não é mais considerado resíduos radioactivos excessiva após esta etapa.
- Desidratar o suporte e as lâminas em 70%, 95% e EtOH a 100% durante 2 min cada.
- Seque as lâminas na capa, pelo menos, 30 min.
5. Visualização do sinal radioativo
- Coloque as lâminas secas em um cassete de filme e em um quarto escuro, coloque o filme de raios-X (Kodak BioMax filme MR) sobre as lâminas, e fechar a gaveta. Certifique-se que os slides estão virados para o lado da emulsão do filme x-ray. Expor as lâminas para ~ 1-7 dias, dependendo abundância esperado das transcrições.
- Desenvolver o filme x-ray no padrão desenvolvedor e fixador. O sinal de hibridação mostra-se como negro (expostos grãos de prata na emulsão) sobre a película (Fig. 1).
- (Opcional) Para determinar a resolução do sinal celular e encontra sobre o tecido, as lâminas necessitam de ser mergulhado em emulsão fotográfica e contracoradas.Se vai para eventualmente contracoloração com violeta de cresilo, em seguida, delipidize secções por incubação em xileno durante 5 min à temperatura ambiente, duas vezes, re-hidratar 1 min cada um em 100%, 100%, 95%, 95%, 70%, e 50% de EtOH e, em seguida, em água desionizada. Se você não está indo para corar com um corante que não requer delipidization, então delipidization não é necessário. Lâminas secas bem sob uma capa de pelo menos 2-3 horas.
- Na sala escura com uma luz de segurança, retirar o suficiente Kodak NTB emulsão para cobrir metade do comprimento de slides (tecido, por exemplo) em recipiente de vidro de imersão, e depois derreter em banho-maria a 42 ° C por 20-30 min. Em seguida, dilui-lo com água destilada para relação de 1:1. O nível da emulsão deve agora cobrir todas as secções de um slide quando a lâmina é mergulhada no mesmo. Se você tem um monte de slides, você pode precisar preparar emulsão extra.
- Dip desliza para dentro da emulsão diluída em 42 ° C banho de água e secas as lâminas mergulhadas em um recipiente fechado apertado luz overnight na sala escura, ou numa estufa a 37 ° C durante 2-3 horas, com luzes desligadas.
- Transferir as lâminas em slots prateleiras em caixas-pretas contendo dessecadores, tendo o cuidado para as lâminas não se toquem e, assim, criar artefatos. Selar as arestas das caixas com fita isolante preta lentamente para evitar faíscas estático induzidas leves e, em seguida, embrulhar as caixas em folha de alumínio. Armazenar as caixas a 4 ° C a partir de vários dias a semanas (sinal a partir de 1 dia em filme de raios X é semelhante a 5 dias sob emulsão).
- Aquecer as caixas deslizantes até à temperatura ambiente durante 1 hora.
- Na câmara escura, remova a grelha com os slides (diapositivos ou coloque em uma prateleira de metal se usar caixas com aberturas de slides) das caixas e desenvolvê-los no D-19 Kodak desenvolvedor em 16 ° C por 3,5 min.
- Lavar as lâminas desenvolvidos na água da torneira à temperatura ambiente durante 1 min.
- Incubar as lâminas duas vezes em fixador a 19 ° C durante 6 min cada. As luzes podem ser ativados durante a incubação fixador segundo.
- Lavar as lâminas em água corrente, à temperatura ambiente durante pelo menos 30 min e raspar a emulsão a partir do lado de trás da lâmina, enquanto 'molhado' com uma lâmina de barbear para evitar riscar o vidro.
- Stain tecido com 0,3% de violeta de cresilo em água da torneira durante 5 min.
- Lave solução de violeta cresil extras na água da torneira fresco para cerca de 15 mergulhos.
- Desidratar as lâminas para ~ 15 depressões em cada solução de álcool: 50%, 70%, 95%, 95%, 100% e EtOH a 100%.
- Incubar as lâminas em xileno durante 5 min à temperatura ambiente por duas vezes.
- Lamínula com meio Permount no slide e seque a lâmina coberta no bairro durante a noite (> 16 horas), que irá demorar vários dias até que a cola é resistente o suficiente para limpar as lâminas mais.
6. Geração de imagens coloridas de campo escuro
- Se necessário, ainda mais limpo emulsão excesso na parte de trás das lâminas (não-coversliped lado sem tecido) molhando-o com água e raspagem com uma lâmina de barbear.
- Lavar as lâminas com solução EtOH 80% e limpe 1-2 vezes, com cuidado para se livrar dos detritos e poeira.
- Tire fotos em campo escuro ou iluminação de campo claro. Passos 6.2 e 6.3 pode ter que ser repetido 2-3 vezes a fim de obter uma boa imagem sem partículas de poeira, que são facilmente vistos em campo escuro sob um microscópio de dissecação.
7. Os resultados representativos
A duas principais formas de visualização dos resultados de hibridização in situ em cortes de tecido hibridizaram com S 35 sondas radioativas: 1) x-ray do filme que foi colocado sobre as lâminas ou 2) emulsão que foi revestido nos slides. Uma terceira abordagem é usar uma tela Phosphorimager colocado sobre as lâminas, mas não ter ficado satisfeita com a resolução desta abordagem. X-ray filmes oferecem um resultado rápido e análises da condição geral da hibridação. O filme de raios X de dados também revela resolução anatómica amplo e pode ser usado para Analys quantitativosé de 10. Exemplos de imagens película de raios X de tardios cabeças embrião aviário hibridaram com as sondas anti-sentido para FoxP1 e CoupTF2 a expressão do gene são nas Figuras 1A e B. Ambos os genes são muito abundantes em subdivisões específicas do cérebro. A boa qualidade do resultado radiografia deve ser acentuada (não embaçado) e tem relação sinal-background-alta. Uma imagem desfocada pode ser devido ao contacto desigual entre uma película de raios-x e da lâmina de vidro com o tecido hibridizada.
Para lâminas de emulsão de cruzamento, a emulsão contém de luz sais de prata sensíveis revestidas sobre o tecido como apposed de ser sobre o plástico do filme de raios-x. Durante o desenvolvimento, o S 35-expostos sais de prata são convertidos em grãos de prata metálica, assim como no filme de raios-X. No entanto, os depósitos de prata são directamente visível sobre as células que representam a expressão do gene que pode ser observado e medido qualitativamente sob um microscópio. Os grãos de prata metálica bloquear a luz direta por meioe aparecem como os pontos pretos em vista de campo claro. O violeta de cresilo contracorante aparece na cor púrpura (Fig. 3A, 3C, e Fig. 4). Em campo escuro, os grãos de prata refletem a luz que vem do lado e aparecem como os pontos brancos (Fig. 1C, 1D, 3B e 3D). Nesta situação, o cresil violeta mancha aparece na cor vermelha. Em brightfield, o sinal de hibridação é mais fácil de visualizar sob a ampliação de alta resolução em celular, enquanto que no campo escuro, além disso, o sinal de hibridação pode ser visto sob inferior ampliação sobre o tecido inteiro. A visão de campo escuro é a abordagem que normalmente usam para mostrar o padrão de expressão gênica global. No entanto, em relação ao resultado obtido a partir de rápida de raios-x filmes, as lâminas de emulsão de cruzamento leva mais tempo (uma a várias semanas) e é mais sensível para a obtenção de fundo.
Há quatro fontes comuns de fundo forte: 1) Antecedentes todo o filme x-ray é geralmente fazer paraproblemas com o desenvolvedor ou fixador, ou filme parcialmente exposto, 2) de fundo sobre as lâminas de vidro geralmente é devido a problemas com a lavagem ou vidro preparação da lâmina, como silination indevido dos slides da fonte comercial ou auto-preparados; 3) Emulsão de exposição e de fundo de desenvolvimento, e 4) de base sobre a secção devido quer à falta de cuidado passos de pós-hibridação de lavagem, muito baixa de uma temperatura de hibridização, a má qualidade da solução de hibridação, a contaminação paraformaldeído em pratos de lavagem causando sondas para permanentemente ligação cruzada para a degradação riboprobe tecido, conduzindo a pequenas moléculas de etiquetagem do tecido não-especificamente, DTT inactiva ou β-mercaptoetanol, resultando em ligação cruzada de S 35-RNA sondas em di-sulfeto de títulos para o tecido, e espera demasiado longo para acetilação. É crítico para ter as lâminas na solução acetilação dentro de segundos de mistura do anidrido acético e trietanolamina. Se passar vários minutos without adicionando a solução para as lâminas, em seguida, os grupos acetilo, não irá ser eficientemente removido e, em seguida, se ligam ao RNA não-especificamente. Outros factores incluem hibridação mais de 20 hr, o que pode gerar demasiado forte de um sinal, e gotículas de óleo em excesso sobre as lâminas, o que sequestram solução de hibridação sobre as lâminas durante lavagens aquosas, resultando em manchas radioactivas sobre o tecido e desliza dando sinais fundo escuro. Se trabalhar com muitos slides (mais de 100 fatias), adicionar uma 3 ª clorofórmio lavar ou alterar as lavagens de clorofórmio, para evitar que partículas de óleo em excesso de permanecerem nos slides. Tratamento de tecidos descuidada, ou seja, não congelação de forma suficientemente rápida (dentro de 5-10 minutos após a dissecação) ou descongelação e congelação re-aumenta também de fundo devido ao mRNA de degradação. Tenha cuidado para não confundir fundo para exposição sobre.
Para o fundo da emulsão sobre as lâminas de cruzamento é possível porque é altamente sensível à luz e requer exposição de longa no escuro. Com problemas de fundo comuns incluem muito alto de temperatura para o revelador e fixador. Quando a temperatura é maior do que 19 ° C, próximo ou mais quente do que a temperatura ambiente, de fundo mais grãos de prata é obtido. A exposição a baixos níveis de luz vazando para uma câmara escura irá causar fundo emulsão. Não lavagem fixador tempo suficiente (pelo menos 30 min em água corrente), que vai deixar o fixador em seguida, reage com violeta de cresilo para gerar um precipitado acastanhado em toda a emulsão. No entanto, se as lâminas são lavadas em mais água do que 90 min após a fixação antes de coloração violeta de cresilo, o que pode causar a emulsão para se tornar solto e as secções para corar mal. Se não há tempo suficiente para corar as lâminas dentro de uma janela min 30-90 após a fixação e lavagem, após a lavagem de 30 min, secar as lâminas durante a noite e prosseguir com violeta de cresilo coloração no dia seguinte. Geralmente, a maioria dos mRNAs têm padrões de expressão de genes específicos, ao passo que o sinal de fundo é mais uniforme.
e_content "> tecido dobrado pode ser enganosa por resultados de expressão gênica em película de raio X, levando a uma região com mais escura do sinal. Para determinar se o tecido é dobrado, examine não-coradas em campo escuro ou seções cresil violeta manchadas em campo claro. cresil contracoloração violeta fornece a melhor maneira de analisar a condição dos tecidos.Para uma melhor interpretação da especificidade da sonda, sondas senso de controle deve ser aplicado em várias seções adjacentes. Sondas mais sentido não mostram um sinal, mas alguns fazem, e quando o fazem, descobrimos que muitas vezes é diferente do sinal antisense. Acreditamos que esta poderia estar relacionada com anti-sentido de síntese ou outro gene na cadeia anti-sentido do genoma. Nós apresentamos um exemplo Pax6 sentido e sondas antisense (Fig. 2A e B). A cadeia anti-sentido revela etiquetagem ao longo da zona ventricular do prosencéfalo cerebelo, e do olho como esperado (Fig. 2A), mas o sentido revela laBeling na camada de pigmento da retina (Fig. 2B).
Para os tamanhos de sonda, usamos sondas de cDNA em qualquer lugar na gama de 300-5000 bps. Sondas menos de 300 bps de trabalho, mas os sinais são geralmente mais fraca. Nós não tentamos sondas maiores que 5000 bps. O melhor é a utilização de sondas sobre o tecido das mesmas espécies, se possível. Se não, vamos cruzar-hibridizar sondas em seções de outras espécies e diminuir a hibridação e lavar as temperaturas em 3-5 ° C incrementos com base na identidade de seqüência, se conhecido. Se não for conhecido, então realizamos hibridização tentativa e erro e lavar as temperaturas. Se a temperatura for demasiado baixa, a sonda de cDNA pode atravessar-hibridizar com as sequências de mRNAs outros semelhantes em todas as espécies ou na mesma espécie 11. Na prática, descobrimos que os cDNAs que são ~ 95% idêntica ou superior para o alvo do mRNA no tecido, a hibridação rigorosas e temperatura de lavagem (65 ° C) condições funciona bem. Para sequências que estão na tocoues de ~ 85 a 94% idêntica, a hibridação ea temperatura de lavagem pode necessitar de ser reduzida na gama de ~ 50 a 60 ° C.
A razão para usar uma prateleira de metal na maior parte dos passos é a utilização de lavagens de clorofórmio e xileno. Ambos os produtos orgânicos derreter muitos tipos de plásticos. Vidro e alguns tipos de plásticos são resistentes a esses compostos orgânicos. Mas o vidro é mais fácil de quebrar, e alguns plásticos que inicialmente são resistentes irá derreter mais longo de períodos de exposição aos compostos orgânicos.
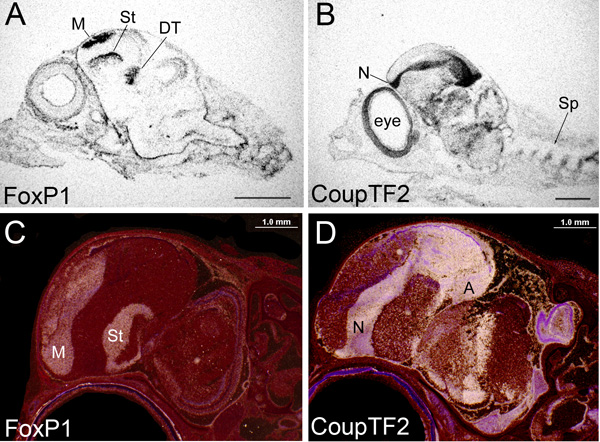
Figura 1. Autorradiografia de imagens hibridização in-situ de x-ray filmes e slides de emulsão mergulhados. (AB) filme de raios-X de imagens de sagital secções da cabeça inteiras da Finch zebra songbird em embrionário dia 10, hibridado com riboprobes anti-sentido para (A) FoxP1 ou (B) CoupTF2, tomado com iluminação de campo claro sob um microscópio de dissecação. Preto, grãos expostos em filme mostrando mRNA expressoion. Barra de escala = 500 mm. (CD) Emulsão mergulhado imagens de slides de sagital secções da cabeça inteiras da Finch zebra no dia pós escotilha 6, hibridado com riboprobes anti-sentido para (C) FoxP1 e (D) CoupTF2, tomado com iluminação de campo escuro sob um microscópio de dissecção. White, exposta grãos de prata em emulsão acima tecido mostrando a expressão do mRNA. Violeta, vermelho cresil mancha. Barra de escala = 200 mM. Para todos os imagem, o bico é rostral para a esquerda. As imagens película de raios X foram expostas durante um dia, as lâminas de cruzamento durante 3 dias. A sonda é FoxP1 178 bps para a parte pb 1544-1711 do ARNm; CoupTF2 é 545 bps para a parte pb 1-545 do mRNA. Como pode ser visto, dentro do prosencéfalo mRNA FoxP1 é enriquecido no mesopallium (M), striatum (St) e tálamo dorsal (DT), enquanto que CoupTF2 é enriquecido no nidopallium (N), arcopallium (A), e tálamo mais ventral . Não há consistência na expressão entre os tipos de exposição (e idades). Com as lâminas de cruzamento, no entanto, uma maior resolução de rotulagem é visto, umalimites de tecido d e subdivisões são diretamente identificados. Estas e todas as outras imagens mostradas no papel são a partir de secções, utilizando o padrão de 65 ° C de hibridação de elevado rigor.
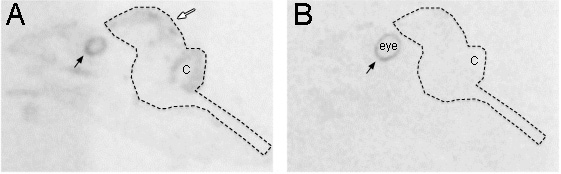
Figura 2. Comparação de anti-sentido e rotulagem sentido de que mostra diferentes padrões. (A) A cadeia anti-sentido de Pax6 foi expressa no cérebro, especialmente a zona ventricular (seta branca). É mostrado autoradiografia em filmes de raios-x de fatias sagitais cabeça inteira tiradas zebra finch no dia embrionário 12. (B) secção adjacente hibridizada com a cadeia de sentido de Pax6 revela nenhuma expressão de fundo em toda a cabeça do embrião, mas a expressão aparente na camada de pigmento da retina (setas pretas) como a cadeia anti-sentido. A linha tracejada indica o contorno de todo o cérebro. C: Cerebelo.
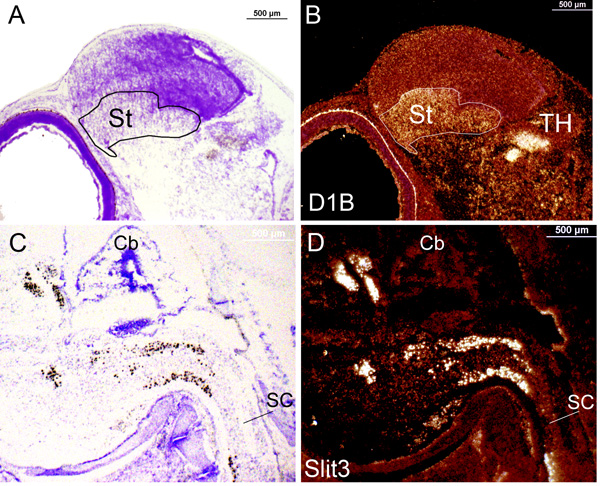
rong> Figura 3. Em sinais in situ de expressão gênica em lâminas de emulsão mergulhados tomadas de zebra finch cérebro durante o final estágios embrionários sob a visão de campo claro e campo escuro. (A) da imagem Brightfield de expressão em D1b embrionário dias 10 a partir de uma exposição normal para a emulsão. A etiqueta (preto) mal pode ser visto sob esta ampliação. Sonda é de 625 bps para a parte pb 1-625 do mRNA. (B) secção idêntica e ampliação, como em (A), mas comutado para o modo de campo escuro apresentando etiqueta (branco) no corpo estriado (St) e tálamo (TH). (C) da imagem de campo claro Slit3 expressão a 12 dia embrionário a partir de uma exposição sobre a emulsão. Label (preto) pode ser facilmente visto. Sonda é 779 bps para a parte 1243-2021 do ARNm. (B) e secção idêntica de ampliação, como em (A) ligado ao campo escuro rótulo vista mostrando (branco) na medula espinal (SC) que corresponde à imagem de campo claro. Rostral é orientada para a esquerda. Cb: Cerebelo.
/ 3764/3764fig4.jpg "alt =" Figura 4 "/>
Figura 4. Prata de grãos resolução ao nível celular. É mostrado FoxP1 rótulo mRNA no tentilhão prosencéfalo com grãos de prata (pontos pretos) acima de células (violeta cresil) em diferentes regiões cerebrais e idades comparados com imagens de baixa potência da Figura 1A e 1C. (A) expressão abundância de alta sobre as células individuais (setas pretas) na tentilhão adulto mesopallium. (B) Expressão baixa abundância sobre as células individuais (pontas de seta preta) no nidopallium adjacente da mesma secção. (C) de alta expressão de mRNA FoxP1 sobre as células (setas pretas) na embrionário mesopallium dia 12. (D) expressão baixa abundância sobre as células (pontas de seta pretas) no nidopallium adjacente da mesma secção. Exemplo as células estão circuladas com uma linha amarela. As células embrionárias (C e D) são menores e mais apertada em comparação com as células adultas (A e B). A uma vez que existe algum espaço entre as células e de emulsão, a área de Si expostogrãos lver a partir do 35 S sonda são ligeiramente grande do que a área dos corpos celulares. Barra de escala = 10 mM.
Discussão
Radioactivos hibridação in situ da expressão do mRNA é amplamente utilizado para fins múltiplos, incluindo para estudar organização do tecido regional, tipos de células, e actividade funcional do cérebro 2-5,10,12-14. O uso mais tarde está em genes cuja expressão de mRNA no cérebro é dependente da atividade neural maior, muitas vezes chamado de atividade dependentes de genes ou genes imediatos iniciais. Com estas utilizações, o nosso método foi aplicado em várias espécies múltiplas, incluindo, em aves, mamíferos (por exemplo, humanos), peixes e anfíbios; em múltiplos tecidos, incluindo o cérebro, pele, músculo e; e idades múltiplas, incluindo hatchlings / recém-nascidos, juvenis , os adultos, e aqui em seções inteiras de embriões 2,3,5,15-17. As características especiais do nosso protocolo incluem: (1) Ela produz um equilíbrio entre a especificidade anatômica e especificidade quantitativa. Para quantificar a expressão gênica no filme de raios-x, tiramos fotos digitais das imagens (ex:. Figura 1A e 1B), use o Photoshop função de histograma (Adobe) para medir a densidade de pixels nas regiões de interesse e subtrair os níveis de fundo sobre o filme exterior do tecido, mas ainda na lâmina de vidro 2,4. Para quantificar a expressão a nível celular, tomamos imagens de grãos de prata sobre as células sob ampliação elevada (40-100X; Fig. 4). Em seguida, usamos o limiar e medindo funções de J Imagem por Wayne Rasband no NIH para contar o número de grãos de prata na imagem, subtrair a contagem de fundo em uma área similar sem células sobre a lâmina de vidro, dividir pelo número de células, para obter um valor de expressão por célula. 4,18 (2) Pode ser rendimento relativamente elevado, permitindo o processamento de 100-200 lâminas simultaneamente, devido à vedação lamela apertado criado pelo banho de óleo mineral. Métodos padrão de hibridização in-situ demorar mais tempo para selar as lâminas com parafilme, unha polonês, e outros meios, onde lâminas ocupam muito espaço, (3) É altamentesensível para as transcrições baixa abundância devido ao sulfato de dextrano e solução de Denhardt no tampão de hibridação 2,13, (4) A abordagem de imagem em campo escuro produz imagens de alto contraste devido a fotografar sob iluminação de campo escuro sobre um microscópio de dissecação 4,5. Além disso, permite a detecção sensível de pequenas alterações na expressão do gene, tais como na actividade dependente da expressão do gene para identificar regiões específicas do cérebro activadas durante a percepção e produção de comportamentos específicos 19. As limitações em relação ao não-radioactivos protocolos são que estas últimas são mais clara na resolução celular e da localização do mRNA no interior da célula, e trabalhando com a emulsão é muito sensível à manipulação e da luz. É possível combinar o nosso método com outros métodos, tais como não radioactivo em hibridização in situ para rótulo de expressão de mRNA de mais de um gene no mesmo tecido 10,17,20. Ele pode ser combinado com imunocitoquímicapara rotular RNA e expressão da proteína sobre a mesma amostra, a fim de co-localizar o ARNm com certos tipos de células 10. As modificações protocolo necessário para tais experiências dupla marcação são descritas nas referências citadas.
Em resumo, a nossa abordagem facilita a compreensão do tempo e localização celular da expressão do gene, a fim de entender organização região, a atividade do tecido funcional e da função do gene.
Divulgações
Não temos nada a divulgar.
Agradecimentos
Os autores gostariam de agradecer a todos os membros Jarvis laboratório que melhoraram o protocolo ao longo dos anos.
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente | Companhia | Número de catálogo | Comentários |
| Anidrido acético | VWR | MK242002 | |
| Clorofórmio | VWR | BDH1109 | |
| Acetato de cresil violeta | Sigma | C5042 | |
| Criostato | Thermo Scientific | Microm HM550 | |
| Formamida deionizada | Sigma | F9037 | |
| TDT | Promega | P1171 | 100mM |
| EDTA | Sigma | ED | |
| Incorporação de molde | VWR | 15160-215 | |
| Fisher marca Superfrost mais slides | Fisher Scientific | 22-034-979 | |
| Formaldeído | VWR | BDH0506-4LP | |
| Formamida | Sigma | F7508 | |
| GENECLEAN kit | Q-Bio gene | 1001-200 | |
| Kodak BioMax MR filme | Sigma | Z350370 | |
| Kodak NTB Emulsão | A Carestream Health | 8895666 | |
| Kodak Professional desenvolvedor D19 | Câmera fotográfica | 1462593 | |
| Kodak profissional Fixer | Câmera fotográfica | 1971746 | |
| β-mercaptoetanol | Calbiochem | 444203 | |
| Óleo mineral | VWR | IC15169491 | |
| NaOH | VWR | SX0600-1 | |
| Paraformaldeído | Sigma | 76240 | |
| Poli A | Invitrogen | POLYA.GF | |
| rATP | Promega | P1132 | 10mM |
| RCTP | Promega | P1142 | 10mM |
| rGTP | Promega | P1152 | 10mM |
| RNasin | Promega | N2111 | 40Units/μl |
| S 35 UTP | PerkinElmer | NEG039C001MC | |
| Safety-Solve solução | Segurança Resolva Research Products International | 111177 | |
| Acetato de Sódio | Sigma | S7899 | 3M |
| Fosfato de sódio dibásico | Sigma | S3264 | |
| Fosfato de sódio monobásico | Sigma | S3139 | |
| SP6RNA polimerase | Promega | P1085 | |
| Coloração prateleira de metal | Electron Microscopy Sciences | 70312-54 | |
| T7 RNA polimerase | Promega | P2075 | |
| Tissue-Tek outubro | Sakura | 4583 | |
| Transcrição tampão 5x | Promega | P1181 | |
| Trietanolamina | VWR | IC15216391 | |
| Tris-HCl (1 M, pH 8,0) | VWR | 101449-446 | |
| tRNA | Roche | 10109509001 |
Soluções:
- Reagentes para fazer de riboprobes: 1,5 modelo DNA ul (0,2-0,3 mg / mL), 2 ul de tampão de transcrição 5x otimizado, 1 ml de DTT 100mM (vem com Polimerase de Promega, mixbem à temperatura ambiente), 0,3 ul de RNasin (40 unidades / uL), 1,5 ul de solução de AGC mistura ribonucleótido, 3,5 ul de S 35 UTP, e uma polimerase de ARN uL. Traga até 10 ul com nuclease livre de água.
- AGC solução mix ribonucleotídeo: misture quantidades iguais de 10 mM ATP, GTP e CTP juntos.
- Solução de acetato de sódio para EtOH precipitação para remover 35 S livre UTP: 40 ul de ARNase e ADNase água livre, 5 ul de acetato de sódio 3M, e 125 uL de EtOH a 100%.
- Tampão de hibridação (10 mL de stock): 5 ml de 100% de formamida desionizada, 600 ul de NaCl 5M, 1M Tris-HCl (pH = 8,0), 240 ul de EDTA 0,5 M (pH = 8,0), 100 uL de solução de Denhart 100x, o 100 ul de 1M de DTT, 250 uL de 20mg/ml tRNA, 125 uL de 20 mg / ml de poli A, e 1 g de sulfato de sódio dextrano. Adicionar DEPC tratada com água para levar o volume total para 10 ml. Agita-se vigorosamente e depois incubar a 55 ° C até que todo o sulfato de sódio dextrano é dissolvido. Armazenar o tampão de hibridação em-20 ° C, que é bom para ~ 6 meses.
- 4% tamponado solução de paraformaldeído: Adicionar 40 g de paraformaldeído em 760 ml de água destilada em um balão de designado para paraformaldeído, o calor a 50 ° C sobre uma placa quente com agitação. Adicionar 320 uL de NaOH 10N para ajudar a dissolver o paraformaldeído. Depois de se dissolver (~ 10 min), adicionar 100 ml de PBS 10x, e trazer o volume com água destilada até 1 litro. Agita-se e aquecer a solução até paraformaldeído é dissolvido. O pH deve ser de 7,4.
- Tampão de acetilação: 13,6 ml de trietanolamina, mais 2,52 ml de anidrido acético em 1 litro de água destilada.
- 20x SSPE solução: 3M NaCl, 200 mM de NaH 2 PO 4-H2O, e 200 mM de EDTA em água destilada. Ajuste a solução para pH 7,4 com NaOH 10N.
- 10x tampão PBS: 80g de NaCl, 2,0 g de KCl, 14,4 g de Na 2 HPO 4, e 2,4 g de KH PO 2 4 em água destilada. Ajuste a solução a pH 7,0 e trazer o volume total a 1 Liter.
- Solução de lavagem segundo: 0,1% β-mercaptoetanol e formamida a 50% em 2x SSPE
- Solução de acetato de cresilo violeta: acetato de violeta de cresilo 3% em água da torneira (água destilada impede coloração bom), dissolvido durante a noite, com agitação num balão à temperatura ambiente. Filtrar por sucção a vácuo através de um Whatman 1 milímetro filtro de papel e um funil de Buchner.
Referências
- Clayton, D. F., Huecas, M. E., Sinclair-Thompson, E. Y., Nastiuk, K. L., Nottebohm, F. Probes for rare mRNAs reveal distributed cell subsets in canary brain. Neuron. 1, 249-261 (1988).
- Wada, K., Sakaguchi, H., Jarvis, E. D., Hagiwara, M. Differential expression of glutamate receptors in avian neural pathways for learned vocalization. J. Comp. Neurol. 476, 44-64 (2004).
- Haesler, S. FoxP2 expression in avian vocal learners and non-learners. J. Neurosci. 24, 3164-3175 (2004).
- Jarvis, E. D., Nottebohm, F. Motor-driven gene expression. Proc. Natl. Acad. Sci. U.S.A. 94, 4097-4102 (1997).
- Holzenberger, M. Selective expression of insulin-like growth factor II in the songbird brain. J. Neurosci. 17, 6974-6987 (1997).
- Mahmood, R., Mason, I. In-situ hybridization of radioactive riboprobes to RNA in tissue sections. Methods Mol. Biol. 461, 675-686 (2008).
- Wilkinson, D. G., Nieto, M. A. Detection of messenger RNA by in situ hybridization to tissue sections and whole mounts. Methods Enzymol. 225, 361-373 (1993).
- Acloque, H., Wilkinson, D. G., Nieto, M. A. In situ hybridization analysis of chick embryos in whole-mount and tissue sections. Methods Cell Biol. 87, 169-185 (2008).
- Moorman, A. F., Houweling, A. C., de Boer, P. A., Christoffels, V. M. Sensitive nonradioactive detection of mRNA in tissue sections: novel application of the whole-mount in situ hybridization protocol. J. Histochem. Cytochem. 49, 1-8 (2001).
- Horita, H., Wada, K., Rivas, M. V., Hara, E., Jarvis, E. D. The dusp1 immediate early gene is regulated by natural stimuli predominantly in sensory input neurons. J. Comp. Neurol. 518, 2873-2901 (2010).
- Braissant, O., Wahli, W. A simplified in situ hybridization protocol using non-radioactively labelled probes to detect abundant and rare mRNAs on tissue sections. Biochemica. 1, 10-16 (1998).
- Kubikova, L., Wada, K., Jarvis, E. D. Dopamine receptors in a songbird brain. J. Comp. Neurol. 518, 741-769 (2010).
- Wada, K. A molecular neuroethological approach for identifying and characterizing a cascade of behaviorally regulated genes. Proc. Natl. Acad. Sci. U.S.A. 103, 15212-15217 (2006).
- Jarvis, E. D. Avian brains and a new understanding of vertebrate brain evolution. Nat. Rev. Neurosci. 6, 151-159 (2005).
- Hoke, K. L., Ryan, M. J., Wilczynski, W. Social cues shift functional connectivity in the hypothalamus. Proc. Natl. Acad. Sci. U.S.A. 102, 10712-10717 (2005).
- Burmeister, S. S., Jarvis, E. D., Fernald, R. D. Rapid behavioral and genomic responses to social opportunity. PLoS. Biol. 3, e363 (2005).
- Jarvis, E. D., Schwabl, H., Ribeiro, S., Mello, C. V. Brain gene regulation by territorial singing behavior in freely ranging songbirds. Neuroreport. 8, 2073-2077 (1997).
- Jarvis, E. D., Scharff, C., Grossman, M. R., Ramos, J. A., Nottebohm, F. For whom the bird sings: context-dependent gene expression. Neuron. 21, 775-788 (1998).
- Feenders, G. Molecular mapping of movement-associated areas in the avian brain: a motor theory for vocal learning origin. PLoS One. 3, e1768 (2008).
- Chen, C. C., Fernald, R. D. Distributions of two gonadotropin-releasing hormone receptor types in a cichlid fish suggest functional specialization. J. Comp. Neurol. 495, 314-323 (2006).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados