Method Article
Reverse-Genetics vermittelte Wiedergewinnung eines infektiösen Murine Norovirus
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Noroviren sind eine bedeutende Ursache von Gastroenteritis noch molekulare Techniken zu ihrer Charakterisierung sind noch relativ neu. Hier berichten wir über zwei verschiedene Ansätze der reversen Genetik für die effiziente Gewinnung von murinen Norovirus (MNV), das einzige Mitglied dieser Gattung, die in Zellkultur vermehrt werden kann.
Zusammenfassung
Menschliche Noroviren sind verantwortlich für die meisten Fälle von menschlichen Gastroenteritis (GE) weltweit und sind immer wiederkehrendes Problem in Umgebungen, in denen nahe stehende Person zu Person Kontakt nicht 1, 2 vermieden werden können. In den letzten Jahren ein Anstieg der Häufigkeit der Ausbrüche in Krankenhäusern wurde berichtet, was zu erheblichen Störungen ihrer operativen Leistungsfähigkeit sowie große wirtschaftliche Verluste. Die Identifizierung von neuen antiviralen Ansätzen wurde aufgrund der Unfähigkeit des menschlichen Noroviren, um eine produktive Infektion in Zellkultur 3 vervollständigen begrenzt. Die kürzliche Isolierung eines murinen Norovirus (MNV), eng mit den menschlichen Norovirus 4, aber die Suchbegriffe in Zellen 5 werden ausgebreitet hat neue Wege zur Untersuchung dieser Pathogene 6, 7 geöffnet.
MNV Replikation resultiert in der Synthese der neuen positiven Sinne genomischen und subgenomischen RNA-Moleküle, entspricht die letztere zur letzten third des viralen Genoms (Abbildung 1). MNV enthält vier verschiedene offene Leseraster (ORFs), von denen ORF1 nimmt den größten Teil des Genoms kodiert und sieben nicht-strukturelle Proteine (NS1-7) aus einem Polyprotein-Vorläufer-Freigabe. ORF2 und ORF3 innerhalb des subgenomischen RNA-Region enthalten ist, und codieren die Capsid-Proteine (VP1 und VP2, jeweils) (1). Vor kurzem haben wir diese zusätzliche ORF4 überlappenden ORF2 identifiziert, aber in einem anderen Leseraster ist funktional und kodiert für eine mitochondriale lokalisierte Virulenzfaktor (VF1) 8.
Replikation für positive RNA Viren, einschließlich Noroviren, erfolgt im Zytoplasma, was zur Synthese neuer begrenzten RNA-Genome. Um virale Übersetzung zu fördern, zu nutzen Viren unterschiedliche Strategien bei der Einstellung des zellulären Proteinsynthese-Maschinerie 11.09 abzielen. Interessant ist, dass Norovirus-Übersetzung durch die multifunktionale virale Protein-Primer VPg angetriebenkovalent an das 5'-Ende der beiden genomischen und subgenomischen RNAs 12-14 verbunden. Dieser ausgeklügelte Mechanismus der Übersetzung wird wahrscheinlich ein wichtiger Faktor in der geringen Effizienz der viralen Wiederherstellung durch konventionelle Methoden der reversen Genetik sein.
Hier berichten wir zwei verschiedene Strategien, die auf die Generierung von murinen Norovirus-1 (im Folgenden als MNV beiliegenden) Transkripte am 5'-Ende verschlossen. Eines der Verfahren umfasst sowohl in vitro Synthese und Begrenzung der viralen RNA, während die zweite Ansatz beinhaltet die Transkription der cDNA MNV in Zellen, die T7-RNA-Polymerase. Die Verfügbarkeit dieser reversen Genetik zum Studium von MNV und einem Tiermodell eine beispiellose Fähigkeit, die Rolle von viralen Sequenzen in Replikation und Pathogenese von 15 bis 17 zu zerlegen ist.
Protokoll
1. RNA-Transkription und Capping für den Recovery of Infectious MNV
Dieses Protokoll wurde entwickelt, um die effiziente Gewinnung von infektiösen MNV aus cDNA mittels in vitro-Transkription und die anschließende In-vitro-Capping (Abschnitt 1.1) zu ermöglichen. Die daraus resultierenden capped Transkripte werden dann in Zellen transfiziert, um infektiöse MNV (Abschnitte 1.2 und 1.3) zu erholen. Dieser Ansatz bietet die empfindlichste Methode zur Gewinnung von MNV mit typischen Ausbeuten von über 10 5 infektiösen Einheiten pro 35 mm (Durchmesser)-Schale von Zellen für MNV. Das Protokoll wird im Folgenden aufgeführt:
1.1 Synthese von infektiösen capped MNV Transkripte:
- Verdau des Plasmids, welches das Wildtyp MNV cDNA (pT7: MNV 3'Rz) mit NheI, um lineare DNA zu erhalten Nhe I erkennt eine einzigartige Restriktionsstelle nach dem 3 'Ende polyA-Schwanz von MNV Genom (Abbildung 2).. Linearisierte Plasmide gereinigt werden typischerweisemit der Verwendung von Silica-Säulen (z. B. GFX PCR-DNA-Gel Band Purification Kit von GE Healthcare) aufgetragen und in H 2 O.
- In-vitro-Transkription der linearisierte Vektor unter Verwendung von T7-RNA-Polymerase, wie zuvor 17 beschrieben. Viele kommerzielle Kits zur Verfügung zu diesem Zweck und ein reproduzierbares Verfahren große Mengen an RNA-Synthese, wie MEGAscript (Life Technologies) und RiboMAX (Promega). Transkriptionsreaktionen werden typischerweise DNAse vor der weiteren Analyse verdaut, aber in vielen Fällen wird dies nicht als Lithiumchlorid Reinigungen erforderlich, da unten nicht ausfallen DNA effizient beschrieben.
- Analysieren eines kleines Aliquot der RNA-Transkriptions-Reaktion, typischerweise 0,5 ul oder weniger, durch Agarose-Gelelektrophorese, um die Transkription zu gewährleisten, hat effektiv arbeiten RNA voller Länge. Während viele Benutzer wollen laufen denaturierende Gele für volle Größe der RNA, wir verwenden in der Regel nicht-denaturierenden Agarosegel electrophorESIS als schnelle Methode, um RNA-Integrität zu analysieren. Die MNV Genom aus dem infektiösen cDNA-Klon pT7 hergestellt: MNV 3'Rz wird bei etwa 3 kbp relativ zu einer dsDNA Leiter auf einem nicht-denaturierenden Agarosegel (3) ausgeführt.
- Bedenken Sie, dass andere Methoden zur Verfügung stehen als Alternative zu einer schnellen Analyse von RNA Integrität wie mit einem Agilent Bioanalyzer (Abbildung 3) zu erhalten.
- Beachten Sie, dass arme Gel Resolution auftreten können, wenn zu viel RNA wird geladen. Seien Sie besonders vorsichtig, um sicherzustellen, das Agarosegel wird unter Verwendung RNase-freie Reagenzien zur RNA-Abbau während der Elektrophorese, die Band-Auflösung beeinflussen können, zu vermeiden. Heizung RNA auf 65 ° C, gefolgt von Kühlen auf Eis kann auch in einigen Fällen zu helfen.
- Entschlacken RNA-Probe, um die eingebaute Nukleotide zu entfernen. Viele Methoden sind für diese auch Silica-Säule basierend Ansätze vorhanden aber in diesem Protokoll verwenden wir meist Lithiumchlorid als kostengünstige Alternative. FürZu diesem Zweck fügen H 2 O, um ein Endvolumen von 100 ul erreichen und fügen Sie dann 40 ul Lithiumchlorid Niederschläge Lösung (7,5 M LiCl, 50 mM EDTA, pH 8,0, Ambion) und lagern Sie die Probe bei -20 ° C für mindestens 30 min.
- Pellet die RNA durch Zentrifugation bei 12.000 × g bei 4 ° C für 15 min.
- Entfernen Sie den Überstand, dabei nicht um die durchscheinende RNA-Pellet stören und waschen Sie ihn in 150 ul 70% igem Ethanol. Das Röhrchen bei 12.000 × g bei 4 ° C für 15 min.
- Entfernen Sie das Ethanol und an der Luft trocknen die RNA, die Vermeidung der Pellet vollständig austrocknen, da dies zu machen Resuspension schwierig.
- Dann resuspendieren MNV Transkripte in 50-100 ul der RNA-Storage-Lösung (Ambion). Es sollte darauf geachtet werden, alle RNA ordnungsgemäß aufgelöst werden. Sollten die RNA schwierig, vollständig zu lösen erscheinen, Erhitzen der Probe auf 60 ° C kann dazu beitragen, sie zu resuspendieren. Jede unlösliches Material sollte dann entfernt werden, by Zentrifugation vor der Quantifizierung RNA. Die gereinigten Transkripte nicht abgedeckt sind und erfordern eine anschließende In-vitro-Capping-Schritt ansteckend zu sein (ScriptCap m7G Capping-System, Epicentre Biotechnologies).
- Quantifizierung der RNA durch Spektrophotometrie. Je nach Art und Umfang des Transkriptions-Reaktion, reichen typische Ausbeuten von 50 bis 150 ug RNA pro 100 ul Transkriptions-Reaktion. Die Integrität der RNA sollte vor dem Capping-Reaktion, indem Sie aus 100 bis 300 ng der Probe in einer 1% igen Agarosegel (3) analysiert werden.
- Um die Effizienz der RNA-Capping zu verbessern, platzieren Hitze 60 bis 70 pg MNV RNA-Transkripte bei 65 ° C für 10 min und dann das Rohr sofort auf Eis. Dieser Schritt kann jede reduzieren hemmende Wirkung von RNA-Struktur auf der Deckelung. Impuls das Rohr in einem gekühlten Mikrofuge zu Tröpfchen während des Erwärmungsschritts ausgebildet sammeln.
- Bereiten Sie eine Deckelung Reaktionsgemisch, wie vom Hersteller (ScriptCap m7G vorgeschlagenCapping-System, Epicentre Biotechnologies). Kurz gesagt, fügen 60-70 pg MNV-RNA auf ein Endvolumen von 100 ul. Die Capping Reaktion Mix kann 10 ul 10 x Capping-Puffer (500 mM Tris-HCl pH 8,0, 60 mM KCl, 12,5 mM MgCl2), 10 ul 10 mM GTP, 0,5 ul 20 mM S-Adenosylmethionin, 2,5 ul Scriptguard (100 Einheiten) und 4 ul Scriptcap Enzym (40 Einheiten).
- Während der Reaktion einzurichten, halten RNA-Transkripte auf Eis, um den Abbau zu vermeiden. Gut mischen des Reaktionsgemisches und inkubieren bei 37 ° C für 1 h. Beachten Sie die Reaktion eine Größe entsprechend der Menge der begrenzten Transkripts erforderlich skaliert werden.
- Reinigen Sie die RNA durch LiCl Niederschlag wie oben erklärt (siehe Punkt 1.1.6). Das Pellet in 50-100 ul der RNA-Storage-Lösung (Ambion) und zu quantifizieren, die Menge an RNA. Typischerweise werden RNA-Proben nachträglich auf 1 ug / ul normalisiert. , Prüfen Sie nochmal die RNA ordnungsgemäß aufgelöst. Wenn er nicht richtig gelöst worden, Wärme ter Probe auf 60 ° C, um dessen Auflösung zu ermöglichen. Entfernen Sie durch Zentrifugation jegliches unlösliches Material vor der Quantifizierung RNA.
- Überprüfen Sie die Integrität der RNA wieder, bevor mit der Transfektion Schritt. Zu diesem Zweck ausgeführt einer Menge von 100-300 ng der Probe in einer 1% igen Agarosegel (Abbildung 3).
1,2 Wiederherstellung von Neon-vermittelte Transfektion von RNA in RAW 264.7-Zellen:
Zur Bergung von MNV infektiösen Virionen in einer permissiven Zelllinie ist es möglich, die verschlossene MNV-Transkript in RAW 264.7-Zellen mit Neon Transfektion System (Invitrogen) elektroporieren. Raw264.7 Zellen sind anfällig für MNV-Infektion, die Unterstützung mehrerer Runden der Virusreplikation und die anschließende Re-Infektion. Als ein Ergebnis wird typische Ausbeuten von mehr als 10 5 infektiösen Einheiten pro ml bei 24 Stunden nach der Transfektion jedoch Peak bei> 10 7 infektiösen Einheiten nähern nach 48 Stunden.
- Einen Tag vor Transfection, seed RAW 264.7-Zellen bei schätzungsweise 50% Konfluenz. Typischerweise werden zwei T75-Flaschen von Zellen werden für 3 Transfektionen benötigt.
- Der Tag der Transfektion, kratzen die Zell-Monolayer in Dulbecco modifiziertem Eagle Medium (DMEM) mit 10% fötalem Kälberserum (FCS), sodass Sie erzeugen eine einzige Zellsuspension durch wiederholtes Pipettieren.
- Die Konzentration an lebenden Zellen in einem Hämozytometer mit Trypanblau-Ausschluss, die nicht lebensfähigen Zellen zu markieren.
- Pellet die Zellen bei 1200 × g für 5 min und Resuspension sie in DMEM, enthaltend 10% FCS mit einer Endkonzentration von 8 x 10 6 Zellen / ml.
- Unmittelbar vor der Transfektion Aliquot von 1 ml der Zellen pro Transfektion und Pellet sie bei 1200 × g für 2 min. Entfernen Sie die Medien und waschen Sie die Zellen in 500 ul PBS (ohne Mg 2 + / Ca 2 +). Anzentrifugieren die Zellen erneut bei 1.200 X g für 2 min. Beachten Sie, ist es ratsam, Zellen in DMEM zu halten, so lange wie möglichdie Speicherung in PBS für längere Zeit kann dies die Lebensfähigkeit der Zellen und Transfektionsrate.
- Entfernen PBS von Röhrchen und geben 130 ul des Resuspensionslösung (Neon Transfektionssystem Kit, Invitrogen) bis zu einer Endkonzentration von 6 x 10 7 Zellen / ml. Es sollte darauf geachtet, um die Zellen Vermeidung der Bildung von Blasen, die zu Funkenbildung während der Transfektion und Kompromiss das Überleben der Zelle wird resuspendieren werden.
- Fügen Sie den entsprechenden Betrag der begrenzten MNV-Transkript zu den Zellen (Abbildung 2), wird in der Regel 1,3 ug capped RNA zu 130 ul der resuspendierten Zellen und vorsichtig gemischt aufgenommen. Dann sammeln 100 ul der Mischung in der 100 ul Neon Transfektion Spitze. Besondere Vorsicht ist geboten, um sicherzustellen, dass keine Luftblasen in der Elektroporationsküvette (Neon Transfektionssystem Kit 100 ul Spitze) gebildet werden, wie dieses Scheitern des Experiments führen wird.
- Elektroporieren die Zellen unter Verwendung eines einzelnen Pulses bei 1.700 V für 25 ms, enSuring die Abwesenheit von Funken in Pulsen, welche das Vorhandensein von Blasen in der Probe anzuzeigen. In der Fall Funkenbildung auftreten sollten, entsorgen Sie die Probe und wiederholen Sie die Transfektion. Lassen Sie die Zellen in ein Eppendorf-Röhrchen mit 1 ml des Antibiotikums DMEM mit 10% FCS. Beachten Sie, jede Spitze kann bis zu dreimal wiederverwendet werden mit der gleichen RNA-Probe, wenn eine größere Anzahl von transfizierten Zellen benötigt werden.
- Danach verteilen die Zellen aus dem Rohr in unabhängigen Vertiefungen, die eine geeignete Menge von vorgewärmtem Antibiotika DMEM mit 10% FCS. Als allgemeine Orientierung, sind 150 pl der Zellsuspension in Schritt 1.2.8 erzeugt ausreichend für eine einzige Vertiefung einer 24-Teller Platte mit 0,5 ml vorgewärmtem DMEM, während 300 pl für eine Vertiefung einer 12-Teller Platte geeignet sind mit 1 ml vorgewärmtem DMEM.
- Inkubieren der Zellen bei 37 ° C und 10% CO 2 für 24 bis 72 Stunden. Dann, Freigabe infektiösen Virionen von Zellen durch eine (oder mehrere)Frost-Tau-Zyklen und bestimmen Virustiter in der Probe entweder mit Plaque-Assay oder TCID50. Beachten Sie, dass Lysate durch Zentrifugation sollte für 1-2 Minuten bei maximaler Geschwindigkeit oder durch ihre Filtrieren durch ein 0,22 um Porenfilter vor der Titration geklärt werden. Typischerweise erreicht MNV Titer von etwa 1 x 10 6 TCID50/ml bei 24 Stunden nach der Transfektion und bis zu 1 x 10 9 bei 72 Stunden nach der Transfektion.
- Das Vorhandensein und die Stabilität von Mutationen in pT7 eingeführt: MNV 3'Rz werden typischerweise durch Sequenzierung der geretteten Viren, die nach 2 bis 5 zusätzliche Passagen in RAW 264.7-Zellen bestimmt.
1,3 Einziehung durch Lipofektion in BHK-21 Zellen:
Eine direktere und oft kostengünstiger Verfahren zur Rückgewinnung von infektiösen MNV von capped Transkripte ist via Lipofektion (Lipofectamin 2000, Invitrogen). Da RAW 264.7-Zellen schwer zu transfizieren mit Lipid-basierten Ansätzen sind wir in der Regel nutzen other leicht zu transfizieren Zelllinien, wie BHK-21, die eine immortalisierte Linie von Babyhamsternierenzellen Fibroblasten stammt. Als Standardverfahren in unserem Labor verwenden wir BSR-T7-Zellen, ein Derivat der BHK-21-Zelllinie, wie während diese Zellen sind leicht zu transfizieren und unterstützen MNV Replikation sie einen geeigneten Rezeptor fehlt, dass mehrere Runden Reinfektion . Als ein Ergebnis ist die Virusausbeute ins System erzeugt ein Hinweis auf einen einzigen Zyklus der Virusreplikation. Dieser Ansatz ist von besonderem Nutzen bei der Untersuchung der Auswirkung der Mutation auf Virus Erholung, da es mehrere Transfektionen bis zu deutlich niedrigeren Kosten im Vergleich zu Neon-vermittelte Transfektion und benötigt auch keine spezielle Ausrüstung durchgeführt werden können. Es sei darauf hingewiesen, dass andere leicht verfügbare Zelllinien, wie humane embryonale Nierenzellen 293T-Zellen unterstützen die effiziente Gewinnung von MNV jedoch Transfektionsbedingungen zunächst optimiert, um effiziente RNA Lieferung zu gewährleisten.
- TrypsinISE eine Monoschicht von BHK-21-Zellen (oder BSR-T7-Zellen), Samen 7,5 x 10 5 Zellen in 35 mm Durchmesser Schale in Antibiotika-freien Wachstumsmediums und Inkubieren der Zellen bei 37 ° C mit 10% CO 2 über Nacht. Die doppelte Menge an Zellen in jeder Platte, wenn die Transfektionen für den gleichen Tag wie die Aussaat sind geplant, und lassen Zellen auf der Platte für 2-3 Stunden bei 37 ° C halten mit 10% CO 2. Beachten Sie, dass andere Zellen, die geeignet sind, diesem Ansatz sind menschliche 293T-Zellen, humanen hepatozellulären Karzinoms Huh7-Zellen und afrikanischen grünen Meerkatze Cos7-Zellen umfassen.
- Entfernen Sie die Medien aus den Zellen und ersetzen mit 3 ml frisches Medium ohne Antibiotika, um die maximale Effizienz der Transfektion zu gewährleisten.
- Bereiten Sie eine Mischung von 1-2 pg capped MNV-Transkript in 100 mu l Opti-MEM (Invitrogen) und mischen Sie es mit 4 ul Lipofectamine 2000 zuvor in 100 ul Opti-MEM gemischt. Mischen Sie die Probe gründlich durch Pipettieren es rauf und runter 15-mal. Verlassendas Gemisch bei Raumtemperatur für 20 Minuten.
- Fügen Sie die Transfektion Komplexe mit capped MNV-Transkripte in einer Dropdown-weise auf die Art und Weise Zellmonolayer und schütteln Sie die Platte in senkrechten Richtungen.
- Inkubieren der Zellen bei 37 ° C und 10% CO 2 für 24 bis 72 Stunden. Danach loslassen infektiösen Virionen von Zellen durch Einfrieren und Auftauen und bestimmen Virustiter mittels Plaque-Assay oder TCID50. Typische Ausbeuten von etwa 1 x 10 6 TCID50/ml erreicht werden.
2. Direkte Gewinnung von Infectious MNV aus cDNA in Zellen, die T7-RNA-Polymerase
Dieses Protokoll ist für die Rückgewinnung von MNV in Zellen durch die Transkription eines infektiösen Plasmid, die vollständige genomische cDNA-Sequenz von einem T7-Polymerase in den Zellen exprimiert zu ermöglichen. Verschiedene Zelllinien verwendet werden, um infektiöse MNV durch diesen Ansatz erholen werden, obwohl wir erhalten typischerweise die höchsten Ausbeuten mit BHK-21 und BSR-T7 ceLLS 15. Wir verwenden in der Regel BSR-T7-Zellen, da sie schneller als der elterliche BHK-Klon Linie wachsen. Die Zellen werden mit Geflügelpockenvirus (FPV) kodiert für die T7-RNA-Polymerase (FPV-T7) 18, der als Helfervirus-Funktionen, um die Expression der viralen RNA und die anschließende Gewinnung von infektiösen Viren (4) anzutreiben infiziert. Obwohl BSR-T7-Zellen konstitutiv T7-RNA-Polymerase, ist dieser Ausdruck nicht ausreicht, infektiöse MNV nach Transfektion von pT7 retten: MNV 3'Rz in Abwesenheit von Helfer-FPV-T7. Während die typische Ausbeuten von diesem System sind mindestens 10-fach niedriger als die oben beschriebenen, wird bei diesem Verfahren wird eine schnelle Methode zum Screening-Mutanten, die Identifikation von schwächenden Mutationen zu ermöglichen. Typischerweise wird diese Methode wird zuerst, um die Lebensfähigkeit einer cDNA-Konstrukt Beurteilung verwendet. Sollte ein Konstrukt entweder nicht infektiöses Virus produzieren oder Viren scheinen auf den unteren Ebenen als die des Wildtyp-infektiösen Klon nachgeben, dann die RNA-basierte approach oben beschrieben vorgenommen wird.
- Trypsinise eine Monoschicht von BHK-21-Zellen (oder BSR-T7-Zellen) und mit Saatgut 7,5 x 10 5 Zellen in einer 35 mm-Schale in Antibiotika freie Wachstumsmedium und Inkubieren der Zellen bei 37 ° C und 10% CO 2 über Nacht. In die doppelte Menge an Zellen in jeder Platte, wenn die Transfektionen für den gleichen Tag wie die Seeder geplant sind, und ermöglichen Zellen auf der Platte für 2-3 Stunden lang anhaften bei 37 ° C und 10% CO 2.
- Entfernen Sie Zellkulturmedien und fügen Sie 700 ul FPV-T7 in jede Vertiefung (Abbildung 4). Ein Multiplizität der Infektion (MOI) von ~ 0,5 PFU pro Zelle auf Titrationen primären Hühnerembryofibroblasten basiert, wird im Allgemeinen verwendet. Es ist jedoch bemerkenswert, dass neue Präparate von Helfer-Virus in primären Fibroblasten gewachsen sind funktionell titriert, um die Dosis für einen effizienten Viren-Wiederherstellung erforderlich zu bestimmen. Protokolle zur Vermehrung und Titration von FPV-T7 wurden zuvor 18 beschrieben.
- Incubate bei 37 ° C und 10% CO 2 für 1 Stunde, bis FPV-T7, um die Zellen zu infizieren. Dann werden 2 ml des Antibiotikums DMEM, enthaltend 10% FCS und Inkubation der Zellen für eine weitere Stunde bei 37 ° C und 10% CO 2 bis T7-RNA-Polymerase Expression erlauben.
- Um mit der Transfektion des infektiösen Plasmid vorgehen, zunächst entfernen Sie die Medien aus den infizierten Zellen, mit 2 ml Medium (10% FCS in der Antibiotika-freiem DMEM) gewaschen und schließlich decken die Zellrasen mit 3 ml Medium. Antibiotika nicht zu dem Medium zugegeben werden, da sie mit der Effizienz des Lipofectamin 2000 (Invitrogen) stören können.
- Bereiten Sie eine Mischung aus 1 pg Wildtyp MNV infektiösen cDNA-Plasmid (zB pT7: MNV 3'Rz) in 100 ul Opti-MEM (Invitrogen) und mischen Sie es mit 4 ul Lipofectamin 2000 (Invitrogen), die vorher mit 100 ul gemischt Opti-MEM (Invitrogen). Mischen Sie die Reaktion durch Pipettieren gründlich es rauf und runter 15-mal und halten Sie die Mischung bei Raumtemperatur bestimmt wortemperatur für 20 Minuten.
- Die daraus resultierende Transfektionsmix sollte dann gegeben werden, um die Zellmonoschicht tropfenweise und die Platte sollte sanft in senkrechten Richtungen geschüttelt werden.
- Inkubieren Sie das FPV-T7 infizierten, MNV Plasmid-transfizierten Zellen bei 37 ° C und 10% CO 2 für 24 bis 72 Stunden. Zellen, die mit infektiösen Plasmid pT7 transfiziert: MNV 3'Rz normalerweise machen Titer von 1 x 10 4 bis 5 x 10 4 TCID50/ml.
3. Repräsentative Ergebnisse
Beide Ansätze sind reversen Genetik hocheffiziente zur Rückgewinnung von infektiösen MNV in der Zellkultur, wie in Abbildung 5 dargestellt. Infektiöse MNV mit Titern von mehr als 10 5 TCID50/ml bei 24 Stunden nach der Transfektion der begrenzten MNV RNA in RAW 264.7-Zellen gewonnen. Auch die Transfektion von infektiösen Plasmid pT7: MNV 3'Rz in BSR-T7-Zellen zuvor mit Helfer FPV auszudrücken T7 (FPV-T7) führte zu Virustiter weitgehend überschritten infiziertding 10 4 TCID50/ml (Abbildung 5). Diese viralen Titer Werte mit synthetischen RNA und DNA-Moleküle erhalten werden, ähnlich den in Transfektionen mit natürlichen VPg-linked-RNA aus infektiösen Virionen in den gleichen Zellen (5) isoliert wurden. Diese Ergebnisse unterstreichen die hohe Effizienz der reversen Genetik Ansätze hier, um genetisch definierten erholen MNV Varianten in Zellkultur beschrieben.
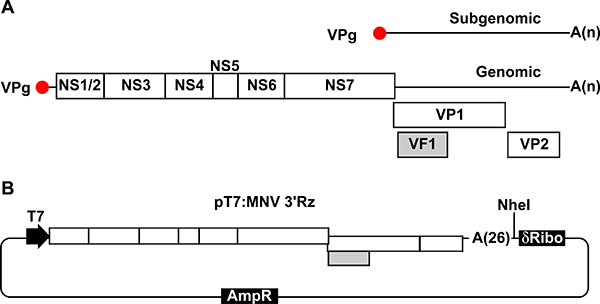
Abbildung 1. Illustration von MNV Genom und Plasmid für die Rückgewinnung des infektiösen Virus. A, Schematische Darstellung der MNV-Genom-Organisation. Jedes Protein kodierende Region wird als ein einzelnes weißes Feld dargestellt. ORF1 in 7 verschiedene nicht-strukturelle Proteine (NS1 / 2 bis NS7), die von Precursor-Polyproteins nach Selbstbestäubung proteolytische Prozessierung freigesetzt übersetzt. ORF 2 kodiert das Major Capsid Protein VP1, ORF 3 kodiert für die kleine Kappesid Protein VP2 und ORF4 Überschneidungen mit ORF2 kodierenden Region kodiert Virulenzfaktor VF1. Genomische und subgenomischen RNAs enthalten polyA-Schwanz am 3'-Enden von variabler Länge. B, Plasmid mit MNV cDNA verwendet in unserem Reverse genetische Ansätze (pT7: MNV 3'Rz). MNV cDNA mit einem polyA-Schwanz von 26 Resten in seinem 3'-Ende fusioniert ist. Die MNV cDNA-Sequenz ist unmittelbar stromabwärts eines abgeschnittenen T7-Promotorsequenz, die T7-Transkription angetriebenen ermöglichen, und vor einer einzigartigen Nhe I-Stelle und eine DNA-Sequenz, die ein selbst-spaltende Ribozym nach. Diese Sequenzen sind für die Sicherstellung instrumentellen RNA-Transkription Kündigungsrecht, nachdem die genomische polyA-Schwanz am 3'-Ende.
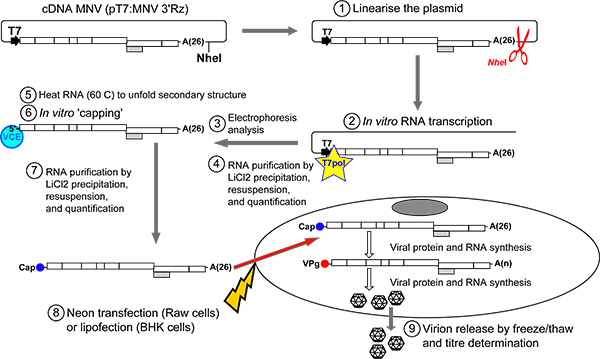
Abbildung 2. Überblick über das Protokoll für die Gewinnung von infektiösen MNV aus RNA transkribiert und in vitro gedeckelt Das Plasmid pT7:. MNV 3'Rz unmittelbar hinter linearisiertder MNV genomische Sequenz mit NheI Restriktionsenzym (Schritt 1). Nach der DNA-Reinigung werden MNV RNA-Transkripte in vitro durch Verwendung von T7-RNA-Polymerase (Schritt 2) erzeugt wird. Transkription Produkte in der Regel mit einem scheinbaren Mobilität von 2,5-3Kb laufen eines nicht-denaturierenden 1% igen Agarosegel (Schritt 3, 3). Die Template-DNA wird unter Verwendung eines kommerziellen eliminiert RNAse-freier DNAse. RNA wird dann aus freien Nukleotiden durch LiCl-Fällung (Schritt 4) gereinigt. Die gereinigte RNA Produkt kann dann in vitro sein gekappt, nachdem er zuvor bei 65 ° C erhitzt, um sekundäre RNA-Strukturen (Stufen 5-6) zu entfalten. Nach Reinigung durch LiCl-Fällung wird die RNA entweder in RAW 264.7-Zellen (Neon Transfektionssystem, Invitrogen) oder BSR-T7-Zellen (Lipofectamine 2000, Invitrogen) (Schritte 7-8) transfiziert. Einmal in der Zelle, wird verkappten RNA-Transkripte in virale Proteine, die virale Replikation Transkripte in neue MNV RNA-Moleküle würden, von übersetzt werdenenthaltend eine ordnungsgemäße VPg Molekül an ihrem 5'-Ende. Aufeinander folgenden Zyklen der Replikation der viralen Übersetzung beigefügt erzeugen würde eine große Zahl von viralen Genomen, die eingekapselt zu infektiösen Virionen generieren wird. Um Virus-Freisetzung aus Zellen zu erleichtern, werden eine oder mehrere Zyklen von Einfrieren und Auftauen durchgeführt (Schritt 9). Virale Erträge können dann durch TCID50 oder Plaque-Assay-Verfahren ermittelt werden.
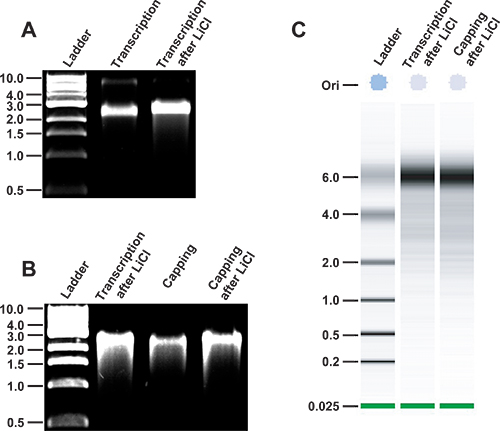
Abbildung 3. Die Analyse der RNA-Transkripte MNV Integrität entlang der Protokoll. A, der Integrität der RNA synthetisiert MNV in vitro. Das Plasmid pT7: MNV 3'Rz wird zunächst linearisiert mit NheI Restriktionsenzym. Nach der DNA-Reinigung werden MNV RNA-Transkripte in vitro durch Verwendung von T7-RNA-Polymerase (Spur 2) erzeugt wird. RNA wird dann purifi ED aus freien Nukleotiden durch LiCl-Fällung (Bahn 3). Transkription Produkte werden auf einem nicht-denaturierenden 1% Agarose-Gel parallel zu 1-Kb-DNA-Leiter (New England Biolabs, Spur 1) verlaufen. Relative Mobilität der viralen Transkripte unter nicht-denaturierenden Bedingungen ist ähnlich einer dsDNA Produkt von 2,5-3 Kb. B, der Integrität der RNA-Transkripte MNV nach Kappung. MNV Transkripte zuvor durch LiCl-Fällung (Spur 2) gereinigt werden, um die enzymatische Abdeckung (Spur 3) und Reinigung durch LiCl-Fällung (Spur 4) unterzogen. C,,-Analyse in einem Agilent RNA 6000 Nano Chip von MNV Transkripte (zweite Spur) und verkappten MNV Transkripte (dritte Spur), die zuvor in LiCl gefällt. Ein ssRNA Leiter wird parallel laufen.
= "Pdflinebreak"> 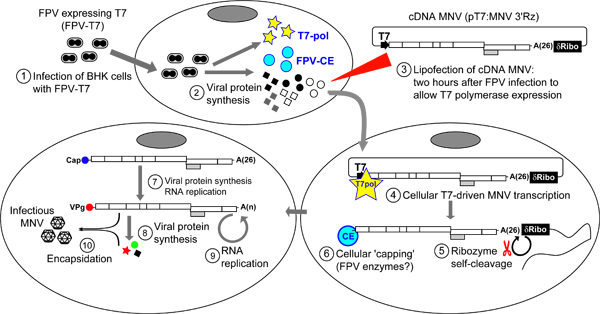
Abbildung 4. Übersicht des Protokolls für die Rückgewinnung von infektiösen MNV aus cDNA. Zunächst BSR-T7 (oder BHK-Zellen) werden mit einem rekombinanten Geflügelpocken-Virus (FPV), in den Bakteriophagen T7-RNA-Polymerase (FPV-T7) (Schritt 1) infiziert. Die infizierten Zellen werden für 2 Stunden inkubiert, bevor eine weitere Behandlung die Expression von FPV Proteine, die das rekombinante T7-RNA-Polymerase (Schritt 2) zu ermöglichen. Danach pT7: MNV wird 3'Rz in die Zellen durch Verwendung von Lipofectamin 2000 (Invitrogen) (Schritt 3) transfiziert. Einmal in der Zelle, pT7: MNV 3'Rz durch T7-RNA-Polymerase, die RNA-Transkripte MNV (Schritt 4) synthetisiert erkannt. Die Gegenwart eines Selbst-spaltende δ-Ribozym-Sequenz am 3'-Ende des Genoms garantiert der Transkript 3 'terminus wird direkt nach dem polyA-Schwanz (Schritt 5). Einige viralen Transkripte werden intrazellulär durch einen FPV Capping-Enzym (Schritt 6) begrenzt. Die daraus resultierenden MNV capped Transkripte werden übersetzt, um Proteine, die MNV MNV Transkripte Replikation katalysieren würden zu generieren. Neu synthetisierte MNV RNA-Moleküle, eine ordnungsgemäße VPg Molekül an ihrem 5'-Ende würde unterzogen aufeinanderfolgenden Zyklen der Replikation von viralen Übersetzung, die schließlich in der Erzeugung von infektiösen Virus führen eingekapselte begleitet. Um Virus-Freisetzung aus Zellen zu erleichtern, werden eine oder mehrere Zyklen von Einfrieren und Auftauen durchgeführt (Schritt 7). Virale Erträge können dann durch TCID50 oder Plaque-Assay-Verfahren ermittelt werden.
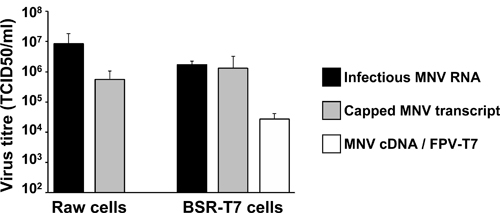
Bild 5. Repräsentative Ergebnisse Virustiter von verschiedenen reversen Genetik nähert im Text beschriebenen Verfahren erhalten. Graue Balken stellen die Virustiter 24 Stunden nach der Neon erhalten-Transfektion von 2 x 10 6 RAW 264.7-Zellen, oder nach der Lipo-Transfektion von 2 x 10 6 BSR-T7-Zellen mit in vitro transkribierten und verkappten MNV-RNA. Weiße Balken stellen den Virustiter typischerweise nach Lipofektion von pT7 erhalten: MNV 3'Rz (MNV cDNA) in 2 x 10 6 BSR-T7-Zellen zuvor für 2 Stunden mit Geflügelpockenvirus exprimierende rekombinante T7-Polymerase (FPV-T7) infiziert. Als positive Kontrolle für die Transfektion in Roh-und BSR-T7cells, wir verwenden in der Regel 2 ug RNA aus Zellen mit MNV infiziert, die ein hohes Maß an VPG-linked MNV-RNA enthalten, extrahiert. Negative Kontrollen wurden entweder mit MNV-RNA oder pT7 durchgeführt: MNV 3'Rz Codieren einer Frameshift-Mutation (F / S), welche die Replikation aufhebt, was zu keiner nachweisbaren Virus (Daten nicht gezeigt).
Diskussion
Hier haben wir zwei verschiedene Reverse genetische Ansätze, die die Rückgewinnung von infektiösen MNV in Zellkultur ermöglichen dargestellt. Beide Ansätze wirksam zu umgehen die absolute Voraussetzung für die Verknüpfung kovalent VPg an das 5'-Ende des viralen RNA-Genoms über die Erzeugung der begrenzten MNV Transkripte, die dann von den zellulären Ribosomen erkannt werden. In-vitro-Transkription durch enzymatisch Capping gefolgt ist effizienter die Rückgewinnung von infektiösen MNV als Transkription von infektiösen Plasmide in Zellen, die T7-RNA-Polymerase, bei dem die Transkripte von den FPV Capping Enzyme verschlossen werden kann. Virustiter mit diesen reversen Genetik zurückgewonnen entsprechen denen, die durch Transfektion von viralen VPg-verknüpfte RNA aus infizierten Zellkulturen 17 (5) gereinigt erhalten. Die Transfektion der begrenzten MNV-RNA in permissiven RAW 264.7-Zellen macht eine Virustiter nur 1-log niedriger als Experimente mit der transfection der Gesamt-RNA aus infizierten Zellen mit viralen VPG-linked-RNA (Abbildung 5). Diese Tatsache unterstützt weitere Untersuchungen zu bestimmen, ob die Zugabe eines VPg Molekül an das 5'-Ende der Transkripte von diesen Systemen erzeugten zu einer erhöhten Virusausbeuten die funktionalen Aspekte zugrunde MNV Infektiosität in Zellen zugeordnet VPg untersucht werden kann, führen. Trotzdem betrachten wir dieses System als reversen Genetik umgekehrt eine hocheffiziente ein vergleichbar mit anderen RNA-Viren Genetik Systeme, die derzeit verwendet werden, in dem die In-vitro-transkribierte RNA die Erholung der Titer nur 10-100 niedriger als im wirklichen Infektionen mit Virionen 19, 20 ermöglicht.
Insgesamt stellen die aktuellen Methoden einen erheblichen Fortschritt auf dem Gebiet der Molekularbiologie und der Norovirus liefern uns die Werkzeuge, um die funktionelle Rolle von Proteinen und RNA-Motive in konservierten Norovirus Genomen zu untersuchen. Diese Konzepte wurden bereits mit C kombinierttrom Mausmodell zur Verfügung und haben gezeigt, dass MNV von infektiösen cDNA erholt ist in der Lage, tödliche Infektion von> 80% STAT1-/ verursachen - Mäuse in weniger als 10 Tagen 4, 21. Die Nutzung dieses Systems haben wir tragfähige murine Norovirus Mutanten des Kapsidprotein und einer Polypyrimidintrakt bei der Bindung von verschiedenen Host-Faktoren (PTB und PCBP), dass eine etwas abgeschwächte Phänotypen zeigen in vivo 21, 22 beteiligten sich erholt haben. Darüber hinaus haben wir vor kurzem gezeigt, dass Viren die Fähigkeit fehlt, die VF1 Protein aus ORF4 effizient ausdrücken in der Zellkultur zu replizieren, aber wieder haben Virulenz in Mäusen, die mit Bezug auf WT MNV 8 reduziert. Diese Studien ermutigen uns, abgeschwächte Versionen von menschlichen Noroviren auf MNV Studien, die als potenzielle Impfstoffe untersucht werden konnte entwerfen.
Offenlegungen
Wir haben nichts zu offenbaren.
Danksagungen
Diese Arbeit wurde durch eine Wellcome Trust Senior Fellowship ausgezeichnet zu Ian Goodfellow, und ein Marie Curie Intra European Fellowship (RP7 European Research Council) ausgezeichnet zu Armando Arias finanziert. Wir möchten Io Hong Cheong, Dr. Rebecca Robey und Dr. Mike Skinner, dass er uns die Erlaubnis, ihre Agilent Bioanalyzer bedienen und hilft mit fließendem RNA-Proben zu danken.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenzes | Firma | Katalog-Nummer | |
| Lithiumchlorid Fällungslösung | Ambion | AM9480 | |
| RNA-Storage-Lösung | Ambion | AM7000 | |
| MEGAscript T7-Skript | Ambion | AM1333 | |
| ScriptCap m7G Capping-System | Epicentre Biotechnologies | SCCE0610 | |
| Neon Transfektionssystem | Invitrogen | MPK5000 | |
| Neon Transfektionssystem Kit | Invitrogen | MPK1025 | |
| Opti-MEM I | Invitrogen | 31985070 | |
| Lipofectamine 2000 Transfektionsreagenz | Invitrogen | 11668-027 | |
| Agilent RNA 600 Nano-Kit | Agilent | 5067-1511 | |
| Agilent 2100 bioanalyzer | Agilent | G2939AA | |
| Illustra GFX PCR DNA und Gel Band Purification Kit | GE Healthcare | 28-9034-70 | |
| RiboMAX Large Scale RNA Production System-T7 | Promega | P1300 |
Referenzen
- . Outbreaks of gastroenteritis associated with noroviruses on cruise ships--United States. MMWR Morb. Mortal. Wkly. Rep. 51, 1112-1115 (2002).
- Lopman, B. A. Epidemiology and cost of nosocomial gastroenteritis, Avon, England, 2002-2003. Emerg. Infect. Dis. 10, 2002-2003 (2004).
- Duizer, E. Laboratory efforts to cultivate noroviruses. J. Gen. Virol. 85, 79-87 (2004).
- Karst, S. M., Wobus, C. E., Lay, M., Davidson, J., Virgin, H. W. T. STAT1-dependent innate immunity to a Norwalk-like virus. Science. 299, 1575-1575 (2003).
- Wobus, C. E. Replication of Norovirus in cell culture reveals a tropism for dendritic cells and macrophages. PLoS Biol. 2, e432 (2004).
- Bok, K. Inhibition of norovirus replication by morpholino oligomers targeting the 5'-end of the genome. Virology. 380, 328-337 (2008).
- Kim, Y., Thapa, M., Hua, D. H., Chang, K. O. Biodegradable nanogels for oral delivery of interferon for norovirus infection. Antiviral Res. 89, 165-173 (2011).
- McFadden, N. Norovirus Regulation of the Innate Immune Response and Apoptosis Occurs via the Product of the Alternative Open Reading Frame 4. PLoS Pathog. 7, e1002413 (2011).
- Kormelink, R., van Poelwijk, F., Peters, D., Goldbach, R. Non-viral heterogeneous sequences at the 5' ends of tomato spotted wilt virus mRNAs. J. Gen. Virol. 73, 2125-2128 (1992).
- Shuman, S., Hurwitz, J. Mechanism of mRNA capping by vaccinia virus guanylyltransferase: characterization of an enzyme--guanylate intermediate. Proc. Natl. Acad. Sci. U.S.A. 78, 187-191 (1981).
- Jang, S. K., Pestova, T. V., Hellen, C. U., Witherell, G. W., Wimmer, E. Cap-independent translation of picornavirus RNAs: structure and function of the internal ribosomal entry site. Enzyme. 44, 292-309 (1990).
- Chaudhry, Y. Caliciviruses differ in their functional requirements for eIF4F components. J. Biol. Chem. 281, 25315-25325 (2006).
- Goodfellow, I. Calicivirus translation initiation requires an interaction between VPg and eIF 4. E. EMBO Rep. 6, 968-972 (2005).
- Herbert, T. P., Brierley, I., Brown, T. D. Identification of a protein linked to the genomic and subgenomic mRNAs of feline calicivirus and its role in translation. J. Gen. Virol. 78 ( Pt5 ), 1033-1040 (1997).
- Chaudhry, Y., Skinner, M. A., Goodfellow, I. G. Recovery of genetically defined murine norovirus in tissue culture by using a fowlpox virus expressing T7 RNA polymerase. J. Gen. Virol. 88, 2091-20100 (2007).
- Ward, V. K. Recovery of infectious murine norovirus using pol II-driven expression of full-length cDNA. Proc. Natl. Acad. Sci. U.S.A. 104, 11050-11055 (2007).
- Yunus, M. A., Chung, L. M., Chaudhry, Y., Bailey, D., Goodfellow, I. Development of an optimized RNA-based murine norovirus reverse genetics system. J. Virol. Methods. 169, 112-118 (2010).
- Arias, A., Perales, C., Escarmis, C., Domingo, E. Deletion mutants of VPg reveal new cytopathology determinants in a picornavirus. PLoS One. 5, e10735 (2010).
- Werf, S. v. a. n. d. e. r., Bradley, J., Wimmer, E., Studier, F. W., Dunn, J. J. Synthesis of infectious poliovirus RNA by purified T7 RNA polymerase. Proc. Natl. Acad. Sci. U.S.A. 83, 2330-2334 (1986).
- Bailey, D., Thackray, L. B., Goodfellow, I. G. A single amino acid substitution in the murine norovirus capsid protein is sufficient for attenuation in vivo. J. Virol. 82, 7725-7728 (2008).
- Bailey, D. Functional analysis of RNA structures present at the 3' extremity of the murine norovirus genome: the variable polypyrimidine tract plays a role in viral virulence. J. Virol. 84, 2859-2870 (2010).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten