Method Article
Génétique inverse de récupération des norovirus murin médiation infectieuses
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Les norovirus sont une cause majeure de gastro-entérite encore des techniques moléculaires pour leur caractérisation sont encore relativement nouvelle. Nous rapportons ici deux différentes approches de génétique inverse pour la récupération efficace des norovirus murin (MNV), le seul membre de ce genre qui peuvent être propagées en culture cellulaire.
Résumé
Norovirus humains sont responsables de la plupart des cas de gastro-entérite humaine (GE) dans le monde entier et sont des problèmes récurrents dans des environnements où près de personne à personne de contact ne peuvent pas être évitées 1, 2. Au cours des dernières années une augmentation de l'incidence des épidémies dans les hôpitaux a été signalée, ce qui provoque d'importantes perturbations de leur capacité opérationnelle ainsi que d'importantes pertes économiques. L'identification de nouvelles approches antivirales a été limitée en raison de l'incapacité des norovirus humains pour compléter une infection productive dans 3 culture cellulaire. L'isolement récent d'un norovirus murin (MNV), étroitement liée à norovirus humain 4, mais qui peut se propager dans les cellules 5 a ouvert de nouvelles voies pour l'enquête de ces agents pathogènes 6, 7.
Les résultats de réplication MNV dans la synthèse de génomique nouveau sens positif et des molécules d'ARN sous-génomique, celui-ci correspond à la dernière thie du génome viral (figure 1). MNV contient quatre différents cadres de lecture ouverts (ORF), dont ORF1 occupe la majeure partie du génome et code sept protéines non structurales (NS1-7) libérés à partir d'un précurseur de polyprotéine. ORF2 et ORF3 sont contenus dans la région sub-génomique de l'ARN et coder les protéines de capside (VP1 et VP2, respectivement) (figure 1). Récemment, nous avons déterminé que l'ORF4 supplémentaires chevauchement ORF2, mais dans un cadre de lecture différent est fonctionnel et code pour un facteur de virulence localisée mitochondrial (VF1) 8.
La réplication des virus à ARN positif pour le sens, y compris les norovirus, se déroule dans le cytoplasme aboutissant à la synthèse de nouveaux génomes ARN non plafonnés. Pour promouvoir la traduction virale, les virus profitent de différentes stratégies visant à recruter la machinerie de synthèse protéique cellulaire 9-11. Fait intéressant, la traduction norovirus est entraîné par le multifonctionnel protéine virale VPg-primaireliée de manière covalente à l'extrémité 5 'de deux ARN génomique et subgénomique 12-14. Ce mécanisme sophistiqué de la traduction est susceptible d'être un facteur majeur dans l'efficacité limitée de la reprise virale par les approches conventionnelles de génétique inverse.
Nous rapportons ici deux stratégies différentes basées sur la génération de souris norovirus-1 (dénommé ci-joint MNV) transcriptions plafonnées à l'extrémité 5 '. Une des méthodes consiste à la fois la synthèse in vitro et le plafonnement de l'ARN viral, tandis que la seconde approche implique la transcription de MNV ADNc dans des cellules exprimant la T7 RNA polymérase. La disponibilité de ces systèmes de génétique inverse pour l'étude de MNV et un petit modèle animal a fourni une capacité sans précédent à disséquer le rôle de séquences virales dans la réplication et la pathogenèse 15-17.
Protocole
1. Transcription de l'ARN et de coiffage pour la récupération des maladies infectieuses MNV
Ce protocole est conçu pour permettre la récupération efficace de MNV infectieuses à partir d'ADNc par transcription in vitro et ses modifications ultérieures in vitro de coiffage (section 1.1). Les transcriptions résultant plafonnés sont ensuite transfectés dans des cellules pour récupérer infectieuse MNV (sections 1.2 et 1.3). Cette approche fournit la méthode la plus sensible pour la récupération de MNV avec des rendements typiques de plus de 10 5 unités infectieuses par 35 mm (de diamètre) des plats de cellules pour MNV. Le protocole est détaillé ci-dessous:
1.1 Synthèse des maladies infectieuses transcriptions MNV plafonnés:
- Digérer le plasmide contenant le type sauvage ADNc MNV (pT7: MNV 3'Rz) avec Nhe I pour obtenir l'ADN linéaire Nhe I reconnaît un site de restriction unique après l'extrémité 3 'polyA queue de MNV génome (Figure 2).. Plasmides linéarisés sont habituellement purifiésavec l'aide de colonnes de silice (par exemple GFX PCR Gel ADN Band kit de purification de GE Healthcare) et élue dans H 2 O.
- In vitro transcrire le vecteur linéarisé en utilisant l'ARN polymérase T7, comme décrit précédemment 17. De nombreux kits commerciaux sont disponibles à cet effet et de fournir une méthode reproductible de grandes quantités de synthèse de l'ARN comme MEGAscript (Life Technologies) et RiboMAX (Promega). Réactions de transcription sont généralement DNAse digéré avant de l'analyser, mais dans de nombreux cas ce n'est pas nécessaire que les purifications de chlorure de lithium comme décrit ci-dessous ne précipitent pas l'ADN de manière efficace.
- Analyser une aliquote de la réaction de transcription d'ARN, typiquement 0,5 pi ou moins, par électrophorèse sur gel d'agarose pour assurer la réaction de transcription fonctionne efficacement et d'ARN est pleine longueur. Alors que de nombreux utilisateurs peuvent exécuter des gels dénaturants correctement la taille de l'ARN, nous utilisons généralement non dénaturant sur gel d'agarose electrophorSIAE comme une méthode rapide pour analyser l'intégrité de l'ARN. Le génome MNV que produit à partir du clone d'ADNc infectieux pT7: MNV 3'Rz fonctionnera à environ 3 Kbp par rapport à une échelle ADNdb sur un gel non dénaturant d'agarose (figure 3).
- Considérez que d'autres méthodes sont disponibles comme une alternative pour obtenir une analyse rapide de l'intégrité ARN tels que l'aide d'un Bioanalyser Agilent (Figure 3).
- Notez que la résolution de gel pauvres peuvent être rencontrés si trop de l'ARN est chargé. Prenez grand soin afin d'assurer le gel d'agarose est préparé en utilisant la RNAse-libres réactifs pour éviter la dégradation des ARN pendant l'électrophorèse qui peuvent influer sur la résolution de bande. Chauffage ARN à 65 ° C suivi d'un refroidissement sur de la glace peut également aider dans certains cas.
- Purifier l'échantillon d'ARN pour éliminer les nucléotides non incorporés. De nombreuses méthodes sont disponibles pour cette approche, y compris colonne de silice en fonction cependant, dans ce protocole nous utilisons habituellement le chlorure de lithium comme une alternative rentable. PourA cette fin, ajouter la lettre H 2 O pour atteindre un volume final de 100 pl puis ajouter 40 ul de solution de précipitation du chlorure de lithium (7,5 M LiCl, 50 mM, EDTA pH 8,0, Ambion) et stocker l'échantillon à -20 ° C pendant au moins au moins 30 min.
- Pellet l'ARN par centrifugation à 12.000 X g à 4 ° C pendant 15 min.
- Eliminer le surnageant, en prenant soin de ne pas perturber le culot d'ARN translucide et le laver dans 150 pi d'éthanol à 70%. Centrifuger le tube à 12.000 X g à 4 ° C pendant 15 min.
- Retirer de l'éthanol et sécher à l'air de l'ARN, en évitant le culot de sécher complètement, car cela rendrait la remise en suspension difficile.
- Puis, remettre les relevés de notes dans MNV 50-100 ul d'ARN solution de stockage (Ambion). Des précautions doivent être prises pour s'assurer que tous les ARN a dissous correctement. Si les ARN semblent difficiles à dissoudre complètement, chauffer l'échantillon à 60 ° C peut aider à la remettre en suspension. Toute matière insoluble devrait alors être retiré bcentrifugation y avant la quantification d'ARN. Les transcriptions purifiés sont plafonnés et exigent une ultérieure à l'étape in vitro plafonnement d'être infectieux (Système M7G ScriptCap Bouchage, Biotechnologies Epicentre).
- Quantifier les ARN par spectrophotométrie. Selon la nature et l'ampleur de la réaction de transcription, les rendements typiques vont de 50 à 150 ug d'ARN par 100 ul réaction de transcription. L'intégrité de l'ARN doit être analysé avant la réaction de coiffage en exécutant à 100-300 ng d'échantillon dans un gel d'agarose 1% (Figure 3).
- Pour améliorer l'efficacité de l'ARN plafonnement, la chaleur de 60 à 70 pg de transcrits d'ARN MNV à 65 ° C pendant 10 min puis placer le tube immédiatement sur la glace. Cette étape peut réduire tout effet inhibiteur de la structure de l'ARN sur le plafonnement. Impulsions du tube dans une micro-réfrigéré pour collecter des gouttelettes formées au cours de l'étape de chauffage.
- Préparer un mélange réaction de coiffage comme suggéré par le fabricant (ScriptCap M7GSystème de plafonnement, Biotechnologies Epicentre). En bref, ajouter 60 à 70 pg de MNV ARN à un volume final de réaction de 100 pl. Le mélange réaction de coiffage peut contenir 10 ul de 10 x plafonnement tampon (500 mM Tris-HCl pH 8,0, KCl 60 mM, 12,5 mM MgCl2), 10 pl de 10 mM de GTP, 0,5 pi de 20 mM S-adénosyl méthionine, ul 2.5 sur Scriptguard (100 unités), et 4 pl de l'enzyme Scriptcap (40 unités).
- Au cours de la réaction mis en place, maintenir l'ARN transcrits sur la glace pour éviter la dégradation. Mélangez bien le mélange réactionnel, puis incuber à 37 ° C pendant 1 h. Notez la taille de la réaction peut être mise à l'échelle en fonction du montant de la transcription plafonné nécessaire.
- Purifier l'ARN par précipitation de LiCl comme expliqué ci-dessus (voir point 1.1.6). Dissoudre le culot dans 50-100 ul d'ARN solution de stockage (Ambion) et de quantifier la quantité d'ARN. En règle générale, les échantillons d'ARN sont ensuite normalisés à 1 ug / ul. Encore une fois, vérifiez tous les ARN a dissous correctement. Si elle n'a pas été correctement dissous la chaleur, til échantillon à 60 ° C pour permettre la dissolution. Retirer par centrifugation tout matériel insoluble avant ARN quantification.
- Vérifier l'intégrité de l'ARN à nouveau avant de procéder à l'étape de transfection. Dans ce but, exécutez un montant de 100-300 ng d'échantillon dans un gel d'agarose 1% (Figure 3).
1.2 Recouvrement par Neon-transfection médiée par l'ARN dans les cellules RAW264.7:
Pour la récupération des virions infectieux dans MNV une lignée cellulaire permissive, il est possible de électroporer la transcription plafonné MNV en RAW264.7 cellules en utilisant le système de transfection Neon (Invitrogen). RAW264.7 sont des cellules sensibles à l'infection MNV, en soutenant de multiples séries de la réplication du virus et la suite ré-infection. En conséquence, les rendements typiques approchera de plus de 10 5 unités infectieuses par ml à 24 heures, mais après la transfection pic à plus de 10 7 unités infectieuses après 48 heures.
- Un jour avant transfection, graines RAW264.7 cellules à environ 50% de confluence. En général, deux flacons T75 de cellules sont nécessaires pour 3 transfections.
- Le jour de la transfection, gratter les monocouches de cellules dans un milieu de Eagle modifié par Dulbecco (DMEM) contenant 10% de sérum de veau foetal (FCS), en vous assurant de générer une seule suspension cellulaire par pipetage répété.
- Déterminer la concentration de cellules viables dans un hémocytomètre en utilisant exclusion du bleu trypan pour étiqueter les cellules non-viables.
- Pellet les cellules à 1200 X g pendant 5 min et les remettre en suspension dans du DMEM contenant 10% de SVF à une concentration finale de 8 x 10 6 cellules / ml.
- Juste avant la transfection, aliquote de 1 ml de cellules par transfection et le culot eux à 1200 X g pendant 2 min. Retirez le support et laver les cellules dans 500 ul de PBS (sans Mg 2 + / Ca 2 +). Isoler les cellules à nouveau à 1200 X g pendant 2 min. Notez qu'il est conseillé de garder les cellules dans du DMEM pour aussi longtemps que possibleque le stockage dans du PBS pendant de longues périodes de temps peut compromettre la viabilité des cellules et le taux de transfection.
- Retirez du PBS à partir de tubes et ajouter 130 ul d'une solution de remise en suspension (Neon kit système de transfection, Invitrogen) à une concentration finale de 6 x 10 7 cellules / ml. Des précautions doivent être prises pour remettre les cellules en évitant la formation de bulles qui provoquent des étincelles lors de la transfection et la survie cellulaire compromis.
- Ajouter la quantité appropriée de la transcription MNV plafonné aux cellules (Figure 2), généralement 1,3 mg d'ARN coiffée est ajouté à 130 ul de cellules remises en suspension et mélangés doucement. Ensuite, recueillir 100 ul du mélange dans les 100 pointe Neon transfection ul. Une attention particulière devrait être prises pour veiller à ce qu'aucun des bulles se forment dans la cuvette d'électroporation (Neon kit système de transfection 100 pointe pl) car cela peut provoquer une panne de l'expérience.
- Électroporer les cellules en utilisant une seule impulsion à 1.700 V pour 25 ms, enSuring l'absence d'étincelles lors de pulsation qui indiquent la présence de bulles dans l'échantillon. Dans le cas des étincelles se produisent, jeter l'échantillon et répéter la transfection. Relâchez les cellules dans un tube Eppendorf contenant 1 ml d'antibiotique sans DMEM contenant 10% de FCS. Notez chaque extrémité peut être réutilisé jusqu'à trois fois avec le même échantillon d'ARN si un plus grand nombre de cellules transfectées sont nécessaires.
- Par la suite, de distribuer les cellules du tube dans les puits contenant indépendants une quantité appropriée d'antibiotiques préchauffé DMEM sans contenant 10% de FCS. A titre d'indication générale, 150 pi de la suspension cellulaire généré lors de l'étape 1.2.8 sont suffisants pour un seul puits d'une plaque de 24 plat contenant 0,5 ml de DMEM préchauffé, tandis que 300 pl sont appropriés pour un puits d'une plaque 12-plat contenant 1 ml de DMEM préchauffé.
- Incuber les cellules à 37 ° C et 10% de CO 2 pendant 24 à 72 heures. Puis, libérer virions infectieux à partir de cellules par une (ou plusieurs)de geler et de dégel et de déterminer titre du virus dans l'échantillon en utilisant un essai sur plaque soit ou DICT50. Notez que lysats doivent être clarifiés par centrifugation pendant 1-2 minutes à vitesse maximale, soit par leur filtration à travers un filtre de 0,22 um pores avant le titrage. En règle générale, MNV atteint titres de l'ordre de 1 x 10 6 TCID50/ml moins 24 heures post-transfection et jusqu'à 1 x 10 9 à 72 heures post-transfection.
- La présence et la stabilité des mutations introduites dans pT7: 3'Rz MNV sont généralement déterminés par le séquençage des virus secourus au bout de 2 à 5 passages supplémentaires dans les cellules RAW264.7.
1.3 Récupération par lipofection en cellules BHK-21:
Une méthode plus directe et souvent plus rentable pour la récupération de MNV infectieux à partir de transcriptions plafonnés est par lipofection (Lipofectamine 2000, Invitrogen). Étant donné que les cellules RAW264.7 sont difficiles à transfecter en utilisant des approches basées sur des lipides que nous normalement faire usage de OTHfacile er pour transfecter des lignées cellulaires telles que BHK-21, qui est une ligne immortalisée dérivée à partir de fibroblastes de rein de hamster bébé. Comme une approche standard dans notre laboratoire, nous utilisons BSR-T7, un dérivé de cellules de la ligne de cellules BHK-21, comme tout ces cellules sont faciles à transfecter et de soutenir la réplication MNV, ils manquent un récepteur apte à permettre plusieurs cycles de réinfection . En conséquence, le rendement en virus généré à partir de ce système est une indication d'un seul cycle de réplication du virus. Cette approche est particulièrement utile lors de l'examen de l'effet de la mutation sur le recouvrement des virus, car il permet de multiples transfections à effectuer à des coûts considérablement réduits par rapport à Neon-transfection médiée et n'exige pas non plus l'équipement spécialisé. Il est à noter que d'autres lignées cellulaires facilement disponibles tels que embryonnaires humaines de rein cellules 293T également soutenir la reprise efficace des conditions de transfection MNV ne devrait toutefois d'abord être optimisé pour garantir l'efficacité de la prestation d'ARN.
- Trypsineise une monocouche de cellules BHK-21 (ou BSR-T7 cellules), les graines 7,5 x 10 5 cellules dans un plat de diamètre 35 mm dans les médias de croissance sans antibiotique et incuber les cellules à 37 ° C avec 10% de CO2 durant la nuit. Doublez la quantité de cellules dans chaque assiette, si les transfections sont prévues pour le même jour que l'ensemencement, et permettent aux cellules d'adhérer à la plaque pendant 2-3 heures à 37 ° C avec 10% de CO 2. Notez que d'autres cellules qui conviennent à cette approche comprennent des cellules 293T, l'homme du carcinome hépatocellulaire Huh7 cellules et de singe vert africain cellules COS7.
- Retirez le support à partir des cellules et les remplacer par 3 ml de milieu frais sans antibiotiques pour assurer le maximum d'efficacité de la transfection.
- Préparer un mélange de 1-2 pg de transcription MNV plafonné dans 100 ul d'Opti-MEM (Invitrogen) et le mélanger avec 4 pi de Lipofectamine 2000 préalablement mélangés dans 100 ul d'Opti-MEM. Mélanger soigneusement l'échantillon en le pipetant 15 fois. Quitterle mélange à température ambiante pendant 20 minutes.
- Ajouter les complexes de transfection contenant plafonnés transcriptions MNV dans un mode goutte à goutte à la monocouche de cellules et secouez doucement la plaque dans des directions perpendiculaires.
- Incuber les cellules à 37 ° C et 10% de CO 2 pendant 24 à 72 heures. Ensuite, relâchez virions infectieux à partir de cellules par le gel et le dégel et de déterminer le titre viral par dosage de plaque ou DICT50. Les rendements typiques de l'ordre de 1 x 10 6 TCID50/ml sont atteints.
2. Recouvrement direct des MNV infectieuses à partir d'ADNc dans les cellules exprimant la T7 RNA polymérase
Ce protocole est conçu pour permettre la récupération de MNV dans les cellules par la transcription d'un hébergement infectieuse plasmide séquence génomique complète d'ADNc par une polymérase T7 exprimée dans les cellules. Différentes lignées cellulaires peuvent être utilisés pour récupérer MNV infectieux par cette approche, bien que nous obtiennent généralement les meilleurs rendements avec BHK-21 et BSR-T7 CElls 15. Nous utilisons généralement BSR-T7 cellules car ils grandissent plus vite que la ligne BHK clone parental. Les cellules sont infectées par Fowlpox (FPV) codant pour l'ARN polymérase T7 (FPV-T7) 18 qui fonctionne comme un virus auxiliaire pour diriger l'expression de l'ARN viral et de récupération subséquente du virus infectieux (figure 4). Bien que BSR-T7 cellules expriment constitutivement la T7 RNA polymérase, cette expression n'est pas suffisant pour sauver MNV infectieux après transfection de pT7: MNV 3'Rz en l'absence de l'auxiliaire FPV-T7. Alors que les rendements typiques de ce système sont au moins 10 fois plus faible que ceux décrits ci-dessus, cette approche fournit une méthode rapide de mutants de dépistage afin de permettre l'identification de mutations débilitantes. Typiquement, cette méthode est utilisée en premier pour évaluer la viabilité d'une construction d'ADNc. Si une construction, soit ne parviennent pas à produire des virus infectieux ou semblent produire le virus à des niveaux inférieurs celle du clone infectieux de type sauvage, puis l'ARN basé sur approach décrit ci-dessus est entrepris.
- Trypsinise une monocouche de cellules BHK-21 (ou BSR-T7 cellules) et des semences de 7,5 x 10 5 cellules dans un plat de 35 mm dans des milieux de croissance antibiotiques libres et incuber les cellules à 37 ° C et 10% de CO 2 pendant une nuit. Ajouter le double de la quantité de cellules dans chaque assiette, si les transfections sont prévues pour le même jour que l'ensemencement, et permettent aux cellules d'adhérer à la plaque pendant 2-3 heures à 37 ° C et 10% de CO 2.
- Retirez le support de culture cellulaire et ajouter 700 ul de FPV-T7 à chaque puits (Figure 4). Une multiplicité d'infection (MOI) de ~ 0,5 UFP par cellule, sur la base de titrages sur des fibroblastes primaires d'embryons de poulet, est généralement utilisé. Toutefois, il est intéressant de noter que de nouvelles préparations de virus helper cultivé dans des fibroblastes primaires sont fonctionnellement titré pour déterminer la dose requise pour la récupération efficace du virus. Protocoles pour la propagation et le titrage des FPV-T7 ont été décrites précédemment 18.
- Danscubate à 37 ° C et 10% de CO 2 pendant 1 heure pour permettre FPV-T7 pour infecter les cellules. Ensuite, ajoutez 2 ml d'antibiotique sans DMEM contenant 10% de FCS et incuber les cellules pendant une heure supplémentaire à 37 ° C et 10% de CO 2 pour permettre l'expression d'ARN polymerase T7.
- Pour procéder à la transfection du plasmide infectieuse, d'une part éliminer les médias à partir des cellules infectées, se laver avec 2 ml de milieu (10% de FCS dans du DMEM sans antibiotiques), et enfin couvrir la monocouche de cellules avec 3 ml de milieu. Les antibiotiques ne doivent pas être ajouté aux médias, car ils peuvent interférer avec l'efficacité de Lipofectamine 2000 (Invitrogen).
- Préparer un mélange de 1 ug de type sauvage MNV ADNc infectieux plasmide (par exemple pT7: MNV 3'Rz) dans 100 ul d'Opti-MEM (Invitrogen) et mélanger avec 4 pi de Lipofectamine 2000 (Invitrogen) préalablement mélangés avec 100 pi de Opti-MEM (Invitrogen). Mélanger soigneusement la réaction en le pipetant 15 fois et de garder le mélange à température ambiante Temperature pendant 20 minutes.
- Le mélange de transfection obtenue doit être ensuite ajouté goutte à goutte à la monocouche de cellules et la plaque doit être agitée doucement dans des directions perpendiculaires.
- Incuber les personnes infectées FPV-T7, MNV plasmide cellules transfectées à 37 ° C et 10% de CO 2 de 24 à 72 heures. Les cellules transfectées avec le plasmide pT7 infectieuse: MNV 3'Rz normalement rendre des titres de 1 x 10 4 à 5 x 10 4 TCID50/ml.
3. Les résultats représentatifs
Les deux approches de génétique inverse sont très efficaces pour la récupération de MNV infectieuses en culture cellulaire, comme indiqué dans la figure 5. Infectieuses MNV avec des titres dépassant 10 5 TCID50/ml sont récupérés à 24 heures après transfection de l'assiette MNV ARN dans les cellules RAW264.7. De même, la transfection de plasmide pT7 infectieuse: MNV 3'Rz en BSR-T7 cellules préalablement infectées par le FPV exprimant aide T7 (FPV-T7) a conduit à des titres viraux largement dépasding 10 4 TCID50/ml (Figure 5). Ces valeurs titre viral obtenue avec de l'ARN de synthèse et de molécules d'ADN sont similaires à ceux obtenus dans des transfections impliquant naturelles VPg-linked ARN isolé à partir de virions infectieux dans les mêmes cellules (Figure 5). Ces résultats mettent en évidence la grande efficacité des approches de génétique inverse décrites ici pour récupérer génétiquement défini variantes MNV en culture cellulaire.
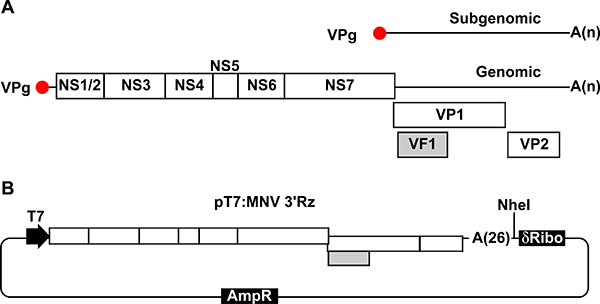
Figure 1. Illustration du génome MNV et le plasmide pour la récupération de virus infectieux. Une représentation, Schéma d'organisation du génome MNV. Chaque protéine région codante est représentée comme une seule boîte blanche. ORF1 est traduit en 7 différentes protéines non structurelles (NS1 / 2 à NS7) qui sont libérés à partir polyprotéine précurseur après auto-protéolytique de traitement. ORF 2 code pour la protéine majeure de capside VP1, ORF 3 codant pour le bouchon de mineureprotéines VP2 sid, et ORF4 chevauchement avec ORF2 région codante code VF1 facteur de virulence. ARN génomiques et subgénomiques contiennent une queue polyA à leurs extrémités 3 'de longueur variable. B, plasmide contenant l'ADNc MNV utilisé dans nos inverse des approches génétiques (pT7: MNV 3'Rz). ADNc NVM est fusionné à une queue polyA de 26 résidus dans son extrémité 3 '. La séquence d'ADNc NVM est situé immédiatement en aval d'une séquence promoteur T7 tronquée, pour permettre T7-entraînée de transcription, et en amont d'un Nhe I unique emplacement et une séquence d'ADN codant pour un ribozyme auto-clivage après. Ces séquences sont déterminants pour assurer la terminaison de transcription ARN juste après la présente queue polyA génomique à l'extrémité 3 '.
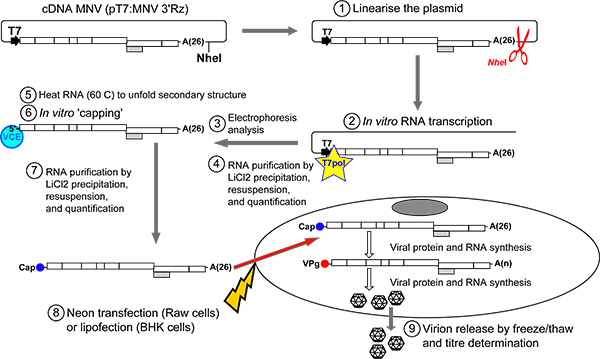
Figure 2. Vue d'ensemble du protocole pour la récupération des maladies infectieuses à partir d'ARN transcrits MNV et plafonné in vitro Le plasmide pT7:. MNV 3'Rz est linéarisé immédiatement en avalde la séquence génomique MNV en utilisant l'enzyme de restriction Nhe I (étape 1). Après purification de l'ADN, MNV transcrits d'ARN sont générés in vitro en utilisant l'ARN polymérase T7 (étape 2). Produits de transcription généralement exécuté avec une mobilité apparente de 2,5-3Ko sur une base non-dénaturant d'agarose 1% gel (étape 3, figure 3). La matrice d'ADN est éliminé en utilisant un commercial RNAse free DNAse. ARN est ensuite purifié à partir de nucléotides libres par LiCl précipitations (étape 4). Le produit purifié l'ARN peuvent alors être in vitro plafonné après avoir été préalablement chauffé à 65 ° C à déplier structures secondaires d'ARN (étapes 5-6). Après purification par précipitation de LiCl, l'ARN est transfecté dans des cellules (soit RAW264.7 système de transfection Neon, Invitrogen) ou BSR-T7 cellules (Lipofectamine 2000, Invitrogen) (étapes 7-8). Une fois à l'intérieur de la cellule, plafonnés transcrits d'ARN seront traduits en protéines virales qui catalysent la réplication virale transcriptions de nouvelles molécules d'ARN MNVcontenant une molécule VPg appropriée à leur extrémité 5 '. Les cycles successifs de réplication virale, accompagnés de la traduction serait de générer un grand nombre de génomes viraux qui sera encapsidé pour générer des virions infectieux. Pour faciliter la libération du virus à partir de cellules, une ou plusieurs cycles de gel et de dégel sont effectuées (étape 9). Rendements viraux peuvent être ensuite déterminée par DICT50 ou les procédures d'essai de plaque.
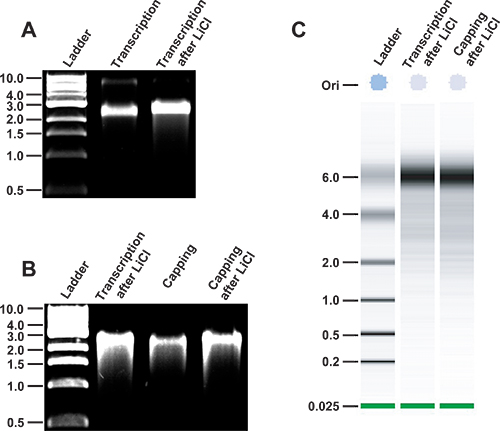
Figure 3. L'analyse des transcriptions d'ARN MNV intégrité tout au long du protocole. Une intégrité, de MNV ARN synthétisé in vitro. Le plasmide pT7: MNV 3'Rz est tout d'abord linéarisé en utilisant l'enzyme de restriction Nhe I. Après purification de l'ADN, MNV transcrits d'ARN sont générés in vitro en utilisant l'ARN polymérase T7 (piste 2). L'ARN est ensuite purification ed de nucléotides libres par LiCl précipitations (piste 3). Produits de transcription sont gérées sur une base non-gel dénaturant d'agarose 1% en parallèle à 1-Ko échelle d'ADN (New England Biolabs, piste 1). La mobilité relative des transcrits viraux sous des conditions non dénaturantes est similaire à un produit ADNdb de 2,5-3 Kb. B, l'intégrité de la transcription de l'ARN MNV après écrêtement. Transcriptions MNV purifiées précédemment par LiCl précipitations (piste 2) sont soumis au plafonnement enzymatique (piste 3) et la purification par précipitation de LiCl (piste 4). C,, de l'analyse dans un Agilent RNA 6000 Nano puce de transcriptions MNV (deuxième voie) et plafonnés transcriptions MNV (troisième voie), qui ont déjà été précipitées dans LiCl. Une échelle à ARN simple brin est exécuté en parallèle.
= "Pdflinebreak"> 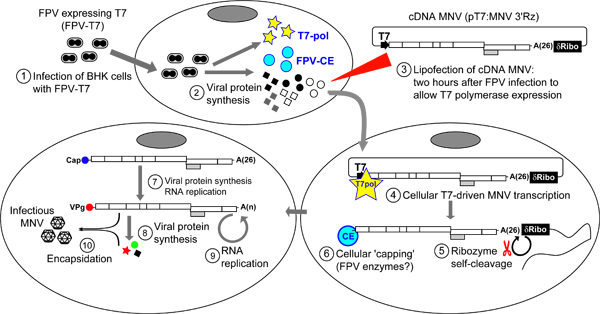
Figure 4. Vue d'ensemble du protocole pour la récupération de MNV infectieuses à partir d'ADNc. Initialement, BSR-T7 (ou BHK) sont infectés par un virus de variole aviaire recombinant (FPV) exprimant le bactériophage T7 RNA polymérase (FPV-T7) (étape 1). Les cellules infectées sont incubées pendant 2 heures avant le traitement en outre de permettre l'expression de protéines FPV recombinant qui comprend le T7 ARN polymérase (étape 2). Ensuite, pT7: MNV 3'Rz est transfecté dans les cellules en utilisant Lipofectamine 2000 (Invitrogen) (étape 3). Une fois à l'intérieur de la cellule, pT7: MNV 3'Rz est reconnu par l'ARN polymérase T7 qui synthétise MNV transcription de l'ARN (étape 4). La présence d'un auto-clivage δ-ribozyme séquence à l'extrémité 3 'du génome garantit 3 Transcription du' tErminus est située juste après la queue polyA (étape 5). Certains transcrits viraux sont plafonnés intracellulaire par une enzyme de coiffage FPV (étape 6). Les transcriptions résultant MNV plafonnés seront traduits pour générer des protéines qui catalysent MNV MNV réplication transcriptions. Nouvellement synthétisées MNV molécules d'ARN contenant une molécule VPg appropriée à leur extrémité 5 'serait soumis à des cycles successifs de réplication virale, accompagnés par une traduction qui peut finalement aboutir à la génération de virus encapsidé infectieux. Pour faciliter la libération du virus à partir de cellules, une ou plusieurs cycles de gel et de dégel sont effectuées (étape 7). Rendements viraux peuvent être ensuite déterminée par DICT50 ou les procédures d'essai de plaque.
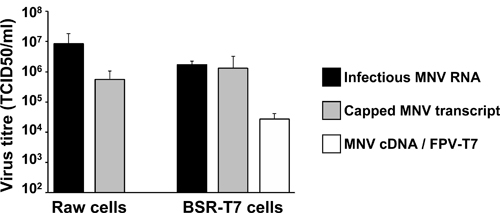
Figure 5. Les résultats représentatifs de titres de virus obtenus à partir de différentes approches de génétique inverse décrit dans le texte. Barres grises représentent les titres de virus obtenus à 24 heures après Neon-Transfection de 2 x 10 6 cellules RAW264.7 ou après lipo-transfection de 2 x 10 6 cellules BSR-T7 avec transcrit in vitro et d'ARN coiffée NVM. Les barres blanches représentent le titre du virus généralement obtenu après lipofection de pT7: MNV 3'Rz (MNV ADNc) en 2 x 10 6 BSR-T7 cellules déjà infectées pendant 2 heures avec le virus de la variole aviaire exprimant la T7 polymerase recombinante (FPV-T7). Comme un contrôle positif pour la transfection dans Raw et BSR-T7cells, nous utilisons généralement 2 ug d'ARN extrait de cellules infectées par le MNV qui contiennent des niveaux élevés de VPg liée MNV ARN. Les contrôles négatifs ont été réalisées soit avec MNV ARN ou pT7: MNV 3'Rz codant pour une mutation du cadre de lecture (F / S) qui abroge la réplication, ce qui entraîne pas de virus détectable (données non présentées).
Discussion
Ici, nous avons illustré deux différentes approches génétiques inverse qui permettent la récupération de MNV infectieuses en culture cellulaire. Les deux approches de manière efficace de contourner l'exigence absolue pour la liaison covalente de VPg à l'extrémité 5 'du génome ARN viral via la génération de transcrits MNV plafonnés, qui sont ensuite reconnus par les ribosomes cellulaires. La transcription in vitro suivie par voie enzymatique de coiffage est plus efficace dans la récupération des NVM infectieuse de la transcription de plasmides infectieuses dans des cellules exprimant l'ARN polymérase T7, dans lequel les transcrits peut être coiffé par les enzymes FPV coiffage. Titres de virus récupérés avec ces systèmes de génétique inverse sont semblables à celles obtenues par transfection de VPg virales liées ARN purifiés à partir de cultures de cellules infectées 17 ans (figure 5). La transfection de l'assiette MNV ARN dans les cellules permissives RAW264.7 rend un titre seul virus de 1 log inférieur expériences impliquant l'transfection de l'ARN total à partir de cellules infectées par le virus contenant des VPg liée ARN (Figure 5). Ce fait encourage de nouvelles investigations pour déterminer si l'ajout d'une molécule VPg à l'extrémité 5 'des transcrits générés par ces systèmes pourrait entraîner des rendements de virus a augmenté ce qui peut révéler des aspects fonctionnels qui sous-tendent l'infectiosité MNV dans les cellules associées à VPg. Néanmoins, nous considérons que ce système de génétique inverse comme un très efficace comparable à d'autres virus à ARN inverser systèmes de génétique actuellement utilisé dans laquelle le transcrit in vitro d'ARN permet la récupération de titres seulement 10-100 inférieurs à ceux des infections réelles avec des virions 19, 20.
Dans l'ensemble, les méthodologies actuelles constituent une avancée significative dans le domaine de la biologie moléculaire et norovirus nous fournir les outils nécessaires pour enquêter sur les rôles fonctionnels des protéines et des motifs conservés dans les génomes ARN norovirus. Ces approches ont déjà été combiné avec cmodèle de souris ACTUEL disponibles et ont montré que MNV récupéré à partir d'ADNc infectieux est en mesure de provoquer une infection mortelle de> 80% STAT1-/ - souris en moins de 10 jours 4, 21. Faisant usage de ce système, nous avons récupéré viables mutants norovirus murin de la protéine de capside et d'un tube polypyrimidine impliqué dans la liaison des facteurs de l'hôte différents (PTB et PCBP) qui affichent un peu atténués phénotypes in vivo 21, 22. En outre, nous avons récemment démontré que les virus qui n'ont pas la capacité d'exprimer la protéine VF1 de ORF4 efficacement répliquer en culture cellulaire, mais encore une fois ont réduit la virulence chez la souris par rapport à WT MNV 8. Ces études nous incitent à concevoir des versions atténuées de norovirus humains fondées sur des études MNV qui pourraient être étudiés en tant que candidats vaccins potentiels.
Déclarations de divulgation
Nous n'avons rien à communiquer.
Remerciements
Cette recherche a été financée par une bourse du Wellcome Trust principal décerné à Ian Goodfellow, et une Marie Curie intra European Fellowship (7e PC European Research Council) a décerné à Armando Arias. Nous tenons à remercier Io Hong Cheong, le Dr Rebecca Robey et le Dr Mike Skinner de nous donner la permission d'utiliser leur Bioanalyser Agilent et en aidant à l'exécution des exemples d'ARN.
matériels
| Name | Company | Catalog Number | Comments |
| Nom du réactif | Entreprise | Numéro de catalogue | |
| Solution de précipitation au chlorure de lithium | Ambion | AM9480 | |
| Solution de stockage d'ARN | Ambion | AM7000 | |
| MEGAscript T7 de script | Ambion | AM1333 | |
| ScriptCap M7G Plafonnement du système | Biotechnologies Epicentre | SCCE0610 | |
| Système de transfection néon | Invitrogen | MPK5000 | |
| Neon kit système de transfection | Invitrogen | MPK1025 | |
| Opti-MEM I | Invitrogen | 31985070 | |
| Réactif de transfection Lipofectamine 2000 | Invitrogen | 11668-027 | |
| Agilent ARN 600 Nano kit | Agilent | 5067-1511 | |
| Agilent 2100 bioanalyzer | Agilent | G2939AA | |
| Illustra GFX PCR de l'ADN et Kit Gel purification Band | GE Healthcare | 28-9034-70 | |
| RiboMAX production à grande échelle du système ARN-T7 | Promega | P1300 |
Références
- . Outbreaks of gastroenteritis associated with noroviruses on cruise ships--United States. MMWR Morb. Mortal. Wkly. Rep. 51, 1112-1115 (2002).
- Lopman, B. A. Epidemiology and cost of nosocomial gastroenteritis, Avon, England, 2002-2003. Emerg. Infect. Dis. 10, 2002-2003 (2004).
- Duizer, E. Laboratory efforts to cultivate noroviruses. J. Gen. Virol. 85, 79-87 (2004).
- Karst, S. M., Wobus, C. E., Lay, M., Davidson, J., Virgin, H. W. T. STAT1-dependent innate immunity to a Norwalk-like virus. Science. 299, 1575-1575 (2003).
- Wobus, C. E. Replication of Norovirus in cell culture reveals a tropism for dendritic cells and macrophages. PLoS Biol. 2, e432 (2004).
- Bok, K. Inhibition of norovirus replication by morpholino oligomers targeting the 5'-end of the genome. Virology. 380, 328-337 (2008).
- Kim, Y., Thapa, M., Hua, D. H., Chang, K. O. Biodegradable nanogels for oral delivery of interferon for norovirus infection. Antiviral Res. 89, 165-173 (2011).
- McFadden, N. Norovirus Regulation of the Innate Immune Response and Apoptosis Occurs via the Product of the Alternative Open Reading Frame 4. PLoS Pathog. 7, e1002413 (2011).
- Kormelink, R., van Poelwijk, F., Peters, D., Goldbach, R. Non-viral heterogeneous sequences at the 5' ends of tomato spotted wilt virus mRNAs. J. Gen. Virol. 73, 2125-2128 (1992).
- Shuman, S., Hurwitz, J. Mechanism of mRNA capping by vaccinia virus guanylyltransferase: characterization of an enzyme--guanylate intermediate. Proc. Natl. Acad. Sci. U.S.A. 78, 187-191 (1981).
- Jang, S. K., Pestova, T. V., Hellen, C. U., Witherell, G. W., Wimmer, E. Cap-independent translation of picornavirus RNAs: structure and function of the internal ribosomal entry site. Enzyme. 44, 292-309 (1990).
- Chaudhry, Y. Caliciviruses differ in their functional requirements for eIF4F components. J. Biol. Chem. 281, 25315-25325 (2006).
- Goodfellow, I. Calicivirus translation initiation requires an interaction between VPg and eIF 4. E. EMBO Rep. 6, 968-972 (2005).
- Herbert, T. P., Brierley, I., Brown, T. D. Identification of a protein linked to the genomic and subgenomic mRNAs of feline calicivirus and its role in translation. J. Gen. Virol. 78 ( Pt5 ), 1033-1040 (1997).
- Chaudhry, Y., Skinner, M. A., Goodfellow, I. G. Recovery of genetically defined murine norovirus in tissue culture by using a fowlpox virus expressing T7 RNA polymerase. J. Gen. Virol. 88, 2091-20100 (2007).
- Ward, V. K. Recovery of infectious murine norovirus using pol II-driven expression of full-length cDNA. Proc. Natl. Acad. Sci. U.S.A. 104, 11050-11055 (2007).
- Yunus, M. A., Chung, L. M., Chaudhry, Y., Bailey, D., Goodfellow, I. Development of an optimized RNA-based murine norovirus reverse genetics system. J. Virol. Methods. 169, 112-118 (2010).
- Arias, A., Perales, C., Escarmis, C., Domingo, E. Deletion mutants of VPg reveal new cytopathology determinants in a picornavirus. PLoS One. 5, e10735 (2010).
- Werf, S. v. a. n. d. e. r., Bradley, J., Wimmer, E., Studier, F. W., Dunn, J. J. Synthesis of infectious poliovirus RNA by purified T7 RNA polymerase. Proc. Natl. Acad. Sci. U.S.A. 83, 2330-2334 (1986).
- Bailey, D., Thackray, L. B., Goodfellow, I. G. A single amino acid substitution in the murine norovirus capsid protein is sufficient for attenuation in vivo. J. Virol. 82, 7725-7728 (2008).
- Bailey, D. Functional analysis of RNA structures present at the 3' extremity of the murine norovirus genome: the variable polypyrimidine tract plays a role in viral virulence. J. Virol. 84, 2859-2870 (2010).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon