Method Article
Recuperação de reversão genética mediada por norovírus Murina Infecciosas
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Os norovírus são a principal causa de gastroenterite ainda técnicas moleculares para sua caracterização são ainda relativamente novo. Aqui relatamos duas diferentes abordagens genética reversa para a recuperação eficiente de norovírus murino (MNV), o único membro do gênero que podem ser propagadas em cultura de células.
Resumo
Norovírus humanos são responsáveis pela maioria dos casos de gastrenterite humana (GE) em todo o mundo e são um problema recorrente em ambientes onde o contato pessoa-a-corpo não podem ser evitados 1, 2. Durante os últimos anos um aumento na incidência de focos nos hospitais tem sido relatada, causar perturbações significativas para a sua capacidade operacional, bem como grandes perdas económicas. A identificação de novas abordagens antivirais tem sido limitada devido à incapacidade de norovírus humanos para completar uma infecção produtiva em cultura de células 3. O isolamento recente de um norovírus murino (MNV), estreitamente relacionada com norovírus humano 4, mas que pode ser propagado em células 5 abriu novas vias para a investigação de agentes patogénicos estes 6, 7.
Resultados de replicação MNV na síntese de genómico novo sentido positivo e moléculas de RNA subgenómico, o último dos quais corresponde ao thi últimaestr do genoma viral (Figura 1). MNV contém quatro diferentes estruturas de leitura abertas (ORFs), dos quais ORF1 ocupa a maior parte do genoma e codifica sete proteínas não estruturais (NS1-7) libertados por um precursor de poliproteínas. ORF2 e ORF3 estão contidos dentro da região de RNA subgenómico e codificar as proteínas da cápside (VP1 e VP2, respectivamente) (Figura 1). Recentemente, identificou-se que ORF4 adicional sobreposição ORF2 mas de uma grelha de leitura diferente é funcional e codifica para um factor de virulência mitocondrial localizada (VF1) 8.
A replicação de vírus de sentido positivo de RNA, incluindo norovírus, tem lugar no citoplasma resultando na síntese de novos genomas não niveladas de RNA. Para promover a tradução viral, vírus explorar diferentes estratégias destinadas a recrutar a maquinaria de síntese protéica celular 9-11. Curiosamente, a tradução norovírus é impulsionada pelo multifuncional VPG proteína iniciador-viralcovalentemente ligada à extremidade 5 'de ambos os RNAs genómicas e subgenómico 12-14. Este sofisticado mecanismo de tradução é provável que seja um fator importante na eficiência limitada de recuperação viral por convencionais genética reversa.
Aqui relatamos duas estratégias diferentes com base na geração de murino norovírus-1 (MNV referido como anexo) transcrições seladas na extremidade 5 '. Um dos métodos envolve tanto a síntese in vitro e capping de RNA viral, enquanto que a segunda abordagem implica a transcrição de MNV ADNc em células que expressam-polimerase de T7 RNA. A disponibilidade destes sistemas de genética inversa para o estudo da MNV e um modelo animal pequeno forneceu uma capacidade sem precedentes para dissecar o papel de sequências virais em replicação e patogénese 15-17.
Protocolo
1. RNA Transcrição e nivelamento de Recuperação de MNV Infecciosas
Este protocolo é projetado para permitir a recuperação eficiente de MNV infeccioso de cDNA através de transcrição in vitro e sua posterior in vitro de cobertura (seção 1.1). As transcrições resultantes são então cobertas transfectado em células de recuperar MNV infecciosa (secções 1.2 e 1.3). Esta abordagem proporciona o método mais sensível para a recuperação de MNV com rendimentos típicos em excesso de 10 5 unidades infecciosas por 35 mm (de diâmetro)-prato de células para MNV. O protocolo é detalhado abaixo:
Síntese de 1,1 infecciosas transcrições MNV cobertas:
- Digerir o plasmídeo contendo o cDNA do tipo selvagem MNV (pT7: MNV 3'Rz) com Nhe I para obter ADN linear Nhe I reconhece um local de restrição único após a extremidade 3 'poliA cauda de MNV genoma (Figura 2).. Plasmídeos linearizados são tipicamente purificadascom o uso de colunas de sílica (por exemplo, GFX PCR Gel DNA Kit de Purificação da faixa da GE Healthcare) e eluída em H 2 O.
- In vitro transcrever o vector linearizado usando polimerase de ARN T7, como descrito anteriormente 17. Muitos kits comerciais estão disponíveis para este fim e fornecer um método reprodutível de grandes quantidades de ARN de síntese tais como MEGAScript (Life Technologies) e RiboMAX (Promega). As reacções de transcrição são tipicamente DNAse digerido antes de posterior análise, no entanto em muitos casos, isso não é necessário como purificações de cloreto de lítio, como descrito a seguir não precipitar o DNA de forma eficiente.
- Analise uma pequena alíquota da reacção de transcrição de ARN, tipicamente 0,5 uL ou menos, por electroforese em gel de agarose para assegurar a reacção de transcrição trabalhou de forma eficiente e RNA de comprimento completo. Embora muitos usuários podem querer executar géis corretamente o tamanho do RNA, que normalmente usam não desnaturante em gel de agarose electrophoresis como um método rápido para analisar a integridade do RNA. O genoma MNV como produzido a partir do clone de cDNA infeccioso pT7: MNV 3'Rz será executado em aproximadamente 3 Kbp relativa a uma escada dsDNA em um não desnaturante em gel de agarose (Figura 3).
- Considere-se que outros métodos estão disponíveis como uma alternativa para a obtenção de uma análise rápida de integridade de RNA tais como a utilização de um Bioanalyzer Agilent (Figura 3).
- Observe que a resolução gel pobre pode ser encontrado se RNA demais é carregado. Tomar cuidado para assegurar que o gel de agarose é preparado utilizando RNase reagentes para evitar a degradação de RNA durante a electroforese que pode afectar a resolução banda. Aquecimento de RNA para 65 ° C seguido de arrefecimento em gelo pode também ajudar em alguns casos.
- Purifica-se amostra de RNA para remover os nucleótidos não incorporados. Muitos métodos estão disponíveis para este abordagens de sílica, incluindo coluna com base no entanto, neste protocolo que geralmente usam cloreto de lítio como uma alternativa rentável. Paraesta finalidade, adicionar H2O para alcançar um volume final de 100 uL e depois adicionar 40 ul de solução de precipitação com cloreto de lítio (7,5 M LiCl, 50 mM de EDTA, pH 8,0, Ambion) e armazenar a amostra à temperatura de -20 ° C durante no pelo menos 30 min.
- Pellet do RNA por centrifugação a 12.000 x G a 4 ° C durante 15 min.
- Retire o sobrenadante, tomando cuidado para não perturbar o pellet de RNA translúcido e lavá-lo em 150 ul de etanol 70%. Centrifugar o tubo a 12.000 x G a 4 ° C durante 15 min.
- Remover o etanol e ar seco o RNA, evitando o sedimento para secar completamente para fora como isto fará com que a ressuspensão difícil.
- Em seguida, ressuspender as transcrições MNV em 50-100 ul de RNA solução de armazenamento (Ambion). Cuidados devem ser tomados para garantir que todos os RNA se dissolveu corretamente. No caso de os ARN parecem ser difíceis de dissolver completamente, aquecendo a amostra a 60 ° C pode ajudar a ressuspender-lo. Qualquer material insolúvel deve então ser removido by centrifugação antes para o RNA quantificação. As transcrições purificadas são estreantes e requerem uma posterior in vitro etapa de nivelamento para ser infecciosa (ScriptCap Sistema M7G nivelamento, Biotecnologias Epicentre).
- Quantificar o RNA por espectrofotometria. Dependendo da natureza e da escala da reacção de transcrição, os rendimentos típicos variam de 50-150 ug de RNA por 100 uL reacção de transcrição. A integridade de RNA deve ser analisado antes da reacção de capping por esgotar-se 100-300 ng de amostra num gel de agarose a 1% (Figura 3).
- Para melhorar a eficiência de RNA nivelamento, ug de calor 60-70 de transcritos de RNA MNV a 65 ° C durante 10 min e depois colocar o tubo imediatamente em gelo. Este passo pode reduzir qualquer efeito inibitório da estrutura do RNA no nivelamento. Pulsar o tubo numa microcentrífuga refrigerada para recolher gotas formadas durante o passo de aquecimento.
- Prepara-se uma mistura de reacção capping como sugerido pelo fabricante (ScriptCap M7GSistema de nivelamento, Biotecnologias Epicentre). Resumidamente, adicionar 60-70 ug de RNA MNV para um volume de reacção final de 100 uL. A mistura de reacção capping pode conter 10 uL de 10 x limitação de tampão (500 mM TrisHCl, pH 8,0, 60 mM de KCl, 12,5 mM de MgCl2), 10 uL de 10 mM GTP, 0,5 ul de 20 uL mM S-adenosil-metionina, 2,5 de Scriptguard (100 unidades), e 4 uL de enzima Scriptcap (40 unidades).
- Durante a reação configurar, manter RNA transcritos no gelo para evitar a degradação. Misturar bem a mistura de reacção e, em seguida, ela incubar a 37 ° C durante 1 h. Observe o tamanho reacção pode ser dimensionada de acordo com a quantidade de transcrição capped necessário.
- Purificar o RNA por precipitação LiCl como explicado acima (ver ponto 1.1.6). Dissolve-se o pellet em 50-100 ul de RNA solução de armazenamento (Ambion) e quantificar a quantidade de RNA. Tipicamente, as amostras de RNA são subsequentemente normalizado para 1 ug / uL. Novamente, verifique toda a RNA foi dissolvido corretamente. Se não foi adequadamente dissolvido, o calor tele amostra a 60 ° C para permitir a sua dissolução. Remover por centrifugação qualquer material insolúvel antes da RNA quantificação.
- Verifique a integridade do RNA novamente antes de prosseguir com a etapa de transfecção. Para este objectivo, executar uma quantidade de 100-300 ng de amostra num gel de agarose a 1% (Figura 3).
1,2 Recuperação por néon transfecção mediada de RNA em Raw264.7 células:
Para a recuperação de viriões infecciosos em MNV uma linha celular permissiva é possível electroporate a transcrição capped MNV em Raw264.7 células usando o sistema de transfecção de néon (Invitrogen). Raw264.7 são células susceptíveis a infecção MNV, suportando múltiplos ciclos de replicação de vírus e subsequente re-infecção. Como resultado, os rendimentos típicos se aproximará em excesso de 10 5 unidades infecciosas por ml a 24 horas pós transfecção mas pico em mais de 10 7 unidades infecciosas após 48 horas.
- Um dia antes de transemente sfection, Raw264.7 células em uma estimativa de 50% de confluência. Tipicamente dois frascos T75 de células são necessárias para 3 transfecções.
- O dia da transfecção, raspar as monocamadas de células em Dulbecco modificado por Eagle (DMEM) contendo 10% de soro fetal de vitelo (FCS), assegurando-lhe gerar uma única suspensão celular por pipetagem repetida.
- Determinar a concentração de células viáveis em um hemocitómetro usando exclusão de azul de tripano para rotular as células não viáveis.
- Sedimentar as células a 1.200 X g durante 5 min e ressuspender-los em DMEM contendo FCS a 10% a uma concentração final de 8 x 10 6 células / ml.
- Pouco antes da transfecção, 1 ml alíquota de células por transfecção e pellet-los em 1.200 X g durante 2 min. Remover os meios de comunicação e lavar as células em 500 uL de PBS (sem + / Ca 2 + Mg 2). Girar para baixo as células novamente em 1.200 X g durante 2 min. Note que é aconselhável manter as células em DMEM por tanto tempo quanto possívelcomo armazenamento em PBS durante longos períodos de tempo pode comprometer a viabilidade das células ea taxa de transfecção.
- Remover PBS a partir de tubos e adicionar 130 uL de solução de ressuspensão (kit de sistema de néon transfecção, Invitrogen) para uma concentração final de 6 x 10 7 células / ml. Deve ser tomado cuidado para ressuspender as células, evitando a formação de bolhas que irá causar faíscas durante a transfecção ea sobrevivência das células de compromisso.
- Adicionar a quantidade apropriada de transcrição MNV capped para as células (Figura 2), geralmente ug de RNA 1,3 capped é adicionado a 130 ul de células ressuspensas e misturados suavemente. Em seguida, recolher 100 uL da mistura de transfecção na ponta 100 uL de néon. Especial cuidado deve ser tomado para assegurar que não existem bolhas são formadas na cubeta de eletroporação (kit sistema Neon transfecção 100 ul ponta), pois isso causará a falha do experimento.
- Electroporate as células utilizando um único pulso 1700 V durante 25 mseg, ensuring a ausência de faíscas durante pulsando que irá indicar a presença de bolhas na amostra. No caso faíscas deve ocorrer, descarte da amostra e repetir a transfecção. Libertar as células em um tubo de Eppendorf contendo 1 ml de antibiótico livre DMEM contendo FCS a 10%. Observe cada ponta pode ser reutilizado até três vezes com a amostra de RNA mesmo se grandes números de células transfectadas são necessários.
- Posteriormente, distribuir as células do tubo em poços independentes contendo uma quantidade apropriada de pré-aquecido antibiótico livre DMEM contendo FCS a 10%. Como orientação geral, 150 uL da suspensão de células gerado durante o passo 1.2.8 são suficientes para um único poço de uma placa de 24-prato contendo 0,5 ml de DMEM pré-aquecido, enquanto que 300 uL são apropriados para um poço de uma placa de 12-prato contendo 1 ml de DMEM pré-aquecido.
- Incubar as células a 37 ° C e 10% de CO2 durante 24 a 72 horas. Em seguida, solte viriões infecciosos a partir de células por um (ou mais)congelar e descongelação e determinar título de vírus na amostra usando um ensaio de placa ou TCID50. Note-se que os lisados deve ser clarificada por centrifugação durante 1-2 minutos à velocidade máxima ou pela sua filtração através de um filtro de poro de 0,22 um antes da titulação. Tipicamente, MNV atinge títulos de cerca de 1 x 10 6 TCID50/ml menos 24 horas após a transfecção, e até 1 x 10 9, 72 horas após a transfecção.
- A presença e estabilidade de mutações introduzidas na pT7: 3'Rz MNV são tipicamente determinada por sequenciação dos vírus recuperados após 2 a 5 passagens adicionais em Raw264.7 células.
1.3 Recuperação por lipofecção em células BHK-21:
Um método mais directo e muitas vezes mais rentável para a recuperação de MNV infeccioso a partir de transcritos de casquilho é através de lipofecção (Lipofectamine 2000, Invitrogen). Dado que Raw264.7 células são difíceis para transfectar utilizando abordagens baseadas lipídicas que normalmente fazem uso de OTHer fácil para transfectar as linhas celulares, tais como BHK-21 que é uma linha imortalizada derivado de fibroblastos de rim de hamster bebé. Como uma abordagem padrão no nosso laboratório usamos BSR-T7 células, um derivado da linha de células BHK-21, tal como enquanto estas células são fáceis para transfectar e suportar a replicação MNV, falta-lhes um receptor adequado para permitir que vários ciclos de re-infecção . Como resultado, o rendimento do vírus gerado a partir deste sistema é uma indicação de um único ciclo de replicação do vírus. Esta abordagem é de particular utilidade quando se examina o efeito da mutação sobre a recuperação do vírus uma vez que permite transfecções múltiplas para ser realizada a custos substancialmente reduzidos em comparação com néon transfecção mediada e também não requer equipamento especializado. É importante notar que outras linhagens de células prontamente disponíveis, tais como rim embrionário humano 293T também apoiar a recuperação eficiente de condições MNV porém transfecção deve primeiro ser otimizados para garantir a entrega RNA eficiente.
- Tripsinaise uma monocamada de células BHK-21 (ou BSR-T7 células), sementes de 7,5 x 10 5 células em um prato de diâmetro 35 mm de meios de comunicação sem antibiótico de crescimento e incubam as células a 37 ° C, com 10% de CO 2 durante a noite. O dobro da quantidade de células em cada placa, se as transfecções estão previstas para o mesmo dia que a semeadura, e permitir que as células adiram à placa durante 2-3 horas a 37 ° C, com 10% de CO 2. Note-se que outras células que são adequados para esta abordagem incluem células 293T, humanos carcinoma hepatocelular Huh7 células e macaco verde Africano Cos7 células.
- Remova a mídia a partir das células e substituir com 3 ml de meio fresco sem antibióticos para garantir a máxima eficiência de transfecção.
- Prepara-se uma mistura de 1-2 ug de transcrição MNV capped em 100 uL de Opti-MEM (Invitrogen) e misturá-lo com 4 ul de Lipofectamine 2000 previamente misturados em 100 uL de Opti-MEM. Misturar a amostra completamente por pipetagem para cima e para baixo de 15 vezes. Deixara mistura à temperatura ambiente durante 20 minutos.
- Adicionar os complexos de transfecção contendo transcrições cobertas MNV de forma gota a gota, a monocamada de células e agitar suavemente a placa em direções perpendiculares.
- Incubar as células a 37 ° C e 10% de CO2 durante 24 a 72 horas. Depois, libertar viriões infecciosos a partir de células por congelamento e descongelamento e determinar título de vírus por ensaio de placa ou TCID50. Rendimentos típicos de cerca de 1 x 10 6 TCID50/ml são atingidos.
2. Recuperação directa de MNV Infecciosa a partir de ADNc em células que expressam polimerase de ARN T7
Este protocolo é concebido para permitir a recuperação de MNV em células através da transcrição de um guarida infecciosa plasmídeo a sequência genómica completa de ADNc por uma polimerase de T7 expressa nas células. Linhas de células diferentes podem ser usados para recuperar MNV infecciosa por esta abordagem embora tipicamente obter os maiores rendimentos com BHK-21 e BSR-T7 cells 15. Normalmente usamos BSR-T7 células, uma vez que crescem mais rapidamente do que a linha parental clone BHK. As células são infectadas com bouba codificação (FPV) para polimerase de ARN T7 (FPV-T7) 18 que funciona como um vírus auxiliar para dirigir a expressão do RNA viral e subsequente recuperação do vírus infeccioso (Figura 4). Embora BSR-T7 células constitutivamente expresso polimerase de ARN T7, esta expressão não é suficiente para resgatar MNV infeccioso após transfecção de pT7: MNV 3'Rz na ausência de auxiliar FPV-T7. Embora os rendimentos típicos deste sistema são, pelo menos, 10 vezes mais baixa do que os descritos acima, esta abordagem faz fornecer um método rápido de mutantes de rastreio para permitir a identificação de mutações debilitantes. Tipicamente este método é utilizado em primeiro lugar para avaliar a viabilidade de uma construção de cDNA. No caso de uma construção ou falham para produzir vírus infecciosos ou parecem produzir vírus em níveis mais baixos do que a do clone de tipo selvagem infecciosa, em seguida, o RNA baseado approach descrito acima é realizado.
- Trypsinise uma monocamada de células BHK-21 (ou BSR-T7 células) e sementes de 7,5 x 10 5 células num prato de 35 mm em meio de crescimento de antibióticos livres e incubam as células a 37 ° C e 10% de CO 2 durante a noite. Adicionar o dobro da quantidade de células em cada placa, se as transfecções estão previstas para o mesmo dia que a semeadura, e permitir que as células adiram à placa durante 2-3 horas a 37 ° C e 10% de CO 2.
- Remover os meios de cultura de células e adicionar 700 uL de FPV-T7 a cada poço (Figura 4). Uma multiplicidade de infecção (MOI) de ~ célula PFU 0,5 por, com base em titulações de fibroblastos de embrião de primárias de galinha, é geralmente utilizado. No entanto, é importante notar que as preparações de novos de vírus auxiliar crescido em fibroblastos primários são funcionalmente titulada para determinar a dose necessária para a recuperação de vírus eficiente. Os protocolos para a propagação e titulação de FPV-T7 foram previamente descritos 18.
- EmCubate a 37 ° C e 10% de CO2 durante 1 hora para permitir que FPV-T7 para infectar as células. Em seguida, adicionar 2 ml de antibiótico livre DMEM contendo FCS a 10% e incubar as células durante uma hora adicional a 37 ° C e 10% de CO 2 para permitir a expressão de T7 RNA polimerase.
- Para prosseguir com a transfecção do plasmídeo infecciosa, em primeiro lugar remover os meios de comunicação a partir das células infectadas, lava-se com 2 ml de meio (FCS 10% em DMEM livre de antibiótico) e, finalmente, cobrir a monocamada de células com 3 ml de meio. Os antibióticos não devem ser adicionados aos meios de comunicação, uma vez que pode interferir com a eficácia de Lipofectamine 2000 (Invitrogen).
- Prepara-se uma mistura de 1 ug de tipo selvagem MNV cDNA infeccioso plasmídeo (por exemplo, pT7: MNV 3'Rz) em 100 uL de Opti-MEM (Invitrogen) e mistura-lo com 4 ul de Lipofectamine 2000 (Invitrogen), previamente misturada com 100 ul de Opti-MEM (Invitrogen). Misture a reação completamente pipetando cima e para baixo 15 vezes e manter a mistura em sala de temperatura durante 20 minutos.
- A mistura de transfecção resultante deve ser então adicionado gota a gota, à monocamada de células e da placa deve ser agitada suavemente em direcções perpendiculares.
- Incubar a infectada FPV-T7, MNV plasmídeo-transfectadas células a 37 ° C e 10% de CO2 durante 24 a 72 horas. As células transfectadas com o plasmídeo pT7 infecciosa: MNV 3'Rz normalmente tornar títulos de 1 x 4 a 5 x 10 10 4 TCID50/ml.
3. Os resultados representativos
Ambas as abordagens de genética inversa são altamente eficientes para a recuperação de MNV infecciosa em cultura de células, como mostrado na Figura 5. MNV infeccioso com títulos superiores a 10 TCID50/ml 5 são recuperados durante 24 horas após a transfecção de capped MNV RNA em Raw264.7 células. Do mesmo modo, a transfecção de plasmídeo pT7 infecciosa: MNV 3'Rz em BSR-T7 células previamente infectadas com o auxiliar de FPV expressando T7 (FPV-T7), levou à títulos virais em grande parte exceeDing 10 4 TCID50/ml (Figura 5). Estes valores de titulação virais obtidos com RNA sintéticas e moléculas de DNA são semelhantes aos obtidos no transfecções envolvendo naturais VPG-linked RNA isolado a partir de viriões infecciosos nas mesmas células (Figura 5). Esses resultados destacam a alta eficiência dos genética reversa descritos aqui para recuperar geneticamente definido variantes MNV em cultura de células.
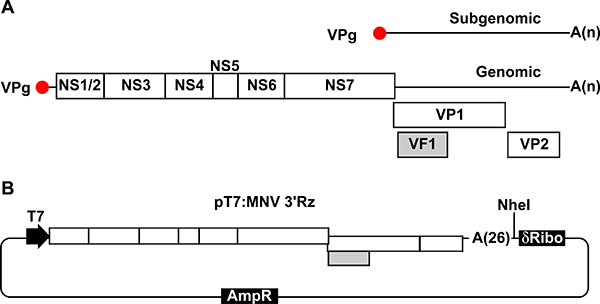
Figura 1. Ilustração de MNV genoma e plasmídeo para a recuperação de vírus infeccioso. Uma representação, esquemático de MNV organização do genoma. Cada proteína região codificante está ilustrado como uma única caixa branca. ORF1 é traduzida em 7 diferentes proteínas não-estruturais (NS1 / 2 a NS7) que são libertados a partir de poliproteína precursora após auto-proteolítica de processamento. ORF 2 codifica a proteína da cápside grande VP1, ORF 3 codifica a tampa menorsid proteína VP2, e ORF4 sobreposição com ORF2 codifica região codificadora fator de virulência VF1. RNAs genómicas e subgenómico conter uma cauda poliA nas suas extremidades 3 'de comprimento variável. B, plasmídeo contendo ADNc MNV utilizado nos nossos reversa abordagens genéticas (pT7: MNV 3'Rz). MNV ADNc está fundido com uma cauda poliA de 26 resíduos em sua extremidade 3 '. A sequência de cDNA MNV está localizado imediatamente a jusante de uma sequência truncada do promotor T7, para permitir que T7-driven, transcrição e a montante de um único Nhe I local e uma sequência de ADN que codifica para um ribozima auto-clivagem depois dela. Estas sequências são decisivos para assegurar a terminação da transcrição de RNA logo após a presente genómico cauda poliA à extremidade 3 '.
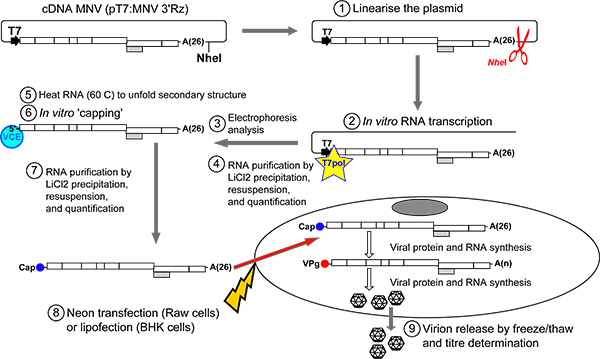
Figura 2. Visão geral do protocolo para a recuperação de MNV infeccioso a partir de RNA transcrito e tampado in vitro O plasmídeo pT7:. MNV 3'Rz é linearizado imediatamente a jusanteda sequência genómica MNV utilizando a enzima de restrição Nhe I (passo 1). Depois de purificação de ADN, os transcritos de RNA MNV são gerados in vitro, utilizando polimerase de ARN T7 (passo 2). Produtos de transcrição geralmente executado com uma mobilidade aparente de 2,5-3Kb em um não desnaturante 1% em gel de agarose (passo 3, a Figura 3). O DNA molde é eliminado usando um comercial RNAse livre de DNAse. RNA é então purificado a partir de nucleótidos livres por precipitação com LiCl (passo 4). O produto purificado pode então ser RNA in vitro após ser limitado previamente aquecida a 65 ° C para se desdobrar estruturas secundárias de RNA (passos 5-6). Após purificação por precipitação com LiCl, o ARN é transfectado em células quer Raw264.7 (néon sistema de transfecção, Invitrogen) ou BSR-T7 (células Lipofectamine 2000, Invitrogen) (passos 7-8). Uma vez dentro da célula, transcrições de RNA cobertas será traduzido em proteínas virais que catalisam a replicação viral em transcrições de novas moléculas de RNA MNVcontendo uma molécula de VPG adequada no final sua 5 '. Sucessivos ciclos de replicação viral acompanhados de tradução geraria um grande número de genomas virais que irá ser encapsidado para gerar viriões infecciosos. Para facilitar a libertação do vírus a partir de células, um ou vários ciclos de congelamento e descongelamento são executadas (passo 9). Rendimentos virais pode ser então determinada por TCID50 ou procedimentos de ensaio de placa.
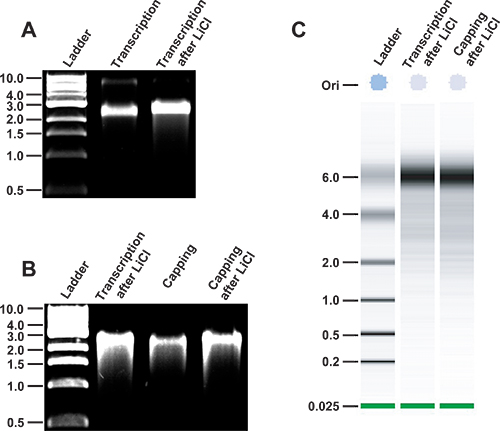
Figura 3. Análise de MNV integridade transcrições de RNA ao longo do protocolo. Uma integridade, de MNV RNA sintetizados in vitro. O plasmídeo pT7: MNV 3'Rz é em primeiro lugar linearizado usando Nhe I a enzima de restrição. Depois de purificação de ADN, os transcritos de RNA MNV são gerados in vitro, utilizando polimerase de ARN T7 (pista 2). O RNA é então purificação ed a partir de nucleótidos livres por precipitação com LiCl (pista 3). Produtos de transcrição são executados em um não desnaturante em gel de agarose a 1% em paralelo com 1-Kb escada de ADN (New England Biolabs, pista 1). Mobilidade relativa dos transcritos virais sob condições não desnaturantes é semelhante a um produto dsDNA de 2,5-3 Kb. B Integridade, de transcrições de RNA MNV após nivelamento. Transcritos MNV purificados anteriormente por precipitação com LiCl (pista 2) são submetidos a capping enzimática (pista 3) e purificação por precipitação com LiCl (pista 4). C,, Análise, em um chip de 6000 RNA Agilent Nano de transcritos MNV (pista segundo) e de casquilho transcrições MNV (pista terceiro) que tenham sido previamente precipitados em LiCl. Uma escada ssRNA é executado em paralelo.
= "Pdflinebreak"> 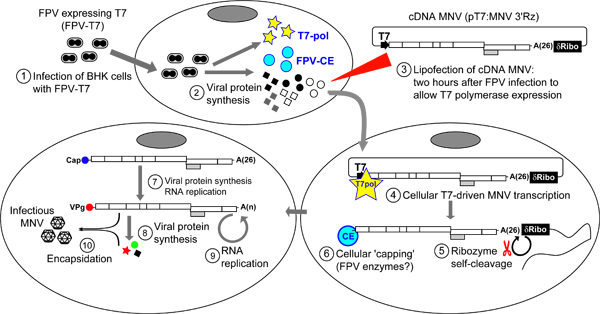
Figura 4. Descrição geral do protocolo para a recuperação de MNV infeccioso a partir de cDNA. Inicialmente, BSR-T7 (ou BHK), as células são infectadas com um vírus recombinante bouba (FPV) que expressa o bacteriófago T7 RNA polimerase (FPV-T7) (passo 1). As células infectadas são incubadas durante 2 horas antes do tratamento adicional para permitir a expressão de proteínas que inclui o FPV recombinante polimerase de ARN T7 (passo 2). Depois, pT7: MNV 3'Rz é transfectado para as células, utilizando Lipofectamine 2000 (Invitrogen) (passo 3). Uma vez no interior da célula, pT7: MNV 3'Rz é reconhecido por T7 RNA polimerase que sintetiza transcrições de RNA MNV (passo 4). A presença de um auto-clivagem sequência δ-ribozima no extremo 3 'do genoma garante a transcrição 3' terminus está localizado apenas após a cauda poliA (passo 5). Alguns transcritos virais são intracelularmente tapado por uma enzima FPV capping (passo 6). As transcrições resultantes MNV cobertas serão traduzidos para gerar proteínas MNV que catalisam a replicação transcrições MNV. Recém-sintetizado moléculas de RNA MNV contendo uma molécula de VPG adequada no final sua 5 'passariam por ciclos sucessivos de replicação viral acompanhados por tradução que podem finalmente resultar na geração do vírus infeccioso encapsidado. Para facilitar a libertação do vírus a partir de células, um ou vários ciclos de congelamento e descongelamento são executadas (passo 7). Rendimentos virais pode ser então determinada por TCID50 ou procedimentos de ensaio de placa.
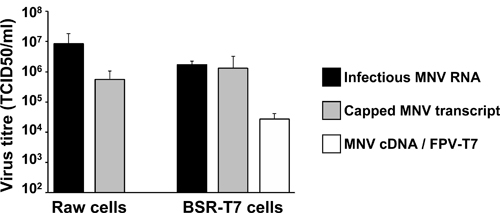
Figura 5. Os resultados representativos de títulos de anticorpos de vírus obtidas a partir de diferentes genética inversa aproxima descrito no texto. Barras cinzentas representam os títulos de vírus obtidas menos 24 horas após néon-Transfecção de 2 x 10 6 Raw264.7 células, ou após lipo-transfecção de 2 x 10 6 BSR-T7 células com transcrito in vitro e capped MNV RNA. As barras brancas representam o título de vírus normalmente obtido após lipofecção de pT7: MNV 3'Rz (MNV cDNA) em 2 x 10 6 BSR-T7 células previamente infectadas durante 2 horas com vírus expressando bouba recombinante polimerase de T7 (FPV-T7). Como um controlo positivo para a transfecção em bruto e BSR de T7cells, nós usam tipicamente 2 ug de RNA extraído de células infectadas com MNV que contêm níveis elevados de VPG-linked MNV RNA. Os controlos negativos foram efectuados com um MNV RNA ou pT7: MNV 3'Rz que codifica uma mutação frameshift (F / S), que anula a replicação, resultando em nenhum vírus detectável (dados não mostrados).
Discussão
Aqui temos ilustrado duas diferentes abordagens reversa genéticos que permitem a recuperação de MNV infecciosa em cultura de células. Ambos aproxima eficazmente ignorar o requisito absoluto para a ligação covalente de VPG para a extremidade 5 'do genoma de RNA viral através da geração de transcritos de casquilho MNV que são, então, reconhecidos pelos ribossomas celulares. Transcrição in vitro seguido de enzimaticamente capping é mais eficiente na a recuperação de MNV infeccioso que a transcrição de plasmídeos infecciosas em células que expressam polimerase de ARN T7, em que as transcrições podem ser tapados pelas enzimas FPV nivelamento. Os títulos de vírus recuperados com estes sistemas de genética inversa são semelhantes a estes obtidos por transfecção de virais VPG-linked RNA purificadas a partir de culturas de células infectadas 17 (Figura 5). A transfecção de capped MNV RNA em células permissivas Raw264.7 torna um título vírus apenas 1 log mais baixa do que as experiências que envolvem a transfection de RNA total de células infectadas e contendo viral VPG-linked RNA (Figura 5). Este facto encoraja novas investigações para determinar se a adição de uma molécula de VPG para a extremidade 5 'dos transcritos gerados por esses sistemas podem resultar em rendimentos de vírus aumento que podem revelar aspectos funcionais subjacentes infecciosidade MNV em células associadas à VPG. No entanto, consideramos este sistema de genética inversa como um um altamente eficiente comparável com outros vírus de RNA inverter sistemas genética actualmente utilizado em que o RNA transcrito in vitro permite a recuperação de títulos de anticorpos apenas 10-100 mais baixas do que as infecções reais com viriões 19, 20.
Em geral, as metodologias atuais constituem um passo significativo no campo da biologia molecular e norovírus nos fornecer as ferramentas para investigar os papéis funcionais de proteínas e motivos conservadas nos genomas de RNA de norovírus. Estas abordagens têm já sido combinados com cmodelo de rato ACTUAL disponíveis e têm mostrado que MNV recuperado a partir de cDNA infeccioso é capaz de causar infecção letal de> 80% STAT1-/ - ratinhos em menos de 10 dias 4, 21. Fazendo uso deste sistema que se recuperaram viáveis mutantes norovírus murinos da proteína da cápside e de um tracto polipirimidina envolvido na ligação de factores do hospedeiro diferentes (PTB e PCBP) que exibem um fenótipos um tanto in vivo atenuado 21, 22. Além disso, demonstraram recentemente que os vírus que não possuem a capacidade de expressar a proteína a partir de VF1 ORF4 eficientemente replicar em cultura de células, mas novamente ter reduzido a virulência em ratinhos com respeito a WT MNV 8. Estes estudos nos incentivam a projetar versões atenuadas de norovírus humanos com base em estudos MNV que poderiam ser investigadas como candidatos vacinais potenciais.
Divulgações
Não temos nada a divulgar.
Agradecimentos
Esta pesquisa foi financiada por uma bolsa Wellcome Senior confiança atribuído a Ian Goodfellow, e Marie Curie Intra Europeia Fellowship (FP7 Conselho Europeu de Investigação) concedido a Armando Arias. Gostaríamos de agradecer a Io Hong Cheong, Dr Rebecca Robey e Dr. Mike Skinner para dar-nos permissão para usar seu Bioanalyzer Agilent e ajudando na execução de amostras de RNA.
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente | Companhia | Número de catálogo | |
| Lítio solução de cloreto de precipitação | Ambion | AM9480 | |
| RNA solução de armazenamento | Ambion | AM7000 | |
| MEGAscript T7 roteiro | Ambion | AM1333 | |
| ScriptCap M7G nivelamento Sistema | Biotecnologias Epicentre | SCCE0610 | |
| Sistema de transfecção Neon | Invitrogen | MPK5000 | |
| Kit sistema Neon transfecção | Invitrogen | MPK1025 | |
| Opti-MEM I | Invitrogen | 31985070 | |
| Lipofectamine Reagente Transfection 2000 | Invitrogen | 11668-027 | |
| Agilent RNA 600 Nano kit | Agilent | 5067-1511 | |
| Agilent 2100 bioanalyzer | Agilent | G2939AA | |
| Illustra GFX PCR de DNA e Gel Kit de Purificação da faixa | GE Healthcare | 28-9034-70 | |
| RiboMAX grande escala de produção de RNA System-T7 | Promega | P1300 |
Referências
- . Outbreaks of gastroenteritis associated with noroviruses on cruise ships--United States. MMWR Morb. Mortal. Wkly. Rep. 51, 1112-1115 (2002).
- Lopman, B. A. Epidemiology and cost of nosocomial gastroenteritis, Avon, England, 2002-2003. Emerg. Infect. Dis. 10, 2002-2003 (2004).
- Duizer, E. Laboratory efforts to cultivate noroviruses. J. Gen. Virol. 85, 79-87 (2004).
- Karst, S. M., Wobus, C. E., Lay, M., Davidson, J., Virgin, H. W. T. STAT1-dependent innate immunity to a Norwalk-like virus. Science. 299, 1575-1575 (2003).
- Wobus, C. E. Replication of Norovirus in cell culture reveals a tropism for dendritic cells and macrophages. PLoS Biol. 2, e432 (2004).
- Bok, K. Inhibition of norovirus replication by morpholino oligomers targeting the 5'-end of the genome. Virology. 380, 328-337 (2008).
- Kim, Y., Thapa, M., Hua, D. H., Chang, K. O. Biodegradable nanogels for oral delivery of interferon for norovirus infection. Antiviral Res. 89, 165-173 (2011).
- McFadden, N. Norovirus Regulation of the Innate Immune Response and Apoptosis Occurs via the Product of the Alternative Open Reading Frame 4. PLoS Pathog. 7, e1002413 (2011).
- Kormelink, R., van Poelwijk, F., Peters, D., Goldbach, R. Non-viral heterogeneous sequences at the 5' ends of tomato spotted wilt virus mRNAs. J. Gen. Virol. 73, 2125-2128 (1992).
- Shuman, S., Hurwitz, J. Mechanism of mRNA capping by vaccinia virus guanylyltransferase: characterization of an enzyme--guanylate intermediate. Proc. Natl. Acad. Sci. U.S.A. 78, 187-191 (1981).
- Jang, S. K., Pestova, T. V., Hellen, C. U., Witherell, G. W., Wimmer, E. Cap-independent translation of picornavirus RNAs: structure and function of the internal ribosomal entry site. Enzyme. 44, 292-309 (1990).
- Chaudhry, Y. Caliciviruses differ in their functional requirements for eIF4F components. J. Biol. Chem. 281, 25315-25325 (2006).
- Goodfellow, I. Calicivirus translation initiation requires an interaction between VPg and eIF 4. E. EMBO Rep. 6, 968-972 (2005).
- Herbert, T. P., Brierley, I., Brown, T. D. Identification of a protein linked to the genomic and subgenomic mRNAs of feline calicivirus and its role in translation. J. Gen. Virol. 78 ( Pt5 ), 1033-1040 (1997).
- Chaudhry, Y., Skinner, M. A., Goodfellow, I. G. Recovery of genetically defined murine norovirus in tissue culture by using a fowlpox virus expressing T7 RNA polymerase. J. Gen. Virol. 88, 2091-20100 (2007).
- Ward, V. K. Recovery of infectious murine norovirus using pol II-driven expression of full-length cDNA. Proc. Natl. Acad. Sci. U.S.A. 104, 11050-11055 (2007).
- Yunus, M. A., Chung, L. M., Chaudhry, Y., Bailey, D., Goodfellow, I. Development of an optimized RNA-based murine norovirus reverse genetics system. J. Virol. Methods. 169, 112-118 (2010).
- Arias, A., Perales, C., Escarmis, C., Domingo, E. Deletion mutants of VPg reveal new cytopathology determinants in a picornavirus. PLoS One. 5, e10735 (2010).
- Werf, S. v. a. n. d. e. r., Bradley, J., Wimmer, E., Studier, F. W., Dunn, J. J. Synthesis of infectious poliovirus RNA by purified T7 RNA polymerase. Proc. Natl. Acad. Sci. U.S.A. 83, 2330-2334 (1986).
- Bailey, D., Thackray, L. B., Goodfellow, I. G. A single amino acid substitution in the murine norovirus capsid protein is sufficient for attenuation in vivo. J. Virol. 82, 7725-7728 (2008).
- Bailey, D. Functional analysis of RNA structures present at the 3' extremity of the murine norovirus genome: the variable polypyrimidine tract plays a role in viral virulence. J. Virol. 84, 2859-2870 (2010).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados