Method Article
Ein Visual Assay zur T6SS-vermittelte bakterielle Wettbewerb überwachen
In diesem Artikel
Zusammenfassung
Wir beschreiben einen qualitativen Test auf bakterielle Wettbewerb durch die vermittelten überwachen Pseudomonas aeruginosa Type VI Sekretionssystem (T6SS). Der Assay beruht auf der Überlebensrate / Abtötung von Escherichia coli eine tragende Zielzellen LacZ-Reporter. Diese Technik ist einstellbar, um die bakteriziden / Bakteriostase Aktivität T6SS-kompetente Mikroorganismen einzuschätzen.
Zusammenfassung
Typ VI Sekretionssysteme (T6SSs) sind molekulare Nanomaschinen ermöglichen Gram-negativen Bakterien zu transportieren und zu injizieren Proteine in einer Vielzahl von Zielzellen 1,2. Die T6SS besteht aus 13 Kern-Komponenten und zeigt strukturelle Ähnlichkeiten mit dem Schwanz-Rohr von Bakteriophagen 3. Der Phage verwendet ein Rohr und ein Stechgerät die Zellhülle von Ziel-Bakterien eindringen und injizieren DNA. Es wird vorgeschlagen, dass die T6SS ein invertiertes Bakteriophagen Vorrichtung Schaffung eines spezifischen Pfades in der bakteriellen Zellwand und Toxine Effektoren an die Oberfläche zu treiben. Das Verfahren könnte weiterhin entnommen werden, und die Vorrichtung könnte T6SS anderen Zellen, mit denen das Bakterium in Kontakt zu perforieren, wodurch die Injektion der Effektoren in diesen Zielen. Der Schwanz Rohr und Stechgerät Teile der T6SS mit Hcp und VgrG Proteinen bzw. 4,5.
Die Vielseitigkeit des T6SS wurde durch s nachgewiesenTUDIEN mit verschiedenen bakteriellen Erregern. Die Vibrio cholerae T6SS enthaltene Zytoskelett eukaryontischer Wirtszellen durch Einspritzen eines "entwickelt" VgrG Tragen eines C-terminalen Aktin Vernetzen Domäne 6,7 umzugestalten. Ein weiteres eindrucksvolles Beispiel wurde kürzlich dokumentiert mit Pseudomonas aeruginosa in der Lage, anzugreifen und zu töten Bakterien in einem T6SS-abhängigen Weise, also die Förderung der Errichtung von Bakterien in spezifischen mikrobiellen Nischen und Wettbewerbsumfeld 8,9,10 ist.
Im letzteren Fall sind drei T6SS-sekretierte Proteine, nämlich Tse1, Tse2 und TSE3 wie die Toxine in den Zielbakterien (Abbildung 1) injiziert identifiziert. Donorzelle ist aus der schädlichen Wirkung dieser Effektoren über einen Anti-Toxin-Mechanismus, durch den TSI1, TSI2 und Tsi3 Immunität Proteine vermittelt 8,9,10 geschützt. Dieses antimikrobielle Aktivität kann überwacht werden, wenn T6SS-kompetenten Bakterien zu n cokultiviert werdenolid Oberflächen im Wettbewerb mit anderen Bakterienarten oder mit T6SS-inactive Bakterien der gleichen Art 8,11,12,13.
Die verfügbaren Daten betonte eine numerische Annäherung an die bakterielle Kompetitionsassay, einschließlich zeitaufwändig CFU Zählen, das hängt stark von Antibiotika Entscheidungsträger. Im Fall von antibiotikaresistenten Stämmen wie P. aeruginosa, können diese Methoden ungeeignet sein. Ferner mit der Identifizierung von etwa 200 verschiedenen T6SS Loci in mehr als 100 Bakteriengenome 14, ist eine bequeme Screening-Werkzeug sehr wünschenswert. Wir entwickelten einen Test, der einfach zu bedienen ist und erfordert Standard-Labor-Material und Reagenzien. Das Verfahren bietet eine schnelle und qualitative Technik, um die T6SS-abhängige bakterizide / Bakteriostase-Aktivität durch Verwendung eines Reporter-Stamm als Beute (in diesem Fall Escherichia coli DH5a) Zulassen einer-Komplementation des lacZ-Gens zu überwachen. Insgesamt ist dieses Verfahren graphic und ermöglicht eine schnelle Identifizierung von T6SS-bezogene Phänotypen auf Agarplatten. Dieses experimentelle Protokoll kann auf andere Stämme oder Bakterienspezies unter Berücksichtigung der besonderen Bedingungen wie Wachstumsmedium, Temperatur oder Zeit des Kontaktes angepasst werden.
Protokoll
Ein. Bakterienstämme und Kulturen
- Engineering einer Escherichia coli Empfängerzelle (Prey, P) durch Transformieren (unter Verwendung von Standardverfahren CaCl 2-Behandlung oder Elektroporation) 15 E. coli DH5a Zellen mit einem Plasmid, so dass der a-Komplementation des lacZ-Gens (Tab. 1). Platte die transformierten Zellen auf Luria-Bertani-Agar-Platten (LBA, 1,5% Agar), das 5-Brom-4-chlor-indolyl-β-D-galactopyranosid (X-gal) bei 40 mg / ml Endkonzentration und geeigneten Antibiotikum. Inkubation bei 37 ° C über Nacht und am nächsten Morgen wählen Sie für Blau Transformanten (vgl. § 1,3).
- Wachsen die Pseudomonas aeruginosa Spenderzellen, die T6SS-aktiv sind (D +) oder T6SS-inactive (D-) (Tabelle 1) auf LBA über Nacht bei 37 ° C. In dem hier vorgestellten Beispiel verwendeten wir ein P. aeruginosa RETs belasten besitzeneine konstitutiv aktive H1-T6SS 16 und einen isogenen Mutante mit einer Deletion des H1-T6SS Gencluster (Tabelle 1).
- Am nächsten Tag vorzubereiten eine Übernachtung flüssigen Kultur in belüfteten Kolben durch Impfen einer hellblauen Klon unter den E. coli-Transformanten (P) von der Platte in § 1.1 beschrieben, in 5 ml tryptischer Sojabrühe (TSB) mit dem entsprechenden Antibiotikum ergänzt. Wachsen unter Rühren bei 37 ° C. Verfahren gleichermaßen mit einer einzelnen Kolonie von (D +) und von (D -) von der Platte in § 1.2 beschrieben.
2. Wettbewerb Assay
- Bereiten LBA Platten für den nächsten Tag Experiment. Achten Sie darauf, diese Platten richtig getrocknet (entweder in der Nähe der Bunsenbrenner oder in Reinraumgehäuse). Bereiten Sie ein "Assay-Eingang" (A-Eingang) Platte für die Stämme (D +), (D -) und (P ). Vorbereiten eines "Assay-Ausgang" (A-Output)-Platte für die Stämme (D - + P) und (D + + P). Teilen Sie und beschriften Sie die Platten entsprechend.
- Nach Wachstum über Nacht (siehe § 1.3), messen die optische Dichte (OD 600nm) des Eingangs-Bakterienkultur (D - D + und P) und Berechnen der Volumen benötigt, um eine Zelldichte äquivalent zu 1 Einheit OD 600nm jedes Stammes erhalten . Jedes "Eingang" Zellkultur (D - D + und P) anfänglich in ein steriles 1.5 ml Eppendorf-Röhrchen gesammelt.
- Zentrifugation der Proben bakteriellen bei 13.000 rpm während 1 min bei Raumtemperatur und der Überstand verworfen.
- Resuspendieren der Pellets des D - inokulieren D + und P Kulturen, in 100 ml frisches TSB durch vorsichtiges Pipettieren und 10 ml jeder entspreentsprechenden Stamm einer einzigen Stelle auf der "A-Eingang" Platte (hergestellt in § 2,1).
- Inokulieren "A-Ausgabe" Platte (hergestellt in § 2,1). Genauer gesagt, vorsichtig mischen 30 ul (D +) mit 30 ul (P) und 30 ul (D -) mit 30 ml (P) in zwei getrennte Eppendorf-Röhrchen (beachten Sie, dass die Kulturen verwendet, die in § 2.4 beschrieben sind) . Beimpfen 20 ul des Mixes (D + / P) und (D - / P) als einzelne Punkte auf der "A-Ausgabe" Platte. Lassen Sie die Spots in der Nähe einen Bunsenbrenner trocknen und legen Sie die Platte in einem Brutschrank bei 37 ° C für einen angemessenen Zeitraum, während dessen der Abtöten der Bakterien stattfindet. Im Fall von S. aeruginosa wird eine effiziente Bakterienabtötung beobachtet nach 5-stündiger Inkubation bei einem Vergleich eines aktiven T6SS mit einem T6SS defekte Stamm.
3. Qualitative Beobachtung von ter Abtöten der Bakterien
- Planen LBA Platten, enthaltend 40 ug / ml X-gal für das Auslesen des Assays. Die Platten werden als "Readout Eingang" oder "R-Eingang" zu isolierten Bakterien oder "Readout Ausgang" oder "R-Ausgang" auf gemischte Bakterienkulturen zu erkennen und so gelesen das Töten Leistung erkennen werden. Diese Platten müssen auch richtig getrocknet werden. Teilen Sie die Platte in vier gleiche Teile auf seinem Rücken und kommentieren diese Teile 0, 10 -1, 10 -2 und 10 -3, Bestimmung der Verdünnungen der Bakterienkultur auf den Agarplatten erkennen (vgl. § 3,3). Die Verdünnung wird eine semi-quantitative Auswertung der blau / weiß Bakterien-Verhältnis innerhalb der gleichen Stelle zu ermöglichen.
- Sammeln Sie mit einer sterilen Schleife die einzelnen bakteriellen Spots aus dem "A-Eingang" Platte (vgl. § 2,4) und "A-Ausgabe" Platte (vgl. § 2,5) und resuspendieren jeden Ort in verschiedenen 1,5 ml Eppendorf-Röhrchen mit 1 ml TSB. Zeigen in einer Eppendorf-Schüttler Block während 30 min, um effizient die resuspendierenBakterien.
- Bereiten Sie eine Serie von 5 mal 3 Eppendorf Röhrchen mit 900 ul TSB und gehen für jeden resuspendierten Ort zu zehn fache serielle Verdünnungen bis zu 10 -3. Achten Sie darauf, Pipettenspitzen zu ändern und Vortex 5 Sekunden zwischen jedem Verdünnungsschritt.
- Fahren Sie mit dem Spotten Ihre bakterielle Verdünnungen in § 3,3 auf gut getrockneten LBA-Platten mit 40 ug / ml X-gal, indem Sie mit der meisten verdünnten dem unverwässerten Suspension hergestellt, so dass diese die "R-Eingang" Platten für die "A -input "-Spots und die" R-Ausgang "Platten für die" A-Ausgabe "Flecken (Abbildung 2). Vortex kurz jedes Rohr, bevor Sie mit dem Spotting, um eine homogene Bakteriensuspension zu halten. Für die Reproduzierbarkeit der Ergebnisse, spot 20 ul in dreifacher Ausfertigung innerhalb eines Quadranten (Abbildung 2). Die relative Trockenheit Ihrem Teller ist in diesem Stadium wichtig, da die Flecken bleiben einzeln auf der Platte getrennt und nicht aufeinander gleiten müssen.
Eine Quantifizierung des Kompetitionstest ist auch in dieser Phase des Experiments möglich. Nach dem Schritt 3,3, verteilt 100 ul der Verdünnung 10 -3 auf LBA-Platten mit 40 ug / ml X-gal. Für die Reproduzierbarkeit der Ergebnisse zu einer Beschichtung in dreifacher Ausfertigung für jeden Punkt aus dem "A-Ausgabe" Platte gehen. Platzieren Sie die Verdünnungsplatten in einem 37 ° C Inkubator über Nacht (entsprechend 16 h Inkubation). Fahren Sie mit dem Zählen der blauen Kolonien (die P-Zellen, die am Leben geblieben). Typische Ergebnisse nach 5 Stunden von Wettbewerb erhaltenen Ergebnisse sind in Abbildung 3 dargestellt. - Hinterlegen der Platte offen (ohne Deckel) innerhalb eines sterilen Zone, um die Absorption der Flüssigkeit in Überschuß innerhalb jeder Stelle zu ermöglichen.
- Legen Sie die Platte in einem 37 ° C Inkubator über Nacht. Während dieser Inkubationszeit wird die Abtötung noch auftritt in der Mischung (D + / P), da beide Stämme sind noch in Kontakt.
- Nehmen Sie ein Bildoder Scannen Ihrer Platten für die Output-Analyse (Abbildung 2). Wo Flecken weitgehend blauen bleibt es zeigt, dass E. coli nicht durch P. getötet worden aeruginosa. Das ist der Fall, wenn E. coli wird mit einem T6SS defekten P. gemischt aeruginosa Stamm (D-) (Abbildung 2, Platte unten rechts).
Ergebnisse
Typische Ergebnisse sind in Abbildung 1 mit den Stämmen und Reagenzien in Tabelle 1 beschrieben dargestellt. Die Platten in dieser Figur gezeigt sind, wurden nach Inkubation über Nacht gescannt. Die "Auslese-Input" Platten zeigen eine Verdünnungsreihe Muster für die Stämme in diesem Assay verwendet. Wie erwartet ist der E. coli Beute Flecken (P) Überexprimieren das lacZ-Gen erscheinen blau auf Medien mit X-gal ergänzt, während der Spender P. aeruginosa-Stämme (D +, T6SS aktiv) und (D -, T6SS inaktiv) weiß bleiben. Die "Auslese-Ausgang" Platten, auf denen die Mischung zwischen dem Opfer und einem T6SS aktiven Stamm (D + / P) gesichtet wurde zeigen, das Verschwinden der blauen Beute damit angibt, dass es getötet wurde. Dies zeigt die Fähigkeit des Spenders, um die Beute outcompete. Die Persistenz der blauen Farbe auf dem (D - / P)-Platte zeigt die Unfähigkeit eines inaktiven T6SS Spender, um den blauen Beute zu töten.
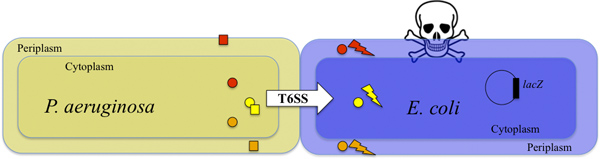
Abbildung 1. Tötung von E. coli durch T6SS-kompetent P. aeruginosa. P. aeruginosa spritzt Toxine in den E. coli Zielzelle in einem T6SS-abhängigen Weise (durch den weißen Pfeil). Zwei Toxine Tse1 und TSE3 (orange und rote Kreise) in den E. eingespritzt coli Periplasma und verschlechtern die Peptidoglycan 9. Die Tse2 Toxin (gelber Kreis) wird in den E. eingespritzt coli Zytoplasma und hat eine bakteriostatische Wirkung 8,10. Die kombinierte Wirkung der Toxine tötet die Zielzellen (Blitz und Schädel). Die Überlebensrate von Zielzellen kann durch Überwachen der Aktivität des hergestellten β-Galactosidase (siehe auch Figur 2) erfaßt werden. P. eineruginosa gegen die Aktivität der Toxine durch die Immunität Proteine TSI1, TSI2 und Tsi3 (orange, gelb und rot Plätzen, jeweils) 8,9,10 geschützt.
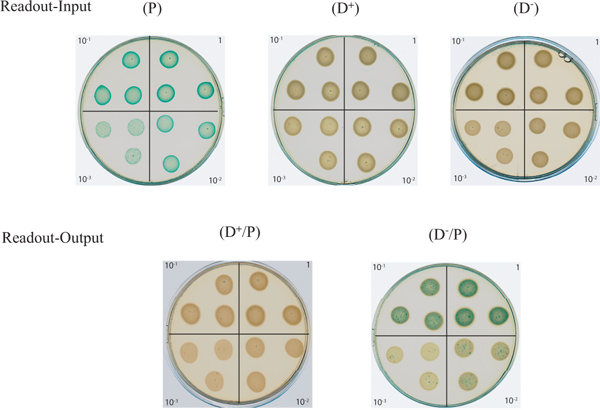
Abbildung 2. Agar-Platten-Assay zu überwachen T6SS-abhängige Abtöten der Bakterien In dieser Zahl sind auf dem oberen Teil der "Auslese-Input" Platten, bestehend aus den Verdünnungsreihen der D gezeigt -., D + und P-Eingang Zellen. Die P Eingangszellen sind blau aufgrund des α-Komplementation des lacZ-Gens und damit produzierten β-Galactosidase, dass spaltet X-Gal. Im unteren Teil wird die "Auslese-Output" Platten aus Verdünnungsreihen der bakteriellen Mischung zwischen einem aktiven (D + / P) gezeigt, , oder ein inaktives (D - / P), T6SS Donor P. aeruginosa Stamm mit der E. coli Beute. Klicken Sie hier für eine größere Abbildung zu sehen .
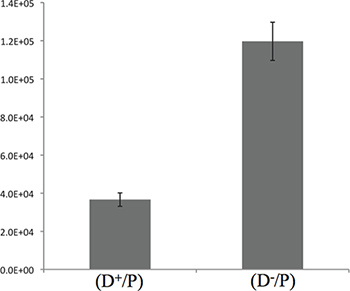
Abbildung 3. Die Quantifizierung der Tötung von E. coli durch P. aeruginosa nach 5 h Inkubation. Die Grafik zeigt die CFU Zählung von E. coli beschrieben in der 3,4 Schritt. Die hier vorgestellten Ergebnisse zeigen eine 3-fache Differenz zwischen dem T6SS + und den T6SS-Stämmen, was darauf hindeutet, dass die meisten der Tötung stattfindet während der 5 ersten Stunden des Kontakts.
Diskussion
Die Methode in diesem Artikel vorgestellt ermöglicht eine visuelle Beobachtung der T6SS-vermittelte bakterizide / Bakteriostase Aktivität. Der Assay basiert auf der Oberfläche einer Agarplatte durchgeführt. Es wurde zuvor gezeigt, dass T6SS-abhängigen Abtötungsassay mit gemischten bakteriellen flüssigen Kultur durchgeführt nicht effizient ist, wahrscheinlich wegen der fehlenden stationären Kontakt zwischen den beiden Bakterien 8. Die T6SS wird angenommen, dass mit einem Mechanismus ähnlich dem von Bakteriophagen verwendet, um DNA in Zielzellen 17 einzuspritzen betreiben. In flüssiger Kultur, die röhrenförmige Struktur des T6SS kann leichter brechen können inter-bakterielle Kontakt verloren, und die Giftstoffe nicht effizient bereitgestellt.
In Bezug auf die Inkubationszeiten, sind die 5 ersten Stunden der Kontakt, den wir beschreiben, zwischen dem Donorstamm und die Beute aus, um bakterielle Tötung zwischen P. beobachten aeruginosa und E. coli, wie in Abbildung 3 dargestellt . Trotzdem ist es empfehlenswert, die Inkubationszeit durch Durchführung einer kinetischen um die Versuchsbedingungen zu optimieren einzustellen.
Da dieses Verfahren eine Farbe basierten Technik können die Ausgabeergebnisse vom Pigmentierung der Donorstamm beeinträchtigt werden. Zum Beispiel werden im Fall von S. aeruginosa, produzieren einige Stämme hohe Buntpigmente wie Pyocyanin und Pyoverdin, das mit dem Assay Auslesen stören können, wodurch der Unterschied zu der Beute relativ schwierig. Andere chromogene β-Galactosidase-Substrate, wie der Magenta-gal oder die rot-gal, kann anstelle des X-gal (Tabelle 1) verwendet werden.
Der Wettbewerb Assay kann Verwendung anderer Reportergene für das Auslesen machen. So hat ähnlichen Assay auch durch Verwendung grün fluoreszierendes Protein-markierten preys 12 durchgeführt.
Unser Test zwar nicht quantitativ, gibt einen guten Hinweisder T6SS Aktivität, da sie auf das Überleben oder die Tötung eines Reporters Beute basiert. Diese Technik bietet den Vorteil, dass sie einfach und bequem, die bakterizides / Bakteriostase Aktivität T6SSs aus beliebigen Bakterien-Spezies zu evaluieren. Bisher war die Aktivität des T6SS hat gegen Gram-negative Bakterien und keine klare Beispiels T6SS-sensitive gram-positive Bakterien gezeigt wurde berichtet noch 12. Es ist auch offensichtlich, dass in der Kultur Inkompatibilität der verschiedenen Bakterienarten zu testen (zB Wachstumstemperatur, Sauerstoffzufuhr, spezifischer Medien) zu berücksichtigen.
Unserem Assay kann auch verwendet werden, um auszuwerten, welches der T6SS Komponenten sind unbedingt erforderlich, da bereits Spuren von einem sekretierten Toxin könnte ausreichend sein, um die Beute zu töten. Auch schwache Aktivität der T6SS könnte dann deutlich von unseren Test nachgewiesen werden, dass Standard-Verfahren Tests T6SS-abhängige Sekretion mit Kulturüberstand und Western-Blot-VergleichAnalyse. Allerdings ist eine richtige koloniebildenden Einheit (CFU) Zählen noch für eine genaue Quantifizierung dieses T6SS Aktivität erforderlich.
Offenlegungen
Wir haben nichts zu offenbaren.
Danksagungen
Diese Arbeit wurde durch den Wellcome Trust Gewährung WT091939MA finanziert. Alain Filloux wird von der Royal Society unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenz / Material | Firma | Katalog-Nummer | Kommentare |
| P. aeruginosa PAKΔ RETs (D +) (Aktiv T6SS) | Laborstamm | Beschrieben in Reference 16 | |
| P. aeruginosa PAKΔ RETs Δ H1-T6SS (D -) (inaktive T6SS) | Diese Studie | Die H1-T6SS Cluster (umfasst der Gene PA0070 um PA0095) durch allelischen Austausch wurde nach dem Verfahren in Referenz 18 beschrieben gelöscht. Die Bis-Fragment-Primer: Die Mutator-Fragment wurde mit dem folgenden Satz von Primern erzeugt: 5'-ATGGTCAACGACATGGAGCTGGAG-3 ', und 5'CGAGGCCGATCAGGCCTTCAGAACTGA-3 '. Die Down-Fragment Primer: 5'-TCAGGCCTTCAGAACTGAAGCGGCGCA-3 ', 5'-GGTGGCGTTCAACAGTTCCATGTC-3 ' | |
| E.coli DH5 | Invitrogen | 18258-012 | F-φ80 lac ZΔM15 Δ (lac ZYA-arg F) U169 rec A1 Ende A1 hsd R17 (r k -, m k +) pho A sup E44 λ-thi -1 gyr A96 rel A1 |
| pBluescript II SK (+) | Agilent | 212205 | Dieser Vektor drückt die α-Peptid der β-Galactosidase für α-Komplementation verwendet. |
| X-gal | Invitrogen | 15520-018 | Verwenden Sie bei 40 pg / ml |
| Luria Bertani-Agar | Merck-Chemikalien | 1.10283.0500 | |
| TSB (Kasein Soja Brühe) | Oxoid | CM109 | |
| Vortex Schüttler Genius 3 | IKA | 3340000 | |
| Scanner | Epson | V700 | |
| Spektrophotometer | WPA Biowave | CO8000 Zelldichte Meter | |
| Magenta-gal | Bioworld | 30350001-1 (715241) | |
| Red-gal | Research Organics | 1364c | |
Tabelle 1. Strain, Plasmid, Material und Reagenz verwendet. |
Referenzen
- Filloux, A., et al. The bacterial type VI secretion machine: yet another player for protein transport across membranes. Microbiology. 154 (6), 1570 (2008).
- Cascales, E., Cambillau, C. Structural biology of type VI secretion systems. Philos. Trans. R. Soc. Lond. B. Biol. Sci. 367 (1592), 1102 (2012).
- Leiman, P. G., et al. Morphogenesis of the T4 tail and tail fibers. Virol. J. 7, 355 (2010).
- Ballister, E. R., et al. In vitro self-assembly of tailorable nanotubes from a simple protein building block. Proc. Natl. Acad. Sci. U.S.A. 105 (10), 3733 (2008).
- Leiman, P. G., et al. Type VI secretion apparatus and phage tail-associated protein complexes share a common evolutionary origin. Proc. Natl. Acad. Sci. U.S.A. 106 (11), 4154 (2009).
- Pukatzki, S., et al. Type VI secretion system translocates a phage tail spike-like protein into target cells where it cross-links actin. Proc. Natl. Acad. Sci. U.S.A. 104 (39), 15508 (2007).
- Ma, A. T., et al. Translocation of a Vibrio cholerae type VI secretion effector requires bacterial endocytosis by host cells. Cell Host Microbe. 5 (3), 234 (2009).
- Hood, R. D., et al. A type VI secretion system of Pseudomonas aeruginosa targets a toxin to bacteria. Cell Host Microbe. 7 (1), 25 (2010).
- Russell, A. B., et al. Type VI secretion delivers bacteriolytic effectors to target cells. Nature. 475 (7356), 343 (2011).
- Li, M., et al. Structural basis for type VI secretion effector recognition by a cognate immunity protein. PLoS Pathog. 8 (4), e1002613 (2012).
- Zheng, J., et al. Genetic analysis of anti-amoebae and anti-bacterial activities of the type VI secretion system in Vibrio cholerae. PLoS One. 6 (8), (2011).
- Schwarz, S., et al. Burkholderia type VI secretion systems have distinct roles in eukaryotic and bacterial cell interactions. PLoS Pathog. 6 (8), e23876 (2010).
- Murdoch, S. L., et al. The opportunistic pathogen Serratia marcescens utilizes type VI secretion to target bacterial competitors. J. Bacteriol. 193 (21), 6057 (2011).
- Boyer, F., et al. Dissecting the bacterial type VI secretion system by a genome wide in silico analysis: what can be learned from available microbial genomic resources?. BMC Genomics. 10, 104 (2009).
- Maniatis, T., et al. . Molecular cloning: A Laboratory Manual. , (1982).
- Mougous, J. D., et al. A virulence locus of Pseudomonas aeruginosa encodes a protein secretion apparatus. Science. 312 (5779), 1526 (2006).
- Records, A. R., et al. The type VI secretion system: a multipurpose delivery system with a phage-like machinery. Mol. Plant Microbe Interact. 24 (7), 751 (2011).
- Hachani, A., et al. Type VI secretion system in Pseudomonas aeruginosa: secretion and multimerization of VgrG proteins. J. Biol. Chem. 286 (14), 12317 (2011).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten