Method Article
Un test per il monitoraggio visivo T6SS-mediata Concorso batterica
In questo articolo
Riepilogo
Descriviamo un dosaggio qualitativo per monitorare la concorrenza batterica mediata dalla Pseudomonas aeruginosa Sistema di secrezione di tipo VI (T6SS). Il saggio si basa sulla sopravvivenza / uccisione di cellule di Escherichia coli che trasportano un bersaglio LacZ-Reporter. Questa tecnica è regolabile per valutare l'attività battericida / bacteriostasis di T6SS-abili microrganismi.
Abstract
Sistemi di secrezione di tipo VI (T6SSs) sono nanomacchine molecolari permettendo batteri Gram-negativi per trasportare e iniettare proteine in una varietà di cellule bersaglio 1,2. Il T6SS è composto da 13 componenti di base e mostra somiglianze strutturali con la coda-tubo di batteriofagi 3. Il fago utilizza un tubo e un dispositivo di puntura di penetrare dell'involucro cellulare di batteri bersaglio e iniettare DNA. Si propone che il T6SS è un dispositivo batteriofago invertita creando un percorso specifico nella busta cellula batterica guidare effettori e tossine alla superficie. Il processo potrebbe proseguire e il dispositivo potrebbe perforare T6SS altre cellule con cui il batterio è in contatto, quindi iniettare gli effettori in questi obiettivi. Il tubo di coda e parti punzonamento dei dispositivi dei T6SS sono realizzati con Hcp e proteine VgrG, rispettivamente, 4,5.
La versatilità del T6SS è stata dimostrata attraverso sTUDI utilizzando vari batteri patogeni. Le T6SS Vibrio cholerae può rimodellare citoscheletro delle cellule ospite eucariotiche iniettando un VgrG "evoluto" portando un C-terminale actina reticolazione domini 6,7. Un altro esempio lampante è stato recentemente documentato con Pseudomonas aeruginosa, che è in grado di colpire e uccidere i batteri in un T6SS-dipendente, quindi, favorire la creazione di batteri in nicchie microbiche specifiche e il contesto concorrenziale 8,9,10.
In quest'ultimo caso, tre T6SS-proteine secrete, cioè Tse1, Tse2 TSE3 e sono state identificate come le tossine iniettati nei batteri bersaglio (Figura 1). La cellula donatrice è protetta dagli effetti deleteri di questi effettori attraverso un meccanismo anti-tossina, mediato da proteine di immunità TSI1, TSI2 e Tsi3 8,9,10. Questa attività antimicrobica può essere monitorata quando T6SS-abili batteri sono co-coltivati su sOlid superfici in competizione con altre specie batteriche o con T6SS-inattivi i batteri della stessa specie 8,11,12,13.
I dati disponibili hanno sottolineato un approccio numerico al saggio di competizione batterica, anche in termini di tempo di conteggio CFU che dipende in gran parte produttori di antibiotici. Nel caso di ceppi resistenti agli antibiotici come P. aeruginosa, questi metodi possono essere appropriati. Inoltre, con l'identificazione di circa 200 differenti loci T6SS in più di 100 genomi batterici 14, uno strumento di screening conveniente è altamente desiderabile. Abbiamo sviluppato un saggio che è facile da usare e richiede materiale standard di laboratorio e reagenti. Il metodo offre una tecnica rapida e qualitativa per monitorare la T6SS-dipendente battericida / bacteriostasis attività utilizzando un ceppo reporter come una preda (in questo caso Escherichia coli DH5a) permettendo a-complementazione del gene lacZ. Nel complesso, questo metodo è graphic e permette la rapida identificazione di T6SS legati fenotipi su piastre di agar. Questo protocollo sperimentale può essere adattato ad altri ceppi o specie batteriche tenendo conto delle condizioni specifiche come mezzi di crescita, la temperatura o il tempo di contatto.
Protocollo
1. Ceppi batterici e Culture
- Ingegnere una cellula di Escherichia coli destinatario (Prey, P), trasformando (utilizzando standard di CaCl 2 trattamento o elettroporazione) 15 E. coli DH5a cellule con un plasmide permette l'a-complementazione del gene lacZ (Tabella 1). Piastra le cellule trasformate di Luria-Bertani piastre di agar (LBA, 1,5% agar) contenente 5-bromo-4-cloro-indolil-β-D-galattopiranoside (X-gal) a 40 mg / ml concentrazione finale e antibiotico appropriato. Incubare a 37 ° C durante la notte e la mattina seguente per selezionare trasformanti blu (vedi § 1.3).
- Crescere le cellule donatrici Pseudomonas aeruginosa che sono T6SS-attiva (D +) o T6SS-inattivo (D-) (Tabella 1) per tutta la notte LBA a 37 ° C. Nell'esempio qui presentato è stato utilizzato un P. RET aeruginosa ceppo possessoun H1-T6SS costitutivamente attivo 16 ed un mutante isogenico con una delezione della H1-T6SS cluster genico (Tabella 1).
- Il giorno successivo, preparare un liquido di coltura durante la notte in matraccio aerato inoculando un clone blu luminoso tra la E. trasformanti coli (P) dalla piastra descritto in § 1.1, in 5 ml di Tryptic Soy Broth (TSB) integrato con l'antibiotico appropriato. Crescere sotto agitazione a 37 ° C. Procedere anche con una singola colonia di (D +) e di (D -) dalla piastra descritto in § 1.2.
2. Concorso Saggio
- Preparare piatti LBA per l'esperimento giorno successivo. Assicurarsi che questi piatti è asciugato correttamente (sia vicino al becco Bunsen o in cabinet a flusso laminare). Preparare un "test di ingresso" (A-Input) Piastra per i ceppi (D +), (D -) e (P ). Preparazione di un "Assay-output" (A-output) piastra per i ceppi (D - P +) e (D + + P). Dividere ed etichettare le piastre di conseguenza.
- Dopo una crescita overnight (cfr. § 1.3), misurare la densità ottica (OD 600nm) della coltura batterica di ingresso (D -, D + e P) e calcolare il volume richiesto per ottenere una densità cellulare equivalente ad 1 unità OD 600nm di ogni ceppo . Ogni coltura cellulare "input" (D -, D + e P) è inizialmente raccolto in un contenitore sterile da 1,5 ml provette Eppendorf.
- Centrifugare i campioni batterici a 13.000 rpm per 1 min a temperatura ambiente e scartare il surnatante.
- Risospendere il pellet di D -, D + culture, e P, in 100 ml di fresca TSB pipettando dolce e inoculare 10 ml di ciascuna corricorrispondente ceppo come una singola applicazione sulla piastra "A-input" (preparato in § 2.1).
- Inoculare la piastra "A-output" (preparato in § 2.1). Più precisamente, mescolare delicatamente 30 ml di (D +) con 30 microlitri (P) e 30 pl di (D -) con 30 ml (P) in due tubi separati Eppendorf (nota che le colture usate sono quelle descritte in § 2.4) . Inoculare 20 microlitri della miscele (D + / P) e (D - / P) come punti singoli sulla piastra "A-output". Lasciare le macchie asciugare vicino un becco Bunsen e posizionare la piastra in un incubatore a 37 ° C per un adeguato periodo di tempo durante il quale l'uccisione batterica avviene. Nel caso di P. aeruginosa, un delitto d'efficiente batterica si osserva dopo 5 ore di incubazione quando si confrontano un T6SS attivi con un ceppo T6SS difettoso.
3. Osservazione qualitativa di tRapina a mano armata ha batterica
- Preparare lastre LBA contenenti 40 pg / ml di X-gal per la lettura del saggio. Le piastre si chiamerà "ingresso Lettura" o "R-input" per individuare i batteri isolati o "uscita Lettura" o "R-output" per individuare miste colture batteriche e quindi leggere le prestazioni uccidere. Queste piastre devono anche essere adeguatamente essiccata. Dividere il piatto in quattro parti uguali sulla sua schiena e annotare queste parti 0, 10 -1, 10 -2 e 10 -3, che designa le diluizioni di coltura batterica per individuare le piastre di agar (vedi § 3.3). La diluizione consentirà una valutazione semi-quantitativa del blu / bianco rapporto di batteri all'interno del punto stesso.
- Raccogliere con un'ansa sterile le macchie singole batterica dalla piastra "A-input" (vedi § 2.4) e "A-output" piastra (vedi § 2.5) e risospendere ogni punto in distinte da 1,5 ml provette Eppendorf contenenti 1 ml di TSB. Mettere in un agitatore Eppendorf blocco durante 30 minuti per risospendere in modo efficiente ilbatteri.
- Preparare una serie di 5 volte 3 provette Eppendorf contenente 900 ml di TSB e procedere per ciascuna macchia risospeso a dieci diluizioni seriali fino a 10 -3. Assicurarsi di modificare i puntali per pipette e il vortex 5 secondi tra ogni fase di diluizione.
- Procedere alla individuazione delle diluizioni batteriche preparate in § 3.3 a ben secchi LBA piastre contenenti 40 mg / ml di X-gal iniziando con la più diluita alla sospensione non diluito, rendendo questi i "R-input" piastre per la "A -input "macchie e le" R-uscita "per i piatti" A-"punti di uscita (Figura 2). Vortex brevemente ogni tubo prima di procedere alla individuazione di mantenere una sospensione omogenea batterica. Per la riproducibilità dei risultati, posto 20 pl in triplicato all'interno di un quadrante (Figura 2). La secchezza relativa del piatto è importante in questa fase, in quanto i punti devono rimanere separati singolarmente sul piatto e non scivolare verso l'altro.
Una quantificazione del saggio di competizione è anche possibile in questa fase dell'esperimento. Dopo la fase 3.3, diffuso 100 ul della diluizione 10 -3 LBA su piastre contenenti 40 mg / ml di X-gal. Per la riproducibilità dei risultati, procedere ad una placcatura in triplicato per ogni punto della piastra "A-output". Posizionare le piastre di diluizione in un incubatore ° C durante la notte (corrispondente a 16 ore di incubazione) 37. Procedere al conteggio delle colonie blu (le cellule P che sono rimasti in vita). Risultati tipici ottenuti dopo 5 ore di competizione sono mostrati in Figura 3. - Lasciare aperta la piastra (senza coperchio) all'interno di una zona sterile per consentire l'assorbimento del liquido in eccesso all'interno di ciascun punto.
- Porre la piastra in un incubatore a 37 ° C durante la notte. Durante questo periodo di incubazione, l'uccisione è ancora in corso nel mix (D + / P) in quanto entrambi i ceppi sono ancora in contatto.
- Scattare una fotoo acquisire le piastre per l'analisi di uscita (figura 2). Dove i punti restano in gran parte blu indica che E. coli non è stato ucciso da P. aeruginosa. Questo è il caso in cui E. coli viene miscelato con un T6SS-difettoso P. aeruginosa ceppo (D-) (Figura 2, piastra in basso a destra).
Risultati
Risultati tipici sono mostrati in figura 1 con i ceppi e reagenti descritti nella Tabella 1. Le piastre mostrate in questa figura sono stati analizzati dopo una notte di incubazione. Le "Lettura-Input" piatti mostrano un modello di serie di diluizione per i ceppi utilizzati in questa analisi. Come previsto, la E. coli preda punti (P) iperespressione del gene lacZ appaiono blu su un supporto integrato con X-gal, mentre il donatore P. ceppi aeruginosa (D +, T6SS attivo) e (D -, inattivo T6SS) rimangono bianco. Le "Lettura-output" piatti in cui il mix tra la preda e un ceppo T6SS attivo (D + / P) è stata avvistata mostrano la scomparsa del blu preda indicando così che è stato ucciso. Questo dimostra la capacità del donatore per competere con la preda. La persistenza del colore blu sulla (D - / P) piastra dimostra l'incapacità di un donatore T6SS inattivo per uccidere la preda blu.
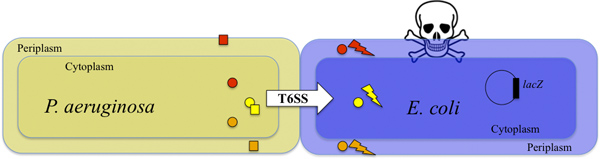
Figura 1. Uccisione di E. coli di T6SS-abile P. aeruginosa. P. aeruginosa inietta tossine nel E. coli in una cellula bersaglio T6SS-dipendente (indicato dalla freccia bianca). Due tossine Tse1 e TSE3 (arancione e cerchi rossi) sono iniettati nel E. coli periplasma e degradare il peptidoglicano 9. La tossina Tse2 (cerchio giallo) viene iniettato nella E. coli citoplasma ed ha una attività batteriostatica 8,10. L'azione combinata delle tossine uccide le cellule bersaglio (lampo e del cranio). La sopravvivenza delle cellule bersaglio può essere rilevata monitorando l'attività del prodotto β-galattosidasi (vedi anche figura 2). P. uneruginosa è protetto contro l'attività delle tossine dalle proteine immunità TSI1, TSI2 e Tsi3 (arancio, quadrati gialli e rossi, rispettivamente) 8,9,10.
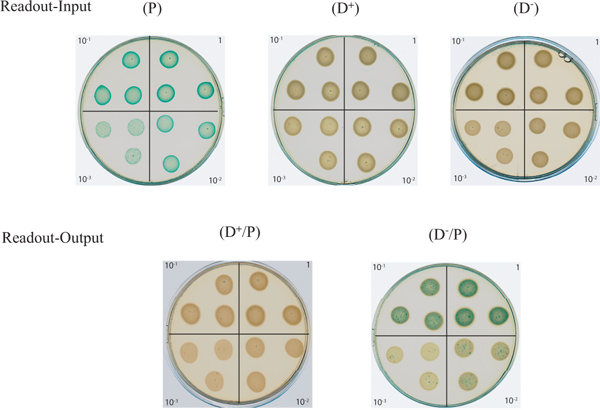
Figura 2. Piastra di saggio Agar per monitorare T6SS-dipendente uccisione batterica In questa figura sono mostrati nella parte superiore dei "Lettura-Input" come piastre delle diluizioni seriali del D -., D +, e le celle di input P. Le celle di immissione P sono blu dovuto alla α-complementazione del gene lacZ e dunque a β-galattosidasi che taglia X-gal. Nella parte inferiore viene visualizzata la "Lettura-uscita" piastre delle diluizioni seriali del mix batterica tra un attivo (D + / P) , o un inattivo (D - / P), T6SS donatore P. aeruginosa con il ceppo E. coli preda. Clicca qui per ingrandire la figura .
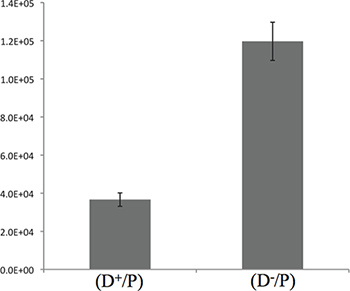
Figura 3. Quantificazione della uccisione di E. coli di P. aeruginosa dopo 5 ore di incubazione. Il grafico presenta il conteggio di E. CFU coli descritto nel punto 3.4. I risultati qui presentati mostrano un 3-fold differenza tra il T6SS + e le T6SS-ceppi, suggerendo che la maggior parte della uccisione avviene durante le prime 5 ore di contatto.
Discussione
Il metodo presentato in questo articolo permette una osservazione visiva di T6SS mediata da attività battericida / bacteriostasis. Il test viene eseguito sulla superficie di una piastra di agar. È stato precedentemente dimostrato che T6SS-dipendente uccisione saggio eseguito con liquido di coltura batterica mista non è efficiente, probabilmente a causa della mancanza di contatto stabile tra i due batteri 8. Il T6SS è creduto per funzionare con un meccanismo simile a quello utilizzato da batteriofagi per iniettare DNA nelle cellule bersaglio 17. In coltura liquida, il tubo-come la struttura del T6SS può rompere più facilmente, inter-batterica contatto può essere perso, e le tossine non sono efficientemente consegnati.
In termini di tempi di incubazione, le 5 prime ore di contatto che ci descrivono la tensione tra donatore e la preda è sufficiente osservare l'uccisione dei batteri tra P. aeruginosa e E. coli, come illustrato in figura 3 . Tuttavia, è opportuno regolare il tempo di incubazione eseguendo una cinetica al fine di ottimizzare le condizioni sperimentali.
Poiché questo metodo è una tecnica basata colore, i risultati di uscita può essere compromessa dalla pigmentazione del ceppo donatore. Per esempio, nel caso di P. aeruginosa, alcuni ceppi producono alti livelli di pigmenti colorati come piocianina e pyoverdine, che possono interferire con la lettura del saggio, rendendo la distinzione dalla preda relativamente difficile. Altri cromogeni β-galattosidasi substrati, come il magenta-gal o il rosso-gal, può essere usato al posto di X-gal (Tabella 1).
Il saggio di competizione può fare uso di altri geni reporter per la lettura. Per esempio, analisi analoga è stata eseguita utilizzando la proteina verde fluorescente marcato prede 12.
La nostra analisi, pur non quantitativa, fornisce una buona indicazionedell'attività T6SS poiché si basa sulla sopravvivenza o l'uccisione di una preda reporter. Questa tecnica presenta il vantaggio di essere facile e conveniente per valutare l'attività battericida / bacteriostasis di T6SSs da qualsiasi specie batteriche. Finora, l'attività del T6SS ha dimostrato contro batteri Gram-negativi e non chiaro esempio di T6SS sensibili batteri Gram-positivi è stato segnalato ancora 12. È anche ovvio che l'incompatibilità nella cultura delle diverse specie batteriche (ad esempio la crescita temperatura, ossigenazione, supporti specifici) è da considerare.
La nostra analisi può anche essere utilizzato per valutare quali componenti T6SS sono assolutamente essenziale poiché anche tracce di una tossina secreta potrebbe essere sufficiente per uccidere la preda. Anche debole attività dei T6SS potrebbe quindi chiaramente essere rilevato dalla nostra analisi, rispetto al normale di test procedura T6SS-dipendente della secrezione con blot supernatante di coltura e occidentaleanalisi. Tuttavia, una corretta unità formanti colonie (CFU) conteggio è ancora necessaria per quantificazione accurata di questa attività T6SS.
Divulgazioni
Non abbiamo nulla da rivelare.
Riconoscimenti
Questo lavoro è stato finanziato dal Wellcome Trust sovvenzione WT091939MA. Alain Filloux è sostenuta dalla Royal Society.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reagente / materiale | Azienda | Numero di catalogo | Commenti |
| P. aeruginosa PAKΔ RET (D +) (T6SS attivi) | Lab ceppo | Descritto in riferimento 16 | |
| P. aeruginosa PAKΔ RET Δ H1-T6SS (D -) (T6SS inattivi) | Questo studio | L'H1-T6SS cluster (che comprende i geni PA0070 a PA0095) è stato cancellato dallo scambio allelico seguendo la procedura descritta nel Manuale 18. Il frammento mutatore è stato generato con il seguente set di primer: I primer frammento Up: 5'-ATGGTCAACGACATGGAGCTGGAG-3 ', e 5'CGAGGCCGATCAGGCCTTCAGAACTGA-3 '. I primer frammento di Down: 5'-TCAGGCCTTCAGAACTGAAGCGGCGCA-3 ', 5'-GGTGGCGTTCAACAGTTCCATGTC-3 ' | |
| E.coli DH5a | Invitrogen | 18258-012 | F-φ80 lac ZΔM15 Δ (lac ZYA-arg F) U169 rec A1 A1 fine hsd R17 (r k -, k + m) pho A supporto E44 λ-thi -1 Gyr A96 rel A1 |
| pBluescript II SK (+) | Agilent | 212205 | Questo vettore esprime il peptide di α β-galattosidasi utilizzato per α-complementazione. |
| X-gal | Invitrogen | 15520-018 | Utilizzare a 40 mcg / ml |
| Luria Bertani agar | Merck-chimici | 1.10283.0500 | |
| TSB (caseina di soia brodo) | Oxoid | CM109 | |
| Vortex shaker Genius 3 | IKA | 3340000 | |
| Scanner | Epson | V700 | |
| Spettrofotometro | WPA Biowave | CO8000 cellulare densimetro | |
| Magenta-gal | Bioworld | 30350001-1 (715241) | |
| Red-gal | La ricerca Organics | 1364c | |
Tabella 1. Strain, plasmide, materiali e reagenti utilizzati. |
Riferimenti
- Filloux, A., et al. The bacterial type VI secretion machine: yet another player for protein transport across membranes. Microbiology. 154 (6), 1570 (2008).
- Cascales, E., Cambillau, C. Structural biology of type VI secretion systems. Philos. Trans. R. Soc. Lond. B. Biol. Sci. 367 (1592), 1102 (2012).
- Leiman, P. G., et al. Morphogenesis of the T4 tail and tail fibers. Virol. J. 7, 355 (2010).
- Ballister, E. R., et al. In vitro self-assembly of tailorable nanotubes from a simple protein building block. Proc. Natl. Acad. Sci. U.S.A. 105 (10), 3733 (2008).
- Leiman, P. G., et al. Type VI secretion apparatus and phage tail-associated protein complexes share a common evolutionary origin. Proc. Natl. Acad. Sci. U.S.A. 106 (11), 4154 (2009).
- Pukatzki, S., et al. Type VI secretion system translocates a phage tail spike-like protein into target cells where it cross-links actin. Proc. Natl. Acad. Sci. U.S.A. 104 (39), 15508 (2007).
- Ma, A. T., et al. Translocation of a Vibrio cholerae type VI secretion effector requires bacterial endocytosis by host cells. Cell Host Microbe. 5 (3), 234 (2009).
- Hood, R. D., et al. A type VI secretion system of Pseudomonas aeruginosa targets a toxin to bacteria. Cell Host Microbe. 7 (1), 25 (2010).
- Russell, A. B., et al. Type VI secretion delivers bacteriolytic effectors to target cells. Nature. 475 (7356), 343 (2011).
- Li, M., et al. Structural basis for type VI secretion effector recognition by a cognate immunity protein. PLoS Pathog. 8 (4), e1002613 (2012).
- Zheng, J., et al. Genetic analysis of anti-amoebae and anti-bacterial activities of the type VI secretion system in Vibrio cholerae. PLoS One. 6 (8), (2011).
- Schwarz, S., et al. Burkholderia type VI secretion systems have distinct roles in eukaryotic and bacterial cell interactions. PLoS Pathog. 6 (8), e23876 (2010).
- Murdoch, S. L., et al. The opportunistic pathogen Serratia marcescens utilizes type VI secretion to target bacterial competitors. J. Bacteriol. 193 (21), 6057 (2011).
- Boyer, F., et al. Dissecting the bacterial type VI secretion system by a genome wide in silico analysis: what can be learned from available microbial genomic resources?. BMC Genomics. 10, 104 (2009).
- Maniatis, T., et al. . Molecular cloning: A Laboratory Manual. , (1982).
- Mougous, J. D., et al. A virulence locus of Pseudomonas aeruginosa encodes a protein secretion apparatus. Science. 312 (5779), 1526 (2006).
- Records, A. R., et al. The type VI secretion system: a multipurpose delivery system with a phage-like machinery. Mol. Plant Microbe Interact. 24 (7), 751 (2011).
- Hachani, A., et al. Type VI secretion system in Pseudomonas aeruginosa: secretion and multimerization of VgrG proteins. J. Biol. Chem. 286 (14), 12317 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon