Method Article
Sonde-basierte Echtzeit-PCR Ansätze zur quantitativen Messung von microRNAs
In diesem Artikel
Zusammenfassung
Zirkulierende microRNAs haben in letzter Zeit als vielversprechend und neue Biomarker für verschiedene Krebsarten und anderen Krankheiten. Das Ziel dieses Artikels ist es, drei verschiedene Sonden-basierten Echtzeit-PCR-Plattformen und Methoden, die zur Verfügung zu quantifizieren und zu bestimmen, die Fülle der zirkulierenden microRNAs sind zu diskutieren.
Zusammenfassung
Sondenbasierten quantitative PCR (qPCR) ist ein bevorzugtes Verfahren zum Messen Transkript Fluss, da es eines der am meisten empfindlichen Nachweisverfahren, die eine genaue und reproduzierbare Analyse liefert. Sondenbasierte Chemie bietet den geringsten Grundfluoreszenz im Vergleich zu anderen (Farbstoff) Chemien. Gegenwärtig gibt es verschiedene Betriebssysteme, dass die Verwendung sondenbasierte Chemie Transkript Fluss zu quantifizieren. qPCR in einer 96-Well-Platte ist die routinemßig verwendete Verfahren ist jedoch nur ein Maximum von 96 Proben oder miRNAs kann in einem einzigen Durchlauf getestet werden. Dies ist zeitraubend und mühsam, wenn eine große Anzahl von Proben / miRNAs analysiert werden. Hochdurchsondenbasierten Plattformen wie Mikrofluidik (zB TaqMan Array Card) und nanofluidics Arrays (zB Openarray) bieten eine einfache reproduzierbar und effizient die Fülle von mehreren mikroRNAs detektieren in einer großen Anzahl von Proben in einer kurzen Zeit. Hier zeigen wir, den Versuchsaufbau einnd Protokoll für die miRNA-Quantifizierung von Serum oder Plasma-EDTA-Proben, mit Sonde-basierte Chemie und drei verschiedenen Plattformen (96-Well-Platte, Mikrofluidik und Nanofluidik Arrays) mit steigendem Durchsatz.
Einleitung
Mikro-RNAs (miRNAs) sind ~ 22 Nukleotid nicht-kodierenden (NC-) RNAs, die als Regulatoren der Genexpression 1-3. Die meisten miRNAs bei Tieren funktionieren durch sequenzspezifische Basenpaarung mit einer mRNA, zur Förderung der 3'-UTR, die negative Regulation der Genexpression 2-4 führt. Dies erfolgt in der Regel über eine Hemmung der mRNA-Translation oder durch ribosomale Drop-off. miRNAs Umlauf ist gezeigt worden, Biomarkern in Forschungs- und klinischen Gebieten für eine Vielzahl von Krankheiten, wie Diabetes 5-7, Eierstock- 8, 9 Prostata und Brustkrebs 10,11, Hepatitis B 12 und anderen Autoimmunerkrankungen 13 sein. Es wurden Untersuchungen durchgeführt, um reichlich miRNAs in verschiedenen Zellen oder Gewebe zu identifizieren, ebenso wie im Umlauf aus menschlichem Plasma und Serumproben, die besser zugänglich und weniger invasiv ist 9,11-15.
Verschiedene Methoden der miRNA Quantifizierung wurden established mit mehreren Plattformen, wie beispielsweise die Standard-96-Well-Platte Plattform 4,12,16-18, der Mikrofluidik Kartenplattform 12,18-23 und der Nanofluidik Array-Plattform 17,24. Quantitative real-time PCR (qPCR) bietet die Möglichkeit, relative oder absolute Zahl der Transkripte Messung mit mehreren (Farbstoff- oder Sonde-basierte) Chemie. Sonde-basierte Echtzeit-PCR-Chemie bietet den Vorteil der niedrigen Hintergrundfluoreszenz und eine hohe Empfindlichkeit, eine einzige Abschrift Kopie zu erkennen. Es ist relativ kostengünstig, einfach zu bedienen und sehr gut reproduzierbar, so dass es eine bevorzugte Methode zur Quantifizierung und Bestimmung miRNA-Expression 25. Probe qPCR-basierte Verfahren umfasst im Allgemeinen zwei Schritte: die reverse Transkription (RT) und qPCR 4,26,27. RT ist, wo der Stamm-Schleifen-RT-Primer an einem reifen oder primären miRNA-Molekül hybridisiert und komplementären (c) DNA umgewandelt. Quantifizierung der cDNA-Produkt wird dann unter Verwendung miRNA-spezifischen PCR Primern26-28. Das Prinzip der sondenbasierten qPCR basiert auf dem Nachweis von Komplementärstrangverlängerung in Echtzeit, was die Hydrolyse des fluoreszenzmarkierten Sonde beinhaltet bezogen. Diese Sonden wurden entwickelt, um einen fluoreszierenden Reporter und einen Quencher, die nur voneinander entfernt sind, um FRET (Fluorescence Resonance Energy Transfer) erlauben. Detektion der Emission von der Fluoreszenzreporter (Emitter) von der Nähe des Löschermolekül maskiert. Wenn Taq-Polymerase (pol) sich von der Stromaufwärts-Primer und erreicht eine Gabel (das 5'-Ende der Sonde), hydrolysiert das Taq Pol-Exonukleaseaktivität der Sonde, was zu einer physikalischen Dissoziations / Trennung des fluoreszierenden Emitters vom Quencher. Diese Version von einem einzigen Molekül Fluoreszenzemitter wird durch den Detektor erfasst und als eine inkrementelle Erhöhung des Fluoreszenzsignals von diesem gut / Reaktions dargestellt. Der Anstieg in der Fluoreszenz ist proportional zu der Menge an PCR-Produkt erzeugt wird, so dass eine genaue quantification des amplifizierten Ziel 26,28.
Mit zunehmender Nachfrage miRNA Quantifizierung mittlere bis hohe Durchsatz-Technologien wurden entwickelt, damit eine größere Anzahl von Proben in einer kurzen Zeit verarbeitet werden. TaqMan Low Density Array (TLDA) ist ein mittelDurch innovative mikrofluidische Design auf Sondenbasis qPCR Chemie bietet eine Erhöhung der Zahl von miRNAs auf einer Platte analysiert. TLDA beinhalten die Verwendung eines vorgegebenen Pool von RT-Primer, die verwendet werden, um die cDNA zu synthetisieren. Diese cDNAs werden anschließend in einen kunden 384er mikrofluidischer Karte gesponnen, um die Expression mehrerer miRNAs mittels qPCR 22,26,29 bestimmen. Jede Vertiefung der Karte enthält getrockneten Primer und Sonden zur spezifischen miRNA (en) zu verstärken, also bis zu 384 Reaktionen können in Einzel TLDA Karte 26 verarbeitet werden.
Die nanofluidics Array ein Hochdurchsatz-Plattform, die zum Nachweis von Gen-Transkripte verwendet wird 24 unter Verwendung der gleichen Sonde basierenden Chemie. Es nutzt eine proprietäre Matrix mit hydrophoben hydrophilen Wechselwirkungen einfache Laden der 33 Nanoliter-Reaktionsmischung in einer Anordnung von 3072 Durchgangslöcher auf einem Edelstahl-Schlitten 24 zu erleichtern. Dieser Artikel befasst sich zeigen, wie diese Methoden zur Quantifizierung von miRNAs im Serum / Plasma durchgeführt werden und die kritischen Faktoren, die berücksichtigt werden müssen bei der Durchführung und Interpretation dieser Daten. Getroffen, um Konto, werden ihre individuellen Vor- und Nachteile in diesem Artikel beschrieben werden.
Protokoll
Die Gesamt-RNA aus Serum mit einem in unserem Labor 30 oder mit anderen kommerziell erhältlichen Kits festgelegten Protokoll isoliert werden.
HINWEIS: Zusätzliche interaktive Tabellen für die Berechnung der Reaktionsvolumina in jedem Experiment (mit 5% Überschussvolumen entfielen Pipettieren im Lieferumfang enthalten) zur Verfügung gestellt.
1. Sonde-basierte Echtzeit-qPCR mit einem Standard-96-Well-Platte Platform
- cDNA-Synthese (reverse Transkription) auf Serum / Plasma-RNA-Proben für die miRNA-
- Berechnen Sie und nehmen Sie 10 ng RNA für jede cDNA-Synthesereaktion. Hinzufügen Nuklease-freies Wasser bis zu einem Endvolumen von 10 ng RNA auf 1,67 & mgr; l (für ein 5 & mgr; l-Reaktion) zu bringen. Halten Sie Proben auf Eis.
- Wählen Sie das miRNA und tauen ihre RT Primer.
- Auftauen RT Reagenzienmix Komponenten: RT Buffer (10X), dNTPs (100 mM), RNase Inhibitor (20 U / ul). Bewahren Sie niemals statt Enzym (rekombinante Moloney Murine Leukemia Virus(RMoMuLV)) (recombinanat Moloney murine leukemia virus (rMoMuLV) Reverse Transkriptase (50 U / & mgr; l) auf Eis. Bei -20 ° C, bis sie gebraucht oder in einem Gefrierschrank Block.
- Bereiten RT Reagenzienmix auf Eis, wie in Tabelle 1A beschrieben. Es wird empfohlen, mindestens 5% herzustellen überschüssiges Volumen für Pipettierfehlers kompensieren.
- Hinzufügen 2,33 ul des RT Reagenzienmix jeder in eine 0,2 ml PCR-Rohr / Boden.
- Vortex die RT-Primer Röhrchen bei 10.000 × g mischen, dann kurz zentrifugieren für 10 Sekunden. 1 ul der miRNA spezifische RT-Primer an ihre jeweiligen PCR-Gefäße oder Platte. Hinzufügen 1,67 ul der verdünnten RNA ihren jeweiligen PCR-Röhrchen oder Vertiefungen in der Platte. Führen Sie alle Ergänzungen auf Eis.
- Zentrifugieren Sie das Reaktionsrohr oder Platte bei 1950 xg für 5 Minuten bei 4 ° C.
- 16 ° C für 30 min, 42 ° C für 30 min, 85 °: die miRNA cDNA Syntheseprogramm mit folgenden Einstellbedingungen Auf ThermocyclerC für 5 min und 4 ° C gehalten.
- Eingestellt Reaktionsvolumen auf 10 ul. Legen Sie die Reaktionsröhrchen oder Platte in Thermocycler. Starten Sie den RT laufen. Speichern die cDNA (RT) Reaktion bei -20 ° C, wenn Echtzeit-PCR-Amplifikation nicht unmittelbar fort.
- Sonde-basierte Echtzeit-qPCR zum Nachweis von reifen miRNAs
- Tauen Sie die markierten Sonde-basierte qPCR-Assays (20X) für die jeweilige Produkt RT.
- Mischen Sie den Anstand alleinige Lösung von Echtzeit-PCR (qPCR Reagenzienmix) durch Schwenken der Flasche.
- Bereiten Sie die qPCR Reagenzienmix für jedes miRNA analysiert werden. Zur Herstellung der qPCR erhalten eine sterile 1,5-ml-Mikrozentrifugenröhrchen für jede Probe miRNA und fügen Sie die Komponenten in jedes Röhrchen, wie in Tabelle 1B beschrieben. Es wird empfohlen, mindestens 5% herzustellen überschüssiges Volumen für Pipettierfehlers kompensieren.
- Hinzufügen 4,2 ul der jeweiligen qPCR Reagenzgemisch in jede Vertiefung einer optischen Platte mit 96 Vertiefungen (oder Röhren). In 0,8 ulder jeweiligen cDNA-Reaktion auf jeweilige gut (in Schritt 1.1 für jede miRNAs synthetisiert).
- Verschließen Sie die Platte mit den entsprechenden optischen Abdeckung. Zentrifugieren Sie die Platte (oder Rohre) bei 1950 xg für 5 Minuten bei 4 ° C
- Schalten Sie den Computer, 96-Well-Platte / Mikrofluidik Array Leser und schließlich die Software zu starten. Stellen Sie sicher, dass die Maschinen korrekt angeschlossen sind und korrekt Block und Heizdeckel (für schnelle Platte mit 96 Vertiefungen) gegeben sind.
- Wählen Sie Experiment als 96-Loch schnell Block (0,1 ml), Standardkurve, TaqMan-Reagenzien und Schnelllauf. 95 ° C für 20 sec, 50 Zyklen (95 ° C für 1 s, 60 ° C für 30 sec): bis qPCR auf der Echtzeit-PCR-System mit den folgenden Programmzyklusbedingungen eingestellt.
- Eingestellt Reaktionsvolumen auf 10 ul. Laden Sie das Reaktionsrohr oder Platte in das Gerät. Drücken Sie "Start Run". Das Programm dauert etwa 1 Stunde in Anspruch nehmen.
2. Sondenbasierten Mikrofluidik Array-Karte (Card A und B)
HINWEIS: Sonde-basierte miRNA-Panel kommt als ein Satz von zwei 384-Well-Mikrofluid-Karten (Array Karte A und Array Karte B). Jede Karte enthält getrockneten Primer und Sonden für bis zu 380 miRNAs und Kontrollen. cDNA-Produkt (mit oder ohne Vorverstärkung), spezifisch für A-Karte oder einer Karte B auf die jeweilige Anordnung zur Echtzeit-PCR geladen.
- Reverse Transkription (RT)
- Verwenden Sie dieses Protokoll für eine Gesamt-RNA-Eingang 1-1.000 ng. Ist der Eingang zwischen 1 bis 350 ng, führen Sie eine Prä-Amplifikation (Vorverstärker) Schritt. Für Eingaben über 350 ng, laden cDNA direkt auf den Array-Karten ohne Vorverstärker.
HINWEIS: Serum miRNA Profiling, mit 100 ng Gesamt-RNA zu starten. Eine vollständige miRNA-Profil, laufen zwei vordefinierten Pools von RT-Primer-Sets von RT-Reaktionen (Pool A und Pool B) pro Probe. - Tauen Sie die folgenden Reagenzien auf Eis. RT Primer (10x): Pool A und Pool B, dNTPs mit dTTP (100 mM), RT Buffer (10x), MgCl 2 (25 mM), RNase Inhibitor (20 U / &# 181; l). Halten Sie das Enzym (rekombinante Moloney Murine Leukemia Virus (rMoMuLV)) Reverse-Transkriptase (50 U / ul) auf Eis. Bewahren Sie es bei -20 ° C, bis sie benötigt.
- Vorsichtig vortexen alle Reagenzien, mit Ausnahme des Enzyms, und dann kurz die Röhrchen bei 10.000 × g zentrifugieren für 10 Sekunden.
- Kombinieren Reagenzien, wie in Tabelle 2A beschrieben, in zwei Röhrchen; ein Rohr für Pool A, die andere für Pool B. Jedes Rohr wird lediglich eine vordefinierte Pool von RT-Primer-Set enthalten, die entweder aus Pool A oder aus Pool B. Es wird empfohlen, mindestens 5% herzustellen überschüssiges Volumen um Pipettierfehler zu kompensieren .
- Kehren Sie zu mischen und dann kurz zentrifugieren bei 10.000 × g für 10 Sekunden. Aliquoten 100 ng jeder RNA-Probe in ein neues Röhrchen, dann fügen Sie eine entsprechende Volumen Nuklease-freies Wasser, um insgesamt 3 ul machen.
- Aliquot 4,5 ul der geeigneten RT Reagenzienmix in ihre Röhrchen. Kehren Sie zu mischen und dann kurz bei 10.000 × g zentrifugieren für 10 Sekunden. Inkubieren auf Eisfür 5 min.
- Platz Proben in einem Thermocycler und starten Sie die RT unter den folgenden Bedingungen: 40 Zyklen (16 ° C für 2 min, 42 ° C für 1 min, 50 ° C für 1 sec), 85 ° C für 5 min bei 4 ° halten C. Speicher cDNA unter Verwendung dieser vordefinierten Pool von RT-Primern bei -15 bis -25 ° C oder sofort verwendet.
- Verwenden Sie dieses Protokoll für eine Gesamt-RNA-Eingang 1-1.000 ng. Ist der Eingang zwischen 1 bis 350 ng, führen Sie eine Prä-Amplifikation (Vorverstärker) Schritt. Für Eingaben über 350 ng, laden cDNA direkt auf den Array-Karten ohne Vorverstärker.
- Vorverstärkung
- Auftauen vordefinierten Pool von Vorverstärker Primer auf Eis. Vorsichtig Vortex Primer und dann kurz zentrifugieren bei 10.000 × g für 10 Sekunden. Swirl TaqMan PreAmp Reagenzienmix (2X) durch leichtes Ausschlagen, um zu mischen.
- Kombinieren Reagenzien, wie in Tabelle 2B beschrieben, in zwei Röhrchen; ein Rohr für Pool A, die andere für Pool B.
HINWEIS: Wie oben beschrieben, wird Pool A Vorverstärker-Primer an Rohr A gehen und Pool B Primer sollte Rohr B. gehen Es wird empfohlen, mindestens 5% herzustellen überschüssiges Volumen um Pipettierfehler zu kompensieren. - Kehren Sie zu mischen und dann kurz bei 10.000 × g zentrifugieren für 10 Sekunden.Aliquoten 22,5 ul der entsprechenden PreAmp Reagenzienmix in neue Schläuche. 2,5 ul Aliquot der cDNA-Probe (im RT-Schritt hergestellt) in das jeweilige Rohr.
- Kehren Sie zu mischen und dann kurz bei 10.000 × g zentrifugieren für 10 Sekunden. Inkubieren auf Eis für 5 Minuten. Platz Proben in einem Thermocycler und starten Sie die Pre-Amp-Zyklus. Zyklusbedingungen: 95 ° C für 10 min, 55 ° C für 2 min, 72 ° C für 2 min, 12 Zyklen (95 ° C für 15 sec, 60 ° C für 4 min), 99,9 ° C für 10 min, halten bei 4 ° C.
- Invert vorverstärkt cDNA zu mischen, und dann kurz Zentrifuge bei 10.000 × g für 10 Sekunden. Hinzufügen 75 ul 0,1x TE-Puffer (pH 8,0) vor, amplifizierte cDNA (1: 4-Verdünnung). Invert verdünnter Vorverstärker Proben zu mischen, und dann kurz bei 10.000 × g zentrifugieren für 10 Sekunden. Speicher verdünnt vorverstärkte cDNA bei -15 bis -25 ° C für bis zu einer Woche, oder sofort verwendet.
- Laden Mikrofluidik-Karten und Durchführung qPCR
- Halten Sie die MikrofluidikmiRNA Karten außerhalb für mindestens eine halbe Stunde auf Raumtemperatur kommen. Auftauen verdünnter vorverstärkte cDNA auf Eis und mischen durch Umdrehen der Röhrchen, gefolgt von kurzen Spin. Mischen Sie qPCR Reagenzienmix durch Schwenken der Flasche.
- In neuen Schlauch mit 450 ul der qPCR Reagenzienmix bis 9 & mgr; l der verdünnten vorverstärkte cDNA. Für Karte A, verwenden vorverstärkte Produkt hergestellt unter Verwendung von Primer Pool A. Fügen Sie 441 ul Nuklease-freies Wasser zu bilden das Endvolumen auf 900 ul. Das Röhrchen zu mischen, und dann kurz bei 10.000 × g zentrifugieren für 10 Sekunden.
- Entfernen Sie die TLDA Karte aus der Verpackung (wenn es Raumtemperatur erreicht hat) und legen Sie sie auf eine saubere Fläche mit Folienseite nach unten. Füge 100 & mgr; l PCR-Reaktionsmischung in jeder der 8 Füllöffnungen auf der Karte. Es gibt 2 Ports an jedem der Behälter (8 Reservoirs insgesamt auf jeder Karte).
HINWEIS: Die Einfüllöffnung ist das größere Loch, wo der Reaktionsmischung zugefügt, während die Belüftungsöffnung ist das kleinere Loch. Jedes miRNA TLDA Karte hat 8 Reservoirs, die jeweils zu 48 Vertiefungen (24 Vertiefungen in einer Spalte x 2 Spalten), wodurch das Laden alle 384 Vertiefungen in der Karte mit dem gleichen PCR-Reaktionsmischung. Stellen Sie sicher, dass die Gruppe A und Gruppe B von jeder Probe auf entsprechenden TLDA Karten geladen. Setzen Sie den Array-Karte in den Fach Mikrofluidik Array Kartenhalter Eimer in der Zentrifuge. - Für die Mikrofluidik Array-Karten, mit einem geeigneten Zentrifuge (wie Heraeus Multifuge 3SR, 230V Zentrifuge) und spezifische Eimer (wie "TaqMan Array Card"). Jede Schaufel kann bis zu 3 Karten (beladen / leer) zu halten. Achten Sie immer darauf, dass alle 3 Slots eines Becher belegt sind und die Schaufel, indem ähnliche Eimer (Eimer dies sollte auch 3 Karten, entweder leer oder voll enthalten) in die entgegengesetzte Schlitz der Zentrifuge ausgeglichen. Beim Aufsetzen der Karte in der Eimerhalterung, stellen Sie sicher, dass die 8 Stauseen projizieren nach oben und Reaktionsfeldern vor der Außenwand der Zentrifuge.
- Dreh die Karten bei 331 × g für 1 min bei Raumtemperatur. Nachersten Spin, öffnen Sie die Zentrifuge und optisch sicher, dass die Reaktionsmischung wurde durch die 384 Vertiefungen verzichtet. Wiederholen Sie den Spin bei gleichen Einstellungen für ein weiteres Mal. Entfernen Sie die Karte aus Eimer und sicherzustellen, dass das Niveau der Reaktionsmischung in jedem der acht Stauseen einheitlich ist. Etwaige Unstimmigkeiten bei den in den Behältern links Flüssigkeitsvolumen machen die Karte ungeeignet zum weiteren Gebrauch.
- Mikrofluidik Array Karten benötigen eine spezielle Versiegelung, die eine Präzisionsstiftaufbau (Schlitten), die Fluidverteilungskanäle des Arrays zu versiegeln und die Reaktionsmischung gleichmäßig verteilt in alle Vertiefungen (Gesamtreaktionsvolumen 1 ul / Vertiefung) hat.
- Bringen Sie den Wagen in die Ausgangsposition und legen Sie die Karte in den geladenen Siegelgerät mit Folienseite nach oben und standen Schlange, um den Stift Stifte auf der Versiegelung. In einer langsamen, stetigen und einzigen einheitlichen Schlaganfall, muss der Führungsschlitten über die Karte, bis sie den Endpunkt der Versiegelung erreicht. Nehmen Sie den verschlossenen Array-Karte und dann schneiden Sie die Behälters von der Karte mit einer Schere.
- Überprüfen, dass der Block, beheizte Deckel und Probenträger in der Mikrofluidik Array Echtzeit-PCR-System / Maschine installiert.
- Schalten Sie den Computer, dann die Mikrofluidik-Array-System und schließlich die Software zu starten. Stellen Sie sicher, dass die Maschinen korrekt angeschlossen sind. Wählen Sie Experiment als Array Karte, Standardkurve, TaqMan-Reagenzien und Standard-Modus. Importieren Sie die Setup-Dateien für eine der Karten A und B. Karte Speichern Sie die Datei.
- Das verschlossene Karte im Gerät Tablett mit gut A1 an der oberen linken Ecke und Barcode in Richtung der Vorderseite des Gerätes. Drücken Sie "Start Ausführen". Das Programm wird etwa 2 Stunden dauern.
3. Probe basierte Nanofluidik Menschen miRNA-Panel
- Reverse Transkription (RT)
- Verwenden Sie dieses Protokoll für eine Gesamt-RNA Eingang von 50-200 ng jedoch 100 ng ist optimal für die meisten Proben. Eine vollständige miRNA-Profil, laufen zwei vordefined Pools von RT-Primer-Sets von RT-Reaktionen (Pool A und Pool B) pro Probe.
- Tauen Sie die Reagenzien auf Eis: vordefinierten Pool von RT-Primer-Sets (10X), dNTPs mit dTTP (100 mM), RT Buffer (10x), MgCl 2, (25 mM), RNase Inhibitor (20 U / ul). Das Enzym (rekombinantes Moloney Murine Leukemia Virus (rMoMuLV) Reverse-Transkriptase (50 U / ul) auf Eis aufbewahren. Bewahren Sie es bei -20 ° C, bis sie benötigt. Vorsichtig vortexen alle Reagenzien, mit Ausnahme des Enzyms, und kurz zentrifugieren bei 10.000 g für 10 sec.
- Kombinieren Reagenzien, wie in Tabelle 3A beschrieben, in zwei Röhrchen; ein Rohr für Pool A, die andere für Pool B. Jedes Rohr wird lediglich eine vordefinierte Pool von RT-Primer-Set enthalten, die entweder aus Pool A oder aus Pool B. Es wird empfohlen, mindestens 5% herzustellen Überschussvolumen für Pipettieren auszugleichen Fehler.
- Pipette zu mischen und dann kurz zentrifugieren bei 10.000 × g für 10 Sekunden. Aliquoten 100 ng jeder RNA-Probe in ein neues Röhrchen, dann fügen Sie eine geeignete Lautstärkevon Nuklease-freies Wasser, um insgesamt 3 ul machen. Aliquot 4,5 ul der geeigneten RT Reagenzienmix in das jeweilige Rohr.
- Kehren Sie zu mischen und dann kurz bei 10.000 × g zentrifugieren für 10 Sekunden. Inkubieren auf Eis für 5 min. Platz Proben in einem Thermocycler und starten Sie die RT-Programm. Zyklusbedingungen: 40 Zyklen (16 ° C für 2 min, 42 ° C für 1 min, 50 ° C für 1 sec), 85 ° C für 5 min, Halten bei 4 ° C. Shop cDNA, die unter Verwendung dieser vordefinierten Pool von RT-Primer bei -15 bis -25 ° C eingestellt und sofort nutzen.
- Vorverstärkung (Vorverstärker)
- Auftauen vordefinierten Pool von Vorverstärker Primer auf Eis gesetzt. Vorsichtig Vortex Primer, dann kurz zentrifugieren bei 10.000 × g für 10 Sekunden. Swirl TaqMan PreAmp Reagenzienmix (2X) zu mischen.
- Kombinieren Reagenzien, wie in Tabelle 3B beschrieben, in zwei Röhrchen; ein Rohr für Pool A, die andere für Pool B. Jedes Röhrchen wird lediglich eine vordefinierte Pool von RT-Primersätze enthalten, die entweder from Pool A oder aus Pool B. Es wird empfohlen, mindestens 5% herzustellen überschüssiges Volumen um zum Pipettieren Fehler zu kompensieren.
- Pipette zu mischen, und dann kurz zentrifugieren bei 10.000 × g für 10 Sekunden. Aliquoten 22,5 ul der entsprechenden PreAmp Reagenzienmix in neue Schläuche. 2,5 ul Aliquot der cDNA-Probe in die entsprechenden Röhrchen. Kehren Sie zu mischen und dann kurz die Röhrchen bei 10.000 × g zentrifugieren für 10 Sekunden. Inkubieren auf Eis für 5 min.
- Platz Proben in einem Thermocycler und starten Sie die Pre-Amp. Stellen Reaktionsvolumen auf 25 ul. Zyklusbedingungen: 95 ° C für 10 min, 55 ° C für 2 min, 72 ° C für 2 min, 12 Zyklen (95 ° C für 15 sec, 60 ° C für 4 min), 99,9 ° C für 10 min, halten bei 4 ° C.
- Invert vorverstärkt cDNA zu mischen, und dann kurz Zentrifuge bei 10.000 × g für 10 Sekunden. In neuen Röhren hinzufügen 4 ul vorverstärkte cDNA bis 156 & mgr; l von 0,1 x TE-Puffer pH 8,0 (1:40 Verdünnung). Invert verdünnter Vorverstärker Proben zu mischen, und dann kurz centrifuge bei 10.000 × g für 10 sec. Shop verwässert und unverwässert vorverstärkte cDNA bei -15 bis -25 ° C bis zu einer Woche, oder sofort ab.
- Laden Nanofluidik Arrays und darstellende qPCR
- Laden Sie die entsprechenden Plattendatei (.tpf) von der Website 31 mit dem Nanofluidik Array Schiebeseriennummer. Diese enthält die Laufinformationen für die spezifischen Nanofluidik Array Objektträger.
- Thaw verdünnter vorverstärkte cDNA und TaqMan Real-Time qPCR Reagenzienmix (wenn Sie zum ersten Mal) auf Eis. Mischen Sie qPCR Reagenzienmix durch Schwenken der Flasche. In neuen Röhren fügen 22,5 ul der qPCR Reagenzienmix auf 22,5 ul verdünnter vorverstärkte cDNA. Vorsichtig Vortex mischen, und dann kurz bei 10.000 × g zentrifugieren für 10 Sekunden.
- Aliquot 5 ul jeder Probe in 8 Vertiefungen (2 Spalten, 4 Zeilen) des Nanofluidik Array Workflow 384er Probenplatte. Stellen Sie sicher, dass die Gruppe A und Gruppe B von jeder Probe sind in benachbarten 8-Well-Blocks (siehe FigurE 1 oder Ergänzungs Excel-Tabelle für das Layout).
- Jede Probenplatte kann bis zu acht Dias im Wert von Proben enthalten. Allerdings kann das System für die Nanofluidik Array nur verarbeiten 4 Nanofluidik Arrays in einem einzigen Durchlauf. Wenn mehr als vier Dias im Wert von Proben werden auf einer Probenplatte geladen werden, stellen Sie sicher, bleiben die übrigen Teile sind versiegelt. Dichtung mit Openarray Probenfolie verschließen.
HINWEIS: Es ist ratsam, vorgeschnitten die Versiegelung in die benötigten Teile, so dass die Abschnitte können versiegelt / werden individuell entsiegelt, um Verdunstung zu reduzieren. Alternativ dazu kann die Platte mit einer intakten Versiegelung versiegelt werden, und dann Abschnitte können einzeln heraus beim Laden geschnitten werden.
- Jede Probenplatte kann bis zu acht Dias im Wert von Proben enthalten. Allerdings kann das System für die Nanofluidik Array nur verarbeiten 4 Nanofluidik Arrays in einem einzigen Durchlauf. Wenn mehr als vier Dias im Wert von Proben werden auf einer Probenplatte geladen werden, stellen Sie sicher, bleiben die übrigen Teile sind versiegelt. Dichtung mit Openarray Probenfolie verschließen.
- Zentrifugenplatte bei 490 × g für 1 min bei 4 ° C. Laden Sie die Nanofluidik Array gleitet innerhalb 1 Stunde. Aufgrund der begrenzten Zeit erlaubt, um die Folien zu versiegeln, bitte nur eine Folie gleichzeitig zu laden. Entfernen Sie die Nanofluidik Array Rutsche aus dem Gefrierschrank und lassen Sie ihn auf Raumtemperatur kommen,peratur (~ 15 min).
- Überprüfen, dass der Block, beheizte Deckel und Probenträger in der Nanofluidik Array-System installiert. Schalten Sie den Computer und Echtzeit-PCR-System und dem Ladesystem. Zugriff auf die jeweilige Software und stellen Sie sicher, dass die Maschinen verbunden sind. Entfernen Sie die Ladesystem Verbrauchsmaterialien (Array Schiebedeckel, Plug-and-Immersionsflüssigkeit) aus der Verpackung.
- Ziehen Sie vorsichtig am Kolben der Spritze Immersionsflüssigkeit zu lockern. Entfernen Sie die Kappe, setzen Spitze auf und bündig Luft von der Spitze. Platzieren Sie die Ladesystem-Tipps in der Maschine entfernen und Deckel. Zeigen Probenplatte in PCR-System.
- Handschuhe anziehen. Stellen Sie sicher, sie eng anliegende, um das Risiko einer versehentlichen Kennzeichnung der Schiebedeckel zu minimieren. Vorsichtig öffnen Rutsche Verpackung. Langsam Spitze Abgleiten in die Hand. Berühren Sie nicht den oberen Rand der Folie.
- Zeigen Rutsche in den PCR-System, mit dem Strichcode auf der linken Seite. Entfernen Versiegelung von dem Teil der Probenplatte für die Beladung vorgesehen. Verwenden Sie das LadesystemSoftware, um die Diashow Barcode einzugeben, schieben Position, Probenposition und Spitzenkonfiguration.
- Wenn alle relevanten Prüfungen abgeschlossen haben, drücken Lastschlitten. Während die PCR-System lädt die Rutsche, entfernen Sie die klare und roten Kunststoff von der Unterseite des Schlittens Deckel. Wenn Sie fertig sind Laden, sorgfältig zu entfernen und verschließen Sie die Folie innerhalb von 90 sec.
- Den Objektträger in der Plattenklemme. Stellen Sie den Schiebedeckel auf die Folie. Klammer für 30 Sek. Sicherstellen, dass der Deckel so angeordnet, daß Barcode korrekt angezeigt wird. Entfernen Sie die Baugruppe aus dem Plattenklemme.
- Position Sionsflüssigkeit Spritze innerhalb des Schiebers, so dass die Spitze gegen den Deckel drücken. Langsam füllen Rutsche mit Immersionsflüssigkeit, wodurch die Flüssigkeit verläuft entlang der Deckel. Einmal voll, versiegeln Sie die Folie mit dem Stecker, Drehen der Schraube, bis der Griff abbricht.
- Die Kunststoffabdeckung auf der Oberseite des Schiebers Deckel und dann vorsichtig in den Objektträgerhalter von der Echtzeit-PCR-System zu setzen. Stellen Sie sicher, es gibt support am unteren Rand der Folie, wie sie abgesenkt wird, so dass es nicht plötzlich fallen, und der Oberseite des Schlittens nicht berühren. Es ist OK, um die Seiten des Objektträgers / cassette.Initialize die PCR-System für qPCR innerhalb von 1 Stunde zu berühren und das Programm starten.
- Wähle "Openarray" innerhalb des PCR-Systemsoftware. Drücken Sie auf "Suchen Slide-IDs". Dies kann einige Minuten dauern. Wenn die Software den Teller ID nicht finden können, werden Sie gefragt, für sie manuell eingegeben werden.
- Drücken Sie auf "Bestätigen Plattenzentren". Auch hier wird ein paar Minuten dauern. Überprüfen Sie, ob sich der rote Punkt in der Mitte, und dass es keine Fingerabdrücke / Markierungen auf der Oberseite des Schlittens. Laden Sie die jeweiligen .tpf Datei für jede Folie, und geben Sie das Ergebnis Dateinamen und Speicherort. Drücken Sie "Start Ausführen". Das Programm wird etwa 2 Stunden dauern.
Ergebnisse
Die empfohlene Volumen für eine miRNA-Sonde-basierte Assays qPCR Reaktion ist 20 ul. Anmerkung: Wir haben bestätigt, dass ein Reaktionsvolumen von 5 ul der Lage ist, ähnliche Ergebnisse wie die unter Verwendung von 20 ul Volumen 4,7,30 erreicht herzustellen. Absenken des Reaktionsvolumen auf 5 ul ermöglicht eine 75% ige Abnahme der Kosten für Reagenzien, ohne merklichen Verlust der Empfindlichkeit. Wie in Abbildung 2 dargestellt, Reaktionsvolumen von 20 ul und 5 ul zeigen eine starke Co-Verhältnis bis 39 Zyklen (mit r 2 von 0,92, p = 0,0002).
Mikrofluidik Array stellt ein Werkzeug zur Gewinnung von Daten über 754 miRNAs in einer Probe in etwa 5 Stunden (für Karte A und die Karte B), die eine effizientere Methode zur Analyse mehrerer Proben im Vergleich zu herkömmlichen Platte mit 96 Vertiefungen PCR ist ausgedrückt. Wir verglichen miRNA Mikrofluidik Array Karten für die gleiche Probe (Probe A und Probe A Wiederholung) 3A -. B zeigt eine Bland-Altman-Plot ( 3A) und Korrelation Grundstück (3B) für alle 754 miRNAs für diese Proben getestet. Es gibt 3 verschiedene Steuer miRNAs (U6, RNU44 und RNU48) nach dem Zufallsprinzip auf beiden Karten (Karten A und B) an verschiedenen Standorten platziert. Wenn Werte U6 Zyklus Schwelle (Ct) zwischen den 2 läuft im Vergleich haben wir nicht beobachten, signifikante Unterschiede zwischen den Werten (Tabelle 4). Es ist auch wichtig zu beachten, dass U6 ist in größerer Fülle (niedrigere Ct-Wert) in der Probe beurteilt ausgedrückt. Dann verglichen wir alle miRNAs, die Ct-Werte in beiden Läufen (n = 150) haben zwischen 0-19,99, die ähnlich Ausdruck miRNAs insgesamt mit einem Koeffizient der Bestimmung von 98% (3C-D) hatte. Von allen 277 miRNAs, die Ct-Werte zwischen 20 und 29.99 in den beiden Läufen haben, unterschieden sich 16 miRs deutlich zwischen den ursprünglichen und wiederholen Läufe (3E - F) Die Zahl der miRNAs mit signifikanten Unterschied zwische.n die Durchläufe erhöht (89 von 327), wenn die Ct-Werte wurden zwischen 30-40 für beide Läufe (Abbildung 3G - H) ausgewählt.
Die nanofluidics Array Plattform liefert die Daten für 754 miRNAs von jedem Serum / Plasma-Probe getestet, wie in 4A dargestellt ist es wichtig, diese Verstärkungskurven zu untersuchen. - Wie es bei allen qPCR - sicherzustellen, dass das Ergebnis anzeigt wahre Verstärkung. Jedes der 48 Untergruppen (Abbildung 1) enthält auch einen Test für die drei beliebtesten "Hauswirtschaft" ncRNAs. U6, RNU44 und RNU48 4B veranschaulicht eine typische Clustering von U6 repliziert aus einer einzigen Probe. Diese Wiederholungen anzuzeigen niedrige Standardabweichung (SD <0,5) und so sind ein Indikator für die Zuverlässigkeit. Alternativ 4C zeigt die erhöhte Variabilität U6 Replikate (SD> 0.5) in einer zweiten Probe. Dies schließt nicht die Gültigkeit o negierenf die übrigen Tests, obwohl es notwendig eine gründliche Kritik. U6, wie bei den meisten "Hauswirtschaft" miRNAs in biologischen Flüssigkeiten, können eine Variable Ausdruck haben. Es sei darauf hingewiesen, daß eine der Proben, in 4C skizziert, wird 4-fach weniger U6 Gehalt als die in Figur 4B dargestellt. Da die Höhe der U6 in Probe in 4C dargestellt ist, 75% weniger als mit der, in der Platte 4B dargestellt zu beginnen, wird eine verstärkte technische Variabilität aufgrund der Poisson-Verteilung von Transkripten, die von der kleinen Reaktionsvolumen 17 verstärkt wird erwartet.
Ein weiteres nützliches Tool ist die Qualitätskontrolle (QC) Bilder, für den Export zur Verfügung, sobald der Lauf beendet ist. Eine Auswahl davon ist der Fluoreszenz von ROX, der passive Farbstoff in der qPCR Reagenzgemisch gefunden, um zu bestätigen, dass jedes Durchgangsloch wurde korrekt (Abbildung 5) geladen. Ein Durchgangsloch oder gar ein eigenes Reifen Subarray kann nicht geladen werden aufgrund einer unzureichenden Probenvolumen, Verdampfung, in die Vertiefungen der 384er-Probenplatte, Versagen der Probe Plattendichtung, oder Defekte in dem Accufill Systems oder seiner Spitzen vollständig entfernen vorliegende Blasen. Jede entladen Durchgangsbohrungen müssen identifiziert werden, um die Kennzeichnung miRNAs als "nicht nachweisbar", zu vermeiden, wenn in Wirklichkeit der Test wurde nicht geladen. Wenn dieses Problem auftritt, zu bestätigen, dass mindestens 5 & mgr; l Probe / Mastermix in jedem Well der 384-well-Probenplatte geladen wird, die Probenplatte ordnungsgemäß vor der Beladung zentrifugiert, ist die Siegelfolie vollständig entfernt, und die beladene Openarray Schieber verschlossen und innerhalb der vorgegebenen Zeit für alle aufeinanderfolgenden Durchläufen ausführen. Wenn Lade Problem nicht beseitigen, können diese werden eher die sich auf bestimmte Partie oder des Loses der Arrays oder ähnlichen Verbrauchsmaterial und weitere Unterstützung sollte durch den Hersteller eingeholt werden.
"Src =" / files / ftp_upload / 52.586 / 52586fig1.jpg "/>
Abbildung 1: Layout von Proben für die Nanofluidik Array-Workflow: (A) Jeder 384-Well-Probenteller können Proben für bis zu 8 Nanofluidik Arrays zu halten. (B) Verwässert ist vorverstärkt cDNA in 8 Vertiefungen (2 Spalten von 4 Zeilen) in benachbarten 8-Well-Gruppen gelegt, mit Pool A und Pool B. Jeder Kreis steht für eine gut. (C) Jede Vertiefung der Probenplatte wird zu einem Unterfeld des Nanofluidik Array geladen werden. Jedes kleine Quadrat stellt einen Teilfeld.
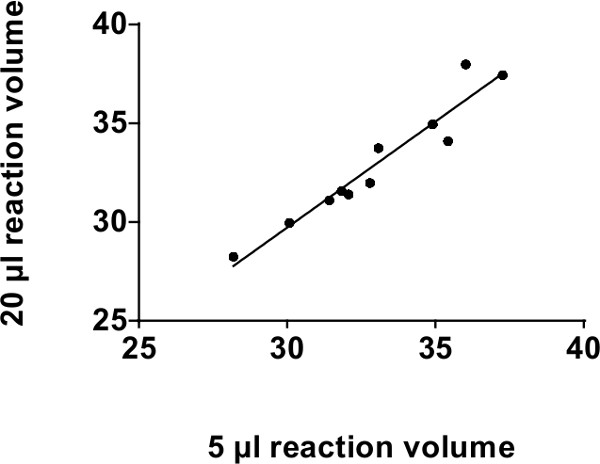
Abbildung 2: Co-Verhältnis-Analyse für konventionelle 96-well PCR-Plattformen: Co-Verhältnis von 20 ul und 5 ul Reaktionsvolumina auf TaqMan Real-time qPCR mit einem Standard-96-Well-Platte-Plattform in CT-Werte (39 Zyklen). Wir verglichen 4 verschiedene microRNAs (miR-375, miR-30c, 30d und miR-miR-7) in 4 verschiedenen menschlichen Serum- und Plasmaproben. Nur 11 Datenpunkte aufgezeichnet, da die anderen waren nicht nachweisbar. R 2 = 0,92, p = 0,0002.
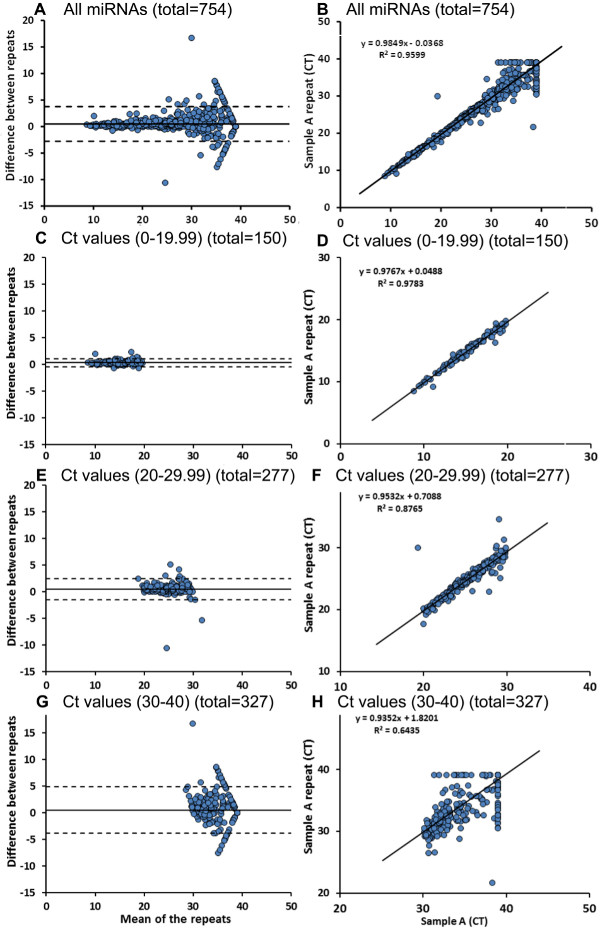
. Abbildung 3: Die Umlauf miRNA Profiling mit Mikrofluidik Array Karten Mit 2 Mikrofluidik-Karten (Karten-A und-B-Karte), ein Profil von 754 miRNAs erzeugt (A - B). Wie hier gezeigt, haben wir dieselbe Probe für 2 Mikrofluidik Array läuft, um die Reproduzierbarkeit der Mikrofluidik Karte Ergebnisse zu überprüfen. Wir beobachteten einen ähnlichen Ausdruck miRNAs insgesamt mit Ct-Werten zwischen 0 bis 19,99 (C - D). Es gibt nur wenige miRNAs (16 von 277) mit Ct-Werten zwischen 20 bis 29,99 und signifikante Unterschiede zwischen den Wiederholungsläufe (E - F). Achtzig neun von 327microRNAs mit höheren Ct-Werte (30-40) zeigten signifikante Unterschiede zwischen den beiden Durchgängen (G - H). Die Daten werden mit Hilfe gepaart T-Test analysiert. Bitte klicken Sie hier, um eine größere Version dieses Bild anzuzeigen.
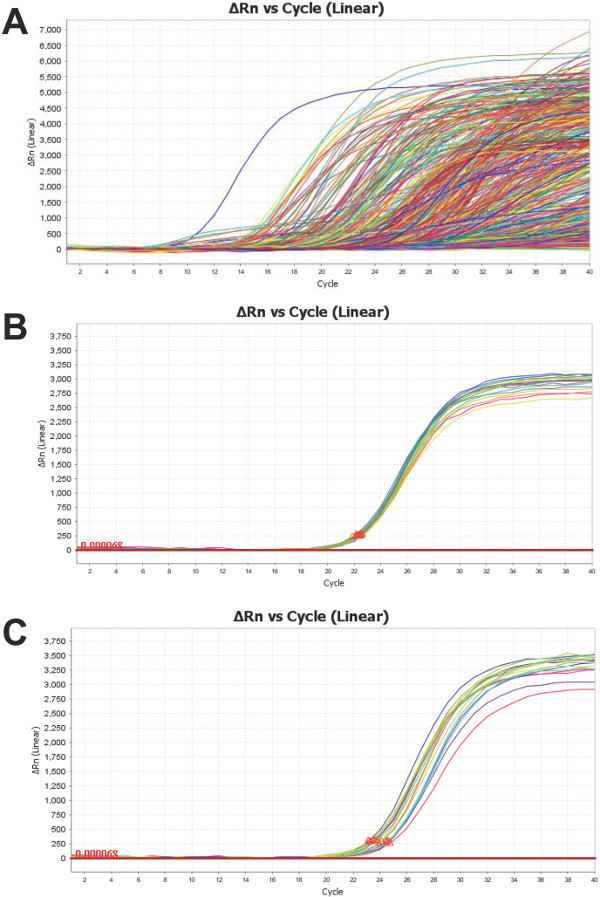
Abbildung 4: Vertreter Profil von PCR-Produkt Amplifikationskurven: Ist eine repräsentative Figur der kombinierten (A) Amplifikationskurven aller miRNA Ziele für eine menschliche Plasmaprobe. Ein Test auf U6 (eine gemeinsame Steuer ncRNA) ist in jedem Unterfeld platziert. Die Probe in (B) zeigt eine geringe Variabilität (SD <0,5), während (C) zeigt eine hohe Standardabweichung (SD> 0,5) innerhalb U6 repliziert. Beide Proben sind total RNA aus menschlichem Plasma isoliert. Bitte klicken Sie hier, um eine größere Version dieses Bild anzuzeigen.

Abbildung 5: QC Analyse Nanofluidik Arrays: Qualitätskontrolle (QC) Bilder einer richtig eingelegt Nanofluidik Array (links) und einer falsch eingelegt Nanofluidik Array (rechts). Die passive Farbstoff ROX (in der qPCR Reagenzienmix vorhanden), fluoresziert, um eine korrekt geladen Durchgangsbohrung angeben. Die Anordnung auf der rechten Seite hat mehrere Subarrays / Durchgangslöcher, die nicht mit der qPCR Reagenzgemisch geladen werden, und diese sollten als falsch-negative PCR-Reaktionen identifiziert werden.
| Components | Volumen pro 5 ul-Reaktion (ul) |
| RT-Puffer (10x) | 0,5 |
| dNTPs (100 mM) | 0,05 |
| RNase Inhibitor | 0.6 |
| Nuklease-freies Wasser | 1.39 |
| Reverse-Transkriptase- | 0.33 |
| Gesamtvolumen | 2.33 |
Tabelle 1A: RT Reagenzienmischung Komponenten in einer 5 & mgr; l RT-Reaktionsvorbereitung TaqMan Echtzeit qPCR unter Verwendung eines Standard-96-Well-Platte-Plattform.
| Components | Volumen pro 5 ul-Reaktion (ul) | Volumen pro 20 ul Reaktions (ul) |
| Schnelle PCR-Mastermix (2x) | 2.5 | 10 |
| TaqMan qPCR Assay (20x) * | 0,25 | 1 |
| Nuklease-freiesWasser | 1,45 | 5.8 |
| Gesamtvolumen: | 4.2 | 16,8 |
* Testkomponente auf Grundlage des ausgewählten miRNA getestet.
Tabelle 1B: qPCR Reagenzienmix Komponenten in einem 5 oder 20 & mgr; qPCR Reaktionsansatz für TaqMan Real-time qPCR mit einem Standard-96-Well-Platte Plattform.
| Components | Volumen pro Reaktion (ul) |
| Megaplex RT Primer (10x) | 0.8 |
| dNTPs mit dTTP (100 mM) | 0.2 |
| MultiScribe Reverse Transcriptase (50 U / ul) | 1.5 |
| 10X RT-Puffer | 0.8 |
| MgCl 2 (25 mM) | 0.9 |
| RNase Inhibitor & # 160; (20 U / ul) | 0.1 |
| Nuklease-freiem Wasser | 0.2 |
| Gesamt | 4.5 |
Tabelle 2A: RT Reagenzienmix Komponenten in einer 5 ul RT-Reaktion Vorbereitung TLDA miRNA-Panel.
| Components | Volumen pro Reaktion (ul) |
| TaqMan PreAmp Mastermix (2x) | 12,5 |
| Megaplex PreAmp Primers (10x) | 2.5 |
| Nuklease-freies Wasser | 7.5 |
| Gesamt | 22,5 |
Tabelle 2B: PreAmp Reagenzienmix Komponenten in einem 25 & mgr; pre-Amplifikation Reaktions Vorbereitung TLDA miRNA-Panel.
"Cellspacing =" 0 "> Components Volumen pro Reaktion (ul) Megaplex RT Primer (10x) 0.75 dNTPs mit dTTP (100 mM) 0,15 MultiScribe Reverse Transcriptase (50 U / ul) 1.5 10X RT-Puffer 0.75 MgCl 2 (25 mM) 0.9 RNase Inhibitor (20 U / ul) 0,09 Nuklease-freiem Wasser 0,35 Gesamt 4.5Tabelle 3A: RT Reagenzienmix Komponenten in einer 5 ul RT-Reaktion Vorbereitung sondenbasierten Nanofluidik miRNA-Panel.
| Komponentes | Volumen pro Reaktion (ul) |
| TaqMan PreAmp Mastermix (2x) | 12,5 |
| Megaplex PreAmp Primers (10x) | 2.5 |
| Nuklease-freies Wasser | 7.5 |
| Gesamt | 22,5 |
Tabelle 3B:. PreAmp Reagenzienmix Komponenten in einem 25 & mgr; pre-Amplifikation Reaktionsansatz für sondenbasierten Nanofluidik miRNA Platte HINWEIS: Die interaktive Zusatz Excel-Tabellen für die drei Plattformen zur Verfügung gestellt, die bereits einen Anteil von 5% zusätzlich zu zum Pipettieren Fehler zu kompensieren.
| Probe A | Probe A wiederholen |
| 15,535 | 16,156 |
| 15,471 | 15,652 |
| 15.623 | 16,063 |
| 15,963 | 15,889 |
| 14,006 | 13,993 |
| 14,502 | 14,623 |
| 14,907 | 14,384 |
| 13,732 | 14,946 |
Tabelle 4: Die Umlauf miRNA U6 Profilerstellung mithilfe der Mikrofluidik Array Karte. Mit zwei Array Mikrofluidik-Karten (A und B). Ct-Werte U6 Steuer miRNA aus Probe A (insgesamt = 8). Konsequente U6 Steuer miRNA Fluss zwischen den Läufen beobachtet.
Diskussion
Die kritischen Schritte innerhalb der Sonde qPCR-basierte Protokolle, um genaue und reproduzierbare Ergebnisse zu erhalten, sind Sie sicher, dass es 1) das gleiche Volumen und die Konzentration der RT-Produkt wird in jedem qPCR Reaktions geladen, 2) die richtigen Verhältnisse und Mengen der Komponenten benötigt qPCR Reaktion vorbereitet und gut gemischt, 3) die korrekte und einheitliche Volumina sind miteinander qPCR Reaktion gegeben, und 4) die Vorbereitung und Laden von jeder Probe und Reaktionsgemisch wird in kürzester Zeit wie möglich durchgeführt, während immer noch darauf achten, die obigen kritischen Schritte bereits erwähnt.
Die Mikrofluidik Array Karten müssen geeignete Zentrifuge und spezifische Eimer. Jede Schaufel kann bis zu 3 Karten (beladen / leer) zu halten. Achten Sie immer darauf, dass alle 3 Slots eines Becher belegt sind und die Schaufel, indem ähnliche Eimer (Eimer dies sollte auch 3 Karten, entweder leer oder voll enthalten) in die entgegengesetzte Schlitz der Zentrifuge ausgeglichen. Beim Aufsetzen der Karte in der bucket Halter, stellen Sie sicher, dass die 8 Stauseen projizieren nach oben und Reaktionsfeldern vor der Außenwand der Zentrifuge. Dreh die Karten bei 331 × g für 1 min bei Raumtemperatur. Nach dem ersten Durchlauf, öffnen Sie die Zentrifuge und optisch sicher, dass die Reaktionsmischung wurde durch die 384 Vertiefungen verzichtet. Wiederholen Sie den Spin bei gleichen Einstellungen für ein weiteres Mal. Entfernen Sie die Karte aus Eimer und sicherzustellen, dass das Niveau der Reaktionsmischung in jedem der acht Stauseen einheitlich ist. Etwaige Unstimmigkeiten bei den in den Behältern links Flüssigkeitsvolumen machen die Karte ungeeignet zum weiteren Gebrauch.
Um die gleiche Konzentration der RT-Produkt haben, wird dieselbe Eingangskonzentration von RNA in jedem RT-Reaktion gegeben. Die Gesamt-RNA-Konzentration wird unter Verwendung eines Mikro volumespectrophotometer gemessen. Die beobachtete 260/280 Verhältnis so niedrig wie 1.3 für die RNA aus Plasma / Serum isoliert werden; dies scheint nicht zu wirken auf den nachgelagerten qPCR bezogene Verfahren oder Daten erzeugt 30. Ebenso ist die 260/280 Verhältnisdie hier getesteten RNA-Proben lagen zwischen 1,3 bis 1,7 ohne abnormale Effekte in der qPCR beobachtet.
Bei Verwendung einer geringen Gehalt an RNA-Proben, wie die aus biologischen Flüssigkeiten, kann es schwierig werden, um RNA vor der Verarbeitung zu quantifizieren. Wir empfehlen die Verwendung von synthetischen Spike-in an der RNA-Isolierung sowie die reverse Transkription Stufen. Nach unserer Erfahrung Arabidopsis thaliana miRNA-Kandidaten (ath-miR-159a und ath-miR-172a) über Caenorhabditis elegans miRNAs (wie cel-miR-39 oder cel-miR-54), die in unserer Erfahrung können höher sind bevorzugt Homologie als die von A. thaliana. Die Verwendung einer solchen stufenspezifischen Spike-in kann für die Normalisierung der miRNA Daten über mehrere Proben zu verschiedenen Zeitpunkten getestet ausmachen. Mit einem festen Eingangslautstärke von RNA für die cDNA-Synthese-Reaktion wird auch empfohlen 32,33.
Die drei Sonden-basierte Protokolle für miRNA Quantifizierung hier beschriebenen erfordern unterschiedliche MengenGesamt-RNA-Eingang, unterschiedliche Workflows und Kosten. Jeder der Abläufe sind entworfen, um unterschiedliche Durchsätze, basierend auf der Anzahl der miRNA und der Anzahl der zu analysierenden Proben zu bieten. Mit zunehmendem Durchsatz (96 rxn 384 rxn 3072 rxn), die Kosten pro Reaktion mit einer Zunahme in der Menge an Daten über eine Zeiteinheit erhalten wird. Da alle diese Plattformen nutzen TaqMan Chemie, kann die Qualität der erhaltenen Daten erwartungsgemäß ähnlich sein werden. TaqMan qPCR ist ein etabliertes Verfahren zur Identifizierung der Fülle von miRNAs im Serum / Plasma Proben 9,11,27. Obwohl die drei Plattformen hier diskutierten dieselbe Chemie, eine Abnahme der Reaktionsvolumen führt zu einer Verringerung des dynamischen Bereichs des Transkripts Detektion (Farr RJ et al., Unveröffentlichte Daten). Der 96-Loch-qPCR-Plattform ist eine geringeren Durchsatz, aber hohe Empfindlichkeit Plattform und unserer Ansicht nach der "Goldstandard" für alle sondenbasierten (oder dye based) PCR-Plattformen. Dies kann jedochdie wirtschaftlichste und effiziente Plattform nicht sein, wenn mehrere Hundert oder Tausende von Proben, die untersucht und für mehrere microRNAs. Mikrofluidik (TLDA) und nanofluidics (OA) Plattformen mit hohem Durchsatz / Content-Plattformen zur Akquisition von größeren Daten in kürzerer Zeit zu ermöglichen. Obwohl Chargenunterschiede wurden in den TLDA Karten beobachtet worden ist, kann diese durch Anfordern TLDA Karten aus dem gleichen Ansatz minimiert werden. Wir beobachten, dass TLDA Plattform (Abbildung 3) zeigten signifikante Unterschiede in 17% der Mitte - geringe Häufigkeit miRNAs bei der Prüfung mit dem gleichen Probe auf unterschiedliche Chargen TLDA Karten. Wir empfehlen daher die gleichen Chargennummern für die Analyse mit TLDA Karten. Diese Variante könnte auch aufgrund der technischen Variabilität inklusive eventueller Beladung und Pipettierfehlern sein. Wir empfehlen jedoch, Bestellung / Anforderung der gleichen Charge von TLDA Karten. Keine signifikante Batch-Variante wurde auf die OA-Plattform beobachtet. Trotzdem TaqMan basierend experimental qPCR nähert Angebot Leichtigkeit, um die Fülle von miRNAs im Plasma / Serum Proben zu messen. Die niedrigen, mittleren und hohen Durchsatz Ansätze hier nicht an die Flexibilität, um eine Reihe von Proben und miRNAs mit einem hocheffiziente, reproduzierbare und sauber (geringem Hintergrundrauschen) Chemie analysieren.
Offenlegungen
Artikel Verarbeitung / Veröffentlichungskosten wurden von Life Technologies fallen, nach der Annahme des Manuskripts zur Veröffentlichung.
Danksagungen
Alle Autoren erkennen die Infrastruktur Unterstützung der NHMRC CTC, University of Sydney, der Juvenile Diabetes Research Foundation (JDRF), Australien und den Rebecca Cooper Foundation, Australien. Diese Forschung wurde unterstützt durch Zuschüsse aus dem Australian Research Council (FT110100254) und der JDRF, Australien (CRN201314) finanziert, um WW, RJF AAH und MVJ durchgeführt alle Nasslabor Experimente durchgeführt ASJ Datenanalyse. WW schrieb den ersten Entwurf. AAH geplant das Studium und die Daten analysiert. Alle Autoren lesen und einigten sich auf die endgültige Fassung der Handschrift, Zahlen und Arbeitsblätter zur Veröffentlichung eingereicht.
Materialien
| Name | Company | Catalog Number | Comments |
| 96 well-platform | |||
| For Reverse transcription | |||
| TaqMan MicroRNA Assays INV SM | Applied Biosystems | 4427975 | https://www.lifetechnologies.com/au/en/home/life-science/pcr/real-time-pcr/real-time-pcr-assays/mirna-ncrna-taqman-assays/single-tube-mirna-taqman-assays.html?ICID=search-4427975 The assays comes as a pack of RT primers and PCR primer. |
| TaqMan MicroRNA Reverse Transcription Kit | Applied Biosystems | 4366597 or 4366596 | https://www.lifetechnologies.com/order/catalog/product/4366597?ICID=search-4366597 or http://www.lifetechnologies.com/order/catalog/product/4366596?ICID=search-4366596 The TaqMan MicroRNA Reverse Transcription Kit is the same kit used for reverse transcription in all the threeTaqMan platform- 96-well platform, TLDA and OpenArray. |
| For qPCR run on a 96-well platform | |||
| 2x TaqMan Fast Universal PCR Master Mix, no AmpErase UNG | Applied Biosystems | 4367846 | https://www.lifetechnologies.com/order/catalog/product/4367846?ICID=search-4367846 The 2X TaqMan Fast Universal PCR Master Mix is used for qPCR on the two TaqMan platforms-96-well platform and TLDA. |
| MicroAmp Optical Adhesive Film | Applied Biosystems | 4311971 | https://www.lifetechnologies.com/order/catalog/product/4311971?ICID=search-product |
| MicroAmp Fast Optical 96-Well Reaction Plate, 0.1 ml | Applied Biosystems | 4346907 | https://www.lifetechnologies.com/order/catalog/product/4346907?ICID=search-product |
| Microfluidics platform (TLDA) | |||
| For Reverse transcription | |||
| Refer to 96-well platform TaqMan MicroRNA Reverse Transcription Kit kit (4366597 or 4366596) (as shown above). | |||
| Megaplex RT Primers, Human Pool A v2.1 | Applied Biosystems | 4399966 | https://www.lifetechnologies.com/order/catalog/product/4399966?ICID=search-4399966 The Megaplex RT Primers, Human Pool A v2.1 is used for RT on the two TaqMan platforms- TLDA and OpenArray. |
| Megaplex RT Primers, Human Pool B v3.0 | Applied Biosystems | 4444281 | https://www.lifetechnologies.com/order/catalog/product/4444281?ICID=search-4444281 The Megaplex RT Primers, Human Pool B v3.0 is used for RT on the two TaqMan platforms- TLDA and OpenArray. |
| OR | OR | ||
| Megaplex Primer Pools, Human Pools Set v3.0 | Applied Biosystems | 4444750 | http://www.lifetechnologies.com/order/catalog/product/4444750?ICID=search-4444750 The Megaplex Primer Pools, Human Pools Set v3.0 is used for RT on the two TaqMan platforms- TLDA and OpenArray. |
| For Pre-amplification | |||
| TaqMan PreAmp Master Mix | Applied Biosystems | 4391128 or 4488593 | https://www.lifetechnologies.com/order/catalog/product/4391128?ICID=search-4391128 or http://www.lifetechnologies.com/order/catalog/product/4488593 The TaqMan PreAmp Master Mix is used for pre-amplification of qPCR on the two TaqMan platforms- TLDA and OpenArray. |
| Megaplex PreAmp Primers, Human Pool B v3.0 | Applied Biosystems | 4444303 | https://www.lifetechnologies.com/order/catalog/product/4444303?ICID=search-4444303 The Megaplex PreAmp Primers, Human Pool B v3.0 is used for pre-amplification of qPCR on the two TaqMan platforms- TLDA and OpenArray. |
| Megaplex PreAmp Primers, Human Pool A v2.1 | Applied Biosystems | 4399233 | https://www.lifetechnologies.com/order/catalog/product/4399233?ICID=search-4399233 The Megaplex PreAmp Primers, Human Pool A v2.1 is used for pre-amplification of qPCR on the two TaqMan platforms- TLDA and OpenArray. |
| 1x TE Buffer (100 ml) | Invitrogen | 12090-015 | https://www.lifetechnologies.com/order/catalog/product/12090015?ICID=search-product The 1x TE Buffer is used for pre-amplification of qPCR on the two TaqMan platforms- TLDA and OpenArray. |
| To load and run the 384 well microfluidics (TLDA) card | |||
| TaqMan Array Human MicroRNA A+B Cards Set v3.0 | Applied Biosystems | 4444913 | https://www.lifetechnologies.com/order/catalog/product/4444913 |
| Nanofluidics platform (OpenArray) | |||
| For Reverse transcription | |||
| Refer to 96-well platform TaqMan MicroRNA Reverse Transcription Kit kit (4366597 or 4366596) (as shown above). | |||
| For Pre-amplification | |||
| Refer to TLDA pre-amplification reagents (as shown above) | |||
| To load and run the slides | |||
| TaqMan OpenArray Human MicroRNA Panel, QuantStudio 12K Flex (1 panel) | Applied Biosystems | 4470187 | https://www.lifetechnologies.com/order/catalog/product/4470187?ICID=search-4470187 |
| QuantStudio 12K Flex OpenArray Accessories Kit | Applied Biosystems | 4469576 | https://www.lifetechnologies.com/order/catalog/product/4469576?ICID=search-4469576 |
| OpenArray 384-well Sample Plates | Applied Biosystems | 4406947 | https://www.lifetechnologies.com/order/catalog/product/4406947?ICID=search-4406947 |
| TaqMan OpenArray Real-Time PCR Master Mix | Applied Biosystems | 4462159 | https://www.lifetechnologies.com/order/catalog/product/4462159?ICID=search-4462159 |
| OpenArray AccuFill System Tips | Applied Biosystems | 4457246 | https://www.lifetechnologies.com/order/catalog/product/4457246?ICID=search-4457246 |
| Others | |||
| Nuclease-Free Water | Qiagen | 129117 | http://www.qiagen.com/products/catalog/lab-essentials-and-accessories/nuclease-free-water |
Referenzen
- Ambros, V. The functions of animal microRNAs. Nature. 431, 350-355 (2004).
- Alvarez-Garcia, I., Miska, E. A. MicroRNA functions in animal development and human disease. Development. 132, 4653-4662 (2005).
- Bartel, D. P. MicroRNAs: genomics, biogenesis, mechanism, and function. Cell. 116, 281-297 (2004).
- Joglekar, M. V., Wei, C., Hardikar, A. A. Quantitative estimation of multiple miRNAs and mRNAs from a single cell. Cold Spring Harb Protoc. 2010, pdb prot5478(2010).
- Joglekar, M. V., Parekh, V. S., Hardikar, A. A. Islet-specific microRNAs in pancreas development, regeneration and diabetes. Indian J Exp Biol. 49, 401-408 (2011).
- Farr, R. J., Joglekar, M. V., Taylor, C. J., Hardikar, A. A. Circulating non-coding RNAs as biomarkers of beta cell death in diabetes. Pediatr Endocrinol Rev. 11, 14-20 (2013).
- Joglekar, M. V., Joglekar, V. M., Hardikar, A. A. Expression of islet-specific microRNAs during human pancreatic development. Gene Expr Patterns. 9, 109-113 (2009).
- Iorio, M. V., Croce, C. M. MicroRNA profiling in ovarian cancer. Methods Mol Biol. 1049, 187-197 (2013).
- Mitchell, P. S., et al. Circulating microRNAs as stable blood-based markers for cancer detection. P Natl Acad Sci USA. 105, 10513-10518 (2008).
- Mattie, M. D., et al. Optimized high-throughput microRNA expression profiling provides novel biomarker assessment of clinical prostate and breast cancer biopsies. Mol Cancer. 5, 24(2006).
- Zhu, W., Qin, W., Atasoy, U., Sauter, E. R. Circulating microRNAs in breast cancer and healthy subjects. BMC Res Notes. 2, 89(2009).
- Zhu, H. T., et al. Identification of suitable reference genes for qRT-PCR analysis of circulating microRNAs in hepatitis B virus-infected patients. Mol Biotechnol. 50, 49-56 (2012).
- Yamada, H., Itoh, M., Hiratsuka, I., Hashimoto, S. Circulating microRNAs in autoimmune thyroid diseases. Clin Endocrinol (Oxf). , (2014).
- Liu, R., et al. Serum MicroRNA Expression Profile as a Biomarker in the Diagnosis and Prognosis of Pancreatic Cancer. Clinical Chemistry. 58, 610-618 (2012).
- Gilad, S., et al. Serum microRNAs are promising novel biomarkers. PLoS One. 3, e3148(2008).
- Joglekar, M. V., Parekh, V. S., Mehta, S., Bhonde, R. R., Hardikar, A. A. MicroRNA profiling of developing and regenerating pancreas reveal post-transcriptional regulation of neurogenin3. Dev Biol. 311, 603-612 (2007).
- Hardikar, A. A., Farr, R. J., Joglekar, M. V. Circulating microRNAs: understanding the limits for quantitative measurement by real-time PCR. J Am Heart Assoc. 3, e000792(2014).
- Wu, C., et al. Diagnostic and Prognostic Implications of a Serum miRNA Panel in Oesophageal Squamous Cell Carcinoma. PLoS One. 9, e92292(2014).
- McDonald, M. K., Capasso, K. E., Ajit, S. K. Purification and microRNA profiling of exosomes derived from blood and culture media. J Vis Exp. , e50294(2013).
- Joglekar, M. V., et al. The miR-30 family microRNAs confer epithelial phenotype to human pancreatic cells. Islets. 1, 137-147 (2009).
- Genda, Y., et al. microRNA changes in the dorsal horn of the spinal cord of rats with chronic constriction injury: A TaqMan(R) Low Density Array study. Int J Mol Med. 31, 129-137 (2013).
- Wang, B., et al. Systematic evaluation of three microRNA profiling platforms: microarray, beads array, and quantitative real-time PCR array. PLoS One. 6, e17167(2011).
- Cuk, K., et al. Circulating microRNAs in plasma as early detection markers for breast cancer. Int J Cancer. 132, 1602-1612 (2013).
- Morrison, T., et al. Nanoliter high throughput quantitative PCR. Nucleic Acids Res. 34, e123(2006).
- Git, A., et al. Systematic comparison of microarray profiling, real-time PCR, and next-generation sequencing technologies for measuring differential microRNA expression. RNA. 16, 991-1006 (2010).
- Benes, V., Castoldi, M. Expression profiling of microRNA using real-time quantitative PCR, how to use it and what is available. Methods. 50, 244-249 (2010).
- Chen, C., et al. Real-time quantification of microRNAs by stem-loop RT-PCR. Nucleic Acids Res. 33, e179(2005).
- Schmittgen, T. D., et al. Real-time PCR quantification of precursor and mature microRNA. Methods. 44, 31-38 (2008).
- Kodani, M., et al. Application of TaqMan low-density arrays for simultaneous detection of multiple respiratory pathogens. J Clin Microbiol. 49, 2175-2182 (2011).
- Taylor, C. J., Satoor, S. N., Ranjan, A. K., Pereirae Cotta,, V, M., Joglekar, M. V. A protocol for measurement of noncoding RNA in human serum. Exp Diabetes Res. 2012, 168368(2012).
- TPF & SPF File Download Options [Internet]. , Available from: http://www.appliedbiosystems.com/absite/us/en/home/products/tpf-spf-download.html (2014).
- Turchinovich, A., Weiz, L., Langheinz, A., Burwinkel, B. Characterization of extracellular circulating microRNA. Nucleic Acids Res. 39, 7223-7233 (2011).
- Li, Y., Kowdley, K. V. Method for microRNA isolation from clinical serum samples. Anal Biochem. 431, 69-75 (2012).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten