Method Article
Baseado-Probe-PCR em tempo real Abordagens para a medição quantitativa de microRNAs
Neste Artigo
Resumo
MicroRNAs circulam surgiram recentemente como biomarcadores promissoras e inovadoras para vários tipos de câncer e outras doenças. O objetivo deste artigo é discutir três diferentes plataformas baseadas em sonda de PCR em tempo real e métodos que estão disponíveis para quantificar e determinar a abundância de microRNAs em circulação.
Resumo
Baseada em sonda de PCR quantitativa (qPCR) é um método preferido para medir a abundância de transcritos, uma vez que é um dos métodos de detecção mais sensíveis que fornece uma análise precisa e reprodutível. Química baseada em sonda oferece a menor fluorescência de fundo em comparação com outros (à base de corantes) químicos. Atualmente, existem várias plataformas disponíveis que a química baseada em uso de sonda para quantificar abundância transcrição. qPCR em uma placa de 96 poços é o método mais utilizada de rotina, no entanto, apenas um máximo de 96 amostras ou miARN pode ser testada num único ensaio. Isto é muito demorado e fastidioso se um grande número de amostras / miARNs estão a ser analisados. Plataformas baseadas em sonda de alto rendimento, como microfluídica (por exemplo, cartão matriz TaqMan) e matrizes nanofluidics (por exemplo OpenArray) oferecem facilidade para reprodutível e eficiente detectar a abundância de vários microRNAs em um grande número de amostras em um curto espaço de tempo. Aqui, demonstramos a uma configuração experimentalnd miARN protocolo para a quantificação de amostras de soro ou plasma com EDTA, usando produtos químicos à base de sonda e três plataformas diferentes (placa de 96 poços, e matrizes de microfluidos nanofluidics) oferecendo níveis crescentes de rendimento.
Introdução
Os microRNAs (miRNAs) são ~ 22 nucleotídeos não-codificante (NC) RNAs, funcionando como reguladores da expressão gênica 1-3. A maioria dos animais em miARNs funcionar através específica da sequência emparelhamento de bases com um mRNA, tendo como alvo a UTR 3 ', o que conduz a regulação negativa da expressão do gene 2-4. Isso geralmente ocorre através da inibição da tradução do mRNA ou por ribossômica drop-off. miARNs em circulação têm sido mostrados para ser novos biomarcadores em campos clínicos e de pesquisa para uma variedade de doenças, tais como diabetes 5-7, 8 ovário, próstata e cancro da mama 9 10,11, hepatite B e 12 outras doenças auto-imunes 13. A investigação foi conduzida para identificar miARNs abundantes em diferentes células ou tecidos, bem como em circulação a partir do plasma humano e amostras de soro, o que é mais acessível e menos invasiva 9,11-15.
Diferentes métodos de quantificação foram miARN eriado usando várias plataformas, como a plataforma padrão em placas de 96 poços 4,12,16-18, a plataforma de cartão de microfluídica 12,18-23 ea plataforma matriz nanofluidics 17,24. -PCR quantitativo em tempo real (qPCR) oferece a capacidade de medir números relativos ou absolutos de transcrições usando múltipla (dye- ou à base de sonda) químicas. Baseada em sonda em tempo real química PCR oferece a vantagem de baixa fluorescência de fundo e elevada sensibilidade para detectar uma única cópia de transcrição. É relativamente eficientes, simples de usar e altamente reprodutível, tornando-se um método preferido para a quantificação e determinação miRNA expressão 25. QPCR método baseia-sonda geralmente envolve dois passos: transcrição (RT) e qPCR 4,26,27 reversa. RT é onde o iniciador de haste-laçada RT é hibridado com uma molécula de miARN madura ou primário e convertido em (c) ADN complementar. A quantificação do produto de cDNA é então levada a cabo usando iniciadores de PCR específicos de miARN26-28. O princípio baseia-qPCR de sonda baseia-se na detecção da extensão de cadeia complementar, em tempo real, o que envolve a hidrólise da sonda fluorescente marcada com. Estas sondas são concebidas para conter um repórter fluorescente e um extintor que são apenas para além de permitir TERF (transferência de energia de fluorescência de ressonância). Detecção da emissão do repórter de fluorescência (emissor) é mascarada pela proximidade da molécula de extintor. Quando a Taq polimerase (pol) estende-se desde o iniciador a montante e chega a uma bifurcação (extremidade 5? Da sonda), a actividade de exonuclease de pol Taq hidrolisa a sonda, o que leva a uma dissociação física / separação do emissor do quencher fluorescente. Esta libertação de uma única molécula de fluorescência emissor é registado pelo detector e apresentado como um aumento gradual no seu sinal de fluorescência a partir desse bem / reacção. O aumento de fluorescência é proporcional à quantidade de produto de PCR gerado, permitindo que um qu precisasantification do alvo amplificado 26,28.
Com o aumento da procura de miARN quantificação, médio e tecnologias de elevado rendimento têm sido desenvolvidos para permitir que um maior número de amostras a ser processado em um curto espaço de tempo. TaqMan matriz de Baixa Densidade (TLDA) é uma concepção inovadora de microfluidos de médio rendimento com base em baseada em sonda qPCR química oferecendo um aumento no número de miARNs analisadas sobre um prato. TLDA envolvem a utilização de uma piscina predefinido de RT-iniciadores que são utilizados para sintetizar o ADNc. Estes ADNc são depois girado em um cartão personalizado 384 bem micro-fluídico para determinar a expressão de vários miARNs utilizando qPCR 22,26,29. Cada poço da placa contém seca iniciadores e sondas para amplificar miARN (s) específico, até 384, por conseguinte, as reacções podem ser transformados em cartão único TLDA 26.
A matriz nanofluidics é uma plataforma de alto rendimento, que é utilizado para a detecção de transcritos do gene 24 usando a mesma química à base de sonda. Ele utiliza uma matriz de propriedade oferecendo interacções hidrofóbicas-hidrófila para facilitar o carregamento da mistura de reacção de 33 nanolitros em uma matriz de 3072 por meio de furos de passagem sobre uma lâmina de aço inoxidável 24. Este artigo centra-se na demonstração de como esses métodos de quantificação miRNAs no soro / plasma são realizados e os fatores críticos que devem ser considerados quando se realizar e interpretar esses dados. Levado para a conta, seus benefícios e limitações individuais será discutido neste artigo.
Protocolo
O RNA total pode ser isolado do soro utilizando um protocolo estabelecido no nosso laboratório utilizando 30 ou outros kits disponíveis comercialmente.
NOTA: planilhas interativas suplementares para o cálculo dos volumes de reação em cada experimento (com o excesso de volume 5% responderam por pipetagem incluídas) são fornecidos.
1.-Probe-qPCR em tempo real, utilizando uma plataforma da placa de 96 poços Padrão
- a síntese de cDNA (transcrição reversa) em amostras de ARN de soro / plasma para miARN
- Calcule e tirar 10 ng de RNA para cada reação de síntese de cDNA. Adicionar água isenta de nuclease para levar o volume final de 10 ng de ARN de 1,67 uL (para uma reacção 5 ul). Manter as amostras em gelo.
- Selecione o miRNA e descongelar os seus iniciadores de RT.
- Descongele as RT componentes misturar reagentes: RT Buffer (10X), dNTPs (100 mM), RNase Inhibitor (20 U / l). Nunca manter enzima lugar (moloney vírus da leucemia murina recombinante(RMoMuLV)) (recombinanat vírus da leucemia murina de Moloney (rMoMuLV) transcriptase reversa (50 U / mL) em gelo. Armazenar a -20 ° C até ser necessário ou em bloco de um congelador.
- Preparar a mistura reagente sobre gelo RT, tal como descrito na Tabela 1A. Recomenda-se para preparar, pelo menos, 5% de excesso de volume para compensar erros de pipetagem.
- Adicionar 2,33 mL da mistura reagente RT cada um em um tubo de 0,2 ml PCR / placa.
- Vortex os tubos de iniciadores RT a mistura, em seguida, brevemente centrifugar a 10.000 xg durante 10 seg. Adicionar 1 ml de miARN iniciador RT específico aos seus respectivos tubos ou placa de PCR. Adicionar 1,67 mL da ARN diluída para os seus respectivos tubos de PCR ou poços na placa. Executar todas as adições no gelo.
- Centrifuga-se o tubo de reacção ou em placas a 1.950 xg durante 5 min a 4 ° C.
- Defina-se o programa de síntese de ADNc miARN no termociclador com as seguintes condições de ajuste: 16 ° C durante 30 min, 42 ° C durante 30 min, 85 °C durante 5 minutos e 4 ° C em espera.
- Ajuste do volume de reacção de 10 ul. Coloque os tubos ou placa de reação em termociclador. Comece a correr RT. Armazenar as reacções de cDNA (RT) a -20 ° C, se a amplificação por PCR em tempo real não é imediatamente seguido.
- Probe baseada em tempo real qPCR para detecção de miRNAs maduros
- Descongelar os ensaios de sondas seleccionadas com base no qPCR (20X) para o respectivo produto de RT.
- Misture a solução única propriedade de PCR em tempo real (qPCR mix reagente) agitando a garrafa.
- Preparar a mistura de reagente para cada um miARN qPCR a ser analisado. Para preparar o qPCR obter um tubo de microcentrífuga de 1,5 ml estéril para cada amostra miARN e adicionar os componentes dentro de cada tubo, tal como descrito na Tabela 1B. Recomenda-se para preparar, pelo menos, 5% de excesso de volume para compensar erros de pipetagem.
- Adicionar 4,2 mL da respectiva mistura reagente qPCR a cada poço de uma placa de 96 poços óptica (ou tubos). Adicionar 0,8 mLda respectiva reacção de cDNA (sintetizado no passo 1.1 para cada miARNs) a respectiva bem.
- Selar a placa com tampa óptico adequado. Centrifugar a placa (ou tubos) a 1950 xg durante 5 min a 4 ° C
- Ligue o computador, 96 poços leitor matriz placa / microfluídica e, finalmente, lançar o software. Certifique-se de que as máquinas estejam conectados corretamente e bloco correto e tampa aquecida (para rápida placa de 96 poços) estão no local.
- Seleccionar experimento como bloco de 96 poços rápido (0,1 ml), a curva padrão, os reagentes TaqMan e modo rápido. Configurar o sistema de qPCR em PCR em tempo real usando as seguintes condições de ciclo programa: 95 ° C durante 20 seg, 50 ciclos (95 ° C durante 1 seg, 60 ° C durante 30 seg).
- Ajuste do volume de reacção de 10 ul. Carregar o tubo de reacção ou da placa no instrumento. Pressione o botão "Iniciar run". O programa levará aproximadamente 1 hora para ser concluído.
2. Cartão de microfluídica matriz baseada em Probe (Caestr A e B)
NOTA: base-Probe painel miRNA vem como um conjunto de duas placas de 384 poços microfluídicos (cartão matriz A e matriz de cartão B). Cada cartão contém Primers seca e sondas para até 380 miRNAs e controles. produto de ADNc (com ou sem pré-amplificação) específico para um cartão ou cartão de B é carregado para o respectivo arranjo para PCR em tempo real.
- Transcrição Reversa (RT)
- Utilize este protocolo para a entrada de RNA total de 1-1,000 ng. Se a entrada está entre 1-350 ng, realizar uma etapa de pré-amplificação (pré-amp). Para entradas acima de 350 ng, carregar cDNA diretamente nos cartões de matriz sem pré-amplificador.
NOTA: Para miRNA soro profiling, comece com 100 ng de RNA total. Para um perfil completo miRNA, executar duas piscinas pré-definidos de conjuntos RT-iniciadores de reações de RT (Grupo A e Grupo B) por amostra. - Descongelar os seguintes reagentes em gelo. RT Primers (10x): Grupo A e Grupo B, dNTPs com dTTP (100 mM), RT Buffer (10x), MgCl2 (25 mM), RNase Inhibitor (20 U / &# 181; l). Não manter a enzima (recombinante moloney vírus da leucemia murina (rMoMuLV)) transcriptase reversa (50 U / mL) em gelo. Armazená-lo a -20 ° C até ser necessário.
- Suavemente vortex todos os reagentes, excepto a enzima, e depois centrifugar brevemente os tubos a 10.000 xg durante 10 seg.
- Combinar os reagentes, tal como descrito na Tabela 2A, em dois tubos; um tubo para Pool A, o outro para Piscina B. Cada tubo irá conter apenas uma piscina pré-definido de RT conjunto de primers, quer a partir de Pool A ou do exterior B. Recomenda-se a preparar, pelo menos, 5% de excesso de volume para compensar erros de pipetagem .
- Inverta para misturar, e brevemente, em seguida, centrifugar a 10.000 xg por 10 s. Alíquota de 100 ng de cada amostra de ARN para um novo tubo, em seguida, adicionar um volume apropriado de água isenta de nuclease para fazer um total de 3 ul.
- Alíquota de 4,5 mL da mistura reagente RT adequado para os respectivos tubos. Inverta a mistura, e depois centrifugar brevemente a 10.000 xg durante 10 seg. Incubar em gelodurante 5 min.
- Colocar as amostras em um termociclador e iniciar a RT utilizando as seguintes condições: 40 ciclos (16 ° C durante 2 min, 42 ° C durante 1 min, 50 ° C durante 1 seg), 85 ° C durante 5 min, mantido a 4 ° C. ADNc da loja gerada usando estes piscina predefinido de RT-iniciadores à temperatura de -15 a -25 ° C ou usado imediatamente.
- Utilize este protocolo para a entrada de RNA total de 1-1,000 ng. Se a entrada está entre 1-350 ng, realizar uma etapa de pré-amplificação (pré-amp). Para entradas acima de 350 ng, carregar cDNA diretamente nos cartões de matriz sem pré-amplificador.
- Pré-amplificação
- Descongele a piscina predefinido de primers pré-amp no gelo. Delicadamente, primers de vórtices e, em seguida, centrifugar-los brevemente a 10.000 xg por 10 s. Swirl TaqMan PreAmp mix reagente (2X) agitando ligeiramente, para misturar.
- Combinar os reagentes, tal como descrito na Tabela 2B, em dois tubos; um tubo para Pool A, o outro para Piscina B.
NOTA: Como descrito acima, Piscina primers pré-amplificador irá para tubo A e primers Grupo B deve ir para o tubo B. Recomenda-se a preparar, pelo menos, 5% de excesso de volume para compensar erros de pipetagem. - Inverta a mistura, e depois centrifugar brevemente a 10.000 xg durante 10 seg.Alíquota de 22,5 mL da mistura reagente PreAmp apropriado para novos tubos. Aliquota de 2,5 ul da amostra de cDNA (preparado no passo de RT) para o respectivo tubo.
- Inverta a mistura, e depois centrifugar brevemente a 10.000 xg durante 10 seg. Incubar em gelo durante 5 minutos. Colocar as amostras em um termociclador e iniciar o ciclo de pré-amp. Condições de ciclo: 95 ° C durante 10 min, 55 ° C durante 2 min, 72 ° C durante 2 min, 12 ciclos (95 ° C durante 15 seg, 60 ° C durante 4 min), 99,9 ° C durante 10 min, manter a 4 ° C.
- Inverter pré-amplificados de ADNc para misturar, e, em seguida, brevemente centrifugar a 10.000 xg durante 10 seg. Adicionar 75 ul de 0,1 x tampão TE (pH 8,0) para pré-amplificado ADNc (diluição 1: 4). Inverta as amostras de pré-amp diluída para misturar, e depois centrifugar brevemente a 10.000 xg por 10 s. Loja diluído pré-amplificado cDNA a -15 a -25 ° C durante até uma semana, ou utilizado imediatamente.
- Colocando cartões de microfluídica e realizando qPCR
- Mantenha a microfluídicamiRNA cartões de fora por pelo menos meia hora para atingir a temperatura ambiente. Descongele diluído cDNA pré-amplificada no gelo e misture invertendo os tubos seguidos de breve giro. Misture mix reagente qPCR agitando a garrafa.
- No novo tubo de adicionar 450 ul de mistura reagente qPCR a 9 ul de cDNA diluído pré-amplificado. Para card A, usar o produto pré-amplificada preparada com cartilha Piscina A. Adicionar 441 mL de água livre de nuclease para compor o volume final de 900 mL. Inverter o tubo de mistura, e então centrifugar brevemente a 10.000 xg durante 10 seg.
- Remova o cartão TLDA de sua embalagem (uma vez que atinge a temperatura ambiente) e coloque-o em uma área limpa, com o lado da folha para baixo. Adicionar 100 uL de mistura de reacção de PCR em cada um dos oito portas de enchimento sobre o cartão. Há 2 portas em cada reservatório (8 reservatórios no total em cada cartão).
NOTA: A porta de preenchimento é o maior buraco onde é adicionado à mistura de reação, enquanto que a abertura de ventilação é o buraco menor. Cada cartão tem miARN TLDA 8 reservatórios, cada um conduzindo a 48 poços (placas com 24 poços em uma coluna x 2 colunas), carregando, assim, todos os 384 poços na placa com a mesma mistura de reacção de PCR. Certifique-se de que Poule A e Grupo B de cada amostra são carregados nos respectivos cartões TLDA. Coloque o cartão matriz na matriz microfluidics baldes card holder especializados na centrífuga. - Para os cartões de matriz microfluídica, use uma centrífuga adequado (como Heraeus Multifuge 3SR, 230V centrífuga) e caçambas específicas (tais como titulares de "Cartão Matriz TaqMan"). Cada balde pode conter até 3 cartões (carregado / vazio). Sempre garantir que todos os 3 slots de um balde são ocupados eo balde é equilibrado, colocando balde semelhante (esta balde também deve conter três cartões, vazios ou cheios) no slot oposto da centrífuga. Ao colocar o cartão no suporte do balde, certifique-se de que os reservatórios 8 projectar para cima e poços de reacção frente para a parede externa do centrifugador.
- Rodar as placas a 331 xg durante 1 min à temperatura ambiente. Depoisprimeira rodada, abra a centrífuga e visualmente garantir a mistura de reacção foi distribuída através dos 384 poços. Repita o spin em mesmas configurações para mais uma vez. Remova a placa do balde e assegurar que o nível de mistura de reacção em cada um dos 8 reservatórios é uniforme. Qualquer inconsistência nos volumes líquidos deixados nos reservatórios fazer o cartão inapropriado usar mais.
- Placas de matriz Microfluidics precisa de um selador especializado que tem uma montagem de precisão estilete (transporte) para selar os canais de distribuição de fluido da matriz e igualmente distribuir a mistura de reacção em todas as cavidades (volume total de reacção de 1 mL / poço).
- Traga o carro para a posição inicial e insira o cartão carregado no selador com o lado da folha e alinhados aos pinos caneta sobre o cimento. Em um golpe uniforme lento, constante e único, empurre o carro através do cartão até que se atinja o ponto final do selante. Remova o cartão matriz selado e depois cortar o reservatórios do cartão com uma tesoura.
- Certifique-se de que a transportadora bloco correto, tampa aquecida e amostra está instalado na matriz microfluidics tempo real sistema de PCR / máquina.
- Ligue o computador, então o sistema de matriz microfluídica e, finalmente, lançar o software. Certifique-se de que as máquinas estão conectados corretamente. Escolha um experimento como o cartão matriz, curva padrão, reagentes TaqMan e modo padrão. Importe os arquivos de instalação para qualquer um o cartão A e B. cartão Salve o arquivo.
- Colocar a placa selada na bandeja do instrumento com o poço A1 no canto superior esquerdo e do código de barras para a frente do instrumento. Pressione o botão "Start Run". O programa terá cerca de 2 horas para ser concluído.
Baseada em sonda 3. Painel Nanofluidics miARN Humano
- Transcrição Reversa (RT)
- Utilize este protocolo para a entrada de RNA total de 50-200 ng, porém 100 ng é o ideal para a maioria das amostras. Para um perfil completo miRNA, executar dois predefipools NED de conjuntos RT-iniciadores de reações de RT (Grupo A e Grupo B) por amostra.
- Descongelar os reagentes em gelo: piscina predefinido de conjuntos de iniciadores de RT (10X), com dTTP dNTPs (100 mM), tampão de RT (10x), MgCl2 (25 mM), ARNase Inibidor (20 U / ul). Não manter a enzima (recombinante do vírus da leucemia murina de Moloney (rMoMuLV) transcriptase reversa (50 U / mL) em gelo. Armazenar a -20 ° C até ser necessário. Vortex suavemente todos os reagentes, excepto a enzima, e centrifugar para brevemente a 10.000 xg durante 10 seg.
- Combinar os reagentes, tal como descrito na Tabela 3A, em dois tubos; um tubo para Pool A, o outro para Piscina B. Cada tubo irá conter apenas uma piscina conjunto pré-definido de RT-primer, quer a partir de Pool A ou do exterior B. Recomenda-se a preparar, pelo menos, 5% de excesso de volume para compensar pipetagem erro.
- Pipeta para misturar e brevemente em seguida, centrifugar a 10.000 xg por 10 s. Alíquota de 100 ng de cada amostra de ARN para um novo tubo, em seguida, adicionar um volume apropriadode água isenta de nuclease para fazer um total de 3 ul. Aliquota 4,5 ul da mistura reagente RT adequado para o respectivo tubo.
- Inverta a mistura, e depois centrifugar brevemente a 10.000 xg durante 10 seg. Incubar em gelo durante 5 min. Colocar as amostras em um termociclador e iniciar o programa RT. Condições de ciclo: 40 ciclos (16 ° C durante 2 min, 42 ° C durante 1 min, 50 ° C durante 1 seg), 85 ° C durante 5 min, mantido a 4 ° C. ADNc da loja gerado usando estes piscina predefinido de RT-conjunto de iniciadores de -15 a -25 ° C ou utilizar imediatamente.
- Pré-amplificação (pré-amp)
- Descongele a piscina pré-definido de primer pré-amp definido no gelo. Gentilmente primers vórtice, em seguida, centrifugar-los brevemente a 10.000 xg por 10 s. Swirl TaqMan PreAmp mix reagente (2X) para misturar.
- Combinar os reagentes, tal como descrito na Tabela 3B, em dois tubos; um tubo para Pool A, o outro para Piscina B. Cada tubo irá conter apenas uma piscina predefinido de conjuntos RT-iniciadores, ou from Pool A ou do exterior B. Recomenda-se a preparar, pelo menos, 5% de excesso de volume para compensar pipetagem erro.
- Pipeta para misturar, e, em seguida, brevemente centrifugar para a 10.000 xg durante 10 seg. Alíquota de 22,5 mL da mistura reagente PreAmp apropriado para novos tubos. Aliquota de 2,5 ul da amostra de cDNA para o respectivo tubo. Inverta a mistura, e depois centrifugar brevemente os tubos a 10.000 xg durante 10 seg. Incubar em gelo durante 5 min.
- Colocar as amostras em um termociclador e executar o pré-amp. Ajuste do volume de reacção de 25 ul. Condições de ciclo: 95 ° C durante 10 min, 55 ° C durante 2 min, 72 ° C durante 2 min, 12 ciclos (95 ° C durante 15 seg, 60 ° C durante 4 min), 99,9 ° C durante 10 min, manter a 4 ° C.
- Inverter pré-amplificados de ADNc para misturar, e, em seguida, brevemente centrifugar a 10.000 xg durante 10 seg. Em novos tubos adicionar 4 mL de ADNc pré-amplificado com 156 ul de 0,1 x tampão TE a pH 8,0 (diluição 1:40). Inverta as amostras de pré-amp diluída a mistura, e, em seguida, brevemente centrifuge a 10.000 xg durante 10 seg. Loja diluído e ADNc pré-amplificado não diluída à temperatura de -15 a -25 ° C durante até uma semana, ou usar imediatamente.
- Carregando Nanofluidics Arrays e Performing qPCR
- Baixe o arquivo placa relevante (.tpf) a partir do website 31 usando a matriz nanofluidics slides número de série. Este contém as informações de funcionamento para o slide matriz nanofluidics específico.
- Mistura pré-amplificado diluído Descongelar ADNc e em tempo real TaqMan qPCR reagente (se utilizar pela primeira vez) em gelo. Misture mix reagente qPCR agitando a garrafa. Em novos tubos de adicionar 22,5 mL de mistura de reagente de qPCR de 22,5 ul de cDNA diluído pré-amplificado. Gentilmente vortex para misturar, e depois centrifugar brevemente a 10.000 xg por 10 s.
- Alíquota de 5 ul de cada amostra em 8 poços (2 colunas, 4 filas) da chapa de amostra, o fluxo de trabalho matriz nanofluidics de 384 poços. Certifique-se de que Poule A e Grupo B de cada amostra estão em blocos de 8 poços adjacentes (ver Figure 1 ou Suplementar folha de excel para layout).
- Cada placa de amostra pode conter até oito lâminas valor de amostras. No entanto, o sistema de matriz nanofluidics só pode processar 4 matrizes nanofluidics em uma única corrida. Se mais de quatro slides no valor de amostras devem ser carregados em uma placa de amostra, certifique-se as seções restantes são selados. Selo com OpenArray placa de amostra selador.
NOTA: É aconselhável a pré-cortar o cimento para as seções necessárias, para que as seções podem ser selados / não selada individualmente para reduzir a evaporação. Alternativamente, a chapa pode ser selado com um selante intacto, e então secções podem ser cortadas individualmente para fora durante o carregamento.
- Cada placa de amostra pode conter até oito lâminas valor de amostras. No entanto, o sistema de matriz nanofluidics só pode processar 4 matrizes nanofluidics em uma única corrida. Se mais de quatro slides no valor de amostras devem ser carregados em uma placa de amostra, certifique-se as seções restantes são selados. Selo com OpenArray placa de amostra selador.
- Centrifugar a placa 490 xg durante 1 min a 4 ° C. Coloque os nanofluidics corrediças da disposição dentro de 1 hora. Devido ao tempo limitado permitido para selar os slides, por favor, carregue apenas uma lâmina de cada vez. Remover o slide matriz nanofluidics do congelador e deixe-o vir a temperatura ambienteratura (~ 15 min).
- Certifique-se de que a transportadora bloco correto, tampa aquecida e amostra é instalado no sistema de matriz nanofluidics. Ligue o computador, e em tempo real sistema de PCR e do sistema de carregamento. Acesse o respectivo software e garantir que as máquinas estão conectadas. Retire os consumíveis sistema de carregamento (array de slides tampa, plug and fluido de imersão) da embalagem.
- Com cuidado, puxe o êmbolo da seringa fluido de imersão para soltar. Retire a tampa, coloque na ponta e lave o ar da ponta. Coloque as pontas do sistema de carga no interior da máquina e retire a tampa. Coloque placa de amostra dentro do sistema de PCR.
- Coloque luvas. Certifique-se de que eles estão bem ajustados para minimizar o risco de marcar acidentalmente a tampa slide. Embalagem Deslize cuidadosamente aberto. Lentamente ponta slide em mão. Não toque na parte superior do slide.
- Colocar a lâmina para o sistema de PCR, com o código de barras do lado esquerdo. Remover selador a partir da porção da placa de amostra destinada a ser carregada. Use o sistema de carregamentosoftware para inserir o código de barras slide, slide posição, a posição da amostra e configuração de ponta.
- Quando todos os controlos necessários sejam concluídos, escorrega imprensa carga. Enquanto o sistema de PCR está a carregar o diapositivo, remover a película de plástico e vermelho da parte inferior da tampa corrediça. Quando terminar o carregamento, retire cuidadosamente e selar o slide dentro de 90 segundos.
- Colocar a lâmina dentro do grampo de chapa. Coloque a tampa deslizante para o slide. Grampo para 30 seg. Certifique-se de que a tampa é posicionada de modo que código de barras é exibido corretamente. Remova o conjunto da placa de fixação.
- Posição imersão seringa de fluido no interior da corrediça, de modo que a ponta é pressionada contra a tampa. Lentamente encher de slides com fluido de imersão, garantindo o fluido corre ao longo da tampa. Uma vez completo, selar o slide com a ficha, girar o parafuso até que a alça quebra.
- Retire a tampa de plástico na parte superior da tampa slide, e em seguida, coloque cuidadosamente no suporte de slides do sistema de PCR em tempo real. Verifique se há support na parte inferior da corrediça, uma vez que está a ser reduzido, de modo que não cai repentinamente, e não toque na parte superior da lâmina. É OK para tocar os lados da lâmina de sistema / cassette.Initialize a PCR e iniciar o programa para qPCR dentro de 1 hora.
- Selecione "OpenArray" dentro do software PCR-sistema. Pressione o botão "Localizar IDs de slides". Isso vai demorar alguns minutos. Se o software não consegue encontrar a placa de identificação, ele vai pedir para que seja inserido manualmente.
- Pressione o botão "Confirmar Centros de placa". Novamente, isso vai demorar alguns minutos. Verificar que o ponto vermelho é no centro e que não há impressões digitais / marcas no topo da corrediça. Coloque o respectivo arquivo .tpf para cada slide e especificar um nome de arquivo resultado e localização. Pressione o botão "Start Run". O programa terá cerca de 2 horas para ser concluído.
Resultados
O volume recomendado para uma reacção qPCR ensaio à base de miARN sonda é de 20 ul. NOTA: Confirmou-se que um volume de reacção de 5 ul é capaz de produzir resultados semelhantes aos obtidos usando 20 ul de volume 4,7,30. Diminuindo o volume de reacção para 5 ul permite uma redução de 75% nos custos de reagentes sem perda significativa de sensibilidade. Como apresentado na Figura 2, os volumes de reacção de 20 ul e 5 ul mostram uma forte correlação até 39 ciclos (com r 2 de 0,92, p = 0,0002).
Microfluidics matriz fornece uma ferramenta para a obtenção de dados sobre 754 miRNAs expressos em uma amostra em cerca de 5 horas (para o cartão e um cartão B), que é uma forma mais eficiente de analisar amostras múltiplas em comparação com 96 PCRs placa convencionais bem. Comparamos miRNA cartões matriz microfluídica para a mesma amostra (amostra A e amostra A repetição) Figura 3A -. B mostra um gráfico de Bland-Altman ( 3A) e curva de correlação (3B) para todos os 754 miRNAs testados para estas amostras. Há 3 miRNAs de controle diferentes (U6, RNU44 e RNU48) colocadas aleatoriamente em ambos os cartões (CARD A e B) em vários locais. Quando os valores de limite de ciclo U6 (TC) são comparados entre as duas corridas, não observamos diferenças significativas entre os valores (Tabela 4). É também importante notar aqui que U6 é expressa em maior abundância (menor valor de Ct) na amostra avaliada. Em seguida, comparou todas miARNs que têm valores de Ct entre 0-19,99 em ambas as passagens (n = 150), que tinham de expressão semelhante miARNs global com um coeficiente de determinação de 98% (Figura 3C-D). De todos os 277 miRNAs que têm valores Ct entre 20 e 29,99 em ambas as corridas, 16 miRs diferiu significativamente entre os originais e repita runs (Figura 3E - F) O número de miRNAs com diferença significativa betwee.n as corridas aumentou (89 de 327), quando foram selecionados os valores Ct entre 30-40 para ambas as corridas (Figura 3G - H).
A plataforma matriz nanofluidics fornece dados para miARNs 754 de cada amostra de soro / plasma testado, tal como representado na Figura 4A É importante examinar estas curvas de amplificação -. Tal como acontece com todos qPCR - para assegurar que o resultado é indicativo da verdadeira amplificação. Cada um dos 48 subarrays (Figura 1) contém também um ensaio para as três mais populares "limpeza" ncRNAs:. U6, RNU44 e RNU48 Figura 4B ilustra um agrupamento típico da U6 replica a partir de uma única amostra. Essas repetições exibir baixo desvio-padrão (SD <0,5) e por isso são um indicador de confiabilidade. Alternativamente, Figura 4C demonstra o aumento da variabilidade de repetições U6 (DP> 0,5) em uma segunda amostra. Isso não nega a validade of os ensaios restantes, embora não necessitam de uma crítica mais aprofundada. U6, como acontece com a maioria dos miRNAs "limpeza" em fluidos biológicos, pode ter uma expressão variável. Deve notar-se que uma das amostras, descritas na Figura 4C exibe, 4 vezes menos do que o conteúdo U6 apresentado na Figura 4B. Uma vez que o nível de U6 em amostra apresentada na 4C é 75% menos do que começar com o apresentado no painel 4B, uma maior variabilidade técnica é esperado devido à distribuição de Poisson de transcritos, que é agravada pelo pequeno volume de reacção 17.
Outra ferramenta útil é as imagens de controle de qualidade (QC), disponíveis para a exportação uma vez que o prazo foi concluída. Uma selecção destes usa a fluorescência de ROX, o corante passiva encontrado na mistura reagente qPCR, para confirmar que cada orifício foi colocado correctamente (Figura 5). A-buraco através, ou mesmo uma casa pneu sub-disposição, não pode carregar em função do volume de amostra insuficiente, evaporação, bolhas presentes nos poços da placa de amostra de 384 poços, a incapacidade de remover completamente a junta da placa de amostra, ou defeitos no interior do sistema Accufill ou suas pontas. Qualquer descarregado orifícios devem ser identificados para evitar miRNAs rotulagem como "indetectável", quando na realidade o ensaio nunca foi carregado. Se este problema é encontrada, confirmam que, pelo menos, 5 ul de amostra / mastermix é carregado em cada poço da placa de amostra de 384 poços, a placa de amostra é centrifugada adequadamente antes do carregamento, a folha de vedação é completamente removido, e o OpenArray carregado deslizante é vedado e executado dentro do tempo estipulado para todas as corridas sucessivas. Se os problemas de carregamento ainda persistem, estes podem ser mais propensos pertencente ao lote específico ou lote das matrizes ou consumíveis relacionados e mais assistência deve ser procurada através do fabricante.
"Src =" / files / ftp_upload / 52586 / 52586fig1.jpg "/>
Figura 1: Esquema de amostras para o fluxo de trabalho da Matriz Nanofluidics: (A) Cada placa de amostra de 384 poços pode conter amostras para até 8 matrizes nanofluidics. (B) Diluído, cDNA pré-amplificada é colocado em 8 poços (2 colunas por 4 linhas), com tanque e Pool B em grupos de 8 poços adjacentes. Cada círculo representa um poço. (C) A cada poço da placa de amostra irá ser carregado em um sub-disposição da matriz nanofluidics. Cada pequeno quadrado representa um subarray.
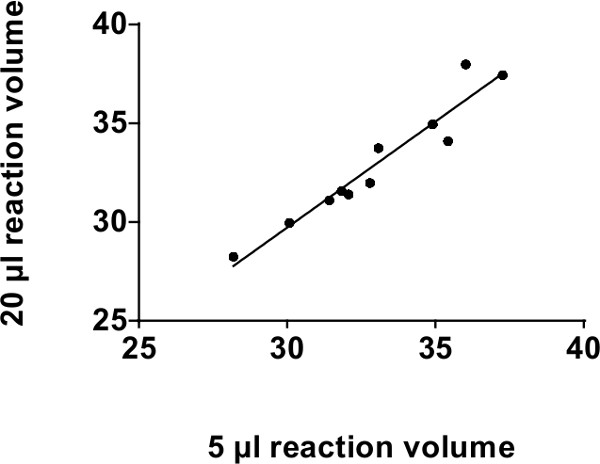
Figura 2: A análise de co-relação de 96 poços convencionais plataformas de PCR: A co-relação entre 20 ul e 5 ul em volumes de reacção de TaqMan qPCR em tempo real, utilizando uma plataforma padrão em placas de 96 poços em valores de CT (39 ciclos). Foram comparados quatro microRNAs diferentes (miR-375, miR-30c, 30d e miR-miR-7) em 4 amostras de soro e plasma humanos diferentes. Apenas os 11 pontos de dados estão representados graficamente uma vez que os outros eram indetectáveis. R 2 = 0,92, p = 0,0002.
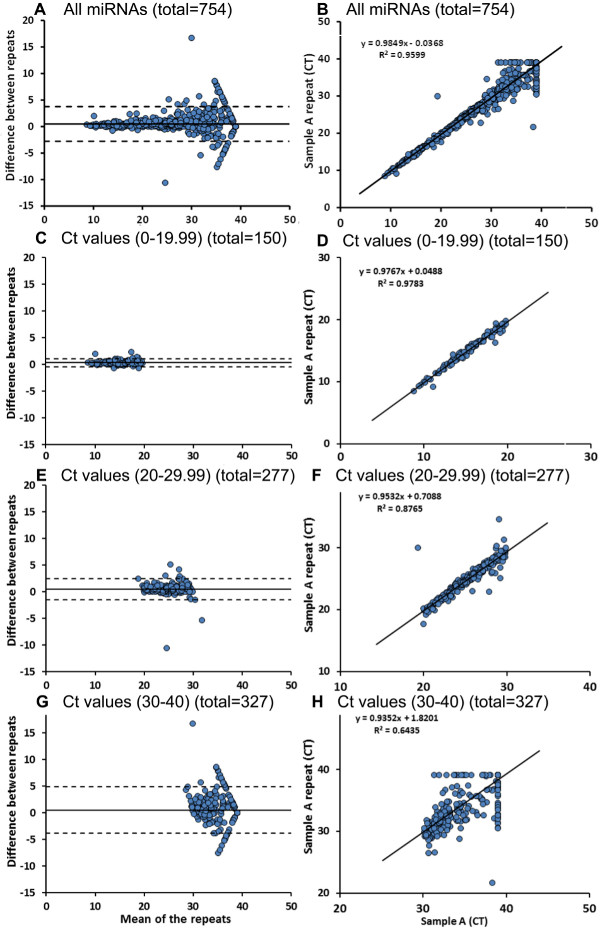
. Figura 3: Circulação miRNA profiling usando cartões de matriz microfluidics Usando 2 cartões microfluidics (cartão-A e cartão-B), um perfil de 754 miRNAs é gerado (A - B). Como mostrado aqui, usamos mesma amostra para 2 matriz microfluidics corre para verificar a reprodutibilidade dos resultados de cartão de microfluídica. Observou-se uma expressão similar de miRNAs no geral, com valores Ct entre 0-19,99 (C - D). Há poucos miRNAs (16 de 277) com valores Ct entre 20-29,99 e significativas diferenças entre repetição runs (E - F). Oitenta e nove de 327microRNAs com valores mais elevados de Ct (30-40) apresentaram diferenças significativas entre os dois runs (G - H). Os dados são analisados por meio do teste T pareado. Por favor, clique aqui para ver uma versão maior desta figura.
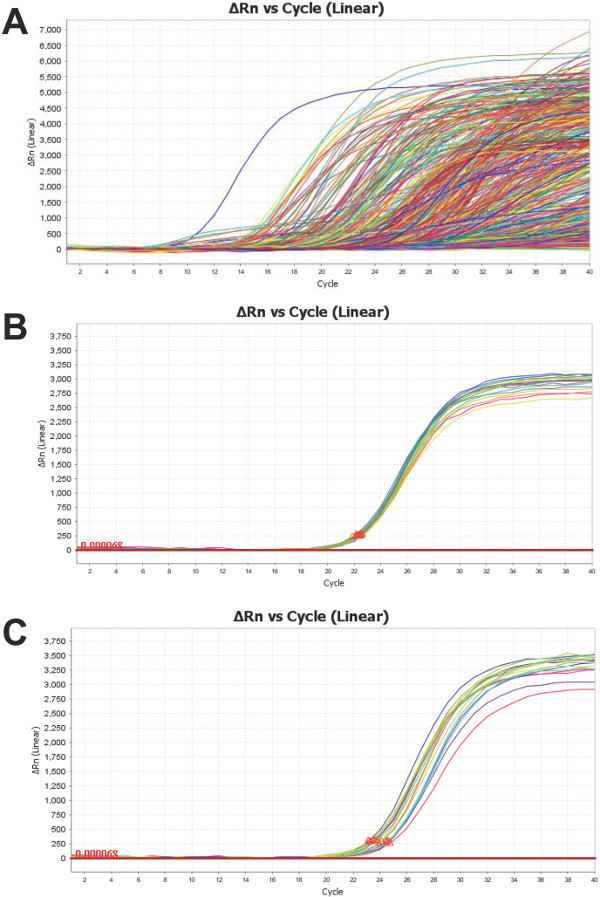
Figura 4: Perfil Representante da Curves PCR amplificação do produto: é uma figura representativa das curvas (A) de amplificação combinados de todos os alvos miRNA para uma amostra de plasma humano. Um ensaio para U6 (a controle comum ncRNA) é colocado em cada subarray. A amostra em (B) demonstra baixa variabilidade (SD <0,5), enquanto que (C) mostra alto desvio padrão (SD> 0,5) dentro U6 replica. Ambas as amostras são total RNA isolado a partir de plasma humano. Por favor, clique aqui para ver uma versão maior desta figura.

Figura 5: Análise do QC Nanofluidics Arrays: controle de qualidade (QC) imagens de uma matriz nanofluidics carregado corretamente (esquerda) e uma matriz nanofluidics carregado incorretamente (à direita). O corante passiva, ROX (presente na mistura reagente qPCR), fluorescência para indicar um carregado corretamente através de buracos. A matriz da direita tem vários subarrays / furos de passagem que não são carregados com a mistura reagente qPCR e estes devem ser identificados como reações de PCR falso-negativos.
| Componentes | Volume por reacção 5 ul (uL) |
| Tampão RT (10x) | 0,5 |
| dNTPs (100 mM) | 0,05 |
| RNase inibidor | 0,6 |
| Água livre de nuclease | 1,39 |
| Transcriptase Reversa | 0.33 |
| O volume total | 2.33 |
Tabela 1A: RT componentes da mistura reagente em 5 ul de uma reacção de RT preparação para TaqMan qPCR em tempo real, utilizando uma plataforma padrão placa de 96 poços.
| Componentes | Volume por reacção 5 ul (uL) | Volume por reacção de 20 ul (uL) |
| Rápido mastermix PCR (2x) | 2,5 | 10 |
| Ensaio TaqMan qPCR (20x) * | 0,25 | 1 |
| Livre de nucleaseágua | 1,45 | 5,8 |
| Volume Total: | 4.2 | 16.8 |
* Componente de ensaio com base na miARN seleccionado testado.
Tabela 1B: qPCR componentes da mistura reagente em um de 5 ou 20 uL de reacção para preparação qPCR em tempo real TaqMan qPCR utilizando uma plataforma padrão placa de 96 poços.
| Componentes | Volume por reacção de (l) |
| Megaplex RT Primers (10x) | 0,8 |
| dNTPs com dTTP (100 mM) | 0,2 |
| MultiScribe transcriptase inversa (50 U / ul) | 1,5 |
| 10X RT Tampão | 0,8 |
| MgCl2 (25 mM) | 0,9 |
| Inibidor de RNase & # 160; (20 U / ul) | 0,1 |
| Água livre de nuclease | 0,2 |
| Total | 4,5 |
Quadro 2A: RT componentes da mistura reagente em um 5 jul RT preparação reação para o painel TLDA miRNA.
| Componentes | Volume por reacção de (l) |
| TaqMan PreAmp Mastermix (2x) | 12.5 |
| Megaplex pré-amplificador Primers (10x) | 2,5 |
| Água livre de nuclease | 7,5 |
| Total | 22.5 |
Quadro 2B: pré-amplificador componentes da mistura reagente em uma preparação de reacção 25 mL de pré-amplificação para o painel TLDA miRNA.
"Cellspacing =" 0 "> Componentes Volume por reacção de (l) Megaplex RT Primers (10x) 0,75 dNTPs com dTTP (100 mM) 0,15 MultiScribe transcriptase inversa (50 U / ul) 1,5 10X RT Tampão 0,75 MgCl2 (25 mM) 0,9 Inibidor de RNase (20 U / ul) 0,09 Água livre de nuclease 0,35 Total 4,5Tabela 3A: RT componentes da mistura reagente em um 5 jul RT preparação reação para o painel nanofluidics miRNA-base da sonda.
| Componentes | Volume por reacção de (l) |
| TaqMan PreAmp Mastermix (2x) | 12.5 |
| Megaplex pré-amplificador Primers (10x) | 2,5 |
| Água livre de nuclease | 7,5 |
| Total | 22.5 |
Tabela 3B:. Pré-amplificador componentes da mistura reagente em uma preparação de reacção 25 mL de pré-amplificação para o painel nanofluidics miRNA-base sonda NOTA: O complementar interativo excel planilhas fornecidas para as três plataformas, já representa a adição de 5% para compensar a pipetagem erro.
| Amostra A | Amostra A repetição |
| 15,535 | 16,156 |
| 15,471 | 15,652 |
| 15.623 | 16,063 |
| 15,963 | 15,889 |
| 14,006 | 13.993 |
| 14,502 | 14,623 |
| 14,907 | 14,384 |
| 13,732 | 14,946 |
Tabela 4: Circulação miRNA profiling U6 usando microfluidics cartão matriz. Usando duas placas de matriz microfluidos (A e B). Ct-valores de miRNA controle U6 da Amostra A (total = 8). Controle U6 consistente abundância miRNA observada entre as corridas.
Discussão
Os passos críticos dentro dos protocolos de qPCR à base de sondas para obter resultados precisos e reprodutíveis são certificar-se 1) o mesmo volume e concentração de RT-produto é carregado em cada reacção qPCR, 2) as proporções correctas e volumes dos componentes necessários para reação qPCR são preparados e bem misturado, 3) os volumes corretos e coerentes são adicionados a cada reação qPCR, e 4) a preparação e carregamento de cada mistura de amostra e reação se completa no menor espaço de tempo possível, enquanto ainda consciente de as etapas críticas acima mencionado anteriormente.
Os cartões de matriz microfluidos precisa centrífuga adequado e baldes específicos. Cada balde pode conter até 3 cartões (carregado / vazio). Sempre garantir que todos os 3 slots de um balde são ocupados eo balde é equilibrado, colocando balde semelhante (esta balde também deve conter três cartões, vazios ou cheios) no slot oposto da centrífuga. Além de colocar o cartão no bucketitular t, certifique-se de que os oito reservatórios projetar para cima e poços de reacção enfrentar a parede externa da centrífuga. Rodar as placas a 331 xg durante 1 min à temperatura ambiente. Após a primeira rodada, abra a centrífuga e visualmente garantir a mistura de reacção foi distribuída através dos 384 poços. Repita o spin em mesmas configurações para mais uma vez. Remova a placa do balde e assegurar que o nível de mistura de reacção em cada um dos 8 reservatórios é uniforme. Qualquer inconsistência nos volumes líquidos deixados nos reservatórios fazer o cartão inapropriado usar mais.
Para ter a mesma concentração do produto de RT, mesma concentração de entrada de ARN é adicionado em cada reacção de RT. A concentração de ARN total é medido usando um micro-volumespectrophotometer. A relação observada 260/280 pode ser tão baixo como 1.3 para o ARN isolado a partir de plasma / soro; isto não parece ter um efeito sobre o processo relacionados com qPCR jusante ou dados gerados 30. Da mesma forma, a razão de 260/280as amostras de ARN foram testadas aqui entre 1,3-1,7 com nenhuns efeitos anormais no qPCR observados.
Quando o conteúdo de RNA usando amostras baixos, tais como aqueles a partir de fluidos biológicos, pode ser difícil de quantificar o ARN antes do processamento. Recomendamos o uso de sintéticos spike-in no isolamento do RNA, bem como as etapas de transcrição reversa. Em nossa experiência, os candidatos Arabidopsis thaliana miRNA (ATH-miR-159A e ATH-miR-172a) têm preferência sobre Caenorhabditis elegans miRNAs (como cel-miR-39 ou cel-miR-54), que, em nossa experiência pode ter maior homologia do que aqueles a partir de A. thaliana. O uso de tais específicos do estágio spike-in pode ser responsável pela normalização dos dados de miRNA em múltiplas amostras testadas em momentos diferentes. Utilizando um volume de entrada fixa de ARN para a reacção de síntese de ADNc também é recomendado 32,33.
Os três protocolos baseados sonda para miRNA quantificação descrito aqui requerem quantidades variáveisdo total de entrada de RNA, diferentes fluxos de trabalho e custos. Cada um dos fluxos de trabalho são concebidos para atender a diferentes processamentos com base no número de alvos de miARN e o número de amostras a serem analisadas. Com aumento da taxa de transferência (96 rxn 384 rxn 3072 rxn), o custo por reacção diminui com um aumento na quantidade de dados obtidos através de uma unidade de tempo. Uma vez que todas estas plataformas utilizar TaqMan química, a qualidade dos dados obtidos pode ser esperado que seja semelhante. TaqMan qPCR é um método bem estabelecido para identificação da abundância de miARNs em amostras de soro / plasma 9,11,27. Embora as três plataformas discutidos aqui partilham a mesma composição química, uma diminuição no volume de reacção conduz a uma diminuição da gama dinâmica de detecção de transcrição (RJ Farr et ai., Dados não publicados). A 96 poços plataforma qPCR é um rendimento mais baixo, mas plataforma de alta sensibilidade e, em nossa opinião, o "padrão ouro" para todos (ou corante base) à base de sonda plataformas de PCR. No entanto, isso podenão ser a plataforma mais econômica ou eficiente se várias centenas ou milhares de amostras estão sendo analisadas e para vários microRNAs. Microfluídica (TLDA) e nanofluidics plataformas (OA) são plataformas de alto rendimento / conteúdos projetados para permitir a aquisição de dados maior em um tempo menor. Embora as diferenças de lote foram observadas nas placas de TLDA, isto pode ser minimizado pela requerente TLDA cartões do mesmo lote. Observa-se que a plataforma TLDA (Figura 3) mostrou variação significativa em 17% dos mid - miRNAs de baixa abundância, quando testado utilizando a mesma amostra em diferentes lotes de TLDA cartões. Recomendamos, portanto, usando os mesmos números de lote para análise utilizando TLDA cartões. Essa variação também pode ser devido à variabilidade técnica, incluindo quaisquer erros de carga e de pipetagem potenciais. No entanto, recomendamos ordenação / solicitando o mesmo lote de TLDA cartões. Não houve variação significativa lote foi observada na plataforma OA. Apesar disso, TaqMan experimen basetal qPCR abordagens oferecem facilidade para medir a abundância de miRNAs em amostras de plasma / soro. As abordagens de rendimento baixo, médio e alto discutidos aqui oferecem a flexibilidade para analisar uma série de amostras e miRNAs usando um (baixo ruído de fundo) química altamente eficiente, reprodutível e limpo.
Divulgações
Artigo custos de processamento / publicação foram cobertos pela Life Technologies, na sequência da aceitação do manuscrito para publicação.
Agradecimentos
Todos os autores agradecem o apoio de infra-estrutura do NHMRC CTC, da Universidade de Sydney, a Juvenile Diabetes Research Foundation (JDRF), Austrália e Cooper Fundação Rebecca, Austrália. Esta pesquisa foi financiada através de doações do Conselho Australiano de Investigação (FT110100254) eo JDRF, Austrália (CRN201314) para AAH WW, RJF e MVJ realizada toda a experimentação laboratório molhado, ASJ realizada análise dos dados. WW escreveu o primeiro rascunho. AAH planejou o estudo e análise dos dados. Todos os autores leram e aprovaram a versão final do manuscrito, figuras e planilhas submetidos para publicação.
Materiais
| Name | Company | Catalog Number | Comments |
| 96 well-platform | |||
| For Reverse transcription | |||
| TaqMan MicroRNA Assays INV SM | Applied Biosystems | 4427975 | https://www.lifetechnologies.com/au/en/home/life-science/pcr/real-time-pcr/real-time-pcr-assays/mirna-ncrna-taqman-assays/single-tube-mirna-taqman-assays.html?ICID=search-4427975 The assays comes as a pack of RT primers and PCR primer. |
| TaqMan MicroRNA Reverse Transcription Kit | Applied Biosystems | 4366597 or 4366596 | https://www.lifetechnologies.com/order/catalog/product/4366597?ICID=search-4366597 or http://www.lifetechnologies.com/order/catalog/product/4366596?ICID=search-4366596 The TaqMan MicroRNA Reverse Transcription Kit is the same kit used for reverse transcription in all the threeTaqMan platform- 96-well platform, TLDA and OpenArray. |
| For qPCR run on a 96-well platform | |||
| 2x TaqMan Fast Universal PCR Master Mix, no AmpErase UNG | Applied Biosystems | 4367846 | https://www.lifetechnologies.com/order/catalog/product/4367846?ICID=search-4367846 The 2X TaqMan Fast Universal PCR Master Mix is used for qPCR on the two TaqMan platforms-96-well platform and TLDA. |
| MicroAmp Optical Adhesive Film | Applied Biosystems | 4311971 | https://www.lifetechnologies.com/order/catalog/product/4311971?ICID=search-product |
| MicroAmp Fast Optical 96-Well Reaction Plate, 0.1 ml | Applied Biosystems | 4346907 | https://www.lifetechnologies.com/order/catalog/product/4346907?ICID=search-product |
| Microfluidics platform (TLDA) | |||
| For Reverse transcription | |||
| Refer to 96-well platform TaqMan MicroRNA Reverse Transcription Kit kit (4366597 or 4366596) (as shown above). | |||
| Megaplex RT Primers, Human Pool A v2.1 | Applied Biosystems | 4399966 | https://www.lifetechnologies.com/order/catalog/product/4399966?ICID=search-4399966 The Megaplex RT Primers, Human Pool A v2.1 is used for RT on the two TaqMan platforms- TLDA and OpenArray. |
| Megaplex RT Primers, Human Pool B v3.0 | Applied Biosystems | 4444281 | https://www.lifetechnologies.com/order/catalog/product/4444281?ICID=search-4444281 The Megaplex RT Primers, Human Pool B v3.0 is used for RT on the two TaqMan platforms- TLDA and OpenArray. |
| OR | OR | ||
| Megaplex Primer Pools, Human Pools Set v3.0 | Applied Biosystems | 4444750 | http://www.lifetechnologies.com/order/catalog/product/4444750?ICID=search-4444750 The Megaplex Primer Pools, Human Pools Set v3.0 is used for RT on the two TaqMan platforms- TLDA and OpenArray. |
| For Pre-amplification | |||
| TaqMan PreAmp Master Mix | Applied Biosystems | 4391128 or 4488593 | https://www.lifetechnologies.com/order/catalog/product/4391128?ICID=search-4391128 or http://www.lifetechnologies.com/order/catalog/product/4488593 The TaqMan PreAmp Master Mix is used for pre-amplification of qPCR on the two TaqMan platforms- TLDA and OpenArray. |
| Megaplex PreAmp Primers, Human Pool B v3.0 | Applied Biosystems | 4444303 | https://www.lifetechnologies.com/order/catalog/product/4444303?ICID=search-4444303 The Megaplex PreAmp Primers, Human Pool B v3.0 is used for pre-amplification of qPCR on the two TaqMan platforms- TLDA and OpenArray. |
| Megaplex PreAmp Primers, Human Pool A v2.1 | Applied Biosystems | 4399233 | https://www.lifetechnologies.com/order/catalog/product/4399233?ICID=search-4399233 The Megaplex PreAmp Primers, Human Pool A v2.1 is used for pre-amplification of qPCR on the two TaqMan platforms- TLDA and OpenArray. |
| 1x TE Buffer (100 ml) | Invitrogen | 12090-015 | https://www.lifetechnologies.com/order/catalog/product/12090015?ICID=search-product The 1x TE Buffer is used for pre-amplification of qPCR on the two TaqMan platforms- TLDA and OpenArray. |
| To load and run the 384 well microfluidics (TLDA) card | |||
| TaqMan Array Human MicroRNA A+B Cards Set v3.0 | Applied Biosystems | 4444913 | https://www.lifetechnologies.com/order/catalog/product/4444913 |
| Nanofluidics platform (OpenArray) | |||
| For Reverse transcription | |||
| Refer to 96-well platform TaqMan MicroRNA Reverse Transcription Kit kit (4366597 or 4366596) (as shown above). | |||
| For Pre-amplification | |||
| Refer to TLDA pre-amplification reagents (as shown above) | |||
| To load and run the slides | |||
| TaqMan OpenArray Human MicroRNA Panel, QuantStudio 12K Flex (1 panel) | Applied Biosystems | 4470187 | https://www.lifetechnologies.com/order/catalog/product/4470187?ICID=search-4470187 |
| QuantStudio 12K Flex OpenArray Accessories Kit | Applied Biosystems | 4469576 | https://www.lifetechnologies.com/order/catalog/product/4469576?ICID=search-4469576 |
| OpenArray 384-well Sample Plates | Applied Biosystems | 4406947 | https://www.lifetechnologies.com/order/catalog/product/4406947?ICID=search-4406947 |
| TaqMan OpenArray Real-Time PCR Master Mix | Applied Biosystems | 4462159 | https://www.lifetechnologies.com/order/catalog/product/4462159?ICID=search-4462159 |
| OpenArray AccuFill System Tips | Applied Biosystems | 4457246 | https://www.lifetechnologies.com/order/catalog/product/4457246?ICID=search-4457246 |
| Others | |||
| Nuclease-Free Water | Qiagen | 129117 | http://www.qiagen.com/products/catalog/lab-essentials-and-accessories/nuclease-free-water |
Referências
- Ambros, V. The functions of animal microRNAs. Nature. 431, 350-355 (2004).
- Alvarez-Garcia, I., Miska, E. A. MicroRNA functions in animal development and human disease. Development. 132, 4653-4662 (2005).
- Bartel, D. P. MicroRNAs: genomics, biogenesis, mechanism, and function. Cell. 116, 281-297 (2004).
- Joglekar, M. V., Wei, C., Hardikar, A. A. Quantitative estimation of multiple miRNAs and mRNAs from a single cell. Cold Spring Harb Protoc. 2010, pdb prot5478(2010).
- Joglekar, M. V., Parekh, V. S., Hardikar, A. A. Islet-specific microRNAs in pancreas development, regeneration and diabetes. Indian J Exp Biol. 49, 401-408 (2011).
- Farr, R. J., Joglekar, M. V., Taylor, C. J., Hardikar, A. A. Circulating non-coding RNAs as biomarkers of beta cell death in diabetes. Pediatr Endocrinol Rev. 11, 14-20 (2013).
- Joglekar, M. V., Joglekar, V. M., Hardikar, A. A. Expression of islet-specific microRNAs during human pancreatic development. Gene Expr Patterns. 9, 109-113 (2009).
- Iorio, M. V., Croce, C. M. MicroRNA profiling in ovarian cancer. Methods Mol Biol. 1049, 187-197 (2013).
- Mitchell, P. S., et al. Circulating microRNAs as stable blood-based markers for cancer detection. P Natl Acad Sci USA. 105, 10513-10518 (2008).
- Mattie, M. D., et al. Optimized high-throughput microRNA expression profiling provides novel biomarker assessment of clinical prostate and breast cancer biopsies. Mol Cancer. 5, 24(2006).
- Zhu, W., Qin, W., Atasoy, U., Sauter, E. R. Circulating microRNAs in breast cancer and healthy subjects. BMC Res Notes. 2, 89(2009).
- Zhu, H. T., et al. Identification of suitable reference genes for qRT-PCR analysis of circulating microRNAs in hepatitis B virus-infected patients. Mol Biotechnol. 50, 49-56 (2012).
- Yamada, H., Itoh, M., Hiratsuka, I., Hashimoto, S. Circulating microRNAs in autoimmune thyroid diseases. Clin Endocrinol (Oxf). , (2014).
- Liu, R., et al. Serum MicroRNA Expression Profile as a Biomarker in the Diagnosis and Prognosis of Pancreatic Cancer. Clinical Chemistry. 58, 610-618 (2012).
- Gilad, S., et al. Serum microRNAs are promising novel biomarkers. PLoS One. 3, e3148(2008).
- Joglekar, M. V., Parekh, V. S., Mehta, S., Bhonde, R. R., Hardikar, A. A. MicroRNA profiling of developing and regenerating pancreas reveal post-transcriptional regulation of neurogenin3. Dev Biol. 311, 603-612 (2007).
- Hardikar, A. A., Farr, R. J., Joglekar, M. V. Circulating microRNAs: understanding the limits for quantitative measurement by real-time PCR. J Am Heart Assoc. 3, e000792(2014).
- Wu, C., et al. Diagnostic and Prognostic Implications of a Serum miRNA Panel in Oesophageal Squamous Cell Carcinoma. PLoS One. 9, e92292(2014).
- McDonald, M. K., Capasso, K. E., Ajit, S. K. Purification and microRNA profiling of exosomes derived from blood and culture media. J Vis Exp. , e50294(2013).
- Joglekar, M. V., et al. The miR-30 family microRNAs confer epithelial phenotype to human pancreatic cells. Islets. 1, 137-147 (2009).
- Genda, Y., et al. microRNA changes in the dorsal horn of the spinal cord of rats with chronic constriction injury: A TaqMan(R) Low Density Array study. Int J Mol Med. 31, 129-137 (2013).
- Wang, B., et al. Systematic evaluation of three microRNA profiling platforms: microarray, beads array, and quantitative real-time PCR array. PLoS One. 6, e17167(2011).
- Cuk, K., et al. Circulating microRNAs in plasma as early detection markers for breast cancer. Int J Cancer. 132, 1602-1612 (2013).
- Morrison, T., et al. Nanoliter high throughput quantitative PCR. Nucleic Acids Res. 34, e123(2006).
- Git, A., et al. Systematic comparison of microarray profiling, real-time PCR, and next-generation sequencing technologies for measuring differential microRNA expression. RNA. 16, 991-1006 (2010).
- Benes, V., Castoldi, M. Expression profiling of microRNA using real-time quantitative PCR, how to use it and what is available. Methods. 50, 244-249 (2010).
- Chen, C., et al. Real-time quantification of microRNAs by stem-loop RT-PCR. Nucleic Acids Res. 33, e179(2005).
- Schmittgen, T. D., et al. Real-time PCR quantification of precursor and mature microRNA. Methods. 44, 31-38 (2008).
- Kodani, M., et al. Application of TaqMan low-density arrays for simultaneous detection of multiple respiratory pathogens. J Clin Microbiol. 49, 2175-2182 (2011).
- Taylor, C. J., Satoor, S. N., Ranjan, A. K., Pereirae Cotta,, V, M., Joglekar, M. V. A protocol for measurement of noncoding RNA in human serum. Exp Diabetes Res. 2012, 168368(2012).
- TPF & SPF File Download Options [Internet]. , Available from: http://www.appliedbiosystems.com/absite/us/en/home/products/tpf-spf-download.html (2014).
- Turchinovich, A., Weiz, L., Langheinz, A., Burwinkel, B. Characterization of extracellular circulating microRNA. Nucleic Acids Res. 39, 7223-7233 (2011).
- Li, Y., Kowdley, K. V. Method for microRNA isolation from clinical serum samples. Anal Biochem. 431, 69-75 (2012).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados