Method Article
Ein 3D-humanem Lungengewebe Modell für funktionelle Studien auf
In diesem Artikel
Zusammenfassung
Human tuberculosis infection is a complex process, which is difficult to model in vitro. Here we describe a novel 3D human lung tissue model that recapitulates the dynamics that occur during infection, including the migration of immune cells and early granuloma formation in a physiological environment.
Zusammenfassung
Tuberkulose (TB) hält immer noch eine große Bedrohung für die Gesundheit der Menschen weltweit, und es besteht ein Bedarf an kostengünstigen, aber zuverlässigen Modelle, die uns helfen zu verstehen, die Krankheitsmechanismen und vorab die Entdeckung neuer Behandlungsmöglichkeiten. In-vitro-Zellkulturen von Monoschichten oder Co-Kulturen fehlen die dreidimensionale (3D) Umgebung und Gewebereaktionen. Hier beschreiben wir eine innovative In-vitro-Modell eines menschlichen Lungengewebe, das Versprechen hält, um ein wirkungsvolles Werkzeug für die Untersuchung der komplexen Ereignisse, die während einer Infektion mit Mycobacterium tuberculosis (M. tuberculosis) auftreten. Das Gewebemodell 3D besteht aus gewebespezifischer Epithelzellen und Fibroblasten, die in einer Matrix aus Kollagen auf der Oberseite einer porösen Membran kultiviert werden. Bei Lufteinwirkung die Epithelzellen stratify und sezer Schleim an der apikalen Seite. Durch die Einführung von primären humanen Makrophagen, die mit M. infiziert tuberculosis zur Gewebemodusl, haben wir gezeigt, dass die Immunzellen wandern in das infizierte Gewebe und frühen Stadien der TB Granulome bilden. Diese Strukturen rekapitulieren die besonderes Merkmal der menschlichen Tuberkulose, der Granulom, die sich grundlegend oder nicht gewöhnlich in weit verbreiteten experimentellen Tiermodellen festgestellt ist. Das organotypische Kultur Verfahren ermöglicht die 3D-Visualisierung und robuste quantitative Analyse, die entscheidenden Informationen über räumliche und zeitliche Merkmale der Wirtszelle-Pathogen-Interaktionen bietet. Zusammengenommen bietet das Lungengewebe-Modell einen physiologisch relevanten Gewebemikroumgebung für Untersuchungen an TB. Somit hat das Lungengewebe-Modell möglichen Auswirkungen auf sowohl grundlegende mechanistische und angewandten Studien. Wichtiger ist, ermöglicht das Modell zusätzlich oder Manipulation einzelner Zelltypen, die dadurch ihre Verwendung aufweitet zum Modellieren einer Vielzahl von Infektionskrankheiten der Lungen auf.
Einleitung
Bei Menschen, Antworten auf eine Infektion, Entzündung im Gewebe, Zellrekrutierung, Gewebeumbau und die Regulierung der Gewebehomöostase sind komplexe Veranstaltungen mit verschiedenen Zelltypen. Daher sind diese Verfahren am besten in der lokalen Gewebeumgebung untersucht. Bisher hat dies vor allem möglich unter Verwendung von experimentellen Tiermodellen. Jedoch die weit verbreiteten Versuchstieren halten viele Grenzen, da sie oft zu antworten Pathogenen in einer anderen Weise als Menschen und auch einen anderen Verlauf der Erkrankung 1 anzuzeigen. Ein menschlicher Lunge in vitro Gewebemodell hält der Möglichkeiten, um spezifische Immunantworten in der menschlichen Lunge zu untersuchen.
Menschlicher Tuberkuloseinfektion (TB) ist hauptsächlich eine Erkrankung der Lunge. Mycobacterium tuberculosis (M. tuberculosis), den Erreger der Tuberkulose, die Lunge über Aerosol-Tröpfchen, die an den Alveolarraum, wobei die Bakterien durch Lungen dendri verschlungen transportiert werden erreichttic Zellen und Alveolarmakrophagen als Teil der angeborenen Immunantwort auf die Infektion 2,3. Phagozytose der Erreger führt zur Abschottung des Fehlers innerhalb eines Phagosom und führt zur Neutralisation und Tötung des Erregers durch die Phagozyten ideal. Bis zu 50% der Personen, um M. ausgesetzt Tuberkulose wird angenommen, dass in der Lage, die Infektion durch die angeborene Immunantwort 4 klar sein. Weitere Ergebnisse der Infektion sind Freigabe durch das adaptive Immunsystem in einem späteren Stadium, latente Infektion oder im schlimmsten Fall chronisch aktiver Erkrankung 5.
Zuvor gab es für das Studium der menschlichen TB wurden keine In-vitro-Gewebemodelle. Einzelzellkulturen von menschlichen Makrophagen oder andere Zellen des peripheren Blutes wurden häufig verwendet 6,7. Der Nachteil dieses Ansatzes ist, dass sie die Bewegungen der verschiedenen Zelltypen gleichzeitig zusammen in einem Lungengewebe zu M. ausgesetzt gewesen sein kann Tuberkulose . Somit besteht ein Bedarf nach einem in vitro-Modell, um funktionelle und mechanistische Studien über TB auszuführen. Die zellbasierten in vitro hier beschriebenen menschlichen Lungengewebe-Modell wurde ursprünglich von unserer Gruppe für Studien über dendritische Zellfunktionen 8 etabliert. Wir haben dieses Verfahren für die Untersuchung von TB angepasst.
Das menschliche Lungengewebe Modell hier präsentierten aus gewebespezifischer Epithelzellen und Fibroblasten 8. Diese Zellen sind in einer Matrix aus Kollagen auf der Oberseite der porösen Membran in einem Transwell-Einsatz und bilden Strukturen ähnlich normalen menschlichen Lungengewebes (1) kultiviert. An der Luft beginnen die Zellen, um Schleim in der apikalen Seite 8 sekretieren. Durch Implantation von humanen primären Makrophagen, die mit M. infiziert tuberculosis zum Modell haben wir beobachtet, wie die Immunzellen wandern in das Gewebe und frühen Stadien der TB Granulome 9 bilden. Dies ist die erste menschliche Gewebemodell described für TB, und es stellt ein vielversprechendes Werkzeug für die Untersuchung angeborenen Immunantwort auf Tuberkulose und anderen Erkrankungen der Lunge. Bisher haben wir nur Monozyten und Makrophagen als Immunzellen im Modell verwendet, aber der Grad der Komplexität kann durch Aufnahme zusätzlicher relevanter Zelltypen erhöht werden.
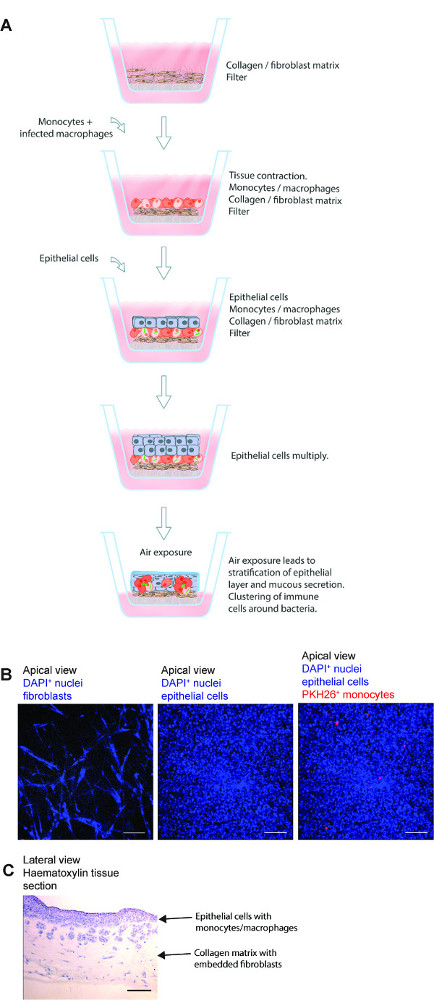
Abbildung 1. Schematische Darstellung des Lungengewebes Modell. (A) wird das Modell der menschlichen Lunge spezifische Epithelzellen besteht, M. Tuberkulose infizierten primären Makrophagen und roten Farbstoff markiert Monozyten auf Kollagen eingebetteten Fibroblasten auf einem Transwell-Filter hergestellt ausgesät. Exposition der Gewebemodell, um Luft initiiert Produktion von extrazellulären Matrixproteinen, Schleimabsonderung und Schichtung nach dem Epithel. Das so entwickelte 3D-Gewebemodell ist ein nützliches Tool, um M. studieren tuberculosis-Infektion in einer Umgebung, die closely ähnelt einem menschlichen Lunge. (B) Repräsentative mikroskopische Bilder der verschiedenen Schritte bei der Herstellung der Gewebemodell. (C) die komplette Struktur des Lungenmodell Gewebeschnitt. Scale -. 100 & mgr; m Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
Protokoll
Hinweis: Menschliche periphere Blut von gesunden Blutspendern anonym an der Blutbank der Linköping University Hospital gekauft wurde Schweden als Quelle der Immunzellen für diese Studie verwendet. Dieses Protokoll wird für 24 mm 6-Well-Platte Einsätze konzipiert. Direkte Anpassung an andere Formate auch nicht, da die Gewebemusterverträge vertikal und horizontal während der Entwicklung empfohlen.
1. Herstellung von Materialien, Medien und Kultur der Bakterien / Zelllinien
- Bakterienkultur:
- Wachsen die Mykobakterien-Stamm M. tuberculosis H37Rv tragenden pFPV2 Plasmid zu exprimieren konstitutiv das grün fluoreszierende Protein (GFP), in Middlebrook 7H9-Medium, das 0,05% Tween-80, 0,5% Glycerin, Kanamycin (20 ug / ml), ergänzt mit Middle Albumin, Dextrose und Anreicherung Katalase ( Middle ADC Enrichment), bei 37 ° C mit 5% CO 2 für 7-10 Tage.
Hinweis: Alle Versuchsschritts mit Live-virulenten M. tuberculosis-Stämme sollten in einem BSL-3-Anlage durchgeführt werden.
- Wachsen die Mykobakterien-Stamm M. tuberculosis H37Rv tragenden pFPV2 Plasmid zu exprimieren konstitutiv das grün fluoreszierende Protein (GFP), in Middlebrook 7H9-Medium, das 0,05% Tween-80, 0,5% Glycerin, Kanamycin (20 ug / ml), ergänzt mit Middle Albumin, Dextrose und Anreicherung Katalase ( Middle ADC Enrichment), bei 37 ° C mit 5% CO 2 für 7-10 Tage.
- Vorbereitung 1x Dulbecco-modifiziertes Eagle-Medium (DMEM) vollständigem Medium (ergänzt mit 1 mM Natriumpyruvat, 2 mM L-Glutamin, 100 U / ml Penicillin, 100 ug / ml Streptomycin, 10 mM HEPES, 0,1 mM nicht-essentielle Aminosäuren und 10 % hitzeinaktiviertem fötalem Rinderserum (FBS)). Bereiten Sie auch antibiotikafreies DMEM Vollmedium.
- 1fach Minimum Essential Medium (MEM) vollständigem Medium (1 mM Natriumpyruvat, 2 mM L-Glutamin, 100 U / ml Penicillin, 100 ug / ml Streptomycin, 10 mM HEPES, 0,1 mM nicht-essentiellen Aminosäuren und 10% hitze inaktiviertes fötales Rinderserum (FBS)).
- Herstellung von Fibronektin / Collagen-beschichteten Kolben (insgesamt 10 ml):
- Pipette 8,8 ml sterile 1x phosphatgepufferter Kochsalzlösung (PBS) in ein sauberes Röhrchen. 1 ml Rinderserumalbumin (1 mg / ml), 100 ul Typ I Rinderkollagen (3 mg / ml) und 100 ul rekombinanter human Fibronectin (1 mg / ml).
- Mischen Sie die Lösung durch Drehen des Rohres Upside-Down-5-mal. Kolben werden mit Fibronectin / Kollagen-Lösung (für eine T-25 und 2 ml für eine T-75-Kolben 1 ml) beschichtet. Lassen Sie es O / N bei 37 ° C. Nach der Inkubation, entfernen Sie die Lösung und speichern die beschichteten Kolben bei RT.
Anmerkung: Die gesammelte Lösung kann bei 4 ° C gelagert und dreimal wiederverwendet werden. Lagerung für mehr als 2 Wochen können die Flüssigkeits bis braun (Kristallbildung), wobei auf sie weggeworfen werden muß drehen.
- Kultur von Fibroblasten:
- Aufbau und zur Pflege MRC-5, (eine humane Lungenfibroblasten-Zelllinie von normalem Lungengewebe eines 14 Wochen alten männlichen Fötus stammen), in vollständigem DMEM in 5% CO 2 bei 37 ° C. Verwenden Sie die Fibroblasten an Passagen 24-26 und wachsen, bis 70-80% konfluent.
Hinweis: MRC-5-Zelllinie bei Durchgang> 30 sind in der Regel, um die Morphologie zu verlieren und ist nicht für die Verwendung in der Gewebemodell empfohlen.
- Aufbau und zur Pflege MRC-5, (eine humane Lungenfibroblasten-Zelllinie von normalem Lungengewebe eines 14 Wochen alten männlichen Fötus stammen), in vollständigem DMEM in 5% CO 2 bei 37 ° C. Verwenden Sie die Fibroblasten an Passagen 24-26 und wachsen, bis 70-80% konfluent.
- Kultur von Epithelzellen:
- Erhalten 16HBE14o- (16HBE), einer immortalisierten menschlichen bronchialen Epithelzellen Zelllinie, die das differenzierte Morphologie und Funktion des normalen menschlichen Atemwegsepithelien behält, (dies war ein Geschenk von Dr. Dieter Gruenert, Mt. Zion Cancer Center, University of California, San Francisco , USA. 10). Kultur 16HBE Zellen in Fibronectin / Collagen-beschichteten Kolben und Aufrechterhaltung der Zellen in komplettem MEM in 5% CO 2 bei 37 ° C.
- Herstellung von 5x DMEM
- Vorbereitung 5x DMEM durch Auflösen von 13,4 g DMEM-Pulver und 3,7 g Natriumbicarbonat in 150 ml steriles destilliertes Wasser. Der pH-Wert des Mediums auf 7,3, auf ein Volumen von 200 ml und filtern es mit 0,22 um Membranfilter. Sammeln des gefilterten Mediums in einem sterilen Behälter und bis zur Verwendung bei Raumtemperatur gelagert.
2. Herstellung von Kollagen eingebetteten Fibroblasten
- Tauwetter gefrorenen Teilmengen von FBS und L-Glutamin in einem 37 ° C Wasserbad.Nach dem Auftauen halten die Proben auf Eis. Platzieren Natriumbicarbonat (71,2 mg / ml) und Gentamicin (50 mg / ml) bei 4 ° C. Pre-cool 50 ml Zentrifugenröhrchen und 10 ml sterilen Pipetten bis 4 ° C.
Anmerkung: Alle eingesetzten (mit Ausnahme des 5x DMEM) Materialien werden auf Eis vor der Verwendung gekühlt und alle Schritte werden auf Eis durchgeführt. Typ-I-Kollagen (1,1 mg / ml) muß kühl gehalten werden, da dies verhindert, dass die Verfestigung des Kollagens. - Bereiten Sie die Fibroblasten:
- Warm Trypsin in einem 37 ° C Wasserbad inkubieren ausreichender Menge mit Lungenfibroblasten (MRC-5-Zellen) für 10 min bei 5% CO 2 bei 37 ° C. Neutralisieren die Trypsin durch Zugabe von DMEM 1x komplett. Saugen Sie das Zellsuspension und Zentrifuge bei 300 xg für 5 min. Aspiration des Überstands und Resuspendieren der Zellen bei 2,3 × 10 5 Zellen / ml in DMEM-Voll. Legen Sie die Zellen auf Eis bis zum Gebrauch.
- Bereiten Sie die Pre-Mix:
- Fügen Sie den folgenden in ein Röhrchen "Pre-Mix" gekennzeichnet; 395ul 5x DMEM, 40 & mgr; L-Glutamin, 120 & mgr; l NaHCO 3 (71,2 mg / ml), 440 ul FBS, 5 & mgr; l Gentamicin (50 mg / ml), Volumen gesamt 1.000 & mgr; l, dann wirbeln die Vormischung gut und legte auf Eis.
Hinweis: Die angegebenen Volumina sind für einen 24 mm 6-Well-Kultureinsatzes. Berechnen Sie die für die Gesamtzahl der Einsätze erforderlichen spezifischen Mengen, aber fügen Sie eine zusätzliche, um sicher zu sein, ist genug Vormischung vorbereitet.
- Fügen Sie den folgenden in ein Röhrchen "Pre-Mix" gekennzeichnet; 395ul 5x DMEM, 40 & mgr; L-Glutamin, 120 & mgr; l NaHCO 3 (71,2 mg / ml), 440 ul FBS, 5 & mgr; l Gentamicin (50 mg / ml), Volumen gesamt 1.000 & mgr; l, dann wirbeln die Vormischung gut und legte auf Eis.
- Bereiten Sie die azellulären Kollagenmischung:
- Zu jeder Kultur 1 ml azellulären Kollagenmischung. Fügen Sie den folgenden, um einen 50 ml konischen Röhrchen auf Eis in der angegebenen Reihenfolge; 686 & mgr; l von 1,1 mg / ml Kollagen, 250 ul Pre-Mix und 64 ul 1x komplettem DMEM zu einem Gesamtvolumen von 1000 & mgr; l. Mischen Sie die Lösung auch die Gewährleistung und keine Luftblasen. Arbeiten Sie schnell und fügen Sie die Collagen an der Wand der Röhre, um Luftblasen zu vermeiden.
- 1 ml des zellfreien Schichtmischung an dem Einsatz in der Platte mit 6 Vertiefungen gelegt. Nicht jedes Medium zu der auch hinzufügen nicht außerhalb des Einsatzes. Imcubate für 30 min in einem 37 ° C Inkubator. Stellen Sie sicher, die azellulären Mischung umfasst den gesamten Einsatz ohne Luftblasen.
- Bereiten Sie das Mobil Kollagen-Mischung:
- Mischungskomponenten der Zellschicht in einem 50 ml konischen Röhrchen auf Eis in der folgenden Reihenfolge gehalten werden; 2 ml Collagen, 615 & mgr; Pre-Mix, 58 & mgr; l 1x komplettes DMEM und 327 & mgr; l Zellsuspension Lungenfibroblasten (MRC-5) bis zu 3.000 ul bilden das Gesamtvolumen. Jede Kultur erfordert 3 ml der Zell Kollagen-Mischung.
Entscheidender Schritt: Stellen Sie sicher, das Kollagen und Vormischung sorgfältig vor Zugabe der Zellsuspension mischen. Dadurch wird der pH-Wert der Kollagen neutralisieren toxische Wirkung auf die Fibroblasten zu vermeiden. - Hinzufügen der Zellschicht (3 ml) auf der Oberseite des azelluläre Kollagenschicht und Inkubation für 2 Stunden in einem 37 ° C Inkubator. Arbeiten Sie schnell und fügen Sie die Collagen an der Wand der Röhre, um Luftblasen zu vermeiden.
- Nach der Polymerisation 2 ml DMEM Komplett bis ter unten auf der 6-Well-Platte (unter der Einlage) und 24 h inkubiert.
HINWEIS: Wenn die Polymerisation trat nicht auf, entsorgen Sie die Platteneinsätze, die das Fibroblasten-Kollagenmatrix und Inbetriebnahme immer wieder. Der wahrscheinlichste Grund ein Fehler in der Addition oder falscher Volumen von einem der oben genannten Reagenzien.
- Mischungskomponenten der Zellschicht in einem 50 ml konischen Röhrchen auf Eis in der folgenden Reihenfolge gehalten werden; 2 ml Collagen, 615 & mgr; Pre-Mix, 58 & mgr; l 1x komplettes DMEM und 327 & mgr; l Zellsuspension Lungenfibroblasten (MRC-5) bis zu 3.000 ul bilden das Gesamtvolumen. Jede Kultur erfordert 3 ml der Zell Kollagen-Mischung.
3. Kontinuierliche Kultur des Fibroblasten-Kollagenmatrix
- Heben Sie den Einsatz mit einem sauberen Pinzette und saugen Sie den Kulturmedien aus dem Boden des Bohrlochs. 2 ml komplettem DMEM mit dem Boden der Vertiefung, gefolgt von 2 ml komplettem DMEM innerhalb des Einsatzes. Vermeidung der Einführung von Luftblasen unter dem Einsatz, da dies die Diffusion von Nährstoffen zwischen den äußeren und inneren Kammern zu verhindern. Entfernen Sie Luftblasen mit einer Mikropipettenspitze.
- Ändern Sie den Kulturmedien (innerhalb und unterhalb des Einsatzes) jeden zweiten Tag und Kultur für etwa 5-7 Tage. Achten Sie darauf, in der Sie das Medium aus dem Einsatz. Um zu vermeiden, contact mit dem Fibroblasten-Kollagenmatrix, leicht kippen Sie den Einsatz mit einem sauberen Pinzette und saugen Sie den Datenträger aus den Einsatzwänden.
Entscheidenden Schritt: Die Fibroblasten in der Kollagenmatrix sollte eine längliche Phänotyp zu bekommen, und renovieren das Kollagen, die dann Aufträge. In etwa 5-7 Tagen hat sich die Matrix zusammengezogen, um eine Plattform (10-14 mm im Durchmesser) in der Mitte des Einsatzes zu bilden. Die vertraglich Matrix ist bereit für den Einsatz in der nächsten Schritt. Um eine gleichmäßige Kontraktion der Matrix ist es entscheidend, zu erhalten, dass die Fibroblasten sind gut mit dem Kollagen vor der Aussaat (Schritt 2.6.1) gemischt.
4. Setzen von Immunzellen (Infizierte / Nicht infizierte Monozyten-Makrophagen-Gemisch)
Hinweis: Die folgenden experimentellen Schritte beinhalten virulenten Mykobakterien und müssen daher in einer BSL-3-Anlage durchgeführt werden.
- Herstellung von primären Monozyten und Makrophagen:
- Isolieren Monozyten im peripheren Blut von Spenderblut mit Hilfe eines establiSchuppen-Protokoll. Isolieren Monozyten am selben Tag wie die Einrichtung des Gewebemodells und Kultur und differenzieren sich zu Makrophagen etwa 7 Tage vor der Infektion mit M. Tuberkulose.
HINWEIS: Dadurch wird sichergestellt, sowohl die Makrophagen und die vertraglich Fibroblasten-Kollagenmatrix sind nach 7 Tagen zur Verfügung. Auch isolieren frischen Monocyten, die zusammen mit den infizierten Makrophagen aufgenommen wird.
- Isolieren Monozyten im peripheren Blut von Spenderblut mit Hilfe eines establiSchuppen-Protokoll. Isolieren Monozyten am selben Tag wie die Einrichtung des Gewebemodells und Kultur und differenzieren sich zu Makrophagen etwa 7 Tage vor der Infektion mit M. Tuberkulose.
- Herstellung von M. Tuberkulose infizierten Makrophagen
- Ernten Sie die kultivierten Bakterien, wäscht mit 1x PBS mit 0,05% Tween-80, resuspendieren in antibiotikafreiem DMEM komplette, durchlaufen einen abgeschnittenen sterile 27 G-Nadel, um bakterielle Klumpen zu zerstreuen und messen die optische Dichte.
Hinweis: Pre-Bestimmung der M. tuberculosis koloniebildende Einheit Mittel der optischen Dichte im Labor. Dies gibt eine Schätzung der Anzahl der Bakterien, für Infektion verwendet werden. - Die Makrophagen Inkubieren für 4 Stunden mit M. Tuberkulose beim Multiplizität der Infektion (MOI) 10). Nach der Infektion waschen 3x mit 1x PBS, um die extrazelluläre Bakterien zu entfernen. Verwenden uninfizierten Makrophagen in der gleichen Weise, aber ohne M. kultiviert Tuberkulose, als Kontrolle.
- Trennen der mit Makrophagen von der Kulturplatte durch Behandlung mit 2 mM EDTA für 10 Minuten bei 37 ° C resuspendiert und die Zellen in antibiotikafreiem komplettem DMEM.
- Ernten Sie die kultivierten Bakterien, wäscht mit 1x PBS mit 0,05% Tween-80, resuspendieren in antibiotikafreiem DMEM komplette, durchlaufen einen abgeschnittenen sterile 27 G-Nadel, um bakterielle Klumpen zu zerstreuen und messen die optische Dichte.
- Kennzeichnung von Monozyten
Hinweis: Isolierung und Markierung von Monozyten in der BSL-2 Bank durchgeführt werden und dann in BSL-3 zur weiteren Verarbeitung entnommen.- Fleck frisch hergestellten Monozyten (2 x 10 7 Zellen) mit einer Endkonzentration von 2 uM PKH26-Farbstoff für 5 min, nach den Anweisungen des Herstellers. Waschen 3x und Resuspendieren der Zellen, die mit antibiotikafreiem komplettem DMEM mit einer Dichte von 1 x 10 7 Zellen / ml.
- Zugabe von Immunzellen Fibroblasten Kollagenmatrix
- EINfter 5-7 Tagen Kultur der Fibroblasten-Kollagenmatrix absaugen Kulturmedien aus den äußeren und inneren Kammern und 1,5 ml frischer antibiotikafreies DMEM-Voll in die äußere Kammer.
- Vorbereitung eines markierten Monozyten-Makrophagen (nichtinfizierten / infizierten) in Mischung mit einem Verhältnis von MO: MQ (5: 1) in 50 & mgr; l DMEM-Voll. Für 50.000 Makrophagen zu nehmen 250000 markierten Monozyten.
- Dann werden 50 ul MO: MQ Mischung auf die Fibroblasten-Kollagen-Matrix und Inkubation für 1 Stunde in 5% CO 2 bei 37 ° C. Nach der Inkubation vorsichtig 2 ml Kulturmedium in den Einsatz und Inkubation für weitere 24 h in 5% CO 2 bei 37 ° C.
Hinweis: Da Zellen gegeben sind lose befestigt ist, sollte die Zugabe von Medien langsam durch vorsichtiges Hinzufügen von an den Wänden des Einsatzes sein.
5. Setzen von Lungenepithelzellen (16HBE)
Hinweis: Die folgenden Schritte müssen in einer Anlage BSL-3 durchgeführt werden.
- Seed Lungen epithelial Zellen (16HBE) auf der Oberseite der MO: MQ-Fibroblasten-Kollagenschicht. Um dies durchzuführen, erste dissoziieren 16HBE Zellen aus dem Kolben durch Behandlung mit Trypsin (wie in 2.2.1.) Und resuspendieren in 4 × 10 6 Zellen / ml in antibiotikafreiem DMEM.
- Saugt den Kulturmedien innerhalb und außerhalb des Einsatzes. Dann fügen Sie 1,5 ml antibiotikafreies DMEM Komplett im Boden der Vertiefung außerhalb des Einsatzes.
- Dann werden 50 ul 16HBE oben auf der Immunzelle-Fibroblasten Kollagenmatrix. Lassen Sie für 2 min in der Haube und Inkubation für 1 h bei 37 ° C Inkubator mit 5% CO 2. Nach Inkubation geben vorsichtig 2 ml antibiotikafreies DMEM-Voll innerhalb des Einsatzes und die Kultur bei 37 ° C für 3 Tage. Die Kulturschritt erleichtert Proliferation von Epithelzellen in der Gewebemodell.
Hinweis: Da Zellen gegeben sind lose befestigt ist, sollte die Zugabe von Medien langsam und sanft durch Gleiten durch die Wände des Einsatzes sein.
6. Luftexposition des 3D-LungModell
Hinweis: Nach Tag 5 nach Zugabe von infizierten Makrophagen sind die Gewebemodelle Luft ausgesetzten und die folgenden Schritte müssen in einer Anlage BSL-3 durchgeführt werden.
- Saugt den Kulturmedien innerhalb und außerhalb des Einsatzes.
Anmerkung: In diesem Schritt können die Überstände zum Nachweis von sezernierten Faktoren gesammelt werden. Zentrifuge, sterile Filter und speichern Überstände bei -70ºC. - Hinzuzufügen 1,8 ml antibiotikafreies DMEM-Voll in der äußeren Kammer und inkubiere in 5% CO 2 bei 37 ° C Inkubator für 2 Tage. Haben Kulturmedien hinzuzufügen nicht innerhalb des Einsatzes.
HINWEIS: Die Luft Anheben des Gewebemodells erleichtert die Bildung von geschichteten Epithelien und Schleimsekretion, die die Festigkeit des Gewebes und physiologische Ähnlichkeit mit humanem Lungengewebe liefert.
7. Ernte und Montage des 3D Lungengewebemodell
Hinweis: Die folgenden Schritte müssen in einer Anlage BSL-3 durchgeführt werden.
- Am Tag 7 nach der Implantation von infizierten Makrophagen sind die Gewebemodelle bereit für die Ernte. Entfernen Sie die Kulturmedien vollständig aus dem Gewebemodell. Befestigen Sie die Gewebemodell mit 4% Paraformaldehyd für 30 Minuten im Dunkeln bei RT. Dieser Schritt tötet die Bakterien und fixiert das Gewebe / Zellmorphologie zur weiteren Verarbeitung.
- Mit einem Skalpell, trennen Sie die Membran aus dem Brunnen Einsatz. Übertragen Sie die Membran in das Gewebe zu einer gut aus 1x PBS mit.
- Schneiden Sie und entfernen Sie die Seiten des Gewebemodell mit einem sauberen Skalpell. Dann schneiden Sie die Gewebemodell in 4 ungefähr gleich große quadratische Stücke. Bringen Sie ein Stück Gewebe auf einen Glasobjektträger Superfrost. Die Gewebestücke in 1x PBS bei 4 ° C.
- Trocknen Sie das Gewebe 5 min und montieren mit ProLong Gold-Antifade mit DAPI und Deckglas. Lassen Sie die Folien, ohne in der Dunkelheit zu stören bei RT bis trocken.
Hinweis: Die Dicke des Gewebes zwischen dem Zentrum und Peripherie zu variieren, was zu leichten Tilting des Deckglases. Um dies zu vermeiden, kann ein Abstandshalter (zum Beispiel Parafilm) auf der Ecke des Deckglases gebracht wird. - Bewerben Nagellack auf die Ränder des Deckglases und trocknen lassen. Tauchen Sie die Objektträger in 70% Ethanol sie sicher aus dem BSL-3-Anlage zu bringen, um zu machen.
8. Visualisierung, Erfassung und Quantitative 3D Analyse
- Visualisieren die Gewebeschnitte mit einem konfokalen Mikroskopsystem mit Lasern bei 488 nm für die Anregung von GFP (grün-Kanal), 420 nm für DAPI (blau) und 555 nm für die PKH26-markierten Monozyten (rot) auf.
- Erwerben 3D-Bilder mit einer Auflösung von 512x512 mit Z-Stapel abdeckt bei einem Minimum von 20 um Dicke und mit 1 bis 1,5 & mgr; m Abstand zwischen den Stapeln. Erwerben 5-10 verschiedenen Bereichen, die den gesamten Gewebestück.
Hinweis: Verwenden Sie die Konfiguration wie Nyqvist zur optimalen Einstellung der optischen Auflösung (Wellenlänge, Laserleistung / Belichtung, Pixelgröße und Zoom). Vermeiden Sie unDer oder Übersättigung von Pixeln. - Analysieren Sie die konfokale Bilder mit 3D-Bildverarbeitungs-Software. Für 3D-Quantifizierung von Zellclustern, werden die folgenden Schritte für eine optimale Analyse empfohlen.
- Öffnen Sie die 3D-Bildverarbeitungs-Software, und laden Sie das Bild. Messung der Dimensionen von Objekten in dem Bild untersucht werden, beispielsweise die Größe eines Nukleus, individuelle Monozyten- und einzelne Bakterien. Diese Beobachtungen sind nützlich für die Definition oder Filterung der Objekte.
- Wenn ein Wiedergabeanpassungstool optimieren Volume Rendering indem jeder Kanal für Bildkontrast, Helligkeit und Opazität zu mischen. Dieser Schritt ist, die Interferenz von Rauschen in Volume Rendering minimieren.
Anmerkung: Die Gammakorrektur kann die Manipulation von Bildern führen und sollten daher vermieden werden. - Erstellen Sie Flächen, indem Sie den roten Kanal (Monozyten) und stellen Sie die Schwelle (automatische oder manuelle Auswahl). Falls erforderlich, verwenden Sie Filter, um die Auswahl von roten Monozyten zu beschränken oder die b auszuschließenintergrund. Ebenso erstellen Flächen für grün (M. tuberculosis) und Blau (Kerne) Kanäle, wie oben beschrieben.
- Exportieren Sie die Daten in MS-Excel-Dateien. Es ist möglich, einen bestimmten Parameter oder alle Daten zu exportieren. Die Parameter, die für die Analyse von Zellclusterbildung relevant sind, sind Volumen, Intensität, Anzahl der Objekte, Anzahl der Voxel und Kugelgestalt.
- Speichert und exportiert die Bilder in einem geeigneten Bildformat TIFF bevorzugt.
HINWEIS: Animationen können auch mit dem Animationsmenü vorgenommen und als Medien-Datei gespeichert werden. - Speichern Sie die Analyse-Einstellungen der einzelnen Kanäle mit der Option Parameter hinzufügen und können später mit der Rebuild-Funktion abgerufen werden. Gleichzeitig analysiert mehrere Dateien mit Hilfe der Batch-Verarbeitung-Tool.
Anmerkung: Alle zu vergleichenden Bilder erworben werden müssen, verarbeitet und in der gleichen Weise analysiert werden. Beispielsweise ein 8 Bit-Bild (256 integer) sollte nicht mit einem 12-Bit-Bild (4096 integer) verglichen werden.
Ergebnisse
Ein 3D-Lungengewebe Modell für menschliche TB effektiv genutzt, um die Wirt-Pathogen-Interaktionen in M. zu studieren Tuberkulose-Infektion. Die grundlegenden Schritte dieses Verfahrens werden repräsentative mikroskopische Bilder verschiedener Stufen und einer Gesamt mikroskopische Struktur einer Gewebeschnitt in 1 dargestellt. Das Modell besteht aus mehreren Komponenten von humanem Lungengewebe, einschließlich Lungenfibroblasten, bronchiale Epithelzellen und primäre Monozyten / Makrophagen in der 3D-Gewebeumgebung eingebettet. Neben der Einbeziehung Komponenten der menschlichen Lungengewebe, das Modell ähnelt physiologischen Bedingungen zwar Schichtung der Epithelien und Schleimabsonderung.
Ein Beispiel für die Verwendung des Lungengewebes Modell bei der Überwachung einer TB-Infektion ist in Abbildung 2 dargestellt. Zur Visualisierung der M. Tuberkulose-Immun-Zell-Migration und Interaktion führten wir Makrophagen, die mit M. infiziert Tuberkulose, die GFP exprimieren(grün) zusammen mit den frisch isolierten PKH26-markierten Monozyten (rot) in das Gewebe-Modell (blau, befleckt DAPI für Kerne). Am Tag 7 nach der Zugabe von M. Tuberkulose infizierten Zellen an den Gewebemodell, zeigt konfokale Mikroskopie Clustering von roten Monozyten an den Ort der Infektion (grün) (Abbildung 2), die das Markenzeichen Läsionen des menschlichen TB 9 nachahmt.
Eine Reihe von repräsentativen Bildern für die 3D-Visualisierung von M. Tuberkulose infizierten Gewebemodell und Quantifizierung von Zellclustern ist in Abbildung 3 dargestellt. Die 3D-Visualisierung gibt dem Benutzer die Flexibilität, zu interagieren, zu untersuchen und zu quantifizieren, mehrere Funktionen in einem 3D-Bild. Die räumliche Anordnung der grünen und roten Bakterien Monozyten Cluster von der apikalen, Dreh- und Seitenansicht gesehen, wie in 3B, die Clusterbildung von Monozyten an der Stelle zeigt, M. illustriert Tuberkulose. Die Cluster nicht OBbei nicht infizierten Geweben (3A) serviert. Quantifizierten wir die Größe und Anzahl der Monozyten Zellcluster und festgestellt, dass die Größe (Volumen) von Zellclustern verbessert (p <0,001), während die Anzahl der Einzel Monozyten verringert (p <0,01) in M. Tuberkulose infizierten Geweben im Vergleich zu nicht infizierten Gewebemodelle (3C und 3D). Diese Daten bestätigt unsere frühere Feststellung der frühen Granulombildung in M. im Lungengewebe-Modelle in 2D Gewebeschnitte 9 analysiert Tuberkulose-Infektion beobachtet.
Unsere Daten weisen darauf hin, dass die Gewebemodell bietet eine natürliche 3D-Lebensraum zu untersuchen, die komplexe Host-Zell-M. tuberculosis Kommunikationsnetz. Wir fanden auch, dass 3D-Visualisierung und quantitativen Analyse sind bessere Werkzeuge für die Untersuchung der Funktionen im Gewebe-Modell (Abbildung 3). Die Quantifizierung der Zellcluster (Granulom zum Beispiel) oft stretc hes zu mehreren Zellschichten und kann vollständig durch eine quantitative 3D-Analyse erfasst werden. Außerdem Visualisierung der genauen räumlichen und zeitlichen Eigenschaften einzelner Zellen oder Bakterien im Modell ermöglichen Live-Bildgebung, Migration und Tracking-Studien in einem autorisierten Labor.
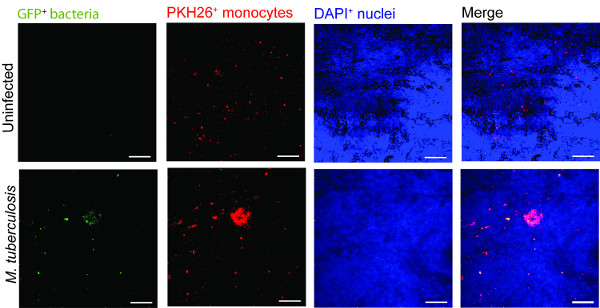
Abbildung 2. Monozyten im Gewebemodell Cluster rund virulenten M. Tuberkulose. Repräsentative konfokale Bilder von nicht infizierten und M. tuberculosis infiziert Gewebemodell wird vorgestellt. Panels von grün (M. tuberkulose- GFP), rot (PKH26-markierten Monozyten), blau (DAPI-gefärbten Zellkernen) und fusionierte Kanäle zeigen die Rekrutierung von Monozyten im infizierten Gewebe im Vergleich zu nicht infizierten Gewebe. Scale - 100 um.large.jpg "style =" font-size: 14px; line-height: 28px; "target =" _ blank "> Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
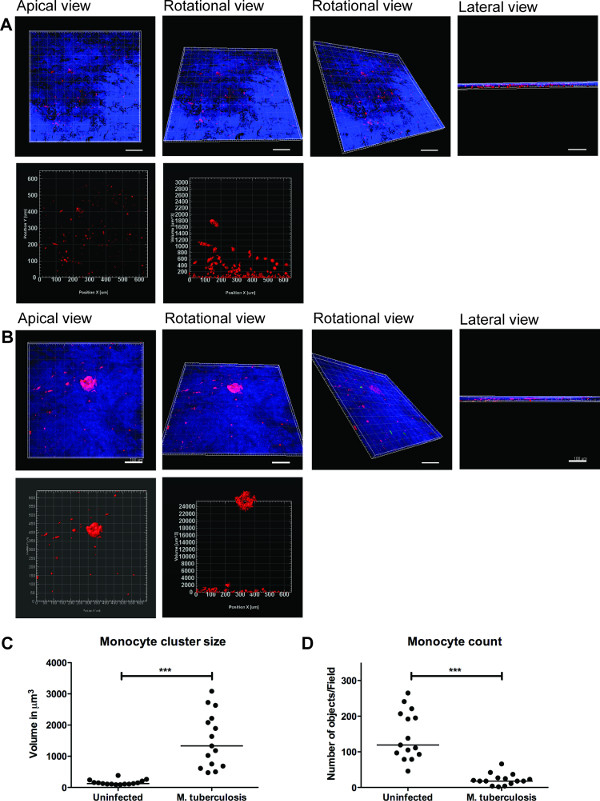
Abbildung 3. 3D-Visualisierung und quantitativen Analyse von Gewebemodell nützliche Informationen. Repräsentative Bilder der 3D-Visualisierung des gesamten Gewebemodell (A) nicht infizierten Gewebe, (B) mit M. infiziert Tuberkulose, durch optische Schnitte mit Zeiss LSM700 konfokalen Mikroskop und quantitative Analyse von Imaris Bildverarbeitungs-Software (Version 7.6.8). Diese Bilder wurden bei 20-facher Vergrößerung erworben, 14 Z-Stapel bedeckt eine Gewebedicke von 19,5 & mgr; m mit 1,5 um Abstand, so dass die Visualisierung von apikal, Rotations horizontale Dreh vertikale und seitliche Ansicht (A und B). (C) QuaGröße ntitative Analyse der Monozyten-Zellcluster offenbaren verbesserte (p <0,0001) der frühen Granulom Clustern nach M. Tuberkulose-Infektion im Vergleich zur Abwesenheit von Infektion. (D) Quantifizierung der Zahl der Monozyten zeigten eine Abnahme (p <0,01) im infizierten Gewebe im Vergleich zu nicht infizierten Gewebe wiederholte mehrere Cluster im infizierten Gewebe. Green - M. Tuberkulose -GFP, Rot - PKH26-markierten Monozyten, Blue - Zellkernen, Scale -. 100 & mgr; m Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
Diskussion
The ability to recruit and form organized cell clusters at the site of infection is the hallmark of human TB 11. These dynamic structures known as tubercle granulomas primarily consist of immune cells (macrophages, monocytes, T-cells and B-cells) and multi-nucleated giant cells surrounding M. tuberculosis. The role of the granuloma has long been considered to wall off the infection, preventing local spread of bacteria. However, more recent studies show that granuloma formation is critical for early bacterial survival, growth and dissemination 12. A strategy of new studies is to identify molecules or pathways that could efficiently be targeted to inhibit the cellular migration in granuloma formation and/or TB dissemination.
A caveat for novel studies on TB is the lack of models that recapitulate human TB. The most widely used experimental animals do not form true granuloma upon M. tuberculosis infection, and are therefore not appropriate choices for studies of TB 13-16. Non-human primates have the closest resemblance to human TB 17, but are not the preferred choice owing to high operational costs and ethical issues. Human TB is a complex immunological process and is difficult to model in vitro. Cell cultures of monolayers or co-cultures lack the 3D environment and tissue responses. Therefore, we have developed an innovative lung tissue model based on human primary immune cells and human lung-specific cell lines 8,9. The model displays characteristic features of human lung tissue, including epithelia with evenly integrated macrophages, formation of extracellular matrix, stratified epithelia and mucus secretion 9.
The 3D human lung tissue model has several benefits over the in vitro single or co-cultures seeded on tissue culture plates or transwell inserts. First, the human lung-specific cells (fibroblasts and epithelial cells) are not commonly included in the in vitro single or co-cultures. Second, the immune cells and lung-specific cells are embedded in a 3D physiological context (collagen rich extra-cellular matrix products). The response of cells to a stimulus/infection and the migratory behaviour of cells, for instance formation of a granuloma, differ significantly between a 2D and 3D environment. Furthermore, the described method enables the 3D visualization and robust 3D quantitative analysis that provides pivotal information on spatial distribution and intricate cellular interactions.
Experimental infection in the model tissue with M. tuberculosis resulted in clustering of macrophages at the site of infection, reminiscent of early TB granuloma (Figure 2 and 3). We have recently demonstrated that mutant strains defective in the ability to secrete the virulence factor ESAT-6 or Mycobacterium bovis BCG that lacks ESAT-6 did not induce the clustering of monocytes (no early granuloma), in contrast to the virulent M. tuberculosis 9. These data are consistent with the observations made from Mycobacterium marinum-infected zebrafish embryos, whose transparency allows for elegant live imaging of granuloma formation 12. As there is no gold-standard model for TB, we took advantage of the surgically resected tissue biopsies from TB patients for validation of the method 9. Our in vitro tissue model shares several characteristics with the lung and lymph node biopsies from TB patients, including the aggregation of macrophages in granuloma, the presence of both intra- and extracellular bacteria 18 and induction of necrosis 11.
Although the described model has physiological relevance to human TB and has several advantages over other in vitro models, it has some limitations. For instance, out of more than 20 collagen proteins identified in humans, only type I is included to the model to mimic the extra-cellular matrix. However, type I collagen is a complex mixture of extra-cellular matrix products and is the most abundant collagen in the human body. Further, we have demonstrated the presence of collagen IV and several extra-cellular matrix proteins such as tropoelastin, vimentin and laminin, which are produced by the epithelial cells and fibroblasts in the tissue model, indicating the synthesis of new collagen 8. Presently, the lung tissue model only has monocytes and macrophages, besides lung-specific cells. It lacks neutrophils and lymphocytes that are also known to be present in the granuloma. Remarkably the model is not limited to the introduction of additional immune cells and is of interest to explore how they contribute to the complex cellular interactions in human TB. Implantation of primary alveolar macrophages, skin-specific cells and lung carcinoma cells has already been tested in the model. Since our objective was to use a model that closely resembles human TB, introduction of mouse cells have not been attempted.
In summary, the lung tissue model has implications for both basic mechanistic and applied studies. Potential applications of the lung model include the study of innate immunity, investigating mechanistic aspects of host defences such as phagosomal maturation, autophagy, production of cytokines, chemokines and anti-microbial peptides, and functional characterization of individual cell types. Strikingly, the in vitro tissue model allows manipulation of one or more cells types and provides a relevant tissue micro-environment, not only for studies on TB, but for a variety of infectious and non-infectious diseases that affect the lungs.
Offenlegungen
The authors declare no competing financial interests.
Danksagungen
The authors acknowledge the Microscopy core facility at the Faculty of Health Sciences, Linköping University for providing access to advanced imaging systems; Karl-Eric Magnusson (Emeritus Scientist) at the Dept. of Clinical and Experimental Medicine, Linköping University for providing access to Imaris 3D/4D image processing software (Bitplane, Switzerland); and S. Braian for his help with the lung model cartoon. This work was supported by funds from the Swedish Research Council (Alternatives to animal research, 2012-1951) and Swedish Research Council (2012-3349) to M.L. and Swedish Foundation for Strategic Research to S.B. S.B. receive grants from the Karolinska Institutet, Swedish Research Council, the Swedish International Development Cooperation Agency (Sida) and the Swedish Civil Contingencies Agency (MSB), and the Swedish Heart and Lung Foundation (HLF). M.S. received grants from the Karolinska Institutet and Stockholm County Council.
Materialien
| Name | Company | Catalog Number | Comments |
| Cell culture inserts | BD Falcon | 353092 | |
| 6-well culture plates | BD Falcon | 353046 | |
| MRC-5 cells, lung fibroblasts | ATCC#CCL-171 | ||
| 16HBE cells, lung epithelial cells | Gift from Dr. Dieter Gruenert, Mt. Zion Cancer Center, University of California, San Fransisco, USA | ||
| 5 x Dulbecco’s modified Eagle’s medium (5 x DMEM) | Gibco | 12800-082 | Made from powder but add 5 times less water. Adjust pH to 7.3 and filter it using a 0.2 µm filter. |
| Dulbecco’s modified Eagle’s medium with glucose (DMEM) 1x | Gibco | 41965-039 | |
| Minimum Essential Medium (MEM) 1x with Earle’s salts | Sigma | M4655 | |
| Non-Essential Amino Acids Solution, 100x | Life Technologies | 11140-035 | |
| L-glutamine 200 mM (100x) | Gibco | 25030-024 | |
| Sodium Pyruvate | Life Technologies | 11360-039 | |
| NaHCO3 (71.2 mg/ml) | Prepared in house | ||
| Heat inactivated Fetal Bovine Serum (FBS) | Gibco | 10270-106 | Heat inactivated for 30 min, 56 °C |
| Gentamicin (50 mg/ml) | Gibco | 15750-060 | |
| Hepes buffer solution 1M | Gibco | 15630-056 | |
| Penicillin Streptomycin (Pen Strep) | Gibco | 15140-122 | |
| Lymphoprep | Axis-Shield | 7801 | |
| Ultrapure 0.5 M EDTA | Gibco | 15575 | |
| Bovine Collagen PA treated (500 ml) | Organogenesis | 200-055 | |
| Pure col purified Bovine Collagen solution (100 ml) | Advanced biomatrix | 5005-B | |
| Extracellular matrix protein, Fibronectin (1 mg) | BD | 354008 | |
| Primary human monocytes/macrophages | Isolated from human whole blood or buffy coats. | ||
| PKH26 Red fluorescent cell linker | Sigma | MINI26 | |
| Mycobacterium tuberculosis H37Rv expressing green fluorescent protein | M. tuberculosis H37Rv wild type was transformed with the pFPV2 plasmid constitutively expressing GFP. | ||
| Middlebrook 7H9 medium | Difco | 271310 | |
| BBL Middlebrook ADC Enrichment | BBL | 211887 | |
| Tween-80 | |||
| Glycerol | |||
| Kanamycin B sulfate (20 µg/ml) | Sigma | B5264 | |
| Prolong Gold anti=-fade reagent with DAPI | Invitrogen | P36935 | |
| Trypsin -EDTA | |||
| Bovine serum albumin | |||
| Paraformaldehyde | |||
| DAPI | |||
| LSM700 Confocal microscope | Zeiss | ||
| iMaris Scientific 3D/4D image processing software, version 7.6.8 | Bitplane AG |
Referenzen
- Sakamoto, K. The pathology of Mycobacterium tuberculosis infection. Veterinary pathology. 49, 423-439 (2012).
- Saunders, B. M., Britton, W. J. Life and death in the granuloma: immunopathology of tuberculosis. Immunol Cell Biol. 85, 103-111 (2007).
- Kaufmann, S. H. New issues in tuberculosis. Ann Rheum Dis. Ann Rheum Dis. 63, ii50-ii56 (2004).
- Morrison, J., Pai, M., Hopewell, P. C. Tuberculosis and latent tuberculosis infection in close contacts of people with pulmonary tuberculosis in low-income and middle-income countries: a systematic review and meta-analysis. Lancet Infect Dis. 8, 359-368 (2008).
- Barry 3rd, C. E., et al. The spectrum of latent tuberculosis: rethinking the biology and intervention strategies. Nat Rev Microbiol. 7, 845-855 (2009).
- Puissegur, M. P., et al. An in vitro dual model of mycobacterial granulomas to investigate the molecular interactions between mycobacteria and human host cells. Cell Microbiol. 6, 423-433 (2004).
- Kapoor, N., et al. Human Granuloma In Vitro Model, for TB Dormancy and Resuscitation. PLoS One. 8, e53657 (2013).
- Nguyen Hoang, ., T, A., et al. Dendritic cell functional properties in a three-dimensional tissue model of human lung mucosa. Am J Physiol Lung Cell Mol Physiol. 302, L226-L237 (2012).
- Parasa, V. R., et al. Modeling Mycobacterium tuberculosis early granuloma formation in experimental human lung tissue. Dis Model Mech. 7, 281-288 (2014).
- Cozens, A. L., et al. CFTR expression and chloride secretion in polarized immortal human bronchial epithelial cells. Am J Respir Cell Mol Biol. 10, 38-47 (1994).
- Brighenti, S., Andersson, J. Local immune responses in human tuberculosis: learning from the site of infection. J Infect Dis. 205, S316-S324 (2012).
- Davis, J. M., Ramakrishnan, L. The role of the granuloma in expansion and dissemination of early tuberculous infection. Cell. 136, 37-49 (2009).
- Gupta, U. D., Katoch, V. M. Animal models of tuberculosis. Tuberculosis (Edinb). 85, 277-293 (2005).
- Kashino, S. S., Napolitano, D. R., Skobe, Z., Campos-Neto, A. Guinea pig model of Mycobacterium tuberculosis latent/dormant infection. Microbes Infect. 10, 1469-1476 (2008).
- Singhal, A., et al. Experimental tuberculosis in the Wistar rat: a model for protective immunity and control of infection. PLoS One. 6, e18632 (2011).
- Subbian, S., et al. Phosphodiesterase-4 inhibition alters gene expression and improves isoniazid-mediated clearance of Mycobacterium tuberculosis in rabbit lungs. PLoS Pathog. 7, e1002262 (2011).
- Lin, P. L., et al. Tumor necrosis factor neutralization results in disseminated disease in acute and latent Mycobacterium tuberculosis infection with normal granuloma structure in a cynomolgus macaque model. Arthritis Rheum. 62, 340-350 (2010).
- Rahman, S., et al. Compartmentalization of immune responses in human tuberculosis: few CD8+ effector T cells but elevated levels of FoxP3+ regulatory t cells in the granulomatous lesions. Am J Pathol. 174, 2211-2224 (2009).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten