Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Automatische Quantifizierung und Analyse von Zellzählung Schritte bei der Verwendung ImageJ Plugins
In diesem Artikel
Zusammenfassung
This paper describes the quantification of hemocytometer and migration/invasion micrographs through two new open-source ImageJ plugins Cell Concentration Calculator and migration assay Counter. Furthermore, it describes image acquisition and calibration protocols as well as discusses in detail the input requirements of the plugins.
Zusammenfassung
Das National Institute of Health ImageJ ist ein leistungsfähiges, frei verfügbaren Bildverarbeitungs-Software-Suite. ImageJ hat umfassende Partikelanalyse-Algorithmen, die effektiv genutzt werden kann, verschiedene biologische Partikel zu zählen. Wenn eine große Anzahl von Zellproben zu zählen, stellt der Zählkammer einen Engpass in Bezug auf die Zeit. Ebenso Membranen von Migration / Invasion Assays mit dem ImageJ Plug-Zellzähler, obwohl eine genaue Zählung, ist außerordentlich arbeitsintensiv, subjektiv und berüchtigt für verursachen Schmerzen am Handgelenk. Um diesem Bedarf zu begegnen, haben wir zwei Plugins innerhalb ImageJ für die einzige Aufgabe der automatisierten Hämozytometer (oder bekannten Volumen) und Migration / invasion Zellzählung. Beide Plugins verlassen sich auf die Fähigkeit, qualitativ hochwertige Mikroaufnahmen mit minimalen Hintergrund zu erfassen. Sie sind einfach für den schnellen Zählen und Analyse großer Probengrößen mit integrierten Analysetools zu verwenden und optimierte Kalibrierung von Zählungen zu helfen. Durch die Kombination der Kernprinzips von Zellzähler mit einem automatisierten Zählalgorithmus und post-counting Analyse Dies erhöht die Leichtigkeit, mit der Migrationsassays kann ohne Verlust an Genauigkeit bearbeitet werden.
Einleitung
In - vitro - Zellzählung ist eine wichtige Grundtechnik in einem weiten Bereich von Gewebekulturexperimenten. Exakt die Anzahl der Zellen in einer Kultur , die Bestimmung ist von wesentlicher Bedeutung für die experimentelle Reproduzierbarkeit und Standardisierung 1,2. Zellzählung kann ein Hämozytometer manuell über als auch unter Verwendung einer Vielzahl von automatisierten Verfahren, jede mit ihren eigenen Vorteilen und Nachteilen 3,4,5 ausgeführt werden. Die meisten der automatisierten Verfahren zur Zellzählung gehören zu einer von zwei Klassen, diejenigen, die das Coulter-Prinzip verwenden oder Durchflusszytometrie. Coulter Zähler nutzen Sie Zellen elektrische Widerstand der Zellzahl und Größe zu bestimmen. Sie sind schnell, präzise und kostengünstiger als Durchflusszytometer. Jedoch werden sie selten für nur Zellzählung aufgrund ihrer beträchtlichen Kosten im Vergleich zu manuellen Zählung 3 verwendet. Durchflusszytometer, auf der anderen Seite, sind teuer, aber sie haben viele Anwendungen, wie beispielsweise Zellzählung, Analyse der Zellen Form, structure und Mess interne Zellmarker 4,5. Maschinen, die eine dieser beiden Prinzipien verwenden, sind von vielen Herstellern erhältlich. Manuelle Zählen ist erschwinglich , aber zeitaufwendig und unterliegt Bias , während die automatisierten Verfahren mit einem Bruchteil der Zeit kommen für die manuelle Zählung erforderlich , aber mit teuren Maschinen 6.
Andere häufige Zellkulturverfahren sind in - vitro - Zellbeweglichkeit Assays, nämlich die Zellmigration und Invasion 7. Migration und Invasion Assays werden üblicherweise zur Untersuchung der Zellbeweglichkeit und Invasivität in Antwort auf eine chemotaktische Reaktion verwendet. Darüber hinaus sind sie häufig zu studieren Embryonalentwicklung, Differenzierung, Entzündungsreaktion, und die Metastasierung von mehreren Zelltypen 7-11 verwendet. Zellen, die durch die poröse Membran aus einem Migrationstest migriert oder eingedrungen kann auf zwei verschiedene Weisen quantifiziert werden. Erstens, indem die Zellen mit einem fluoreszierenden Farbstoff Anfärben Dissoziation from die Membran und Quantifizierung eines Fluoreszenzleser 12. Eine Einschränkung dieser Methode der Quantifizierung ist , dass keine Aufzeichnung der Membranen zurückgehalten werden kann , und es besteht keine Möglichkeit zur weiteren Analyse 13. Die zweite Quantifizierungsverfahren wird für Zellen migrierten / eingedrungen mit Fluoreszenzfarbstoff oder häufiger fixiert und gefärbt werden, wobei zytologischen Farbstoffe wie Kristallviolett, Toluidinblau oder Hämatoxylin-Farbstoff; dann werden die Zellen manuell mit invertierten Mikroskopbilder dieser Membranen quantifiziert , die eine sehr zeitraubende Aufgabe ist 12,13.
Um die Nachteile der manuellen Zellzählung, zwei zuverlässigen und genauen automatischen Zellzähler für Zellkonzentration und für den Migrationstest überwunden wurden entwickelt. Diese automatisierten Zellzähler Algorithmen wurden für ImageJ als Plugin unter Verwendung von Oracle Java-Computer entwickelt. ImageJ ist ein öffentlicher und weit verbreiteten Bildbearbeitungswerkzeug durch das National Institute of Hea entwickeltlth (NIH) 14,15; Somit erleichtert diese Plugins für ImageJ Schreiben eine einfache Integration in die biologische Gemeinschaft.
Automatisierung der Zellzählung sorgt für einen hohen Durchsatz und Reproduzierbarkeit im Vergleich zu manuellen Zählung. Obwohl andere verfügbare Software und Plugins verwendet werden kann , die Zellkonzentration durch Bildanalyse 5,16,17, Zellkonzentration Calculator Plugin ist schnell und kann auch behandeln Verdünnungen von Zellen und Behandlungen zu berechnen. Darüber hinaus können alle Ergebnisse und Berechnungen aus diesen beiden Zähler gespeichert und exportiert werden. Die beiden Plugins in diesem Dokument beschrieben sind, für die Verwendung eines Phasenkontrastmikroskop für Live Cell Imaging und großes Sichtfeld (gesamte Membran capture) Bildgebung für Migration Assay-Membranen durch die Verwendung eines Binokular optimiert. Die Plugins sind frei zum Download zur Verfügung mit Installationsanweisungen aus: http://peng.lab.yorku.ca/imagej-plugins.
Protokoll
1. Verbindung Mikroskop und Kamera-Setup (Zellkonzentration Calculator)
- Erhöhen Glühbirne Helligkeit voll mit dem Licht Einstellknopf, wechseln Sie auf die 4X Objektivlinse und gewährleisten Phasenkontrastfilter ausgewählt werden.
HINWEIS: Jede invertierte Phasenkontrastmikroskop für die Gewebekultur mit einem dunklen Hintergrund, zum Beispiel PhP Phasenkontrast, können folgende Standard - Mikroskop und Kamera Verfahren verwendet werden. - Innerhalb der Software des Mikroskops eingestellt Bildaufnahmeeinstellungen auf die Standardwerte.
HINWEIS: Lesen Sie in der Bedienungsanleitung des Mikroskops die Lage dieser Einstellungen zu finden.- Set 'Helligkeit "," Kontrast "und" Sättigung "auf 100% und" Gamma "und" Gain "bis 1,0.
HINWEIS: Je nach Software, "Helligkeit" und "Kontrast" auf einen Wert von 0% Standard anstelle von 100%. - Set Bilder in schwarz und weiß mit der höchsten Auflösung availabl erfasst werdene (1.600 x 1.200 Pixel (px) oder höher).
HINWEIS: A 'Sättigung' von 0% reicht aus, wenn ein Schwarz-Weiß-Einstellung nicht verfügbar ist.
- Set 'Helligkeit "," Kontrast "und" Sättigung "auf 100% und" Gamma "und" Gain "bis 1,0.
- Legen Sie eine Standard Hemocytometer auf den Mikroskoptisch und erfassen ein Bild wie in (Abbildung 1A) dargestellt; dies ist das "Volume Kalibrierung Bild '. Stellen Sie Belichtungszeit je nach Bedarf.
2. Bildvolumenkalibrierung
- Offene ImageJ und aus dem Plugins - Menü, starten Sie die Zellkonzentration Rechner (CCC) Plugin, zB Plugins> Analyzer> 'Zellkonzentration Calculator ".
- Wenn das Panel 'Bild Volume Kalibrierung "der rechten Seite nicht sichtbar ist, klicken Sie auf" Kalibrieren ", es zu zeigen.
- In ImageJ, öffnen Sie die "Volume Kalibrierung Bild 'aus Schritt 1.3 (' Datei '>' Open ') und CCC, klicken Sie auf die Schaltfläche" Bild Dimension Get'.
HINWEIS: Diese sowohl in Bild füllt Breite undHöhe Textfelder mit der Bildauflösung in Pixeln automatisch. - In ImageJ, wählen Sie die nächste "Straight Line Tool 'auf die Auswahl - Werkzeuge und eine gerade Linie über die gesamte Länge des Hemocytometer primären (P) zeichnen -square in (Abbildung 1B) gezeigt , durch Klicken und Ziehen Sie den Cursor.
- Drücken Sie die Taste "M" das Ergebnisfenster mit den geraden Linie Messungen angezeigt werden soll. Geben Sie den Wert aus der Spalte Länge in die "P-Quadrat Length 'Textbox in CCC (Abbildung 1B).
- Klicken Sie auf "Berechnen Bild Volume 'Taste zur Ausgabe des Bildvolumen in das Bildvolumen Textbox. Alternativ, wenn das Volumen des Bildes bereits bekannt ist, geben Sie die Lautstärke in nL in das Bildvolumen Textbox.
- Klicken Sie auf die Schaltfläche "Speichern".
HINWEIS: Das Plugin ist nun kalibriert.
3. Kamera Belichtung Kalibrierung
- Nach der Zellernte fürZählen über Hemocytometer, Last 10 & mgr; l Zellen in eine Kammer des Hämozytometer und legen Sie es auf den Mikroskoptisch.
- Mit den gleichen Einstellungen von Schritt 1.2, stellen Sie die Belichtungszeit, so dass die Hintergrundlinien des Hemocytometer verschwinden.
- Stellen Sie den Fokus, so dass das Innere der Zellen dunkler als der Zellmembran ist, was anzeigt Fokus innerhalb des zentralen Querschnitts der Zelle und nicht die Pole.
- Genauer einstellen , die Belichtung , so dass die Zellen nicht überbelichtet sind , und diejenigen , dargestellt in (1C) ähneln.
HINWEIS: Ein wenig sichtbar Hemocytometer Linien akzeptabel sind. Es wird empfohlen, diese Einstellungen zu speichern oder aufzeichnen Genauigkeit und Reproduzierbarkeit zu erhalten.
4. Image Acquisition
- Für jede Zellprobe, Last 10 & mgr; l in beiden Kammern des Hemocytometer statistische Inferenz Leistung 2 zu erhöhen. Legen Sie die Hemocytometer auf dem Mikroskoptischfür die Bildgebung.
HINWEIS: Die Auflösung und Vergrößerung jedes Bild muss die gleiche wie die "Volume Kalibrierung Bild" sein. Das Plugin zählt alle Bilder von jedem ausgewählten Ordner; halten Bilder im selben Ordner zusammengezählt werden.- Wenn ein Dateiname Autoinkrement Funktion verfügbar ist, schalten Sie ihn ein und stellen Sie sicher, dass jedes Bild nicht nach der Aufnahme den Durchsatz zu erhöhen gezeigt.
HINWEIS: Manuelles Speichern und jedes Bild Schließen wird der Prozess drastisch verlangsamen. Lesen Sie in der Bedienungsanleitung des Mikroskops für Informationen über die Verfügbarkeit eines Autoinkrement-Funktion. - Erfassen mindestens drei nicht überlappenden Bilder des zentralen Bereichs des Hämozytometer obwohl mehr (5-10) wird empfohlen, die Genauigkeit zu erhöhen.
HINWEIS: Vermeiden sowohl die oberen und unteren Bereiche der Kammern als Zellen an beiden Stellen in der Dichte zu erhöhen, neigen. Nehmen Sie die gleiche Anzahl von Bildern für jede Kammer. Dies wird bei der Zählung für die ordnungsgemäße Funktion des Plugins erforderlich.
- Wenn ein Dateiname Autoinkrement Funktion verfügbar ist, schalten Sie ihn ein und stellen Sie sicher, dass jedes Bild nicht nach der Aufnahme den Durchsatz zu erhöhen gezeigt.
5. Bild Zählen und Verdünnungen
- In CCC, klicken Sie auf "Zellen Count" und beobachten Sie das Verzeichnis Wählen Sie im Dialogfeld. Wählen Sie einen Ordner gezählt werden.
- Beachten Sie die Probennummer Eingabefeld nach Auswahl eines Ordners. Geben Sie die Anzahl der Bilder pro Kammer genommen, das heißt, 4.1.2, und klicken Sie auf "OK". Das Plugin wird zählen nun alle JPG, TIFF und PNG-Bilder im ausgewählten Ordner in alphabetischer Reihenfolge.
Hinweis: Mit einem Klick auf die Schaltfläche "Sample-Viewer", wird der Sample Viewer-Fenster die Anzeige von Informationen über die gezählten Proben bringen. Probenkonzentration ist die durchschnittliche Konzentration aller pro Kammer aufgenommenen Bilder. Proben mit einheitslose Konzentrationen können zu dieser Liste, wie die Zugabe eines Arzneimittels oder kleines Molekül Behandlung zugesetzt werden.- Neuauszählung der Zellproben, wenn die Konzentrationen deutlich innerhalb zählt der gleichen Proben (Abschnitt 4) variieren.
HINWEIS: Um Verdünnungen für alle berechnen ter zählte-Proben, CCC verwendet automatisch die Formel C 1 V 1 = C 2 V 2.
- Neuauszählung der Zellproben, wenn die Konzentrationen deutlich innerhalb zählt der gleichen Proben (Abschnitt 4) variieren.
- Verwenden Sie das Szenario der Aussaat 15.000 Zellen pro 200 & mgr; l in 30 Wells einer 96-Well-Platte + 1 Extra:
- Stellen Sie die Textbox rechts von der C2-Etikett auf 15.000 und die Konzentration Volumen Textbox bis 200, die benachbarte Kombinationsfeld Einheit 'ul' zu ändern.
HINWEIS: Das Plugin wird letztlich die Konzentration in Zellen / ml berechnen. - Stellen Sie sicher, dass das Volumen Kombinationsfeld hat V2 ausgewählt (endgültige Volumen) und in der Textbox rechts eingeben 6200 (200 & mgr; L x (30 + 1)), "ul" in der Volumeneinheit Kombinationsfeld auswählen.
- Stellen Sie die Textbox rechts von der C2-Etikett auf 15.000 und die Konzentration Volumen Textbox bis 200, die benachbarte Kombinationsfeld Einheit 'ul' zu ändern.
- Klicken Sie auf "berechnen Verwässerung" der aktuell eingegebenen Verdünnung auf die linke untere Listenfeld hinzuzufügen.
HINWEIS: Für jede Verdünnung hinzugefügt wird für jede Probe und angezeigt in dem Baumdiagramm nach rechts gelöst werden. Klicken Sie doppelt auf jeden Eintrag zu erweitern. - Durch Klicken auf ter Schaltfläche "Speichern" unter dem "Sample-Viewer", um zu schreiben, alle Beispieldaten und Verdünnungen einzureichen.
- Hinweis: Diese Daten können die gespeicherte Datei, indem Sie auf "Load" und die Auswahl jederzeit wiederhergestellt werden.
6. Migration und Invasion (Counter)
- Führen Sie die Migration und Invasion Assays , die die Standard - Boyden - Kammer - Methode 7-9 verwendet wird .
- Nachdem die Zellen gewandert sind / eingedrungen, entfernen Sie sorgfältig die Medien innerhalb des Einsatzes durch Umdrehen und leichtes Antippen. Docht entfernt jegliche überschüssige Medien an der Unterseite der Membran verklebt, indem der Rand mit einem Papiertuch zu berühren.
HINWEIS: Berühren Sie nicht die Membran selbst auf das Handtuch, das adhärierten Zellen lösen kann. - Fix und färben die Zellen wie berichtet 7-9 in einer 24-Well - Platte mit jeder Zeile mit ~ 500 ul einer anderen Lösung einrichten, zum Beispiel Fixativ, Stain 1, Fleck 2 und doppelt destilliertem Wasser (ddH 2 O). Füllen Sie eine zweite Platte mit 1x PBS to Stellen Sie die Einsätze in vor dem Schneiden.
- Waschen Sie die Einsätze von ihnen in die Vertiefungen gefüllt mit ddH 2 O und füllen Sie den Einsatz mit ddH 2 O. Lassen Sie das Wasser vor dem Abtupfen entfernt Zellen durch Umdrehen platzieren.
- Verwenden Sie einen sauberen Baumwollapplikator un-migrierten / un-Invasion Zellen von der Oberseite der Membran die Pflege zu entfernen die Membran nicht beschädigt wird. Sein gründliches um die Ränder der Membran.
- Schneiden Sie die Membran mit einer Rasierklinge oder einem Skalpell und sorgfältig übertragen die Membran (Unterseite nach oben) auf einen sauberen Glasobjektträger.
- Fügen Sie einen kleinen Tropfen Befestigungslösung unter und auf der Oberseite der Membran und die Abdeckung mit einer dünnen Deckglas.
HINWEIS: Vermeiden Sie Blasen innerhalb der Befestigungslösung Trapping Genauigkeit der Zählungen zu erhalten.
7. Binokular und Kamera-Setup
- Schalten Sie die Lichtquelle des Mikroskops und der Kamera.
HINWEIS: Lesen Sie in der Bedienungsanleitung für detaillierte Anweisungen des Mikroskops. - WitzHin der Software die Bildaufnahmeeinstellungen des Mikroskops auf die Standardwerte.
HINWEIS: Wenn eine Mittelungsfunktion vorhanden ist, wird ein Wert von vier empfohlen. Dies ist ein guter Kompromiss zwischen dem Grad der Mittelung und Bildaufnahmezeit. Ebenso kann ein kleiner Grad der Schärfbildtreue erhöhen.- Set 'Helligkeit "," Kontrast "und" Sättigung "auf 100% und" Gamma "und" Gain "bis 1,0.
HINWEIS: Je nach Software, "Helligkeit" und "Kontrast" auf einen Wert von 0% Standard anstelle von 100%. - Set-Display (Echtzeit) und Capture Auflösung bis zu ihrer maximalen Einstellungen (1.600 x 1.200 Pixel und 2.592 x 1.944 Pixel, respectively).
HINWEIS: Display-Auflösung kann je nach Bedarf angepasst werden, wenn die Refresh-Rate zu langsam ist. Niedrigere Auflösungen werden es schwieriger machen, genau zu konzentrieren, sondern die Aktualisierungsrate zu erhöhen.
- Set 'Helligkeit "," Kontrast "und" Sättigung "auf 100% und" Gamma "und" Gain "bis 1,0.
- Für die Phase des Binokular, verwenden Sie einen festen weißen background; eine schwarze oder Glas Hintergrund ist unzureichend.
- Verwenden Sie die oben Bühne Lichtquelle, vorzugsweise aus zwei flexiblen LED-Leuchten auf der rechten Seite und von der Bühne gelassen.
HINWEIS: Eine unten stufigen Lichtquelle werden die Poren innerhalb der Migrationstest Membran beleuchten, die sich negativ auf die Genauigkeit der Folgezählungen beeinflussen können. - Legen Sie eine abgeschlossene Migration Assaymembran Schieber auf die Bühne. Mit Blick auf das Bild in Echtzeit durch die Software angezeigt, stellen Sie die Vergrößerung mit dem Knopf Zoom-Einstellung, so dass die Kanten einer einzigen Membran nur innerhalb der Kamera Sichtfeld sind.
HINWEIS: Platzieren Sie die Folie auf einer Glasplatte overtop der weiße Hintergrund der Bühne macht es leichter, die Folie für die Bildgebung zu manövrieren. - Stellen Sie die Lichtquellenpositionen (7.6.1) und Belichtungszeiten so eng wie möglich um das ideale Bild in 2A gezeigt zu reproduzieren. In Abhängigkeit von der Helligkeit, Belichtungszeiten von 5-60 ms ausreichen.
HINWEIS:Das Ziel ist , ein Bild mit so wenig Hintergrundfarbe wie möglich und eine gleichmäßig beleuchtete Membran ohne Reduktionsbildtreue, das heißt, eine Überbelichtung , die zu einem Verlust von sichtbaren Zellen zu erzeugen.- Positionieren Sie die linke und rechte Lichtquellen in einem niedrigen Winkel relativ zum Schlitten. Dies wird helfen, Hintergrundflecken und Farbfehler entfernen. Versuchen Sie, jede Lichtquelle direkt gegenüber dem anderen zu halten.
HINWEIS: Während jede Lichtquelle in Position manövriert, drehen Sie den anderen ab. Dies hilft, sich auf den Bereich der Beleuchtung auf die Membran zu zentrieren einfacher und genauer.
- Positionieren Sie die linke und rechte Lichtquellen in einem niedrigen Winkel relativ zum Schlitten. Dies wird helfen, Hintergrundflecken und Farbfehler entfernen. Versuchen Sie, jede Lichtquelle direkt gegenüber dem anderen zu halten.
- Entfernen Sie die Folie und Weißabgleich das Bild einen einzigen Knopfdruck in das Mikroskop-Software.
HINWEIS: Das Mikroskop ist nun kalibriert. Speichern Sie so viele Einstellungen wie möglich für die zukünftige Verwendung und beachten Sie die Lichtquellenpositionen und Intensität.
8. Bildaufnahme und Flatfield
- Innerhalb der Software dieder Capture-Ordner und Capture ein Bild pro Membran. Nennen Sie die Bilder nach dem allgemeinen Vorlage von: Name - ###, zB Steuerung - 001.tif, Kontrolle - 002.tif, Test Droge - 001.tif, und so weiter.
HINWEIS: Für die beste Bildergebnisse, die Bilddateityp als TIFF über andere verlustbehaftete Formate wie JPEG speichern.
HINWEIS: Die Bilder im Anschluss an die allgemeine Vorlage nicht noch gezählt werden, aber nicht auf automatisierte Gruppierung und Flatfield unterliegen.- Wenn Flatfield-Korrektur gewünscht wird, für jede Folie in einen leeren Bereich zu finden und ein leeres Bild im Anschluss an die Namenskonvention nehmen: Name - Blank.
HINWEIS: Eine leere ein Bereich auf der Folie ist, die keine Membran enthält und stellt die Hintergrundbeleuchtung. - Unmittelbar vor der Bildgebung, glätten jede Membran durch Druck über die Deck Anwendung und so viele gefangen Blasen wie möglich zu entfernen.
- Wenn Flatfield-Korrektur gewünscht wird, für jede Folie in einen leeren Bereich zu finden und ein leeres Bild im Anschluss an die Namenskonvention nehmen: Name - Blank.
- In ImageJ, gehen die TC-Plugin, um Plugins und öffnen; zB Plugins> Analysieren> & #39; Transwell-Counter '.
- Innerhalb TC, klicken Sie auf die Schaltfläche 'Flatfield' und das Dialogfeld Verzeichnis auswählen beobachten. Wählen Sie den Ordner, in dem die Membran Bilder gespeichert wurden.
HINWEIS: Es werden nur Bilder mit der allgemeinen Vorlage gespeichert oben wird automatisch korrigiert und in einem neuen Ordner namens Flatfield im ausgewählten Ordner gespeichert Flatfield werden. Siehe 2B ein Beispiel eines Flatfield korrigiertes Bild.
9. Konfigurationseinstellungen
- Öffnen Sie eine Migration Assaymembran Bild in ImageJ ( 'Datei'> 'Open') und wählen Bild> Anpassen> 'Farbschwellwert ...'. Beachten Sie die Farbschwelle Fenster einstellen, welche Farben werden aus dem Bild herausgefiltert werden.
- Am unteren Rand des Fensters gesetzt Thresholding Methode 'Shanbhag', Threshold Farbe "Weiß" und Farbraum auf "RGB" (rot, grün, blau); Deaktivieren Sie die Option Dunkler Hintergrund, wenn ausgewählt.
- Stellen Sie die obereSchieber durch Klicken und die Markierungen auf 0 und die unteren Schieberegler, um 255 ziehen (die unteren Schieber bei 255 verlassen). Stellen Sie sicher, dass das Bild vollständig weiß ist.
- Stellen Sie die grünen und roten oberen Schieber, bis nur noch die Kerne sichtbar sind.
HINWEIS: Die Einstellungen werden verwendet, vollständig auf die Zelle Fleck ausgewählt variieren. Siehe Abbildung 2C. - Im TC-Plugin, geben Sie die Werte der RGB oberen Schieber in die "RGB Threshold" Konfigurationseinstellungen zugehörigen Textfelder. Klicken Sie auf die "Hinzufügen / Ändern" klicken und die Konfiguration überschreiben.
- Lassen Sie die Größenbereich Untere und Obere Werte bei 1 - Infinity.
- Im Panel-Konfigurationseinstellungen, klicken Sie auf "Speichern", um die Einstellungen auf die Festplatte zu schreiben.
10. Zählen Bilder und Kalibrierung
- Öffnen Sie das TC-Plugin (8.2) und klicken Sie auf 'Count Ordner' und beobachten Sie das Verzeichnis auswählen Dialogfeld. Wählen Sie den Ordner zu zählen einnd warten, bis es zu beenden.
- Jede gezählte-Probe wird auf die Haupttabelle automatisch hinzugefügt. Schlüsselspalten sind 'Count "," Qualität "und" Kalibrieren Sie? ".
HINWEIS: Die nicht-abgeglichenen Count ist die Anzahl der Partikel pro Membran in dem Bereich in der Spalte 'Area Bereich' angezeigt.
HINWEIS: Qualität reicht von etwa -0,8 bis 1,7; Q ≥ 0,5 ist akzeptabel.- Beachten Sie ein Häkchen in das "Kalibrieren? ' Spalte, wenn ein Bild markiert ist der ideale Metriken auf seiner Ähnlichkeit zur Kalibrierung basiert.
HINWEIS: Sowohl Qualität und Kalibrierung könnte darauf hindeuten, dass die aktuellen Einstellungen möglicherweise nicht ausreichen. Wenn nach dem richtigen "Farbe Thresholding" und "Größenbereichen" wurden ausgewählt und Kalibrierung wird noch vorgeschlagen, Inspektion des Originals und gezählt Bilder sollte die endgültige Determinante eines gültigen Zählung sein.
- Beachten Sie ein Häkchen in das "Kalibrieren? ' Spalte, wenn ein Bild markiert ist der ideale Metriken auf seiner Ähnlichkeit zur Kalibrierung basiert.
- Wählen Sie alle Proben in der Tabelle Markiert für die Kalibrierung.
- Rechtsklick in ter Tisch und wählen Recount> 'Empfohlene Größe ". Dies wird erzählen die Bilder mit einem empfohlenen Mindestkornfläche.
- Rechts klicken Sie erneut auf und wählen Sie "Show Plot '. Beachten Sie die Frequenz Streudiagramm der Partikelbereiche. Ein ideales Bild wird eine Kurve , die eine lange rechten tailed - Normalverteilung ähnlich, dh der Glockenkurve. Siehe Abbildung 3B.
- Stellen Sie den unteren Größenbereich, falls erforderlich, durch die Auswahl der Stichprobe, der rechten Maustaste, und wählen Sie Recount> 'Manuelle Einstellungen ". Beachten Sie die manuellen Einstellungen-Dialog. Geben Sie die gewünschten Einstellungen und klicken Sie auf Count mit den neuen Einstellungen auf das Bild erzählen.
- Zum Einstellen der Zählungen manuell wählen Sie die Proben, rechts der Tabelle klicken, und wählen Sie "Bild öffnen mit zählt". Beachten Sie das Originalbild mit roten Markierungen repräsentiert jedes Teilchen durch das Plugin gezählt.
HINWEIS: Recount> 'Show gezählt Binärbild' nützlich sein kann, wie gut zu überprüfen iNDIVIDUALSTIPENDIEN Zellen werden durch die Farbe Schwellwertbildung gelöst.- Um einen Zähler hinzuzufügen, halten Sie "Strg" und klicken Sie links. Beachten Sie einen Marker an der Cursor-Position, die zu den Proben Gesamtzahl hinzugefügt werden. Um eine Markierung zu entfernen, klicken Sie rechts auf das Bild. Das Plugin wird die Markierung am Cursor zu entfernen.
- Um eine Gruppe von Markierungen zu entfernen, ein Auswahlwerkzeug in ImageJ verwenden, um eine Region of Interest (ROI) zu wählen. Digi-Key, rechts klicken Sie auf das Bild Halten und alle Marker innerhalb der ROI werden entfernt. Beachten Sie die Zellzahl in der "Count" Spalte.
11. Speichern / Öffnen Ergebnisse und Exportieren in CSV
- In TC, gehen Sie in der Menüleiste und klicken Sie auf "Datei"> "Ergebnisse speichern". Beachten Sie die Ergebnisse speichern Dialogfeld. Wählen Sie einen Namen und das Ziel und klicken Sie auf "Speichern".
- Verwenden Sie 'Datei'> 'Open Ergebnisse' eine Datei zu laden, die alle die in der Haupttabelle angezeigten Daten, including die Handlung.
HINWEIS: Die Ergebnisdatei speichert die Verzeichnisse die Bilder in gespeichert sind, wenn die Originalbilder bewegt werden nach dem Speichern, die 'Open Originalbild "und" Bild öffnen mit Zählungen' Funktionen fehl.. - Um das Bild Verzeichnis zurückzusetzen, um die Proben auszuwählen, klicken Sie rechts in der Tabelle, und wählen Sie "Reset Bildverzeichnis '. Beachten Sie das Verzeichnis auswählen Dialogfeld. Wählen Sie den neuen Ordner, und wenn die Bilder vorhanden sind, werden die Verzeichnisse zurückgesetzt werden. Re-save die Ergebnisdatei.
- Verwenden Sie 'Datei'> 'Open Ergebnisse' eine Datei zu laden, die alle die in der Haupttabelle angezeigten Daten, including die Handlung.
- So speichern Sie gehen, um eine Comma Separated Values (CSV) Datei, in der Menüleiste auf "Datei"> "Export to CSV". Dies erzeugt eine Datei mit Proben in statistischer Gruppen mit der mittleren organisiert zählen und Standardfehler des Mittelwerts; das Layout ist für eine schnelle Grafik gemeinsam Grafikprogramme.
HINWEIS: Die statistische Gruppen basieren auf den Gruppen innerhalb TC angelegt.- Wenn die Proben der allgemeinen Benennungsvorlage folgen, wählen Sie die Proben gr seinouped rechten Maustaste und wählen Sie "Auto-Gruppierung". Dies fügt Proben mit dem gleichen 'Name' zu der gleichen Gruppe. Am Beispiel Steuerung - 1, Kontrolle - 2, Behandlung - 1, Behandlung - 2: beide Kontrollen der Gruppe "Control" und Behandlungen "Behandlung" hinzugefügt würde.
- In Proben manuell Gruppen durch einen Doppelklick auf das die Probe 'Gruppe' Zelle und der Eingabe in die Gruppennamen. Wählen Sie die Proben zu dieser Gruppe hinzugefügt werden soll, klicken Sie rechts und wählen Sie "Zur Gruppe hinzufügen".
Ergebnisse
Zellkonzentration Rechner
Abbildung 1 zeigt den Gesamtprozess der CCC - Kalibrierung und zählbar Bildaufnahme. 1A und 1B zeigen die P-Quadrat Kalibrierungsbild und Berechnung von P-Quadrat Länge in Pixeln. CCC bestimmt Zellkonzentration in einem bestimmten Volumen nach der Formel:
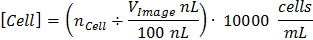
Diskussion
Kritische Schritte, Fehlerbehebung und Einschränkungen
Das Wesen der automatisierten Rechenverfahren, insbesondere die der Partikelanalyse erfordert die mathematische Fähigkeit, diese Partikel zu definieren. Folglich ist die Genauigkeit der beiden Zellkonzentrationsrechner und Migrationsassay Zähler majorly abhängig von Bildwiedergabetreue, das heißt, wie genau das übernommene Bild das Membranzellprobe oder Migrationstest ähnelt. Es ist daher von größter Bedeutung Mikroskop und zugehö...
Offenlegungen
The authors declare that they have no competing financial interests.
Danksagungen
This work was supported by the Canadian Institute of Health Research to CP (OR 142730 and OR 89931). We would like to thank Jelena Brkic for her initial idea of binary particle analysis in ImageJ.
Materialien
| Name | Company | Catalog Number | Comments |
| HyClone Classical Liquid Media: RPMI 1640 - With L-Glutamine | Fisher Scientific | SH3002702 | Cell culturing media |
| Fetal bovian serum (FBS) | GIBCO BRL | P00015 | Media suppliment |
| HTR8/SVneo trophoblast cell line | Cells were obtained from Dr. Charles Graham (Queen’s University, Kingston, Canada) | Software is designed to work with any cell line. | |
| Trypsin | GIBCO BRL | 27250-018 | Prepared as 0.20% (w/v) in 10 µM EDTA 1x PBS |
| Accutase | Innovative Cell Technologies | AT104 | |
| 10 cm cell culture plates | SARSTEDT | 833902 | Any tissue culture treated plates will be suitable |
| Transwell Polyester Membrane Inserts - 8.0 µm Pore size | Costar 3422 ordered from Fisher Scientific | 7200150 | For 24-well plates; Pore size: 8.0 µm; 6.5 mm diameter; 0.33 cm2 growth area |
| HARLECO Hematology Stains and Reagents, EMD Millipore - Soluntions 1, 2 & 3 | EMD Millipore and ordered from VWR | 65044A, B, & C | Hemacolor stain set consists of three 500 ml (16.9 oz.) poly bottles & includes a methanol fixative (Solution 1), an eosin or acid stain (Solution 2), and a methylene blue or basic stain (Solution 3) |
| Cotton Tipped Applicator | Puritan Medical | 806-WC | |
| Single-edge industrial razor blades | VWR | 55411 - 055 | Thickness: 0.30 mm (0.012") |
| Microscope Slides - Precleaned/Plain | Fisher Scientific | 12550A3 | Dimentions: 25 mm x 75 mm x 1.0 mm |
| Fisherbrand Cover Glasses - Rectangles no. 1 | Fisher Scientific | 12-545E | Thickness: 0.13 to 0.17 mm; Size: 50 mm x 22 mm |
| Fisher Chemical Permount Mounting Medium | Fisher Scientific | SP15-500 | |
| Leica Stereo dissecting microscope | Leica Microsystems | The microsope is equipped with Leica microscope camera Model MC170 HD & camera software is Leica App. Suite (LAS E2) Version 3.1.1 [Build: 490]. Microscope parts: LED3000 Spot Light Illumination Model: MEB126, Leica M80 Optic Carrier Model M80, Objective achromat 1.0X, WD=90 mm Model: MOB306 & Objective achromat 0.32X, WD=303 mm Model: MOB315, Video Objective 0.5X Model: MTU-293 | |
| Hemacytometer | Assistant Germany | 0.100 mm Depth - 0.0025 mm2 | |
| Olympus inverted light microscope | Olympus Corporation | CKX41SF | The microsope is equipped with Lumenera Infinity 1-2 2.0 Megapixel CMOS Color Camera & camera software is Infinity analyze Version 6.5.2 |
| Laminar flow cabinet 1300 Series A2 | Thermo Scientific | Model: 1375 | Any laminar flow cabinet for cell culture work will be suitable |
| Cell culture incubator | Thermo Scientific | Model: 370 | Any cell culture incubator will be suitable - Cells were cultured under humidefied environment, 5% CO2, 37 °C |
| ImageJ | NIH | Version 1.50e | Minimum version required |
| Java Runtime Environment | Oracle | Version 1.8.0_66 | Minimum version required |
Referenzen
- Ricardo, R., Phelan, K. Counting and Determining the Viability of Cultured Cells. J. Vis. Exp. (16), e752 (2008).
- Using a Hemacytometer to Count Cells. Basic Methods in Cellular and Molecular Biology Available from: https://www.jove.com/science-education/5048/using-a-hemacytometer-to-count-cells (2016)
- Graham, M. D. The coulter principle: foundation of an industry. J. Lab. Autom. 8 (6), 72-81 (2003).
- Ormerod, M. G., Imrie, P. R., Walker, J. M., Pollard, J. W. Flow cytometry. Animal Cell Culture. , 543-558 (1990).
- Eliceiri, K. W., et al. Biological Imaging Software Tools. Nat. Methods. 9 (7), 697-710 (2013).
- Louis, K. S., Siegel, A. C., Stoddart, J. M. Cell viability analysis using Trypan blue: manual and automated methods. Mammalian Cell Viability: Methods and Protocols. , 7-12 (2011).
- Scott, W. N., Langdon, S. P. Invasion and motility assays. Cancer Cell Culture: Methods and Protocols. , 225-229 (2004).
- Justus, C. R., Leffler, N., Ruiz-Echevarria, M., Yang, L. V. In vitro Cell Migration and Invasion Assays. J. Vis. Exp. (88), e51046 (2014).
- Luo, L., et al. MicroRNA-378a-5p promotes trophoblast cell survival, migration and invasion by targeting Nodal. J. Cell Sci. 125, 3124-3132 (2012).
- Adorno, M., et al. A Mutant-p53/Smad Complex Opposes p63 to Empower TGFbeta-Induced Metastasis. Cell. 137, 87-98 (2009).
- Chung, T. K. H., et al. Dysregulation of microRNA-204 mediates migration and invasion of endometrial cancer by regulating FOXC1. Int. J. Cancer. 130, 1036-1045 (2012).
- Kramer, N., et al. et al. In vitro cell migration and invasion assays. Mutat. Res./Rev. Mutat. 752, 10-24 (2013).
- Al-Khazraji, B. K., Medeiros, P. J., Novielli, N. M., Jackson, D. N. An automated cell-counting algorithm for fluorescently-stained cells in migration assays. Biol. Proced. Online. 13, 1-6 (2011).
- Abramoff, M. D., Magalhaes, P. J., Ram, S. J. Image processing with ImageJ. Biophotonics Intern. 11, 36-42 (2004).
- Grishagin, I. V. Automatic cell counting with ImageJ. Anal. Biochem. 473, 63-65 (2015).
- Thomas, C. R., Paul, G. C. Applications of image analysis in cell technology. Curr. Opin. Biotech. 7 (1), 35-45 (1996).
- . Appreciating data: warts, wrinkles and all. Nat. Cell Biol. 8, 203 (2006).
- Rossner, M., Yamada, K. M. What's in a picture? The temptation of image manipulation. J. Cell Biol. 166, 11-15 (2004).
- . Count objects in an image in Adobe Photoshop. Helpx.adobe.com. , (2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten