Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Produktion, Kristallisation und Strukturbestimmung
In diesem Artikel
Zusammenfassung
Proline-proline endopeptidase-1 (PPEP-1) is a secreted metalloprotease and promising drug-target from the human pathogen Clostridium difficile. Here we describe all methods necessary for the production and structure determination of this protein.
Zusammenfassung
New therapies are needed to treat Clostridium difficile infections that are a major threat to human health. The C. difficile metalloprotease PPEP-1 is a target for future development of inhibitors to decrease the virulence of the pathogen. To perform biophysical and structural characterization as well as inhibitor screening, large amounts of pure and active protein will be needed. We have developed a protocol for efficient production and purification of PPEP-1 by the use of E. coli as the expression host yielding sufficient amounts and purity of protein for crystallization and structure determination. Additionally, using microseeding, highly intergrown crystals of PPEP-1 can be grown to well-ordered crystals suitable for X-ray diffraction analysis. The methods could also be used to produce other recombinant proteins and to study the structures of other proteins producing intergrown crystals.
Einleitung
Clostridium difficile ist eine der Hauptursachen für nosokomiale Antibiotika-assoziierter Diarrhoe Infektionen 1. Diese grampositive anaerobe Bakterium wird durch seine Sporenform über die fäkal-oral übertragen. In den letzten zehn Jahren neue '' Epidemie '' oder '' hyper '' Stämme (zB BI / NAP1 / 027) führte zu einer drastischen Zunahme der Neuinfektionen und Todesraten in Nordamerika und Europa 2. C. difficile - assoziierten Erkrankungen (CDAD) ist eine lebensbedrohliche Kolon Entzündung mit einer hohen Sterblichkeitsrate 3. Die Symptome reichen von Durchfall 4 bis Pseudomembrankolik 5 und der oft tödlich verlauf toxisches Megakolon 6.
Die Behandlung von CDAD ist schwierig , da die virulente Stämme multiresistente und die Rate Wiederholung sind hoch 7. Im Moment Therapie umfasst die Antibiotika Metronidazol, Fidaxomicin oder Vancomycin oder in repetitively rezidivierende Fälle fäkale Mikrobiota Transplantation. Neue therapeutische Strategien sind dringend 8 benötigt. Einige Fortschritte als therapeutische monoklonale Antikörper Bezlotoxumab aufgezeichnet zielt, C. difficile Toxin B 9, wurde vor kurzem erfolgreich Phase III der klinischen Studien bestanden und wurde für die Zulassung bei der FDA und EMA eingereicht. Zusätzlich neue Antibiotika in verschiedenen Stadien der klinischen Versuche im Augenblick geprüft 10 werden.
Zur Entwicklung wirksame Behandlung neue therapeutische Targets identifiziert werden müssen. Die vor kurzem entdeckte C. difficile - Protease - Prolin-Prolin - Endopeptidase-1 (PPEP-1; CD2830 / Zmp1; EC 3.4.24.89) ist so ein vielversprechendes Ziel, wie der Mangel an PPEP-1 in einem Knock-out - Stamm nimmt die Virulenz von C . difficile in vivo 11. PPEP-1 ist ein sekretiertes Metalloprotease 12,13 Abspalten zwei C. difficile Adhäsine an ihrem C-Terminus 13 somit die Freigabe der anhaftenden bacteria aus dem menschlichen Darmepithel. Daher ist es bei der Aufrechterhaltung des Gleichgewichts zwischen dem sessile und motile Phänotyps von C. difficile beteiligt. Zur Entwicklung selektiver Inhibitoren gegen PPEP-1 und zu verstehen, wie sie ihre Substrate intime Kenntnis der dreidimensionalen Struktur erkennt ist unverzichtbar. Wir haben die erste Kristallstruktur PPEP-1 allein und im Komplex mit einem Peptid - Substrat 14 gelöst. PPEP-1 ist der erste bekannte Protease spaltet Peptidbindungen zwischen zwei Prolinreste 15 selektiv. Es bindet das Substrat in einem zwei geknickte Weise und stabilisiert sie über einen längeren aliphatisch-aromatischen Netzwerk von Resten in der S-Schleife angeordnet, die die Protease - aktiven Stelle 14 erstreckt. Dieses Substrat-Bindungsmodus ist einzigartig für PPEP-1 und nicht in der menschlichen Proteasen bisher gefunden. Dies macht es ein vielversprechendes Medikament Ziel, und Off-Target-Effekte von Inhibitoren sehr unwahrscheinlich.
Zur Entwicklung und Bildschirm selektive PPEP-1-INHibitors in der Zukunft eine große Menge an reinem und monodisperse PPEP-1-Protein benötigt. Weiterhin ist die Art der Bindung der ersten Inhibitoren, Co-Kristallstrukturen mit PPEP-1 zu bestimmen, wird bestimmt werden müssen. In unseren Händen PPEP-1 produziert ständig verwachsene Kristalle. So haben wir ein Optimierungsverfahren zur Herstellung von einzelnen Beugungs Qualität Kristalle von PPEP-1 entwickelt. In diesem Protokoll beschreiben wir ausführlich in der Produktion, Reinigung, Kristallisation und Strukturlösung von PPEP-1 14. Wir verwenden die intrazelluläre Expression in Escherichia coli einer PPEP-1 - Variante die Sekretions - Signalsequenz, Affinitätschromatographie und Grßenausschlußchromatographie unter Entfernung des Reinigungsmarkierung fehlt, gefolgt von 16 microseeding in einem Optimierungs Bildschirm und Strukturbestimmung mittels Zink Einwellenlängen-anomale Dispersion (Zink-SAD) 17. Dieses Protokoll kann für die Produktion und die Strukturbestimmung von anderen Proteinen (zB angepasst werden </ Em> Metalloproteasen), insbesondere für Proteine verwachsene Kristalle zu erzeugen. Auf Wunsch Plasmid-DNA des Konstruktes (pET28a-NHis-rPPEP-1) und Beugungsdaten können für Bildungszwecke zur Verfügung gestellt werden.
Protokoll
1. Klonierung und Konstruieren Entwurf
- Klonen Sie die Codon-optimierte Sequenz (für E. coli) von C. difficile PPEP-1 ohne das Signalpeptid [Aminosäuren 27-220, nachstehend genannten rekombinanten PPEP-1 (rPPEP-1) 11] in den pET28a Vektor mit NdeI und Xho I - Restriktionsstellen (Figur 1) mit einem Stopp - Codon am 3'-Ende (resultierende Vektor pET28a-NHis-rPPEP-1). Dies erzeugt eine N-terminal 6xHis-markierte Protein (NHis-rPPEP-1) mit einer Website Thrombinschnittstelle so dass der Tag während der Reinigung zu entfernen (Abbildung 1). Das Plasmid enthält ein Kanamycin-Resistenzkassette zur Auswahl. Die Primer für die Klonierung verwendet werden an anderer Stelle beschrieben 14.
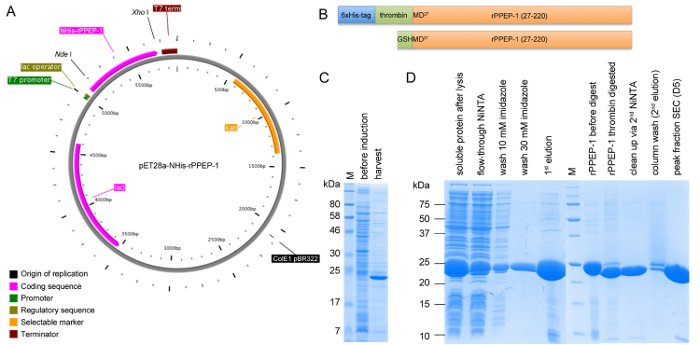
Abbildung 1: Schematische Darstellung der Konstruktion pET28a-NHis-rPPEP-1 und SDS-PAGE - Analyse von expression und alle Reinigungsschritte. (A) Vektorkarte von NHis-rPPEP-1 kloniert in pET28a Vektor mit NdeI / XhoI mit PlasMapper erstellt. (B) Schematische Darstellung des NHis-rPPEP-1 - Konstrukt (oberes Feld) und das endgültige Konstrukt nach Thrombin-Spaltung des 6xHis-Tag mit der daraus resultierenden zusätzlichen GSHM-Sequenz am N-Terminus (unteres Bild). SDS-PAGE - Analyse (C) des Ausdrucks in BL21 (DE3) Star bei 37 ° C für 4 Stunden und (D) von Proben aus allen Reinigungsschritten (M: Molekulargewichtsmarker). Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
2. Expression und Reinigung von rPPEP-1
- Die Expression von NHis-rPPEP-1
- Make-up und Autoklaven LB (LB-Medium) Medium (10 g / l Trypton, 5 g / l Hefeextrakt, 10 g / l NaCl, adjust 7,5 mit NaOH auf pH-Wert). Supplement mit Kanamycin-Sulfat (50 ug / ml) unmittelbar vor der Anwendung (LB / Kan-Medium).
- Impfen ein 200 ml über Nacht Kultur von frisch transformierten E. coli in LB / Kan - Medium. Wachsen über Nacht bei 37 ° C mit bei 220 Umdrehungen pro Minute schütteln.
- Am nächsten Morgen, überprüfen Sie die OD 600 (optische Dichte bei 600 nm Wellenlänge) der über Nacht - Kultur. Mit 1 L LB / Kan - Medium jeweils mit der Nacht - Kultur auf eine OD 600 von 0,1 beimpft und zwei 2,8 - l - Flaschen verwirrt. Nahrungsergänzungsmittel mit drei Tropfen wässrigen Silikonemulsion übermäßige Schaumbildung zu verhindern. Wachsen Zellen bei 37 ° C bei 180 rpm , bis die OD 600 0,6 Schütteln erreicht.
- Nehmen Sie eine Vorinduktion Probe für die SDS-PAGE - Analyse (Äquivalent von 1 ml aus einer Kultur bei einer OD 600 = 1); IPTG bis 0,5 mM Endkonzentration hinzufügen Expression von NHis-rPPEP-1 zu induzieren. Weiterhin bei 37 ° C / 180 rpm für 4 Stunden wachsen.
- Bestimmen Sie die OD 600 in einem 10 - fach dilution und (aus einer Kultur bei OD - Äquivalent von 1 ml 600 = 1) eine Ernte Probe nehmen.
- Sammle die Zellen durch Zentrifugation für 20 min bei 7000 xg und 4 ° C. Rest LB Medium resuspendieren Zellpellets aus 1 l Kultur in 40 ml TBS-Puffer (Tris-gepufferte Kochsalzlösung: 20 mM Tris-HCl, pH 7,5, 200 mM NaCl) zu entfernen und in ein 50 ml Zentrifugenröhrchen. Sammeln Sie die Zellen durch Zentrifugation für 10 min bei 10.000 × g und 4 ° C und bei -80 ° C bis zur Verwendung. Analysieren Ausdruck (Gesamtlysate und löslichen Fraktionen) über SDS-PAGE 18.
- Reinigung von unmarkiert rPPEP-1
- Bis zu 50 & mgr; l Proben jeder Reinigungsschritt für die SDS-PAGE-Analyse. Resuspendieren des Zellpellets aus 1 l Kultur in TBS-Puffer, ergänzt mit 10 & mgr; g / ml DNaseI. Verwenden Sie 5 ml TBS / DNaseI pro g Zellen.
- Lyse der Zellen durch Beschallung auf Eis / Wasser unter Verwendung von 30% Amplitude für 15 min (2 sec Impulse mit 2 sec Pause). Entfernen Sie die Ablagerungen von centrifugation für 10 min bei 10.000 × g und 4 ° C und Überstand in ein Ultrazentrifugenröhrchen. Klare Lysat in einer Ultrazentrifuge 30 Minuten lang bei 165.000 xg und 4 ° C.
- Arbeiten bei 4-6 ° C. Unter Verwendung einer peristaltischen Pumpe oder Chromatographiesystem äquilibrieren 2 ml Nickel-Nitrilotriessigsäure (NiNTA-Harz) in einer Glassäule mit Puffer TBS, supplementiert mit 10 mM Imidazol pH 7,5. Alternativ können Sie die Schwerkraft fließen.
- Stellen Sie das geklärte Lysat mit 1 M Imidazol, pH 7,5, auf eine Endkonzentration von 10 mM. Anzuwenden das Lysat auf die Säule und wasche schrittweise mit Puffer TBS, supplementiert mit 10 mM und 30 mM Imidazol, bzw. bis die UV-Absorption bei 280 nm die Basislinie erreicht hat.
- Eluieren des Proteins mit Puffer TBS plus 250 mM Imidazol. Re-Äquilibrierung der Säule zu TBS mit 10 mM Imidazol ergänzt und über Nacht speichern.
- Bestimmen Sie die Proteinkonzentration entweder bei 280 nm durch das Aussterben koeffi mitzient von 25.900 M -1 cm -1 oder durch jedes andere Verfahren (zB Bradford - Methode 19). Fügen Sie 2 Einheiten Thrombin pro mg Protein und dialysieren der Proteinlösung über Nacht bei 4 ° C gegen einen 50x Volumen von TBS (50x des NiNTA Elutionsvolumen).
HINWEIS: Nehmen Sie die richtigen Rohling zur Bestimmung der Proteinkonzentration, wie Imidazol stark bei 280 nm absorbiert. - Übergeben Sie die Proteinlösung über die äquilibrierte NiNTA Harz ungespaltenen Protein zu entfernen. Als nächstes gilt das gleiche Volumen an TBS mit 10 mM Imidazol zu der Säule alle abgespaltenen Protein wiederzugewinnen ergänzt. Um die Säule zu reinigen, eluieren alle restlichen Protein mit 250 mM Imidazol. Analysieren Sie die Proben über SDS-PAGE (Abbildung 1).
- Konzentriere die Proteinlösung zu 4 ml in 10 min-Intervallen bei 4000 xg und 4 ° C eine zentrifugale Ultrafiltrationseinheit verwendet wird. Mischen Sie die Konzentration Protein nach jedem Intervall Ausfällung und Aggregation zu verhindern. Bei diesem Schritt gelegentlicherly einige Fällung für rPPEP-1 trotz des Mischvorgangs beobachtet.
- Übernehmen der konzentrierten Protein auf eine voräquilibriert Grßenausschluß-Chromatographie-Säule (in TBS-Puffer) bei 4-6 ° C. Eluieren der Säule mit TBS-Puffer, sammeln 1-ml-Fraktionen und je 5 ul jeder zweiten Fraktion auf SDS-PAGE-Analyse. rPPEP-1 eluierte in einem einzigen Peak , entsprechend einem Monomer (Abbildung 2). Gelegentlich wird ein kleinerer Peak bei höherem Molekulargewicht beobachtet (Front peak), das auf ein Dimer des Proteins entspricht. Die Ausbeute sollte etwa 50 mg reines Protein pro Liter Kultur. Analyse aller Proben über SDS-PAGE 18 (Abbildung 2).
- Bis zu 50 & mgr; l Proben jeder Reinigungsschritt für die SDS-PAGE-Analyse. Resuspendieren des Zellpellets aus 1 l Kultur in TBS-Puffer, ergänzt mit 10 & mgr; g / ml DNaseI. Verwenden Sie 5 ml TBS / DNaseI pro g Zellen.
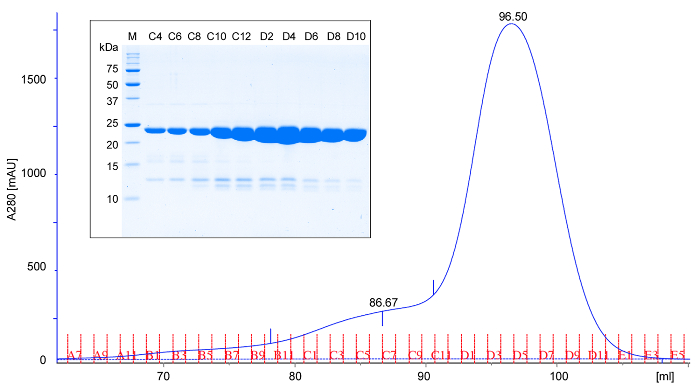
Abbildung 2: Representative Grßenausschlußchromatographie und SDS-PAGE - Analyse von rPPEP-1. Größenausschlusschromatogramm (A280; Extinktion bei 280 nm) von gereinigtem unmarkierte rPPEP-1 A (16/600) Säule in Tris-HCl, pH 7,5, 200 mM NaCl bei 6 ° C verwendet wird. Bezogen auf das Elutionsvolumen als rPPEP-1 wandert für ein 22 kDa-Protein erwartet, was nahe legt, dass es vorwiegend monomer ist. Selten ein kleiner fronting Spitze scheint, dass zu einem Dimer entspricht. (Kleines Bild) SDS-PAGE-Analyse der Fraktionen von Grßenausschlußchromatographie (M; Molekulargewichtsmarker). Jede zweite Fraktion wird angewendet. Die schwachen Banden unter dem Haupt rPPEP-1-Band entsprechen gelegentlich geringfügige Verunreinigungen auftreten. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
3. Kristallisation und Kristall Optimierung Microseeding Verwendung
HINWEIS: rPPEP-1 kristallisiert aus Bedingungen , die ständig produzieren sehr verwachsene Kristalle nicht geeignet für die Röntgenbeugungsanalyse (Abbildung 3). Daher wurde eine Optimierungsstrategie (Abbildung 4) entwickelt , um Kristalle hoher Qualität (Abbildung 5) zu erhalten.
- Das anfängliche Screening von rPPEP-1 unter Verwendung von kommerziellen Bildschirme
HINWEIS: Führen Sie die Kristallisationsversuche in der Sitzung Drop-Format mit im Handel erhältlichen Standard-Bildschirme und einen Kristallisationsroboter.- Konzentriere das gereinigte Protein zu 12 mg / ml einer zentrifugalen Ultrafiltrationsvorrichtung in Intervallen von 5 Minuten bei 4.000 × g und 4 ° C verwendet wird. Mischen Sie die Konzentration Protein nach jedem Intervall Ausfällung und Aggregation zu verhindern. Bestimmung der Proteinkonzentration entweder bei 280 nm unter Verwendung des Extinktionskoeffizienten von 25.900 M -1 cm -1 oder durch jedes andere Verfahren (zB Bradford - Methode). Äquilibrieren das Protein auf 20 ° C. Wegzuräumen alle Partikel und Staub durch Zentrifugation für 10 min bei 16.000 xg und 20 ° C.
- Verwenden Sie entweder bereits vorgefüllten Kristallisationsplatten sealed und bei 4 ° C gelagert oder die Reservoir-Wells der Platten mit 70 & mgr; l jeder Kristallisationsbedingung zu füllen. Äquilibrieren alle Kristallisationsplatten auf 20 ° C. Arbeiten Sie schnell, da die kleinen Volumina schnell austrocknen. Verwenden Sie eine Feuchtigkeitskammer rund um das Dock des Roboters, wenn möglich.
HINWEIS: Verwenden Sie die folgenden Bildschirme als Standardverfahren: SaltRx, Index, PEG / Ion, Kristall, Wizard, PACT ++, JCSG ++. - Stellen Sie den Bildschirm durch Pipettieren von Protein und Reservoir in subwells 2-4. Tropfenvolumen von 300 nl und die Verhältnisse (Protein: Reservoir) sind 200: 100 (subwell 2), 150: 150 (subwell 3) und 100: 200 (subwell 4) (in nl). Unmittelbar Abdichtung der Platte und in einer Kammer bei 20 ° C.
- Überprüfen Sie Tabletts unmittelbar nach Set-up, und dann in der ersten Woche jeden Tag zu inspizieren, die wöchentliche Überprüfung gefolgt.
- Co-Kristallisation von rPPEP-1 - Liganden mit
- Für Co-Kristallisation von Substratpeptid-rPPEP-1-Komplexe mischen rPPEP-1bei 24 mg / ml in einem Verhältnis 1: 1 (v / v) mit einem 7-fachen molaren Überschuss an Peptidlösung (Ac-EVNPPVPD-NH 2) lyophilisiertes Pulver in TBS - Puffer solubilisiert), die eine Endkonzentration von 12 geben mg / ml r-PPEP-1-Protein und einen 7-fachen molaren Überschuss an Peptid über PPEP-1. Inkubieren für 30 min bei 20 ° C und Löschen aller Partikel und Staub durch Zentrifugation für 10 min bei 16.000 xg und 20 ° C entfernt. Weiter mit der Kristallisation unter Verwendung des microseeding Verfahrens wie für die ungebundene r-PPEP-1-Protein beschrieben.

Abbildung 3: Repräsentative Kristalle von der ersten Bildschirme. Verwachsene Kristalle, die aus rPPEP-1 bei 12 mg / ml in Zustand gezüchtet. (A) Kristall Bildschirm I / 38 (1,4 M Natriumcitrat dreibasisches entwässern, 0,1 M HEPES Natrium pH 7,5; 200 nl: 100 nl). (B) SaltRx Bildschirm / 52 (2,4 M Ammoniumphosphat dibasic, 0,1 M Tris, pH 8,5; 100 nl: 200 nl) und (C) (200 nl: 100 nl). (D) SaltRx Bildschirm / 96 (60% v / v Tacsimate pH 7,0, 0,1 M Bis-Tris - Propan pH 7,0; 200 nl: 100 nl). Der Maßstabsbalken = 0,2 mm. Die Volumenverhältnisse sind immer Protein: Reservoir. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
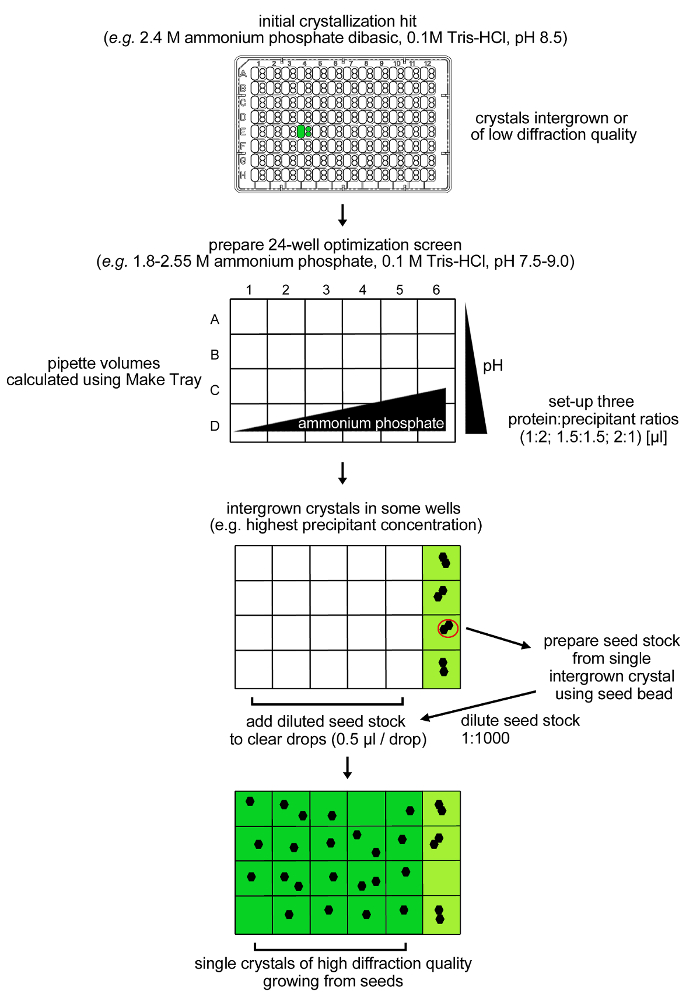
Abbildung 4: Optimierungsverfahren für rPPEP-1 Kristallisation. Erste Kristalle aus rPPEP-1 bei 12 mg / ml niedriger Beugungs Qualität und mit mehreren Gittern (verwachsene) wurden in einem 24-Zustand Optimierung Bildschirm wiedergegeben. enthaltend 2,55 M Ammoniumhydrogenphosphat Wieder nur verwachsene Kristalle wurden in Bedingungen beobachtet. Ein Saatgut wurde aus einem einzigen verwachsenen Kristall hergestellt und verdünnt 1: 1000 in die gleiche conditIon (microseeding). Ein Volumen von 0,5 ul des verdünnten Saatgut wurde in den verbleibenden klaren tropft und Einkristalle wuchsen in fast allen Bedingungen. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
- Kristall Optimierung microseeding mit
HINWEIS: In hohem Grade verwachsene Kristalle von rPPEP-1 erscheinen nach zwei Tagen in einem Zustand , die 2,4 M Ammoniumhydrogenphosphat, 0,1 M Tris-HCl, pH - Wert 8,5 (SaltRx Bildschirm, Zustand E4, alle drei subwells) (Abbildung 3). Ein Optimierungsverfahren ein Gitter Bildschirm um die Anfangsbedingung in Kombination mit microseeding angewendet wurde (Abbildung 4).- Vorbereiten eines Gittersiebes (Abbildung 4), die 24 Bedingungen mit 1,8 bis 2,55 M Ammoniumphosphat dibasisches (in Schritten von 0,15 M) und 0,1 M Tris-HCl pH 7,5-9,0 (in Schritten von 0,5 pH-Einheiten) von entspreaß Stammlösungen (4 M Ammoniumphosphat und 1 M Tris-Puffer).
HINWEIS: Verwenden Sie das Make Tray Applet (http://hamptonresearch.com/make_tray.aspx) Schema der Mengen und Pipettieren zu berechnen, um 2 ml jeder Bedingung, die 10 Optimierung Bildschirme ausführen können. Das 4 M Ammoniumphosphat-Stammlösung ist schwierig herzustellen. Die Lösung wird unter Rühren vollständig um das Pulver in Wasser auflösen. - Je 200 & mgr; l jedes Raster-Bildschirm-Lösung in die Vertiefungen einer 24-Well-Platte und ins Gleichgewicht bei 20 ° C.
- Sie manuell die Kristallisationsplatte einzurichten. Das Tropfenvolumen beträgt 3 & mgr; l und die Verhältnisse (Protein: Reservoir) sind 2: 1, 1,5: 1,5 und 1: 2 (in ul). Hier verwenden, um eine Verdrängerpipette Luftblasenbildung zu vermeiden. Unmittelbar Abdichtung der Platte und in einer Kammer bei 20 ° C. Vermeiden Sie Luftblasenbildung.
HINWEIS: Nach einem bis vier Tagen hoch erscheinen verwachsene Kristalle in den vier Bedingungen enthält, 2,55 M Ammonium phophosphat dibasischen und 0,1 M Tris-HCl pH 7,5 bis 9,0 (Figur 4). Keine Kristalle werden in den verbleibenden 20 Bedingungen mit Ammoniumphosphatkonzentrationen unter 2,55 M. Die microseeding Verfahren gebildet verwendet Einkristalle rPPEP-1 unter diesen Bedingungen erhalten. - Bereiten Sie eine microseed Lager durch einen einzigen verwachsenen Kristall aus einer der beiden Bedingungen mit 2,55 M Ammoniumhydrogenphosphat Ernte und 0,1 M Tris-HCl pH 8,0 oder 8,5. Die Kristalle können auf der Kunststoffoberfläche befestigt sein. Eine sorgfältige Verformung des umgebenden Kunststoff mit einer Akupunktur-Nadel hilft, die Kristalle zu lösen.
- Dann werden 50 ul der jeweiligen Mutterlauge in ein 1,5-ml-Röhrchen eine kleine hochpolierte Glasperlen (Perlen-für-Samen) enthalten. ein bereitgestelltes Nylonschlaufe übertragen den Kristall in 1 ul der Mutterlauge auf eine Glasabdeckung Folie platziert werden.
- Die Flüssigkeit der Kristall in das Rohr und vortex bei hoher Geschwindigkeit für 30 Sekunden enthält. Machen Sie eine 1:1000 Verdünnung des Saatguts in ein neues 1,5-ml-Röhrchen die gleiche frisch zubereiteten Zustand und Vortex gründlich für 5 Sekunden enthält.
HINWEIS: Seeds Bestände können bei -80 ° C für die spätere Verwendung gespeichert werden.
- Entfernen Sie die Dichtung der Platte bedeckt die 20 Bedingungen mit klaren Tropfen und Pipette 0,5 ul des Saatguts (1: 1000 Verdünnung) in die Vertiefungen. Verschließen Sie die Platte und in einer Kammer bei 20 ° C. Einkristalle von hoher Beugungsqualität erscheinen in 2-7 Tagen (Abbildung 5).
- Vorbereiten eines Gittersiebes (Abbildung 4), die 24 Bedingungen mit 1,8 bis 2,55 M Ammoniumphosphat dibasisches (in Schritten von 0,15 M) und 0,1 M Tris-HCl pH 7,5-9,0 (in Schritten von 0,5 pH-Einheiten) von entspreaß Stammlösungen (4 M Ammoniumphosphat und 1 M Tris-Puffer).
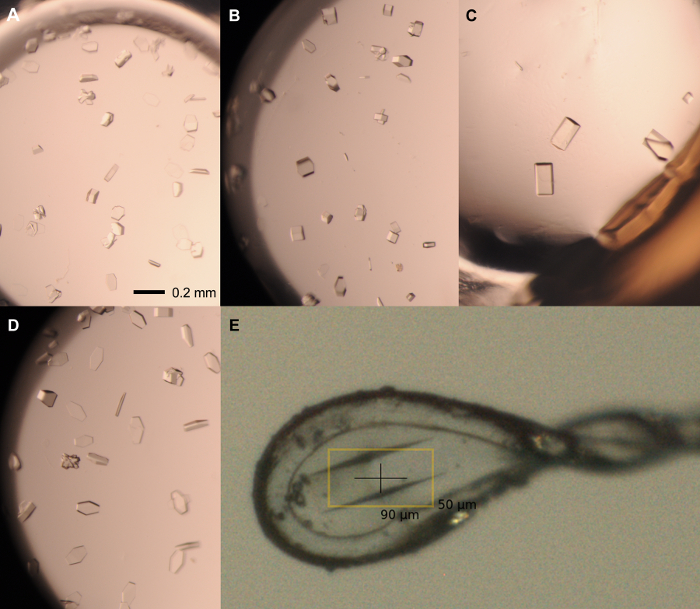
Abbildung 5: Repräsentative Kristalle von der Optimierung Bildschirm. Einkristalle aus rPPEP-1 bei 12 mg / ml ausgesät mit 1: 1000-Verdünnung Saatgut in den folgenden Bedingungen gezüchtet: (A) 2,1 M Ammoniumhydrogenphosphat, 0,1 M Tris , pH 7,5; 1,5 ul: 1,5 ul; (B) 2,1 M Ammoniumhydrogenphosphat, 0,1 M Tris, pH 7,5; 2 & mgr; l 1 & mgr; l; (C) 2,25 M Ammoniumphosphat dibasisches, 0,1 M Tris pH 8; 2 & mgr; l 1 & mgr; l; (D) 2,1 M Ammoniumhydrogenphosphat, 0,1 M Tris pH 8; 1 & mgr; l 2 & mgr; l. (E) Mounted Kristall in 0,1-0,2 um Nylon - Schleife, in 2,1 M Ammoniumhydrogenphosphat gewachsen, 0,1 M Tris pH 8 (2 ul: 1 & mgr; l) und Kryo-geschützt in 2,1 M Ammoniumhydrogenphosphat, 0,1 M Tris pH 8, 20% Glycerin. Der Maßstabsbalken = 0,2 mm (AD). Volumenverhältnis sind immer Protein: Reservoir. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
4. Kristall Montage und Datenerfassung
HINWEIS: Um die beste Qualität der Beugungsdaten Kristalle erhalten sollte auf dem Höhepunkt ihrer Qualität und Größe montiert werden. Kristalle können in flüssigem Stickstoff gelagert werden, bis sie are Daher bei 100 K. Röntgenbeugungsanalyse unterzogen, aus denen sie die Bedingung Kryo-Bedingungen eingestellt werden, stammen. rPPEP-1-Kristalle können entweder 20% Glycerol oder 30% Saccharose (Ersatz von Wasser in dem Zustand, der durch das Kryo-Schutzmittel) cryo geschützt werden durch Zugabe von.
- Kristallmontage
HINWEIS: Alle Kristallmanipulationsschritte unter dem Stereomikroskop durchgeführt werden sollte.- Wählen Sie die optimale Größe der Nylonschlaufe für die maximale Länge der ausgewählten Kristalle. Die typische längste Achse rPPEP-1 - Kristalle beträgt etwa 100-200 um (Abbildung 5). Bereiten Sie eine Abdeckschlitten und die entsprechende Kryo-Zustand (zB 2,1 M Ammoniumhydrogenphosphat, 0,1 M Tris, pH 8,0, 20% Glycerin).
- Füllen Sie die Schaum Dewar mit flüssigem Stickstoff, laden Sie das Fläschchen Klemme mit einem Fläschchen und vorzukühlen es in den flüssigen Stickstoff gefüllten 800 ml Schaum Dewar. Legen Sie eine Kryo Hülse und ein Kryo Stockhalter mit einem geeigneten Kennung markiertin flüssigem Stickstoff gefüllten 2 l Schaum Dewar. Legen Sie den Magnetstab mit aufgesetztem Nylonschlaufe.
HINWEIS: Schutzkleidung tragen (eyeshield / Brille, Handschuhe), wenn sie mit flüssigem Stickstoff zu arbeiten. Warme Objekte in flüssigen Stickstoff getaucht kann Leckagen erzeugen.
- Füllen Sie die Schaum Dewar mit flüssigem Stickstoff, laden Sie das Fläschchen Klemme mit einem Fläschchen und vorzukühlen es in den flüssigen Stickstoff gefüllten 800 ml Schaum Dewar. Legen Sie eine Kryo Hülse und ein Kryo Stockhalter mit einem geeigneten Kennung markiertin flüssigem Stickstoff gefüllten 2 l Schaum Dewar. Legen Sie den Magnetstab mit aufgesetztem Nylonschlaufe.
- Schneiden Sie öffnen Sie das Dichtungsband mit einem scharfen Skalpell und entfernen Sie sie mit der Zange. Pipette 1 ul der Kryo-Zustand auf den Abdeckschieber (oder alternativ in einer leeren Mulde auf der gleichen Platte) und entfernen Sie den Kristall aus dem Drop indem sie sie mit der montierten Nylonschlaufe Fischerei (Abbildung 5). Befestigt Kristalle können leicht vom Boden gelöst werden, indem die umgebenden Kunststoff mit einer Akupunktur-Nadel zu verformen.
- Schnell übertragen den Kristall auf den Rückgang der Kryo-Zustand und lassen Sie es für 1 Sekunde ins Gleichgewicht. Fisch den Kristall so schnell wie möglich und Tauchfrost in flüssigem Stickstoff.
- Wenn der flüssige Stickstoff um die montierte Schleife stoppt Kochen, legen Sie die Schleife in dem Fläschchen.Legen Sie das Fläschchen auf dem Kryo Stockhalter und, wenn sie mit 6 Flaschen legen Sie eine Kryo Hülse um den Halter geladen. Speichern der Kristalle in einem Behälter mit flüssigem Stickstoff bis zum Gebrauch gefüllt.
- Wählen Sie die optimale Größe der Nylonschlaufe für die maximale Länge der ausgewählten Kristalle. Die typische längste Achse rPPEP-1 - Kristalle beträgt etwa 100-200 um (Abbildung 5). Bereiten Sie eine Abdeckschlitten und die entsprechende Kryo-Zustand (zB 2,1 M Ammoniumhydrogenphosphat, 0,1 M Tris, pH 8,0, 20% Glycerin).
- Datensammlung
HINWEIS: Die Datenerfassung kann im Hause Diffraktometer durchgeführt werden, wenn verfügbar, oder an einem Synchrotron-Strahlrohr. Für rPPEP-1 Daten wurden auf der beamline X06DA der Synchrotron Lichtquelle Schweiz, Paul-Scherrer-Institut, Villigen, Schweiz mit einem Hybrid-Photon-Counting-Detektor gesammelt. Die Originaldaten und alle Dateien in Strukturbestimmung verwendet wird, kann auf Anfrage zur Verfügung gestellt werden.- Eingerichtet, um die Wellenlänge des Strahls auf 1.282 Å (9667 keV), die die charakteristische Röntgenabsorptionskantenenergie (peak) des Elements Zink. rPPEP-1 ist eine Metalloprotease, die ein einzelnes Zink pro Molekül in der aktiven Stelle enthält.
- Sammeln von Daten bei 100 K in dem Inversen des abgeblendeten Modus in 10 ° Keilen für insgesamt 270 ° in jeder direction. Die Belichtungszeit beträgt 0,1 sec bei 0,1 ° -Drehung pro Bild. Stellen Sie die Übertragung auf 14% (0,14).
- Um eine native hochauflösenden Datensatzes aus einem zweiten Kristall sammeln aus dem gleichen Kristallisationsbedingung Ursprung gesetzt, die Wellenlänge des Strahls auf 1,00 Å bis (12.398 keV). Sammeln von Daten bei 100 K. Die Belichtungszeit 0,1 s mit 0,1 ° Drehung pro Bild ist. Stellen Sie die Übertragung auf 70% (0,7).
5. Strukturbestimmung über Zink-SAD
HINWEIS: Um die Struktur von rPPEP-1 über Zink-SAD einige grundlegende kristallographischen Wissen , um zu bestimmen , sowie die Software - Pakete XDS benötigt 20, Phenix 21 und das Programm Blässhuhn 22. Zur Visualisierung von Strukturen das Programm PyMOL 23 oder 24 Chimera benötigt. Daten bei der Wellenlänge gesammelt entsprechend dem Peak bei der Absorptionskante des Elements Zink kann zur Einzel Wellenlänge anomale disp verwendet werdenersion (SAD) 25 , um Phaseninformationen zu erhalten , die für alle Proteinatome verlängert werden kann.
- Datenverarbeitung
- Verarbeiten Sie die beiden Spitzen Datensätze (normal und invers) mit Hilfe der Software XDS (alternativ iMosflm oder HKL3000) in der Raumgruppe P2 1 2 1 2 1 (Raumgruppe 19) die Friedel Geschwister (anomale Daten) zu trennen. Die Einheitszellparameter sollten um a, b, c (A) = 43.17, 71.68, 117.70 und α = β = γ (°) = 90. Das gibt zwei HKL-Dateien (Reflexions-Dateien).
- Überprüfen Sie die Datei CORRECT.LP. Verwenden Sie Daten bis zu der Auflösung , in dem die CC 1/2 beträgt mindestens 50%. Skalieren zusammen beide Datensätze / Reflexion Dateien (HKL-Dateien) mit XSCALE. Überprüfen Sie die Datei XSCALE.LP. Prüfen Sie, wie weit das anomale Signal erstreckt (SigAno) und beachten Sie die Auflösung mit einer anomalen Korrelation (anomal Corr) von etwa 30%, was im Fall der Daten 2 A verwendet hier auf 1,67 Å gesammelt. Dies ist dasAuflösung cut-off für das anomale Signal verwendet in Phenix autosol.
- Konvertieren Sie die (skaliert) HKL-Datei in ein CCP4-Format Reflexion Datei (benannt, zum Beispiel peak_anom.mtz) mit XDSCONV eine R freie Teilmenge von 5% zu schaffen und halten die anomale Daten (FRIEDEL'S_LAW = FALSE). Schauen Sie sich die MTZ-Datei für die Übereinstimmung mit dem Programm mtzdmp die Zellparameter Inspektion, die Raumgruppe und die Existenz der R freie Teilmenge (Label FreeRflag) und die anomalen Daten (Etiketten DANO / SIGDANO). Bereiten Sie auch eine zusätzliche MTZ-Datei mit XDSCONV ohne die anomalen Daten zu extrahieren (FRIEDEL'S_LAW = TRUE; genannt, zum Beispiel peak_native.mtz) zur Verfeinerung zu einem späteren Zeitpunkt.
- Unterbau - Lösung (Phasenbestimmung)
- Führen Sie Phenix autosol die Reflexion Datei peak_anom.mtz verwenden. Wählen Sie SAD / MAD Spitze als Datentyp und wählen Sie 2 Zink-Websites (wie es zwei Moleküle pro asymmetrische Einheit). Wählen Sie entweder die genauere Erfahgeistige Werte für die f '/ f' 'Parameter (in einem Fluoreszenz-Scan an der Beamline bestimmt) oder die theotetical Werte f' = -8,245 und f '' = 3,887. Auch laden die FASTA-Datei mit der Aminosäuresequenz des kristallisierten Protein enthält.
- Stellen Sie die Auflösungsgrenze der Auflösung mit einer anomalen Korrelation (anomal Corr) von etwa 30% (bestimmt in 5.1.2), in diesem Fall 2 Å und wählen Sie die "Autobuild-Modell" Option. Mit den Phasen der beiden Zink-Websites von Phenix HYSS (Teil der Pipeline Phenix autosol) fanden die Phasen für das gesamte Protein abgeleitet und baute das Modell werden konnte (von Phenix RESOLVE) in die Elektronendichte. Das beste Modell ist "overall_best.pdb" genannt.
- Modellbau, Optimierung und Validierung
- Wählen Sie die Option "Autobuild-Modell", um den größten Teil der automatisch rPPEP-1-Modell bauen. Überprüfen Sie die Elektronendichte bei 1,0 σ Kontur Ebene der progra mitm Blässhuhn (Abbildung 6). Es sollte die Atome des Modells Binde- und umgebenden sein. Im Idealfall sollten auch einige Wassermoleküle in das Modell eingebaut werden (bei einer Auflösung besser als 2,5 Å). Bulk-Wasser (Raum zwischen den Molekülen) sollte keine Dichte enthalten.
- Überprüfen Sie, ob das ganze Modell ist vollständig (alle Aminosäuren in die Elektronendichte gebaut). Wenn nicht, bauen sie manuell die Werkzeuge von Blässhuhn zur Verfügung gestellt werden. die Struktur Filtern nach mit 5 Verfeinerung iterative Runden von Phenix verfeinern Laufrunden jeweils die overall_best.pdb Modelldatei, die peak_native.mtz Reflexion Datei und die FASTA-Sequenz-Datei; und manuelle Modellbau in Blässhuhn.
- Überprüfen Sie die Qualität des Strukturmodells mit den entsprechenden Werkzeugen in Blässhuhn.
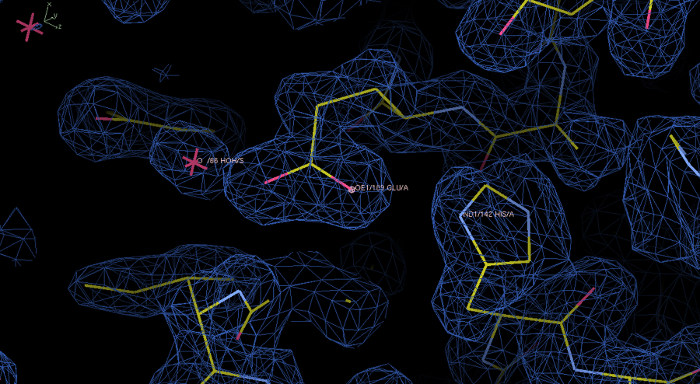
Abbildung 6: Experimentelle Elektronendichtekarte und das Modell des rPPEP-1 nach demPhenix Autosol laufen. Elektronendichte in Blau bei einer Konturebene von 1,0 σ im Programm Blässhuhn angezeigt. In dieser ersten Karte ist die Elektronendichte gut gelöst und das Modell zu bauen in die Elektronendichte. Die Zoom-in zeigt die Reste His142 und Glu189 sowie ein Wassermolekül. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
6. Strukturbestimmung zu Hohe Auflösung über Molecular Replacement
HINWEIS: Um hochauflösende strukturelle Informationen über rPPEP-1 eine native Datensatzes zu erhalten, wird gesammelt. Dann wird eine molekulare Ersetzungsprozedur der Software Phaser 26,27 (innerhalb der Software - Paket Phenix) verwendet , wird verwendet , um die Struktur über Zink-SAD als Modell bestimmt. Dieses Verfahren kann auch nachträglich eingesetzt werden, wenn Strukturen rPPEP-1 im Komplex mit kleinen Molekülen zu lösen.
- Um eine Kristallstruktur mit höherer Auflösung (in diesem Fall bis zu 1,4 Å) verarbeiten die native Datensatzes mit Hilfe der Software XDS (alternativ iMosflm oder HKL3000) in der Raumgruppe P2 1 2 1 2 1 (Raumgruppe 19) zu erhalten. Die Einheitszellparameter sollten um a, b, c (A) = 43.17, 71.77, 117.80 und α = β = γ (°) = 90. Das gibt eine HKL-Dateien (Reflexions-Datei).
- Überprüfen Sie die Datei CORRECT.LP. Verwenden Sie Daten bis zu der Auflösung , in dem die CC 1/2 beträgt mindestens 50%. Konvertieren Sie die HKL-Datei in ein CCP4-Format Reflexion Datei (benannt, zum Beispiel native.mtz) mit XDSCONV eine R freie Teilmenge von 5% zu schaffen. Schauen Sie sich die MTZ-Datei für die Übereinstimmung mit dem Programm mtzdmp die Zellparameter Inspektion, die Raumgruppe und die Existenz der R freie Teilmenge (Label FreeRflag).
- Bereiten Sie die PDB-Datei , um das Modell aus overall_best.pdb bestimmt früher enthält , und entfernen Sie alle Wassermoleküle und alle Liganden (dh. das Zinkatom). Auch laden die FASTA-Datei mit der Aminosäuresequenz des kristallisierten Protein enthält. Führen Sie Phaser in Phenix die Reflexion Datei native.mtz verwenden. Suche nach zwei Molekülen pro asymmetrischer Einheit.
- Nach der erfolgreichen Strukturlösung (TFZ-Score größer als 8, hier 10.2) inspizieren das Modell (genannt native_phaser.1.pdb) und die Elektronendichtekarte in Blässhuhn. Erstellen Sie die Struktur zu verfeinern, indem Sie iterative Runden von Phenix verfeinern mit 5 Verfeinerung rundet jeweils die native_phaser.1.pdb Modell-Datei, die native.mtz Reflexion Datei und die FASTA-Sequenz-Datei; und manuelle Modellbau in Blässhuhn.
- Überprüfen Sie die Qualität des Strukturmodells mit den entsprechenden Werkzeugen in Blässhuhn.
Ergebnisse
rPPEP-1 wird in verschiedene E. coli - Stämmen überexprimiert, mit der höchsten Ausbeute in E. coli BL21 (DE3) Star (Abbildung 1C). Nach dem ersten Chromatographieschritt NiNTA Affinität der 6xHis-Tag erfolgreich von den meisten werden können des Proteins und in dem zweiten Schritt NiNTA unverdaute Protein vollständig von Thrombin-verdautem Protein (1D) abgespalten getrennt werden können. Auf einer S200 - Säule 16/600 unmarkiert...
Diskussion
Röntgenkristallographie ist immer noch die schnellste und genaueste Methode zur Bestimmung dreidimensionaler nahe Strukturen mit atomarer Auflösung von Proteinen 28. Es erfordert jedoch das Wachstum von gut geordneten Einkristallen. Diese sind oft schwer zu bekommen und der kristalline Zustand ist künstlich. Ein Vergleich der Proteinstrukturen durch Röntgenkristallographie mit den durch andere Verfahren bestimmt jedoch, insbesondere NMR, zeigt im Allgemeinen eine sehr gute Übereinstimmung. Im Falle von P...
Offenlegungen
The authors have nothing to disclose.
Danksagungen
Wir danken dem Personal an der beamline X06DA an der Synchrotron Lichtquelle Schweiz, Paul-Scherrer-Institut, Villigen, Schweiz, für Unterstützung bei der Synchrotron-Datenerfassung. Wir sind für einen ausgezeichneten technischen Support zu Monika Gompert dankbar. Das Projekt wurde von der Universität zu Köln unterstützt und gewähren INST 216 / 682-1 fugg von der Deutschen Forschungsgemeinschaft. Ein Promotionsstipendium von der International Graduate School in Entwicklung Gesundheit und Krankheit zu CP quittiert. Die Forschung zu diesen Ergebnissen geführt haben, wurden von der Europäischen Gemeinschaft Siebten Rahmenprogramm (FP7 / 2007-2013) im Rahmen der Finanzhilfevereinbarung Nr 283570 (BioStruct-X) erhalten.
Materialien
| Name | Company | Catalog Number | Comments |
| Genes / Vectors / cell strains | |||
| pET28a vector | Merck-Millipore | 69864 | Thrombin cleavable N-terminal His-tag |
| E. coli strain BL21 (DE3) Star | ThermoFisher Scientific | C601003 | RNase H deficient |
| Codon-optimized gene (for E. coli) of PPEP-1 (CD630_28300) | Geneart (Thermo Fisher Scientific) | custom | amino acids 27-220 |
| Name | Company | Catalog Number | Comments |
| Chemicals | |||
| Yeast extract | any | ||
| Tryptone | any | ||
| Antifoam B | Sigma-Aldrich | A5757 | aqueous-silicone emulsion |
| Agar | any | ||
| Kanamycin | any | ||
| IPTG | AppliChem | A1008 | |
| Tris-HCl | AppliChem | A1087 | Buffer grade |
| NaCl | any | Buffer grade | |
| DNaseI | AppliChem | A3778 | |
| Imidazole | AppliChem | A1073 | Buffer grade |
| Thrombin | Sigma-Aldrich | T4648 | |
| Ammonium phosphate dibasic | Sigma-Aldrich | 215996 | |
| Glycerol 100% | any | purest grade | |
| Sucrose | Sigma-Aldrich | 84097 | |
| Liquid nitrogen | any | for storage and cryocooling of crystals | |
| Name | Company | Catalog Number | Comments |
| Equipment (general) | |||
| Shaking incubator | any | providing temperatures of 20 °C - 37 °C | |
| Glassware | any | baffled Erlenmeyer flasks (50 ml - 2.8 L) | |
| Centrifuge for large culture volumes | any | centrifuge for processing volumes up to 12 L | |
| Sonicator Vibra-Cell VCX500 | Sonics | SO-VCX500 | or any other sonicator / cell disruptor |
| Ultracentrifuge | any | centrifuge providing speeds up to 150,000 x g | |
| NiNTA Superflow resin | Qiagen | ||
| Empty Glass Econo-Column | Bio-Rad | 7371007 | or any other empty glass or plastic column |
| Size exclusion chromatography column HiLoad Superdex 200 16/600 | GE Healthcare | 28989335 | |
| Chromatography system Äkta Purifier | GE Healthcare | 28406264 | or any other chromatography system |
| Dialysis tubing Spectra/Por 3 | Spectrum Labs | 132724 | |
| Dialysis tubing closures | Spectrum Labs | 132738 | |
| Ultrafiltration units (concentrators) 10,000 NWCO | any | ||
| UV-Vis spectrophotometer | any | ||
| Name | Company | Catalog Number | Comments |
| Equipment (crystallography) | |||
| Low volume pipette 0.1-10 µl | any | ||
| Positive displacement pipette Microman M10 | Gilson | F148501 | |
| Crystallization robot | any | ||
| 96-well crystallization plates TTP IQ with three protein wells | TTP | 4150-05810 | or any other 96-well crystallization plate |
| 24-well CombiClover Junior Plate | Jena Bioscience | EB-CJR | |
| Crystal Clear Sealing Tape | Hampton Research | HR3-511 | |
| Siliconized Glass Cover Slides | Hampton Research | HR3-225 | |
| Commercial crystallization screens: SaltRx, Index, PEG/Ion, Crystal | Hampton Research | diverse | |
| Commercial crystallization screens: Wizard, PACT++, JCSG++ | Jena Bioscience | diverse | |
| JBS Beads-for-Seeds | Jena Bioscience | CO-501 | |
| CrystalCap SPINE HT (nylon loops) | Hampton Research | diverse | loop sizes 0.025 mm - 0.5 mm |
| CrystalCap Vial | Hampton Research | HR4-904 | |
| Cryogenic Foam Dewar 800 ml | Hampton Research | HR4-673 | |
| Cryogenic Foam Dewar 2 L | Hampton Research | HR4-675 | |
| Vial Clamp, Straight | Hampton Research | HR4-670 | |
| CrystalWand Magnetic, Straight | Hampton Research | HR4-729 | |
| CryoCane 6 Vial Holder | Hampton Research | HR4-711 | |
| CryoSleeve | Hampton Research | HR4-708 | |
| CryoCane Color Coder - White | Hampton Research | HR4-713 | |
| Scalpel | any | ||
| Straight microforcep | any | for manipulation of sealing tape. etc. | |
| Acupuncture needle | any | e.g. from a pharmacy | |
| Stereo microscope | any | for inspection of crystallization plates and crystal mounting, magnification up to 160X |
Referenzen
- Bouza, E. Consequences of Clostridium difficile infection: understanding the healthcare burden. Clin Microbiol Infect. 18 (Suppl 6), 5-12 (2012).
- O'Connor, J. R., Johnson, S., Gerding, D. N. Clostridium difficile infection caused by the epidemic BI/NAP1/027 strain. Gastroenterology. 136 (6), 1913-1924 (2009).
- Mitchell, B. G., Gardner, A. Mortality and Clostridium difficile infection: a review. Antimicrob Resist Infect Control. 1 (1), (2012).
- George, W. L., Sutter, V. L., Finegold, S. M. Antimicrobial agent-induced diarrhea--a bacterial disease. J Infect Dis. 136 (6), 822-828 (1977).
- George, R. H., et al. Identification of Clostridium difficile as a cause of pseudomembranous colitis. Br Med J. 1 (6114), 695 (1978).
- Bartlett, J. G. Narrative review: the new epidemic of Clostridium difficile-associated enteric disease. Ann Intern Med. 145 (10), 758-764 (2006).
- Kelly, C. P., LaMont, J. T. Clostridium difficile--more difficult than ever. N Engl J Med. 359 (18), 1932-1940 (2008).
- Ünal, C. M., Steinert, M. Novel therapeutic strategies for Clostridium difficile infections. Expert Opin Ther Targets. 20 (3), 269-285 (2016).
- Kelly, C. P., et al. The Monoclonal Antibody, Bezlotoxumab Targeting C. difficile Toxin B Shows Efficacy in Preventing Recurrent C. difficile Infection (CDI) in Patients at High Risk of Recurrence or of CDI-Related Adverse Outcomes. Gastroenterology. 150 (4), S122 (2016).
- Tsutsumi, L. S., Owusu, Y. B., Hurdle, J. G., Sun, D. Progress in the discovery of treatments for C. difficile infection: A clinical and medicinal chemistry review. Curr Top Med Chem. 14 (1), 152-175 (2014).
- Hensbergen, P. J., et al. Clostridium difficile secreted Pro-Pro endopeptidase PPEP-1 (ZMP1/CD2830) modulates adhesion through cleavage of the collagen binding protein CD2831. FEBS Lett. 589 (24), 3952-3958 (2015).
- Cafardi, V., et al. Identification of a novel zinc metalloprotease through a global analysis of Clostridium difficile extracellular proteins. PLoS One. 8 (11), e81306 (2013).
- Hensbergen, P. J., et al. A novel secreted metalloprotease (CD2830) from Clostridium difficile cleaves specific proline sequences in LPXTG cell surface proteins. Mol Cell Proteomics. 13 (5), 1231-1244 (2014).
- Schacherl, M., Pichlo, C., Neundorf, I., Baumann, U. Structural Basis of Proline-Proline Peptide Bond Specificity of the Metalloprotease Zmp1 Implicated in Motility of Clostridium difficile. Structure. 23 (9), 1632-1642 (2015).
- Rawlings, N. D., Waller, M., Barrett, A. J., Bateman, A. MEROPS: the database of proteolytic enzymes, their substrates and inhibitors. Nucleic Acids Res. 42 (Release 10.0), D503-D509 (2014).
- Bergfors, T. Seeds to crystals. J Struct Biol. 142 (1), 66-76 (2003).
- Dauter, Z., Dauter, M., Dodson, E. Jolly SAD. Acta Crystallogr D Biol Crystallogr. 58 (Pt 3), 494-506 (2002).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 227 (5259), 680-685 (1970).
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Anal Biochem. 72, 248-254 (1976).
- Kabsch, W. XDS. Acta Crystallogr D Biol Crystallogr. 66 (Pt 2), 125-132 (2010).
- Adams, P. D., et al. PHENIX: a comprehensive Python-based system for macromolecular structure solution. Acta Crystallogr D Biol Crystallogr. 66, 213-221 (2010).
- Emsley, P., Lohkamp, B., Scott, W. G., Cowtan, K. Features and development of Coot. Acta Crystallogr D Biol Crystallogr. 66 (Pt 4), 486-501 (2010).
- . . The PyMOL Molecular Graphics System. , (2002).
- Pettersen, E. F., et al. UCSF Chimera--a visualization system for exploratory research and analysis. J Comput Chem. 25 (13), 1605-1612 (2004).
- Wang, B. C. Resolution of phase ambiguity in macromolecular crystallography. Methods Enzymol. 115, 90-112 (1985).
- McCoy, A. J., Grosse-Kunstleve, R. W., Adams, P. D., Winn, M. D., Storoni, L. C., Read, R. J. Phaser crystallographic software. J Appl Crystallogr. 40 (Pt 4), 658-674 (2007).
- Zwart, P. H., et al. Automated structure solution with the PHENIX suite. Methods Mol Biol. 426, 419-435 (2008).
- Zheng, H., Handing, K. B., Zimmerman, M. D., Shabalin, I. G., Almo, S. C., Minor, W. X-ray crystallography over the past decade for novel drug discovery - where are we heading next?. Expert Opin Drug Discov. 10 (9), 975-989 (2015).
- Rubino, J. T., et al. Structural characterization of zinc-bound Zmp1, a zinc-dependent metalloprotease secreted by Clostridium difficile. J Biol Inorg Chem. 21 (2), 185-196 (2016).
- Carson, M., Johnson, D. H., McDonald, H., Brouillette, C., Delucas, L. J. His-tag impact on structure. Acta Crystallogr D Biol Crystallogr. 63 (Pt 3), 295-301 (2007).
- Gasteiger, E., Walker, J. M., et al. Protein Identification and Analysis Tools on the ExPASy Server. The Proteomics Protocols Handbook. , 571-607 (2005).
- Dummler, A., Lawrence, A. M., de Marco, A. Simplified screening for the detection of soluble fusion constructs expressed in E. coli using a modular set of vectors. Microb Cell Fact. 4, 34 (2005).
- Stura, E. A., Wilson, I. A. Applications of the streak seeding technique in protein crystallization. J Crys Growth. 110 (1), 270-282 (1991).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten