Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Производство, Кристаллизация и структура Определение
В этой статье
Резюме
Proline-proline endopeptidase-1 (PPEP-1) is a secreted metalloprotease and promising drug-target from the human pathogen Clostridium difficile. Here we describe all methods necessary for the production and structure determination of this protein.
Аннотация
New therapies are needed to treat Clostridium difficile infections that are a major threat to human health. The C. difficile metalloprotease PPEP-1 is a target for future development of inhibitors to decrease the virulence of the pathogen. To perform biophysical and structural characterization as well as inhibitor screening, large amounts of pure and active protein will be needed. We have developed a protocol for efficient production and purification of PPEP-1 by the use of E. coli as the expression host yielding sufficient amounts and purity of protein for crystallization and structure determination. Additionally, using microseeding, highly intergrown crystals of PPEP-1 can be grown to well-ordered crystals suitable for X-ray diffraction analysis. The methods could also be used to produce other recombinant proteins and to study the structures of other proteins producing intergrown crystals.
Введение
Clostridium несговорчивый является одной из основных причин внутрибольничных антибиотик-ассоциированной диареи инфекций 1. Этот грамположительных анаэробной бактерии передается через споровой форме через фекально-оральным путем. В последнее десятилетие, новая '' эпидемия '' или '' гипервирулентных '' штаммов (например , BI / NAP1 / 027) вызвало резкое увеличение числа новых инфекций и летальности в Северной Америке и Европе 2. C. несговорчивый -associated болезнь (РЕТЕНЦИИ) является опасным для жизни воспаление толстой кишки с высоким уровнем смертности 3. Симптомы варьируются от диареи 4 до псевдомембранозного колита 5 и часто со смертельным исходом токсического мегаколон 6.
Лечение РЕТЕНЦИИ трудно , как вирулентные штаммы с множественной лекарственной устойчивостью и частота рецидивов высока 7. В данный момент терапия включает в себя антибиотики метронидазол, fidaxomicin или ванкомицин, или в repetitiVely рецидивирующие случаи фекальные трансплантации микрофлора. Новые терапевтические стратегии срочно необходимы 8. Определенный прогресс записывается в качестве терапевтического моноклональных антител Bezlotoxumab, нацеливание C. несговорчивый токсин B 9, недавно успешно прошел III фазы клинических испытаний и был подан на утверждение с FDA и EMA. Кроме того, новые антибиотики испытываются в данный момент на разных стадиях клинических испытаний 10.
Для разработки эффективного лечения новые терапевтические мишени должны быть идентифицированы. Недавно обнаруженный C. несговорчивый протеазы пролин-пролина эндопептидазы-1 (PPEP-1; CD2830 / Zmp1; EC 3.4.24.89) является таким перспективной мишенью, как отсутствие PPEP-1 в нокаут-штамм уменьшает вирулентность C . несговорчивый в естественных условиях 11. PPEP-1 представляет собой секретируемый металлопротеазы 12,13 расщепляющие два C.difficile , адгезинов на их С-конца 13 , таким образом , освобождая клейкий BacterИ. из человеческого кишечника эпителия. Таким образом, она участвует в поддержании баланса между сидячим и мотильных фенотипа C. несговорчивый. Для разработки селективных ингибиторов против PPEP-1 и понять, как он признает свои субстраты глубокое знание его трехмерной структуры является необходимым условием. Мы решили первую кристаллическую структуру PPEP-1 по отдельности и в комплексе с субстратом пептида 14. PPEP-1 является первой известной протеазы , которые селективно расщепляет пептидные связи между двумя остатками пролина 15. Он связывает субстрат в двойном перекручены образом и стабилизирует его через расширенную жирноароматических сеть остатков , расположенных в S-петле , которая покрывает протеазы активного сайта 14. Этот режим субстратсвязывающих является уникальным для PPEP-1 и не нашли в человеческих протеаз на сегодняшний день. Это делает его перспективным объектом наркотиков, и вне мишени эффекты ингибиторов очень маловероятно.
Для разработки и экрана селективного PPEP-1 вдыханиеibitors в будущем требуется большое количество чистой и монодисперсному PPEP-1 белка. Кроме того, для определения режима связывания первых ингибиторов, со-кристаллических структур с PPEP-1 должны быть определены. В наших руках PPEP-1 постоянно производит сросшиеся кристаллы. Таким образом, мы разработали процедуру оптимизации для получения монокристаллов дифракционного качества PPEP-1. В этом протоколе мы опишем подробно производства, очистки, кристаллизации и структуру раствора PPEP-1 14. Мы используем внутриклеточную экспрессию в кишечной палочки из более PPEP-1 варианта , лишенного сигнальной последовательности секреции, аффинной хроматографии и гель -проникающей хроматографии с удалением метки очистки, а затем microseeding 16 в экране оптимизации и определения структуры с помощью цинка монохромному аномальной дисперсии (цинк-SAD) 17. Этот протокол может быть адаптирован для определения производства и структуры других белков (например , </ EM> металлопротеаз) и, в частности, для белков, производящих сросшиеся кристаллы. По запросу ДНК плазмиды конструкции (pET28a-Nhis-rPPEP-1) и дифракционные данные могут быть предоставлены для образовательных целей.
протокол
1. Клонирование и строительства Проектирование
- Клон кодон-оптимизированная последовательность (для кишечной палочки) С. несговорчивый PPEP-1 без сигнального пептида [аминокислоты 27-220, названные далее рекомбинантный PPEP-1 (rPPEP-1) 11] в вектор pET28a с использованием NDE I и сайты рестрикции Xho I (рисунок 1) с стоп - кодон на 3'-конец (результирующего вектора pET28a-Nhis-rPPEP-1). Это приводит к N-терминально 6xHis-меченого белка (Nhis-rPPEP-1) с сайтом расщепления тромбином , позволяющей извлекать тег во время очистки (Рисунок 1). Плазмида содержит канамицин кассету сопротивления для выбора. Праймеры , используемые для клонирования описаны в другом месте 14.
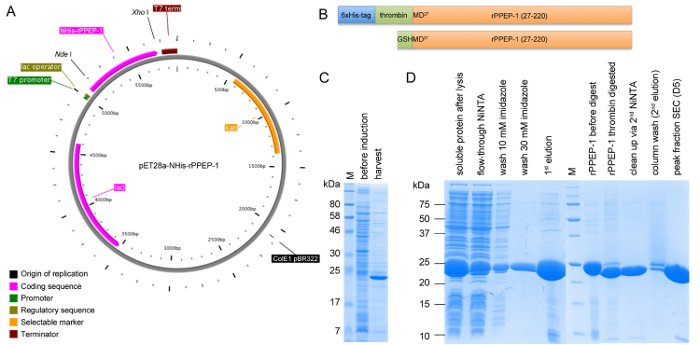
Рисунок 1: Схематическое представление построения pET28a-Nhis-rPPEP-1 и SDS-PAGE анализа expression и все этапы очистки. (A) Векторная карта Nhis-rPPEP-1 клонировали в вектор pET28a использованием NDE I / Xho I , созданный с PlasMapper. (Б) Схематическое представление НСМСИ-rPPEP-1 конструкцию (верхняя панель) и конечная конструкция после того, как тромбин-расщеплением 6xHis-тега с результирующей дополнительной ОСА-последовательности на N-конце (нижняя панель). Анализ SDS-PAGE (С) экспрессии в BL21 (DE3) Star при 37 ° С в течение 4 ч и (D) образцов из всех стадий очистки (М: маркер молекулярной массы). Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
2. Экспрессия и очистка rPPEP-1
- Выражение Nhis-rPPEP-1
- Составляют и автоклав LB (лизогении бульон) среда (10 г / л триптона, 5 г / л дрожжевого экстракта, 10 г / л NaCl, AdjuSt до рН 7,5 с помощью NaOH). Дополнение с канамицина сульфата (50 мкг / мл) непосредственно перед использованием (среды LB / Kan).
- Привить 200 мл ночной культуры из свеже трансформированной Е.coli в LB / Kan среде. Расти в течение ночи при 37 ° C при встряхивании со скоростью 220 оборотов в минуту.
- На следующее утро, проверьте OD 600 (оптическая плотность при 600 нм) ночной культуры. Привить два 2,8 л озадачены колбы , содержащие 1 л LB / Кан среды каждый с ночной культуры до OD 600 , равной 0,1. Дополнение с тремя каплями водно-силиконовой эмульсии для предотвращения чрезмерного образования пены. Рост клеток при 37 ° С встряхивании при 180 оборотах в минуту до OD 600 достигает 0,6.
- Возьмите образец предварительно индукционную для анализа SDS-PAGE (эквивалент 1 мл из культуры при OD 600 = 1); добавить IPTG до конечной концентрации 0,5 мМ, чтобы индуцировать экспрессию НСМСИ-rPPEP-1. Продолжить расти при 37 ° С / 180 оборотов в минуту в течение 4 часов.
- Определить OD 600 в 10х дилюция и взять пробу урожая (эквивалент 1 мл из культуры при OD 600 = 1).
- Собирают клетки центрифугированием в течение 20 мин при 7000 х г и 4 ° С. Для того, чтобы удалить остатки LB гранул среднего ресуспендирования клеток из 1 л культуры в 40 мл TBS буфера (Трис-солевой буфер: 20 мМ Трис-HCl, рН 7,5, 200 мМ NaCl) и перенос т в центрифужную пробирку на 50 мл. Собирают клетки центрифугированием в течение 10 мин при 10000 х г и 4 ° C и хранят при -80 ° С до использования. Анализ выражения (лизате и растворимые фракции) с помощью SDS-PAGE 18.
- Очистка немаркированной rPPEP-1
- Принимать по 50 мкл пробы каждой стадии очистки для анализа SDS-PAGE. Ресуспендируют осадок клеток из 1 л культуры в буфере TBS с добавлением 10 мкг / мл DNAseI. С помощью 5 мл TBS / DNAseI на г клеток.
- Лизиса клеток путем обработки ультразвуком на льду / воде с применением амплитуды 30% в течение 15 мин (2 секунды импульсов с 2 сек паузы). Удалите мусор с помощью CENTRifugation в течение 10 мин при 10000 х г и 4 ° С и передачи супернатант в ультрацентрифужную пробирку. Очистить лизат в ультрацентрифуге в течение 30 мин при 165000 х г и 4 ° С.
- Работа при температуре 4-6 ° C. С помощью перистальтического насоса или с помощью хроматографии систему уравновешивания 2 мл никелевой-нитрилтриуксусная кислоты (NiNTA) смолы в стеклянную колонку с TBS буфера с добавлением 10 мМ имидазола, рН 7,5. В качестве альтернативы, используйте самотеком.
- Регулировка очищенного лизата с помощью 1 М имидазола, рН 7,5 до конечной концентрации 10 мМ. Нанесите лизата на колонку и не приведут к рассасыванию ступенчато с TBS буфером с добавлением 10 мМ и 30 мМ имидазола, соответственно, пока УФ-поглощение при 280 нм не достигла базового уровня.
- Элюции белка с TBS буфером плюс 250 мМ имидазола. Повторно уравновешивают колонку к TBS с добавлением 10 мМ имидазола и хранят в течение ночи.
- Определить концентрацию белка либо при 280 нм с помощью экстинкции coeffiциент 25900 М -1 см -1 или любым другим способом (например , метод Бредфорд 19). Добавляют 2 единиц тромбина на мг белка и диализ раствора белка в течение ночи при 4 ° С против 50-кратном объеме TBS (50x объема элюирования NiNTA).
Примечание: Примите правильную заготовку для определения концентрации белка, а имидазола сильно поглощает при длине волны 280 нм. - Пропускают раствор белка над уравновешенную NiNTA смолой для удаления нерасщепленный белка. Далее, применить тот же объем TBS с добавлением 10 мМ имидазола в колонну, чтобы восстановить все расщепленный белок. Для очистки колонны, элюировать все оставшиеся белок с 250 мМ имидазола. Анализ образцов посредством SDS-PAGE (Рисунок 1).
- Концентрат раствора белка до 4 мл с интервалом 10 мин при 4000 х г и 4 ° С с использованием центробежного блока ультрафильтрации. Смешайте белок с концентрацией внимания после каждого интервала, чтобы предотвратить осаждение и агрегацию. На этом этапе от времениLY некоторое количество осадков наблюдается для rPPEP-1, несмотря на процедуру смешивания.
- Применение концентрированного белка на предварительно уравновешенную колонку гель-проникающей хроматографии (в буфере TBS) при 4-6 ° С. Элюируют колонку буфером TBS, собирают 1 мл фракций и при условии 5 мкл каждой второй фракции в анализе SDS-PAGE. rPPEP-1 элюируется в одном пике , соответствующем мономере (рисунок 2). Время от времени незначительный пик при большей молекулярной массы наблюдается (противостоя пик), что соответствует димер белка. Выход должен быть около 50 мг чистого белка на литр культуры. Анализ всех образцов посредством SDS-PAGE 18 (рисунок 2).
- Принимать по 50 мкл пробы каждой стадии очистки для анализа SDS-PAGE. Ресуспендируют осадок клеток из 1 л культуры в буфере TBS с добавлением 10 мкг / мл DNAseI. С помощью 5 мл TBS / DNAseI на г клеток.
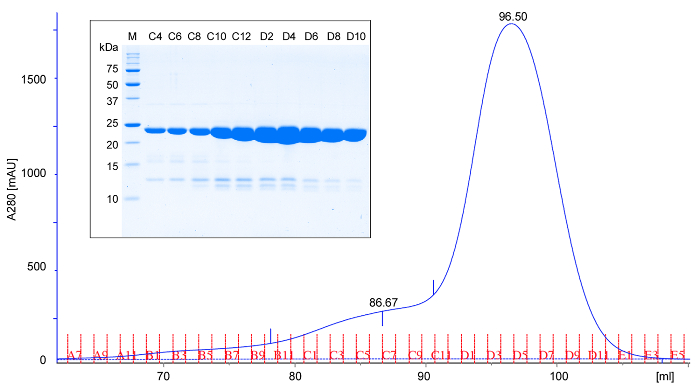
Рисунок 2: представитель вытеснительной хроматографии и электрофореза в ДСН-ПААГ rPPEP-1. Вытеснительная хроматограмма (A280; оптическую плотность при длине волны 280 нм) очищенного немаркированной rPPEP-1 с использованием 16/600) колонку (в Трис-HCl, рН 7,5, 200 мМ NaCl при 6 ° C. На основании объема элюирования, rPPEP-1 мигрирует, как ожидается, в течение 22 белка кД, предполагая, что он является преимущественно мономерные. Редко незначительные Фронтирование появляется пик, который соответствует димер. (Вставка) ДСН-ПААГ-анализ фракций из вытеснительной хроматографии (M; маркер молекулярной массы). Каждая вторая фракция применяется. Слабые полосы ниже главного rPPEP-1 группы соответствуют иногда происходят незначительные примеси. Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
3. Кристаллизация и Кристалл Оптимизация Используя Microseeding
Примечание: rPPEP-1 кристаллизуется из условий , которые постоянно производят высоко сросшиеся кристаллы , которые не подходят для анализа дифракции рентгеновских лучей (рисунок 3), Таким образом, была разработана стратегия оптимизации (Рисунок 4) для получения кристаллов высокого качества (рисунок 5).
- Первоначальный скрининг rPPEP-1 с использованием коммерческих экранов
Примечание: Проведение испытаний кристаллизации в сидячем формате падение с использованием стандартных коммерчески доступных экранов и кристаллизации робота.- Концентрат очищенный белок до 12 мг / мл с использованием центробежного устройства ультрафильтрации через 5-минутные интервалы при 4000 х г и 4 ° С. Смешайте белок с концентрацией внимания после каждого интервала, чтобы предотвратить осаждение и агрегацию. Определить концентрацию белка либо при 280 нм с использованием коэффициента экстинкции 25900 М -1 см -1 или любым другим способом (например , методом Брэдфорда). Равновесие белка до 20 ° С. Расчищать все частицы и пыль центрифугированием в течение 10 мин при 16000 х г и 20 ° C.
- Либо использовать уже предварительно заполненные кристаллизации плиты как таковыеАлед и хранили при 4 ° С или заполнить резервуар лунки пластин с 70 мкл каждого условия кристаллизации. Равновесие все кристаллизации пластин до 20 ° C. Работают быстро, поскольку небольшие объемы быстро высыхают. Используйте влажную камеру вокруг дока робота, если это возможно.
Примечание: Используйте следующие экраны в качестве стандартной процедуры: SaltRx, индекс, PEG / Ion, Crystal, мастера, ПАКТ ++, JCSG ++. - Настройка экрана с помощью пипетки белок и резервуар в subwells 2-4. объем капли составляет 300 нл и соотношения (белок: резервуар) являются 200: 100 (subwell 2), 150: 150 (subwell 3) и 100: 200 (subwell 4) (в NL). Сразу запечатать пластину и место в камере при температуре 20 ° С.
- Осмотрите подносы сразу же после установки, а затем проверять каждый день в течение первой недели с последующим еженедельным осмотром.
- Co-кристаллизация rPPEP-1 с лигандами
- Для совместной кристаллизации пептида-субстрата rPPEP-1 комплексов смешивать rPPEP-1при 24 мг / мл в соотношении 1: 1 (об / об) с 7-кратным мольным избытком раствора пептида (Ас-EVNPPVPD-NH 2) лиофилизированный порошок растворили в буфере TBS), который даст конечную концентрацию 12 мг / мл р-PPEP-1 белка и 7-кратный молярный избыток пептида над PPEP-1. Инкубировать в течение 30 мин при 20 ° С и рассеивать все частицы и пыль центрифугированием в течение 10 мин при 16000 х г и 20 ° C. Продолжайте кристаллизацией с использованием процедуры microseeding, как описано для несвязанного R-PPEP-1 белка.

Рисунок 3: Типичные кристаллы из начальных экранов. Сросшиеся кристаллы из rPPEP-1 при 12 мг / мл выращивают в состоянии. (A) экран Кристалл I / 38 (цитрат натрия 1,4 М трехосновная обезвоживают, 0,1 М HEPES рН натрия 7,5; 200 п: 100 NL). (B) SaltRx экран / 52 (2,4 М фосфата аммония DIBСИС, 0,1 М Трис, рН 8,5; 100 нл: 200 нл) и (С) (200 нл: 100 нл). (D) SaltRx экран / 96 (60% об / об Tacsimate рН 7,0, 0,1 М трис-пропан рН 7,0; 200 нл: 100 нл). Бар Scale = 0,2 мм. объемные соотношения всегда белок: резервуар. Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
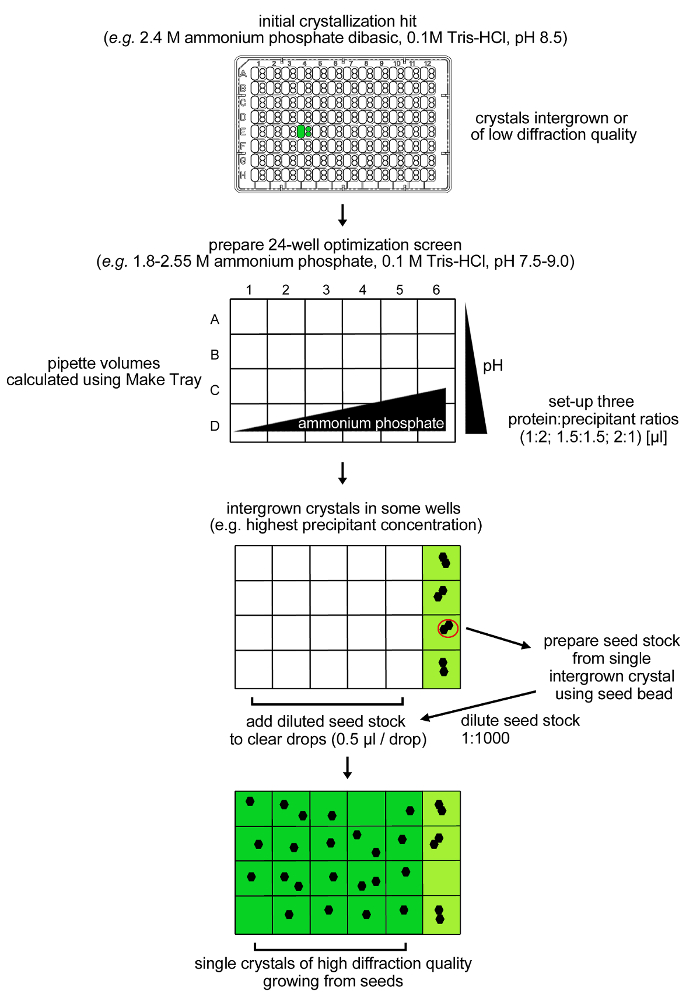
Рисунок 4: Процедура оптимизации для rPPEP-1 кристаллизации. Исходные кристаллы из rPPEP-1 при 12 мг / мл низкого качества дифракции и с несколькими решетками (сросшихся) были воспроизведены в экране оптимизации 24-состояния. Опять же, наблюдались только сросшиеся кристаллы в условиях, содержащих 2,55 М фосфата аммония двухосновной. Семенного материала был получен из одного сросшихся кристаллов и разводили 1: 1000 в том же Conditиона (microseeding). Объем 0,5 мкл разведенных семенного материала был добавлен в остальных прозрачных капель и монокристаллы выросли почти во всех условиях. Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
- Оптимизация кристалла с использованием microseeding
ПРИМЕЧАНИЕ: Высоко сросшиеся кристаллы rPPEP-1 появляются после двух дней в состоянии , содержащем 2,4 М фосфата аммония двухосновный 0,1 М Трис-HCl, рН 8,5 (экран SaltRx, состояние E4, все три subwells) (рисунок 3). Процедура оптимизации с помощью экранной сетки вокруг начального условия в сочетании с microseeding был применен (Рисунок 4).- Подготовьте экран сетки (рисунок 4) , содержащий 24 условий с 1,8 - 2,55 М фосфата аммония двухосновный (с шагом 0,15 м) и 0,1 М Трис-HCl рН 7,5-9,0 (с шагом 0,5 единицы рН) от appropriели маточные растворы (4 М фосфата аммония и 1 М Трис буферы).
Примечание: Используйте лоток апплет Марка (http://hamptonresearch.com/make_tray.aspx~~HEAD=dobj) для расчета объемов и схемы пипетирования для получения 2 мл каждого условия, что позволяет выполнить 10 экранов оптимизации. 4 М фосфата аммония раствор трудно приготовить. Раствор нагревают при перемешивании до полного растворения порошка в воде. - Пипетка по 200 мкл каждого экрана сетки раствора в лунки 24-луночного планшета и уравновешивают при температуре 20 ° С.
- Вручную настроить кристаллизационную пластину. Объем капли составляет 3 мкл и отношения (белка: резервуар) являются 2: 1, 1,5: 1,5 и 1: 2 (в мкл). При этом использовать положительный пипетки смещение, чтобы избежать образования пузырьков воздуха. Сразу запечатать пластину и место в камере при температуре 20 ° С. Избегайте образования пузырьков воздуха.
Примечание: После того, как от одного до четырех дней высоко сросшиеся кристаллы появляются в четырех условиях, содержащих фо 2,55 М аммонийsphate двузамещенный и 0,1 М Трис-HCl , рН 7,5 - 9,0 (рис 4). Кристаллы не образуются в остальных 20 условиях с концентрацией фосфата аммония ниже 2,55 M. используется процедура microseeding для получения монокристаллов rPPEP-1 в этих условиях. - Подготовьте microseed запас путем сбора одного сросшуюся кристалл из одного из двух условий с 2,55 М аммоний двухосновного фосфата и 0,1 М Трис-HCl, рН 8,0 или 8,5. Кристаллы могут быть прикреплены к пластиковой поверхности. Тщательное деформация окружающего пластика с акупунктурной иглой помогает отделить кристаллы.
- Передача 50 мкл соответствующего маточного раствора в 1,5 мл трубки, содержащие небольшое высоко полированного стекла шарик (шарики-для-семян). Используя установленный петлю нейлона передачи кристалл в 1 мкл маточного раствора помещают на стеклянную крышку слайд.
- Перенести жидкость, содержащую кристалл в пробирку и вихря на высокой скорости в течение 30 сек. Сделать 1:1000 разбавление семенного материала в новый 1,5 мл пробирку, содержащую тот же свежеприготовленной состояние и вихрь полностью в течение 5 секунд.
Примечание: Семена запасы можно хранить при -80 ° С для последующего использования.
- Снять уплотнение пластины, покрывающей 20 условий с ясными каплями и пипеткой 0,5 мкл семенного материала (1: 1000 разведение) в лунки. Уплотнение пластины и место в камере при температуре 20 ° С. Монокристаллы высокого качества дифракции появляются через 2-7 дней (рисунок 5).
- Подготовьте экран сетки (рисунок 4) , содержащий 24 условий с 1,8 - 2,55 М фосфата аммония двухосновный (с шагом 0,15 м) и 0,1 М Трис-HCl рН 7,5-9,0 (с шагом 0,5 единицы рН) от appropriели маточные растворы (4 М фосфата аммония и 1 М Трис буферы).
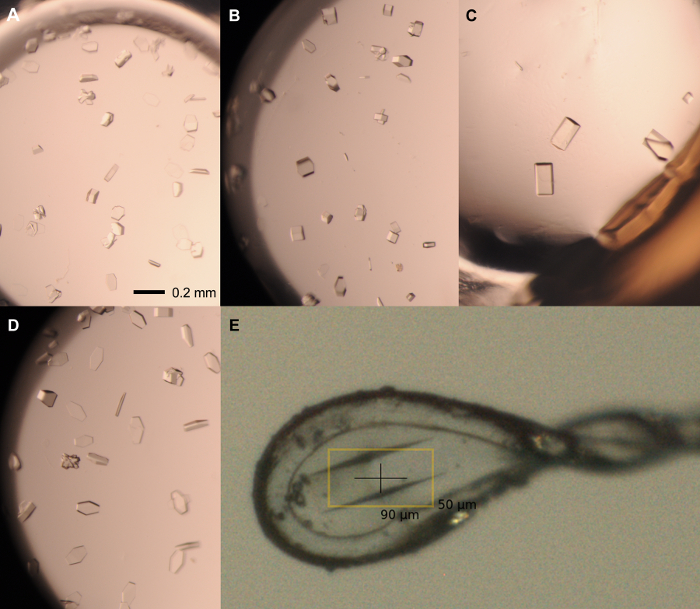
Рисунок 5: Типичные кристаллы с экрана оптимизации. Монокристаллы от rPPEP-1 при 12 мг / мл засевают 1: 1000 разбавлением семенного материала , выращенного в следующих условиях: (а) 2,1 М аммоний фосфат двухосновной, 0,1 М Трис , рН 7,5; 1,5 мкл: 1,5 мкл; (В) 2,1 М аммоний фосфат двузамещенный, 0,1 М Трис, рН 7,5; 2 мкл: 1 мкл; (С) 2,25 М аммоний фосфат двузамещенный, 0,1 М Трис , рН 8; 2 мкл: 1 мкл; (D) , 2,1 М аммоний фосфат двузамещенный, 0,1 М Трис , рН 8; 1 мкл: 2 мкл. (Е) Установленный кристалл в 0,1-0,2 мкм петли нейлон, выращенных в 2,1 М аммоний двухосновного фосфата, 0,1 М Трис , рН 8 (2 мкл: 1 мкл) и крио-защищенный в 2,1 М аммоний двухосновного фосфата, 0,1 М Трис , рН 8, 20% глицерина. Бар Scale = 0,2 мм (AD). Объем рациона всегда белка: резервуар. Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
4. Кристалл Монтаж и сбор данных
ПРИМЕЧАНИЕ: Для получения наилучшего качества кристаллов дифракционных данных должны быть установлены на пике их качества и размера. Кристаллы можно хранить в жидком азоте до тех пор, пока они аре подвергают анализу дифракции рентгеновских лучей при 100 К. Таким образом, условие, из которых они должны быть скорректированы в крио-условиях. rPPEP-1 кристаллы могут быть криоконсервации защищены добавлением либо 20% глицерина или 30% сахарозы (замена воды в состоянии с помощью крио-протравитель).
- Кристалл монтаж
Примечание: Все шаги манипуляции кристалл должны быть выполнены под стереомикроскопа.- Выберите оптимальный размер петли нейлона для максимальной длины выбранных кристаллов. Типичная длинная ось кристаллов rPPEP-1 составляет около 100-200 мкм (рисунок 5). Подготовьте крышку слайд и соответствующий крио-состояние (например , 2,1 М аммоний фосфат двузамещенный, 0,1 М Трис, рН 8,0, 20% глицерина).
- Заполните пенопластовые Дьюара с жидким азотом, загрузите флакон зажим с флакона и предварительно охладить его в жидком наполненным азотом 800 мл пены Дьюара. Поместите крио муфту и держатель крио тростника отмеченную с подходящим идентификаторомв жидком азоте заполненном 2 л пены Дьюара. Загрузите магнитную палочку с смонтированной петли нейлона.
Примечание: Носите защитную одежду (EyeShield / очки, перчатки) при работе с жидким азотом. Теплые объекты погрузилась в жидкий азот может привести к разливы.
- Заполните пенопластовые Дьюара с жидким азотом, загрузите флакон зажим с флакона и предварительно охладить его в жидком наполненным азотом 800 мл пены Дьюара. Поместите крио муфту и держатель крио тростника отмеченную с подходящим идентификаторомв жидком азоте заполненном 2 л пены Дьюара. Загрузите магнитную палочку с смонтированной петли нейлона.
- Разрежьте уплотнительную ленту с острым скальпелем и удалить его с помощью пинцета. Пипетка 1 мкл крио-состояния на крышку слайд (или в качестве альтернативы в пустой хорошо на той же пластинке) и удалите кристалл из выпадающего списка рыболовецких его с смонтированной петли нейлона (рисунок 5). Присоединенные кристаллы могут быть легко отделены от земли, деформируя окружающий пластик с акупунктуры иглы.
- Быстро перенесите кристалл каплю крио-состояния, и пусть он уравновешиваться в течение 1 секунды. Рыба кристалл, как быстро, как это возможно, и погружной замораживание в жидком азоте.
- Когда жидкий азот вокруг смонтированной петли прекращает кипеть, поместите петлю в ампуле.Поместите флакон на держателе крио тростника и при загрузке с 6 флаконов поместить крио втулку вокруг держателя. Хранить кристаллы в резервуаре, заполненном жидким азотом до использования.
- Выберите оптимальный размер петли нейлона для максимальной длины выбранных кристаллов. Типичная длинная ось кристаллов rPPEP-1 составляет около 100-200 мкм (рисунок 5). Подготовьте крышку слайд и соответствующий крио-состояние (например , 2,1 М аммоний фосфат двузамещенный, 0,1 М Трис, рН 8,0, 20% глицерина).
- Сбор данных
Примечание: Сбор данных может осуществляться на дому дифрактометра, если таковая имеется, или на синхротронное пучкового. Для того чтобы данные rPPEP-1 были собраны на пучкового X06DA швейцарского источника света, Поль-Шерера-института, Villigen, Швейцария с использованием детектора подсчета гибридного фотона. Исходные данные и все файлы, используемые в определении структуры могут быть предоставлены по запросу.- Настройка длины волны луча до 1,282 Å (9667 кэВ), что характеристическое рентгеновское края поглощения энергии (пик) элемента цинка. rPPEP-1 представляет собой металлопротеазы, который содержит один цинка на молекулу в активном сайте.
- Сбор данных при 100 К в режиме обратного луча в 10 ° клиньев в общей сложности на 270 ° в каждом directioп. Время экспозиции составляет 0,1 сек при 0,1 ° вращения на одно изображение. Установите коробку передач на 14% (0,14).
- Для того, чтобы собрать нативный данных высокого разрешения от второго кристалла, происходящих из того же состояния кристаллизации установить длину волны луча до 1,00 Å (12398 кэВ). Сбор данных при 100 К. Время экспозиции составляет 0,1 сек при 0,1 ° вращения на одно изображение. Установите коробку передач на 70% (0,7).
5. Структура Определение с помощью цинка-SAD
Примечание: Для того , чтобы определить структуру rPPEP-1 с помощью цинк-SAD необходимы некоторые основные кристаллографические знания, а также пакетов программного обеспечения XDS 20, Phenix 21 и программа Кут 22. Для визуализации структур программа PyMOL 23 или химера 24 необходим. Данные, собранные на длине волны, соответствующей пику поглощения на краю элемента цинка может быть использован для одной длины волны аномальной ИЗОБersion (SAD) 25 для получения информации о фазе , которая может быть расширена для всех атомов белка.
- Обработка данных
- Процесс два пика наборов данных (нормальные и обратные) с помощью программного обеспечения XDS (альтернативно iMosflm или HKL3000) в пространственной группе P2 1 2 1 2 1 (пространственная группа 19) , разделяющие Соседи Фриделя (аномальные данные). Параметры элементарной ячейки должны быть вокруг, b, c (A) = 43,17, 71,68, 117,70 и α = β = γ (°) = 90. Это дает два HKL-файлов (файлы отражения).
- Проверьте файл CORRECT.LP. Использование данных до разрешения , в котором СС 1/2 составляет по меньшей мере 50%. Масштаб вместе Оба набора данных / файлов отражения (HKL-файлы) с помощью XScale. Проверьте файл XSCALE.LP. Проверьте, насколько далеко простирается аномальный сигнал (SigAno) и принять к сведению резолюцию с аномальной корреляции (Аномальные Corr) около 30%, что на 2 Å в случае данных, используемых здесь, собранных на 1,67 Å. ЭтоРазрешение отсечения для аномального сигнала, используемого в Phenix Autosol.
- Преобразование (масштабируется) HKL-файл в файл отражения CCP4-формата (названный, например, peak_anom.mtz) с использованием XDSCONV создания R свободное подмножество 5% и сохранение аномальных данных (FRIEDEL'S_LAW = FALSE). Проверьте MTZ-файл для согласованности с программой mtzdmp инспектирующих параметры элементарной ячейки, пространственную группу и существование свободного подмножества R (метка FreeRflag) и аномальные данные (метки DANO / SIGDANO). Подготовьте также дополнительный MTZ-файл с XDSCONV без извлечения аномальных данных (FRIEDEL'S_LAW = TRUE, названный, например, peak_native.mtz) для уточнения на более позднем этапе.
- Несущая решение (определение фазы)
- Запуск Phenix Autosol с помощью файла отражения peak_anom.mtz. Выберите SAD / MAD пик как тип данных и выбрать 2 цинка сайтов (как есть две молекулы на асимметричный элемент). Выберите либо более точную Experiментальные значения для F '/ F' 'параметров (определено при сканировании флуоресценции в пучкового) или theotetical значений F' = -8.245 и F '' = 3,887. Также загрузить файл FASTA, содержащий аминокислотную последовательность, кристаллизованного белка.
- Установите предел разрешения до разрешения с аномальной корреляции (Аномальные Corr) около 30% (определено в 5.1.2), в данном случае 2 A и выберите опцию "автосборки модель". Используя фазы двух цинковых участков найденных Phenix Хисс (часть трубопровода Phenix Autosol) фазы для всего белка может быть прослежена, и модели, построенной (по Phenix RESOLVE) в электронной плотности. Лучшая модель называется "overall_best.pdb".
- Построение модели, уточнение и проверка
- Выберите опцию "модель AutoBuild", чтобы построить большую часть модели rPPEP-1 автоматически. Проверить электронную плотность на 1,0 σ уровне контура с использованием Prograм Кут (рисунок 6). Она должна быть соединительно и окружающие атомы модели. В идеале также некоторые молекулы воды должны быть встроены в модели (с разрешением лучше, чем 2,5 Å). Объемная вода (пространство между молекулами) не должен содержать плотности.
- Проверьте, если вся модель полно (все аминокислоты, встроенные в электронной плотности). Если нет, то строить их вручную с помощью инструментов, предоставляемых Кут. Уточнить структуру, выполнив итерационных раундов Phenix Уточнить с 5 утонченности раундов каждый из которых использует файл модели overall_best.pdb, файл peak_native.mtz отражения и файл последовательности FASTA; и ручное построение модели в Кут.
- Проверка качества структурной модели с соответствующими инструментами в Кут.
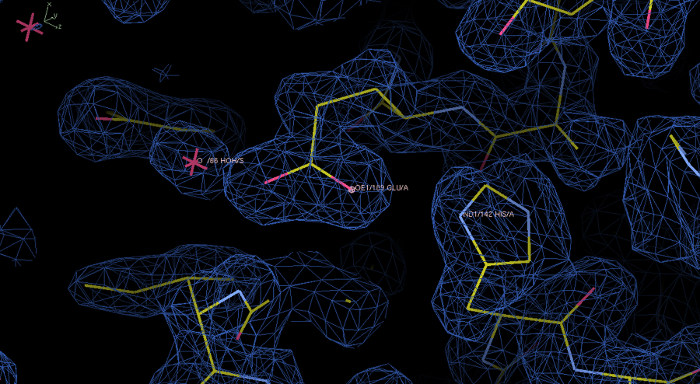
Рисунок 6: Экспериментальная карта плотности электронов и модель rPPEP-1 после того , какPhenix Autosol бежать. Электронная плотность в синем на уровне контура 1,0 сг отображается в программе Кут. В этой начальной карте плотность электронов красиво решена и модель построения в электронной плотности. Увеличения масштаба показывает остатки His142 и Glu189, а также молекулы воды. Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
6. Структура Определение с высоким разрешением с помощью молекулярной замены
ПРИМЕЧАНИЕ: Для получения высокого разрешения структурную информацию о rPPEP-1 дикорастущего набора данных собирают. Затем процедура молекулярного замещения с использованием программного обеспечения Phaser 26,27 ( в пределах пакета программного обеспечения Phenix) применяется с использованием структуры определяется с помощью цинк-SAD в качестве модели. Эта процедура может быть также использована позже при решении структуры rPPEP-1 в виде комплекса с малыми молекулами.
- Чтобы получить кристаллическую структуру с более высоким разрешением (в данном случае до 1,4 Å) обрабатывать родной набор данных с помощью программного обеспечения XDS (альтернативно iMosflm или HKL3000) в пространственной группе P2 1 2 1 2 1 (пространственная группа 19). Параметры элементарной ячейки должны быть вокруг, b, c (A) = 43,17, 71,77, 117,80 и α = β = γ (°) = 90. Это дает один HKL-файлов (файлов отражения).
- Проверьте файл CORRECT.LP. Использование данных до разрешения , в котором СС 1/2 составляет по меньшей мере 50%. Преобразование HKL-файл в файл отражения CCP4-формата (названный, например, native.mtz) с использованием XDSCONV создания R свободное подмножество 5%. Проверьте MTZ-файл для согласованности с программой mtzdmp инспектирующих параметры элементарной ячейки, пространственную группу и существование свободного подмножества R (метка FreeRflag).
- Подготовьте PDB-файл , содержащий модель из overall_best.pdb , определенной ранее , и удалить все молекулы воды и все лиганды (т.е., атом цинка). Также загрузить файл FASTA, содержащий аминокислотную последовательность, кристаллизованного белка. Запуск Phaser в Phenix с помощью файла отражения native.mtz. Поиск двух молекул на единицу асимметричной.
- После успешного структура раствора (TFZ-балл больше, чем 8; здесь 10.2) инспектировать модель (с именем native_phaser.1.pdb) и карту электронной плотности в Кут. Построить и уточнить структуру, запустив итерационных раундов Phenix Уточнить с 5 утонченности раундов каждый из которых использует файл native_phaser.1.pdb модели, native.mtz файл отражения и файл последовательности FASTA; и ручное построение модели в Кут.
- Проверка качества структурной модели с соответствующими инструментами в Кут.
Результаты
rPPEP-1 избыточно экспрессируется в нескольких штаммов E.coli, с самым высоким выходом в E.coli BL21 (DE3) Star (Рисунок 1С). После первого этапа NiNTA аффинной хроматографии 6xHis-метка может быть успешно отщепляется от большей части белка и во второй стадии NiNTA неперева...
Обсуждение
Рентгеновская кристаллография по- прежнему является самым быстрым и точным методом для определения трехмерных вблизи атомных структур разрешением белков 28. Тем не менее, это требует роста хорошо упорядоченных монокристаллов. Они часто трудно получить и кристаллическое состоян...
Раскрытие информации
The authors have nothing to disclose.
Благодарности
Мы благодарим сотрудников на пучкового X06DA в швейцарском Источник света, Поль-Шерера-института, Villigen, Швейцария для поддержки во время сбора синхротронное данных. Мы благодарны Monika Гомперт за отличную техническую поддержку. Проект был поддержан Кельнского университета и предоставить INST 216 / 682-1 FUGG из Научно-исследовательского совета Германии. Она является доктором наук стипендия от Международной высшей школы в области здравоохранения и развития болезни к CP признается. Исследований, приведших к этим результатам получил финансирование из Седьмой рамочной программы Европейского Сообщества (FP7 / 2007-2013) по гранту договору № 283570 (BioStruct-X).
Материалы
| Name | Company | Catalog Number | Comments |
| Genes / Vectors / cell strains | |||
| pET28a vector | Merck-Millipore | 69864 | Thrombin cleavable N-terminal His-tag |
| E. coli strain BL21 (DE3) Star | ThermoFisher Scientific | C601003 | RNase H deficient |
| Codon-optimized gene (for E. coli) of PPEP-1 (CD630_28300) | Geneart (Thermo Fisher Scientific) | custom | amino acids 27-220 |
| Name | Company | Catalog Number | Comments |
| Chemicals | |||
| Yeast extract | any | ||
| Tryptone | any | ||
| Antifoam B | Sigma-Aldrich | A5757 | aqueous-silicone emulsion |
| Agar | any | ||
| Kanamycin | any | ||
| IPTG | AppliChem | A1008 | |
| Tris-HCl | AppliChem | A1087 | Buffer grade |
| NaCl | any | Buffer grade | |
| DNaseI | AppliChem | A3778 | |
| Imidazole | AppliChem | A1073 | Buffer grade |
| Thrombin | Sigma-Aldrich | T4648 | |
| Ammonium phosphate dibasic | Sigma-Aldrich | 215996 | |
| Glycerol 100% | any | purest grade | |
| Sucrose | Sigma-Aldrich | 84097 | |
| Liquid nitrogen | any | for storage and cryocooling of crystals | |
| Name | Company | Catalog Number | Comments |
| Equipment (general) | |||
| Shaking incubator | any | providing temperatures of 20 °C - 37 °C | |
| Glassware | any | baffled Erlenmeyer flasks (50 ml - 2.8 L) | |
| Centrifuge for large culture volumes | any | centrifuge for processing volumes up to 12 L | |
| Sonicator Vibra-Cell VCX500 | Sonics | SO-VCX500 | or any other sonicator / cell disruptor |
| Ultracentrifuge | any | centrifuge providing speeds up to 150,000 x g | |
| NiNTA Superflow resin | Qiagen | ||
| Empty Glass Econo-Column | Bio-Rad | 7371007 | or any other empty glass or plastic column |
| Size exclusion chromatography column HiLoad Superdex 200 16/600 | GE Healthcare | 28989335 | |
| Chromatography system Äkta Purifier | GE Healthcare | 28406264 | or any other chromatography system |
| Dialysis tubing Spectra/Por 3 | Spectrum Labs | 132724 | |
| Dialysis tubing closures | Spectrum Labs | 132738 | |
| Ultrafiltration units (concentrators) 10,000 NWCO | any | ||
| UV-Vis spectrophotometer | any | ||
| Name | Company | Catalog Number | Comments |
| Equipment (crystallography) | |||
| Low volume pipette 0.1-10 µl | any | ||
| Positive displacement pipette Microman M10 | Gilson | F148501 | |
| Crystallization robot | any | ||
| 96-well crystallization plates TTP IQ with three protein wells | TTP | 4150-05810 | or any other 96-well crystallization plate |
| 24-well CombiClover Junior Plate | Jena Bioscience | EB-CJR | |
| Crystal Clear Sealing Tape | Hampton Research | HR3-511 | |
| Siliconized Glass Cover Slides | Hampton Research | HR3-225 | |
| Commercial crystallization screens: SaltRx, Index, PEG/Ion, Crystal | Hampton Research | diverse | |
| Commercial crystallization screens: Wizard, PACT++, JCSG++ | Jena Bioscience | diverse | |
| JBS Beads-for-Seeds | Jena Bioscience | CO-501 | |
| CrystalCap SPINE HT (nylon loops) | Hampton Research | diverse | loop sizes 0.025 mm - 0.5 mm |
| CrystalCap Vial | Hampton Research | HR4-904 | |
| Cryogenic Foam Dewar 800 ml | Hampton Research | HR4-673 | |
| Cryogenic Foam Dewar 2 L | Hampton Research | HR4-675 | |
| Vial Clamp, Straight | Hampton Research | HR4-670 | |
| CrystalWand Magnetic, Straight | Hampton Research | HR4-729 | |
| CryoCane 6 Vial Holder | Hampton Research | HR4-711 | |
| CryoSleeve | Hampton Research | HR4-708 | |
| CryoCane Color Coder - White | Hampton Research | HR4-713 | |
| Scalpel | any | ||
| Straight microforcep | any | for manipulation of sealing tape. etc. | |
| Acupuncture needle | any | e.g. from a pharmacy | |
| Stereo microscope | any | for inspection of crystallization plates and crystal mounting, magnification up to 160X |
Ссылки
- Bouza, E. Consequences of Clostridium difficile infection: understanding the healthcare burden. Clin Microbiol Infect. 18 (Suppl 6), 5-12 (2012).
- O'Connor, J. R., Johnson, S., Gerding, D. N. Clostridium difficile infection caused by the epidemic BI/NAP1/027 strain. Gastroenterology. 136 (6), 1913-1924 (2009).
- Mitchell, B. G., Gardner, A. Mortality and Clostridium difficile infection: a review. Antimicrob Resist Infect Control. 1 (1), (2012).
- George, W. L., Sutter, V. L., Finegold, S. M. Antimicrobial agent-induced diarrhea--a bacterial disease. J Infect Dis. 136 (6), 822-828 (1977).
- George, R. H., et al. Identification of Clostridium difficile as a cause of pseudomembranous colitis. Br Med J. 1 (6114), 695 (1978).
- Bartlett, J. G. Narrative review: the new epidemic of Clostridium difficile-associated enteric disease. Ann Intern Med. 145 (10), 758-764 (2006).
- Kelly, C. P., LaMont, J. T. Clostridium difficile--more difficult than ever. N Engl J Med. 359 (18), 1932-1940 (2008).
- Ünal, C. M., Steinert, M. Novel therapeutic strategies for Clostridium difficile infections. Expert Opin Ther Targets. 20 (3), 269-285 (2016).
- Kelly, C. P., et al. The Monoclonal Antibody, Bezlotoxumab Targeting C. difficile Toxin B Shows Efficacy in Preventing Recurrent C. difficile Infection (CDI) in Patients at High Risk of Recurrence or of CDI-Related Adverse Outcomes. Gastroenterology. 150 (4), S122 (2016).
- Tsutsumi, L. S., Owusu, Y. B., Hurdle, J. G., Sun, D. Progress in the discovery of treatments for C. difficile infection: A clinical and medicinal chemistry review. Curr Top Med Chem. 14 (1), 152-175 (2014).
- Hensbergen, P. J., et al. Clostridium difficile secreted Pro-Pro endopeptidase PPEP-1 (ZMP1/CD2830) modulates adhesion through cleavage of the collagen binding protein CD2831. FEBS Lett. 589 (24), 3952-3958 (2015).
- Cafardi, V., et al. Identification of a novel zinc metalloprotease through a global analysis of Clostridium difficile extracellular proteins. PLoS One. 8 (11), e81306 (2013).
- Hensbergen, P. J., et al. A novel secreted metalloprotease (CD2830) from Clostridium difficile cleaves specific proline sequences in LPXTG cell surface proteins. Mol Cell Proteomics. 13 (5), 1231-1244 (2014).
- Schacherl, M., Pichlo, C., Neundorf, I., Baumann, U. Structural Basis of Proline-Proline Peptide Bond Specificity of the Metalloprotease Zmp1 Implicated in Motility of Clostridium difficile. Structure. 23 (9), 1632-1642 (2015).
- Rawlings, N. D., Waller, M., Barrett, A. J., Bateman, A. MEROPS: the database of proteolytic enzymes, their substrates and inhibitors. Nucleic Acids Res. 42 (Release 10.0), D503-D509 (2014).
- Bergfors, T. Seeds to crystals. J Struct Biol. 142 (1), 66-76 (2003).
- Dauter, Z., Dauter, M., Dodson, E. Jolly SAD. Acta Crystallogr D Biol Crystallogr. 58 (Pt 3), 494-506 (2002).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 227 (5259), 680-685 (1970).
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Anal Biochem. 72, 248-254 (1976).
- Kabsch, W. XDS. Acta Crystallogr D Biol Crystallogr. 66 (Pt 2), 125-132 (2010).
- Adams, P. D., et al. PHENIX: a comprehensive Python-based system for macromolecular structure solution. Acta Crystallogr D Biol Crystallogr. 66, 213-221 (2010).
- Emsley, P., Lohkamp, B., Scott, W. G., Cowtan, K. Features and development of Coot. Acta Crystallogr D Biol Crystallogr. 66 (Pt 4), 486-501 (2010).
- . . The PyMOL Molecular Graphics System. , (2002).
- Pettersen, E. F., et al. UCSF Chimera--a visualization system for exploratory research and analysis. J Comput Chem. 25 (13), 1605-1612 (2004).
- Wang, B. C. Resolution of phase ambiguity in macromolecular crystallography. Methods Enzymol. 115, 90-112 (1985).
- McCoy, A. J., Grosse-Kunstleve, R. W., Adams, P. D., Winn, M. D., Storoni, L. C., Read, R. J. Phaser crystallographic software. J Appl Crystallogr. 40 (Pt 4), 658-674 (2007).
- Zwart, P. H., et al. Automated structure solution with the PHENIX suite. Methods Mol Biol. 426, 419-435 (2008).
- Zheng, H., Handing, K. B., Zimmerman, M. D., Shabalin, I. G., Almo, S. C., Minor, W. X-ray crystallography over the past decade for novel drug discovery - where are we heading next?. Expert Opin Drug Discov. 10 (9), 975-989 (2015).
- Rubino, J. T., et al. Structural characterization of zinc-bound Zmp1, a zinc-dependent metalloprotease secreted by Clostridium difficile. J Biol Inorg Chem. 21 (2), 185-196 (2016).
- Carson, M., Johnson, D. H., McDonald, H., Brouillette, C., Delucas, L. J. His-tag impact on structure. Acta Crystallogr D Biol Crystallogr. 63 (Pt 3), 295-301 (2007).
- Gasteiger, E., Walker, J. M., et al. Protein Identification and Analysis Tools on the ExPASy Server. The Proteomics Protocols Handbook. , 571-607 (2005).
- Dummler, A., Lawrence, A. M., de Marco, A. Simplified screening for the detection of soluble fusion constructs expressed in E. coli using a modular set of vectors. Microb Cell Fact. 4, 34 (2005).
- Stura, E. A., Wilson, I. A. Applications of the streak seeding technique in protein crystallization. J Crys Growth. 110 (1), 270-282 (1991).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены