Method Article
Erzeugung von nativen, unmarkierten Huntingtin Exon1 Monomer und Fibrillen mit einem SUMO-Fusion-Strategie
In diesem Artikel
Zusammenfassung
Hier präsentieren wir Ihnen eine robuste und optimierte Protokoll zur Herstellung von Milligramm-Mengen von native, Tag-freie Monomere und Fibrillen von der exon1 des Huntingtin-Proteins (Httex1), basierend auf die vorübergehende Verschmelzung von kleinen Ubiquitin Verwandte Modifikator (SUMO).
Zusammenfassung
Chorea Huntington (HD) ist eine vererbte tödliche Neurodegenerative Erkrankung, die durch eine CAG-Erweiterung (≥36) in der ersten Exon des Huntington-Gens, wodurch des Ausdrucks des Huntingtin-Proteins (Htt) oder N-terminale Fragmente davon mit einer erweiterten Trinukleotiderkrankungen ( PolyQ) Strecke.
Die exon1 des Huntingtin-Proteins (Httex1) ist die kleinste Htt-Fragment, das viele der Funktionen von HD in zellulären rekapituliert und Tier-Modelle und ist eines der am häufigsten untersuchten Fragmente des Htt. Die geringe Größe des Httex1 macht es experimentell zugänglicher, biophysikalische Charakterisierung mittels Standard- und hochauflösenden Techniken im Vergleich zu längeren Fragmente oder Full-Length Htt. Allerdings hat die hohe Aggregation-Neigung der Mutant Httex1 (mHttex1) mit erhöhten PolyQ Inhalt (≥42) machte es schwierig, entwickeln effiziente Ausdruck und Reinigungssysteme, diese Proteine in ausreichender Menge zu produzieren und ihnen zugänglich zu machen Wissenschaftler aus verschiedenen Disziplinen ohne den Einsatz von Fusionsproteinen oder andere Strategien, die die native Sequenz des Proteins verändern. Wir stellen Ihnen hier eine robuste und optimierte Methode zur Herstellung von Milligramm-Mengen von einheimischem, Tag-freie Httex1 basierend auf die vorübergehende Verschmelzung von kleinen Ubiquitin Verwandte Modifikator (SUMO). Einfachheit und Effizienz der Strategie beseitigt die Notwendigkeit nicht-Native Sequenzen des Httex1, so macht dieses Protein leichter zugänglich für Forscher und verbessert die Reproduzierbarkeit der Experimente in verschiedenen Labors verwendet. Wir glauben, dass diese Fortschritte erleichtert auch zukünftige Studien zur Aufklärung der Struktur-Funktions-Beziehung der Htt und entwickeln neuartige Diagnose-Tools sowie Therapien zur Behandlung oder das Fortschreiten der Huntington-Krankheit.
Einleitung
Htt ist ein 348-kDa-Protein und hat in mehreren physiologischen Funktionen1verwickelt worden. Wenn Htt eine erweiterte PolyQ-Region von mehr als 36-Rückständen in der N-Terminus enthält, verursacht es HD2,3. HD-Pathologie ist geprägt von zellulären Einschlüsse im Striatum und Kortex, die zu neuronalen Tod und Atrophie der betroffenen Gewebe4,5führt. Mehrere N-terminale Htt-Fragmente, die den PolyQ wiederholen Trakt enthalten in Post-Mortem Gehirn von Huntington-Patienten erkannt wurden und werden gedacht, um durch proteolytische Verarbeitung von Huntingtin Protein6erzeugt werden. Neuere Studien legen nahe, dass Httex1 auch durch aberrante mRNA Spleißen gebildet werden könnte. Httex1 enthält die pathologische PolyQ-Mutation und seine Überexpression bei Tieren kann viele der wichtigsten Features von HD7, damit eine mögliche zentrale Rolle dieses Fragments in HD Pathologie und Krankheit Fortschreiten6Hervorhebung rekapitulieren, 8,9.
Durch die hohe Aggregation Neigung der Mutant Httex1 (mHttex1) mit erweiterten PolyQ-Darm-Trakt, die Mehrheit der bestehenden Expressionssysteme basieren auf die vorübergehende Verschmelzung von Httex1 Proteinen (z. B. Glutathion-S-Transferase (GST), Thioredoxin (TRX) oder Maltose-Binding-Protein (MBP) und/oder Peptide (Poly-Histidin), das seinen Ausdruck, Stabilität, Reinigung und/oder Löslichkeit10,11,12,13,14 differentiell zu verbessern ,15,16,17,18,19,20,21,22,23 ,24,25,26,27,28. Der Fusion-Partner ist im Zusammenhang mit Httex1 mit einer kurzen Sequenz mit einer spaltstelle für Proteasen wie Trypsin, Tabacco Ätzen Virus (TEV) Protease oder PreScission, um die Spaltung und die Freigabe der Httex1 vor der Einleitung der Aggregation zu ermöglichen oder Reinigung. Mängel dieser Methoden umfassen die Möglichkeit, zusätzliche Rückstände aufgrund nicht spurlos Spaltung und die Schaffung von abgeschnittenen Fragmente durch Miscleavage innerhalb der Sequenz des Httex1, neben der Heterogenität aufgrund unvollständiger Spaltung ( Siehe Vieweg Et Al. für tiefer gehende Diskussion über die Vorzüge und Grenzen dieses Ansatzes)10. Um diese Einschränkungen zu beheben, haben wir vor kurzem eine Ausdruck Strategie entwickelt so dass die Generation der Tag-freie native Httex1 zum ersten Mal durch den Einsatz einer transienten N-terminale Verschmelzung von Synechocystis SP. (Ssp) DnaB Intein bis Httex110. Während die Intein Spaltung spurlos und spezifische ist und mg Menge von Proteinen liefert, es leidet noch zwei Nachteile, die den Ertrag zu reduzieren konnte: nämlich, vorzeitiger Spaltung Intein die auftreten können, während der Ausdruck und die Tatsache, das Dekolleté über Eintritt mehrere Stunden, die zum Verlust des Proteins durch Aggregation, vor allem für Httex1 mit erweiterten PolyQ Wiederholungen führen können.
Um diese Einschränkungen zu beheben und unsere Strategie für die Herstellung von Native zu verfeinern, Tag-freie Httex1, entwickelten wir ein neues System Ausdruck auf die vorübergehende Verschmelzung von SUMO, genauer gesagt die Hefe Homolog Smt3, Httex1. Die Anwendung des SUMO-Systems für die Produktion von rekombinanten Proteinen erschien erstmals im Jahr 200429, wo eine erhöhte Rate von Ausdruck und Löslichkeit von SUMO Schmelzverfahren Protein nachgewiesen wurde. Das SUMO-Tag kann durch Ubiquitin wie Protein-spezifische Protease 1 (ULP1), die erfordert keiner Anerkennung-Website, sondern erkennt die Tertiärstruktur des SUMO und praktisch verhindert, dass Miscleavage30abgespalten werden. Darüber hinaus ULP1-vermittelte Spaltung ist schnell und spurlos und nicht zusätzliche Rückstände hinterlassen. Die vorzeitige Spaltung des Fusion-Tags ist als mit der autokatalytischen Intein10, beobachtet von der Forderung nach einer externen Protease vollständig vermieden. Während die SUMO-Strategie für rekombinantes Protein Produktion31,32,33heute weit verbreitet ist, zeigen wir in diesem Papier, dass es besonders nützlich zur Erzeugung von einer intrinsisch ungeordneten, Zusammenfassung-anfällig, Amyloidogenic Protein wie Httex1. Wir glauben, dass die Einfachheit, Effizienz und Robustheit unserer SUMO-Fusion-basierte Methode native, Tag-free Httex1 besser zugänglich für Forscher aus unterschiedlichen Disziplinen und die Notwendigkeit beseitigen, gebietsfremden Sequenzen von Httex1 in Vitro zu verwenden . Dies ist ein bedeutender Fortschritt, der zukünftige Studien zur Aufklärung der Struktur-Funktions-Beziehung von Httex1 erleichtert.
Das Protokoll beschreibt die Reinigung des Httex1 von 12 L der bakteriellen Kultur, aber das Protokoll könnte leicht für Produktionen von kleineren oder größeren Maßstab angepasst werden. Das Protokoll beschreibt die Herstellung von Wildtyp Httex1 (wtHttex1) mit einem PolyQ Rapportlänge unter (23Q) und Mutant Httex1 (mHttex1) mit einer PolyQ Rapportlänge oben (43Q) die pathogene Schwelle (36Q).
Protokoll
1. Ausdruck der rekombinanten Httex1 23Q und 43Q
- Bereiten Sie den erforderlichen Puffer und Lösungen. Bereiten Sie 1000 x Stammlösung Ampicillin (AMP, 100 mg/mL), Filter (0,2 µm), aliquoten und Speicher bei-20 ° C. Bereiten Sie Lysogeny Brühe (LB) Medium (25 g LB Miller pro 1 L H2O), Autoklaven. Vorbereitung der Stammlösung 1 M Isopropyl-ß-D-1thiogalactopyranoside (IPTG), Filter (0,2 µm), aliquoten und Shop bei-20 ° C.
- Verwandeln Sie chemisch kompetenter E. Coli B ER 2566 mit einem pTWIN1-Vektor mit menschlichen Httex1 verschmolzen zu einem N-terminalen His6-SUMO-Tag mit der Hitze Schock Methode34.
Hinweis: Die E. Coli BL21 DE3-Stamm wurde auch verwendet. Allerdings war in diesem Fall eine erhöhte Menge an Trunkierungen beobachtet. - Impfen Sie 200 mL LB-Medium mit 1 x AMP in einem 1 L konischen Kolben durch eine einzige Kolonie von der Agarplatte mit einer sterilen PIPETTENSPITZE hinzufügen. Inkubieren Sie die Kultur bei 30 ° C und 180 u/min für 20 h (über Nacht) in einem bakteriellen Inkubator.
- Nehmen Sie eine 1 mL Probe der Kultur mit einer sterilen Pipette. Messung die optische Dichte bei 600 nm (OD600) der Probe eine Einweg-Plastik-Küvette mit einem Photometer (Achtung die Messung im Bereich zwischen 0,1 und 1, verdünnen mit LB-Medium bei Bedarf). Berechnen Sie die Menge der Vorkultur, die einen Start OD600 von 0,05 in einer 3 L Kultur führen (mit einer Vorkultur OD600 = 3, die 50 mL bedeuten würde).
- Vier Kulturen impfen (je 3 L LB-Medium mit 1 x AMP in eine 5 L Flasche), indem man den berechneten Betrag der Vorkultur mit einer sterilen Pipette. Inkubieren Sie die Kulturen bei 37 ° C und 180 u/min in einem bakteriellen Inkubator.
- Nehmen Sie alle 30 Minuten eine 1 mL Probe der Kultur mit einer sterilen Pipette. Messen Sie die OD600 der Probe eine Einweg-Plastik-Küvette mit einem Photometer. OD600 0,1 (in der Regel nach 1-2 h) erreicht hat, stellen Sie die Temperatur des bakteriellen Inkubators auf 14 ° C und die Inkubation bei der Kühlung weiter. Nehmen Sie alle 30 Minuten eine 1 mL Probe der Kultur mit einer sterilen Pipette. Messen Sie die OD600 der Probe eine Einweg-Plastik-Küvette mit einem Photometer.
Hinweis: Die Zeit zum Abkühlen der Kulturen variieren mit dem Inkubator verwendet, damit die Kühlung beginnen möglicherweise angepasst werden, je nach Art des Inkubators verwendet können. Ändern der Temperaturgradient sollte jedoch nur einen kleinen Einfluss auf den Ertrag haben, wie die SUMO-Fusionsprotein scheint recht stabil zu sein. - Wenn OD600 0,3-0,4 (in der Regel nach 1-2 h) erreicht hat, nehmen Sie eine Pre-Induktion-Probe der Kultur für die SDS-PAGE-Analyse von der Überexpression. Berechnen Sie die Größe der Stichprobe, die eine vergleichbare Menge von Zellen gibt und ein gutes Signal auf Coomassie gefärbt SDS-PAGE: gut Gel für ein 10: Volumen = 0,2 mL/OD600; nehmen Sie die Hälfte für ein 15 gut Gel.
- Für eine Bakterienkultur mit einer OD600= 0,4, nehmen Sie 500 µL. Nehmen Sie das berechnete Volumen der Bakterienkultur mit einer sterilen Pipette. Spin-down der Probe (18000 x g, 4 ° C, 2 min) und den überstand verwerfen. Halten Sie die Pellets bei-20 ° C bis zum Gebrauch für die Analyse (Schritt 1.11).
- Proteinexpression durch pipettieren 1,2 mL einer 1 M IPTG Vorratslösung zu jeder 3 L Kulturlösung induzieren (Endkonzentration 0,4 mM). Weiter Inkubation der Kultur bei 14 ° C 16 h (über Nacht).
Hinweis: Die Temperatur wird in der Regel ~ 20 ° C durch die Zeit erreicht haben, die IPTG, abhängig von der Leistungsfähigkeit des Inkubators hinzugefügt wird. - Nehmen Sie eine Post-Induktion-Probe der Kultur für die SDS-PAGE-Analyse der Überexpression, nach dem Verfahren in Schritt 1.7 beschrieben.
- Ernte der Zellen durch Zentrifugation in 1 L-Röhren (3993 x g, 4 ° C, 10 min). Verwerfen Sie den überstand, halten Sie die Zelle Pellet auf Eis und fahren Sie direkt mit der Reinigung.
- Analysieren Sie die Überexpression von SDS-PAGE35,36. Aufzuwirbeln Sie die Pre- und Post-Induktion Proben in 20 µL Puffer und 20 µL 2 x laden Farbstoff. Erwärmen Sie die Proben für 5 min bei 95 ° C in einem Block Hitze und laden Sie 20 µL auf einem 15 % Gel noch heiß. Führen Sie das Gel für 90 min bei 180 V. Fleck das Gel mit Coomassie färben gemäß den Anweisungen des Herstellers. Vergleichen Sie die Ergebnisse mit repräsentative Ergebnisse in Abbildung 1C.
Hinweis: Das Protokoll kann hier gestoppt werden, die Zelle Pellet eingefroren und bei-80 ° C für mehrere Wochen gespeichert werden kann. Um optimale Ergebnisse zu erzielen empfiehlt es sich, verwenden Sie das frische bakterielle Pellet und Einfrieren zu vermeiden. Frost-Tau-wechseln könnte zur Lyse der Zellen und Abbau des Httex1 führen. Dies könnte den Ertrag und die Qualität des Proteins reduzieren.
(2) Zell-Lyse und Reinigung von seiner6-SUMO-Httex1-Fusionsprotein immobilisiert durch Metall Affinitätschromatographie (IMAC)
- Bereiten Sie 2 L Puffer A in einer Glasflasche (50 mM Tris (Hydroxymethyl)-Aminomethan (Tris), 500 mM NaCl, 15 mM imidazol). Bereiten Sie 1 L Puffer B (50 mM Tris, 500 mM NaCl, 500 mM Imidazol pH-Wert 7,4) in einer Glasflasche. Passen Sie nach der Auflösung der Salze den pH-Wert mit 10 N HCl und Filtern Sie die Lösungen in frische Flaschen mit einer Flasche Top Filter (0,65 µm). Bereiten Sie eine 1000-X phenylmethylsulfonyl-Fluorid (PMSF, 0,3 M) Lager Lösung, aliquoten 100 µL und Store bei-20 ° C.
Hinweis: Das Protokoll soll ermöglichen, Abschluss aller Schritte von Lyse der bakteriellen Pellets umgekehrt Phase Hochleistungs-Flüssigkeitschromatographie (RP-HPLC) Reinigung und Lyophilisation innerhalb von 8-9 h. Aggregation und Proteolyse zu begrenzen, empfiehlt es sich, schnell zu arbeiten, ohne Pause, und führen Sie alle Schritte bei 4 ° C oder auf Eis. - 100 mL vorgekühlt Puffer A. Add das bakterielle Pellet in den Puffer 100 µL der Stammlösung PMSF und fünf Tabletten (1 pro 30 mL Endvolumen) der Protease-Inhibitor hinzu und homogenisieren Sie die Aussetzung zu, durch Rühren mit einer magnetischen Stir Bar und nach oben und unten wi pipettieren ten eine sterile 10-mL-Pipette (~ 30 min).
- Teilen Sie die Bakterien Aussetzung in Aliquote von 40 mL in 50 mL Einweg-Kunststoff-Rohre. Beschallen Sie jedes aliquoten in einem Wasser/Eis-Batch für die Lyse der Zellen (70 % Amplitude, total beschallungsdauer 5 min Abständen von 30 s Beschallung, 30 s Pause).
Hinweis: Es ist wichtig, dass die Probe nicht bei der Beschallung aufheizt. Es empfiehlt sich, etwas Wasser hinzufügen, um das Eisbad Wärmeableitung bei Beschallung zu verbessern. Das Ultraschall-Verfahren möglicherweise angepasst werden, wenn ein anderes Instrument verwendet wird. Anderen Lyse Methoden wie ein French-Press oder ein Microfluidizer sollte gut funktionieren und könnte von Vorteil sein, Erwärmung der Probe und Protein Aggregation zu vermeiden. Diese Geräte waren nicht in unserem Labor und wir erhalten gute Ergebnisse mit unserem Beschallung-Protokoll. - Nehmen Sie eine Probe von 50 µL lysate für Sodium Dodecyl Sulfat Polyacrylamid-Gel-Elektrophorese (SDS-PAGE) Analyse. Zentrifugieren der Probe (18000 x g, 4 ° C, 2 min) und die lösliche Fraktion im neuen Reagenzglas pipette. Aufschwemmen Sie den unlöslichen Anteil in 50 µL Puffer A mit einer Pipette. Halten Sie die Proben auf Eis bis zur SDS-PAGE-Analyse (Schritt 2.6).
- Klären Sie die lysate durch Zentrifugieren (39191 x g, 4 ° C, 60 min.).
- Während der Zentrifugationsschritt analysieren Sie die Zelle Lysis Schritt für SDS-PAGE. Fügen Sie 50 µL 2 x Farbstoff auf den löslichen und unlöslichen Bruchteil der lysate bzw. laden. Für 5 min bei 95 ° C erhitzen und 2 µL auf einem 15 % Gel noch heiß zu laden. Führen Sie das Gel für 90 min bei 180 V. Fleck das Gel mit Coomassie färben gemäß den Anweisungen des Herstellers. Vergleichen Sie die Ergebnisse mit repräsentative Ergebnisse in Abbildung 1C.
- Den überstand (0,45 µm, Spritze Filter) zu filtern. Nehmen Sie eine Probe von 20 µL des gefilterten Überstands für SDS-PAGE-Analyse (Schritt 2.11).
Hinweis: In der Regel ergibt sich ein Volumen von 90 bis 100 mL geklärte und gefilterten überstand. 3 Spritze-Filter sind in der Regel ausreichend. Wenn die Filtration umständlich ist, versuchen Sie, die Zentrifugation Geschwindigkeit und/oder Zeit zu erhöhen. - His6-SUMO Httex1 Schmelzverfahren Protein aus der geklärte bakterielle lysate von immobilisierten Metall Affinitätschromatographie (IMAC) auf eine schnelle Performance Flüssigkeitschromatographie (FPLC) System bei 4 ° C37zu isolieren.
- Füllen die geklärte lysate zu einem Superloop und Ladung auf der Ni-NTA-Spalte (abgestreift, gereinigt und neu geladen nach dem Handbuch des Herstellers, eine vorherige leere laufen wird empfohlen) auf 2 mL/min Pass 10 Spalte Volumen (CV, 200 mL) des Puffers A bei 10 mL/min waschen heraus die ungebundene Proteine.
- Eluieren Fusionsprotein mit 2.5 CV (50 mL) von 100 % der Puffer B 2 mL/min. eine Bruchteil Größe 50 mL für Be- und Waschen und 5 mL für Elution verwenden. Vergleichen Sie die Ergebnisse mit repräsentative Ergebnisse in Abbildung 1D.
- Nehmen Sie eine Probe der einzelnen Fraktionen für die SDS-PAGE-Analyse (20 µL) und bündeln Sie die Fraktionen mit der Schmelzverfahren Protein nach dem Höhepunkt der IMAC Chromatogramm zu. Hinzufügen (2 s, 3 s) - 1,4 - Bis (Sulfanyl) Butan-2,3-Diol (DTT) und L-Cystein (Endkonzentration 100 mM) als Pulver und lösen sich durch das Rohr vorsichtig umdrehen.
Hinweis: Nach unserer Erfahrung ist die Reinheit des Fusionsproteins in den verschiedenen Fraktionen vergleichbar. Als Vorsichtsmaßnahme sollten die Fraktionen des gereinigten Fusionsproteins schnell nach IMAC, Aggregation von hochkonzentrierten Brüche zu verhindern gebündelt werden. Darüber hinaus empfiehlt es sich, gehen direkt auf die Spaltung der SUMO-Tag und HPLC-Reinigung. Falls erforderlich, kann das Protokoll hier gestoppt werden. Die verdünnte Lösung des Fusionsproteins wurde in flüssigem Stickstoff eingefroren, bei-80 ° C gelagert und nach dem Auftauen ohne einen erheblichen Rückgang der Rendite gereinigt. Lagerung der verdünnten Lösung des Fusionsproteins bei 4 ° C für 24 h gibt auch ähnliche Ergebnisse. - Analysieren Sie den IMAC durch SDS-PAGE. Fügen Sie 20 µL Farbstoff zu jeder Probe zu laden. Laden Sie 2 µL der Rohmaterial (2,7), der ungebundene Anteil, Waschen und jede Fraktion der Elution Spitze auf einem 15 % Gel. Führen Sie das Gel für 90 min bei 180 V. Fleck das Gel mit Coomassie färben gemäß den Anweisungen des Herstellers. Vergleichen Sie die Ergebnisse mit repräsentative Ergebnisse in Abbildung 1D.
3. Spaltung seiner6-SUMO-Tag und HPLC-Reinigung
Achtung: Trifluoroacetic Säure (TFA) ist eine flüchtige Flüssigkeit und können schwere Verbrennungen Ursache also mit Vorsicht handhaben. Alle Handhabung unter einem Abzug durchzuführen und geeigneten persönlichen Schutzausrüstung (d.h., Einweg-Nitril-Handschuhe, Schutzbrille und einen Laborkittel) zu tragen.
- Fügen Sie 5 mL von TFA mit einer kunststoffspritze in eine 5 L Flasche hinzu 5 L Wasser (Lösungsmittel A: H2O, 0,1 % (TFA). Hinzufügen einer 2,5 L Flasche Acetonitril 2,5 mL von TFA mit einer kunststoffspritze (Lösungsmittel B: Acetonitril, 0,1 % TFA).
- Bereiten Sie das HPLC-System wie vom Hersteller empfohlen. Führen Sie eine leere ausführen, um eine saubere Spalte zu gewährleisten.
- Nehmen Sie eine Probe von 100 µL des Fusionsproteins vor der Zugabe von ULP1, die Spaltung Reaktion UPLC (Schritt 3.5) zu überwachen.
- Eine neue 50 mL Tube 20 mL des Fusionsproteins übermitteln und 0,4 mL Stammlösung ULP1 hinzugeben, Inkubation auf Eis. Halten Sie die restlichen Fusionsproteins auf Eis.
Hinweis: Das sein-tagged katalytische Fragment 403-621 der Ubiquitin-artigen-spezifische Protease 1 (hier als "ULP1" bezeichnet) wurde zur SUMO Tag festhalten. Das Schmelzverfahren Protein ist stabiler als die Httex1 gespalten. Es wird empfohlen, nicht auf das SUMO-Tag für die gesamte Partie zu Spalten. Fahren Sie stattdessen mit Aliquote eine Größe, die direkt und vollständig auf die HPLC-Säule angewendet werden können. - Nehmen Sie alle 10 Minuten eine Probe von 100 µL der Spaltung Reaktion zur Überwachung des Fortschritts von Ultra-Performance liquid Chromatography (UPLC). Zentrifugieren Sie Proben (18000 u/min, 2 min, 4 ° C) und 2 µL des Überstands von UPLC zu analysieren (Steigung von 10 % bis 90 % Lösungsmittel B in A für 0,25 bis 3 min, 10 % B für 1 min, befolgen Sie die Anweisungen des Herstellers für die Nutzung des Instruments). Vergleichen Sie die für die Probe vor der Zugabe des ULP1 und die Proben nach erhaltenen Chromatogramme. Vergleichen Sie die Ergebnisse mit repräsentative Ergebnisse in Abbildung 2B.
- Nach Abschluss der SUMO-Spaltung (der Gipfel des Fusionsproteins in der UPLC-Chromatogramm verschwunden und ist komplett umgestellt, um die neu erscheinenden SUMO und Httex1 Peak), Filtern Sie die Probe mit einer Spritze Filter (0,22 µm).
Hinweis: Die SUMO-Spaltung ist in der Regel sehr schnell (10-20 min bei 4 ° C) UPLC Analyse mit einer Laufzeit von 4 min ist daher ein wertvolles Werkzeug, um die Reaktion zu überwachen. Filtern der Probe vor der HPLC ist Reinigung vor allem eine vorbeugende Maßnahme, um die Lebensdauer der Säule zu erhöhen. Die Probe sollte nicht trüben drehen. - Reinigen Sie die gefilterten Probe mittels RP-HPLC (Steigung von 25-35 % Lösungsmittel B im Lösungsmittel A, über 40 min auf 15 mL/min (0-10 min: 5 %; 10-12,5 min: 5 bis 25 %; 12,5-52,5 min: 25 bis 35 %; 52,5-57,5 min 35 bis 95 %; beziehen sich auf die Anweisungen des Herstellers für die Nutzung des Instruments). Vergleichen Sie die Ergebnisse mit repräsentative Ergebnisse in Abbildung 2C.
Hinweis: Httex1 und seine6-SUMO separate gut mittels RP-HPLC. Jedoch können kleine Mengen von abgeschnittenen Httex1 am Anfang und Ende des Gipfels sein. Sammeln Sie kleine Fraktionen um die maximale Menge des reinen Materials zu erhalten.
Achtung: Verwenden Sie die entsprechende Schutzausrüstung (d.h., Laborkittel, isolierte Handschuhe und einen Gesichtsschutz) beim Umgang mit kryogenen Flüssigkeiten. - Analysieren der HPLC-Fraktionen durch Massenspektrometrie der Electro-Spray-Ionisation (ESI-MS, Autosampler, 10 µL zu injizieren, fließen 0,6 mL/min, Lösungsmittel: 20 % B in A, keine Spalte beziehen sich auf die Anweisungen des Herstellers für die Nutzung des Instruments) und UPLC (Steigung von 10 % bis 90 % Lösungsmittel B in A für 0,25 bis 3 min, 10 % B für 1 min, lesen Sie die Anweisungen des Herstellers für die Nutzung des Instruments). Bruchteile von ähnlichen Reinheit in 50 mL-Kunststoff-Rohre zu bündeln und Einfrieren in flüssigem Stickstoff lyophilize. Wiegen und übertragen die lyophilisierte Protein in 2 mL-Kunststoff-Rohre und Lagerung bei-20 ° C.
- Das gereinigte Material von UPLC, ESI-MS und SDS-PAGE zu charakterisieren. 100 µg des lyophilisierten Httex1 in 8 µL ordentlich TFA in einem 1,5 mL Röhrchen auflösen und 20 min bei Raumtemperatur im verschlossenen Reagenzglas inkubieren. Verdunsten Sie sorgfältig die TFA unter einer Abzugshaube mit einem Strom von Stickstoff oder Argon. Verwenden Sie Niederdruck Stickstoff/Argon, um Verlust der Probe zu vermeiden.
- Das Protein in 100 µL von H2O. analysieren 2 µL von UPLC und 5 µL von ESI-MS wie in Schritt 3.8 auflösen. Mix-20 µL Proteinlösung mit 20 µL 2 x laden Farbstoff.
- Mengen von 1 µg bis 10 µg durch SDS-PAGE zu analysieren. Führen Sie das Gel für 90 min bei 180 V. Fleck das Gel mit Coomassie färben gemäß den Anweisungen des Herstellers. Vergleichen Sie die Ergebnisse mit repräsentative Ergebnisse in Abbildung 2D.
4. Disaggregation und Resolubilization von Httex1 Proteinen
Achtung: TFA ist eine flüchtige Flüssigkeit und können schwere Verbrennungen Ursache also mit Vorsicht handhaben. Alle Handhabung unter einem Abzug durchzuführen und geeigneten persönlichen Schutzausrüstung (z.B. Einweg-Nitril-Handschuhe, Schutzbrille und einen Laborkittel) zu tragen.
- Bereiten Sie 10 mL Dulbeccos Phosphat gepufferte Kochsalzlösung (DPBS) (137 mM NaCl, 2,7 mM KCl, 10 mM Na2HPO4, 2 mM KH2PO4, pH 7,4) aus dem vorgemischten Pulver in ein 50 mL-Tube. Filtern Sie die Lösung des DPBS durch einen 0,2 µm-Filter vor jedem Gebrauch.
- Lösen sich 150 µg des lyophilisierten Httex1 in 12 µL ordentlich TFA in einem 1,5 mL Röhrchen und 20 min bei Raumtemperatur im verschlossenen Reagenzglas inkubieren. Verdunsten Sie sorgfältig die TFA unter einer Abzugshaube mit einem Strom von Stickstoff oder Argon. Verwenden Sie Unterdruck von Stickstoff/Argon, um die Probe38zu vermeiden.
Hinweis: Verwenden Sie In der Regel 4 µL TFA aufzulösen und disaggregieren 50 µg Protein. Bei dieser Vorgehensweise wird einen Film des Proteins erstellt, auf der Innenseite Wände des reagenzglases. Um sofortige Aggregation von Httex1 in den folgenden Schritten zu verhindern, arbeiten mit vorgekühlten Puffer, halten das Protein immer auf Eis und vermeiden hohe Konzentrationen. - Lösen Sie aufgeschlüsselte Proteins in 1 mL vorgekühlte DPBS auf und passen Sie den pH-Wert auf 7,2-7,4 mit 1 M NaOH. Filtern Sie die Proteinlösung durch einen 100 kDa zentrifugale Filter in 1,5 mL Plastikröhrchen (20000 X g, 4 ° C, 20 Minuten).
Hinweis: Die berechnete theoretische Konzentration der Httex1 ist höher als die gewünschte Endkonzentration, um möglichen Verlust zu berücksichtigen. Der Filtrationsschritt ist notwendig, um Aggregate zu entfernen, die während der Auflösung des Proteins gebildet haben könnte. - Bestimmen Sie die Konzentration des Httex1 mit einer UPLC Kalibrationskurve basierend auf Aminosäureanalyse (Erkennung bei λ214) und Aminosäureanalyse zur Validierung der Konzentration102 µg des Proteins an. Berechnen Sie die Menge des DPBS, das muss hinzugefügt werden, um eine Konzentration von 3 µM zu erhalten Httex1.
- Verdünnen Sie das Protein bis 3 µM indem Sie das Reagenzglas den berechneten Betrag des DPBS hinzufügen. Halten Sie das Rohr auf Eis, bis die Einleitung des Protokolls Aggregation.
Hinweis: Httex1 43Q nicht in Lösung gespeichert werden sollte. Bereiten Sie immer ein frisches Proteinlösung basierend auf dem Protokoll oben. Httex1 Proteine sind am besten als lyophilisierte Pulver bei-20 ° c gelagert.
5. Überwachung der Aggregation Kinetik von Httex1 43Q mit UPLC und kreisförmigen Dichroismus (CD)-Spektroskopie und Charakterisierung der Aggregate durch Transmission Electron Microscopy (TEM)
- TEM Uranyl Formiat Lösung als bisher gemeldeten39vorbereiten.
- Die Aggregation von Httex1 zu initiieren 43Q durch Inkubation eine 3 µM-Lösung in DPBS bei 37 ° C (1 mL der Lösung vorbereitet, wie oben beschrieben in die Disaggregation Protokoll verwenden).
Hinweis: Die Aggregation von Httex1 kann bei höheren Konzentrationen je nach den Bedürfnissen und Zielen des Experiments durchgeführt werden. - Die Menge des löslichen Proteins mit UPLC zu angegebenen Zeitpunkten (bei 0, 1, 2, 4, 6, 8, 12, 24, 48 und 120 h) zu quantifizieren. Um dies zu erreichen, eine Aliquote von 35 µL herausnehmen Sie und die unlösliche Aggregate durch Zentrifugieren (20000 x g, 4 ° C, 20 min). Injizieren Sie in der UPLC 4 µL des Überstands. Den Anteil an löslichen Monomer basierend auf die Änderung von Peak mit dem Instrument Software 40zu berechnen. Vergleichen Sie die Ergebnisse mit repräsentative Ergebnisse in Abbildung 3A.
- Charakterisieren Sie die Änderungen in der Sekundärstruktur mit CD-Spektroskopie auf 0 und 48 h. Nehmen Sie eine Aliquote von 100 µL und messen die Ellipticity (1 mm Quarz Küvette, 195 nm bis 250 nm, 20 ° C, Daten weist jedes 0,2 nm Geschwindigkeit 10 nm/min, digitale Integration Zeit 2 s, Bandbreite von 1,0 nm). 6 Spektren der Probe und Durchschnitt zu erwerben und glatt mit einem binomial-Filter mit Faltung Breite von 99. Plotten Sie die Spektren als mittlere Rückstände molaren Ellipticity (θMRE)41. Vergleichen Sie die Ergebnisse mit repräsentativen Ergebnisse in Abbildung 3B.
- Die strukturellen und morphologischen Eigenschaften der Aggregate durch TEM zu charakterisieren. Setzen Sie 3 µL der Proteinlösung auf einem Formvar/Kohlenstoff-beschichtete 200-Mesh, Glut entladen Kupfer Raster für 1 min. Waschen zweimal mit 15 μl Wasser, einmal mit 15 μl von 0,7 % (w/V) Uranyl Formiat und Fleck für 30 s mit 15 μl von 0,7 % w/V Uranyl Formiat. Führen Sie eine TEM-Analyse der Netze. Vergleichen Sie die Ergebnisse mit repräsentativen Ergebnisse in Abbildung 3C.
Ergebnisse
Httex1 drückt sich in E. Coli mit einer N-terminalen seiner6-SUMO-Tag. Die repräsentativen Ergebnisse des Ausdrucks und Reinigung des Fusionsproteins sind in Abbildung 1zusammengefasst. Die Reihenfolge der Httex1 besteht aus Rückständen 2-90 Htt und beginnt mit Ala2, weil Met1 vollständig gespalten in Vivo42ist. Die Nummerierung der Aminosäuren bezieht sich auf die 23Q-Variante, die komplette Sequenz des Fusionsproteins ausgedrückt ist in Abbildung 1Agezeigt. Die Plasmide werden an Addgene in naher Zukunft, mit der Community geteilt werden hinterlegt. Abbildung 1Bzeigt eine schematische Darstellung des Plasmids und ausdrückliche Fusionsproteins. His6-SUMO Httex1 drückt auf einem mittleren Niveau (Abbildung 1C) und die meisten des Fusionsproteins vorliegt in der löslichen Fraktion nach Lyse, sowohl für die 23Q und die 43Q Variante. Das Schmelzverfahren Protein wandert höher als erwartet, basierend auf das Molekulargewicht. Dies ist zum Teil auf die starke Falte des SUMO sondern vor allem durch die ungewöhnliche Reihenfolge Zusammensetzung der Httex1, die vor allem Glutamin und Prolin Rückstände enthalten. Der Wildtyp (23Q) und die Mutante (43Q) Schmelzverfahren Protein kann zu ca. 80 % Reinheit durch IMAC (Abbildung 1D) das Vorhandensein von Co Host Protein Reinigung sich durch die vergleichsweise geringe Expression des Httex1 und der großen Probe lässt angereichert Lautstärke auf die Spalte angewendet.
Die Spaltung seiner6-SUMO-Tag und die Reinigung des Httex1 ist in Abbildung 2Agezeigt. UPLC ist ein effizientes Werkzeug zur Überwachung der Spaltung seiner6-SUMO-Tag (Abbildung 2B). Die ursprüngliche Spitze des Fusionsproteins wird verbraucht und zwei neue und gut getrennt Peaks entspricht seinem6-SUMO-Tag und Httex1 erscheinen. Die Reaktion der Spaltung ist in 10-20 min fertig. Der Western Blot (WB) ist zu langsam, um die Spaltung Reaktion effizient zu überwachen wurde, sondern es in der Abbildung als Referenz und die Vollständigkeit der SUMO-Dekolleté zu zeigen. Beide Httex1 23Q und 43Q können getrennt werden, auch von seinem6-SUMO tag mittels RP-HPLC (Abbildung 2C) und in hoher Reinheit gewonnen wurden, dargestellt durch UPLC, MS und SDS-PAGE-Analyse (Abbildung 2D).
Um zu verdeutlichen, dass die Httex1 Proteine, die von dieser Methode zubereitet behalten die erwarteten Aggregation Eigenschaften der Httex1, wir die Fibrillization Kinetik der Mutant Httex1 bei 37 ° C durch eine Sedimentation Assay bewertet überwacht die Änderungen in der Sekundärstruktur von CD Spektroskopie, und die Morphologie der Aggregate durch TEM charakterisiert. Abbildung 3Azeigt ein repräsentativen Datensatz der Aggregation Kinetik der mHttex1 Fibrille Bildung durch eine Sedimentation-Assay bestimmt. Der Verlust von löslichen Httex1 43Q im Laufe der Zeit durch die Fibrille Bildung durch UPLC quantifiziert wurde. Wir beobachten eine komplette Entleerung des löslichen Proteins nach 48 Stunden Inkubation. Darüber hinaus haben wir die Sekundärstruktur des Proteins durch CD-Spektroskopie (Abbildung 3B) festgestellt. Httex1 43Q Schichten aus unstrukturierten (λmin 205 nm), vor allem β-Sheet reichen Konformation (λmin 215 nm) nach 48 Stunden Inkubation. Dieser Strukturwandel wird durch die Bildung von lange Fibrillären Aggregate als beobachtbare durch TEM 48 Stunden (Abb. 3C) begleitet.
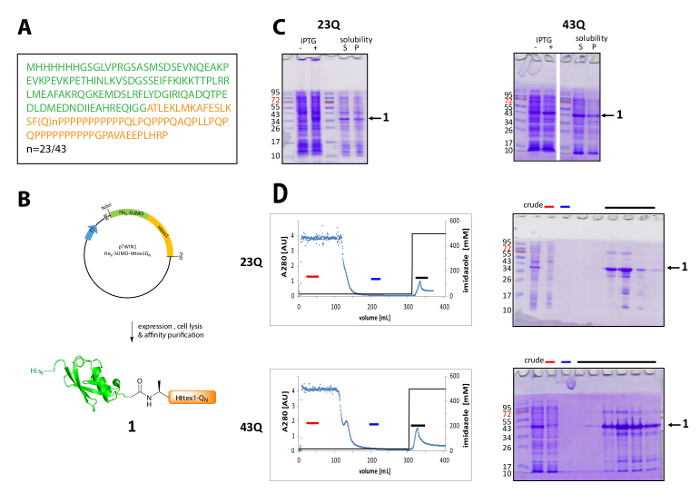
Abbildung 1 . Ausdruck und Reinigung seiner6-Fusionsprotein SUMO Httex1.
(A) die Aminosäure-Sequenz seiner6-SUMO-Httex1-QN Fusion Konstrukte (seine6-SUMO in grün und Httex1-QN in Orange); (B) schematische Übersicht des Ausdrucks und Reinigung des Fusionsproteins; (C) SDS-PAGE-Analyse des Ausdrucks und die Löslichkeit des Fusionsproteins nach Lyse; (D) Chromatogramm der IMAC-Reinigung des Fusionsproteins und Analyse der Fraktionen durch SDS-PAGE (roter Balken: ungebunden, blaue Bruchstrich: Waschen Bruchteil, schwarz bar: Brüche mit der Elution Gipfel); Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 2 . Spaltung von seinem6 SUMO Tag und Reinigung des Tag-freie Httex1-QN Proteine.
(A) schematische Übersicht; (B) Analyse der Spaltung des SUMO-Tags mit ULP1 von UPLC (blau: vor Zugabe des ULP1; schwarz: 20 min (23Q), bzw. 10 min (43Q) nach Zugabe von ULP1) und WB (MAB5492 1: 2000, sekundäre Ziege anti-Maus-Antikörper 1: 5.000); (C) Chromatogramm der präparativen RP-HPLC-Reinigung von Httex1; D: Analyse des gereinigten Httex1 durch UPLC, SDS-PAGE und ESI-MS; das erwartete Molekulargewicht beträgt 9943 Da (23Q) bzw. 12506 Da (43Q). Bitte klicken Sie hier für eine größere Version dieser Figur.
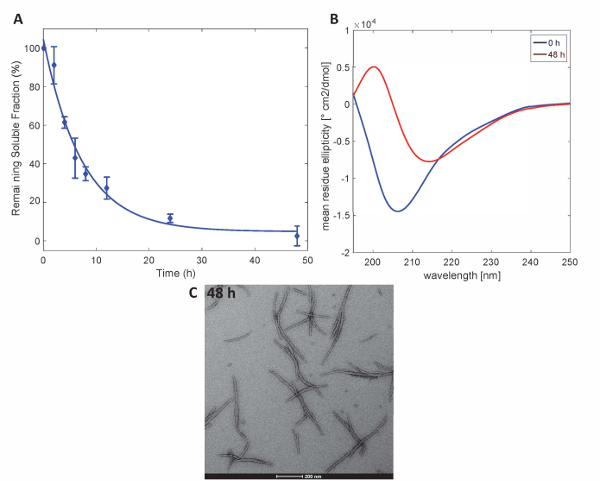
Abbildung 3 . Aggregation von Httex1-43Q: (A) Sedimentation Assay basierend auf UPLC. (B) CD-Spektren von der Sekundärstruktur bei 0 h und 48 h (C) TEM mikrographen der Aggregate bei 48 h (Maßstabsleisten sind 200 nm). Bitte klicken Sie hier für eine größere Version dieser Figur.
Diskussion
In diesem Protokoll haben wir ein effizientes Verfahren für herbeiführende Milligramm-Mengen von native, untagged Httex1 skizziert, die 23 oder 43 Glutamin Rückstände enthalten. Dies gelang durch mit dem Ausdruck Httex1 als eine C-terminale Fusion, eine seiner6-SUMO-Tag, das wird verwendet, um die Schmelzverfahren Protein aus der Zelle lysate isolieren von IMAC und vor der HPLC-Reinigung des Httex1 gespalten ist. Während die SUMO-Strategie bei der Herstellung von mehreren anderen Proteinen verwendet worden ist, zeigt unsere Methode, dass die einzigartigen Eigenschaften SUMO könnte auch verwendet werden, um per se zu generieren, Aggregation neigende, Amyloidogenic Protein, das zuvor erwiesen haben ungeordneten äußerst schwierig zu handhaben und zu produzieren43,44. Wir präsentieren Ihnen eine Protokoll, die unkompliziert, einfach zu bedienen und vergleichbar mit einem Protokoll für die Generation der "brav" Protein ist. Die SUMO-Fusion solubilisiert und Httex1 im Ausdruck und der IMAC Reinigungsstufe stabilisiert. Vorzeitiger Spaltung des Tags, als mit der Intein Strategie10 und Aggregation beobachtet wurden nicht mehr ein Problem.
Intrinsisch ungeordneten Proteine sind besonders anfällig für Abbau. Während N-terminale Abbau der N17-Region kein Problem mit diesem Protokoll ist, können Trunkierungen in der PRD Httex1 auftreten. Da die abgeschnittenen Proteine Httex1 in Hydrophobie, Ladung und Größe sehr ähnlich sind, entfernen sie durch chromatographische Mittel ist eine Herausforderung, so ist es am besten zu ihrer Entstehung in erster Linie zu verhindern. Eng an das Protokoll sollte immer arbeiten auf Eis und mit einer ausreichenden Menge an Protease-Hemmer helfen die beobachteten Abschneiden sehr niedrig zu halten. Anwenden eines Fusion-Tags am C-Terminus des Httex1 könnte Trunkierungen in der PRD leicht entfernen wie die abgeschnittene Protein sowie die Affinität Tag verlieren würde. Allerdings, wenn der native Sequenz muss aufrechterhalten werden, dass diese Option angewendet werden kann, wie Httex1 mit Prolin und nach bestem Wissen und gewissen endet gibt es keine C-terminale Fusion-Tags, die bekanntermaßen spurlos und effiziente Spaltung nach Prolin zu induzieren.
Der wichtigste Teil des Protokolls ist der Umgang mit der Httex1 nach der Spaltung des SUMO-Tags von ULP1 befreit. Das Protein sollte sofort mittels RP-HPLC gereinigt werden. Glücklicherweise ist dies eine effiziente und schnelle Reaktion, die in der Regel in 10-20 min bei 4 ° c abgeschlossen ist Im Gegensatz dazu benötigt Intein Strategie mehrere Stunden für komplette Spaltung von Intein, erfordern somit einen Kompromiss zwischen unvollständiger Spaltung und Anfang Aggregation um den Ertrag zu maximieren. Eine schnelle Aufarbeitung ist erforderlich für Mutant Httex1, wie es beginnt bei der vergleichsweise hohe Konzentration im Dekolleté-Reaktion, zu aggregieren, während die 23Q Variante für einen längeren Zeitraum stabil ist. Während der RP-HPLC-Reinigung, ein weiterer Vorteil des SUMO wird deutlich: während die Ssp DnaB Intein hydrophob ist und hält sich stark an der Spalte, SUMO ist mehr hydrophil und elutes komplett aus der Spalte C4 umgekehrt-Phase. Obwohl kommerzielle ULP1 ziemlich teuer ist, kann das Protein in hoher Ausbeute nach zuvor veröffentlichten Protokolle29leicht hergestellt werden.
Die kritische Bedeutung der Anwendung einer Aufschlüsselung Protokoll vor der Verwendung von Httex1 lässt sich nicht oft genug betont. Lyophilisierten PolyQ Proteine wie Httex1 sind stabil und können gespeicherte lange Zeiträume werden, jedoch nicht vollständig löslich in Wasser und Puffer. Das Vorhandensein von vorgeformten Oligomere oder Fibrillen hätte erhebliche Auswirkungen auf die Aggregation Kinetik und biophysikalischen Eigenschaften des Proteins45. Das hier beschriebene Disaggregation-Protokoll ermöglicht die Aufschlüsselung des Proteins, Entfernung des vorgeformten Aggregate und Erzeugung einer Lösung von Monomeren Httex1 aus einem lyophilisierten Sample. Wir beobachteten ähnliche Aggregation Kinetik und Fibrille Morphologie für Httex1 mit der SUMO und Intein Strategie erhalten.
Im Vergleich zu bisherigen Methoden zur Herstellung von Httex1, die hier beschriebene SUMO-Strategie bietet mehrere Vorteile und erweitert die Palette der möglichen Untersuchungen die Struktur und funktionellen Eigenschaften dieses Proteins in Gesundheit und Krankheit. Die SUMO-Httex1-Fusionsprotein ist einfach zu handhaben, es eingefroren und gespeichert oder in Lösung für 24 h bei Raumtemperatur, gehalten, während die freie mHttex1 schnell zusammenfassen würde. Die Stabilität und die hohe Löslichkeit von SUMO-Httex1-Fusionsproteinen bieten größere Flexibilität um das Protein zu manipulieren und/oder enzymatische und chemische Modifikationen in mHttex1, die sonst nicht möglich nach Spaltung einzuführen. Dazu gehören die Einführung des Post-translationalen Modifikationen, Fluorophore, Spin-Etiketten, Biotin-Tags, etc. sollten die Fortschritte hier vorgestellten 1) erleichtern zukünftige Studien zur Aufklärung von Struktur-Funktions-Beziehungen von Httex1; (2) erzeugen Sie neue Werkzeuge, um Htt Aggregation und Pathologie Verbreitung zu untersuchen; (3) ermöglichen Sie die Entwicklung von neuen Assays Moleküle identifizieren, die Mutant Httex1 zu stabilisieren und zu verhindern, dass die Aggregation; und 4) Wissenschaftler aus anderen Bereichen zu arbeiten auf dieses Protein und verbinden unsere Suche nach Kuren für die Huntington-Krankheit zu fördern.
Offenlegungen
Die Autoren erklären, dass sie keine Interessenkonflikte mit dem Inhalt dieses Artikels.
Danksagungen
Diese Arbeit wurde vor allem durch Zuschüsse von der CHDI-Stiftung und des Schweizerischen Nationalfonds finanziert. Wir danken Dr. Sophie Vieweg für nützliche Gespräche während der Entwicklung dieses neuen Systems Ausdruck und andere Mitglieder der Lashuel Gruppe für ihre Erfahrungen mit diesem Ausdruck System und für ihre ein- und wertvolles Feedback. Wir danken auch Prof. Oliver Hantschel für die Bereitstellung der ULP1 Plasmid. Die Autoren danken Dr. John B. Warner und Dr. Senthil K. Thangaraj für kritische Durchsicht des Manuskripts
Materialien
| Name | Company | Catalog Number | Comments |
| Uranyl formate (UO2(CHO2)2) | EMS | 22450 | |
| Formvar/carbon 200 mesh, Cu 50 grids | EMS | FCF200-Cu-50 | |
| High Precision Cell made of Quartz SUPRASIL 1 mm light path from Hellma Analytics | HellmaAnalytics | 110-1-40 | |
| Buffer Substance Dulbecco's (PBS w/o Ca and Mg) ancinne ref 47302 (RT) SERVA | Witech | SVA4730203 | |
| Ampicillin | AxonLab | A0839.0100 | |
| Luria Broth (Miller's LB Broth) | Chemie Brunschwig | 1551.05 | |
| Isopropyl β-D-1-thiogalactopyranoside (IPTG) | AxonLab | A1008.0025 | |
| E. coli B ER2566 | NEB | NEB# E4130 | |
| Imidazole | Sigma | 56750-500G | |
| cOmplete Protease Inhibitor Cocktail | Roche | 4693116001 | |
| Anti-Huntingtin Antibody, a.a. 1-82 | Merck Millipore Corporation | MAB5492 | |
| IRDye 680RD Goat anti-Mouse IgG (H + L) | Licor | 925-68070 | |
| PMSF | AxonLab | A0999.0005 | |
| HisPrep 16/10 column | GE Healthcare | 28936551 | |
| C4 HPLC column | Phenomenex | 00G-4168.P0 | 10 µm C4 300 Å, LC Column 250 x 21.2 mm, Phenomenex, 19x10 mm guard column, not temperature jacketed |
| Acetonitrile HPLC | MachereyNagel | C2502 | |
| Filtre seringue Filtropur S 0,45 ul sans prefiltre sterile | Sarstedt AG | 83.1826 | |
| Spectrophotometer semi-micro cuvette | Reactolab S.A. | 2534 | |
| Superloop, 1/16" fittings (ÄKTAdesign), 50 ml | GE Healthcare | 18111382 | |
| Trifluoroacetic acid | Sigma | 302031 | |
| GREINER Tubes fo FPLC 16 x 100 mm, cap. 12.0 ml | Greiner Bio-One | 7.160 102 | |
| 100 kD Microcon fast flow filters | Merck Millipore Corporation | MRCF0R100 | |
| Vibra-cell VCX130 ultrasonic liquid processor | Sonics | ||
| Äkta 900 equipped with a fraction collector | GE Healthcare | ||
| Jasco J-815 Circular Dichroism | Jasco | ||
| Waters UPLC system | Waters | C8 BEH acquity 2.1x100 mm 1.7 micron column , preheated column (40 °C), flow rate of 0.6 mL/min, injection volume of 4 μL | |
| waters HPLC system | Waters | 2489 UV detector and 2535 quaternary gradient module, 20 mL loop in a FlexInject housing | |
| ESI-MS: Finnigan LTQ | Thermo Fisher Scientific | ||
| lyophylizer instrument | FreeZone 2.5 Plus | ||
| Tecnai Spirit BioTWIN | FEI | electron microscope equipped with a LaB6 gun and a 4K x 4K FEI Eagle CCD camera (FEI) and operated at 80 kV | |
| 37 °C shaking incubator | Infors HT multitron Standard | ||
| Biophotometer plus | Eppendorf |
Referenzen
- Saudou, F., Humbert, S. The Biology of Huntingtin. Neuron. 89 (5), 910-926 (2016).
- MacDonald, M. E., Gines, S., Gusella, J. F., Wheeler, V. C. Huntington's disease. Neuromolecular Medicine. 4 (1-2), 7-20 (2003).
- Li, S., Li, X. J. Multiple pathways contribute to the pathogenesis of Huntington disease. Molecular Neurodegeneration. 1, 19 (2006).
- DiFiglia, M. Aggregation of Huntingtin in Neuronal Intranuclear Inclusions and Dystrophic Neurites in Brain. Science. 277 (5334), 1990-1993 (1997).
- Atwal, R. S., et al. Huntingtin has a membrane association signal that can modulate huntingtin aggregation, nuclear entry and toxicity. Human Molecular Genetics. 16 (21), 2600-2615 (2007).
- Sathasivam, K., et al. Aberrant splicing of HTT generates the pathogenic exon 1 protein in Huntington disease. Proceedings of the National Academy of Sciences U S A. 110 (6), 2366-2370 (2013).
- Mangiarini, L., et al. Exon 1 of the HD gene with an expanded CAG repeat is sufficient to cause a progressive neurological phenotype in transgenic mice. Cell. 87 (3), 493-506 (1996).
- El-Daher, M. T., et al. Huntingtin proteolysis releases non-polyQ fragments that cause toxicity through dynamin 1 dysregulation. EMBO Journal. 34 (17), 2255-2271 (2015).
- Lunkes, A., et al. Proteases acting on mutant Huntingtin generate cleaved products that differentially build up cytoplasmic and nuclear inclusions. Molecular Cell. 10 (2), 259-269 (2002).
- Vieweg, S., Ansaloni, A., Wang, Z. M., Warner, J. B., Lashuel, H. A. An Intein-based Strategy for the Production of Tag-free Huntingtin Exon 1 Proteins Enables New Insights into the Polyglutamine Dependence of Httex1 Aggregation and Fibril Formation. Journal of Biological Chemistry. 291 (23), 12074-12086 (2016).
- Georgalis, Y., et al. Huntingtin aggregation monitored by dynamic light scattering. Proceedings of the National Academy of Sciences U S A. 95 (11), 6118-6121 (1998).
- Scherzinger, E., et al. Huntingtin-encoded polyglutamine expansions form amyloid-like protein aggregates in vitro and in vivo. Cell. 90 (3), 549-558 (1997).
- Scherzinger, E., et al. Self-assembly of polyglutamine-containing huntingtin fragments into amyloid-like fibrils: implications for Huntington's disease pathology. Proceedings of the National Academy of Sciences U S A. 96 (8), 4604-4609 (1999).
- Muchowski, P. J., et al. Hsp70 and hsp40 chaperones can inhibit self-assembly of polyglutamine proteins into amyloid-like fibrils. Proceedings of the National Academy of Sciences U S A. 97 (14), 7841-7846 (2000).
- Heiser, V., et al. Inhibition of huntingtin fibrillogenesis by specific antibodies and small molecules: implications for Huntington's disease therapy. Proceedings of the National Academy of Sciences U S A. 97 (12), 6739-6744 (2000).
- Bennett, E. J., Bence, N. F., Jayakumar, R., Kopito, R. R. Global impairment of the ubiquitin-proteasome system by nuclear or cytoplasmic protein aggregates precedes inclusion body formation. Molecular Cell. 17 (3), 351-365 (2005).
- Tam, S., et al. The chaperonin TRiC blocks a huntingtin sequence element that promotes the conformational switch to aggregation. Nature Structural & Molecular Biology. 16 (12), 1279-1285 (2009).
- Nekooki-Machida, Y., et al. Distinct conformations of in vitro and in vivo amyloids of huntingtin-exon1 show different cytotoxicity. Proceedings of the National Academy of Sciences U S A. 106 (24), 9679-9684 (2009).
- Wacker, J. L., Zareie, M. H., Fong, H., Sarikaya, M., Muchowski, P. J. Hsp70 and Hsp40 attenuate formation of spherical and annular polyglutamine oligomers by partitioning monomer. Nature Structural & Molecular Biology. 11 (12), 1215-1222 (2004).
- Legleiter, J., et al. Monoclonal antibodies recognize distinct conformational epitopes formed by polyglutamine in a mutant huntingtin fragment. Journal of Biological Chemistry. 284 (32), 21647-21658 (2009).
- Legleiter, J., et al. Mutant huntingtin fragments form oligomers in a polyglutamine length-dependent manner in vitro and in vivo. Journal of Biological Chemistry. 285 (19), 14777-14790 (2010).
- Nucifora, L. G., et al. Identification of novel potentially toxic oligomers formed in vitro. from mammalian-derived expanded huntingtin exon-1 protein. Journal of Biological Chemistry. 287 (19), 16017-16028 (2012).
- Dahlgren, P. R., et al. Atomic force microscopy analysis of the Huntington protein nanofibril formation. Nanomedicine. 1 (1), 52-57 (2005).
- Poirier, M. A., et al. Huntingtin spheroids and protofibrils as precursors in polyglutamine fibrilization. Journal of Biological Chemistry. 277 (43), 41032-41037 (2002).
- Duim, W. C., Chen, B., Frydman, J., Moerner, W. E. Sub-diffraction imaging of huntingtin protein aggregates by fluorescence blink-microscopy and atomic force microscopy. Chemphyschem. 12 (13), 2387-2390 (2011).
- Pieri, L., Madiona, K., Bousset, L., Melki, R. Fibrillar alpha-synuclein and huntingtin exon 1 assemblies are toxic to the cells. Biophysical Journal. 102 (12), 2894-2905 (2012).
- Monsellier, E., Redeker, V., Ruiz-Arlandis, G., Bousset, L., Melki, R. Molecular interaction between the chaperone Hsc70 and the N-terminal flank of huntingtin exon 1 modulates aggregation. Journal of Biological Chemistry. 290 (5), 2560-2576 (2015).
- Isas, J. M., Langen, R., Siemer, A. B. Solid-State Nuclear Magnetic Resonance on the Static and Dynamic Domains of Huntingtin Exon-1 Fibrils. Biochemistry. 54 (25), 3942-3949 (2015).
- Malakhov, M. P., et al. SUMO fusions and SUMO-specific protease for efficient expression and purification of proteins. Journal of Structural Function Genomics. 5 (1-2), 75-86 (2004).
- Mossessova, E., Lima, C. D. Ulp1-SUMO crystal structure and genetic analysis reveal conserved interactions and a regulatory element essential for cell growth in yeast. Molecular Cell. 5 (5), 865-876 (2000).
- Kumari, S., Pal, R. K., Gupta, R., Goel, M. High Resolution X-ray Diffraction Dataset for Bacillus licheniformis Gamma Glutamyl Transpeptidase-acivicin complex: SUMO-Tag Renders High Expression and Solubility. Protein Journakl. 36 (1), 7-16 (2017).
- Zhang, J., Sun, A., Dong, Y., Wei, D. Recombinant Production and Characterization of SAC, the Core Domain of Par-4, by SUMO Fusion System. Applied Biochemistry and Biotechnology. , (2017).
- Reif, A., et al. Semisynthesis of biologically active glycoforms of the human cytokine interleukin 6. Angewandte Chemie International Edition English. 53 (45), 12125-12131 (2014).
- Froger, A., Hall, J. E. Transformation of plasmid DNA into E. coli using the heat shock method. Journal of Visualized Experiments. (6), 253 (2007).
- Smith, B. J. SDS Polyacrylamide Gel Electrophoresis of Proteins. Methods in Molecular Biology. 1, 41-55 (1984).
- Lawrence, A. M., Besir, H. U. Staining of proteins in gels with Coomassie G-250 without organic solvent and acetic acid. Journal of Visualized Experiments. (30), (2009).
- Block, H., et al. Immobilized-metal affinity chromatography (IMAC): a review. Methods in Enzymology. 463, 439-473 (2009).
- Chen, S. M., Wetzel, R. Solubilization and disaggregation of polyglutamine peptides. Protein Science. 10 (4), 887-891 (2001).
- Booth, D. S., Avila-Sakar, A., Cheng, Y. Visualizing proteins and macromolecular complexes by negative stain EM: from grid preparation to image acquisition. Journal of Visualized Experiments. (58), (2011).
- O'Nuallain, B., et al. Kinetics and thermodynamics of amyloid assembly using a high-performance liquid chromatography-based sedimentation assay. Amyloid, Prions, and Other Protein Aggregates, Pt C. 413, 34-74 (2006).
- Greenfield, N. J. Analysis of circular dichroism data. Methods in Enzymology. 383, 282-317 (2004).
- Aiken, C. T., et al. Phosphorylation of threonine 3: implications for Huntingtin aggregation and neurotoxicity. Journal of Biological Chemistry. 284 (43), 29427-29436 (2009).
- Satakarni, M., Curtis, R. Production of recombinant peptides as fusions with SUMO. Protein Expression and Purification. 78 (2), 113-119 (2011).
- Davies, H. A., Wilkinson, M. C., Gibson, R. P., Middleton, D. A. Expression and purification of the aortic amyloid polypeptide medin. Protein Expression and Purification. 98, 32-37 (2014).
- Chen, S., Wetzel, R. Solubilization and disaggregation of polyglutamine peptides. Protein Science. 10 (4), 887-891 (2001).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten