Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Isolieren und die Einbeziehung von Licht-ernten Antennen von Diatomee Cyclotella Meneghiniana in Liposomen mit Thylakoidmembran Lipide
In diesem Artikel
Zusammenfassung
Hier präsentieren wir Ihnen ein Protokoll zur Fucoxanthin Chlorophyll a/c-bindende Proteine (FCP) von Kieselalgen zu isolieren und integrieren sie in Liposomen mit natürlichen Lipid Kompositionen Erregung Energieübertragung auf Ionen-Zusammensetzung Änderungen zu studieren.
Zusammenfassung
Die photosynthetische Leistungsfähigkeit von Pflanzen, Algen und Diatomeen hängt stark von der schnelle und effiziente Regulierung der leichte Ernte und Energie Transferprozesse in der Thylakoidmembran Membran der Chloroplasten. Das Licht, die Ernte Antenne von Kieselalgen, die so genannte Fucoxanthin Chlorophyll a/c-bindende Proteine (FCP), sind erforderlich für die Lichtabsorption und effiziente Übertragung der photosynthetischen Reaktion Zentren sowie für Foto-Schutz vor übermäßig viel Licht. Der Wechsel zwischen diesen beiden Funktionen ist eine langjährige Frage der Forschung. Viele dieser Studien wurden mit FCP in Waschmittel Micellen durchgeführt. Für Studien zu Wechselwirkungen wurden die Reinigungsmittel entfernt, was dazu führte, eine unspezifische Aggregation der FCP-komplexe. Bei diesem Ansatz ist es schwer, zwischen Artefakten und physiologisch relevanten Daten zu unterscheiden. Daher erhalten wertvoller Informationen zu FCP und andere Membran gebunden leichte Ernte komplexe von Protein-Protein-Wechselwirkungen, Energieübertragung und andere spektroskopischen Eigenschaften zu studieren, wenn sie in ihrer Muttersprache Lipid-Umgebung eingebettet sind. Der Hauptvorteil ist, dass Liposomen haben eine definierte Größe und einem definierten Lipid/Protein-Verhältnis durch die das Ausmaß der FCP-clustering gesteuert wird. Darüber hinaus können Veränderungen in der pH und Ionen-Zusammensetzung, das Licht, die Ernte in Vivo regulieren einfach simuliert werden. Im Vergleich zu der Thylakoidmembran Membran sind die Liposomen homogener und weniger komplex, wodurch es einfacher, spektroskopischen Daten zu erhalten. Das Protokoll beschreibt den Ablauf der FCP-Isolierung und Reinigung, Liposomen Vorbereitung und Einbindung der FCP in Liposomen mit natürlichen lipidzusammensetzung. Ergibt sich aus einer typischen Anwendung sind gegeben und diskutiert.
Einleitung
Photosynthetische Organismen wie Kieselalgen müssen mit wechselnden Lichtverhältnissen bewältigen und mit anspruchsvollen Akklimatisierung Mechanismen, die nachhaltig hohe photosynthetischen Leistungsfähigkeit und schützt vor Foto-oxidative Schäden, die durch das übermäßige Licht reagieren. Ein bedeutenden Licht-Schutz-Prozess in den photosynthetischen Eukaryotes ist die hochenergetische abschrecken (QE) absorbiert Licht, das auftritt, als der wichtigste Beitrag zur nicht-photochemische abschrecken (NPQ) unter Lichtstress Bedingungen1,2 ,3. Die leichte Ernte Antennenkomplexe (LHC) sind bei der Regulierung der Übertragung Energiebahnen Erregung beteiligt. Als Reaktion auf hohe Licht induzierten niedriger pH-Wert in den Chloroplasten Lumen, die Antenne System Schalter aus dem Licht, die Ernte in den abschrecken Zustand. Dieses dissipativen Energiezustand verhindert photosysteme (PS) und anderen Anlagen in der Thylakoidmembran Membran Photooxidation. In den photosynthetischen Eukaryotes wird QE in der Regel durch zwei Faktoren1,2,3induziert. Ein Faktor ist das spezielle Licht Ernte Protein, das auf den niedrigen pH-Wertreagiert. Die PsbS-Protein induziert die QE in höheren Pflanzen4. LhcSRs5, moduliert durch PsbS Aktivität induzieren die QE in Grünalgen6. Kieselalgen besitzen Lhcx-ähnliche Proteine, die strukturell verwandt mit LHCSRs7,8,9,10.
Der zweite Faktor qE ist das Xanthophyll Zyklus wo Carotinoide der Antenne in Form eines Foto-Schutz durch de-Epoxidierung umgewandelt und durch Epoxidierung zurückgesetzt. In Pflanzen und Grünalgen wird Violaxanthin in Zeaxanthin konvertiert. In Diatomeen ist Diatoxanthin, Diadinoxanthin konvertiert, die dann mit dem Ausmaß der NPQ11 korreliert. Die Diatomee Licht Ernte Antenne besitzt einige Besonderheiten, obwohl es evolutionär im Zusammenhang mit Pflanzen und Algen LHCs ist. Der Wechsel von Licht, die Ernte bis zur Foto-Schutz ist enorm schnell und die NPQ Kapazität ist höher im Vergleich zu Pflanzen12. Dies könnte ein Grund sein, warum Diatomeen sind sehr erfolgreich in verschiedenen ökologischen Nischen in einer Weise, die sie für bis zu 45 % der ozeanischen Primärproduktion13verantwortlich sind. Diatomee leichte Ernte Systeme sind daher ein interessantes Objekt der Fotosynthese Forschung.
Diatomeen, wie die centric Art Cyclotella Meneghiniana Thylakoidmembran inneren Licht Ernte Systeme benannt nach die Pigmente, die sie besitzen binden - Fucoxanthin, Chlorophyll (Chl) a und C, also FCP Licht Ernte Proteine, wie z. B. FCPs, sind eingebettet in die Thylakoidmembran-Membran-System, bestehend aus mehreren Schichten der Membran. Kieselalgen bilden Bands der drei Thylakoids. Dieser Komplex Situation macht es schwierig, sie zu studieren, auf molekularer Ebene in die Thylakoidmembran Membran. Darüber hinaus tragen viele Komponenten bei der Regulierung von Licht, die Ernte (siehe oben). Daher wurden in vielen Ansätzen, die komplexe isoliert aus der Membran mit Feinwaschmittel, wie n-Dodecyl-β-D-Maltopyranoside (β-DDM), die anderweitig der Membran aber die FCP-komplexe intakt zu halten. Viele spektroskopische Untersuchungen wurden durchgeführt mit solubilisiert FCP, um Intramolekulare Energie Übertragung14,15,16,17zu untersuchen. Jedoch war dieser ehemaligen Ansatz beschränkt, da die Regulierung der Energieübertragung excitonic Interaktion mit anderen Antennenkomplexe oder photosysteme braucht. Daher können nicht diese Art von Studien mit solubilisiert komplexe erfolgen, weil die Interaktion zwischen komplexen verloren geht.
Ein wichtiges Merkmal im Antenne-Verordnung ist die "Molekulare Verdrängung" der Antenne und phototrophen in die Thylakoidmembran Membran18. Früher ein einfacher Ansatz wurde durchgeführt, um diesen Effekt zu simulieren in-vitro-. Das Waschmittel wurde entfernt, was zu zufälligen Aggregation der Antennenkomplexe führt. Obwohl einige vernünftigen Daten von diesem Ansatz17,19erworben wurde, das Reinigungsmittel entfernen spiegelt nicht die Situation in Vivo und hat einige Einschränkungen, da die komplexe nicht in ihre regelmäßigen Tertiär interagieren Struktur.
Die Verwendung von Liposomen überwindet einige der ehemaligen Einschränkungen. Die Tertiärstruktur ist noch völlig intakt. Der liposomenmembran bietet eine quasi-native Umgebung für die Antennenkomplexe. Die Membran trennt das Innere der Liposomen aus der äußeren Umgebung. Auf diese Weise bieten Liposomen zwei Reaktion Fächer für Studien von Ionen und pH Gradienten sowie Transportprozesse. Darüber hinaus können die Parameter des experimentellen Systems leichter für Studien in der Thylakoidmembran Membran gesteuert werden. Liposomen bereits erwiesen sich ein ausgezeichnetes Werkzeug, photosynthetischen komplexe zu studieren. Ein wichtiger Schwerpunkt in der Vergangenheit wurde auf Pflanze LHC, wo die Wirkung der veränderte lipidzusammensetzung am LHC II20getestet wurde. In anderen Ansätzen Proteinprotein Interaktionen zwischen verschiedenen LHC II waren untersuchten21. Darüber hinaus wurden einige Studien in Grünalgen durchgeführt, die beschreiben, spontane clustering zwischen LHC22. In Anbetracht der Bedeutung von Kieselalgen für aquatische Ökosysteme wurden relativ wenige Studien mit Antennenkomplexe von Kieselalgen durchgeführt. Zwei Studien untersuchten die Antennenkomplexe centric Cyclotella Meneghiniana, wo das clustering der FCP Antenne23 und Reaktionsfähigkeit des FCP auf elektrochemischen Gradienten24 wurden gezeigt. Liposomen sind somit ein hervorragendes Instrument Diatomee Antennen und deren Interaktion und Regulierung in fast native Bedingungen zu studieren. Die Liposomen sind vielseitig, da viele Bedingungen wie lipidzusammensetzung, Liposomen Größe, Protein-Dichte und die umgebende wässrige Phase angesteuert werden. Die Methode erfordert darüber hinaus geringe Mengen an Proben. Die experimentelle System ist robust und hoch reproduzierbare. Die Abschottung der Liposomen ermöglicht Studium pH und Ionen-Gradienten, die wichtig sind bei der Regulierung der Antennenkomplexe Faktoren.
Hier beschreiben wir die Isolation der FCP Antennenkomplexe von C. Meneghiniana und ihre Eingliederung in den Liposomen mit natürlichen Thylakoidmembran lipidzusammensetzung. Auch wir bieten beispielhafte Daten für die spektroskopische Charakterisierung von solubilisiert FCP und vergleichen Sie sie mit FCP in Liposomen. Die Methode fasst wissen und standardisierte Protokolle von den Verbesserungen der Gundermann und Büchel 201223, Natali Et Al. 201622und Ahmad und Dietzel 201724erhalten.

Abbildung 1: Schematische Darstellung des Workflows. (1) bezieht sich auf Absatz 1 beschreibt Zellwachstum, Störung und Thylakoidmembran Isolation mit FCP Trennung im Anschluss an Saccharose Dichtegradienten; C. m. -Cyclotella Meneghiniana Zellen. (2) Vorbereitung der natürlichen Thylakoidmembran Lipid Mischung (MGDG, DGDG und SQDG) gemäß Absatz 2 und die Schaffung von Lipid-Reinigungsmittel Micellen mit Octylglycoside (OG). Eine definierte Lipid-Micelle Größe wird durch Extrusion mit Membranen von einer definierten Porendurchmesser erreicht. FCP und Lipid-Mizellen sind vereint in einem vordefinierten Lipid: Protein-Verhältnis und die Reinigungsmittel OG und β-DDM sind entfernte über kontrollierte Dialyse FCP Proteoliposomes bilden. Bitte klicken Sie hier für eine größere Version dieser Figur.
Protokoll
Hinweis: Photosynthetischen komplexe wie FCPs sind sehr anfällig für Licht und Wärme. Arbeiten Sie immer auf dem Eis und unter einem sehr schwachem Licht.
1. Isolierung der FCP aus Zellen
-
Thylakoidmembran Isolation von C. Meneghiniana Zellen
- Wachsen Sie C. Meneghiniana in fünf 500 mL-Flaschen, die jeweils mit 300 mL von ASP-Medium23,25 und 50 Millionen Zellen gefüllt. Stecken Sie die Flaschen mit einem Baumwoll-stopfen und lassen Sie die Zellen wachsen in der exponentiellen Wachstumsphase für etwa eine Woche auf einem Boston-Shaker mit 120 u/min mit einem 16 h Licht und dunkle Phase 8 h 40 µmol photons/(m²s) weißes Licht und eine Temperatur zwischen 15-18 ° C. Überprüfen Sie die Anzahl von Zellen zwischen 1,5 Millionen Zellen/mL mit einer Zelle Zähler Kammer.
- Zentrifugieren Sie die Zellen bei 4.000 X g in eine vorgekühlte Rotor mit 500 mL Zentrifuge Fläschchen (4 ° C) für 15 min in einer High-Speed-Zentrifuge. Wieder aussetzen der Zelle Pellets in 12 mL Homogenisierung Puffer (HB, Tabelle 1) durch pipettieren.
- Übertragen Sie die Aussetzung auf einen einzigen 50 mL-Kunststoff-Rohr. Speichern Sie die Proben bei-80 ° C oder fahren Sie mit Schritt 1.1.3.
- Kühlen Sie Rührwerksmühle und Ausrüstung vor. Füllen Sie die 50 mL-Becherglas von der Rührwerksmühle auf 75 % mit einer Glas-Perle-Mischung und fügen Sie die Zellsuspension. Verwenden Sie für Zellaufschluss, 7 x 45 s Pulse auf Hochtouren mit 30 s Kühlung zwischen jedem Impuls. Nehmen Sie 20 µL des gestörten Zellen für Qualitätsprüfungen in Schritt 1.1.6.
- Filtern der gestörten Zellen über einen Filter Glastrichter und Waschen durch die HB über die Glasperlen Gießen, bis sie klar erscheinen. Pool der Wäsche Bruch mit dem Filtrat. Halten Sie das Endvolumen niedriger als 150 mL.
- Zentrifugieren Sie die Probe für 15 min bei 140 X g mit drei 50 mL-Kunststoff-Rohre zu den zellenrückstand Pellets. Sorgfältig übertragen Sie den überstand auf 20 mL Polycarbonat Ultrazentrifugation Fläschchen und entsorgen Sie das Pellet.
- Füllen Sie die Fläschchen mit HB, equilibrate das Gewicht und in einem geeigneten Rotor für 1 h bei 300.000 x g und 4 ° C in die Thylakoidmembran Membranen pellet Zentrifugieren.
- Nutzen Sie die Zentrifugation Zeit den Anteil der gestörten Zellen durch Mikroskopie bei 400 X Vergrößerung mit 20 µL Probe in 1.1.3 zu überprüfen. Berechnen Sie das Verhältnis zwischen Chloroplasten frei und Chloroplasten mit Frustules (Siliziumdioxid-Schalen).
Hinweis: Diatomee Zellwände bestehen aus Kieselsäure, die als sehr interferenzgitters Stoff im Mikroskop sichtbar ist. Chloroplasten auftritt als grüne Punkte sollten aus den Zellen freigegeben worden sein, wenn der Zellaufschluss gearbeitet. - Wieder aussetzen der Membran-Pellet mit als wenig waschen Puffer wie möglich (0,5-1 mL) mit einem kleinen Maler Pinsel. Füllen Sie die Polycarbonat Ultrazentrifugation Fläschchen mit Waschpuffer (Tabelle 1), equilibrate des Gewichtes und der Zentrifuge für 20 min bei 200.000 x g und 4 ° C.
- Wieder aussetzen Sie, die gewaschenen Membranen mit Pinsel des Malers. Waschen-Puffer nur bei Bedarf hinzugefügt werden, die Thylakoidmembran Konzentration so hoch wie möglich zu halten. Bündeln Sie alle Thylakoids in ein Probengefäß (15 mL).
- Bereits verdünnen Sie die Proben mit 10 µL der Probe mit 90 µL 100 % Aceton. Zentrifugieren sie 12.000 x g für 5 min zu Pellets ausgefälltes Eiweiß. Nehmen Sie 10 µL der Vorverdünnung und mischen Sie es mit 990 µL 90 % Aceton.
- Messung der Extinktion (ABS) von Chlorophyll A und c auf 664 nm und 630 nm in 90 % Aceton. Subtrahieren Sie die ABS-750nm aus beiden Werten. Den insgesamt Chlorophyll-Gehalt anhand der folgenden Formel:24
1)
2)
- Messung der Extinktion (ABS) von Chlorophyll A und c auf 664 nm und 630 nm in 90 % Aceton. Subtrahieren Sie die ABS-750nm aus beiden Werten. Den insgesamt Chlorophyll-Gehalt anhand der folgenden Formel:24
- Aliquoten Thylakoids in Portionen von 0,5 mg insgesamt Chlorophyll in einem 1,5 mL Reaktionsgefäße Einfrieren in flüssigem Stickstoff und bei-80 ° C bis zur weiteren Verwendung aufbewahren.
-
Trennung und Konzentration der FCP-komplexe
- Bereiten Sie einer Saccharoselösung gradient vor und füllen Sie die Ultrazentrifuge Rohre bis oben minus das Ladevolumen (300 – 500 µL). Frieren Sie die Röhrchen bei-20 ° C so lange ein, bis sie vollständig gefroren sind. Lassen Sie die Röhrchen bei + 4 ° C, 3-4 h für einen 17 mL-Tube führt Auftauen.
- Wiederholen Sie den Frost-Tau-Zyklus zweimal, um die Steigung für eine bessere Auflösung zu verfeinern.
- Verwenden Sie die Proben in 1.1.9 entspricht 0,5 mg Chlorophyll und justieren mit Puffer B1 (Tabelle 1) zu einem Endvolumen von 2 mL. Fügen Sie n-Dodecyl-β-D-Maltopyranoside (b-DDM hinzu) Solubilisierung eine Endkonzentration von 20 mM.
- Invertieren der Röhre 3 Mal und legen Sie es auf dem Eis für 20 min mit sanft schütteln um Schaum zu vermeiden. Zentrifuge für 5 min bei 12.000 X g in einer vorgekühlten Tischplatte Zentrifuge bei + 4 ° C.
- Laden Sie den überstand auf der Steigung. Geladen Sie mehr als 125 µg Gesamt Chlorophyll pro Farbverlauf nicht, wenn 17 mL Fläschchen verwendet werden. Zentrifuge für 22 h bei 100.000 x g und + 4 ° C.
- Erholen Sie die gewünschte braun FCP Brüche von der Steigung mit einer Spritze (Abbildung 2A). Nehmen Sie eine 5 µL aliquoten und verdünnen Sie es mit 995 µL B1a.
- Messen Sie das Absorptionsspektrum (ABS) zwischen 370-750 nm im UV-VIS-Spektralphotometer. Verwenden Sie Halbmikrowaagen optisches Glas Küvetten.
- Öffnen Sie die Spektren-Manager-Software, zum Spektrum-Modus gehen und aufzeichnen eine Basislinie von 370 bis 750 nm gefolgt von Probemessungen. Verwenden Sie die folgenden Einstellungen: scan-Geschwindigkeit, 200 nm/min; Daten Tonhöhe, 0,5 nm; Antwort, mittlere.
- Messen Sie das Volumen der wiederhergestellten Probe mit einer Mikropipette. Waschen Sie die FCP-komplexe durch Hinzufügen von zweimal die wiederhergestellten Volumen mit B1-Puffer (Tabelle 1). Konzentrieren Sie sich in einem Membran-Konzentrator mit einem 30 kDa cutoff bei 1.000 X g und + 4 ° C zu einer ABS-672nm von mindestens 20.
Hinweis: Die b-DDM-Konzentration könnte aufgrund der Micelle Anreicherung in den Probenraum steigen. Dies könnte zu weiteren Solubilisierung der FCP komplexe führen! Vermeiden Sie übermäßige Solubilisierung durch Zugabe von Waschmittel freien Puffer B1, wenn weitere Schritte waschen sind erforderlich, um verbleibende Saccharose zu entfernen. - Nehmen Sie eine 20 µL aliquoten für die Steuerelemente. Schock der Proben in flüssigem Stickstoff eingefroren und bei-80 ° c aufbewahren
- Bereiten Sie einer Saccharoselösung gradient vor und füllen Sie die Ultrazentrifuge Rohre bis oben minus das Ladevolumen (300 – 500 µL). Frieren Sie die Röhrchen bei-20 ° C so lange ein, bis sie vollständig gefroren sind. Lassen Sie die Röhrchen bei + 4 ° C, 3-4 h für einen 17 mL-Tube führt Auftauen.

Abbildung 2: Reinigung von FCP, spektroskopische Kontrollen und Reinheit überprüfen. (A) typische Aussehen eines Saccharose-Dichte-Gradienten nach Übernachtung Zentrifugation. Alle braune Bänder enthalten den FCP-Pool bestehend aus FCPa und FCPb. Pigm. - ungebundenen Pigmenten, PS - phototrophen (B) Absorption Spektren von FCP vor (blaue Linie) und nach (Orange gestrichelte Linie) Konzentration 30 kDa cutoff zentrifugale Filter Geräte mit . Carotinoide sind besonders anfällig für Verlust von FCP, die geringere Absorption im Bereich zwischen 500-550 nm zur Folge hätte. Graphen sind normiert auf das Chlorophyll Qy max. ~ 670 nm. (C) Chlorophyll A Emissionsspektren mit Anregung der Chl c (465 nm) für die Prüfung der funktionellen Erregung Energieübertragung. Wenn die Energie von Chl c an Chl Übertragung eine behindert wird, eine zusätzliche Fluoreszenz-Band auf ~ 640 nm (Chl c) auftreten würde. Grafiken sind auf maximale Emission normalisiert. (D) Anregung Spektren aufgenommen bei 675 nm (Chl eine maximale Fluoreszenz) zum Testen der Energieübertragung in Chl eine aus alle Pigmente absorbieren zwischen 370 nm und 600 nm. Wenn die Energie in Chl übertragen eine ist weniger effizient, die Fluoreszenz-Rendite vor allem zwischen 465 und 550 nm verringern würde. Die Diagramme sind normiert auf maximal rund 440 nm. Die Spektren in (B), (C) und (D) sind fast identisch, wenn die Konzentration gut funktioniert. (E) suchen Sie nach Reinheit der isolierten FCP mit einem Tris-Tricine Gel28. FCPa und FCPb haben Untereinheiten zwischen 18-19 kDa. Alle sichtbaren Silber gefärbten Proteine größer als 20 kDa sind Verunreinigungen. Thyl. -Thylakoids Klicken Sie bitte hier, um eine größere Version dieser Figur.
-
Spektroskopische und Gel-basierte Steuerungen
- Notieren Sie die Absorption zwischen 370-750 nm 5 µL des FCP in 995 µL B1a nach Schritt 1.2.5. Vergleichen Sie es mit dem in Schritt 1.2.4 erhaltenen Spektrum. Verwenden Sie die gleichen Instrumente und Einstellungen wie in Schritt 1.2.4 beschrieben.
- Exportieren Sie Daten als *.CSV und importieren Sie die Daten in eine Tabellenkalkulation. Beide Spektren zum Maximum des Chl zu normalisieren einer in der Q-y -Band von rund 672 nm wie in Abbildung 2 bdargestellt.
- Berechnen Sie den Verdünnungsfaktor der Proben aus Schritt 1.2.4 und 1.3.1 durch Teilung der gemessenen ABS-672nm durch die gewünschte ABS-672nm von 0,03. Die Proben entsprechend verdünnen mit B1a und übertragen Sie sie auf Spezialglas Fluoreszenz Küvetten.
- Rekord Chl ein Fluoreszenz-Emissionsspektrum mit einer Spectrofluorometer, intakt Lichtenergie zu offenbaren transfer vom Chl c zum Chl (Abbildung 2).
- Die Spektrometer-Software verwenden und gehen in den Messmodus Spektrum mit den folgenden Einstellungen: Modus, Emission; Schlitz Breite Anregung und Emission, 3 nm; Empfindlichkeit, mittlere; Scan-Geschwindigkeit, 100 nm/min; Daten Tonhöhe, 0,5 nm; Erregung Wellenlänge, 465 nm; Emission, 600-800 nm. Durchführen Sie Autozero und Maßnahme.
- Notieren Sie Anregung Spektren mit der gleichen Probe und Ausrüstung intakt Energieübertragung aus alle Pigmente in Chl offenbaren ein (Abb. 2D). Einstellungen zu ändern: Modus, Erregung; Emissionswellenlänge, 675 nm; Erregung, 370-600 nm. Die Spektren aufnehmen.
- Korrigieren Sie mit einem Rhodamin-Spektrum in der gleichen Größenordnung für die spektralen Eigenschaften der Lampe – Cf. Anweisungen in der Bedienungsanleitung.
- BKP Mix Proben entspricht 1 µg Chl eine mit 10 µL SDS-laden-Puffer. 10 min bei 25 ° c inkubieren Zentrifuge für 5 min bei 12000 X g in einer Table-Top-Zentrifuge.
- Laden Sie den überstand auf eine Tris-Tricine Gel28. 2 h bei 150 V zu trennen Sie und nach Trennung40Silber-Fleck.
Hinweis: Die FCP-Untereinheiten getrennt in zwei prominente Protein Bänder zwischen 18-19 kDa, die Bestandteile des FCPa und FCPb29 (Abbildung 2E) sind.
- Laden Sie den überstand auf eine Tris-Tricine Gel28. 2 h bei 150 V zu trennen Sie und nach Trennung40Silber-Fleck.
- Notieren Sie die Absorption zwischen 370-750 nm 5 µL des FCP in 995 µL B1a nach Schritt 1.2.5. Vergleichen Sie es mit dem in Schritt 1.2.4 erhaltenen Spektrum. Verwenden Sie die gleichen Instrumente und Einstellungen wie in Schritt 1.2.4 beschrieben.
2. Vorbereitung von Liposomen und Eingliederung des FCP
-
Vorbereitung der Lipid-Mischung und Lipid Waschmittel Mizellen
Hinweis: Lipide sind anfällig für warme Temperaturen kombiniert mit oxidativen Bedingungen. Versuchen Sie, die Lipide gekühlt und unter einer N-2 -Atmosphäre zu halten.- Berechnen Sie die gewünschte Thylakoidmembran Lipid-Verhältnisse für C. Meneghiniana laut Vieler Et Al. 200730. Siehe das Beispiel in Supplemental Tabelle 1. Bereiten Sie die Lipid Stammlösungen der vom Hersteller empfohlenen in einem Lösungsmittel Behältnis.
- Pipette die gewünschte Menge von Lipiden in einer 2 mL Reaktionsgefäß und verdunsten der Chloroform mit einem sanften Stickstoff-Flow und versuchen, die Lipide über die gesamte Fläche der Röhre Basis zu verteilen. Lassen Sie die N2 fließen, bis alle Lösungsmittel verdunstet ist.
- Solubilisieren Sie die Lipid-Mischung in 29 µL n-Octyl-β-D-Glucopyranoside-Lösung (OG) bei 4 ° C für 4 h Inkubation die Lipid-Mischung für 10 min bei 30 ° C. Inkubieren Sie die Lipide in einem sonikator Bad für 3 x 3 min bei 25 ° C unterbrochen von 30 s auf dem Eis.
- Fügen Sie 221 µL Trizin--Puffer und 250 µL 4 X Dialyse-Puffer.
- Verwenden Sie einen Extruder mit 0,1 µm-Polycarbonat-Membranen für einen definierten Liposomen-Durchmesser von 50-70 nm. Montieren Sie den Extruder mit der Membrane und Filter Unterstützung. Vermeiden Sie Luftblasen, und ziehen Sie die Baugruppe gründlich.
- Füllen Sie eine Spritze mit 4 X-Dialyse-Puffer und vornässen des Extruders, bis keine Luftblasen in der zweiten Spritze gesehen werden können.
- Gelten die Lipid-Reinigungsmittel-Mizellen für Extruder und drücken Sie die Lösung aus einer Spritze in die andere her und zurück. Wiederholen Sie diesen Schritt 5 Mal, bis die Lösung homogen erscheint.
Hinweis: Diese Lösung kann für mehrere Tage bei 4 ° C aufbewahrt werden. Nicht einfrieren!
-
Einbeziehung der FCP-komplexe und Entfernung von Waschmitteln und Aggregate
Hinweis: In diesem Beispiel verwenden wir ein Lipid/Chl Verhältnis 12:1, was eine Lipid-Eiweiß-Verhältnis von etwa 100: 1 entspricht.- Fügen Sie gleich 20 µg Chl FCP ein in einem Gesamtvolumen von 500 µL B1a Puffer zu 250 μL der extrudierten Lipid-Micellen und 250 µL 4 x Dialyse-Puffer. Inkubieren Sie die Proben 3 x 3 min bei 25 ° C in einem Thermomixer bei 1.500-3.000 u/min, unterbrochen von einer 30 s Pause auf dem Eis.
- Schneiden Sie den Deckel des vier 1,5 mL Reaktionsgefäß knapp unter der Spitze geben einen Ring, der noch auf dem Deckel passt. Bereiten Sie 1,5 x 1,5 cm Stücke der Dialysemembran vor und waschen Sie sie in 20 mL 1 X Dialyse-Puffer
- Füllen Sie 250 µL der Probe zu jeder Deckel. Legen Sie die Membran vorsichtig, auf dem Deckel, damit das Fach mit der Probe vollständig gefüllt ist und keine Luftblasen entstehen. Die Reaktion Rohr Ring auf der Versammlung zu verschärfen, um ein geschlossenes Fach haben.
- Die Proben in 50 mL 1 x Dialyse-Puffer über Nacht (12-16 h) auf Eis auf eine taumelnde Shaker Dialyse. Ersetzen Sie den verwendeten Dialyse-Puffer mit frischen und fügen Sie 7 mg des Adsorbens Perlen, die restlichen Waschmittel für mindestens 6 h zu entfernen.
- Den Dialyse-Puffer wieder ersetzen und Dialyse für eine weitere 12 Std. wieder die Liposomen durch piercing die Dialysemembran mit 200 µL Mikropipette Spitze und Aspirieren Sie alle Liposomen aus dem Reaktion Rohr Deckel.
- Optionaler Schritt: Wenn hoher Reinheit (> 95 %) ist erforderlich. Vorbereiten einer diskontinuierlichen Dichtegradienten in 17 mL Ultrazentrifugation Fläschchen mit Schritten, enthält 6 %, 10 %, 15 % und 20 % Saccharose Hitzesensoren Copolymer in Dialyse-Puffer. Laden Sie die Liposomen auf der Oberseite und Ultrazentrifugen bei 100.000 x g für 4 h in einem schwingenden Eimer-Rotor.
- Erholen Sie die oberen braunen Band mit einer Spritze zu, verdünnen Sie der Probe 1:5 mit DP und mit dem nächsten Schritt fortfahren.
- Zentrifugieren der FCP-Liposomen in mindestens 2 mL 1 x Dialyse-Puffer für 1,5 h bei 100.000 x g und 4 ° C. Die Liposomen durch drehen die Zentrifugenröhrchen in einem Winkel von 45° zu erholen. Lassen Sie die Liposomen für 1 min (Abbildung 3A) nach unten zu bewegen.
- Wiederherstellen der FCP-Liposomen in einem Endvolumen von 25-50 µL. nicht zu stören den Niederschlag.
-
Regler 1: Absorption, Fluoreszenz-Spektroskopie
- Fügen Sie eine Aliquote 3 µL der FCP-Liposomen zu einem Endvolumen von 1 mL 1 x Dialyse-Puffer und Zentrifuge für 5 min bei 12.000 x g.
- Notieren Sie die Absorption zwischen 370 und 750 nm der FCP-Liposomen mit gleicher Ausstattung wie unter 1.2.4. Normalisieren Sie das Spektrum, das Maximum in der Q-y -Region des Chl (670-680 nm) zu und vergleichen sie das normalisierte Spektrum der solubilisiert FCP (Abbildung 3).
- FCP-Liposomen in 1 mL der DP mit einer Extinktion (ABS) vorbereiten = 0,03 in Bezug auf das Maximum zwischen 670-680 nm. Passen Sie die solubilized FCP in Waschmittel aus Schritt 1.2.6 die gleichen ABS verdünnen mit B1a.
- Nehmen Sie 1.2.4 beschrieben Absorption Spektren beider Proben auf. Zeichnen Sie auf, Chl ein Fluoreszenz-Emissionsspektrum beider Proben in 1.3.3 beschrieben.
Hinweis: Die Fluoreszenz-Rendite sinkt in der FCP-Liposomen-Probe (Abbildung 3D und vgl. Diskussion.)
- Fügen Sie eine Aliquote 3 µL der FCP-Liposomen zu einem Endvolumen von 1 mL 1 x Dialyse-Puffer und Zentrifuge für 5 min bei 12.000 x g.
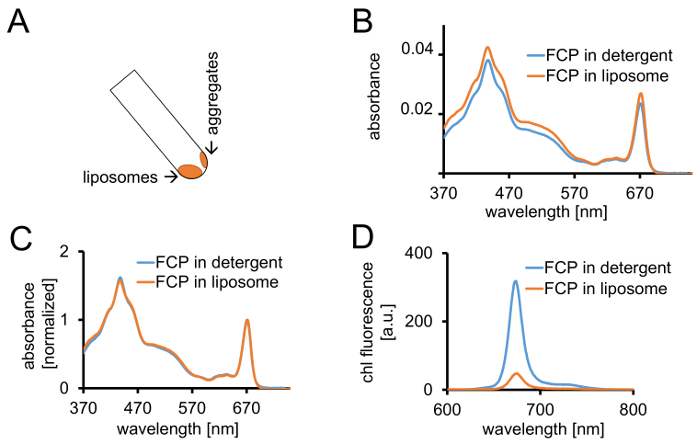
Abbildung 3: Isolierung von FCP Proteoliposomes gefolgt von spektroskopischen Kontrollen und konfokale Bildgebung. (A) Erholung der FCP Liposomen nach Zentrifugation. Drehen Sie die Zentrifugation Röhre auf 45° und warten Sie ca. 1 min - Liposomen bewegt sich nach unten, während der FCP aggregiert die Liposomen-Stick an der Rohrwand nicht berücksichtigt sind. (B) Vergleich der Absorption Spektren von solubilisiert FCP in Waschmittel (blau) und FCP in Liposomen (Orange) (C) die gleiche Spektren wie in (B) normiert auf Chl maximal in den roten Bereich (~ 670 nm - Qy Peak); solubilisiert FCP in Waschmittel (blau) und FCP in Liposomen (Orange). Potenziell, gäbe es ein Pigmentverlust hauptsächlich der Carotinoide sichtbar im Bereich 500-550 nm. Das clustering von FCP in Liposomen führen zu einer Peak Verbreiterung und eine leichte Verschiebung der Chl maximal (~ 670 nm) auf dem roten. (D) Emissionsspektren solubilisiert FCP in Waschmittel und FCP in Liposomen. Clustering von FCP in den Liposomen erhöht energetischen Interaktionen der FCP-komplexe, die senkt der Fluoreszenz-Ausbeute (orange Kurve) und verschiebt sich die Emission Maxima leicht den roten. Bitte klicken Sie hier für eine größere Version dieser Figur.
Ergebnisse
Das Protokoll beschreibt die Isolierung des gesamten FCP-Fraktion aus Cyclotella Meneghiniana und Einbindung in Liposomen mit native lipidzusammensetzung. Die Thylakoidmembran Isolation ist hoch reproduzierbare, aber die Thylakoidmembran Ausbeute kann sich ändern. Das Ergebnis ist akzeptabel, wenn mehr als 50 % aller Pigmente im Schritt 1.1.4 wiederhergestellt werden. Mehr als 80 % ist optimal.
Die Solubilisierung der ...
Diskussion
FCP Liposomen mit natürlichen lipidzusammensetzung bieten eine praktische, einfache und reproduzierbare Werkzeug, um spektroskopische Eigenschaften zu untersuchen in-vitro-. Die Lipid-Umwelt in FCP Liposomen ähnelt die Situation innerhalb der Thylakoidmembran Membran, was zu experimentellen Ergebnissen, die näher an der natürlichen Bedingungen sind.
Es gibt mehrere Vorteile bei der Verwendung von C. Meneghiniana als Modellsystem für FCP-Antenne. Es wächst relativ schnel...
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Wir danken für die Unterstützung bei FCP Reinigung Rana Adeel Ahmad. Prof. Claudia Büchel ist für hilfreiche Diskussionen anerkannt und das Manuskript zu lesen. Diese Arbeit wurde von der Deutschen Forschungsgemeinschaft, LD (DI1956-1/1) und der Humboldt-Stiftung für ein Feodor-Lynen-Forschungsstipendium für LD unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 500 ml centrifuge vials | |||
| high speed centrifuge | Heraeus | ||
| Bead Mill VI 2 | Edmund-Bühler (edmund-buehler.de) | newer version: Vibrogen-Zellmühle Vl 6 | |
| Silibeads S 400 µm | Sigmund-Lindner.com | 5223-7 | |
| Silibeads S 1,-1,3 mm | Sigmund-Lindner.com | 4504 | |
| VitraPOR filter funnel - por1 | ROBU GmbH | 21121 | |
| polycarbonate ultracentrifuagtion vials (30 mL) for T-865 | Beranek Laborgeräte (Laborgeraete-beranek.de) | 314348 | |
| Ultracentrifuge Discovery 90SE | Sorvall | n.a. | |
| rotor T 865 | ThermoFisher Scientific (thermofisher.com) | 51411 | |
| Neubauer Cell Counter Chamber (improved) | Carl Roth Laborbedarf (Carlroth.com) | T729.1 | |
| Zeiss Mikroskop Primostar (7) | Optik-Pro (optik-pro.de) | 51428 | |
| optical glass cuvettes (6040-OG) | Hellma Analytics (hellma-analytics.com) | "6040-10-10" | |
| V-630 UV-VIS Spectrophotometer (incl. software) | Jasco (jasco.de) | V-630 | |
| n-Dodecyl-β-D-Maltopyranoside | ANATRACE (anatrace.com) | D310LA | |
| Ultra-Clear tubes 17 ml for AH629 | Beranek Laborgeräte (Laborgeraete-beranek.de) | 344061 | |
| rotor AH629-17-mL | ThermoFisher Scientific (thermofisher.com) | 54285 | |
| Membrane concentrator_Centriprep 30 kDa cutoff | Millipore (merckmillipore.com) | 4307 | |
| Biometra Minigel-Twin | Analytik Jena AG (analytik-jena.de) | 846-010-100 | |
| Silver Stain Plus Kit | Bio-Rad (bio-rad.com) | 1610449 | |
| libre office spread sheet | The document foundation | https://de.libreoffice.org/download/libreoffice-still/ | |
| special glass cuvettes for fluorescence (101-0S) | Hellma Analytics (hellma-analytics.com) | 101-10-20 | |
| Spectrofluorometer FP-6500 (incl. Software) | Jasco (jasco.de) | FP-6500 | |
| SDS-loading buffer Roti-Load | ROTH (carlroth.com) | K929.1 | |
| n-octyl β-D-glucopyranoside | ANATRACE (anatrace.com) | O311 | |
| Monogalactosyl Diaclyglycerol (MGDG) | Larodan AB (larodan.com) | 59-1300 | make stock solution in chloroform |
| Digalactosyl Diacylglycerol (DGDG) | Larodan AB (larodan.com) | 59-1310 | make stock solution in chloroform |
| Sulphoquinovosyl Diacylglycerol (SQDG) | Larodan AB (larodan.com) | 59-1230 | make stock solution in chloroform |
| L-alpha-Phosphatidylglycerol (PG) | Larodan AB (larodan.com) | 37-0150 | make stock solution in chloroform |
| L-α-Phosphatidylcholine | Sigma-Aldrich (sigmaaldrich.com) | P3782 SIGMA | make stock solution in chloroform |
| sonicator bath S-50TH | Sonicor (getmedonline.com | SONICOR-S-50TH | |
| mini-Extruder | Avanti Polar Lipids (Avanti.com) | 610000 | |
| Nuleopore polycarbonate membrane | Avanti Polar Lipids (Avanti.com) | 610005 | |
| dialysis membrane Visking 14 kDa cutoff | ROTH (carlroth.com) | 0653.1 | boil in destilled water before use |
| Biobeads SM2 Adsorbent | Biorad (Bio-rad.com) | 152-3920 | |
| sucrose epichlorhydrin copolymer - Ficoll 400 | Sigma-Aldrich (sigmaaldrich.com) | F4375 | |
| Polycarbonate ultracentrifuagtion vials (2.7 mL) for TFT 80.4 | Beranek Laborgeräte (Laborgeraete-beranek.de) | 252150 | |
| rotor TFT 80.4 | Millipore (merckmillipore.com) | 54356 | |
| material listed in order of appearance | For specific safety instructions please refer to material safety sheets and repective manuals. Standard lab material and substances are not listed. |
Referenzen
- Eberhard, S., Finazzi, G., Wollman, F. A. The Dynamics of Photosynthesis. Annual Review of Genetics. 42, 463-515 (2008).
- Li, Z. R., Wakao, S., Fischer, B. B., Niyogi, K. K. Sensing and Responding to Excess Light. Annual Review of Plant Biology. 60, 239-260 (2009).
- Niyogi, K. K., Truong, T. B. Evolution of flexible non-photochemical quenching mechanisms that regulate light harvesting in oxygenic photosynthesis. Current Opinion in Plant Biology. 16 (3), 307-314 (2013).
- Li, X. -. P., et al. A pigment-binding protein essential for regulation of photosynthetic light harvesting. Nature. 403 (6768), 391-395 (2000).
- Peers, G., et al. An ancient light-harvesting protein is critical for the regulation of algal photosynthesis. Nature. 462 (7272), 518-521 (2009).
- Correa-Galvis, V., et al. Photosystem II Subunit PsbS Is Involved in the Induction of LHCSR Protein-dependent Energy Dissipation in Chlamydomonas reinhardtii. The Journal of biological chemistry. 291 (33), 17478-17487 (2016).
- Bailleul, B., et al. An atypical member of the light-harvesting complex stress-related protein family modulates diatom responses to light. Proceedings of the National Academy of Sciences of the United States of America. 107 (42), 18214-18219 (2010).
- Taddei, L., et al. Multisignal control of expression of the LHCX protein family in the marine diatom Phaeodactylum tricornutum. Journal of experimental botany. 67 (13), 3939-3951 (2016).
- Lepetit, B., et al. The diatom Phaeodactylum tricornutum adjusts nonphotochemical fluorescence quenching capacity in response to dynamic light via fine-tuned Lhcx and xanthophyll cycle pigment synthesis. New Phytologist. 214 (1), 205-218 (2017).
- Büchel, C. Evolution and function of light harvesting proteins. Journal of Plant Physiology. 172, 62-75 (2015).
- Lavaud, J., Rousseau, B., van Gorkom, H. J., Etienne, A. -. L. Influence of the Diadinoxanthin Pool Size on Photoprotection in the Marine Planktonic Diatom Phaeodactylum tricornutum. Plant Physiology. 129 (3), 1398-1406 (2002).
- Ruban, A., et al. The super-excess energy dissipation in diatom algae: comparative analysis with higher plants. Photosynthesis Research. 82 (2), 165-175 (2004).
- Mann, D. G. The species concept in diatoms. Phycologia. 38 (6), 437-495 (1999).
- Papagiannakis, E., van Stokkum, I. H. M., Fey, H., Büchel, C., van Grondelle, R. Spectroscopic Characterization of the Excitation Energy Transfer in the Fucoxanthin-Chlorophyll Protein of Diatoms. Photosynthesis Research. 86 (1-2), 241-250 (2005).
- Premvardhan, L., Robert, B., Beer, A., Büchel, C. Pigment organization in fucoxanthin chlorophyll a/c2 proteins (FCP) based on resonance Raman spectroscgopy and sequence analysis. Biochimica et Biophysica Acta-Bioenergetics. 1797 (9), 1647-1656 (2010).
- Gildenhoff, N., Herz, J., Gundermann, K., Büchel, C., Wachtveitl, J. The excitation energy transfer in the trimeric fucoxanthin-chlorophyll protein from Cyclotella meneghiniana analyzed by polarized transient absorption spectroscopy. Chemical Physics. 373 (1), 104-109 (2010).
- Ramanan, C., et al. Exploring the mechanism(s) of energy dissipation in the light harvesting complex of the photosynthetic algae Cyclotella meneghiniana. Biochimica et Biophysica Acta-Bioenergetics. 1837 (9), 1507-1513 (2014).
- Haferkamp, S., Kirchhoff, H. Significance of molecular crowding in grana membranes of higher plants for light harvesting by photosystem II. Photosynthesis Research. 95 (2-3), 129-134 (2008).
- Wahadoszamen, M., et al. Stark fluorescence spectroscopy reveals two emitting sites in the dissipative state of FCP antennas. Biochimica et Biophysica Acta-Bioenergetics. 1837 (1), 193-200 (2014).
- Zhou, F., et al. Effect of monogalactosyldiacylglycerol on the interaction between photosystem II core complex and its antenna complexes in liposomes of thylakoid lipids. Photosynthesis Research. 99 (3), 185-193 (2009).
- Moya, I., Silvestri, M., Vallon, O., Cinque, G., Bassi, R. Time-resolved fluorescence analysis of the photosystem II antenna proteins in detergent micelles and liposomes. Biochemistry. 40 (42), 12552-12561 (2001).
- Natali, A., et al. Light-harvesting Complexes (LHCs) Cluster Spontaneously in Membrane Environment Leading to Shortening of Their Excited State Lifetimes. Journal of Biological Chemistry. 291 (32), 16730-16739 (2016).
- Gundermann, K., Büchel, C. Factors determining the fluorescence yield of fucoxanthin-chlorophyll complexes (FCP) involved in non-photochemical quenching in diatoms. Biochimica et Biophysica Acta-Bioenergetics. 1817 (7), 1044-1052 (2012).
- Ahmad, R. A., Dietzel, L. Relaxation of cellular K+ gradients by valinomycin induces diatoxanthin accumulation in Cyclotella meneghiniana cells and alters FCPa fluorescence yield in vitro. Physiologia Plantarum. , 171-180 (2017).
- Provasoli, L., McLaughlin, J. J. A., Droop, M. R. The development of artificial media for marine algae. Archiv für Mikrobiologie. 25 (4), 392-428 (1957).
- Jeffrey, S., Humphrey, G. New spectrophotometry equations for determining chlorophyll a, chlorophyll b, chlorophyll c-1 and chlorophyll c-2 in higher plants, algae and natural phytoplankton. Biochemie und Physiologie der Pflanzen. 167, 191-194 (1975).
- Beer, A., Gundermann, K., Beckmann, J., Büchel, C. Subunit Composition and Pigmentation of Fucoxanthin−Chlorophyll Proteins in Diatoms: Evidence for a Subunit Involved in Diadinoxanthin and Diatoxanthin Binding. Biochemistry. 45 (43), 13046-13053 (2006).
- Schägger, H., von Jagow, G. Tricine-sodium dodecyl sulfate-polyacrylamide gel electrophoresis for the separation of proteins in the range from 1 to 100 kDa. Analytical Biochemistry. 166 (2), 368-379 (1987).
- Büchel, C. Fucoxanthin-Chlorophyll Proteins in Diatoms: 18 and 19 kDa Subunits Assemble into Different Oligomeric States. Biochemistry. 42 (44), 13027-13034 (2003).
- Vieler, A., Wilhelm, C., Goss, R., Süß, R., Schiller, J. The lipid composition of the unicellular green alga Chlamydomonas reinhardtii and the diatom Cyclotella meneghiniana investigated by MALDI-TOF MS and TLC. Chemistry and Physics of Lipids. 150 (2), 143-155 (2007).
- Gundermann, K., Büchel, C. The fluorescence yield of the trimeric fucoxanthin-chlorophyll-protein FCPa in the diatom Cyclotella meneghiniana is dependent on the amount of bound diatoxanthin. Photosynthesis Research. 95 (2-3), 229-235 (2008).
- Miloslavina, Y., et al. Ultrafast fluorescence study on the location and mechanism of non-photochemical quenching in diatoms. Biochimica et Biophysica Acta-Bioenergetics. 1787 (10), 1189-1197 (2009).
- Grouneva, I., Jakob, T., Wilhelm, C., Goss, R. The regulation of xanthophyll cycle activity and of non-photochemical fluorescence quenching by two alternative electron flows in the diatoms Phaeodactylum tricornutum and Cyclotella meneghiniana. Biochimica et Biophysica Acta-Bioenergetics. 1787 (7), 929-938 (2009).
- Chukhutsina, V. U., Büchel, C., van Amerongen, H. Disentangling two non-photochemical quenching processes in Cyclotella meneghiniana by spectrally-resolved picosecond fluorescence at 77 K. Biochimica et Biophysica Acta-Bioenergetics. 1837 (6), 899-907 (2014).
- Ghazaryan, A., Akhtar, P., Garab, G., Lambrev, P. H., Büchel, C. Involvement of the Lhcx protein Fcp6 of the diatom Cyclotella meneghiniana in the macro-organisation and structural flexibility of thylakoid membranes. Biochimica Et Biophysica Acta-Bioenergetics. 1857 (9), 1373-1379 (2016).
- Darley, W. M. Biochemical composition. The biology of diatoms. 13, 198-223 (1977).
- Milsman, M. H. W., Schwendner, R. A., Weder, H. G. Preparation of large single bilayer liposomes by a fast and controlled dialysis. Biochimica Et Biophysica Acta. 512 (1), 147-155 (1978).
- Zumbuehl, O., Weder, H. G. Liposomes of controllable size in the range of 40 to 180 nm by defined dialysis of lipid-detergent-mixed micelles. Biochimica Et Biophysica Acta. 640 (1), 252-262 (1981).
- Verchere, A., Broutin, I., Picard, M. Photo-induced proton gradients for the in vitro investigation of bacterial efflux pumps. Scientific Reports. 2 (306), (2012).
- Chevallet, M., Luche, S., Rabilloud, T. Silver staining of proteins in polyacrylamide gels. Nature Protocols. 1 (4), 1852-1858 (2006).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten