Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Kalibrierung-kostenlose In-vitro- Quantifizierung von Protein Homo-Oligomerisierung mit kommerziellen Instrumentierung und kostenlose Open Source-Helligkeit-Analyse-Software
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt einen Kalibrierung-freie Ansatz zur Quantifizierung der Protein Homo-Oligomerisierung in Vitro basierend auf Fluoreszenzspektroskopie Fluktuation mit kommerziellen Licht Rasterelektronenmikroskopie. Die richtige Übernahme Einstellungen und Analysemethoden werden angezeigt.
Zusammenfassung
Anzahl und Helligkeit ist eine Kalibrierung-freie Fluoreszenz Fluktuation Spektroskopie (FFS) Technik zum Nachweis von Protein Homo-Oligomerisierung. Es kann eingesetzt werden, mit einem herkömmlichen confocal Mikroskop mit digitaler Detektoren ausgestattet. Ein Protokoll für den Einsatz von Technik in Vitro wird mittels einem Use Case gezeigt wo Zahl und Helligkeit zu genau den Oligomeren Stand der mVenus versehenen FKBP12F36V vor und nach der Zugabe der dimerizing Droge AP20187 quantifizieren gesehen werden kann. Die Bedeutung der Verwendung der richtigen Mikroskop Aufnahmeparameter und die korrekten Daten-Vorverarbeitung und Analyse-Methoden werden diskutiert. Insbesondere wird die Bedeutung der Wahl der Immunofluoreszenz Korrektur betont. Diese kostengünstige Methode kann verwendet werden, um Protein-Protein-Interaktionen in vielen biologischen zusammenhängen zu studieren.
Einleitung
Protein-Protein Interaktionen ich n-vitro-
Traditionell sind die Kristallographie und Kernspinresonanz-Experimenten mit Kryo-Elektronenmikroskopie (CryoEM) kombiniert die Technologien entschieden, genau zu beschreiben, die dreidimensionale Architektur der Proteine und ihre Funktion durch Ableiten hinterfragen ihre hochauflösende strukturelle Details. Proteine, jedoch sind keine statischen Strukturen und können eine Vielzahl von Konformationsänderungen und Schwingungen in Zeit und Raum zu unterziehen. Deshalb ist die strukturelle Informationen aus kristallographischen oder CryoEM Daten müssen mit anderen Techniken (z. B. Molekulardynamik-Simulationen und Einzelmolekül-Techniken) ergänzt werden: die Funktion eines Proteins ist im Zusammenhang mit seiner Konformationsänderungen Änderungen und Interaktionen und diese Information ist nicht vorhanden in einer statischen Struktur. Um intramolekularer Dynamik untersuchen, sind Techniken, basierend auf einzelnen Moleküls Forster Resonanz Energietransfer (SmFRET) sehr effektiv1. Diese Ansätze sind verschiedene Subpopulationen von Molekülen in komplexen Medien beurteilen können. Dies ist sehr wichtig, da diese Änderungen schnelle sind und während der Erfassung der Daten (d. h. Nanosekunde, zweite Reihe treten).
Zwei Hauptansätze werden häufig eingesetzt, um zu erkennen und diese Änderungen zu quantifizieren: Proteine in Lösung und Oberfläche-Immobilisierung. Für den Nachweis der Inter molekularen Wechselwirkungen und insbesondere den Prozess der Dimerisierung von Liganden induzierte, ist SmFRET nicht immer das beste Werkzeug. In der Tat Bund richtet sich nicht nur auf der Strecke (≈10 nm), sondern auch auf die Ausrichtung der beiden Dipole (Donor und Akzeptor, χ2) und die Überlappung der Spender-Emission mit den Akzeptor Absorptionsspektren2, aber vielleicht diese letzte Bedingung ist weniger vorausgesetzt, dass der Experimentator kann wählte wichtig, das richtige Paar Bund. Ein besonderer Nachteil von SmFRET zum Sondieren Homo-Dimerisierung stammt aus der Kennzeichnung des Proteins des Interesses: für Hetero SmFRET Dimerisierung kann nur festgestellt, bis zu 50 % (d.h., Hetero-FRET werden nur Donor-Akzeptor zu erkennen und Akzeptor-Spender Homo-Dimere aber nicht Spender Spender oder Akzeptor-Akzeptor, die die anderen 50 % von der Dimere). Die Verwendung von Fluoreszenz Korrelation Spektroskopie (FCS) und Derivaten (FCC, etc.3), Protein Verbreitung Konstanten und verbindliche Konstanten in Vitro zu ermitteln ist eine weitere Alternative. Diese Ansätze sind nicht in der Lage, vollständig Homo-Dimerisierung entweder zu quantifizieren, wie FCS 1 Maßnahmen Konzentration und Diffusion, und den Radius und die Verbreitung Koeffizienten eines diffundierende Teilchen sind sehr schlecht das Molekulargewicht abhängig; zum Beispiel wird ein 10-divisibel Anstieg des Molekulargewichtes nur eine 2.15 Falte Änderung in der Verbreitung Koeffizient4verbunden. Bei zweifarbigen FCS oder FCC werden nur 50 % der Homo-Dimere aus dem gleichen Grund wie oben gesehen. Praktische und quantitative Ansätze, Homo-Dimerisierung in Vitro und in Vivo zu erkennen sind Homo-Bund5 und der Anzahl und der Helligkeit (N & B)6. Angesichts der Tatsache, dass Homo-FRET erfordert spezielle Instrumente Erholung von der Anisotropie-Wert (z. B. optische Elemente/Analyzer, die parallele und senkrechte Polarisation wiederherzustellen), N & B präsentiert hier als eine günstige Methode, um erkennen Sie Protein Homo-Dimerisierung und Aggregation. Es kann sein, beschäftigt, sowohl in Vitro und in Vivo eine kommerzielle Einrichtung.
Anzahl und Helligkeit
N & B wurde erst kürzlich überarbeitete7. Dieser Beitrag konzentriert sich auf die Anwendung der Technik in lebenden Zellen. Es lohnt sich, reproduzieren die mathematische Formalismus hier als diese Gleichungen auf die Daten angewendet werden gesammelt in Vitro. Erstens ist es notwendig, einige Begriffe und mathematische Mengen zu definieren:
- Eine Entität ist eine Reihe von Molekülen, die miteinander verbunden sind.
- Die Helligkeit ε einer Entität ist die Anzahl der Photonen, die es pro Zeiteinheit (pro Frame) aussendet.
- n ist die Anzahl der Elemente vorhanden.
- Für eines bestimmten Pixels im Laufe einer Bilderserie ist < i > seine mittlere Intensität und σ2 die Varianz in ihrer Intensität.
Dann, mit Photon counting Detektoren und vorausgesetzt, mobile Einheiten und keinen Hintergrund,
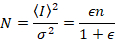
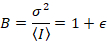
Dabei ist N die scheinbare Zahl und B ist die scheinbare Helligkeit. Dies führt zu
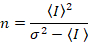
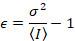
Dalal Et al. 8 zeigte, dass mit analogem Equipment, man drei Korrektur Begriffe braucht: S-Faktor, der Hintergrund- Offsetund die Auslesen Lärm σ02. Dann wieder vorausgesetzt, mobile Einheiten,
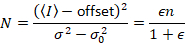
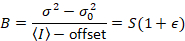
Geben
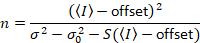

Beachten Sie, dass die obige Gleichung für unterscheidet, die in Dalal Et Al. 8 und einer nachträglichen Prüfung. 7 in Dalal Et Al. S im Nenner wurde durch einen Tippfehler verzichtet und dieser Fehler wurde bei der Überprüfung reproduziert. Die obigen Gleichung ist die richtige. Anweisungen für die Messung von S, Offset und σ02 - zusammen mit einer Erläuterung ihrer Bedeutung - sind gegeben durch Dalal Et Al. 8
Die Helligkeit ε ist proportional zu den Oligomeren Zustand der diffundierende Entitäten: ε werden zweimal für Dimere so groß, wie es für Monomere, dreimal so groß für Trimere ist, wie es für Monomere, für Hexamers doppelt so groß ist wie für Trimere und so weiter. Auf diese Weise messen die Helligkeit ε kann man jede Art von Multimerization quantifizieren.
Gibt es eine Mischung aus Oligomeren Staaten vorhanden, ist Anzahl und Helligkeit nicht in der Lage erholt die Oligomeren Einzelstaaten vorhanden. Dies ist eine Einschränkung der Technik.
Detrend-Algorithmus und Nandb software
Die Bedeutung der Korrektur für Immunofluoreszenz wurde betont zuvor9. Immunofluoreszenz tritt unweigerlich während der Lichtmikroskopie Experimente im Zeitraffer-Modus; sowohl in lebenden Zellen und in Vitro. Viele Ansätze wurden in der Literatur zu korrigieren zum Bleichen7beschrieben. Die exponentielle filternde Technik mit automatischer Wahl der detrending Parameter T ist die derzeit besten. Es ist kostenlos, open Source Software Nandb9integriert. In der Tat kann Software, die die Benutzer manuell wählen ihre detrending Parameter erfordert zu falschen Ergebnissen führen, da diese Parameter Wahl wahrscheinlich willkürlich und falsch sind. Der Automatische Algorithmus prüft die Daten und bestimmt den entsprechenden Parameter, ohne die Notwendigkeit der Benutzer eingreifen9. Auch mit die beste Wahl von glättende Parameter detrending hat seine Grenzen und funktioniert auch nur mit Immunofluoreszenz Prozentsätze niedriger als 25 %, wie abgebildet, mit Simulationen9. Interessant ist, wenn Sie die automatische detrending Routine zu verwenden, die Genauigkeit ist so, dass man mit geringer Helligkeitswerten arbeiten kann (sogar B < 1.01), und somit geringe Intensitäten, und immer noch präzise genug, um Homo-Dimerisierung zu quantifizieren.
Immunofluoreszenz verursacht auch ein weiteres Problem: die Anwesenheit von Photobleached Fluorophore in einem komplexen Multimer. Dies macht z. B. ein Trimer erscheinen wie ein Dimer, wenn eines der drei Geräte in das Trimer nicht fluoreszierenden ist. Hur und Mueller10 zeigte, wie man dies zu korrigieren und diese Korrektur wurde auch in einer nachträglichen Prüfung7hervorgehoben. Die Nandb-Software enthält diese Korrektur-9.
Die FKBP12 F36V System
FKBP12F36V ist ein Protein, das natürlich nicht oligomerize aber bekannt ist, auf den Zusatz von AP20187 Medikamenten (umgangssprachlich bekannt als die BB dimerizing Liganden)11,12Dimere. Dies macht es ein idealer Prüfstein für Anzahl und Helligkeit: mit beschrifteten FKBP12F36V, eine Verdoppelung der Oligomere Zustand nach Zugabe von BB beobachtet werden sollte.
Protokoll
(1) FKBP12 F36V - mVenus Reinigung
- Transformieren Sie (DE3) pLysS Zellen mit pET22b Vektor, monomerized menschlichen FKBP12F36V12 und N-terminale His6 und mVenus-Tags (Vektor auf Anfrage erhältlich) enthält. Platte Zellen auf LB-Agar mit 50 µg/mL Ampicillin und 34 µg/mL Chloramphenicol ergänzt.
- Transformierte Kolonien in 100 mL LB Starterkultur und wachsen für 16-20 Stunden bei 37 ° C mit schütteln.
- Verdünnen Sie dichten Starterkultur (OD600 > (1) 1: 100 in LB-Medium (2 x 500 mL Chargen) und wachsen für 2-3 Stunden zu OD600 = 0,6 - 0,8 (37 ° C, 200 u/min).
- Cool Kulturen auf Eis. Mit 250 µM IPTG induzieren und 16-20 Stunden bei 21 ° C, 200 u/min zu wachsen.
- Ernten Sie Zellen durch Zentrifugation bei 2.000 x g für 20 Minuten.
- Aufschwemmen Sie Pellet in 40 mL IMAC Puffer ein (20 mM Natriumphosphat pH 7.5, 500 mM NaCl, 3 mM Imidazol, 1 mM β-Mercaptoethanol) mit EDTA-freie Protease-Inhibitoren (1 Tablette pro Zelle Pellet) ergänzt.
- Beschallen Zellen (500 Watt, 20 kHz, 40 % Amplitude, 9 s auf, 11 s aus für 15 min) auf Eis und Ernte lösliche Material durch Zentrifugation bei 20.000 x g.
- Transfer lösliche lysate zu einem konischen Kolben und 2 mL Harz (siehe Tabelle der Materialien). 1 Stunde mit 105 u/min inkubieren
Hinweis: Nickel Sepharose kann auch für diesen Schritt IMAC verwendet werden. - Ernten Sie Harz zu und waschen Sie mit 250 mL IMAC Puffer A, gefolgt von 500 mL IMAC Puffer B (20 mM Natriumphosphat pH 7.5, 500 mM NaCl, 7 mM Imidazol, 1 mM β-Mercaptoethanol).
Hinweis: Erhöhen Sie um 50 mM Imidazol wenn Nickel Sepharose Harz verwenden. - Eluieren Sie His6-markierte Protein mit IMAC Puffer C (20 mM Natriumphosphat pH 7.5, 500 mM NaCl, 300 mM Imidazol, 1 mM β-Mercaptoethanol).
- Spritzen auf eine Größe Ausgrenzung Spalte (siehe Tabelle der Materialien) in 10 mM HEPES pH 7.5, 150 mM NaCl, 1 mM DTT equilibriert. FKBP12F36V hat seinen Höhepunkt Elution bei 87,71 mL auf die Spalte, die wir verwendet.
- Reinheit über SDS-PAGE und Pool zu beurteilen und bei Bedarf konzentrieren.
2. Vorbereitung der Multiwell Plate Array
- Auftauen der gereinigten FKBP12F36V (oder mit der Bezeichnung Protein des Interesses) von-80 ° C.
- Bereiten Sie eine Lösung von 100 nM gereinigt FKBP12F36V (mittlere, 10 mM HEPES pH 7.5, 150 mM NaCl, 1 mM DVB-t). Beschallen Sie und Zentrifugieren Sie (schnelle Runde von 13.000 u/min), um die Bildung von Aggregaten zu verhindern.
- Pipette 100-200 µL in eine Kammer 8 gut Beobachtung mit einem Glasboden.
- Hinzu kommt die BB Dimerizer Endkonzentrationen von 10, 20, 40, 80, 100, 150, 300 und 500 nM12,13.
- Als Referenz, bereiten Sie eine Lösung von 100 nM mVenus allein zur Aggregation und Niederschlag Auswirkungen zu bewerten und einen Helligkeitswert für das Monomer mit den gleichen Einstellungen Erwerb zu erholen.
3. Kalibrierung-freie konfokale Erwerb
- Starten Sie die konfokale System (Abbildung 1). Kein Licht Mikroskop konfokale Abtastsystem ausgestattet mit digitaler Detektoren oder gut charakterisierten analogen Detektoren8, und in der Lage zu halten eine Konstante Verweilzeit für jedes Pixel erworben würde funktionieren.
- Legen Sie den Strahlengang Erregung:
- Schalten Sie das 514 nm Laser und setzen es bei 20-100 nW Strom am Ausgang des Ziels (für FKBP12F36V-mVenus).
- Wählen Sie das 63X1.4NA Ziel oder ein Halsband Korrektur Wasser eintauchen Ziel für FCS entwickelt.
- Schalten Sie ein HyD, APD oder kalibrierten PMT-Detektor. Detektoren in der Lage, Photon counting sind vorzuziehen, da in diesem Fall, Berechnung von S, Offset und σ0-2 sind nicht erforderlich.
- Wählen Sie die Emissions-Fenster von 520-560 nm
- 1 luftig Einheit für die entsprechende Emission ~ 545 nm die Lochblende festgesetzt.
- Stellen Sie den Erwerb Modus mit 16 x 16 Pixel
- Die Pixel wohnen Zeit twohnen so eingestellt, dass es tRahmen erfüllt >> TD >> twohnen, wo D die Verweilzeit des diffundierende Protein- und t ist Rahmen ist die Frame-Rate. Das entsprach Einstellen der Haltezeit ~ 13 µs.
Hinweis: Einige kommerzielle Hersteller hatte Scanner, die nicht die Verweildauer pro Pixel konstant hielten. Diese Konstanz ist entscheidend für die Methode funktioniert. - Legen Sie die Pixelgröße bei ~ 120 nm.
- Wählen Sie den Xyt -Übernahme-Modus und wählen Sie die Anzahl der Frames pro Akquisition und gut (zum Beispiel 5.000) erworben werden.
- Wenn das System mit hohem Durchsatz-Modus ausgestattet ist, stellen Sie die Koordinaten jedes gut und die Zahl der Übernahmen pro Bohrloch um den Prozess zu automatisieren.
Hinweis: Achten Sie, die Anwesenheit von einem Wasserspender für Wenn ein Eintauchen-Ziel verwenden. - Wenn das System ist ausgestattet mit eine Perfusion System laden die BB-Lösung und Programmieren der Perfusion beginnen rechts-nach 5000th Rahmen um die Kinetik der Dimerisierung beim Erwerb von z. B. 10.000 Bilder zu bewerten.
- Einen Tropfen Öl in das Öl eintauchen Objektiv / Wasser wenn ein Halsband Korrektur Wasser eintauchen Ziel für FCS entwickelt.
- Montieren Sie die 8 gut Beobachtung Kammer in die Stufe.
- Wählen Sie den richtigen Brunnen und konzentrieren Sie sich auf die Lösung.
Hinweis: Wichtig: vermeiden Sie mit Schwerpunkt in der Nähe der unteren Glas, Reflexion und Streuung zu vermeiden. Wenn tiefer Konzentration in der Lösung, trennen Sie die Option automatische Fokus. - Starten Sie den Erwerb und speichern Sie die resultierenden Stapel Bilder im TIFF-Format zu.
4. detrend und Helligkeit Analyse mit der R-Paket-Nandb
- Als eine Vorverarbeitung Qualitätsprüfung verwenden Sie ImageJ14 werfen einen Blick auf die Bilder und die Intensität Profil wiederherstellen wie in Abbildung 2einegezeigt. Dies ist nützlich, um festzustellen, ob zuviel Immunofluoreszenz aufgetreten ist. Wenn es zu viel bleichen gibt, eignet sich die Daten nicht für weitere Analysen.
Hinweis: ImageJ kann auch Bilder in TIFF aus kommerziellen Formaten konvertieren nützlich sein. Die nachfolgend beschriebene Nandb-Software funktioniert nur mit TIFF-Dateien. - Downloaden Sie und installieren Sie R und RStudio15,16. Es empfiehlt sich, downloaden und installieren Sie R zuerst, dann RStudio.
Hinweis: Was folgt, ist eine Beschreibung, wie das Nandb R-Paket verwenden. R Sprachkenntnisse ist nicht erforderlich, verwenden Sie Nandb, aber es Leben einfacher machen wird. - Installieren Sie das Nandb-Paket.
- Öffnen Sie RStudio und geben Sie in der Konsole, install.packages("nandb") und warten Sie die Installation.
- Lernen Sie Nandb kennen
- Überprüfen Sie die manuelle17.
- Überprüfen Sie die integrierte RStudio-Hilfe für verschiedene Funktionen. Die wahrscheinlichste Funktion verwendet werden wird, mit brightness() werden. Anzeigen die Hilfe-Datei für diese Funktion durch Eingabe? Brightness() an der Konsole.
- Helligkeit zu berechnen
- Also muss man eine Image-Datei auf dem Desktop mit dem Namen img001.tif (Beachten Sie, dass "Nandb" nur mit TIFF-Dateien funktioniert). Man kann die Helligkeit des Bildes berechnen:
b <-Helligkeit ("~/Desktop/img001.tif", Tau = "auto")- Dies weist die Helligkeit des Bildes an die Variable b in R. Das Tau = "Auto" wird sichergestellt, dass das Bild korrekt vor Helligkeit Berechnung trendbereinigte ist. Die häufigste, was hier zu tun ist, berechnen Sie den Mittelwert oder mittlere Helligkeit des Bildes. Dies kann man durch Eingabe von mean(b) oder median(b). Man kann auch die Helligkeit Bild auf dem desktop mit schreiben.
ijtiff::write_tif (b, "~/Desktop/whatever_img_name")
- Dies weist die Helligkeit des Bildes an die Variable b in R. Das Tau = "Auto" wird sichergestellt, dass das Bild korrekt vor Helligkeit Berechnung trendbereinigte ist. Die häufigste, was hier zu tun ist, berechnen Sie den Mittelwert oder mittlere Helligkeit des Bildes. Dies kann man durch Eingabe von mean(b) oder median(b). Man kann auch die Helligkeit Bild auf dem desktop mit schreiben.
- Sagen Sie, man hat Ordner voller Bilder Images_folder auf dem Desktop und muss man die Helligkeiten dieser Bilder zu berechnen und die Helligkeit Bilder als TIFF-Dateien zu schreiben. Dann sehen? brightness_folder(). Diese Funktion verarbeitet einen ganzen Ordner auf einmal:
Brightness_folder ("~/Desktop/images_folder", Tau = "auto")
Dies ist besonders gut für diejenigen, die eine Software, die sie zu R bevorzugen, da aller Dateien in einem einzigen Befehl verarbeitet werden, und dann man weiter mit der Ausgabe Helligkeit TIFF-Bilder in ihrer gewählten Software arbeiten gehen kann, sei es ImageJ14 , Python oder etwas anderes.
- Also muss man eine Image-Datei auf dem Desktop mit dem Namen img001.tif (Beachten Sie, dass "Nandb" nur mit TIFF-Dateien funktioniert). Man kann die Helligkeit des Bildes berechnen:
Ergebnisse
Detrending und Monomeren Helligkeit
Sobald die Daten erhalten hat, kann man die Helligkeit Berechnungen durch, um festzustellen, die Oligomeren Zustand des Proteins des Interesses in der Lösung starten. Auch wenn die Wirkung der Bleiche in Lösung möglicherweise nicht so drastisch wie es sein kann, in Vivo, die Intensität-Ablaufverfolgung wird wahrscheinlich noch nicht stationäre bedeuten, mögliche...
Diskussion
N & B ist eine Technik, um Multimerization mit kommerziellen Licht Scannen konfokale Mikroskopen ausgestattet mit digitaler Detektoren erkennen. Dieser Ansatz ist sehr attraktiv im Vergleich zu single Point FCS, FCC und SmFRET weil es kostenlose Kalibrierung und die Berechnung der Helligkeit ist unkompliziert und Konzentration unabhängigen6. Es ist von großer Bedeutung, jedoch für Bleichen und langfristige intensitätsschwankungen zu korrigieren, bevor Sie Helligkeit Berechnungen
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Diese Arbeit wurde von Wellcome Trust unterstützt gewähren R.N 105278/Z/14/2 Der Wellcome Trust Centre for Human Genetics wird vom Wellcome Trust Core Award 203852/Z/16/2 finanziert. Arbeit in der c.s.-Gruppe wird von Cancer Research UK (C20724/A14414) und des European Research Council unter Horizont 2020 Forschung und Innovation Programm Grant 647278 der Europäischen Union unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| RosettaTM (DE3) pLysS cells | Novagen | 70956-3 | |
| Ampicillin | Sigma Aldrich | PubChem Substance ID 329824407 | |
| Chloramphenicol | Sigma Aldrich | PubChem Substance ID: 24892250 | |
| LB starter culture | QIAGEN | ||
| LB medium | QIAGEN | https://www.sigmaaldrich.com/content/dam/sigma-aldrich/head/search/external-link-icon.gif | |
| IPTG | Sigma Aldrich | PubChem Substance ID 329815691 | |
| IMAC buffer | Medicago | 09-1010-10 | |
| EDTA-free protease inhibitors | Sigma Aldrich | 11873580001 | |
| TALON resin | Clonetech | ||
| Nickel sepharose | GE Healthcare | ||
| S200 16/60 column | GE Healthcare | ||
| Glass bottom 8 well observation dish | Ibidi | 80827 |
Referenzen
- Voith von Voithenberg, L., Lamb, D. C. Single pair forster resonance energy transfer: A versatile tool to investigate protein conformational dynamics. BioEssays. 40, (2018).
- Padilla-Parra, S., Auduge, N., Coppey-Moisan, M., Tramier, M. Quantitative FRET analysis by fast acquisition time domain FLIM at high spatial resolution in living cells. Biophysical Journal. 95, 2976-2988 (2008).
- Padilla-Parra, S., Auduge, N., Coppey-Moisan, M., Tramier, M. Dual-color fluorescence lifetime correlation spectroscopy to quantify protein-protein interactions in live cell. Microscopy Research and Technique. 74, 788-793 (2011).
- Muller, J. D., Chen, Y., Gratton, E. Fluorescence correlation spectroscopy. Methods in Enzymology. 361, 69-92 (2003).
- Tramier, M., Coppey-Moisan, M. Fluorescence anisotropy imaging microscopy for homo-FRET in living cells. Methods in Cell Biology. 85, 395-414 (2008).
- Digman, M. A., Dalal, R., Horwitz, A. F., Gratton, E. Mapping the number of molecules and brightness in the laser scanning microscope. Biophysical Journal. 94, 2320-2332 (2008).
- Nolan, R., Iliopoulou, M., Alvarez, L., Padilla-Parra, S. Detecting protein aggregation and interaction in live cells: A guide to number and brightness. Methods. , (2017).
- Dalal, R. B., Digman, M. A., Horwitz, A. F., Vetri, V., Gratton, E. Determination of particle number and brightness using a laser scanning confocal microscope operating in the analog mode. Microscopy Research and Technique. 71, 69-81 (2008).
- Nolan, R., et al. nandb-number and brightness in R with a novel automatic detrending algorithm. Bioinformatics. , (2017).
- Hur, K. H., et al. Quantitative Measurement of Brightness from Living Cells in the Presence of Photodepletion. PLoS One. 9, (2014).
- Amara, J. F., et al. A versatile synthetic dimerizer for the regulation of protein-protein interactions. Proceedings of the National Academy of Sciences of the United States of America. 94, 10618-10623 (1997).
- Clackson, T., et al. Redesigning an FKBP-ligand interface to generate chemical dimerizers with novel specificity. Proceedings of the National Academy of Sciences of the United States of America. 95, 10437-10442 (1998).
- Rollins, C. T., et al. A ligand-reversible dimerization system for controlling protein-protein interactions. Proceedings of the National Academy of Sciences of the United States of America. 97, 7096-7101 (2000).
- Schindelin, J., Rueden, C. T., Hiner, M. C., Eliceiri, K. W. The ImageJ ecosystem: An open platform for biomedical image analysis. Molecular Reproduction and Development. 82, 518-529 (2015).
- . R: A Language and Environment for Statistical Computing. R Core. , (2017).
- . RStudio: Integrated Development Environment for R. R Team. , (2016).
- . nandb R package Available from: https://CRAN.R-project.org/package=nandb (2017)
- Jung, G., Wiehler, J., Zumbusch, A. The photophysics of green fluorescent protein: influence of the key amino acids at positions 65, 203, and 222. Biophysical Journal. 88, 1932-1947 (2005).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nature Methods. 2, 905-909 (2005).
- Butkevich, A. N., et al. Hydroxylated fluorescent dyes for live-cell labeling: synthesis, spectra and super-resolution STED. Chemistry: A European Journal. 23, 12114-12119 (2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten