Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Калибровка бесплатно в Vitro количественная оценка белка гомо олигомеризации с использованием коммерческих приборов и свободным, открытым исходным кодом программное обеспечение для анализа яркости
В этой статье
Резюме
Этот протокол описывает бесплатная калибровка подход для количественного определения белка гомо олигомеризации в vitro основанные на страницу отдела Флуоресценция колебаний с помощью коммерческого свет, сканирующая микроскопия. Показаны правильные приобретения параметры и методы анализа.
Аннотация
Количество и яркость является методом спектроскопии (FFS) колебания бесплатная калибровка флуоресценции для обнаружения гомо олигомеризации протеина. Он может быть использован с помощью обычных Конфокальный микроскоп оснащен цифровой детекторов. Протокол для использования техники в vitro показано с помощью варианта использования, где можно увидеть номер и яркость точно определить олигомерных состояние mVenus, меченного FKBP12F36V до и после добавления dimerizing препарата AP20187. Обсудили важность использования правильного Микроскоп приобретения параметры и методы предварительной обработки и анализа данных. В частности подчеркнул важность выбора Фотообесцвечивание коррекции. Этот недорогой метод может использоваться для изучения взаимодействия протеин протеина во многих биологических контекстах.
Введение
Белок белковых взаимодействий, я n пробирки
Традиционно технологии выбран точно описать трехмерную архитектуру белков и вывести их функции являются Кристаллография и экспериментов ядерного магнитного резонанса, в сочетании с крио электронной микроскопии (cryoEM) тщательное изучение их структурные детали высокого разрешения. Белки, однако, не являются статичными структурами и можно пройти разнообразные конформационные изменения и вибрации во времени и пространстве. Вот почему структурной информации из кристаллографических или CryoEM данных необходимо дополнить другими методами (например, в моделях молекулярной динамики и одной молекулы методы): функцию белка связано с его конформационную изменения и взаимодействия и эта информация отсутствует в статической структуры. Для того чтобы зондировать для внутри молекулярной динамики, методы, основанные на одной молекулы Форстер резонанса (smFRET) передачи энергии являются очень эффективным1. Эти подходы имеют возможность оценить различные субпопуляции молекул в сложных средах. Это очень важно, как эти изменения быстро и во время приобретения данных (то есть, наносекундных второй диапазон).
Для выявления и количественной оценки этих изменений часто используются два основных подхода: белки в растворе и поверхности иммобилизации. Для обнаружения между молекулярных взаимодействий и, в частности, процесс димеризации индуцированных лигандов, smFRET не всегда является лучшим инструментом. Действительно ладу зависит не только от расстояния (≈10 Нм), но также на ориентацию двух диполей (доноров и акцепторов, χ2) и перекрытие доноров выбросов с акцептором спектров поглощения2, но возможно это Последнее условие меньше важные при том условии, что экспериментатор может выбрал правильный пара ладу. Особый недостаток smFRET для зондирования гомо димеризации приходит от маркировки протеина интереса: Гетеро smFRET, димеризации может только быть обнаружены до 50% (т.е., гетеро Лада будет только в состоянии обнаружить донорно акцепторной и Акцептор доноров гомо димеров но не доноров донора или акцептор акцептор, который другие 50% димеров). Использование корреляции отдела Флуоресценция (FCS) и производных (FCCS,3 и т.д.) для определения белка диффузии константы и привязки константы в пробирке является еще одной альтернативой. Эти подходы не способны полностью количественно гомо димеризации либо, как и коэффициент концентрации и диффузии и радиуса распространения FCS один меры диффундирующих частиц очень слабо зависит от молекулярной массы; к примеру десятикратного увеличения в молекулярном весе только будет означать 2,15 раза изменения коэффициента диффузии4. В случае FCS-двухцветный или FCCS только 50% гомо димеры будет рассматриваться по той же причине, как выше. Наиболее практичных и количественного подходов к обнаружить гомо димеризации в пробирке и в естественных условиях являются гомо Лада5 и номер и яркость (N & B)6. Учитывая тот факт, что гомо лада требует восстановления конкретных приборов значения анизотропии (т.е., оптические элементы/анализаторы для восстановления параллельные и перпендикулярные поляризации), N & B представлен здесь как благоприятный метод Обнаружение белка гомо димеризации и агрегации. Это может быть заняты как in vitro , так и в естественных условиях с коммерческой структуры.
Количество и яркость
N & B был недавно рассмотрели7. Этот обзор был сосредоточен на применение техники в живых клеток. Это целесообразно воспроизвести математического формализма здесь как эти уравнения будут применяться к данным, собранных в пробирке. Во-первых это необходимо для определения некоторых терминов и математических величин:
- Сущность — это набор молекул, которые связаны друг с другом.
- Яркость ε сущности — это количество фотонов, он испускает за единицу времени (в кадре).
- n — это количество сущностей, настоящее.
- Для данного пикселя в течение серии изображений < i > является его средней интенсивности и σ2 вариативность его интенсивности.
Затем с Фотон подсчет детекторы и предполагая мобильных подразделений и без фона
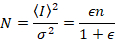
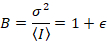
где N — это явное число и B является явно яркость. Это приводит к
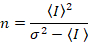
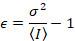
Далаль и др. 8 показал, что с аналогового оборудования, один нуждается три коррекции терминов: S фактор, фон смещениеи шум считывания σ02. Затем опять же при условии мобильных подразделений,
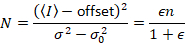
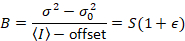
давая
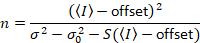

Обратите внимание, что выше уравнение отличается в Далаль и др. 8 и последующий обзор. 7 в Далал et al. S в знаменателе был опущен из-за опечатки, и эта ошибка была воспроизведена в обзоре. Уравнение выше является правильным. Инструкции для измерения S, смещение и σ02 - вместе с объяснением их смысл - выдаются Далаль и др. 8
Ε яркость пропорционален олигомерных состояние диффундирующих образований: ε будет дважды как большой для димеры, как это для мономеров, три раза большим, для тримеры, как это для мономеров, дважды как большой для hexamers, как это для тримеры и так далее. Таким образом, измерения яркости ε один можно количественно определить любой тип multimerization.
Если есть смесь олигомерных государств настоящее время, и яркость не способна восстановление отдельных олигомерных присутствующих государств. Это ограничение техники.
Бестрендовости алгоритма и nandb программного обеспечения
Исправление для Фотообесцвечивание был ранее важность9. Фотообесцвечивание неизбежно происходит во время экспериментов световой микроскопии в режиме покадровой; Оба в живых клеток и в пробирке. Многие подходы были описаны в литературе исправить для отбеливания7. Текущий лучше всего метод экспоненциального фильтрации с автоматического выбора detrending параметра T . Она интегрирована в свободный, Открытый источник программного обеспечения nandb9. Действительно программное обеспечение, которое требует от пользователя вручную выбирать их detrending параметр может привести к неверным результатам потому, что этот параметр выбор, вероятно, будет произвольным и неправильным. Автоматический алгоритм проверяет данные и определяет соответствующие параметры для его, без необходимости вмешательства пользователя9. Даже с самым лучшим выбором параметра сглаживания detrending имеет свои ограничения и работает только с Фотообесцвечивание проценты ниже 25%, как показано с моделирования9. Интересно, что при использовании автоматического detrending рутины, ее точность такова, что можно работать с низкой яркости значения (даже B < 1.01) и следовательно низкой интенсивности и все еще быть достаточно точным, чтобы количественно гомо димеризации.
Фотообесцвечивание также вызывает другая проблема: присутствие флуорофоров photobleached в multimer комплексе. Это делает, например, тример появляются как димер, когда один из этих трех подразделений в тример не люминесцентные. Гур и Мюллер10 показали, как исправить это, и это исправление было также подчеркнуто в последующий обзор7. Nandb программное обеспечение включает в себя этой коррекции9.
FKBP12 F36V системы
FKBP12F36V — это белок, который естественно не oligomerize, но как известно, dimerize после добавления AP20187 наркотиков (известный как разговорно BB, dimerizing лигандом)11,12. Это делает его идеальным тестовый случай для числа и яркость: с маркировкой FKBP12F36V, удвоение олигомерных государства должен соблюдаться при добавлении BB.
протокол
1. FKBP12 F36V - mVenus очистки
- Преобразование (DE3) pLysS клетки с pET22b вектор, содержащий monomerized человека FKBP12F36V12 и His6 и mVenus Теги N-терминальный (вектор по запросу). Плита клетки на LB агар, дополненная 50 мкг/мл Ампициллин и 34 мкг/мл хлорамфеникола.
- Передача преобразованных колоний в 100 мл фунта закваски и расти на 16-20 часов при 37 ° C при встряхивании.
- Разбавить густой закваски (OD600 > 1) 1: 100 в среде фунтов (2 x 500 мл пакетов) и расти на 2-3 часа до OD600 = 0,6 - 0,8 (37 ° C, 200 об/мин).
- Прохладный культур на льду. Побудить с 250 мкм IPTG и расти на 16-20 часов при температуре 21 ° C, 200 об/мин.
- Урожай клетки центрифугированием в 2000 x g за 20 минут.
- Ресуспензируйте Пелле в 40 мл буфера IMAC (20 мм натрия фосфат pH 7.5, 500 мм NaCl, 3 мм имидазола, 1 мм β-меркаптоэтанол) дополнены с ЭДТА бесплатно ингибиторы протеазы (1 таблетка на Пелле клеток).
- Sonicate клетки (500 Вт, 20 кГц, амплитуда 40%, 9 s на 11 s от 15 мин) на льду и урожай растворимых материал центрифугированием на 20000 x g.
- Передача растворимых lysate в коническую колбу и добавить 2 мл смолы (см. Таблицу материалы). Инкубировать 1 час с вращением 105 об/мин
Примечание: Никель sepharose также может использоваться для этого шага IMAC. - Урожай смолы и мыть с 250 мл IMAC буфера A следуют 500 мл IMAC буфера B (20 мм натрия фосфат pH 7.5, 500 мм NaCl, 7 мм имидазола, β-меркаптоэтанол 1 мм).
Примечание: Увеличение до 50 мм имидазола если используется никель sepharose смолы. - Элюировать His6-тегами протеина используя IMAC буфера C (20 мм натрия фосфат рН 7,5, 500 мм NaCl, 300 мм имидазола, β-меркаптоэтанол 1 мм).
- Внедрить на размер исключение столбцов (см. Таблицу материалы) достижение равновесного уровня в 10 мм HEPES pH 7.5, 150 мм NaCl, 1 DTT. FKBP12F36V имеет свой пик элюции в 87.71 мл на столбце, который мы использовали.
- Оценка чистоты через SDS-PAGE и бассейн и сосредоточиться при необходимости.
2. подготовка массива Multiwell пластины
- Оттепель очищенный FKBP12F36V (или помечены протеин интереса) от-80 ° C.
- Приготовляют раствор 100 Нм очищенный FKBP12F36V (средний, 10 мм HEPES pH 7.5, 150 мм NaCl, 1 DTT). Sonicate и центрифуги (быстрый спин 13000 об/мин) для предотвращения образования агрегатов.
- Пипетка 100-200 мкл в камеру 8 хорошо наблюдения с низа стекла.
- Добавьте BB dimerizer окончательного концентрации 10, 20, 40, 80, 100, 150, 300 и 500 Нм12,13.
- Как ссылка, приготовляют раствор 100 Нм mVenus только, чтобы оценить потенциальные последствия агрегации и осадков и восстановить значение яркости на мономера с теми же настройками приобретения.
3. бесплатная калибровка конфокальный приобретение
- Начало конфокальный системы (рис. 1). Свет сканирование системы Конфокальный микроскоп оснащен цифровой детекторы или хорошо изученных аналоговые датчики8, и способны поддержания постоянной продолжительность для каждого пикселя, приобрел будет работать.
- Задайте путь луча возбуждения:
- Включите 514 Нм лазер и набор его на 20-100 nW мощность на выходе цели (для FKBP12F36V-mVenus).
- Выберите цель 63X1.4NA или воротник коррекции цель погружения воды предназначены для FCS.
- Включите один Илья, APD или калиброванные ПЛТ детектор. Детекторы, способные Фотон подсчет предпочтительным, как в этом случае, расчета S, смещение и σ02 являются ненужными.
- Выберите окно выбросов от 520-560 Нм
- Установите обскуры 1 воздушные подразделения для соответствующих выбросов ~ 545 Нм.
- Установите режим приобретения на 16 x 16 пикселов
- Задайте tвремя dwell пикселей,жить таким образом, что он удовлетворяет tкадра >> TD >> tжить, где D — время пребывания диффундирующих белка и t рамка является частота кадров. Это соответствует параметру Продолжительность ~ 13 МКС.
Примечание: Некоторые коммерческие производители имели сканеров, которые не держали время задержки на пиксель постоянно. Это постоянство имеет решающее значение для работы метода. - Задать размер пикселя в ~ 120 Нм.
- Выберите режим приобретения xyt и выберите количество кадров, чтобы быть приобретены за приобретение и хорошо (например, 5000).
- Если система оборудована с высокой пропускной способностью режим, ввести координаты каждой скважины и количество приобретений за скважины для автоматизации процесса.
Примечание: Будьте внимательны для обеспечения присутствия воды Диспенсер для, если с помощью объективного погружения. - Если система оснащена перфузии системы, загрузите решение BB и программа перфузии чтобы начать право после 5000й кадр оценить кинетику димеризации приобретая например, 10 000 изображений.
- Добавить каплю масла в цель погружения нефти / воды если используя воротник коррекции цель погружения воды предназначены для FCS.
- Смонтируйте 8 хорошо наблюдения камеры в сцене.
- Выберите правильный колодец и сосредоточиться на решении.
Примечание: Важно: избежать акцентирования внимания близко к нижней стекла, чтобы избежать отражения и рассеивания. При фокусировке глубже в раствор, отключите параметр автоматической фокус. - Начать приобретение и сохранить полученный стек изображений в формате TIFF.
4. Бестрендовости и яркость анализа с использованием пакета R nandb
- Для предварительной проверки качества используйте ImageJ14 взглянуть на изображения и восстановить интенсивность профиля, как показано на рисунке 2. Это полезно для определения, произошло ли или не слишком много Фотообесцвечивание. Если есть слишком много отбеливания, данных не подходит для дальнейшего анализа.
Примечание: ImageJ также может быть полезным для преобразования изображений TIFF из коммерческих форматов. Nandb программное обеспечение, описанное ниже может работать только с файлами TIFF. - Скачать и установить R и RStudio15,16. Это лучше загрузить и установить R, а затем RStudio.
Примечание: Ниже приводится описание того, как использовать пакет nandb Р. Знание языка R не требуется для использования nandb, но это будет сделать жизнь проще. - Установите пакет nandb.
- Открыть RStudio и в консоли, введите install.packages("nandb") и дождитесь завершения установки.
- Узнать nandb
- Просмотрите руководство17.
- Обзор встроенного RStudio помощь для различных функций. Наиболее вероятным функция использоваться будет использование будет brightness(). Просмотр файла справки для этой функции, введя? Brightness() на консоли.
- Вычислить яркость
- Говорят, что один имеет файл изображения на рабочем столе под названием img001.tif (Обратите внимание, что «nandb» работает только с файлами TIFF). Яркость изображения можно вычислить:
b <-яркость («~/Desktop/img001.tif», Тау = "auto")- Яркость изображения это присваивает переменной b в р. Тау = «auto» гарантирует, что изображение является правильно бестрендового до вычисления яркость. Наиболее распространенные вещи делать здесь является для вычисления среднего значения или средней яркости изображения. Одно может сделать это, набрав mean(b) или median(b). Можно также написать яркость изображения с рабочего стола с помощью
ijtiff::write_tif (b, «~/Desktop/whatever_img_name»)
- Яркость изображения это присваивает переменной b в р. Тау = «auto» гарантирует, что изображение является правильно бестрендового до вычисления яркость. Наиболее распространенные вещи делать здесь является для вычисления среднего значения или средней яркости изображения. Одно может сделать это, набрав mean(b) или median(b). Можно также написать яркость изображения с рабочего стола с помощью
- Говорят, один имеет полный images_folder изображений на рабочем столе папку и необходимо вычислить яркостей эти изображения и писать яркость изображения как TIFF-файлы. Затем посмотреть? brightness_folder(). Эта функция обрабатывает всю папку все сразу:
brightness_folder («~/Desktop/images_folder», Тау = "auto")
Это особенно хорошо для тех, которые имеют программное обеспечение, которое они предпочитают R, потому что все файлы обрабатываются в одной командой, и затем один можно продолжать работать с изображениями TIFF яркость вывода в их выбранного программного обеспечения, будь то ImageJ14 , Python или что-то другое.
- Говорят, что один имеет файл изображения на рабочем столе под названием img001.tif (Обратите внимание, что «nandb» работает только с файлами TIFF). Яркость изображения можно вычислить:
Результаты
Detrending и мономерных яркость
После того, как данные были приобретены, можно начать яркость расчеты для определения олигомерных состояние протеина интереса в решении. Даже если эффект отбеливания в растворе не может быть как рез...
Обсуждение
N & B является методом для обнаружения multimerization с использованием коммерческих свет, сканирование конфокальные микроскопы оснащены цифровыми детекторов. Этот подход является весьма привлекательной по сравнению с одноточечным FCS, FCCS и smFRET потому что это бесплатная калибровка и яркость ра...
Раскрытие информации
Авторы не имеют ничего сообщать.
Благодарности
Эта работа была поддержана Уэллком траст предоставить р.н. 105278/Z/14/2 Добро пожаловать доверять центр по генетике человека финансируется, Добро пожаловать доверять Core награду 203852/Z/16/2. Работа в группе C.S. поддерживается исследований рака Великобритании (C20724/A14414) и Европейский исследовательский совет Европейского союза Horizon 2020 исследования и инновации программа грантов 647278.
Материалы
| Name | Company | Catalog Number | Comments |
| RosettaTM (DE3) pLysS cells | Novagen | 70956-3 | |
| Ampicillin | Sigma Aldrich | PubChem Substance ID 329824407 | |
| Chloramphenicol | Sigma Aldrich | PubChem Substance ID: 24892250 | |
| LB starter culture | QIAGEN | ||
| LB medium | QIAGEN | https://www.sigmaaldrich.com/content/dam/sigma-aldrich/head/search/external-link-icon.gif | |
| IPTG | Sigma Aldrich | PubChem Substance ID 329815691 | |
| IMAC buffer | Medicago | 09-1010-10 | |
| EDTA-free protease inhibitors | Sigma Aldrich | 11873580001 | |
| TALON resin | Clonetech | ||
| Nickel sepharose | GE Healthcare | ||
| S200 16/60 column | GE Healthcare | ||
| Glass bottom 8 well observation dish | Ibidi | 80827 |
Ссылки
- Voith von Voithenberg, L., Lamb, D. C. Single pair forster resonance energy transfer: A versatile tool to investigate protein conformational dynamics. BioEssays. 40, (2018).
- Padilla-Parra, S., Auduge, N., Coppey-Moisan, M., Tramier, M. Quantitative FRET analysis by fast acquisition time domain FLIM at high spatial resolution in living cells. Biophysical Journal. 95, 2976-2988 (2008).
- Padilla-Parra, S., Auduge, N., Coppey-Moisan, M., Tramier, M. Dual-color fluorescence lifetime correlation spectroscopy to quantify protein-protein interactions in live cell. Microscopy Research and Technique. 74, 788-793 (2011).
- Muller, J. D., Chen, Y., Gratton, E. Fluorescence correlation spectroscopy. Methods in Enzymology. 361, 69-92 (2003).
- Tramier, M., Coppey-Moisan, M. Fluorescence anisotropy imaging microscopy for homo-FRET in living cells. Methods in Cell Biology. 85, 395-414 (2008).
- Digman, M. A., Dalal, R., Horwitz, A. F., Gratton, E. Mapping the number of molecules and brightness in the laser scanning microscope. Biophysical Journal. 94, 2320-2332 (2008).
- Nolan, R., Iliopoulou, M., Alvarez, L., Padilla-Parra, S. Detecting protein aggregation and interaction in live cells: A guide to number and brightness. Methods. , (2017).
- Dalal, R. B., Digman, M. A., Horwitz, A. F., Vetri, V., Gratton, E. Determination of particle number and brightness using a laser scanning confocal microscope operating in the analog mode. Microscopy Research and Technique. 71, 69-81 (2008).
- Nolan, R., et al. nandb-number and brightness in R with a novel automatic detrending algorithm. Bioinformatics. , (2017).
- Hur, K. H., et al. Quantitative Measurement of Brightness from Living Cells in the Presence of Photodepletion. PLoS One. 9, (2014).
- Amara, J. F., et al. A versatile synthetic dimerizer for the regulation of protein-protein interactions. Proceedings of the National Academy of Sciences of the United States of America. 94, 10618-10623 (1997).
- Clackson, T., et al. Redesigning an FKBP-ligand interface to generate chemical dimerizers with novel specificity. Proceedings of the National Academy of Sciences of the United States of America. 95, 10437-10442 (1998).
- Rollins, C. T., et al. A ligand-reversible dimerization system for controlling protein-protein interactions. Proceedings of the National Academy of Sciences of the United States of America. 97, 7096-7101 (2000).
- Schindelin, J., Rueden, C. T., Hiner, M. C., Eliceiri, K. W. The ImageJ ecosystem: An open platform for biomedical image analysis. Molecular Reproduction and Development. 82, 518-529 (2015).
- . R: A Language and Environment for Statistical Computing. R Core. , (2017).
- . RStudio: Integrated Development Environment for R. R Team. , (2016).
- . nandb R package Available from: https://CRAN.R-project.org/package=nandb (2017)
- Jung, G., Wiehler, J., Zumbusch, A. The photophysics of green fluorescent protein: influence of the key amino acids at positions 65, 203, and 222. Biophysical Journal. 88, 1932-1947 (2005).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nature Methods. 2, 905-909 (2005).
- Butkevich, A. N., et al. Hydroxylated fluorescent dyes for live-cell labeling: synthesis, spectra and super-resolution STED. Chemistry: A European Journal. 23, 12114-12119 (2017).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены