È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Privo di calibrazione In Vitro quantificazione delle proteine omo-oligomerizzazione utilizzando Strumentazione commerciale e libero, Open Source Software di analisi di luminosità
In questo articolo
Riepilogo
Questo protocollo descrive un approccio privo di calibrazione per la quantificazione della proteina omo-oligomerizzazione in vitro basati sulla spettroscopia di fluorescenza fluttuazione mediante microscopia a scansione di luce commerciale. Le impostazioni di acquisizione corretta e i metodi di analisi sono mostrati.
Abstract
Numero e la luminosità è una tecnica di spettroscopia (FFS) fluttuazione di fluorescenza privo di calibrazione per la rilevazione di proteine omo-oligomerizzazione. Può essere impiegata utilizzando un microscopio confocale convenzionale dotato di rivelatori digitali. Un protocollo per l'uso della tecnica in vitro è mostrato per mezzo di un caso di utilizzo dove il numero e la luminosità può essere visto per quantificare con precisione lo stato oligomerico di FKBP12F36V mVenus-etichettati prima e dopo l'aggiunta del farmaco dimerizing AP20187. L'importanza dell'utilizzo dei parametri di acquisizione corretta microscopio e i metodi di pre-elaborazione e analisi di dati corretti sono discusse. In particolare, l'importanza della scelta della correzione photobleaching è sollecitata. Questo metodo poco costoso può essere impiegato per studiare le interazioni proteina-proteina in molti contesti biologici.
Introduzione
Proteina-proteina interazioni ho n Vitro
Tradizionalmente, cristallografia ed esperimenti di risonanza magnetica nucleare combinati con cRIO-microscopia elettronica (cryoEM) sono le tecnologie scelte di descrivere con precisione l'architettura tridimensionale di proteine e di dedurre la loro funzione di scrutando i dettagli strutturali ad alta risoluzione. Proteine, tuttavia, non sono strutture statiche e possono subire una varietà di cambiamenti conformazionali e vibrazioni nel tempo e nello spazio. Questo è il motivo strutturale informazioni da cristallografici o CryoEM dati devono essere integrata con altre tecniche (ad es., simulazioni di dinamica molecolare e tecniche di singola molecola): la funzione di una proteina è relativo alla sua conformazionali modifiche e interazioni e tale informazione non è presente in una struttura statica. Al fine di sondare per dinamiche intra-molecolari, tecniche basate sulla singola molecola Forster Resonance Energy Transfer (smFRET) sono molto efficaci1. Questi approcci sono in grado di valutare diverse sottopopolazioni di molecole complesse media. Questo è molto importante, poiché questi cambiamenti sono rapidi e si verificano durante l'acquisizione dei dati (cioè, nanosecondo di seconda fascia).
Due approcci principali sono comunemente impiegati per rilevare e quantificare questi cambiamenti: proteine in soluzione e superficie-immobilizzazione. Per la rilevazione delle interazioni inter-molecolari e in particolare, il processo di dimerizzazione indotta da ligandi, smFRET non è sempre il miglior strumento. Infatti, FRET dipende non solo dalla distanza (â10 nm) ma anche l'orientamento dei due dipoli (donatore e accettore, χ2) e la sovrapposizione dell'emissione donatore con spettri di assorbimento2 del ricettore, ma forse quest'ultima condizione è meno importante purché lo sperimentalista può ha scelto la giusta coppia FRET. Una posizione di particolare svantaggio di smFRET per sondare omo-dimerizzazione proviene l'etichettatura della proteina di interesse: per smFRET etero, dimerizzazione può solo essere rilevato fino al 50% (cioè, etero-FRET sarà solo in grado di rilevare il donatore-accettore e donatore-accettore homo-dimeri ma non donatore-donatore o accettore-accettore, che è l'altro 50% dei dimeri). L'uso della spettroscopia di correlazione di fluorescenza (FCS) e derivati (FCC, ecc3) per l'accertamento della proteina diffusione costanti e costanti associazione in vitro è un'altra alternativa. Questi approcci non sono in grado di completamente quantificare sia omo-dimerizzazione, come coefficiente di concentrazione e diffusione e il raggio e la diffusione di misure FCS uno di una particella di diffusione sono molto scarsamente dipendenti dal peso molecolare; ad esempio un aumento di 10 volte il peso molecolare comporterà solo un cambiamento di 2.15 piega in diffusione coefficiente4. Nel caso di due colori FCS o FCCS, solo il 50% di homo-dimeri saranno visibili per lo stesso motivo come sopra. Approcci più pratici e quantitativi per rilevare omo-dimerizzazione in vitro e in vivo sono homo-FRET5 numero e luminosità (N & B)6. Dato il fatto che homo-FRET richiede il ripristino di strumentazione specifica del valore di anisotropia (cioè, ottico elementi/analizzatori per recuperare la polarizzazione parallela e perpendicolare), N & B è presentato qui come una tecnica favorevole per rilevare la proteina omo-dimerizzazione e aggregazione. Può essere impiegato sia in vitro che in vivo con un set-up commerciale.
Numero e luminosità
N & B è stato recentemente rivisto7. Tale revisione focalizzata sull'applicazione della tecnica in cellule vive. Vale la pena di riprodurre il formalismo matematico qui come queste equazioni verrà applicato ai dati raccolti in vitro. Innanzitutto, è necessario definire alcuni termini e quantità matematiche:
- Un'entità è un insieme di molecole che sono legati insieme.
- Il ε di luminosità di un'entità è il numero di fotoni che emette per unità di tempo (per telaio).
- n è il numero di entità presenti.
- Per un dato pixel nel corso di una serie di immagini, < i > è che la media intensità e σ2 è la varianza nella sua intensità.
Quindi, con photon-counting rivelatori e supponendo che entità mobile e senza sfondo,
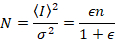
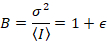
dove N è il numero di apparente e B è la luminosità apparente. Questo si traduce in
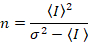
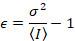
De luca et al. 8 hanno mostrato che con apparecchi analogici, uno ha bisogno di tre termini di correzione: il fattore di S, l' offsetdi sfondo e il rumore di lettura σ02. Quindi, ancora una volta assumendo entità mobile,
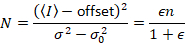
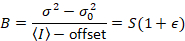
dando
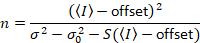

Si noti che l'equazione sopra è diverso che dato a Luca et al. 8 e un successivo riesame. 7 in Luca et al. S nel denominatore è stato omesso a causa di un errore di battitura e questo errore è stato riprodotto nella revisione. L'equazione precedente è quella giusta. Istruzioni per la misura S, offset e σ02 - insieme a una spiegazione del loro significato - sono date da Luca et al. 8
Il ε di luminosità è proporzionale allo stato oligomerico delle entità diffondente: ε sarà due volte più grande per dimeri, come nel caso di monomeri, tre volte più grande per trimeri come nel caso di monomeri, due volte più grande per esameri come lo è per trimeri e così via. In questo modo, il ε di luminosità, di misura nessuno può quantificare qualsiasi tipo di multimerization.
Se ci sono una miscela di oligomeri stati presenti, il numero e la luminosità non è in grado di recuperare i singoli Stati oligomerici presenti. Si tratta di una limitazione della tecnica.
Detrend software algoritmo e nandb
Importanza della correzione per photobleaching è stato sottolineato in precedenza9. Photobleaching inevitabilmente si verifica durante gli esperimenti di microscopia in modalità Time-lapse; sia in cellule dal vivo e in vitro. Molti approcci sono stati descritti nella letteratura di correggere per il candeggio7. La tecnica di filtraggio esponenziale con scelta automatica del parametro detrending T è il migliore corrente. È integrato nel software libero, open source nandb9. In effetti, software che richiede all'utente di scegliere manualmente il parametro detrending può portare a risultati non corretti perché questa scelta di parametro sarà probabilmente arbitrario e non corretto. L'algoritmo automatico controlla i dati e determina il parametro appropriato per esso, senza la necessità di intervento utente9. Anche con la migliore scelta del parametro levigante, detrending ha i suoi limiti e funziona bene solo con photobleaching percentuali inferiori al 25%, come mostrato con simulazioni9. È interessante notare che, quando si utilizza la routine di detrending automatica, l'esattezza è tale che si può lavorare con valori di luminosità bassa (anche B < 1.01) e quindi a bassa intensità e ancora essere abbastanza preciso per quantificare omo-dimerizzazione.
Photobleaching provoca anche un altro problema: la presenza di fluorofori photobleached in un complesso multimer. In questo modo ad esempio, un trimero appaiono come un dimero quando una delle tre unità in un trimero è non fluorescenti. Hur e Mueller10 ha mostrato come correggere questo e questa correzione è stata anche sottolineata in una successiva revisione7. Il software di nandb include questa correzione9.
Il FKBP12 F36V sistema
FKBP12F36V è una proteina che naturalmente non oligomerizzazione, ma è noto che dimerizzano sull'aggiunta di droga (colloquialmente conosciuto come il BB dimerizing ligando)11,AP2018712. Questo lo rende un caso di test ideale per numero e luminosità: con FKBP12F36V etichettati, un raddoppio dello stato oligomerico dovrebbe essere osservato con l'aggiunta di BB.
Protocollo
1. FKBP12 F36V - mVenus purificazione
- Trasformare (DE3) pLysS cellule con pET22b vettoriale contenente monomerized umano FKBP12F36V12 e tag di His6 e mVenus del N-terminale (vettore disponibile su richiesta). Cellule di piastra su agar LB completati con ampicillina 50 µ g/mL e 34 µ g/mL cloramfenicolo.
- Trasferire le colonie trasformate in coltura starter 100 mL LB e crescere per 16-20 ore a 37 ° C con agitazione.
- Diluire la coltura starter denso (OD600 > 1) 1: 100 in mezzo LB (lotti di 2 x 500 mL) e crescere per 2-3 ore per OD600 = 0.6 - 0.8 (37 ° C, 200 giri/min).
- Cool culture sul ghiaccio. Indurre con 250 µM IPTG e crescere per 16-20 ore a 21 ° C, 200 giri/min.
- Raccogliere le cellule mediante centrifugazione a 2.000 x g per 20 minuti.
- Risospendere il pellet in 40 mL di tampone IMAC (20 mM sodio fosfato pH 7.5, 500 mM NaCl, imidazolo di 3 mM, 1 mM β-mercaptoetanolo) completati con EDTA-libero, inibitori della proteasi (1 compressa per pellet cellulare).
- Sottoporre ad ultrasuoni cellule (ampiezza di 500 watt, 20kHz, 40%, 9 s il, 11 s fuori per 15 min) il ghiaccio e raccolto materiale solubile mediante centrifugazione a 20.000 x g.
- Trasferimento solubile lisato in una beuta e aggiungere 2 mL di resina (Vedi Tabella materiali). Incubare per 1 ora con rotazione 105 giri/min
Nota: Nichel sepharose può essere utilizzata anche in questa fase IMAC. - Raccolta della resina e lavare con 250 mL di tampone IMAC A seguita da 500 mL di buffer di IMAC B (20 mM sodio fosfato pH 7.5, 500 mM NaCl, imidazolo di 7 mM, 1 mM β-mercaptoetanolo).
Nota: Aumentare di imidazolo 50mm se usando resina sepharose nichel. - Eluire la proteina His6-etichetta utilizzando IMAC buffer C (20 mM sodio fosfato pH 7.5, 500 mM NaCl, imidazolo 300 mM, 1 mM β-mercaptoetanolo).
- Iniettare su un'esclusione di dimensione colonna (Vedi Tabella materiali) equilibrato in 10 mM a pH 7.5, 150 mM NaCl, 1 millimetro DTT HEPES. FKBP12F36V ha sua eluizione di picco a 87,71 mL sulla colonna che abbiamo usato.
- Valutare la purezza tramite SDS-PAGE e piscina e concentrato come richiesto.
2. preparazione della matrice piastra multipozzetto
- Scongelare il FKBP12F36V purificato (o etichettato proteine di interesse) da-80 ° C.
- Preparare una soluzione di 100 nM purificato FKBP12F36V (media, 10 mM HEPES pH 7.5, 150 mM NaCl, 1 millimetro DTT). Sottoporre ad ultrasuoni e centrifugare (rapido giro di 13.000 giri/min) per evitare la formazione di aggregati.
- Pipettare 100-200 µ l in una camera di osservazione ben 8 con un fondo di vetro.
- Aggiungere il dimerizer BB a concentrazioni finali di 10, 20, 40, 80, 100, 150, 300 e 500 nM12,13.
- Come riferimento, preparare una soluzione di 100 nM di mVenus da soli per valutare potenziali effetti di aggregazione e precipitazione e recuperare un valore di luminosità per il monomero con le stesse impostazioni di acquisizione.
3. calibrazione-libero confocale acquisizione
- Avviare il sistema confocale (Figura 1). Qualsiasi luce microscopio confocale sistema di scansione dotato di rivelatori digitali o ben caratterizzati rivelatori analogici8, e in grado di mantenere un tempo di permanenza costante per ogni pixel acquisito avrebbe funzionato.
- Impostare il percorso del fascio di eccitazione:
- Accendere il 514 nm laser ed insieme a 20-100 nW di potenza all'uscita dell'obiettivo (per FKBP12F36V-mVenus).
- Selezionare l'obiettivo 63X1.4NA o un obiettivo a immersione in acqua correzione collare progettato per FCS.
- Accendere uno HyD, APD o calibrato PMT rivelatore. Rivelatori in grado di photon-counting sono preferibili, come in questo caso, il calcolo di S, offset e σ02 sono inutili.
- Selezionare la finestra di emissione da 520-560 nm
- Impostare il foro stenopeico presso arioso 1 unità per il corrispondente emissione ~ 545 nm.
- Impostare la modalità di acquisizione a 16 x 16 pixel
- Impostare tutte le caratteristiche della a tpixel dwell tempoabitare che soddisfa ttelaio >> TD >> tdimorare, dove D è il tempo di permanenza del diffusore t della proteina e telaio è il frame rate. Ciò ha corrisposto a impostare il tempo di permanenza a ~ 13 µs.
Nota: Alcuni produttori commerciali avevano scanner che non tenevano il tempo di permanenza al pixel costante. Questa costanza è cruciale per il metodo di lavoro. - Impostare la dimensione in pixel a ~ 120 nm.
- Selezionare la modalità di acquisizione xyt e selezionare il numero di fotogrammi da acquisire per acquisizione e bene (ad esempio 5.000).
- Se il sistema è dotato di modalità di alto-rendimento, introdurre le coordinate di ciascun pozzetto e il numero di acquisizioni per pozzetto per automatizzare il processo.
Nota: Fare attenzione a garantire la presenza di un distributore di acqua per se utilizza un obiettivo a immersione. - Se il sistema è equipaggiato con un sistema di perfusione, caricare la soluzione di BB e programmare la perfusione per iniziare a destra-dopo il telaio delth di 5000 per valutare la cinetica di dimerizzazione mentre stava acquistando per esempio, 10.000 immagini.
- Aggiungere una goccia di olio nell'obiettivo a immersione in olio / acqua se utilizzando un obiettivo a immersione in acqua correzione collare progettato per FCS.
- Montare la camera di osservazione ben 8 nella fase.
- Selezionare il corretto pozzo e concentrarsi sulla soluzione.
Nota: Importante: evitare di messa a fuoco vicino il fondo di vetro per evitare la riflessione e dispersione. Quando messa a fuoco più profondo nella soluzione, scollegare l'opzione di messa a fuoco automatica. - Avviare l'acquisizione e salvare lo stack risultante delle immagini in formato TIFF.
4. detrend e analisi di luminosità utilizzando il pacchetto R nandb
- Come controllo di qualità di pre-elaborazione, è possibile utilizzare ImageJ14 per dare un'occhiata alle immagini e ripristinare il profilo di intensità, come illustrato nella Figura 2un. Questo è utile per determinare se o non si è verificato troppo photobleaching. Se c'è troppa sbiancamento, i dati non sono adatti per un'ulteriore analisi.
Nota: ImageJ può anche essere utile per convertire immagini in TIFF da formati commerciali. Il software di nandb descritto di seguito può funzionare solo con i file TIFF. - Scaricare e installare R e RStudio15,16. Si consiglia di scaricare e installare R prima, poi RStudio.
Nota: Quello che segue è una descrizione di come utilizzare il pacchetto di nandb R. Conoscenza del linguaggio di R non è necessario utilizzare nandb, tuttavia si renderà la vita più facile. - Installare il pacchetto nandb.
- Aprire RStudio e nella console, digitare install.packages("nandb") e attendere per l'installazione.
- Conoscere nandb
- Rivedere il manuale17.
- Esaminare la guida RStudio incorporata per varie funzioni. La funzione più probabile da utilizzare sarà utilizzando sarà brightness(). Per visualizzare il file della Guida per questa funzione, digitare? Brightness() alla console.
- Calcolare la luminosità
- Dire che uno ha un file di immagine sul desktop chiamato img001.tif (Nota che 'nandb' funziona solo con i file TIFF). Si può calcolare la luminosità dell'immagine:
b <-luminosità ("~/Desktop/img001.tif", tau = "auto")- Questo assegna la luminosità dell'immagine per la variabile b in R. Il tau = "auto" assicura che l'immagine è correttamente detrended prima del calcolo di luminosità. La cosa più comune da fare da qui consiste nel calcolare la media o mediana luminosità dell'immagine. Uno può fare questo digitando mean(b) o median(b). Si può anche scrivere l'immagine di luminosità per l'utilizzo di desktop
ijtiff::write_tif (b, "~/Desktop/whatever_img_name")
- Questo assegna la luminosità dell'immagine per la variabile b in R. Il tau = "auto" assicura che l'immagine è correttamente detrended prima del calcolo di luminosità. La cosa più comune da fare da qui consiste nel calcolare la media o mediana luminosità dell'immagine. Uno può fare questo digitando mean(b) o median(b). Si può anche scrivere l'immagine di luminosità per l'utilizzo di desktop
- Dire che uno ha la cartella piena di immagini images_folder sul desktop e uno ha bisogno di calcolare la luminosità di queste immagini e scrivere le immagini di luminosità come file TIFF. Poi vedere? brightness_folder(). Questa funzione elabora un'intera cartella tutti in una volta:
brightness_folder ("~/Desktop/images_folder", tau = "auto")
Questo è particolarmente buono per coloro che hanno un software che preferiscono R, perché tutti i file vengono elaborati in un singolo comando, e quindi uno può continuare a lavorare con le immagini TIFF luminosità in uscita nel loro software scelto, sia esso ImageJ14 , Python o qualcos'altro.
- Dire che uno ha un file di immagine sul desktop chiamato img001.tif (Nota che 'nandb' funziona solo con i file TIFF). Si può calcolare la luminosità dell'immagine:
Risultati
Luminosità di detrending e monomerica
Una volta che sono stati acquistati i dati, si possono iniziare i calcoli di luminosità per determinare lo stato oligomerico della proteina di interesse nella soluzione. Anche se l'effetto di sbiancamento in soluzione potrebbe non essere così drastico come esso può essere in vivo, la traccia di intensità probabilmente non avrà ancora stazionaria media, probabi...
Discussione
N & B è una tecnica per rilevare multimerization utilizzando luce commerciale scansione microscopi confocali dotate di rilevatori digitali. Questo approccio è molto attraente rispetto al singolo punto FCS, FCCS e smFRET perché è libero di calibrazione e il calcolo di luminosità è semplice e indipendente di concentrazione6. È di grande importanza, tuttavia, per correggere le fluttuazioni di intensità di imbianchimento e a lungo termine prima di eseguire i calcoli di luminosità
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Questo lavoro è stato supportato da Wellcome Trust concedere 105278/Z/14/2 a R.N. Il Wellcome Trust Centre for Human Genetics è finanziato dalla Wellcome Trust Core premio 203852/Z/16/2. Lavoro nel gruppo C.S. è supportato da Cancer Research UK (C20724/A14414) e il Consiglio europeo della ricerca nell'ambito dell'Unione europea Horizon 2020 ricerca e innovazione programma Grant 647278.
Materiali
| Name | Company | Catalog Number | Comments |
| RosettaTM (DE3) pLysS cells | Novagen | 70956-3 | |
| Ampicillin | Sigma Aldrich | PubChem Substance ID 329824407 | |
| Chloramphenicol | Sigma Aldrich | PubChem Substance ID: 24892250 | |
| LB starter culture | QIAGEN | ||
| LB medium | QIAGEN | https://www.sigmaaldrich.com/content/dam/sigma-aldrich/head/search/external-link-icon.gif | |
| IPTG | Sigma Aldrich | PubChem Substance ID 329815691 | |
| IMAC buffer | Medicago | 09-1010-10 | |
| EDTA-free protease inhibitors | Sigma Aldrich | 11873580001 | |
| TALON resin | Clonetech | ||
| Nickel sepharose | GE Healthcare | ||
| S200 16/60 column | GE Healthcare | ||
| Glass bottom 8 well observation dish | Ibidi | 80827 |
Riferimenti
- Voith von Voithenberg, L., Lamb, D. C. Single pair forster resonance energy transfer: A versatile tool to investigate protein conformational dynamics. BioEssays. 40, (2018).
- Padilla-Parra, S., Auduge, N., Coppey-Moisan, M., Tramier, M. Quantitative FRET analysis by fast acquisition time domain FLIM at high spatial resolution in living cells. Biophysical Journal. 95, 2976-2988 (2008).
- Padilla-Parra, S., Auduge, N., Coppey-Moisan, M., Tramier, M. Dual-color fluorescence lifetime correlation spectroscopy to quantify protein-protein interactions in live cell. Microscopy Research and Technique. 74, 788-793 (2011).
- Muller, J. D., Chen, Y., Gratton, E. Fluorescence correlation spectroscopy. Methods in Enzymology. 361, 69-92 (2003).
- Tramier, M., Coppey-Moisan, M. Fluorescence anisotropy imaging microscopy for homo-FRET in living cells. Methods in Cell Biology. 85, 395-414 (2008).
- Digman, M. A., Dalal, R., Horwitz, A. F., Gratton, E. Mapping the number of molecules and brightness in the laser scanning microscope. Biophysical Journal. 94, 2320-2332 (2008).
- Nolan, R., Iliopoulou, M., Alvarez, L., Padilla-Parra, S. Detecting protein aggregation and interaction in live cells: A guide to number and brightness. Methods. , (2017).
- Dalal, R. B., Digman, M. A., Horwitz, A. F., Vetri, V., Gratton, E. Determination of particle number and brightness using a laser scanning confocal microscope operating in the analog mode. Microscopy Research and Technique. 71, 69-81 (2008).
- Nolan, R., et al. nandb-number and brightness in R with a novel automatic detrending algorithm. Bioinformatics. , (2017).
- Hur, K. H., et al. Quantitative Measurement of Brightness from Living Cells in the Presence of Photodepletion. PLoS One. 9, (2014).
- Amara, J. F., et al. A versatile synthetic dimerizer for the regulation of protein-protein interactions. Proceedings of the National Academy of Sciences of the United States of America. 94, 10618-10623 (1997).
- Clackson, T., et al. Redesigning an FKBP-ligand interface to generate chemical dimerizers with novel specificity. Proceedings of the National Academy of Sciences of the United States of America. 95, 10437-10442 (1998).
- Rollins, C. T., et al. A ligand-reversible dimerization system for controlling protein-protein interactions. Proceedings of the National Academy of Sciences of the United States of America. 97, 7096-7101 (2000).
- Schindelin, J., Rueden, C. T., Hiner, M. C., Eliceiri, K. W. The ImageJ ecosystem: An open platform for biomedical image analysis. Molecular Reproduction and Development. 82, 518-529 (2015).
- . R: A Language and Environment for Statistical Computing. R Core. , (2017).
- . RStudio: Integrated Development Environment for R. R Team. , (2016).
- . nandb R package Available from: https://CRAN.R-project.org/package=nandb (2017)
- Jung, G., Wiehler, J., Zumbusch, A. The photophysics of green fluorescent protein: influence of the key amino acids at positions 65, 203, and 222. Biophysical Journal. 88, 1932-1947 (2005).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nature Methods. 2, 905-909 (2005).
- Butkevich, A. N., et al. Hydroxylated fluorescent dyes for live-cell labeling: synthesis, spectra and super-resolution STED. Chemistry: A European Journal. 23, 12114-12119 (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon