Method Article
CRISPR/Cas9 Ribonucleoprotein-vermittelte präzise Genbearbeitung durch Tube Electroporation
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Hier wird ein Protokoll zur effizienten CRISPR/Cas9 Ribonucleoprotein-vermittelten Genbearbeitung in Säugetierzellen mittels Röhrenelektroporation vorgestellt.
Zusammenfassung
Gen-E-Editing-Nukleasen, vertreten durch CRISPR-assoziiertes Protein 9 (Cas9), werden zu Mainstream-Werkzeugen in der biomedizinischen Forschung. Die erfolgreiche Lieferung von CRISPR/Cas9-Elementen in die Zielzellen durch Transfektion ist Voraussetzung für eine effiziente Genbearbeitung. Dieses Protokoll zeigt, dass die maschinelle Lieferung von CRISPR/Cas9 Ribonucleoprotein (RNP) zusammen mit einsträngigen Oligodeoxynukleotid-Spendervorlagen (ssODN) an verschiedene Arten von Säugetierzellen zu robusten präzise Genbearbeitungsereignisse. Zunächst wurde TE angewendet, um CRISPR/Cas9 RNP und ssODNs zu liefern, um krankheitserregende Mutationen im Interleukin 2-Rezeptor-Subunit-Gamma (IL2RG)-Gen und Sepiapterin-Reduktase-Gen (SPR) in Kaninchen-Fibroblastenzellen zu induzieren. Genaue Mutationsraten von 3,57%-20% wurden erreicht, wie durch bakterielle TA-Klonsequenzierung bestimmt. Die gleiche Strategie wurde dann in menschlichen iPSCs auf mehreren klinisch relevanten Genen verwendet, einschließlich epidermaler Wachstumsfaktorrezeptor (EGFR), Myosin-bindendes Protein C, Herz (Mybpc3) und Hämoglobin-Untereinheit Beta (HBB). Konsequent wurden hochpräzise Mutationsraten erreicht (11,65%-37,92%) durch tiefe Sequenzierung (DeepSeq) bestimmt. Die vorliegende Arbeit zeigt, dass die Röhrenelektroporation von CRISPR/Cas9 RNP ein effizientes Transfektionsprotokoll für die Genbearbeitung in Säugetierzellen darstellt.
Einleitung
CRISPR/Cas9 ist die am häufigsten verwendete programmierbare Nuklease für die Genbearbeitung. Es funktioniert durch Single Guide RNA (sgRNA)-vermittelte Erkennung beider Zielsequenzen und einer angrenzenden protospacer benachbarten Motivsequenz (PAM) im Genom. Die Cas9-Nuklease erzeugt einen doppelsträngigen DNA-Bruch (DSB), der drei Nukleotide vor der PAM-Sequenz1lokalisiert. Die DSBs werden entweder durch fehleranfällige nicht-homologe Endverbindung (NHEJ) oder homologiegesteuerte Reparaturwege (HDR) repariert. Um eine präzise Genbearbeitung über den HDR-Signalweg zu erreichen, werden Spendervorlagen oft im Format plasmid-DNA (pDNA) oder einsträngigen Oligodeoxynukleotid (ssODN) bereitgestellt.
CRISPR/Cas9 und die sgRNA können in drei Formaten an die Zellen abgegeben werden: den Ribonukleoprotein (RNP) Komplex von Cas9-Protein und gRNA2,3; Cas9 mRNA und sgRNA4,5; oder Plasmid-DNA (pDNA), die die notwendigen Promotoren, angetriebene sgRNA und Cas9-Codierungsbereich6,7,8enthält. Viele Gruppen haben gezeigt, dass, wenn CRISPR/Cas9 als RNP geliefert wird, die Gen-Editing-Effizienz oft besser als die in pDNA- oder mRNA-Formaten erreichten, was auf die viel geringere Größe von RNP im Vergleich zu den Nukleinsäuren9zurückzuführen ist. Darüber hinaus hat sich bereits gezeigt, dass eine neuartige Röhrenelektroporation (TE) in Genbearbeitungsanwendungen in verschiedenen Zelltypen9besonders effektiv ist.
In der vorliegenden Arbeit wird ein schrittweises Protokoll zur Verwendung von TE für die Lieferung von CRISPR/Cas9 RNP an Säugetierzellen verschiedener Arten an mehreren klinisch relevanten Loci vorgestellt. Diese neuartige TE-Transfektionstechnik und das Phänomen der hohen HDR-Rate können breite Anwendungen in der biomedizinischen Forschung finden.
Protokoll
Alle Tierpflege-, Pflege- und Verwendungsverfahren wurden vom Institutional Animal Care and Use Committee (IACUC) der University of Michigan überprüft und genehmigt.
1. Vorbereitung der Zellen
-
Erwerben Sie humane iPSCs (ACS-1030) aus der American Type Culture Collection (ATCC). Kultur iPScs auf künstlicher extrazellulärer Matrix mit feederfreiem Zellkulturmedium (siehe Materialtabelle) in einem Zellkultur-Inkubator (5%CO2 bei 37 °C) nach den Anweisungen des Lieferanten.
- 2 h vor der Transfektion, behandeln Sie die iPSCs mit 10 M Rho-assoziierten, gewickelten Spulen enthaltenden Proteinkinase (ROCK) Inhibitor Y27632 (Verwendung von denen reduziert Apoptose von dissoziierten menschlichen HiPSCs und erhöht die Überlebens- und Kloneffizienz von hiPSCs ohne Auswirkungen auf ihre Pluripotenz).
- Dissoziieren Sie bei der Transfezierung iPSCs mit Zellablösung (siehe Materialtabelle)für 5 min. Zählen Sie die Zellzahl.
-
Etablieren Sie eine Kaninchen-Fibroblasten-Zellkultur mit einer primären Kultur von Kaninchen-Ohr-Hautgewebe-Biopsien, wie zuvor beschrieben10.
- Aus der Spitze des Kaninchenohrs wird eine 0,5 cm x 0,5 cm Ohrhautbiopsie gewonnen. Rasieren Sie die Haare vom Ohrgewebe ab.
- 2x mit Dulbecco-Phosphat-gepufferter Saline (DPBS) mit 5% Penicillin-Streptomycin abspülen. Übertragen Sie das Ohrgewebe in eine neue 6 cm Gewebekulturschale, und schneiden Sie das Gewebe dann in kleine Stücke (ca. 1,0 mm x 1,0 mm). Fügen Sie ein paar Tropfen fetales Rinderserum hinzu, um zu verhindern, dass das Gewebe austrocknet.
- Das geschredderte Gewebe auf eine 10 cm lange Gewebekulturschale verteilen und dann 10 ml Kulturmedium hinzufügen. Kaninchen-Fibroblastenzellen werden in Dulbeccoes Modified Eagle es Medium (DMEM) mit 10% fetalem Rinderserum kultiviert. Legen Sie die Gewebekulturschale in den Zellkultur-Inkubator (5%CO2 bei 37 °C).
- Drei bis fünf Tage nach der Beschichtung verwenden Sie trypsin-EDTA, um Zellen bei 37 °C für 2 min zu verdauen. Zählen Sie die Zellzahl.
2. Design und Synthese von gRNAs und Spenderoligos
- Für jedes Gen entwerfen Sie RNA basierend auf der Sequenz des Zielloks mithilfe eines Online-Tools (z. B. ).
- Fügen Sie in die DNA-Sequenz von Interesse.
- Wählen Sie ein Genom und ein protospacer-benachbartes Motiv (PAM). Mögliche Führungssequenzen in Eingabe-DNA-Sequenzen werden auf der Ausgabeseite angezeigt. Es wird empfohlen, gRNA mit höherer vorhergesagter Effizienz und niedrigeren Off-Target-Potenzialen auszuwählen.
- Synthetisieren Sie DNA von einem kommerziellen Anbieter für die Transkribierung von gRNAs. Führen Sie die In-vitro-Transkription von gRNA mit einem gRNA-Synthesekit gemäß den Anweisungen des Herstellers durch.
- Reinigen Sie die gRNA mit einer RNA-Reinigungsmikrosäule, die im gRNA-Synthesekit enthalten ist. Messen Sie die Konzentration, und lagern Sie dann die gRNAs bei -80 °C.
- Entwerfen Sie eine ssODN-Spendervorlage für jede Mutationssite. Die ssODNs können von kommerziellen Anbietern wie IDT synthetisiert werden. Im Allgemeinen ist jede ssODN 120-160 Nukleotide (nt) lang, bestehend aus 60-80 nt im linken Homologiearm und 60-80 nt im rechten Homologiearm. Um eine Nachzerlegung der bearbeiteten DNA zu verhindern, sollte nach Möglichkeit eine stille Mutation am PAM in die ssODN eingeführt werden. Die CRISPR-Schnittstelle sollte so nah wie möglich an der beabsichtigten genomischen Veränderung liegen.
3. Rohrelektroporation von Cas9 RNP und ssODNs
- Bereiten Sie die Zellen wie in Abschnitt 1 beschrieben vor.
- 2-3 x 105 Zellen in 20 l Elektroporationspuffer wieder aufhängen. Pipette vorsichtig nach oben und unten, um eine einzellige Suspension zu erzeugen.
- Für Cas9 RNP-Transfektion 2 g Cas9-NLS-Protein mit 0,67 gRNA bei Raumtemperatur (RT) für 10-15 min vormischen. Als nächstes mischen Sie den gebildeten RNP-Komplex vorsichtig zusammen mit 2 g ssODN mit Zellen.
- Übertragen Sie das Zellgemisch mit universellen Pipettenspitzen des Rohrelektroporationskits auf ein 20-L-Elektroporationsrohr. Um eine bessere Elektroporation zu erreichen, versuchen Sie, die Bildung von Luftblasen während der Übertragung zu vermeiden.
- Legen Sie das Elektroporationsrohr in den Schlitz des Elektroporators und drücken Sie "Go", um zu beenden. Befolgen Sie die vom Hersteller vorgeschlagenen Parameter für jeden Zelltyp. Bei menschlichen iPSCs und Kaninchen-Fibroblastenzellen beträgt der Spannungssatz beispielsweise 420 V und die Pulszeit 30 ms. Ein erfolgreicher Elektroporationszyklus wird durch den Impulsbericht auf dem Bildschirm des Elektroporators angezeigt.
- Übertragen Sie nach der Elektroporation die menschlichen iPS-Zellen auf 1 ml vorgewärmtes Y-27632-haltiges Kulturmedium, das im Zellkulturteil beschrieben wird. Für Kaninchen-Fibroblastenzellen, übertragen Sie sie zu DMEM mit 10% fetalem Rinderserum.
- Die resuspendierten Zellen auf einen Brunnen einer 12 Brunnenzellkulturplatte aufteilen.
- Verändern Sie das Kulturmedium jeden Tag. Y-27632 wird aus dem humanen iPSC-Kulturmedium 24 h Nachelektroporation entfernt.
4. Analyse von Gene Editing Events
- Erntezellen 72 h nach Elektroporation. Verdauungszellen aus der Kulturplatte mit Trypsin-EDTA für Kaninchen-Fibroblastenzellen oder Zellablösung für menschliche iPSCs. Nach Zentrifuge Zellen mit 350 ml Lysepuffer (1 M Tris HCl, 5 M NaCl, 0,5 M EDTA; pH 8,0, 10% SDS, 20 l von 20 mg/ml Proteinase K-Bestand pro 1 ml Lysepuffer hinzufügen) und dann über Nacht bei 55 °C inkubieren.
- Extrahieren Sie die genomische DNA mit Phenol-Chloroform nach Standardverfahren.
- Amplify 100-200 bp DNA-Fragmente mit Zielregion mit High-Fidelity-DNA-Polymerase, dann reinigen Sie die DNA-Fragmente von Gelen mit einem Gel-Extraktions-Kit oder direkt aus PCR-Produkten mit einem PCR SV Mini-Kit.
- Um die Effizienz der Genbearbeitung durch bakterielle Koloniesequenzierung zu bestimmen, ligagen Sie die gereinigten PCR-Produkte mithilfe eines TOPO TA-Klonkits in einen pCR4-TOPO-Vektor. Nehmen Sie zufällig bakterielle Klone auf und sequenzieren Sie die Einsätze dann mit einem universellen Sequenzierungsprimer, der vom TOPO TA-Klonkit bereitgestellt wird.
- Um die Effizienz der Genbearbeitung durch tiefe Sequenzierung zu bestimmen, senden Sie die gereinigten PCR-Produkte (100-200 bp) aus Schritt 4.3 für die CRISPR-Amplicon-Sequenzierung in einem DNA-Sequenzierungskern.
Ergebnisse
TE von Cas9 RNP und ssODNs an Kaninchen-Fibroblastenzellen
Der Gesamtprozess der TE-vermittelten Lieferung von Cas9 RNP an Säugetierzellen ist in Abbildung 1dargestellt. Zunächst wurden C231Y- und Q235X-Mutationen im IL2RG-Gen und die R150G-Mutation im SPR-Gen in Kaninchen-Fibroblastenzellen produziert. Verlust-von-Funktion-Mutationen in IL2RG und SPR-Genen sind dafür bekannt, primäre Immunschwäche11 und motorische und kognitive Defizite12zu verursachen.
Die spezifischen sgRNA-Designs sind in Abbildung 2Adargestellt. Die Primer, mit denen die Zielregionen verstärkt werden, sind in Tabelle 3aufgeführt. Sequenzen von ssODNs sind in Tabelle 1dargestellt. Die Gen-Editing-Raten wurden durch bakterielles TA-Klonen bestimmt (Abbildung 2B). Am IL2RG C231-Lokus wurde von den 28 gesequenzten Klonen einer (3,57%) trug die präzise C231Y-Mutation, vier (14,28%) ein- oder löschende (Indel-) Mutationen und die restlichen 23 (82%) wild-typ. Am IL2RG Q235-Lokus wurden von den 27 gesequenzten Klonen zwei (7,41%) trug die präzise Q235X-Mutation, drei trugen Indelmutationen (11,11%) und die übrigen waren wild-typ. Bei der SPG R150 locus, von den 20 gestaffelten Klonen, fünf (25%) trug die präzise R150G-Mutation, 10 (50%) indel Mutationen getragen, und die übrigen waren Wildtyp.
TE von Cas9 RNP und ssODNs an humane iPSCs
TE wurde dann verwendet, um Cas9 RNP und ssODNs an menschliche iPSCs zu liefern und klinisch relevante Loci in EGFR-, Mybpc3- und HBB-Genen anzuvisieren. Punktmutationen in der proximalen EGFR-T790-Region verleihen EGFR-Tyrosinkinase-Inhibitoren bei Patienten mit nicht-kleinzelligem Lungenkrebs (NSCLC) eine Resistenz, die aktivierende Mutationen von EGFR13enthält. Eine Frameshift-Mutation in Exon 16 in Mybpc3 ist an hypertropher Kardiomyopathie14beteiligt. Die E6V-Punktmutation im HBB-Gen führt zu einer Sichelzellerkrankung15.
Die spezifischen sgRNA-Designs sind in Abbildung 3Adargestellt. Die Primer, mit denen die Zielregionen verstärkt werden, sind in Tabelle 3aufgeführt. Sequenzen von ssODNs sind in Tabelle 1dargestellt. Die Gen-Editing-Raten wurden von DeepSeq bestimmt (Abbildung 3B). Am EGFR-Ort trugen 15,68% der Allele die genauen Punktmutationen (6.315 Lesevorgänge), 22,75% trugen Indelmutationen (9.162 Lesevorgänge) und die restlichen 61,57% waren Wildtyp (24.797 Lesevorgänge). Am Mybpc3-Ort trugen 37,92% die präzise 4-bp TGAA-Deletion (11.654 Lesevorgänge), 2,24% trugen Indelmutationen (410 Lesevorgänge) und die restlichen 59,84% waren Wildtyp (18.692 Lesevorgänge). Am HBB-Ort trugen 11,65% die präzise E6V-Mutation (6.565 Lesevorgänge), 23,35% trugen Indelmutationen (13.163 Lesevorgänge) und die restlichen 65% waren Wildtyp (36.644 Lesevorgänge).

Abbildung1: Flussdiagramm der Rohrelektroporation von Cas9 RNP.

Abbildung 2 : Genbearbeitung von Kaninchen-Fibroblastenzellen. (A) Abbildung von Zielsequenzen. Felder zeigen gezielte Loci an. Unterstrichene Buchstaben entsprechen gRNA-Sequenzen. Rote Buchstaben zeigen PAM-Sequenzen an. (B) TA-Klonergebnisse von Gen-Editing-Ereignissen. Boxen zeigen genau mutierte loci. Die dargestellte Indelsequenz ist nur repräsentativ für einen Alleltyp. Andere Indel-Sequenzen werden nicht angezeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
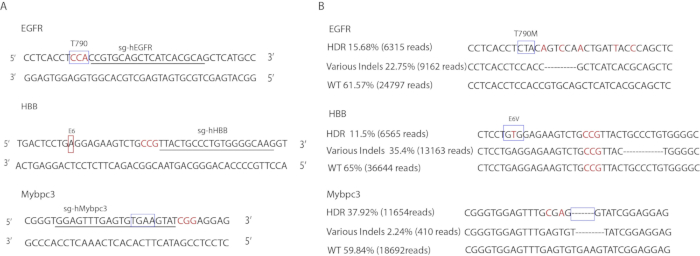
Abbildung 3 : Genbearbeitung menschlicher iPSCs. (A) Abbildung von Zielsequenzen. Felder zeigen gezielte Loci an. Unterstrichene Buchstaben entsprechen der gRNA-Sequenz. Rote Buchstaben zeigen PAM-Sequenzen an. (B) Deepseq Ergebnisse von Gen-Editing-Ereignissen. Boxen zeigen genau mutierte loci. Rot gefärbte Buchstaben weisen auf stille Mutationen hin, die in die Spendervorlagen eingeführt wurden. Die dargestellte Indelsequenz ist nur repräsentativ für einen Alleltyp. Andere Indel-Sequenzen werden nicht angezeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Locus |
Oligo-Sequenz | |||||||
| (gezielte Mutation) | ||||||||
| Kaninchen IL2RG (C231Y) | AGCGTGGATGGGCAGAAACTCTACACGTTCCGAGTCCGGAGCCGTTTTAACCCTTTGTATGGGAGTGCTCAGCTTGGAGT GAATGGAGCCCCCGATCCGGGGGGGAAAACTTCAAAGGGTAAAATGGGCCT | |||||||
| Kaninchen IL2RG (Q235X) | AGCGTGGATGGGCAGAAACTCTACACGTTCCGAGTCCGGAGCCGTTTTAACCCTTTGTGTGGGGGGCTCTTAGCTTGGAGT GAATGGAGCCCCCGATCCGGGGGGGAAAACTTCAAAGGGTAAAATGGGCCT | |||||||
| Kaninchen SPR | gacctccatgctctgcctgacctcctgcatcctgaaggcgtttcctgccagtcctggCctcagcgggctagtgaacatctcgtcgctgtgtgtgtgtgccctgcagcccttcaaagggctggg cgctgtac | |||||||
| (R150G) | ||||||||
| Human EGFR | ACGTGATGGCCAGCGTGGACAACCCCCACGTGTGCCGCCTGCTGGGCTCTGCCTCACCTCTACTACAGTCCAACTGATTACCC AGCTCTCTCTGGCTCTCTCTCTTGGACTATGTCCGGGAACACAAAAGACAATATTGGCTCCCAGTAC | |||||||
| (Punktmutationen in der Nähe von T790) | ||||||||
| Human Mybpc3 | GCCCCCTGTGCTCATCACGCGCTZTTGGAGGACCAGCTGGTGATGGTGGGGCAGCGGGTGGAGTTTGCGAGGT GGAGGGGGCGCAAGTCAAATGGTGAGTTCGAGAAGGGGGCATGGGTGTGGGGGGCAT | |||||||
| (4-bp-Löschung) | ||||||||
| Human HBB | TCTGACACAACTGTGTTCACTAGCAACCTCAAACAGACACCATGGTGCATCTGACTCCTGTGGAGAAGTCTGCAGTTACTGCC CTGTGGGGCAAGGTGAACGGATGAAGTTGGTGGTGAGGCCCTGGGCAG | |||||||
| (E6V) | ||||||||
Tabelle 1: Sequenzen von ssODNs.
| schritt | problem | Mögliche Gründe | Lösungen |
| 2.1 | Niedrige Indel-Rate | Schlechtes Guide-RNA-Design, Guide-RNA-Bestände >6 Monate, niedrige RNA-Konzentration | Redesign Guide RNA, produzieren/bestellen Sie neue Guide RNA. |
| 2,3 | Geringer PGE-Wirkungsgrad | schlechtes Spender-DNA-Design, niedrige effiziente Führungs-RNA, falsche Menge an Spender-DNA oder schlechte DNA-Qualität | Erhöhen Sie die Homologie-Armlänge, führen Sie PAM-Mutationen ein, führen Sie stille Mutationen in die Spender-DNA ein, verwenden Sie eine effizientere Guide-RNA, Optimieren Sie das Verhältnis von Cas9-Protein zu Guide-RNA. |
| 3.4 | Gescheiterte Transfektion | Luftblasen, die sich bei der Übertragung von Zellen-Puffer-Gemisch auf Elektroporationsrohr, falsche Spannungs-/Dauereinstellung gebildet | Versuchen Sie, die Bildung von Luftblasen zu vermeiden, stellen Sie die Spannungs-/Dauereinstellung ein. |
| 3.6 | geringe Zelllebensfähigkeit nach Elektroporation | Geringes Überleben von einzelnen menschlichen ipsc | Fügen Sie ROCK-Hemmer nach der Elektroporation hinzu, erhöhen Sie die Anzahl der Zellen. |
| 4.1 | Fehlerhafte PCR | Hoher GC-Inhalt oder sich wiederholende Sequenz | Optimieren Sie die PCR-Bedingung, fügen Sie DMSO zum PCR-System hinzu. |
Tabelle 2: Anleitungen zur Fehlerbehebung bei häufigen Problemen.
| Primername | folge | notiz |
| Rb-IL2RG-F | CATGACAGTGACAGGGTCCC | Zur Verstärkung des Kaninchens IL2RG DNA-Fragment |
| Rb-IL2RG-R | TGCCAGAGACACAAGCGAAC | |
| Rb-SPR-F | GTACTTTGGAGGGACAGAGG | Zur Verstärkung des Kaninchens SPR DNA-Fragment |
| RB-SPR-R | CTCAGCACCCTGACACTGGG | |
| H-EGFR-F | TGATGGCCAGCGTGGACAAC | Zur Verstärkung des menschlichen EGFR-DNA-Fragments |
| H-EGFR-R | ACCAGTTGAGCAGGTACTGGG | |
| H-Mybpc3-F | ATGCCCCGTGCTTCTGGAAC | Zur Verstärkung des menschlichen Mybpc3-DNA-Fragments |
| H-Mybpc3-R | TCAGGGGAGCCAACCCTCAT | |
| H-HBB-F | TAACCTTGATACCAACCTGC | Zur Verstärkung des menschlichen HBB-DNA-Fragments |
| H-HBB-R | CATTTGCTTCTGACACAACT |
Tabelle 3: In Schritt 4.3 verwendete Grundierungen.
Diskussion
Die Tubeelektroporation Methode war wirksam bei der Bereitstellung von CRISPR/Cas9 RNP und ssODNs an Kaninchen und menschliche Zellen, was zu einer robusten präzisen Genbearbeitung (PGE) führte. Der Hauptunterschied zwischen TE und anderen konventionellen Elektroporationsgeräten ist die Verwendung eines Rohres, in dem sich zwei Elektroden oben und unten des Rohres befinden und die Probe vollständig geladen und bei Elektroporation versiegelt wird (Abbildung 1). Im Gegensatz dazu befinden sich in einer konventionellen Küvette die Elektroden an den Seiten und die Probe wird während der Elektroporation nicht vollständig abgedichtet. Dieses neue Design reduziert die Erzeugung von Luftblasen und komprimiert die Luftblasengröße, was die gleichmäßige Verteilung der elektrischen Spannung verbessert und dadurch zu einem reduzierten Zelltod und einer hohen Transfektionseffizienz9führt. In der vorliegenden Arbeit sind hohe PGE-Raten (15%-37%) EGFR-, Mybpc3- und HBB-Gene in humanen iPSCs erreicht wurden. Diese Ergebnisse stimmen mit einem früheren Bericht überein, in dem hohe PGE-Raten in menschlichen Stammzellen erreicht wurden9.
Krankheitserregende Mutationen wurden in IL2RG- und SPR-Genen in Kaninchenzellen gezielt. Kürzlich wurden IL2RG-Knockout-Kaninchen als Modelle für menschliche X-verknüpfte schwere kombinierte Immunschwäche (SCID-X1)16,17produziert. Die vorliegende Arbeit zeigt, dass Il2RG-Mutationen des Patienten (z. B. C231Y und Q235X) effizient in Kaninchenzellen erzeugt werden können, was die Machbarkeit der Erstellung von SCID-X1-Kaninchenmodellen mit Patientenmutationen demonstriert. Es wurde auch gezeigt, dass SPR R150G Mutationen effizient in Kaninchenzellen erzeugt werden können. Diese Mutation verursacht motorische und kognitive Defizite bei Kindern12. Diese EINMAL erzeugten IL2RG- und SPR-Mutationskaninchenmodelle können als wertvolle präklinische Modelle für translationale Studien dienen. Sie können auch verwendet werden, um Gen-Editing-basierte Therapeutika für diese monogenen Krankheiten zu etablieren.
Ein Anliegen von CRISPR/Cas9-vermittelten Gen-Editing-Anwendungen sind die Off-Target-Bearbeitungsereignisse. Die Indelraten wurden an vorhergesagten Top-Off-Target-Standorten für sgRNAs analysiert, die in dieser Studie verwendet wurden (Tabelle S1), mit Methoden, die zuvor beschrieben wurden9. Insgesamt wurden sieben potenzielle Top-Off-Target-Loci für sg-rb-IL2RG-01, fünf für sg-rb-SPR, sieben für sg-hEGFR, fünf für sg-hMybpc3 und sieben für sg-hHBB) mit den in Tabelle S2aufgeführten Primern analysiert. Die T7E1-Assays (Abbildung S1), die auf minimale Off-Target-Risiken für die CRISPR/Cas9-vermittelte Genbearbeitung mit diesen sgRNAs hindeuten, zeigten keine Off-Target-Indels an. Es zeigt auch an, dass die TubeElektroporation-Methode selbst keine Off-Target-Bearbeitungen verursacht oder erhöht. Dennoch sollten Anstrengungen unternommen werden, um unerwünschte Off-Target-Bearbeitungen zu reduzieren oder zu beseitigen. Eine Sequenzierung des ganzen Genoms kann erforderlich sein, um solche Ereignisse für Zellen auszuschließen, die für die Verwendung in klinischen Anwendungen bestimmt sind.
Auf technischer Ebene werden die folgenden Schlüsselfaktoren für eine effiziente präzise Genombearbeitung durch CRISPR/Cas9 RNP-Röhrenelektroporation betrachtet. Zunächst wird empfohlen, eine effiziente sgRNA mit vorhergesagtem niedrigen Off-Target-Potenzial auszuwählen. Es ist wichtig, die Indel-Effizienz der ausgewählten sgRNA zu validieren, bevor sie für PEG-Anwendungen verwendet wird. Es ist nicht selten, dass eine Software, die eine gute sgRNA vorhergesagt hat, beim Validierungsschritt fehlschlägt.
Zweitens wird empfohlen, dem ssODN-Spender nach Möglichkeit eine PAM-Mutation zu induzieren, um eine hohe PGE zu erreichen. Der Grund dafür ist, dass dadurch crispR/Cas9 nach der Integration von Spendervorlagen erneut geschnitten wird. In bestimmten Fällen führt die PGE selbst PAM-Mutationen ein. In anderen Fällen ist es möglich, stille Mutationen in die PAM-Sequenz einzuführen. Für den Fall, dass eine PAM-Mutation nicht möglich ist, wird empfohlen, mehrere stille Mutationen in den Spender aufzunehmen, die der sgRNA-Sequenz entsprechen.
Drittens, besonders relevant für TE, ist es wichtig, die Bildung von Luftblasen zu vermeiden, wenn Zellen und RNP-Gemisch auf das Elektroporationsrohr übertragen werden. Während das Design eines TE-Rohrs bereits die Luftblasenbildung minimiert, wird eine sorgfältige Handhabung die Bildung von Luftblasen weiter reduzieren und sogar die Luftblasenbildung vollständig vermeiden. Eine Anleitung zur Fehlerbehebung für häufige Probleme, die bei der Anwendung der Rohrelektroporation für CRISPR/Cas9 Ribonucleoprotein vermittelte präzise Genbearbeitung auftreten können, ist in Tabelle 2aufgeführt.
Zusammenfassend lässt sich hier zeigen, dass die Röhrenelektroporation ein wirksames Mittel zur Abgabe von CRISPR/Cas9 RNP und ssODNs an Säugetierzellen ist, um hohe PGE-Raten zu erreichen. Diese neue TE-Transfektionstechnik und ihre robuste präzise Gen-Editing-Rate können die Entwicklung von Gen-Editing-Anwendungen erleichtern.
Offenlegungen
J. C. arbeitet bei Celetrix LLC, Hersteller des Rohrelektroporators. L. M., L. J., J. S., D. Y., J. Z., Y. E. C. und J. X. erklären keine konkurrierenden Interessen.
Danksagungen
Diese Arbeit wurde von den National Institutes of Health (R21OD023194 bis JX) unterstützt. Diese Arbeit nutzte Core Services, die vom Center for Advanced Models for Translational Sciences and Therapeutics (CAMTraST) am University of Michigan Medical Center unterstützt wurden.
Materialien
| Name | Company | Catalog Number | Comments |
| Accutase | STEMCELL Technologies | 792 | Cell detachment solution for human iPSCs, first used in Step 1.1.2. |
| Cas9 Nuclease 3NLS | IDT | 1074182 | Cas9 protein, first used in Step 3.3. |
| DMEM | Thermo Fisher | 11965092 | For cell culture, first used in Step 1.2.3. |
| DPBS | Thermo Fisher | 1708075 | For preparing cell culture, first used in Step 1.2.2. |
| EDTA | Lonza | 51201 | For making lysis buffer, first used in Step 4.1. |
| Electroporation buffer | Celetrix | 13–0104 | The electroporation buffer, first used in Step 3.2. |
| Electroporation tubes | Celetrix | 20 μL: 12–0107; 120 μL: 12–0104 | The electroporation tube, first used in Step 3.4. |
| Electroporator | Celetrix | CTX-1500A LE | The tube electroporation machine, first used in Step 3.5 |
| Fetal bovine serum | Sigma Aldrich | 12003C | For cell culture, first used in Step 1.2.2. |
| Forma CO2 Incubators | Thermo Fisher | Model 370 | For cell culture, first used in Step 1.1. |
| Gel Extraction Kit | Qiagen | 28115 | For gel purification, first used in Step 4.3. |
| Human induced pluripotent stem cells | American Type Culture Collection | ACS-1030 | Human iPSCs, first used in Step 1.1. |
| Matrigel | Corning | 354277 | Artificial extracellular matrix; for precoating cell culture plate, first used in Step 1.1. |
| mTeSR 1 medium | STEMCELL Technologies | 85850 | Feeder-free cell culture medium for human iPSCs, first used in Step 1.1. |
| PCR SV mini | GeneAll | 103-102 | For PCR product purification, first used in Step 4.3. |
| Penicillin-Streptomycin | Thermo Fisher | 15140163 | For preparing cell culture, first used in Step 1.2.2. |
| Phenol-chloroform | Thermo Fisher | 15593031 | For DNA extraction, first used in Step 4.2. |
| Precision gRNA Synthesis Kit | Invitrogen | A29377 | For the generation of full length gRNA (guide RNA), first used in Step 2.4. |
| Proteinase K Solution | Thermo Fisher | AM2548 | For DNA extraction, first used in Step 4.1. |
| Q5 high-fidelity DNA polymerase | NEB | M0491 | For PCR amplification, first used in Step 4.3. |
| Sodium dodecyl sulfate | Sigma Aldrich | L3771 | For making lysis buffer, first used in Step 4.1. |
| TA Cloning Kit | Thermo Fisher | K457502 | For TA clone sequencing, first used in Step 4.4. |
| Tissue Culture Dish (10 cm) | FALCON | 353003 | For cell culture, first used in Step 1.2.3. |
| Tissue Culture Dish (12 well) | FALCON | 353043 | For cell culture, first used in Step 3.7. |
| Tissue Culture Dish (6 cm) | FALCON | 353004 | For cell culture, first used in Step 1.2.2. |
| Tris HCl | Thermo Fisher | BP1757-500 | For making lysis buffer, first used in Step 4.1. |
| Trypsin-EDTA | Thermo Fisher | 25200056 | For cell digestion, first used in Step 1.2. 4. |
| Universal Fit Pipette Tips | Celetrix | 14-0101 | For electroporation, first used in Step 3.4. |
| Y27632 | LC Labs | Y-5301 | The apoptosis inhibotor, first used in Step 1.1.1. |
Referenzen
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Mout, R., et al. Direct Cytosolic Delivery of CRISPR/Cas9-Ribonucleoprotein for Efficient Gene Editing. ACS Nano. 11 (3), 2452-2458 (2017).
- Zuris, J. A., et al. Cationic lipid-mediated delivery of proteins enables efficient protein-based genome editing in vitro and in vivo. Nature Biotechnology. 33 (1), 73-80 (2015).
- Miller, J. B., et al. Non-Viral CRISPR/Cas Gene Editing In Vitro and In Vivo Enabled by Synthetic Nanoparticle Co-Delivery of Cas9 mRNA and sgRNA. Angew Chem Int Ed Engl. 56 (4), 1059-1063 (2017).
- Finn, J. D., et al. A Single Administration of CRISPR/Cas9 Lipid Nanoparticles Achieves Robust and Persistent In Vivo Genome Editing. Cell Reports. 22 (9), 2227-2235 (2018).
- Liang, C., et al. Tumor cell-targeted delivery of CRISPR/Cas9 by aptamer-functionalized lipopolymer for therapeutic genome editing of VEGFA in osteosarcoma. Biomaterials. 147, 68-85 (2017).
- Luo, Y. L., et al. Macrophage-Specific in Vivo Gene Editing Using Cationic Lipid-Assisted Polymeric Nanoparticles. ACS Nano. 12 (2), 994-1005 (2018).
- Wang, H. X., et al. Nonviral gene editing via CRISPR/Cas9 delivery by membrane-disruptive and endosomolytic helical polypeptide. Proceedings of the National Academy of Sciences of the United States of America. 115 (19), 4903-4908 (2018).
- Xu, X., et al. Efficient homology-directed gene editing by CRISPR/Cas9 in human stem and primary cells using tube electroporation. Scientific Reports. 8 (1), 11649 (2018).
- Du, F., et al. Beneficial effect of young oocytes for rabbit somatic cell nuclear transfer. Cloning Stem Cells. 11 (1), 131-140 (2009).
- Allenspach, E., Rawlings, D. J., Scharenberg, A. M., Adam, M. P., et al. . GeneReviews(R). , (1993).
- Friedman, J., et al., Adam, M. P., et al. . GeneReviews(R). , (1993).
- Hidaka, N., et al. Most T790M mutations are present on the same EGFR allele as activating mutations in patients with non-small cell lung cancer. Lung Cancer. 108, 75-82 (2017).
- Ma, H., et al. Correction of a pathogenic gene mutation in human embryos. Nature. 548 (7668), 413-419 (2017).
- Vakulskas, C. A., et al. A high-fidelity Cas9 mutant delivered as a ribonucleoprotein complex enables efficient gene editing in human hematopoietic stem and progenitor cells. Nature Medicine. 24 (8), 1216-1224 (2018).
- Song, J., et al. Bacterial and Pneumocystis Infections in the Lungs of Gene-Knockout Rabbits with Severe Combined Immunodeficiency. Frontiers in Immunology. 9, 429 (2018).
- Song, J., et al. Production of immunodeficient rabbits by multiplex embryo transfer and multiplex gene targeting. Scientific Reports. 7 (1), 12202 (2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten