Method Article
CRISPR/Cas9 Ribonucleoproteína-mediada por Gene preciso de edição por tubo Electroporation
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
É apresentado aqui um protocolo para a edição eficiente do gene de CRISPR/Cas9 ribonucleoprotein-negociado em pilhas de mamíferos usando o electroporation do tubo.
Resumo
As nucleases de edição genética, representadas pela proteína 9 (Cas9) associada à CRISPR, estão se tornando ferramentas mainstream na pesquisa biomédica. A entrega bem-sucedida de elementos CRISPR/Cas9 nas células alvo por transfecção é um pré-requisito para a edição eficiente do gene. Este protocolo demonstra que a entrega mediada por máquina do Electroporation do tubo (TE) do ribonucleoprotein de CRISPR/Cas9 (RNP), junto com moldes único-encalhado do doador do oligodeoxynucleotide (ssODN) aos tipos diferentes de pilhas de mamíferos, conduz a robusto eventos de edição de genes precisos. Primeiramente, o te foi aplicado para entregar crispr/Cas9 RNP e ssodns para induzir mutações causadoras da doença no gene da subunidade gama (IL2RG) do receptor da interleucina 2 e no gene da redutase do sepiapterina (SPR) em pilhas do fibroblasto do coelho. As taxas de mutação precisa de%-20% foram conseguidas como determinado pelo sequenciamento bacteriano da clonagem de TA. A mesma estratégia foi usada então em iPSCs humanos em diversos genes clìnica relevantes que incluem o receptor epidérmico do fator de crescimento (EGFR), a proteína de ligação C do miosina, cardíaco (Mybpc3), e beta da subunidade da hemoglobina (HBB). Consistentemente, as taxas de mutação altamente precisas foram alcançadas (11.65%-37,92%) como determinado pelo seqüenciamento profundo (DeepSeq). O presente trabalho demonstra que o electroporation do tubo de CRISPR/Cas9 RNP representa um protocolo eficiente do transfection para a edição do gene em pilhas de mamíferos.
Introdução
CRISPR/Cas9 é o nuclease programável o mais geralmente usado para a edição do gene. Trabalha com o único RNA do guia (sgRNA)-reconhecimento negociado de ambas as seqüências do alvo e uma seqüência adjacente do motivo do protospacer adjacente (PAM) no genoma. A nuclease Cas9 gera uma ruptura de DNA de duas cordas (DSB) localizada a partir de três nucleotídeos a montante da sequência PAM1. Os DSBs são reparados através das vias de junção de final não homólogas propensas a erros (NHEJ) ou de reparação dirigida por homologia (HDR). Para obter a edição precisa do gene através da via HDR, os modelos de doadores são frequentemente fornecidos no formato de DNA plasmídeo (pDNA) ou oligodeoxinucleotídeo de uma única fita (ssODN).
Crispr/Cas9 e o sgrna podem ser entregues nas células em três formatos: o complexo de ribonucleoproteína (RNP) de proteína Cas9 e grna2,3; Cas9 mRNA e sgrna4,5; ou DNA plasmídeo (PDNA) que contém os promotores necessários, sgrna impulsionado, e região de codificação de Cas96,7,8. Muitos grupos demonstraram que, quando CRISPR/Cas9 é entregue como RNP, a eficiência de edição de genes supera frequentemente as alcançadas nos formatos pDNA ou mRNA, atribuíveis ao tamanho muito menor de RNP em comparação com os ácidos nucleicos9. Além disso, tem sido mostrado previamente que uma máquina nova do Electroporation do tubo (TE) é particular eficaz em aplicações de edição do gene em diversos tipos da pilha9.
Apresentado no presente trabalho é um protocolo passo-a-passo na utilização de TE para a entrega de RNP CRISPR/Cas9 a células de mamíferos de diferentes espécies em vários locos clinicamente relevantes. Esta novela técnica do transfection do TE e o fenômeno elevado da taxa de HDR podem encontrar aplicações largas na pesquisa biomédica.
Protocolo
Todos os procedimentos de manutenção, cuidados e uso de animais foram revisados e aprovados pelo Comitê institucional de cuidado e uso de animais (IACUC) da Universidade de Michigan.
1. preparação de células
-
Adquira iPSCs humanos (ACS-1030) da coleção de cultura de tipo americano (ATCC). Cultura iPScs na matriz extracelular artificial com meio de cultura celular livre de alimentador (ver tabela de materiais) em uma incubadora de cultura celular (5% CO2 a 37 ° c) seguindo as instruções do fornecedor.
- 2 h antes do transfection, trate os iPSCs com 10 μm Rho-associado, coiled-bobina que contem o inibidor da quinase da proteína (rocha) Y27632 (a utilização de que reduz o apoptose de hipscs humanos dissociada e aumenta a eficiência da sobrevivência e do clonagem de hipscs sem que afetam a sua pluripotência).
- Quando transfecting, dissociar iPSCs com solução do destacamento da pilha (veja a tabela dos materiais) às únicas pilhas no ° c 37 por 5 minutos. conte o número da célula.
-
Estabeleça uma cultura da pilha do fibroblasto do coelho usando uma cultura preliminar de biópsias do tecido da pele da orelha de coelho, como descrito previamente10.
- Uma biópsia da pele da orelha de 0,5 cm x de 0,5 cm é obtida da ponta da orelha de coelho. Raspar o cabelo do tecido da orelha.
- Enxague 2x com solução salina tamponada de fosfato de Dulbecco (DPBS) com 5% de penicilina-estreptomicina. Transfira o tecido da orelha a um prato novo da cultura do tecido de 6 cm, a seguir corte o tecido em partes pequenas (~ 1,0 milímetros x 1,0 milímetros). Acrescente algumas gotas de soro fetal bovino para evitar que o tecido seque.
- Espalhe o tecido desfiado a um prato da cultura do tecido de 10 cm, a seguir adicione 10 ml do meio de cultura. As células de fibroblastos de coelhos são cultivadas em meio de águia modificada de Dulbecco (DMEM) com soro bovino fetal a 10%. Põr o prato da cultura do tecido na incubadora da cultura da pilha (5% CO2 em 37 ° c).
- Três a cinco dias após o chapeamento, use Trypsin-EDTA para digerir as células a 37 ° c por 2 min. conte o número da célula.
2. projeto e síntese de gRNAs e oligos doadores
- Para cada gene, o RNA do guia de projeto baseou na seqüência do locus alvejado usando uma ferramenta em linha (por exemplo, < http://crispor.tefor.net/>).
- Colar na seqüência de DNA de interesse.
- Selecione um genoma e um motivo de protoespaçador adjacente (PAM). As sequências de guia possíveis nas sequências de DNA de entrada serão exibidas na página de saída. Recomenda-se selecionar gRNA com maior eficiência prevista e menores potenciais fora do alvo.
- Sintetizar DNA por um vendedor comercial para transcrever gRNAs. Realize a transcrição in vitro do gRNA utilizando um kit de síntese gRNA de acordo com as instruções do fabricante.
- Purify o gRNA usando uma micro coluna da purificação do RNA incluída no jogo da síntese de gRNA. Meça a concentração, a seguir armazene o gRNAs em-80 ° c.
- Projete um molde do doador de ssODN para cada local da mutação. O ssODNs pode ser sintetizado por vendedores comerciais como o IDT. Em geral, cada ssodn é 120-160 nucleotídeos (NT) de comprimento, consistindo de 60-80 NT no braço de homologia esquerda e 60-80 NT no braço de homologia direita. Para impedir a retifica do ADN editado, uma mutação silenciosa no Pam deve ser introduzida no ssodn sempre que possível. O local de corte CRISPR deve ser localizado o mais próximo possível da mudança genômica pretendida.
3. tubo Electroporation de Cas9 RNP e ssODNs
- Prepare as células conforme descrito na seção 1.
- Ressuscitação 2-3 x 105 células em 20 μL de tampão de electroporação. Pipeta acima e para baixo com cuidado para produzir uma suspensão da único-pilha.
- Para transfecção de Cas9 RNP, premix 2 μg de proteína Cas9-NLS com 0,67 μg de gRNA à temperatura ambiente (RT) por 10-15 min. Em seguida, misture suavemente o complexo RNP formado juntamente com 2 μg de ssODN com células.
- Transfira a mistura da pilha a um tubo de Electroporation de 20 μL usando pontas universais da pipeta do ajuste fornecidas pelo jogo do Electroporation do tubo. Para obter uma melhor electroporação, tente evitar a formação de bolhas de ar durante a transferência.
- Coloque o tubo de electroporação na ranhura do electroporador e prima "Go" para terminar. Siga os parâmetros sugeridos do fabricante para cada tipo de célula. Por exemplo, para iPSCs humanos e células de fibroblastos de coelho, o conjunto de tensão é 420 V e o tempo de pulso é de 30 ms. um ciclo de Electroporation bem sucedido é indicado pelo relatório do pulso na tela de exposição do electroporator.
- Após o electroporation, transfira as pilhas humanas do iPS a 1 mL do meio de cultura pre-aquecido de Y-27632-contendo descrito na divisória da cultura da pilha. Para células de fibroblastos de coelhos, transfira-as para DMEM com soro bovino fetal a 10%.
- Placa as pilhas ressuscipended a um poço de uma placa da cultura da pilha de 12 poços.
- Mude o meio de cultura todos os dias. Y-27632 retira-se do meio de cultura humano do iPSC 24 h borne-Electroporation.
4. análise de eventos de edição de genes
- Células de colheita 72 h após o electroporation. Digerir as células da placa de cultura usando Trypsin-EDTA para células de fibroblastos de coelhos ou solução de descolamento de células para iPSCs humanos. Após a centrifugação, as células de ressuscita com 350 mL de tampão de lise (1 M Tris HCl, 5 M NaCl, 0,5 M EDTA; pH 8,0, 10% SDS, adicionar 20 μL de 20 mg/mL de proteases K estoque por 1 mL de tampão de Lise), em seguida, incubar a 55 ° c durante a noite.
- Extraia o DNA genómico com fenol-clorofórmio usando procedimentos padrão.
- Amplify 100-200 BP fragmentos do ADN que contêm a região alvejada usando a polimerase de alta fidelidade do ADN, a seguir purificar os fragmentos do ADN dos géis usando um jogo da extração do gel ou diretamente dos produtos do PCR usando um mini jogo do PCR SV.
- Para determinar a eficiência de edição de genes por sequenciamento de colônias bacterianas, ligase os produtos de PCR purificados em um vetor pCR4-TOPO usando um kit de clonagem TOPO TA. Aleatoriamente pegar clones bacterianos, em seguida, sequenciar as inserções usando uma cartilha seqüenciamento universal fornecido pelo kit de clonagem TOPO TA.
- Para determinar a eficiência da edição do gene pelo seqüenciamento profundo, emita os produtos purified do PCR (~ 100-200 BP) da etapa 4,3 para o sequenciamento do amplicon de CRISPR em um núcleo de sequenciamento do ADN.
Resultados
TE de Cas9 RNP e ssODNs a células de fibroblastos de coelhos
O processo global de fornecimento mediado por TE de Cas9 RNP para células de mamíferos é ilustrado na Figura 1. Primeiramente, as mutações C231Y e Q235X foram produzidas no gene IL2RG, e a mutação R150G foi produzida no gene do SPR em pilhas do fibroblasto do coelho. As mutações da perda--função em genes de IL2RG e de SPR são sabidas para causar a imunodeficiência preliminar11 e os deficits do motor e os cognitivos12, respectivamente.
Os designs sgRNA específicos são ilustrados na Figura 2a. Os primers utilizados para amplificar as regiões visadas estão listados na tabela 3. As sequências de ssODNs são mostradas na tabela 1. As taxas de edição do gene foram determinadas por clonagem de at bacteriana (Figura 2b). No locus IL2RG SP c231, dos 28 clones que foram seqüenciados, um (3,57%) transportou a mutação C231Y precisa, quatro (14,28%) de inserção ou deleção (Indel), e os restantes 23 (82%) eram selvagens-tipo. No locus IL2RG Q235, dos 27 clones que foram seqüenciados, dois (7,41%) carregou a mutação Q235X precisa, três realizaram mutações do Indel (11,11%) e os restantes eram de tipo selvagem. No locus SPG r150, dos 20 clones seqüenciados, cinco (25%) transportou a mutação R150G precisa, 10 (50%) transportou mutações do Indel, e o restante era selvagem-tipo.
TE de Cas9 RNP e ssODNs para iPSCs humanos
O TE foi então utilizado para entregar o RNP e o ssODNs de Cas9 a iPSCs humanos e segmentar os Locos clinicamente relevantes nos genes EGFR, Mybpc3 e HBB. As mutações pontuais na região proximal do EGFR T790 conferem resistência aos inibidores da tirosina quinase do EGFR em pacientes com câncer de pulmão de células não pequenas (NSCLC) que abriam Mutações ativadoras do EGFR13. Uma mutação do Frameshift no exon 16 em Mybpc3 é implicada na cardiomiopatia hipertrófica14. A mutação de ponto E6V no gene de HBB conduz à doença de pilha de Sickle15.
Os designs sgRNA específicos são ilustrados na Figura 3a. Os primers utilizados para amplificar as regiões visadas estão listados na tabela 3. As sequências de ssODNs são mostradas na tabela 1. As taxas de edição do gene foram determinadas pelo DeepSeq (Figura 3B). No locus EGFR, 15,68% dos alelos carregavam as mutações pontuais exatas (6.315 leituras), 22,75% carregavam mutações no Indel (9.162 leituras), e os restantes 61,57% eram de tipo selvagem (24.797 leituras). No locus Mybpc3, 37,92% carregavam o apagamento exato de 4-BP TGAA (11.654 leituras), 2,24% carregavam mutações no Indel (410 leituras) e os restantes 59,84% eram Wild-Type (18.692 leituras). No locus HBB, 11,65% carregavam a mutação E6V precisa (6.565 leituras), 23,35% carregavam mutações do Indel (13.163 leituras) e os restantes 65% eram Wild-Type (36.644 leituras).

Figura 1: fluxograma do Electroporation do tubo de Cas9 RNP.

Figura 2 : Edição do gene de pilhas do fibroblasto do coelho. (A) ilustração das sequências alvo. As caixas indicam loci alvejado. As letras sublinhadas correspondem às sequências gRNA. As letras coloridas vermelhas indicam seqüências de PAM. (B) ta clonagem de resultados de eventos de edição de genes. Caixas indicam precisamente loci mutado. A seqüência de Indel mostrada é somente representativa de um tipo do alelo. Outras sequências Indel não são mostradas. Por favor clique aqui para ver uma versão maior desta figura.
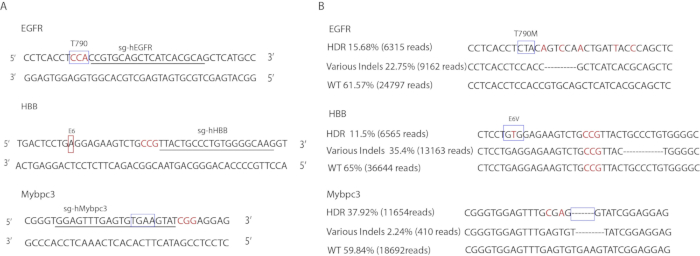
Figura 3 : Edição do gene de iPSCs humanos. (A) ilustração das sequências alvo. As caixas indicam loci alvejado. As letras sublinhadas correspondem à sequência gRNA. As letras coloridas vermelhas indicam seqüências de PAM. (B) resultados deepseq de eventos de edição de genes. Caixas indicam precisamente loci mutado. As letras coloridas vermelhas indicam as mutações silenciosas que foram introduzidas nos moldes do doador. A seqüência de Indel mostrada é somente representativa de um tipo do alelo. Outras sequências Indel não são mostradas. Por favor clique aqui para ver uma versão maior desta figura.
| Locus |
Sequência de oligo | |||||||
| (mutação direcionada) | ||||||||
| Coelho IL2RG (C231Y) | AGCGTGGATGGGCAGAAACTCTACACGTTCCGAGTCCGGAGCCGTTTTAACCCTTTGTATGGGAGTGCTCAGCATTGGAGT GAATGGAGCCACCCGATCCACTGGGGGAGCAAAACTTCAAAGGGTAAAATGGGCCT | |||||||
| Coelho IL2RG (Q235X) | AGCGTGGATGGGCAGAAACTCTACACGTTCCGAGTCCGGAGCCGTTTTAACCCTTTGTGTGGGAGTGCTTAGCATTGGAGT GAATGGAGCCACCCGATCCACTGGGGGAGCAAAACTTCAAAGGGTAAAATGGGCCT | |||||||
| Coelho SPR | gacctccatgctctgcctgacctcctgcatcctgaaggcgtttcctgccagtcctggCctcagcgggactgtggtgaacatctcgtcgctgtgtgccctgcagcccttcaagggctggg o cgctgtac | |||||||
| (R150G) | ||||||||
| EGFR humano | ACGTGATGGCCAGCGTGGACAACCCCCACGTGTGCCGCCTGCTGGGCATCTGCCTCACCTCTACAGTCCAACTGATTACCC AGCTCATGCCCTTCGGCTGCCTCCTGGACTATGTCCGGGAACACAAAGACAATATTGGCTCCCAGTAC | |||||||
| (Mutações pontuais centesimal a T790) | ||||||||
| Human Mybpc3 | GCCCCCTGTGCTCATCACGCGCCCCTTGGAGGACCAGCTGGTGATGGTGGGGCAGCGGGTGGAGTTTGCGAGGTATCGGA O GGAGGGGGCGCAAGTCAAATGGTGAGTTCCAGAAGCACGGGGCATGGGTGTTGGGGGCAT | |||||||
| (supressão de 4 BP) | ||||||||
| HBB humano | TCTGACACAACTGTGTTCACTAGCAACCTCAAACAGACACCATGGTGCATCTGACTCCTGTGGAGAAGTCTGCAGTTACTGCC O CTGTGGGGCAAGGTGAACGTGGATGAAGTTGGTGGTGAGGCCCTGGGCAG | |||||||
| E6V | ||||||||
Tabela 1: sequências de ssODNs.
| Passo | Problema | Razões possíveis | Soluções |
| 2,1 | Baixa taxa de Indel | Projeto pobre do RNA do guia, estoques do RNA do guia > 6 meses, baixa concentração do RNA do guia | Redesenho guia RNA, produzir/encomendar novo guia RNA. |
| 2,3 | Baixa eficiência de PGE | projeto de DNA de doadores pobres, baixo RNA guia eficiente, quantidade incorreta de DNA doador ou DNA de má qualidade | Aumente o comprimento do braço do homologia, introduz a mutação de Pam, introduz mutações silenciosas ao ADN do doador, usa um RNA mais eficiente do guia, otimiza a relação da proteína Cas9 sobre o RNA do guia. |
| 3,4 | Falha na transfecção | Bolhas de ar formadas durante a transferência de células-buffer de mistura para tubo de Electroporation, tensão incorreta/ajuste de duração | Tente evitar a formação de bolhas de ar, ajustar a tensão/ajuste de duração. |
| 3,6 | baixa viabilidade celular após eletroporação | Baixa sobrevida de um único IPSC humano | Adicione o inibidor da rocha após o electroporation, aumente o número de pilhas. |
| 4,1 | PCR falhado | Alto conteúdo GC, ou sequência repetitiva | Otimize a condição do PCR, adicione o sistema de DMSO ao PCR. |
Tabela 2: guias de Troubleshooting para problemas freqüentes.
| Nome da cartilha | Seqüência | Nota |
| RB-IL2RG-F | CATGACAGTGACAGGGTCCC | Para amplificar o fragmento do ADN IL2RG do coelho |
| RB-IL2RG-R | TGCCAGAGACACAAGCGAAC | |
| RB-SPR-F | O GTACTTTGGAGGGACAGAGG | Para amplificar o fragmento do ADN do SPR do coelho |
| RB-SPR-R | O CTCAGCACCCTGACACTGGG | |
| H-EGFR-F | TGATGGCCAGCGTGGACAAC | Para amplificar o fragmento de DNA EGFR humano |
| H-EGFR-R | ACCAGTTGAGCAGGTACTGGG | |
| H-Mybpc3-F | ATGCCCCGTGCTTCTGGAAC | Para amplificar fragmento de DNA Mybpc3 humano |
| H-Mybpc3-R | TCAGGGGAGCCAACCCTCAT | |
| H-HBB-F | TAACCTTGATACCAACCTGC | Para amplificar o fragmento humano do ADN de HBB |
| H-HBB-R | Método CATTTGCTTCTGACACAACT |
Tabela 3: primers usados na etapa 4,3.
Discussão
O método do Electroporation do tubo era eficaz em entregar CRISPR/Cas9 RNP e ssODNs às pilhas do coelho e do ser humano, conduzindo à edição precisa robusta do gene (PGE). A principal diferença entre o TE e outros dispositivos convencionais de eletroporação é o uso de um tubo, no qual dois eletrodos estão na parte superior e inferior do tubo e a amostra é carregada na íntegra, em seguida, selada após a eletroporação (Figura 1). Em contrapartida, em uma cubeta convencional, os eletrodos estão nas laterais e a amostra não está totalmente selada durante a eletroporação. Este projeto novo reduz a geração da bolha de ar e comprime o tamanho da bolha de ar, que melhora conseqüentemente mesmo a distribuição da tensão elétrica, e em conseqüência conduz à morte de pilha reduzida e à eficiência elevada do transfection9. No presente trabalho, taxas elevadas de PGE (15%-37%) foram alcançados visando os genes EGFR, Mybpc3 e HBB em iPSCs humanos. Estes resultados são consistentes com um relatório prévio em que as taxas elevadas de PGE foram conseguidas em pilhas de haste humanas9.
As mutações causadoras da doença foram alvejadas em genes de IL2RG e de SPR em pilhas do coelho. Recentemente, os coelhos IL2RG-Knockout foram produzidos como modelos para a imunodeficiência combinada severa X-lig humana (scid-X1)16,17. O presente trabalho mostra que as mutações do paciente IL2RG (por exemplo, C231Y e Q235X) podem ser eficientemente geradas em células de coelhos, demonstrando a viabilidade da criação de modelos de coelhos SCID-X1 portadores de mutações do paciente. Também foi demonstrado que as mutações da SPR R150G podem ser eficientemente criadas em células de coelhos. Esta mutação provoca déficits motores e cognitivos em crianças12. Esses modelos de coelhos de mutação IL2RG e SPR, uma vez gerados, podem servir como modelos pré-clínicos valiosos para estudos translacionais. Eles também podem ser usados para estabelecer terapêutica baseada na edição de genes para essas doenças monogênicas.
Uma preocupação para aplicativos de edição de genes mediados por CRISPR/Cas9 é os eventos de edição fora do alvo. As taxas de Indel foram analisadas em sítios de topo fora do alvo previstos para sgRNAs utilizados neste estudo (tabela S1), utilizando métodos previamente descritos9. No total, foram analisados sete potenciais loci fora do alvo para SG-RB-IL2RG-01, cinco para SG-RB-SPR, sete para SG-hEGFR, cinco para SG-hMybpc3 e sete para SG-hHBB), utilizando os primers listados na tabela S2. Nenhum indels fora do alvo foi revelado pelos ensaios T7E1 (Figura S1), indicando riscos mínimos fora do alvo para a edição do gene MEDIADO por crispr/Cas9 usando estes sgRNAs. Igualmente indica que o método do Electroporation do tubo próprio não causa ou aumenta edições do fora-alvo. No entanto, os esforços devem ser dedicados para reduzir ou eliminar edições indesejáveis fora do alvo. O sequenciamento do genoma inteiro pode ser necessário para excluir tais eventos para as células que se destinam a ser utilizadas em aplicações clínicas.
No nível técnico, os seguintes são considerados fatores-chave para a obtenção de uma edição eficiente do genoma com a eletroporação de tubo CRISPR/Cas9 RNP. Primeiramente, é aconselhável selecionar um sgRNA eficiente com o baixo potencial fora-alvo previsto. É importante validar a eficiência do Indel do sgRNA selecionado antes de usá-lo para aplicações de PEG. Não é raro que um software previu bom sgRNA falha na etapa de validação.
Em segundo lugar, para conseguir o PGE elevado, recomenda-se induzir uma mutação de PAM ao doador de ssODN sempre que possível. A lógica é que, ao fazê-lo, o recorte CRISPR/Cas9 após a integração do modelo de doador é evitado. Em certos casos, o próprio PGE introduz mutações do PAM. Em outros casos, é possível introduzir mutações silenciosas na sequência do PAM. No caso de uma mutação do PAM não ser possível, é aconselhável tentar incluir várias mutações silenciosas no doador que corresponde à sequência de sgRNA.
Em terceiro lugar, particularmente relevante para TE, é importante evitar a formação de bolhas de ar ao transferir as células e a mistura de RNP para o tubo de eletroporação. Quando o projeto de um tubo do TE já minimiza a formação da bolha de ar, a manipulação cuidadosa reduzirá mais e pode mesmo terminar evita a formação da bolha de ar. Um guia de tiro de problemas para problemas freqüentes que podem ser encontrados na aplicação de eletroporação de tubo para a edição de gene precisa mediada por ribonucleoproteína CRISPR/Cas9 é fornecido na tabela 2.
Em conclusão, é demonstrado aqui que o electroporation do tubo é meios eficazes para a entrega de CRISPR/Cas9 RNP e de ssODNs às pilhas de mamífero para conseguir taxas elevadas de PGE. Esta técnica nova do transfection do TE e sua taxa precisa robusta da edição do gene podem facilitar o desenvolvimento de aplicações de edição do gene.
Divulgações
J. C. trabalha na empresa Celetrix LLC, fabricante do electroporator do tubo. L. M., L. J., J. S., D. Y., J. Z., Y. E. C., e J. X. não declaram interesses concorrentes.
Agradecimentos
Este trabalho foi apoiado pelos institutos nacionais de saúde (R21OD023194 para JX). Este trabalho utilizou os principais serviços apoiados pelo centro de modelos avançados de Ciências translacionais e terapêuticas (CAMTraST) no centro médico da Universidade de Michigan.
Materiais
| Name | Company | Catalog Number | Comments |
| Accutase | STEMCELL Technologies | 792 | Cell detachment solution for human iPSCs, first used in Step 1.1.2. |
| Cas9 Nuclease 3NLS | IDT | 1074182 | Cas9 protein, first used in Step 3.3. |
| DMEM | Thermo Fisher | 11965092 | For cell culture, first used in Step 1.2.3. |
| DPBS | Thermo Fisher | 1708075 | For preparing cell culture, first used in Step 1.2.2. |
| EDTA | Lonza | 51201 | For making lysis buffer, first used in Step 4.1. |
| Electroporation buffer | Celetrix | 13–0104 | The electroporation buffer, first used in Step 3.2. |
| Electroporation tubes | Celetrix | 20 μL: 12–0107; 120 μL: 12–0104 | The electroporation tube, first used in Step 3.4. |
| Electroporator | Celetrix | CTX-1500A LE | The tube electroporation machine, first used in Step 3.5 |
| Fetal bovine serum | Sigma Aldrich | 12003C | For cell culture, first used in Step 1.2.2. |
| Forma CO2 Incubators | Thermo Fisher | Model 370 | For cell culture, first used in Step 1.1. |
| Gel Extraction Kit | Qiagen | 28115 | For gel purification, first used in Step 4.3. |
| Human induced pluripotent stem cells | American Type Culture Collection | ACS-1030 | Human iPSCs, first used in Step 1.1. |
| Matrigel | Corning | 354277 | Artificial extracellular matrix; for precoating cell culture plate, first used in Step 1.1. |
| mTeSR 1 medium | STEMCELL Technologies | 85850 | Feeder-free cell culture medium for human iPSCs, first used in Step 1.1. |
| PCR SV mini | GeneAll | 103-102 | For PCR product purification, first used in Step 4.3. |
| Penicillin-Streptomycin | Thermo Fisher | 15140163 | For preparing cell culture, first used in Step 1.2.2. |
| Phenol-chloroform | Thermo Fisher | 15593031 | For DNA extraction, first used in Step 4.2. |
| Precision gRNA Synthesis Kit | Invitrogen | A29377 | For the generation of full length gRNA (guide RNA), first used in Step 2.4. |
| Proteinase K Solution | Thermo Fisher | AM2548 | For DNA extraction, first used in Step 4.1. |
| Q5 high-fidelity DNA polymerase | NEB | M0491 | For PCR amplification, first used in Step 4.3. |
| Sodium dodecyl sulfate | Sigma Aldrich | L3771 | For making lysis buffer, first used in Step 4.1. |
| TA Cloning Kit | Thermo Fisher | K457502 | For TA clone sequencing, first used in Step 4.4. |
| Tissue Culture Dish (10 cm) | FALCON | 353003 | For cell culture, first used in Step 1.2.3. |
| Tissue Culture Dish (12 well) | FALCON | 353043 | For cell culture, first used in Step 3.7. |
| Tissue Culture Dish (6 cm) | FALCON | 353004 | For cell culture, first used in Step 1.2.2. |
| Tris HCl | Thermo Fisher | BP1757-500 | For making lysis buffer, first used in Step 4.1. |
| Trypsin-EDTA | Thermo Fisher | 25200056 | For cell digestion, first used in Step 1.2. 4. |
| Universal Fit Pipette Tips | Celetrix | 14-0101 | For electroporation, first used in Step 3.4. |
| Y27632 | LC Labs | Y-5301 | The apoptosis inhibotor, first used in Step 1.1.1. |
Referências
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Mout, R., et al. Direct Cytosolic Delivery of CRISPR/Cas9-Ribonucleoprotein for Efficient Gene Editing. ACS Nano. 11 (3), 2452-2458 (2017).
- Zuris, J. A., et al. Cationic lipid-mediated delivery of proteins enables efficient protein-based genome editing in vitro and in vivo. Nature Biotechnology. 33 (1), 73-80 (2015).
- Miller, J. B., et al. Non-Viral CRISPR/Cas Gene Editing In Vitro and In Vivo Enabled by Synthetic Nanoparticle Co-Delivery of Cas9 mRNA and sgRNA. Angew Chem Int Ed Engl. 56 (4), 1059-1063 (2017).
- Finn, J. D., et al. A Single Administration of CRISPR/Cas9 Lipid Nanoparticles Achieves Robust and Persistent In Vivo Genome Editing. Cell Reports. 22 (9), 2227-2235 (2018).
- Liang, C., et al. Tumor cell-targeted delivery of CRISPR/Cas9 by aptamer-functionalized lipopolymer for therapeutic genome editing of VEGFA in osteosarcoma. Biomaterials. 147, 68-85 (2017).
- Luo, Y. L., et al. Macrophage-Specific in Vivo Gene Editing Using Cationic Lipid-Assisted Polymeric Nanoparticles. ACS Nano. 12 (2), 994-1005 (2018).
- Wang, H. X., et al. Nonviral gene editing via CRISPR/Cas9 delivery by membrane-disruptive and endosomolytic helical polypeptide. Proceedings of the National Academy of Sciences of the United States of America. 115 (19), 4903-4908 (2018).
- Xu, X., et al. Efficient homology-directed gene editing by CRISPR/Cas9 in human stem and primary cells using tube electroporation. Scientific Reports. 8 (1), 11649 (2018).
- Du, F., et al. Beneficial effect of young oocytes for rabbit somatic cell nuclear transfer. Cloning Stem Cells. 11 (1), 131-140 (2009).
- Allenspach, E., Rawlings, D. J., Scharenberg, A. M., Adam, M. P., et al. . GeneReviews(R). , (1993).
- Friedman, J., et al., Adam, M. P., et al. . GeneReviews(R). , (1993).
- Hidaka, N., et al. Most T790M mutations are present on the same EGFR allele as activating mutations in patients with non-small cell lung cancer. Lung Cancer. 108, 75-82 (2017).
- Ma, H., et al. Correction of a pathogenic gene mutation in human embryos. Nature. 548 (7668), 413-419 (2017).
- Vakulskas, C. A., et al. A high-fidelity Cas9 mutant delivered as a ribonucleoprotein complex enables efficient gene editing in human hematopoietic stem and progenitor cells. Nature Medicine. 24 (8), 1216-1224 (2018).
- Song, J., et al. Bacterial and Pneumocystis Infections in the Lungs of Gene-Knockout Rabbits with Severe Combined Immunodeficiency. Frontiers in Immunology. 9, 429 (2018).
- Song, J., et al. Production of immunodeficient rabbits by multiplex embryo transfer and multiplex gene targeting. Scientific Reports. 7 (1), 12202 (2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados