Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Phasenwechsel-Dimethyldioctadecylammonium-geschälte Mikrotröpfchen als vielversprechendes Wirkstoffabgabesystem: Ergebnisse an 3D-Sphäroiden von Säugetiertumorzellen
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Decafluorpenttan-Mikrotröpfchen, die mit einer Hülle aus Dimethyldioctadecylammoniumbromid entwickelt wurden, wiesen eine außergewöhnliche kolloidale Stabilität und eine aktraktive Bioschnittstelle auf. DDAB-MDs erwiesen sich als effiziente Wirkstoffreservoire, die sich durch eine hohe Affinität zu Plasmamembranen sowie eine verbesserte Aufnahme und Antitumoraktivität von Doxorubicin gegen das humane dreifach negative Brustkrebsmodell (MDA-MB-231) auszeichnen.
Zusammenfassung
Eine signifikante Verbesserung der Phasenwechsel-Perfluorkohlenstoff-Mikrotröpfchen (MDs) im umfangreichen theranostischen Szenario erfolgt durch die Optimierung der MDs-Zusammensetzung in Bezug auf Syntheseeffizienz, Stabilität und Wirkstoffabgabefähigkeit. Zu diesem Zweck wurden Decafluorpentan (DFP)-MDs entwickelt, die durch eine Hülle aus kationischem Tensid Dimethyldioctadecylammoniumbromid (DDAB) stabilisiert sind. Eine hohe Konzentration von DDAB-MDs konnte innerhalb weniger Sekunden durch gepulste Hochleistungsinsonation erreicht werden, was zu niedrig polydispersen Tröpfchen mit einer Größe von 1 μm führte. Ein sehr positives ζ-Potential, zusammen mit langen, gesättigten Kohlenwasserstoffketten der DDAB-Schale, sind Schlüsselfaktoren zur Stabilisierung des Tröpfchens und der darin befindlichen Wirkstofffracht. Die hohe Affinität der DDAB-Hülle zur Zellplasmamembran ermöglicht eine lokalisierte Verabreichung von Chemotherapeutika, indem sie die Wirkstoffkonzentration an der Tumorzellschnittstelle erhöht und die Aufnahme erhöht. Dies würde DDAB-MDs zu einem relevanten Instrument zur Verabreichung von Medikamenten machen, das eine hohe Antitumoraktivität bei sehr niedrigen Medikamentendosen aufweist.
In dieser Arbeit wurde gezeigt, dass die Wirksamkeit eines solchen Ansatzes die Wirkung von Doxorubicin gegen 3D-Sphäroide von Säugetiertumorzellen, MDA-MB-231, dramatisch verbessert. Die Verwendung von dreidimensionalen (3D) Zellkulturen, die in Form von mehrzelligen Tumorsphäroiden (d. h. dicht gepackten Zellen in kugelförmiger Form) entwickelt wurden, hat zahlreiche Vorteile im Vergleich zu 2D-Zellkulturen: Sie hat nicht nur das Potenzial, die Lücke zwischen herkömmlichen In-vitro-Studien und Tierversuchen zu schließen, sondern verbessert auch die Fähigkeit, in vitro prädiktivere Ergebnisse zu erbringen Screening-Assays für die präklinische Wirkstoffentwicklung oder Bewertung des Potenzials von Off-Label-Medikamenten und neuen Co-Targeting-Strategien.
Einleitung
Wirkstoffverabreichungsvektoren, die in der Lage sind, eine hohe Wirksamkeit gegen Tumore zu gewährleisten und Nebenwirkungen zu reduzieren, sind primäre Ziele, bleiben aber eine schwerwiegende chemisch-pharmazeutische Herausforderung 1,2. Bisher sind ihre Fortschritte zunächst durch den Kontrast zwischen einer unzureichenden In-situ-Wirkstofffreisetzung und einem kritischen Maß an unspezifischer Toxizität begrenzt 3,4,5. In den letzten Jahren wurden mehrere Arzneimittelverabreichungssysteme implementiert, um die Verabreichung von Krebsmitteln zu verbessern, darunter Liposomen, polymere Mizellenund Polymersomen 6,7,8,9,10. Diese Systeme haben das Potenzial, die Zirkulationszeit und die Selektivität von Arzneimitteln zu erhöhen und gleichzeitig die Verteilung und Akkumulation in gesunden Organen und Geweben zu reduzieren. Wie auch immer, die verkapselten Formulierungen von antineoplastischen Chemotherapeutika, wie z. B. Anthrazyklinen, führten zu einer signifikant reduzierten Internalisierungseffizienz des Arzneimittels. In jüngster Zeit haben stimuli-responsive Mikron- und Submikron-Träger wie Mikrobläschen11, Mikrotröpfchen, hybride Gold-Nanopartikel12, Nano-Hydrogele13, PLGA-Gerüste und mesoporöse Plattformen14 aufgrund ihrer hohen Vielseitigkeit bei der Ausrichtung und Ausübung tumorhemmender Wirkungen unter Verwendung von Doxorubicin (Dox) und Docetaxel an pharmakologischem Interesse gewonnen. Bahnbrechende Experimente, um diese Träger in effiziente Anti-Krebs-Soldaten für multimodale Aufgaben (d. h. chemotherapeutische, photothermische und gensynergistische Ansätze) und molekulare Bildgebung zu verwandeln15 haben den Weg für eine personalisierte theranostische Nanomedizin geebnet.
In diesem Szenario wurden Phasenwechsel-Perfluorkohlenstoff-Mikrotröpfchen (MDs) im Hinblick auf die Schlüsselmöglichkeit bewertet, die sie bieten, um eine hohe Wirkstofffrachtbelastung zu konjugieren, die chemische Vielseitigkeit der MDs-Hülle in Bezug auf biologische Barrieren, kolloidale Stabilität und Syntheseeffizienz11,12. Ein weiterer Vorteil ist, dass die Echogenität der MDs, die durch akustische oder optische Verdampfung des Perfluorkohlenstoff (PFC)-Kerns gefördert wird, eine In-situ-Bildgebung und eine vielversprechende therapeutische Wirksamkeit ermöglicht. Darüber hinaus kann die Kernverdampfung von MDs, die durch die Energiefreisetzung ionisierender Teilchenstrahlen erzielt wird, für die Strahlverfolgung und Strahlendosimetrie genutzt werden.
Die vorliegende Studie zielt darauf ab, Decafluorpentan (DFP)-Mikrotröpfchen zu entwickeln, die durch eine mehrfach verwendbare Hülle aus kationischem Tensid Dimethyldioctadecylammoniumbromid (DDAB) stabilisiert werden. DDAB-Schalen-MDs erfüllen sowohl physikalisch-chemische als auch biologische Erwartungen. DFP-basierte Mikrotröpfchen haben sich als wertvolle Phasenwechselkontrastmittel erwiesen, um biokompatible und stabile Perfluorkohlenwasserstoff-MDszu erzielen 16. Das kristalline Gel DDAB sättigt lange Ketten bei physiologischer Temperatur, dringt tief in den hydrophoben Kern ein und stabilisiert das Tröpfchen und die darin enthaltene Wirkstofffracht. Darüber hinaus erhöht das hohe positive ζ-Potential an der Wassergrenzfläche die kolloidale Stabilität der MDs. Die biologische Attraktivität der DDAB-Schalenoberfläche liegt in der Fähigkeit, den Tod von Bakterien und Pilzen in Konzentrationen zu verursachen, die Säugetierzellen kaum beeinflussen, und Plasmamembranen, negativ geladene antigene Proteine, Nukleotide, DNA oder Nanopartikel zu binden. Die oben genannten Eigenschaften können ausgenutzt werden, um eine bemerkenswerte Immunadjuvans-, Gentherapie- und Antitumorwirkung in Säugetierzellen zu erzeugen17.
Dox-beladene DDAB-MDs (Dox@DDAB-MDs), die hierin beschrieben werden, fördern die Wirkstofffreisetzung gegen hochaggressive, invasive und schlecht differenzierte dreifach negative Brustkrebszellen. Im Folgenden wird ein einfaches und schnelles Protokoll beschrieben, das auf der Insonation von Hochleistungssonden basiert, um stabile DDAB-MDs mit hoher Dichte und einer engen Größenverteilung und einer hohen Belastungseffizienz von Dox in einer einstufigen Formulierung zu erhalten. Diese Eigenschaften sind sogar für andere Präparationsverfahren wie mikrofluidische Vorrichtungen und High-Shear-Homogenisatoren16 wettbewerbsfähig.
Das andere große einschränkende Problem bei der Entwicklung effizienter Wirkstoffverabreichungsvektoren besteht darin, dass die Aktivität eines Arzneimittels eine Funktion verschiedener Parameter (z. B. Absorption, Verteilung, Konzentrationen) ist, die in einem tatsächlichen biologischen Ziel erzielt werden können, was von Monolayer-Zellmodellen nicht berücksichtigt werden kann18. Aus diesem Grund ist die Geschichte der Entwicklung neuartiger Antitumorformulierungen gespickt mit In-vitro-Studien , die sich leider bereits auf dem Niveau präklinischer Modelle an Tieren als unwirksam erwiesenhaben 19.
Insbesondere die Notwendigkeit, von Zellkulturen zu einem komplexeren und zuverlässigeren System als In-vivo- und Ex-vivo-Studien überzugehen, hängt mit den inhärenten Einschränkungen pharmakologischer Studien an 2D-Kulturen zusammen. In diesem Zusammenhang sind die in vitro 3D-Systeme eingeschlossen, wie z. B. Sphäroide, Organoide, Organ-on-Chip, die die Morphologie, Aktivität und physiologische Reaktion komplexerer Strukturen als die 2D-Monoschichtensimulieren 20. In präklinischer Sicht bieten 3D-Zellmodelle, die die zelluläre Mikroumgebung nachahmen, die Möglichkeit, komplexe Biologie in einem physiologisch relevanteren Rahmen besser zu verstehen, in dem traditionelle Monolayer-Kulturen nicht wirksam sind21,22.
Nach dem Nachweis, dass DDAB-MDs mit der Zellmembran menschlicher Brustkrebszellen interagieren können, was die Internalisierung von Medikamenten und den Zelltod bei sehr niedrigen (nanomolaren) Dox-Konzentrationen begünstigt, wurde die Wirksamkeit einer solchen Methode gegen 3D-Sphäroide von Säugetiertumorzellen, MDA-MB-231, getestet.
Protokoll
HINWEIS: Alle Reagenzien und Instrumente sind in der Materialtabelle aufgeführt.
1. Herstellung und Charakterisierung von Mikrotröpfchen
- Vorbereiten von Dox-geladenen DDAB-MDs
- Das DDAB-Pulver wird in Ethanol gelöst, um eine Endkonzentration von 10 mM und ein Endvolumen von 1 mL zu erhalten. Bereiten Sie 1 ml Dox-Stammlösung vor, indem Sie 2 mg Dox-Pulver in Ethanol auflösen.
VORSICHT: Es ist bekannt, dass Dox eine akute orale Toxizität der Kategorie 4 und eine Karzinogenität der Kategorie 1B aufweist. Nur unter einem Abzug mit Handschuhen und einer Gesundheitsmaske verwenden. - 250 μl DFP zu 300 μl DDAB-Lösung (Ölphase) in ein 15 ml-Zentrifugenröhrchen aus Kunststoff geben.
HINWEIS: Die Reinheit von DFP beträgt 60 % (GC), die Dichte 1,6 g/ml (20 °C), der Siedepunkt liegt bei 55 °C. Die Dox-Reinheit beträgt 98,0-102,0% (HPLC). Die DDAB-Reinheit beträgt ≥98% (TLC). Die Reinheit des Ethanols beträgt 97%. - Geben Sie vorsichtig 2,15 mL entionisiertes Wasser auf die Ölphase, was zu einem zweiphasigen Wasser-Öl-Gemisch führt. Unmittelbar vor der Insonation werden 10 μl Dox-Lösung vorsichtig direkt in die Ölphase injiziert, um eine Abspaltung einer signifikanten Menge Dox in die Wasserphase zu vermeiden.
- Emulgieren Sie das biphasische Gemisch durch Sondeninsonation (mit einer 1/8 Zoll konischen Mikrospitze aus Titan) unter Verwendung eines hochintensiven Ultraschall-Flüssigkeitsprozessors in einem Impulsmodus (0,7 s Ein und 0,3 s Aus) bei 20 kHz, 100 W für 10 s. Verdünnen Sie frisch zubereitete MDs sofort mit ultrafiltriertem, deionisiertem Wasser (z. B. MilliQ) um den Faktor 1,8.
HINWEIS: Der Verdünnungsfaktor ist indikativ, empirisch gewählt. Die MD-Lösung kann stärker verdünnt werden, solange aus den nachfolgenden Zentrifugationen genügend Pellet gewonnen wird. - Nehmen Sie 1 ml der erhaltenen Suspension und zentrifugieren Sie 3x und resuspendieren Sie sie in ultrafiltriertem, deionisiertem Wasser (z. B. MilliQ), um den Überschuss an Dox und Ethanol zu entfernen. Die erste Zentrifugation bei 25 °C, 360 x g für 3 min und die zweite und dritte bei 280 x g bei der gleichen Temperatur und Zeit durchführen.
- Nach dem letzten Waschen entfernen Sie das überstehende Wasser und ziehen 5 μl der Pellets auf und dispergieren sie in 2 mL des Mediums für die Zellbehandlung, um eine äquivalente Dox-Konzentration von 10 nM zu erhalten.Die Protokollschritte für die Dox@DDAB-MDs-Synthese sind in Abbildung 1 dargestellt.
HINWEIS: Die äquivalente Dox-Konzentration ist definiert als die Menge an freiem Dox, die im gleichen Volumen einer Suspension von Dox@DDAB-MD enthalten ist.
- Das DDAB-Pulver wird in Ethanol gelöst, um eine Endkonzentration von 10 mM und ein Endvolumen von 1 mL zu erhalten. Bereiten Sie 1 ml Dox-Stammlösung vor, indem Sie 2 mg Dox-Pulver in Ethanol auflösen.
- Charakterisierung von Dox-geladenen DDAB-MDs
- Messen Sie die Größenverteilung mit einem Photometer mit dynamischer Lichtstreuung (DLS) und analysieren Sie die erhaltenen Korrelogramme mit dem CONTIN-Algorithmus, um die zugehörigen Abklingzeiten zu extrapolieren. Dann verwenden Sie die Abklingzeiten, um die Verteilung der Diffusionskoeffizienten (D) der Partikel zu bestimmen und diese in eine Verteilung der hydrodynamischen Durchmesser (2RH) umzurechnen, indem Sie die Stokes-Einstein-Beziehung RH = kBT/6πηD verwenden, wobei kBT die thermische Energie des Systems und η die Lösungsmittelviskosität ist.
- Überprüfen Sie die Größe und Größenverteilung auch mit Hilfe der Hellfeldmikroskopie mit einer Bildanalysesoftware (z. B. Image J), messen Sie die Größe von mindestens 100 Tröpfchen pro Bild (für 3 oder 4 Bilder), erhalten Sie einen Durchschnittswert und eine Standardabweichung.
HINWEIS: Für DLS-Messungen verdünnen Sie die MDs-Lösung in ultrafiltriertem, deionisiertem Wasser (z. B. MilliQ), PBS und in Zellkulturmedium, um eine Rückstreuung auf eine feste Konzentration von 1,5 x 108 MDs/ml zu vermeiden. Für ultrafiltriertes, deionisiertes Wasser und PBS in Zellkulturmedium erhielten wir mittlere Größenwerte von 1,1 ± 0,1 μm, 1,1 ± 0,25 μm bzw. 1,2 ± 0,2 μm. MDs, die nach der Zentrifugation aus den Pellets gewonnen werden, zeigen keine Größenänderungen innerhalb der Fehler. - Verwenden Sie einen Objektträger der Zellzählkammer für die Mikroskopie, um die Konzentration von MDs zu beurteilen. Die Kammer setzt sich aus zwei verschiedenen Zählbereichen mit einer Dicke von 0,10 mm zusammen. 10 μl MDs-Suspension über die Kammer geben. Zählen Sie MDs innerhalb des 0,25 x 0,25 nm2 Quadrats der Kammer mit einem optischen Mikroskop mit einem 40x Langstreckenobjektiv und analysieren Sie mit einer Bildanalyse-Freeware.
- Berechnen Sie die MD-Konzentration, ausgedrückt als Anzahl der MDs/ml, gemäß der Gleichung:
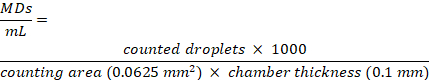
- Überprüfen Sie die internalisierten Dox-Bilder mit dem konfokalen Laser-Scanning-Mikroskop (CLSM) unter Ausnutzung der Dox-Autofluoreszenz bei 590 nm. Messen Sie den Dox-Gehalt mit einem fluorimetrischen Assay, dispergieren Sie die Partikel in ultrafiltriertem, deionisiertem Wasser oder PBS, zentrifugieren Sie sie 3 Minuten lang bei 25 °C, 360 x g und bestimmen Sie dann die Menge des freien Arzneimittels, indem Sie die Fluoreszenz des Überstands bei 590 nm mit einer Kalibrierkurve auswerten (Linearitätsbereich für Dox-Konzentration: 2-20 μmol/ml, R2 = 0,99).
HINWEIS: Subtrahieren Sie den erhaltenen Wert von der Gesamtkonzentration von Dox, um die eingekapselte Menge an Dox zu erhalten. Führen Sie das Experiment in dreifacher Ausführung durch. Die Dox-Verkapselungseffizienz ergibt 28 % ± 2 %, berechnet durch Division der Dox-Wirkstoffmenge in den MDs durch den Gesamtgehalt an eingesetztem Dox. - Es werden Experimente zur Dox-Freisetzung im Zeitverlauf durchgeführt, indem das Verfahren gemäß Schritt 1.2.3 zentrifugiert und wiederholt wird, um den Prozentsatz der im Überstand freigesetzten Dox im Verhältnis zur Anzahl der verkapselten Überstände zu erhalten. Wiederholen Sie nach der ersten Bestimmung den Vorgang, indem Sie die Zentrifugationsgeschwindigkeit auf 280 x g verringern, um ein Brechen der MDs zu vermeiden. Zeichnen Sie eine Release-Kurve im Zeitverlauf. Es ist zu überprüfen, ob die innerhalb von 24 h freigesetzte Dox-Konzentration im Überstand einen Höchstwert von 20 % erreicht.
- Messen Sie das ζ-Potential bei 37 °C mit einem speziellen Gerät (z. B. NanoZetaSizer) und stellen Sie sicher, dass die erhaltenen Werte bei etwa 90-100 mV liegen.
2. Herstellung von Sphäroiden in mikrogeformten, nicht klebenden Substraten
- Gießen von Mikroformen
- Geben Sie kleine 3D-Formen und 1 g reines Agarosepulver in Behälter, die für die Sterilisation geeignet sind. Autoklavieren Sie sie 30 Minuten lang im Trockenzyklus (121 °C).
- Geben Sie in einer Biosicherheitswerkbank 50 ml 0,9%ige Kochsalzlösung (NaCl) in die Glasflasche mit sterilisierter Agarose und stellen Sie sie in eine Mikrowelle, um das Pulver zu kochen und aufzulösen. Lassen Sie die geschmolzene Agarose auf 60 °C abkühlen und fügen Sie 500 μl zu jeder Mikroform hinzu, um die Bildung von Blasen beim Pipettieren zu vermeiden.
WARNUNG: Geschmolzene Agarose kann schwere Hautverbrennungen verursachen. Eine entsprechende persönliche Schutzausrüstung ist erforderlich. - Entfernen Sie die gelierte Agarose, indem Sie die Form vorsichtig biegen und das neu gebildete Substrat entfernen. Legen Sie die Substrate in eine 12-Well-Platte und fügen Sie 2 ml frisches Nährmedium pro Well hinzu. Inkubieren Sie mindestens 15 Minuten lang, um jedes Substrat auszugleichen.
HINWEIS: Bereiten Sie das Nährmedium im Voraus vor und passieren Sie es durch einen 0,22-μm-Filter, um mögliche Partikel oder Verunreinigungen zu entfernen.
- Aussaat der Zellen
- Kultivieren Sie MDA-MB 231-Zellen mit vollständigem Medium (DMEM ergänzt mit 1 % Pen/Strep, 10 % FBS und 1 % L-Glu) in einem Zellkultur-Inkubator bei 37 °C, 5 % CO2. Wenn die Zellen in einem T75-Kolben eine Konfluenz von etwa 80 % erreicht haben, verwerfen Sie das Medium, spülen Sie es mit 4 ml DPBS und fügen Sie 3 ml Trypsin/EDTA hinzu. Stellen Sie den Kolben in den Inkubator und warten Sie, bis sich die Zellen abgelöst haben. Untersuchen Sie die Ablösung unter einem inversen Mikroskop (bei 20x) alle 5 Minuten.
- Sammeln Sie die abgelösten Zellen mit 3 mL DPBS mit 10% FBS in einem 15 mL Röhrchen und zentrifugieren Sie sie bei 235 x g für 10 min. Der Überstand, der sowohl DPBS als auch Trypsin/EDTA enthält, wird verworfen und das Pellet der Zellen in 3 ml frischem Medium resuspendiert. Pipettieren Sie 10 μl Zellsuspension mit 10 μl Trypanblau und zählen Sie die Zellen mit einer Neubauer-Kammer.
HINWEIS: Bei der Entnahme der Zellen ist das Trypsin/EDTA mit DPBS zu neutralisieren, das 10 % FBS enthält, um zu verhindern, dass das Trypsin die Zellen während des Zentrifugationsschritts schädigt. - Um einen nominalen Sphäroiddurchmesser von 50 μm (~15 Zellen/Sphäroid) zu erhalten, verdünnen Sie die Zellsuspension auf eine Endkonzentration von 3.840 Zellen/190 μL, was zu 256 Sphäroiden in der kleinen Form führt. Alternativ können Sie bei einem Sphäroiddurchmesser von 200 μm (~1.000 Zellen/Sphäroid) auf eine Endkonzentration von 81.000 Zellen/190 μl verdünnen, was gemäß den Anweisungen des Herstellers zu 81 Sphäroiden in der großen Form führt.
- Entfernen Sie das Nährmedium von der 12-Well-Platte und kippen Sie die Substrate, um das Medium vorsichtig aus der Zellaussaatkammer zu entfernen. Geben Sie 190 μl Zellsuspension zu jedem Substrat (tropfenweise) und warten Sie, bis sich die Zellen 10 Minuten lang im Gewebekultur-Inkubator abgesetzt haben.
- Fügen Sie 2 ml des umgebenden Mediums pro Vertiefung hinzu und stellen Sie den Multiwell wieder in den Inkubator. Alle 24 Stunden auf Sphäroidbildung prüfen. Bei Bedarf durch ein frisches Medium ersetzen.
- Gewinnung und Verarbeitung von Sphäroiden
- Stellen Sie eine 35-mm-Petrischale mit 2 ml frischem Medium in den Inkubator, um sie 10-15 Minuten lang auszugleichen.
- Entfernen Sie das Zellkulturmedium, das das Substrat umgibt. Entfernen Sie mit einer sterilen Pinzette das Substrat aus der Vertiefung und drehen Sie es in der Petrischale um. Klopfen Sie vorsichtig auf die Unterseite des Substrats, damit die Sphäroide durch die Schwerkraft fallen.
- Stellen Sie die Petrischale mit den Sphäroiden zur weiteren Verarbeitung zurück in den Inkubator. Die Protokollschritte für die Herstellung von Sphäroiden sind in Abbildung 2 dargestellt.
3. Sphäroid-Behandlung
- Entfernen Sie den Überstand aus der Form und ersetzen Sie ihn durch 200 μl Dox@DDAB-MDs-Dispersion, die wie in Schritt 1.1.6 beschrieben hergestellt wurde.
- Nach 5 Minuten fügen Sie 2 ml/Vertiefung des umgebenden Mediums hinzu, um es auszugleichen. Nach der gewünschten Behandlungszeit gehen Sie wie in Schritt 2.1 beschrieben vor.
4. Charakterisierung der Größe und Morphologie der Sphäroide
- Überprüfen Sie die Sphäroidgröße nach der gewünschten Inkubationszeit mit einem 40-fachen Objektiv in Kombination mit der Bildverarbeitungssoftware des Mikroskops.
- Messen Sie die Größe von mindestens 10 Sphäroiden, um genügend Daten für geeignete Statistiken zu sammeln, und analysieren Sie deren Volumina, wie in Schritt 7 beschrieben.
5. Proliferations-/Viabilitätsassay: Fluoreszenzmikroskopie mit Lebendzellfärbung
HINWEIS: Befolgen Sie die Anweisungen für die Herstellung von Sphäroiden bis Schritt 2.2.5.
- Den Überstand aus der Form nehmen und durch 200 μL 4 μM Calcein-AM in PBS ersetzen und 3 h bei Raumtemperatur im Dunkeln inkubieren. Während der letzten 20 Minuten der Inkubation fügen Sie 10 μg/ml Propidiumiodid hinzu, um die abgestorbenen Zellen zu färben.
- Ernten Sie die Sphäroide, indem Sie das Substrat in die Petrischale umdrehen, wie in Schritt 2.3.2 beschrieben.
- Bilden Sie die Sphäroide in Färbelösung mit CLSM mit 40x/60x-Objektiven und einem Ar+-Laser ab, wobei Sie die Verstärkung und die Lochblende auf geeignete Weise einstellen, um scharfe Bilder zu erhalten.
6. Bildanalyse und -erfassung
- Transmissions- und konfokale 2D-Bilder
- Öffnen Sie die Konfokalsoftware (Ergänzungsdatei A) und wählen Sie das Objektiv aus (60x, 40x usw.). Klicken Sie im rechten Bereich auf Trans , um den Übertragungskanal auszuwählen.
- Klicken Sie auf Live , um das Bild zu visualisieren und nach dem optimierten Fokus zu suchen. Klicken Sie auf Einzeln , um die Erfassung zu stoppen.
- Für die konfokalen Bilder klicken Sie je nach verwendetem Fluoreszenzfarbstoff auf rote oder grüne Laserkanäle. Wählen Sie in der Lochblende im Dropdown-Menü die Option S-Blende aus und stellen Sie die Verstärkungssektion auf 6,00 B ein.
- Klicken Sie auf Live und stellen Sie die Lochblende und die Verstärkung ein, um den Kontrast zu optimieren. Klicken Sie auf Einzeln , um die Aufnahme zu stoppen und das Bild zu speichern. Überlappen Sie das Transmissions- und das konfokale Bild, indem Sie in der oberen Symbolleiste den richtigen Kanal auswählen.
- Konfokale 3D-Bilder
- Klicken Sie auf Z im rechten Bereich (Ergänzungsdatei B). Klicken Sie auf die rote Schaltfläche, um die Einstellungen zurückzusetzen.
- Wählen Sie den Abschnitt Ref , klicken Sie auf Live und durchsuchen Sie den Medianplan innerhalb des Objekts, das den Fokus verschiebt. Wählen Sie den oberen Bereich aus und verschieben Sie den Fokus nach oben, bis das Objekt unscharf ist und verschwindet.
- Wiederholen Sie Schritt 6.2.2 für den unteren Bereich, indem Sie den Fokus nach unten verschieben.
- Stellen Sie die Schrittweite auf 0,75 μm ein. Klicken Sie auf Z-Stack und dann auf Single , um den Scanvorgang zu starten. Speichern Sie die Bilder in .ics Format.
- Optische Bilder und Analyse
- Öffnen Sie die Software für das optische Mikroskop und nehmen Sie das Bild mit einem 40-fachen Langzeitfokus oder einem 20-fachen Objektiv auf, indem Sie in der oberen Symbolleiste auf Play drücken. Drücken Sie Stopp und speichern Sie das Bild.
- Klicken Sie in der oberen Symbolleiste auf Ansicht, Analysesteuerung, Anmerkungen und Messungen , um ein Fenster mit verschiedenen Optionen zu öffnen (Zusatzdatei C). Wählen Sie im Bereich Anmerkungen und Messungen die Option Halbachse aus und klicken Sie auf Ellipse.
- Suchen Sie in der Bildersuche nach der Achse, die besser zur Objektform passt, und drücken Sie die Eingabetaste auf der Tastatur, um den erhaltenen Wert auf die rechte Bedienfeldtabelle zu übertragen.
- Wiederholen Sie Schritt 6.3.3 für mindestens 10 Objekte, um genügend Daten zu erhalten. Klicken Sie auf In Excel-Datenblatt exportieren , um die Daten zu exportieren und zu speichern.
- 3D-Z-Stack-Bildvisualisierung und numerische Volumenberechnung
- Öffnen Sie das mit der konfokalen Mikroskopie aufgenommene 3D-Bild in der optischen Mikroskopsoftware (Zusatzdatei D).
- Um die 3D-Rekonstruktion zu sehen, klicken Sie in der Bildsymbolleiste auf Volumenansicht anzeigen . Das Objekt kann durch Klicken mit der linken Maustaste in jede Richtung gedreht werden. Um einen Teil der Lautstärke auszuwählen, halten Sie die Tasten Strg + linke Maustaste gedrückt (Zusatzdatei E).
- Drücken Sie die x-Taste auf der Tastatur, um einen Schnappschuss des 3D-Bildes zu machen und zu speichern.
- Um das Objektvolumen zu berechnen, klicken Sie auf Messen, 3D-Objektmessungen, um ein Panel zu öffnen (Zusatzdatei, Bild F). Klicken Sie in der Symbolleiste des Bedienfelds auf 3D-Schwellenwert definieren und stellen Sie den optimierten Schwellenwert ein, indem Sie auch die Filter für glatte und saubere Elemente (Ergänzende Datei, Bild G) verwenden. Klicken Sie auf OK , um im Bereich 3D-Objektmessungen verschiedene Parameter, einschließlich des Volumens, zu erhalten.
- Klicken Sie auf Nach Excel exportieren , um Daten zu exportieren und zu speichern.
7. Analyse von Sphäroid-Daten
- Für die Berechnung des Sphäroidvolumens nähern Sie sich der 3D-Struktur mit einem prolaten Ellipsoid an und schätzen Sie die Haupt- und Nebenachse durch die 2D-Projektion, wie in der Einfügung in Abbildung 3 gezeigt.
- Validieren Sie die Prolate-Ellipsoid-Approximation durch einen Vergleich zwischen dem numerisch berechneten Volumen, wie in Schritt 6.4 gezeigt, und der Formel
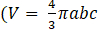 für das Prolate-Ellipsoid-Volumen mit b>a=c, wobei a, b und c die Ellipsoidachse sind).
für das Prolate-Ellipsoid-Volumen mit b>a=c, wobei a, b und c die Ellipsoidachse sind). - Berechnen Sie das mittlere Volumen von mindestens 10 Sphäroiden und die entsprechenden Standardabweichungen. Wenn die Volumenverteilung des Sphäroids normal ist, wenden Sie den Dixon-Test an, um den Ausreißerwert zu identifizieren und zu verwerfen. Danach wird das Volumenverhältnis zwischen den behandelten Proben und den Proben zusammen mit der Fehlerausbreitung berechnet, wie in Abbildung 6A dargestellt.
Ergebnisse
Dox@DDAB-MDs wurden gemäß dem Protokoll (Abschnitt 1) entwickelt, wie in Abbildung 1 schematisch beschrieben. Die erhaltenen MDs bestehen aus einer Monolage aus DDAB, die den DFP-Kern verkapselt (Abbildung 1A). Die kationische Ladung von DDAB und das Beschallungsverfahren verhindern die Bildung von DDAB-Mehrlamellenschichten, die an der Grenzfläche zwischen DFP und Wassergestapelt sind 23
Diskussion
Um die Wirksamkeit von Anthrazyklinen als Antitumormedikamente zu verbessern, wird in dieser Arbeit die Bildung von DDAB-geschälten PFC-Tröpfchen vorgestellt, die das Chemotherapeutikum Doxorubicin (Dox) verkapseln, und die Wirkung einer solchen Formulierung bei der Interaktion mit den hochaggressiven dreifach negativen Brustkrebszellen MDA-MB-231.
Aufbau von DOX@DDAB-MDs
Dox-geladene MDs wurden nach der Insonationsmethode mit einem extre...
Offenlegungen
Interessenkonflikte: Die Autoren erklären keine Interessenkonflikte.
Menschen-/Tierrechte: Dieser Artikel enthält keine Studien mit Menschen oder Tieren, die von einem der Autoren durchgeführt wurden.
Danksagungen
Diese Arbeit wurde durch das Forschungs- und Innovationsprogramm Horizon 2020 der Europäischen Union im Rahmen der Fördervereinbarung AMPHORA (766456) gefördert.
Materialien
| Name | Company | Catalog Number | Comments |
| µ-Petri dish | Ibidi | 81156 | 35mm high, IbiTreat |
| 1,1,1,2,3,4,4,5,5,5-Decafluoropentane | Sigma-Aldrich | 138495-42-8 | b.p. 55°C |
| 12-well culture plate | Corning | ||
| 15 ml centrifuge tube | Falcon | 89039-664 | |
| 3D-Petri dishes 12:256 | Microtissues (Sigma-Aldrich) | Z764000-6EA | Small |
| 3D-Petri dishes 12:81 | Microtissues (Sigma-Aldrich) | Z764019-6EA | Large |
| 5%CO2 culture incubator, 37°C | Thermo Scienific | HERAcell 150i | |
| 50 ml centrifuge tube | Falcon | 352070 | |
| Biological safety cabinet, II level | |||
| Calcein | Sigma-Aldrich | ||
| Calcein-AM | Sigma-Aldrich | 148504-34-1 | 4mM stock solution in DMSO |
| cam sCMOS Andor Zyla 4.2 | Andor Instruments | ||
| Centrifuge Hettich Universal 320R | Hettich Lab. Technology | ||
| DAPI | SIgma-Aldrich | ||
| Dimethyldioctadecylammonium bromide powder | Sigma-Aldrich | 3700-67-2 | |
| DMEM (Dulbecco's Modified Eagle Medium) | Corning | 15-013-CV | |
| Doxorubicin hydrochloride | Sigma-Aldrich | 25316-40-9 | |
| DPBS (Dulbecco's Modified PBS) | Corning | 21-030-CV | pH 7,4 |
| Ethanol 70% | Sigma-Aldrich | ||
| EZ-C1 digital ecliplse | Nikon Instruments | Silver version 3.91 | |
| Fetal Bovine Serum (FBS) | Corning | 35-079-CV | |
| Goniometer BI-200SM | Brookhaven Instruments Corporations | ||
| Laser Ar+ | Spectra Physics | ||
| Laser He-Ne | Melles-Griot | ||
| L-Glutammine | Corning | 25-005-CI | |
| Mcroscope Nikon Eclipse Ti | Nikon Instruments | ||
| MDA-MB 231 cell line | ATCC | ||
| Microsoft Excel | Microsoft | ||
| Microplates reader Spark | Tecan group | ||
| NanoZetaSizer ZS | Malvern Instruments LTD | ||
| Neubauer improved chamber | 718605 | ||
| NIS Elements software | Nikon Instruments | AR 4.30 | |
| Pen/Strepto | Corning | 30-002-CI | |
| Photocorrelator BI-9000 AT | Brookhaven Instruments Corporations | 62927-1 | |
| Photometer HC120 | Brookhaven Instruments Corporations | N° 1275 | |
| Pipettors and tips, various size | Gilson | ||
| Propidium Iodide | SIgma-Aldrich | ||
| Serological pipets, various size | Corning | ||
| Solid-state laser | Suwtech Laser | N° 22368 | |
| T25 Flasks | Sarstedt | 83.3910.002 | |
| T75 Flasks | Sarstedt | 83.3911.002 | |
| Trypsin/EDTA 0.05% | EuroClone | ECB3052D | |
| Vibra-Cell VCX-400 | Sonics & Materials, inc | ||
| Water bath | 37°C |
Referenzen
- Aryal, S., Park, H., Leary, J. F., Key, J. Top-down fabrication-based nano/microparticles for molecular imaging and drug delivery. International Journal of Nanomedicine. 14, 6631-6644 (2019).
- Peng, Y., et al. Research and development of drug delivery systems based on drug transporter and nano-formulation. Asian Journal of Pharmaceutical Sciences. 15, 220-236 (2020).
- Chan, K. S., Koh, C. G., Li, H. Y. Mitosis-targeted anti-cancer therapies: Where they stand. Cell Death and Disease. 3, 411 (2012).
- Raj, S., Franco, V. I., Lipshultz, S. E. Anthracycline-induced cardiotoxicity: A review of pathophysiology, diagnosis, and treatment. Current Treatment Options in Cardiovascular Medicine. 16, 315 (2014).
- Iyer, A. K., Singh, A., Ganta, S., Amiji, M. M. Role of integrated cancer nanomedicine in overcoming drug resistance. Advanced Drug Delivery Reviews. 65, 1784-1802 (2013).
- Blanco, E., Shen, H., Ferrari, M. Principles of nanoparticle design for overcoming biological barriers to drug delivery. Nature Biotechnology. 33, 941-951 (2015).
- Davis, M. E., Chen, Z., Shin, D. M. Nanoparticle therapeutics: An emerging treatment modality for cancer. Nature Reviews Drug Discovery. 7, 771-782 (2008).
- Lammers, T., Hennink, W. E., Storm, G. Tumour-targeted nanomedicines: Principles and practice. British Journal of Cancer. 99, 392-397 (2008).
- Couvreur, P., et al. Polycyanoacrylate nanocapsules as potential lysosomotropic carriers: preparation, morphological and sorptive properties. Journal of Pharmacy and Pharmacology. 31, 331-332 (1979).
- Yordanov, G. G., Dushkin, C. D. Preparation of poly(butylcyanoacrylate) drug carriers by nanoprecipitation using a pre-synthesized polymer and different colloidal stabilizers. Colloid and Polymer Science. 288, 1019-1026 (2010).
- Kooiman, K., Vos, H. J., Versluis, M., De Jong, N. Acoustic behavior of microbubbles and implications for drug delivery. Advanced Drug Delivery Reviews. 72, 28-48 (2014).
- Fasolato, C., et al. Antifolate SERS-active nanovectors: Quantitative drug nanostructuring and selective cell targeting for effective theranostics. Nanoscale. 11, 15224-15233 (2019).
- Cerroni, B., et al. Temperature-tunable nanoparticles for selective biointerface. Biomacromolecules. 16, 1753-1760 (2015).
- Chronopoulou, L., et al. PLGA based particles as "drug reservoir" for antitumor drug delivery: characterization and cytotoxicity studies. Colloids Surfaces B: Biointerfaces. 180, 495-502 (2019).
- Calderó, G., Paradossi, G. Ultrasound/radiation-responsive emulsions. Current Opinion in Colloid and Interface Science. 49, 118-132 (2020).
- Capece, S., et al. Complex interfaces in 'phase-change' contrast agents. Physical Chemistry Chemical Physics. 18, 8378-8388 (2016).
- Mielczarek, L., et al. In the triple-negative breast cancer MDA-MB-231 cell line, sulforaphane enhances the intracellular accumulation and anticancer action of doxorubicin encapsulated in liposomes. International Journal of Pharmaceutics. 558, 311-318 (2019).
- Ravi, M., Paramesh, V., Kaviya, S. R., Anuradha, E., Paul Solomon, F. D. 3D cell culture systems: Advantages and applications. Journal of Cellular Physiology. 230, 16-26 (2015).
- Heinonen, T. Better science with human cell-based organ and tissue models. Alternatives to Laboratory Animals. 43, 29-38 (2015).
- Thoma, C. R., Zimmermann, M., Agarkova, I., Kelm, J. M., Krek, W. 3D cell culture systems modeling tumor growth determinants in cancer target discovery. Advanced Drug Delivery Reviews. 69-70, 29-41 (2014).
- Astashkina, A., Grainger, D. W. Critical analysis of 3-D organoid in vitro cell culture models for high-throughput drug candidate toxicity assessments. Advanced Drug Delivery Reviews. 69-70, 1-18 (2014).
- Weigelt, B., Ghajar, C. M., Bissell, M. J. The need for complex 3D culture models to unravel novel pathways and identify accurate biomarkers in breast cancer. Advanced Drug Delivery Reviews. 69-70, 42-51 (2014).
- Feitosa, E., Karlsson, G., Edwards, K. Unilamellar vesicles obtained by simply mixing dioctadecyldimethylammonium chloride and bromide with water. Chemistry and Physics of Lipids. 140, 66-74 (2006).
- Cancerrxgene. Doxorubicin IC50. Genomics of drug sensitivity in cancer Available from: https://www.cancerrxgene.org/compound/Doxorubicin/133/overview/ic50 (2020)
- Boo, L., et al. Phenotypic and microRNA transcriptomic profiling of the MDA-MB-231 spheroid-enriched CSCs with comparison of MCF- 7 microRNA profiling dataset. PeerJ. 2017, 1-27 (2017).
- Domenici, F., Castellano, C., Dell'unto, F., Congiu, A. Temperature-dependent structural changes on DDAB surfactant assemblies evidenced by energy dispersive X-ray diffraction and dynamic light scattering. Colloids and Surfaces B: Biointerfaces. 95, 170-177 (2012).
- Choosakoonkriang, S., et al. Infrared spectroscopic characterization of the interaction of cationic lipids with plasmid DNA. Journal of Biological Chemistry. 276, 8037-8043 (2001).
- Yin, H., et al. Non-viral vectors for gene-based therapy. Nature Reviews Genetics. 15 (8), 541-555 (2014).
- Lentacker, I., Geers, B., Demeester, J., De Smedt, S. C., Sanders, N. N. Design and evaluation of doxorubicin-containing microbubbles for ultrasound-triggered doxorubicin delivery: Cytotoxicity and mechanisms involved. Molecular Therapy. 18, 101-108 (2010).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten