Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Visualisierung der Konformationsdynamik von Membranrezeptoren mit Einzelmolekül-FRET
In diesem Artikel
Zusammenfassung
Diese Studie stellt ein detailliertes Verfahren zur Durchführung von Einzelmolekül-Fluoreszenz-Resonanzenergietransfer-Experimenten (smFRET) an G-Protein-gekoppelten Rezeptoren (GPCRs) unter Verwendung ortsspezifischer Markierung durch unnatürliche Aminosäureinkorporation (UAA) vor. Das Protokoll bietet eine Schritt-für-Schritt-Anleitung für die Vorbereitung von smFRET-Proben, Experimente und Datenanalysen.
Zusammenfassung
Die Fähigkeit von Zellen, auf externe Signale zu reagieren, ist für die Zellentwicklung, das Wachstum und das Überleben unerlässlich. Um auf ein Signal aus der Umgebung reagieren zu können, muss eine Zelle in der Lage sein, es zu erkennen und zu verarbeiten. Diese Aufgabe beruht hauptsächlich auf der Funktion von Membranrezeptoren, deren Aufgabe es ist, Signale in die biochemische Sprache der Zelle umzuwandeln. G-Protein-gekoppelte Rezeptoren (GPCRs) bilden die größte Familie von Membranrezeptorproteinen beim Menschen. Unter den GPCRs sind metabotrope Glutamatrezeptoren (mGluRs) eine einzigartige Unterklasse, die als obligate Dimere fungieren und eine große extrazelluläre Domäne besitzen, die die Ligandenbindungsstelle enthält. Jüngste Fortschritte in Strukturstudien von mGluRs haben das Verständnis ihres Aktivierungsprozesses verbessert. Die Ausbreitung großräumiger Konformationsänderungen durch mGluRs während der Aktivierung und Modulation ist jedoch kaum verstanden. Der Einzelmolekül-Fluoreszenz-Resonanzenergietransfer (smFRET) ist eine leistungsstarke Technik zur Visualisierung und Quantifizierung der Strukturdynamik von Biomolekülen auf Einzelproteinebene. Um den dynamischen Prozess der mGluR2-Aktivierung zu visualisieren, wurden fluoreszierende Konformationssensoren auf Basis des Einbaus von unnatürlichen Aminosäuren (UAA) entwickelt, die eine ortsspezifische Proteinmarkierung ohne Störung der nativen Struktur von Rezeptoren ermöglichten. Das hier beschriebene Protokoll erklärt, wie diese Experimente durchzuführen sind, einschließlich des neuartigen UAA-Markierungsansatzes, der Probenvorbereitung und der smFRET-Datenerfassung und -analyse. Diese Strategien sind verallgemeinerbar und können erweitert werden, um die Konformationsdynamik einer Vielzahl von Membranproteinen zu untersuchen.
Einleitung
Die Übertragung von Informationen über die Plasmamembran hängt stark von der Funktion der Membranrezeptorenab 1. Die Bindung von Liganden an einen Rezeptor führt zu einer Konformationsänderung und Rezeptoraktivierung. Dieser Prozess ist oft allosterischer Natur2. Mit über 800 Mitgliedern sind G-Protein-gekoppelte Rezeptoren (GPCRs) die größte Familie von Membranrezeptoren beim Menschen3. Aufgrund ihrer Rolle in fast allen zellulären Prozessen sind GPCRs zu wichtigen Zielen für die therapeutische Entwicklung geworden. Im kanonischen Modell der GPCR-Signalübertragung führt die Agonistenaktivierung zu Konformationsänderungen des Rezeptors, die anschließend den heterotrimeren G-Proteinkomplex durch Austausch von GDP gegen GTP an der Nukleotidbindungstasche von Gα aktivieren. Die aktiviertenG-α-GTP- undG-βγ-Untereinheiten steuern dann die Aktivität nachgeschalteter Effektorproteine und propagieren die Signalkaskade 4,5. Dieser Signalprozess hängt wesentlich von der Fähigkeit der Liganden ab, die dreidimensionale Form des Rezeptors zu verändern. Ein mechanistisches Verständnis davon, wie Liganden dies erreichen, ist entscheidend für die Entwicklung neuer Therapeutika und das Design synthetischer Rezeptoren und Sensoren.
Metabotrope Glutamatrezeptoren (mGluR) gehören zur GPCR-Familie der Klasse C und sind wichtig für die langsamen neuromodulatorischen Effekte von Glutamat und die Abstimmung der neuronalen Erregbarkeit 6,7. Unter allen GPCRs sind GPCRs der Klasse C strukturell einzigartig, da sie als obligate Dimere fungieren. mGluRs enthalten drei strukturelle Domänen: die Venusfliegenfalle (VFT), die Cystein-reiche Domäne (CRD) und die Transmembrandomäne (TMD)8. Die Konformationsänderungen während des Aktivierungsprozesses sind komplex und beinhalten lokale und globale Konformationskopplungen, die sich über eine Entfernung von 12 nm ausbreiten, sowie Dimerkooperativität. Die intermediären Konformationen, die zeitliche Ordnung der Zustände und die Übergangsrate zwischen den Zuständen sind unbekannt. Durch die Verfolgung der Konformation einzelner Rezeptoren in Echtzeit ist es möglich, die transienten Zwischenzustände und die Abfolge der Konformationsänderungen während der Aktivierung zu identifizieren. Dies kann durch Anwendung des Einzelmolekül-Fluoreszenzresonanzenergietransfers 9,10 (smFRET) erreicht werden, wie er kürzlich angewendet wurde, um die Ausbreitung von Konformationsänderungen während der Aktivierung von mGluR2 11 zu visualisieren. Ein wichtiger Schritt in FRET-Experimenten ist die Erzeugung von FRET-Sensoren durch ortsspezifisches Einfügen der Donor- und Akzeptorfluorophore in das interessierende Protein. Eine Strategie zur Einbeziehung unnatürlicher Aminosäuren (UAA) wurde 12,13,14,15 angenommen, um die Einschränkungen typischer ortsspezifischer fluoreszierender Markierungstechnologien zu überwinden, die die Schaffung von Cystein-losen Mutanten oder das Einfügen eines großen genetisch kodierten Tags erfordern. Damit konnte die Konformationsumlagerung des essentiellen kompakten allosterischen Linkers beobachtet werden, der die Ligandenbindungs- und Signaldomänen von mGluR2 verband. In diesem Protokoll wird eine Schritt-für-Schritt-Anleitung zur Durchführung von smFRET-Experimenten mit mGluR2 vorgestellt, einschließlich des Ansatzes für die ortsspezifische Markierung von mGluR2 mit UAA zur Bindung von Fluorophoren unter Verwendung der kupferkatalysierten Azidcyclisierungsreaktion. Darüber hinaus beschreibt dieses Protokoll die Methodik für die direkte Erfassung von Membranproteinen und die Datenanalyse. Das hier skizzierte Protokoll ist auch auf die Untersuchung der Konformationsdynamik anderer Membranproteine anwendbar.
Protokoll
Der gesamte Workflow des Protokolls wird in Abbildung 1 beschrieben.
1. Vorbereitung der Probenkammer
- Gleit- und Deckglasreinigung
HINWEIS: Diese Schritte zielen darauf ab, die Oberflächen der Objektträger sowie die Deckgläser zu reinigen und für die Aminosilanisierung vorzubereiten. Eine kritische Voraussetzung für die Durchführung von Einzelmolekül-Fluoreszenzexperimenten an oberflächengebundenen Molekülen ist eine passivierte Oberfläche. Die zuverlässigste und reproduzierbarste Passivierungstechnik besteht darin, inerte Polymerketten kovalent als dichte Schicht an die Glasoberfläche anzubringen. Polyethylenglykol (PEG) ist das effizienteste Polymer für die Oberflächenpassivierung16. Die Details des Passivierungsverfahrens mit PEG (PEGylierung) sind nachfolgend beschrieben:- Markieren Sie die zu bohrenden Löcher auf den Objektträgern mit einem Marker (~6 mm Abstand und vom Rand entfernt). Bohren Sie mit einem Dremel kleine Löcher (1 mm Durchmesser) auf den Glasobjektträger. Tauchen Sie die Objektträger während des Bohrvorgangs in Wasser.
- Waschen Sie die Objektträger mit Aceton, um Resttinte vom Marker zu entfernen.
- Entfernen Sie Aceton und spülen Sie die Objektträger mit Wasser ab, dann Mikrowelle für 5 Minuten in Wasser mit hoher Leistung (700 W).
- Reinigen Sie die Objektträger mit Wasser, bevor Sie sie zur Beschallung in ein Glasfleckenglas legen. Legen Sie die Deckgläser in ein anderes Färbeglas.
- Die Objektträger und Deckgläser in Aceton 30 min in einem Badesonikator bei 23 °C beschallen.
- In der Zwischenzeit reinigen Sie einen Glaskolben zur Vorbereitung der im nächsten Schritt verwendeten Aminosilanisierungslösung. Füllen Sie den Kolben mit 1 M KOH, beschallen Sie den Kolben für 30 min, spülen Sie den KOH gründlich mit Wasser aus und beschallen Sie ihn dann für weitere 30 Minuten in Methanol. Der Kolben wird bis zum Zeitpunkt der Aminosilanisierung in Methanol belassen.
- In der Zwischenzeit das Aminosilan, mPEG und Biotin-PEG aus dem Gefrierschrank (−20 °C) nehmen und im Dunkeln auf Raumtemperatur (RT) kommen lassen.
- Entsorgen Sie Aceton aus den Slide- und Deckglasgläsern im entsprechenden Chemikalienabfallbehälter, spülen Sie ihn gründlich mit Wasser ab und beschallen Sie die Objektträger dann in 5 M KOH für 30 min.
- Spülen Sie die Objektträger und Deckgläser mit Wasser ab und beschallen Sie sie dann 2 min lang mit Methanol (zweimal wiederholen). Lassen Sie die mit Methanol gefüllten Gläser bis zum Aminosilanisierungsschritt.
HINWEIS: In dieser Studie wird entionisiertes Wasser verwendet, sofern nicht anders angegeben. Es wird ein Badsonikator verwendet, der bei 23 °C arbeitet.
- Aminosilanisierung
HINWEIS: Dieser Schritt zielt darauf ab, Aminosilan kovalent mit der sauberen Objektträger- und Deckglasoberfläche zu verbinden. Funktionalisiertes mPEG und Biotin-PEG werden im nächsten Schritt kovalent an diese Oberfläche binden.- Bereiten Sie eine Aminosilanisierungsmischung in einem sauberen Kolben vor (Schritt 1.6). Bereiten Sie die Lösung vor, indem Sie Methanol (150 ml), Essigsäure (7,5 ml, Glaspipette verwenden) und Aminosilan (2,5 ml) mischen.
- Mischen Sie die Lösung vorsichtig im chemischen Abzug und gießen Sie sie dann in die Gläser und Deckgläser. Verwenden Sie 50 mL für die Deckgläser und 100 mL für die Objektträger. Stellen Sie sicher, dass Dias und Deckgläser vollständig in die Lösung eingetaucht sind.
- Inkubieren Sie für 10 min, dann beschallen Sie die Gläser für 1 min (Ultraschall entfernt die Verunreinigungen von der Oberfläche) und inkubieren Sie für weitere 10 Minuten.
- Entsorgen Sie die Aminosilanisierungslösung im Abfallsammelbehälter. Methanol in die Gläser geben, die Gläser mit Deckeln verschließen und vorsichtig von Hand schütteln. Entsorgen Sie das Methanol und füllen Sie die Gläser mit Wasser.
- Geben Sie die Aminosilanflasche in den Gefrierschrank (−20 °C).
- PEGylierung
HINWEIS: Diese Schritte beschreiben das PEGylierungsverfahren.- Bereiten Sie während der Aminosilanisierung den Pegylierungspuffer vor. Wiegen Sie 84 mg Natriumbicarbonat (NaHCO3) und geben Sie es zu 10 ml Wasser (10 mM). Zusätzlich mPEG und Biotin-PEG wiegen und beiseite legen. Verwenden Sie für sechs Objektträger und Deckgläser 96 mg mPEG und 1,2-2,4 mg Biotin-PEG.
HINWEIS: Es ist wichtig, nicht zu viel Biotin-PEG hinzuzufügen, da dies die Anzahl der Hintergrundflecken erhöhen kann. Lösen Sie die PEG-Mischung erst unmittelbar vor dem Auftragen auf Objektträger auf. - Spülen Sie die Objektträger mit Wasser ab, trocknen Sie sie mit einem leichten Luftstoß ab und legen Sie sie dann in die befeuchteten Montageboxen.
HINWEIS: Es ist wichtig, eine saubere Fluggesellschaft zu verwenden. Vermeiden Sie die Verwendung von komprimierter Dosenluft, die Rückstände auf dem Glas hinterlassen kann. - Fügen Sie der PEG-Pulvermischung einen Pegylierungspuffer hinzu und pipettieren Sie mehrmals vorsichtig auf und ab, um sich aufzulösen. Fügen Sie 55 μL Pegylierungspuffer pro Objektträger hinzu (für sechs Objektträger fügen Sie 330 μL Pegylierungspuffer hinzu).
- Zentrifugieren Sie bei 9.600 x g für 1 min bei 23 °C, um ungelöste Partikel auszufällen. Sammeln Sie den Überstand, der im nächsten Schritt verwendet werden soll.
HINWEIS: Biotin-PEG hydrolysiert aufgrund der Anwesenheit der NHS-Gruppe schnell. Es ist wichtig, die Misch- und Zentrifugationsschritte schnell durchzuführen. - Tragen Sie 60 μL PEGylierungslösung auf jeden Objektträger auf und legen Sie dann das Deckglas darauf, so dass die PEGylierungslösung zwischen dem Objektträger und dem Deckglas liegt.
HINWEIS: Vermeiden Sie das Einbringen von Blasen zwischen Objektträger und Deckglas, da dies die Passivierungseffizienz verringert. Entfernen Sie alle Blasen, indem Sie das Deckglas und den Objektträger mit einer Pipettenspitze anpassen. - Legen Sie die Dias in eine flache und dunkle Schublade. Dias können über Nacht gelagert werden; Die Inkubation für 4-6 h führt jedoch zu einer optimalen Passivierung.
HINWEIS: Es ist wichtig, sich daran zu erinnern, welche Seite pegyliert ist. - Markieren Sie die nicht pegylierte Seite vor der Lagerung. Nach der Inkubation die Objektträger und Deckgläser vorsichtig zerlegen und gründlich mit Wasser abspülen
- Trocknen Sie die Pantoletten und Deckgläser durch Luftblasen. Bewahren Sie die Objektträger und Deckgläser in einem sterilen Röhrchen (50 ml) auf, wobei die PEGylierte Oberfläche voneinander abgewandt ist. Bei −20 °C bis zum Tag des Experiments lagern.
HINWEIS: Es ist am besten, Dias und Deckgläser innerhalb von 4 Wochen nach der Zubereitung zu verwenden. Die PEGylierten Oberflächen sollten voneinander abgewandt sein. Die Lagerung der PEGylierten Objektträger und Deckgläser in vakuumversiegelten Beuteln kann ihre Haltbarkeit erhöhen.
- Bereiten Sie während der Aminosilanisierung den Pegylierungspuffer vor. Wiegen Sie 84 mg Natriumbicarbonat (NaHCO3) und geben Sie es zu 10 ml Wasser (10 mM). Zusätzlich mPEG und Biotin-PEG wiegen und beiseite legen. Verwenden Sie für sechs Objektträger und Deckgläser 96 mg mPEG und 1,2-2,4 mg Biotin-PEG.
2. mGluR2-Expression mit inkorporierter unnatürlicher Aminosäure, fluoreszierender Markierung und Extraktion
HINWEIS: Dieses Protokoll beschreibt die Vorbereitung, Reagenzien und Behandlung von Zellen zur Expression von mGluR2, das das UAA-4-Azido-L-phenylalanin (AZP) enthält. Das Verfahren ist für HEK293T-Zellen, die auf 18 mm Glasdeckgläsern gezüchtet werden. Das Verfahren kann bei Bedarf skaliert werden.
- Bekeimung
HINWEIS: Halten Sie die HEK293T-Zellen in DMEM, ergänzt mit 10% (v/v) fetalem Rinderserum, 100 Einheiten·ml-1 Penicillin-Streptomycin und 15 mM HEPES-Puffer (Zusatzdatei 1) (pH 7,4) bei 37 °C unter 5% CO2.- Durchdringen Sie die Zellen mit 0,05% Trypsin-EDTA. Samen Sie HEK293T-Zellen auf Poly-L/D-Lysin (PLL/PDL) Glasdeckgläsern, so dass sie während der Transfektionszeit ≥80% Konfluenz erreichen.
- Transfektion
- Bereiten Sie eine 40 mM Stammlösung von AZP in 0,1 M NaOH vor.
- Bereiten Sie AZP-ergänzte Medien für das Wachstum von Zellen und die mGluR2-Expression vor. Ergänzen Sie Standardmedien (+ FBS, Pen/Streptokokken, 15 mM HEPES) mit 40 mM AZEP-Stammlösung. Die endgültige AZZ-Konzentration wird auf 0,6 mM gebracht. Fügen Sie 1 M HEPES-Lösung hinzu (die Hälfte des Volumens von 40 mM AZZ-Stammlösung hinzugefügt). Um beispielsweise 10 ml mit AZP ergänzte Medien herzustellen, kombinieren Sie 9,775 ml Standardmedien, 150 μL 40 mM AZP-Stammlösung und 75 μL 1 M HEPES-Lösung.
- Filtern (sterilisieren) Sie das Medium mit einem Spritzenfilter (0,2 μm, PES).
- Ersetzen Sie die Standardmedien vor der Transfektion durch Medien, die AZP-ergänzte Medien enthalten.
HINWEIS: Achten Sie darauf, die Zellen während des Austauschs der Medien nicht auszutrocknen. - Transfizieren Sie die Zellen mit Transfektionsreagenz (Table of Materials) gemäß der Bedienungsanleitung des Herstellers. Transfizieren Sie HEK293T-Zellen auf einem 18 mm Deckglas mit insgesamt 2 μg DNA (1000 ng tRNA/Synthetase + 1000 ng bernsteinfarbenes Codon-haltiges Proteinplasmid). In Tabelle 1 finden Sie die Konzentration und das Volumen der verwendeten Komponenten.
- Wechseln Sie das Medium 24 h nach der Transfektion auf frische, AZP-ergänzte Medien und lassen Sie die Zellen für weitere 24 Stunden wachsen.
- Markierung mit Alkincyaninfarbstoffen
- 20 Minuten vor dem Etikettieren die Deckgläser zweimal mit warmem (37 °C) Aufnahmepuffer (RB) (Zusatzdatei 1) waschen und auf warme (37 °C) Standardmedien ohne AZP (+ FBS, Pen/Streptokokken, 15 mM HEPES) verschieben.
- Die Markierungslösung enthält Cy3-Alkin, Cy5-Alkin, BTTES, Kupfer(II)-sulfat (CuSO4), (+) Natrium-L-ascorbat und Aminoguanidin.
- Befolgen Sie die Reihenfolge der Erstellung und des Hinzufügens von Lösungen (Tabelle 2):
- Bereiten Sie 50 mM BTTES vor.
- Bereiten Sie 100 mM Aminoguanidin vor.
- 100 mM Na-Ascorbat vorbereiten.
- Bereiten Sie 655,5 μL RB vor.
- Fügen Sie dem RB Cy3/Cy5-Alkinfarbstoff (10 mM in DMSO-Schaft) hinzu.
HINWEIS: Fügen Sie dem RB Aminoguanidin hinzu. - Bereiten Sie 20 mM CuSO 4 vor.
- Mischen Sie CuSO4 und BTTES in einer neuen Röhre (die Lösung wird blau).
- Die CuSO4- und BTTES-Mischung wird zu RB (2.3.3.6) hinzugefügt.
- Fügen Sie das Na-Ascorbat hinzu.
- Befolgen Sie das in Tabelle 2 angegebene Volumen für ein Deckglas von 18 mm.
- Mischen Sie die Lösung gründlich und inkubieren Sie auf Eis und im Dunkeln für 10 Minuten, bevor Sie die Zellen markieren.
- Bevor Sie die Etikettierlösung zu den Deckgläsern geben, entfernen Sie die Medien und waschen Sie sie mit RB. Die Markierungslösung zugeben und 15 min bei 37 °C bei Dunkelheit inkubieren.
- HINWEIS: Um die Markierung zu verbessern, fügen Sie Glutamat (Endkonzentration ~ 0,5 mM) nach 10 min hinzu und inkubieren Sie für weitere 5 Minuten. Kupfer ist sehr giftig für die Zellen, und die Markierungsreaktion sollte nicht länger als 15 Minuten in vivo fortgesetzt werden. Bereiten Sie alle Komponenten frisch zu. Fügen Sie zuletzt Na-Ascorbate hinzu. Halten Sie die Reaktion während der Zubereitung bei 4 ° C. Nach Zugabe einer Markierungslösung sind die Zellen jedoch bei 37 °C im Inkubator zu lagern.
- Ernte der Zellen und Extraktion der Proteine (Zelllyse)
- Entfernen Sie die Etikettierlösung und waschen Sie das Deckglas (18 mm) mit den mGluR2-transfizierten Zellen zweimal mit dem RB.
- Die Zellen mit einer Pipette vom Deckglas waschen und in RB (1 ml) resuspendieren.
HINWEIS: Minimieren Sie die Lichtexposition der Probe nach diesem Punkt so weit wie möglich. - Pelletieren Sie die Zellen durch Schleudern bei 1.000 x g bei 4 °C für 5 min und entfernen Sie den Überstand. Resuspendieren Sie das Zellpellet in 80-130 μL der Lyselösung.
HINWEIS: Das Zellpellet sollte mit dem Auge sichtbar sein. Das Lysevolumen hängt von der Menge der während des Etikettier- und Waschprozesses verlorenen Probe ab. - Mischen Sie vorsichtig durch Pipettieren, um das Pellet aufzubrechen. In Folie einwickeln und bei 4 °C für 0,5-1 h auf die Wippe legen, um die Zellen zu lysieren.
- Pelletieren Sie die unlösliche Fraktion durch Zentrifugation bei 20.000 x g und 4 °C für 20 min. Übertragen Sie den Überstand in ein frisches kaltes Röhrchen (das lysierte Protein, das das fluoreszierend markierte Protein von Interesse enthält) und lagern Sie es für Experimente auf Eis.
3. Einzelmolekül-Durchflusskammeraufbau und Funktionalisierung
- Nehmen Sie den Objektträger und das Deckglas aus dem Gefrierschrank und lassen Sie sie bei RT bei Dunkelheit (~30 min) erwärmen.
- Montieren Sie die Kammer mit doppelseitigem Klebeband, indem Sie Streifen aus doppelseitigem Klebeband zwischen Objektträger und Deckglas einschließen. Stellen Sie sicher, dass die PEGylierten Oberflächen das Innere der Durchflusskammer bilden.
- Drücken Sie das Deckglas mit einer Pipetenspitze, um sicherzustellen, dass das Band vollständigen Kontakt mit Deckglas und Objektträger hat. Achten Sie darauf, das Deckglas nicht zu zerbrechen. Tragen Sie Epoxidharz auf die Ränder der Objektträger auf.
HINWEIS: Fügen Sie nicht so viel hinzu, dass Epoxidharz die gebohrten Löcher füllt. - Stellen Sie die Durchflusskammer mit der Deckglasseite nach unten in eine befeuchtete dunkle Box, damit das Epoxidharz trocknen kann (~ 30 min).
HINWEIS: Fügen Sie T50-Puffer (Zusatzdatei 1) durch die Bohrlöcher hinzu, um zu verhindern, dass das Epoxidharz die Löcher (10-15 μL) während der Trocknungszeit bedeckt. - Inkubieren Sie jede Kammerbahn mit 500 nM Neutravidin (verdünnt in T50), indem Sie langsam ~ 40 μL auf jede Spur auftragen.
- Inkubieren Sie bei RT für 2 min in einer befeuchteten dunklen Box. Mit ~100 μL T50-Puffer pro Bahn waschen.
- Inkubieren Sie jede Kammerbahn mit 20 nM biotinyliertem Antikörper11. Die Wahl des Antikörpers hängt vom Tag auf dem Protein ab.
HINWEIS: Wenn der primäre Antikörper nicht biotinyliert ist, inkubieren Sie zuerst mit dem biotinylierten sekundären Antikörper für 30 min und inkubieren Sie dann mit dem primären Antikörper. - Inkubieren Sie bei RT für 30 Minuten in einer befeuchteten dunklen Box. Waschen Sie mit ~200 μL T50 pro Bahn.
HINWEIS: Stellen Sie sicher, dass die Bahnen während des Vorbereitungsprozesses niemals austrocknen.
4. Einzelmolekül-Puffer
- Trolox Puffer
HINWEIS: Trolox-Puffer ist der Startpuffer für die Erstellung von Imaging-Puffer. Die Komponenten des Puffers sind abhängig vom Experiment und können je nach interessierendem Protein variieren. Der in dem hier beschriebenen Protokoll verwendete Puffer umfasst Salze (NaCl, KCl, CaCl 2, MgCl2), Puffermittel (HEPES) und Trolox (pH ~ 7,35).- 9-10 mg Trolox werden in 10 ml Einzelmolekül-Aufzeichnungspuffer gelöst (SRB, Zusatzdatei 1)
HINWEIS: Trolox macht den Puffer leicht sauer. Stellen Sie den pH-Wert in diesem Stadium mit einer Natronlauge (NaOH), 10 M (pH 7,35), ein. Die Feineinstellung des pH-Werts erfolgt, nachdem Trolox vollständig aufgelöst ist. Eine Erhöhung des pH-Wertes an dieser Stelle erhöht jedoch die Löslichkeit des Trolox. - Mischen Sie die Lösung bei RT mit einer Tischwippe für 4-8 h (eingewickelt in Aluminiumfolie), um den Trolox vollständig aufzulösen.
- Überprüfen Sie den pH-Wert und passen Sie ihn bei Bedarf an.
- Stellen Sie sicher, dass Trolox vollständig aufgelöst ist. Die Lösung mit einem Spritzenfilter sterilisieren und bei 4 °C lagern.
HINWEIS: Der Puffer sollte nach 2-10 Tagen Alterung verwendet werden. Trolox hilft, das Blinken zu unterdrücken und wird häufig in Einzelmolekülstudien verwendet17. Die blinkhemmenden Eigenschaften stammen von einem oxidierten Derivat von Trolox18; Daher wird empfohlen, es mindestens ein paar Stunden bei RT zu halten, um zu reifen. Darüber hinaus beschleunigt die UV-Strahlung einer frischen Trolox-Lösung den Oxidationsprozess und kann verwendet werden, um die "Alterung" des Trolox-Puffers18 zu beschleunigen.
- 9-10 mg Trolox werden in 10 ml Einzelmolekül-Aufzeichnungspuffer gelöst (SRB, Zusatzdatei 1)
- Bildgebendes Pufferrezept: Mischen Sie Trolox-Puffer + Waschmittel (~ 2-facher CMC-Wert des Waschmittels) + 4 mM Protocatechusäure (PCA).
HINWEIS: Die Waschmittelkonzentration wird in der Nähe von CMC gehalten, da hohe Waschmittelkonzentrationen zu einer erhöhten Proteindenaturierung führen können. Zum Beispiel kann die folgende Mischung 955 μL Trolox + 5 μL 10% DDM + Cholesterin (W%, 10:1) + 40 μL 100 mM PCA-Stammlösung verwendet werden. Hier wirkt PCA als Antioxidans und wurde zuvor in smFRET-Studieneingesetzt 19. DDM ist nichtionisch, wird üblicherweise zur Solubilisierung von Membranproteinen20,21 verwendet und wurde in Einzelmolekülstudien verwendet. DDM ist ein gutes Waschmittel erster Wahl; Wir empfehlen jedoch, mehrere Reinigungsmittel zu testen und sicherzustellen, dass die Ergebnisse konsistent sind.
5. Mikroskopaufbau und smFRET-Datenerfassung
- Schalten Sie den Computer und das Mikroskop ein. Schalten Sie die Laser zum Aufwärmen ein (532 nm für Cy3-Anregung und 640 nm für Cy5-Anregung).
HINWEIS: Hier wurde ein inverses Mikroskop verwendet, das mit einem 100-fachen TIRF-Objektiv (N.A. 1.49), einem Bildteiler und einer EMCCD-Kamera ausgestattet war. Der Aufbau ist mit einem vierzeiligen Laserkombinierer, einem dichroitischen Spiegel, einem Langpass-Emissionsfilter, einem Emissionsdichroitenfilter und einem Kerbfilter ausgestattet. - Schalten Sie die EMCCD-Kamera ein, und öffnen Sie die Kamerasoftware. Warten Sie 20 Minuten, bis die Kamera −69 °C erreicht hat, und stabilisieren Sie sich.
- Montieren Sie die Probenkammer auf dem Mikroskoptisch. Fügen Sie die Proteinprobe schrittweise hinzu, um ~ 400 Moleküle pro Sichtfeld zu erreichen (Schritt 2.4.5). Waschen Sie die Kammer mit 100 μL des Bildpuffers.
- Passen Sie die Verstärkung, die Erfassungsrate und die Laserleistung so an, dass Einzelmolekül-Fluoreszenzsignale sowohl im Donor- als auch im Akzeptorkanal detektiert werden. Passen Sie bei Bedarf die Konzentration der Proteine in der Probenkammer an.
HINWEIS: Mit mehr als 400 Molekülen im Sichtfeld wird die Unterscheidung einzelner Moleküle schwieriger und das Hintergrundrauschen ist höher. - Erregen Sie den Spender und erfassen Sie Zeitspuren, bis mindestens 80% der Spendermoleküle im Sichtfeld photogebleicht sind.
- Schalten Sie am Ende des Films den 640-nm-Laser ein, um den Akzeptor direkt anzuregen, bis einige der Akzeptormoleküle photobleichen, um die Unterscheidung einzelner Moleküle vor Multimeren zu erleichtern.
- Wechseln Sie zu einem anderen Sichtfeld und wiederholen Sie die obigen Schritte, um mindestens drei Filme (technische Replikate) pro Bedingung zu sammeln.
HINWEIS: Verwenden Sie die niedrigstmögliche Laserleistung, wenn Sie einen neuen Bereich von Interesse (ROI) auswählen und fokussieren, um das Photobleaching zu minimieren. Achten Sie bei der Erfassung auf die Stufendrift. Wenn nach dem Wechsel zu einem neuen ROI eine spürbare Drift beobachtet wird, warten Sie 3 Minuten, bevor Sie mit der Erfassung beginnen.
6. Datenanalyse
- Ausrichtung von Donor- und Akzeptorkanälen (Movie Mapping)
- Zeichnen Sie die fluoreszierenden Perlenbilder in den Spender- und Akzeptorkanälen auf.
- Erzeugen Sie die Mapping-Datei unter Verwendung der Bead-Daten, um die Donor- und Akzeptorfluoreszenz von jedem Molekül22,23 zu korrelieren.
HINWEIS: Das Emissionssignal eines einzelnen Moleküls wird durch den dichroitischen Emissionsfilter im Bildteiler in Donor- und Akzeptorsignale aufgeteilt. Die Spender- und Akzeptorbilder werden nebeneinander auf die Kamera projiziert. Um die Donor- und Akzeptorintensitäten eines einzelnen Moleküls genau zwischen den beiden Bereichen zuzuordnen, wird häufig eine Mapping-Datei mit fluoreszierenden Perlenproben erstellt. Mit Hilfe dieser Mapping-Datei werden alle Moleküle, die in den Donor- und Akzeptorkanälen detektiert werden, aufeinander abgebildet. Die Analyse generiert dann die Zeitspuren, die die Donor- und Akzeptorintensitäten im Zeitverlauf für jedes Molekül darstellen.
- Auswahl von Einzelmolekül-FRET-Spuren (Partikelpicking)
HINWEIS: Einzelne Partikelspuren werden untersucht und für die nachgeschaltete Analyse mit MATLAB ausgewählt. Die genauen Auswahlkriterien hängen vom System ab. Allgemeine Richtlinien darüber, was ein Qualitätspartikel ausmacht, werden hier beschrieben. Alle benutzerdefinierten geänderten Codes sind auf GitHub (https://github.com/vafabakhsh-lab) verfügbar.- Wählen Sie die Spuren aus, bei denen die Gesamtintensität der Spuren (Donor + Akzeptor) über die Zeit stabil ist. Wählen Sie die Spuren mit antikorrelierten Änderungen der Spender- und Akzeptorintensitäten aus.
- Wählen Sie die Spender- und Akzeptormoleküle aus, die eine einstufige Photobleiche zeigen. Wählen Sie die Leiterbahnen aus, die >5 s lang sind.
HINWEIS: Der Hintergrund in jedem Kanal nach dem Bleichen sollte auf Null gehen. Die Ablaufverfolgungen sollten nicht viele blinkende Ereignisse aufweisen. Dies erhöht die Schwierigkeit der Analyse. - Berechnen Sie den FRET-Wirkungsgrad mit der Gleichung E = (I A− 0,088 × I D)/(I D + [I A− 0,088 × I D])24,25,26, wobei I D und I A Rohdonor- bzw. Akzeptorintensitäten sind.
HINWEIS: Die Leckage der Donoremission in den Akzeptorkanal wird unter Verwendung einer nur durch Donor markierten Probe bestimmt, die mit einem 532-nm-Laser26 angeregt wurde. Der Leckagekorrekturfaktor von 0,088 kann je nach verwendetem Filtersatz für verschiedene Setups unterschiedlich sein. Es ist wichtig anzumerken, dass die quantitative und robuste Umwandlung von FRET-Wirkungsgraden in absolute Entfernungen die Korrektur der Donor- und Akzeptorintensitäten für mehrere Faktoren erfordert und vor27 ausführlich diskutiert wurde.
- Identifizieren des Konformationszustands durch Hidden-Markov-Modellierung (HMM)
- Führen Sie das Programm vbFRET28 in MATLAB aus und importieren Sie die ausgewählten Traces für eine bestimmte Bedingung. Legen Sie die Einschränkungen für die Anzahl der auszuführenden potenziellen Zustände und Iterationen fest.
HINWEIS: Basierend auf den Rohdaten aus den repräsentativen Ergebnissen wurde die Hypothese aufgestellt, dass der Konformationssensor bis zu vier diskrete FRET-Zustände belegt; So wurde ein Bereich von ein bis vier Staaten bezeichnet. Zuvor wurde festgestellt, dass Verbesserungen in der Anpassung mit >25 Iterationen vernachlässigbar zunehmen; So wurden 25 Iterationen für die Anpassung der repräsentativen Daten verwendet. - Analysieren Sie die smFRET-Traces und exportieren Sie die idealisierten Traces und die Analysesitzung. Speichern Sie die idealisierten Leiterbahnen für die nachgelagerte Analyse in einem separaten Ordner.
ANMERKUNG: Die Programme, die zum Extrahieren von Zustandsübergangs- und Verweilzeitdaten aus idealisierten Traces verwendet werden, wurden in der zuvor veröffentlichten Arbeit29 zur Verfügung gestellt. - Extrahieren Sie mithilfe von MATLAB-Programmen Zustandsübergänge und zeichnen Sie sie als Heatmap, wobei die X-Koordinate die Anfangskonformation und die Y-Koordinate die Endkonformation anzeigt.
HINWEIS: Übergänge sind definiert als Änderungen des FRET-Wertes >0,1 in den hier diskutierten repräsentativen Ergebnissen. Die Schwelle für Übergänge hängt von den hypothetischen Konformationszuständen ab, die ein Protein von Interesse einnimmt (die Übergangsschwelle ist kleiner als die Differenz zwischen den nächsten FRET-Zuständen), sowie von der Auflösung, die der Versuchsaufbau zulässt. Die Untersuchung von Heatmaps für mehrere Bedingungen ermöglicht die Identifizierung der häufigsten Konformationszustände, die ein Sensor durchläuft und somit einnimmt. Für die repräsentativen Ergebnisse wurden vier FRET-Zustände identifiziert (FRET = 0,31, 0,51, 0,71 und 0,89). - Extrahieren Sie mithilfe von MATLAB-Programmen die Verweilzeiten für jeden identifizierten Konformationszustand. Legen Sie einen Bereich von FRET-Werten fest, die jeden Zustand und die Zeitauflösung während der Datenerfassung beschreiben. FRET-Bereiche werden gleichmäßig durch benachbarte FRET-Zustände unterteilt. Exportieren Sie die Verweilzeiten für gegebene Behandlungsbedingungen.
HINWEIS: In den meisten Fällen können Verweilzeitdaten gut durch eine einzige exponentielle Zerfallsfunktion geschätzt werden. Diese Analyse kann in einer Datenanalyse- und Grafiksoftware durchgeführt werden.
- Führen Sie das Programm vbFRET28 in MATLAB aus und importieren Sie die ausgewählten Traces für eine bestimmte Bedingung. Legen Sie die Einschränkungen für die Anzahl der auszuführenden potenziellen Zustände und Iterationen fest.
- Gaußsche Anpassung von smFRET-Populationshistogrammen zur Quantifizierung der Zustandsbelegung
- Importieren Sie Populations-FRET-Histogramme für relevante Bedingungen in die Datenanalyse- und Grafiksoftware für mehrere Peak-Fitting-Analysen.
- Geben Sie die Anzahl der vorhandenen Peaks an (vier Peaks oder Zustände basierend auf HMM-Analyse). Die Anpassung wurde unter Verwendung mehrerer Gaußverteilungen30 durchgeführt, die als definiert sind
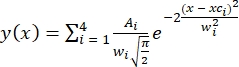 , wobei A die Peakfläche, xc das Peakzentrum und w die Peakbreite für jeden Peak ist.
, wobei A die Peakfläche, xc das Peakzentrum und w die Peakbreite für jeden Peak ist. - Legen Sie die Anpassungsparameter auf A > 0, xc = FRET ± 0,02 und 0,1 ≤w≤ 0,24 fest. Vier FRET-Peaks für einzelne FRET-Histogramme wurden gleichzeitig angepasst. Wenden Sie die definierten Randbedingungen für die Formstücke aller Bedingungen an.
- Berechnen Sie den Belegungszustand (Prozentsatz) als die Fläche von Spitzeninteresse geteilt durch die Gesamtfläche, definiert als Summe aller Spitzen.
Ergebnisse
Expression und fluoreszierende Markierung des UAA-basierten FRET-Sensors
Hier werden beispielhafte Ergebnisse der Insertion und Fluoreszenzmarkierung einer LF (AZP) innerhalb der CRD von mGluR2 (548UAA) diskutiert11. Wie bereits erwähnt, ist zum Einfügen von AZP in mGluR2 die Koexpression der technischen translationalen Maschinerie erforderlich, die eine modifizierte tRNA-Synthetase und komplementäre tRNA (pIRE4-Azi) und mGluR2 mit einem bernsteinfarbenen Codon an Position 5...
Diskussion
GPCRs sind Proteine, die auf der Zellmembran arbeiten, um die Signaltransduktion einzuleiten. Viele GPCRs bestehen aus mehreren Domänen, wobei die Signalisierung von der kooperativen Interaktion zwischen den Domänen abhängt. Um die Eigenschaften dieser Membranrezeptoren zu modulieren, ist es wichtig, das dynamische Verhalten der verschiedenen Domänen zu verstehen. Einzelmolekül-Fluoreszenz-Resonanzenergietransfer (smFRET) ist eine Fluoreszenztechnik, die die Messung von Proteinkonformation und -dynamik in Echtzeit e...
Offenlegungen
Die Autoren erklären keine Interessenkonflikte.
Danksagungen
Wir danken den Mitgliedern des Reza Vafabakhsh Labors für die Diskussionen. Diese Arbeit wurde vom National Institutes of Health Zuschuss R01GM140272 (an R.V.), vom Searle Leadership Fund for the Life Sciences an der Northwestern University und vom Chicago Biomedical Consortium mit Unterstützung der Searle Funds des Chicago Community Trust (an R.V.) unterstützt. B.W.L. wurde vom National Institute of General Medical Sciences (NIGMS) Training Grant T32GM-008061 unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| (+)-Sodium L-Ascorbate | Sigma Aldrich | Cat # 11140-250G | |
| 4-azido-L-phenylalanine | Chem-Impex International | Cat # 06162 | |
| 548UAA | Liauw et al. 2021 | Transfected construct | |
| Acetic Acid | Fisher Chemical | 64-19-7 | |
| Acetone | Fisher Chemical | 67-64-1 | |
| Adobe Illustrator (2022) | https://www.adobe.com/ | RRID:SCR_010279 | Software, algorithm |
| Aminoguanidine (hydrochloride) | Cayman Chemical | 81530 | |
| Aminosilane | Aldrich | 919-30-2 | |
| Bath Sonicator 2.8 L | Fisher Scientific | Ultrasonic Bath 2.8 L | |
| Biotin-PEG | Laysan Bio Inc | Item# Biotin-PEG-SVA-5000-100mg | |
| BTTES | Click Chemistry Tools | 1237-500 | |
| Copper (II) sulfate | Sigma Aldrich | Cat # 451657-10G | |
| Cover slip | VWR | 16004-306 | Sample chamber |
| Cy3 Alkyne | Click Chemistry Tools | TA117-5 | |
| Cy5 Alkyne | Click Chemistry Tools | TA116-5 | |
| DDM | Anatrace | Part# D310 1 GM | Detergent |
| DDM-CHS (10:1) | Anatrace | Part# D310-CH210 1 ML | Detergent with cholecterol |
| Defined Fetal Bovine Serum | Thermo Fisher Scientific | SH30070.03 | |
| Di01-R405/488/561/635 | Semrock | Notch filter | |
| DMEM | Corning | 10-013-CV | |
| EMCCD | Andor | DU-897U | Camera |
| ET542lp | Chroma | Long pass emission filter | |
| FF640-FDi01 | Semrock | Emission dichroic filter | |
| FLAG-tag antibody | Genscript | A01429 | |
| Fluorescent bead | Invitrogen T7279 | TetraSpeck microspheres | Spherical bead |
| Glass slides | Fisherfinest | 12-544-4 | sample chamber |
| Glutamate | Sigma Aldrich | Cat # 6106-04-3 | |
| HEK 293T | Sigma Aldrich | Cat # 12022001 | Cell line |
| HEPES | FisherBioReagents | 7365-45-9 | |
| Image splitter | OptoSplit II | ||
| KOH | Fluka | 1310-58-3 | |
| Laser | Oxxius | 4-line laser combiner | |
| Lipofectamine 3000 Transfection Reagent | Thermo Fisher Scientific | L3000015 | Transfection Reagent |
| Methanol | Fisher Chemical | 67-56-1 | |
| Microscope | Olympus | Olympus IX83 | |
| Milli-Q water | Barnstead | Water Deionizer | |
| m-PEG | Laysan Bio Inc | Item# MPEG-SIL-5000-1g | |
| NF03-405/488/532/635 | Semrock | Dichroic mirror | |
| OptiMEM | Thermo Fisher Scientific | 51985091 | Reduced Serum Medium |
| OptiMEM/Reduced serum medium | Thermo Fisher Scientific | ||
| OriginPro (2020b) | https://www.originlab.com/ | RRID:SCR_014212 | Data analysis and graphing software |
| Penicillin-Streptomycin | Gibco | 15140-122 | |
| pIRE4-Azi | Addgene | Plasmid # 105829 | Transfected construct |
| Poly-L-lysine hydrobromide | Sigma Aldrich | Cat # P2636 | |
| Protocatechuic acid (PCA) | HWI group | 99-50-3 | |
| smCamera (Version 1.0) | http://ha.med.jhmi.edu/resources/ | Camera software | |
| Sodium bicarbonate | FisherBioReagents | 144-55-8 | |
| Sodium hydroxide (NaOH) | Sigma | 1310-73-2 | |
| Syringe filter | Whatman UNIFLO | Cat#9914-2502 | Liquid filtration |
| Trolox | Sigma | 53188-07 |
Referenzen
- Smock, R. G., Gierasch, L. M. Sending signals dynamically. Science. 324 (5924), 198-203 (2009).
- Changeux, J. P., Christopoulos, A. Allosteric modulation as a unifying mechanism for receptor function and regulation. Cell. 166 (5), 1084-1102 (2016).
- Tang, X. -. l., Wang, Y., Li, D. -. l., Luo, J., Liu, M. -. Y. Orphan G protein-coupled receptors (GPCRs): biological functions and potential drug targets. Acta Pharmacologica Sinica. 33 (3), 363-371 (2012).
- Chung, K. Y., et al. Conformational changes in the G protein Gs induced by the β2 adrenergic receptor. Nature. 477 (7366), 611-615 (2011).
- Vafabakhsh, R., Levitz, J., Isacoff, E. Y. Conformational dynamics of a class C G-protein-coupled receptor. Nature. 524 (7566), 497-501 (2015).
- Niswender, C. M., Conn, P. J. Metabotropic glutamate receptors: Physiology, pharmacology, and disease. Annual Review of Pharmacology and Toxicology. 50, 295-322 (2010).
- Pin, J. P., Bettler, B. Organization and functions of mGlu and GABA(B) receptor complexes. Nature. 540 (7631), 60-68 (2016).
- Kniazeff, J., et al. Closed state of both binding domains of homodimeric mGlu receptors is required for full activity. Nature Structural & Molecular Biology. 11 (8), 706-713 (2004).
- Ha, T. Single-molecule fluorescence resonance energy transfer. Methods. 25 (1), 78-86 (2001).
- Schuler, B., Eaton, W. A. Protein folding studied by single-molecule FRET. Current Opinion in Structural Biology. 18 (1), 16-26 (2008).
- Liauw, B. W. -. H., Afsari, H. S., Vafabakhsh, R. Conformational rearrangement during activation of a metabotropic glutamate receptor. Nature Chemical Biology. 17 (3), 291-297 (2021).
- Noren, C. J., Anthonycahill, S. J., Griffith, M. C., Schultz, P. G. A general method for site-specific incorporation of unnatural amino acids into proteins. Science. 244 (4901), 182-188 (1989).
- Presolski, S. I., Hong, V. P., Finn, M. Copper-catalyzed azide-alkyne click chemistry for bioconjugation. Current Protocols in Chemical Biology. 3 (4), 153-162 (2011).
- Huber, T., Naganathan, S., Tian, H., Ye, S. X., Sakmar, T. P. Unnatural amino acid mutagenesis of GPCRs using amber codon suppression and bioorthogonal labeling. G Protein Coupled Receptors: Structure. 520, 281-305 (2013).
- Serfling, R., Coin, I., Pecoraro, V. Chapter Four - Incorporation of Unnatural Amino Acids into Proteins Expressed in Mammalian Cells. Methods in Enzymology. 580, 89-107 (2016).
- Chandradoss, S. D., et al. Surface passivation for single-molecule protein studies. Journal of Visualized Experiments. (86), e50549 (2014).
- Rasnik, I., McKinney, S. A., Ha, T. Nonblinking and long-lasting single-molecule fluorescence imaging. Nature Methods. 3 (11), 891-893 (2006).
- Cordes, T., Vogelsang, J., Tinnefeld, P. On the mechanism of Trolox as antiblinking and antibleaching reagent. Journal of the American Chemical Society. 131 (14), 5018-5019 (2009).
- Aitken, C. E., Marshall, R. A., Puglisi, J. D. An oxygen scavenging system for improvement of dye stability in single-molecule fluorescence experiments. Biophysical Journal. 94 (5), 1826-1835 (2008).
- Lee, S., et al. How do short chain nonionic detergents destabilize G-protein-coupled receptors. Journal of the American Chemical Society. 138 (47), 15425-15433 (2016).
- Cao, A. -. M., et al. Allosteric modulators enhance agonist efficacy by increasing the residence time of a GPCR in the active state. Nature Communications. 12 (1), 1-13 (2021).
- Mancebo, A., Mehra, D., Banerjee, C., Kim, D. -. H., Puchner, E. M. Efficient cross-correlation filtering of one-and two-color single molecule localization microscopy data. Frontiers in Bioinformatics. 1, 739769 (2021).
- Mehra, D., Adhikari, S., Banerjee, C., Puchner, E. M. Characterizing locus specific chromatin structure and dynamics with correlative conventional and super-resolution imaging in living cells. Nucleic Acids Research. , (2022).
- Chen, H., Puhl, H. L., Koushik, S. V., Vogel, S. S., Ikeda, S. R. Measurement of FRET efficiency and ratio of donor to acceptor concentration in living cells. Biophysical Journal. 91 (5), 39-41 (2006).
- Gopich, I. V., Szabo, A. FRET efficiency distributions of multistate single molecules. The Journal of Physical Chemistry B. 114 (46), 15221-15226 (2010).
- Roy, R., Hohng, S., Ha, T. A practical guide to single-molecule FRET. Nature Methods. 5 (6), 507-516 (2008).
- Hellenkamp, B., et al. Precision and accuracy of single-molecule FRET measurements-A multi-laboratory benchmark study. Nature Methods. 15 (9), 669-676 (2018).
- Bronson, J. E., Fei, J., Hofman, J. M., Gonzalez, R. L., Wiggins, C. H. Learning rates and states from biophysical time series: A Bayesian approach to model selection and single-molecule FRET data. Biophysical Journal. 97 (12), 3196-3205 (2009).
- Zhang, J., et al. Specific structural elements of the T-box riboswitch drive the two-step binding of the tRNA ligand. Elife. 7, 39518 (2018).
- Goodman, N. R. Statistical analysis based on a certain multivariate complex Gaussian distribution (an introduction). The Annals of Mathematical Statistics. 34 (1), 152-177 (1963).
- Brown, R. B., Audet, J. Current techniques for single-cell lysis. Journal of the Royal Society Interface. 5, 131-138 (2008).
- Schamber, M. R., Vafabakhsh, R. Mechanism of sensitivity modulation in the calcium-sensing receptor via electrostatic tuning. Nature Communications. 13 (1), 2194 (2022).
- Jain, A., Liu, R., Xiang, Y. K., Ha, T. Single-molecule pull-down for studying protein interactions. Nature Protocols. 7 (3), 445-452 (2012).
- Huang, S. K., et al. Delineating the conformational landscape of the adenosine A(2A) receptor during G protein coupling. Cell. 184 (7), 1884-1894 (2021).
- Wingler, L. M., et al. Angiotensin analogs with divergent bias stabilize distinct receptor conformations. Cell. 176 (3), 468-478 (2019).
- Gordon, C. G., et al. Reactivity of biarylazacyclooctynones in copper-free click chemistry. Journal of the American Chemical Society. 134 (22), 9199-9208 (2012).
- Kim, E., Koo, H. Biomedical applications of copper-free click chemistry: In vitro, in vivo, and ex vivo. Chemical Science. 10 (34), 7835-7851 (2019).
- Pickens, C. J., Johnson, S. N., Pressnall, M. M., Leon, M. A., Berkland, C. J. Practical considerations, challenges, and limitations of bioconjugation via azide-alkyne cycloaddition. Bioconjugate Chemistry. 29 (3), 686-701 (2018).
- Geng, Y., et al. Structural mechanism of ligand activation in human calcium-sensing receptor. Elife. 5, 13662 (2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten