Method Article
Entwicklung eines polymikrobiellen Kolonie-Biofilmmodells zur Erprobung von Antibiotika bei Mukoviszidose
In diesem Artikel
Zusammenfassung
Diese Methodik ermöglicht die Etablierung eines polymikrobiellen Biofilmmodells bei Mukoviszidose für antimikrobielle Sensitivitätstests in Forschungs- und klinischen Laboratorien. Dieses Modell liefert genaue und zuverlässige Ergebnisse über einen Bereich von Ausgaben.
Zusammenfassung
Es gibt eine Reihe von Bakterien-Biofilm-Modellen für die Testung von Antibiotika. Viele davon sind jedoch auf ein einzelnes experimentelles Ergebnis beschränkt, wie z. B. koloniebildende Einheiten oder metabolische Aktivität. Darüber hinaus spiegeln viele Biofilmmodelle nicht die biologischen und physiochemischen Eigenschaften der menschlichen Wirtsumgebung wider. Dies ist ein wichtiges Thema bei vielen Erkrankungen, aber am deutlichsten bei Mukoviszidose (CF). Ein großer Teil der Menschen mit Mukoviszidose leidet sowohl an chronischen als auch an intermittierenden Infektionen, und in vitro korrelieren Antibiotika-Empfindlichkeitstests schlecht mit den Behandlungsergebnissen der Patienten. Einige Biofilmmodelle enthalten CF-Lungen-relevante Medien, einschließlich synthetischer Sputum-Imitationen, berücksichtigen jedoch nicht die polymikrobielle Natur der Umgebung, die die Biofilmarchitektur, die Physiologie und die Art und Weise, wie Mikroben auf die Behandlung reagieren, verändert. Das hier beschriebene Fest-Luft-Grenzflächen-Kolonie-Biofilmmodell ist sehr anpassungsfähig und umfasst sowohl CF-relevante Medien als auch einen polymikrobiellen Kontext. Dieses Modell kann auch für das Screening von Antibiotika im mittleren Durchsatz und zur Untersuchung ihrer Wirkung auf die polymikrobielle Dynamik verwendet werden. Die Ergebnisse des Modells können koloniebildende Einheiten, die Stoffwechselaktivität und die konfokale Mikroskopieanalyse umfassen. Das Modell lässt sich leicht an unterschiedliche Mikroorganismen, Medien, Temperaturen und variable Sauerstoffbedingungen anpassen und kann zur Prüfung einer Vielzahl chemischer, biologischer und physikalischer Behandlungen verwendet werden.
Einleitung
Mukoviszidose (Mukoviszidose) ist eine genetische Erkrankung, von der über 11.000 Menschen in Großbritannien und 162.000 Menschen weltweit betroffensind 1,2. Obwohl Mukoviszidose eine Multiorganerkrankung ist, ist ein Schlüsselsymptom bei Menschen mit Mukoviszidose (pwCF) die Bildung von ungewöhnlich dickem, dehydriertem Schleim in den Atemwegen3. Dies, zusammen mit einer reduzierten Zilienschläge, kann die Besiedlung der Lunge durch eine Vielzahl von Bakterien, Pilzen, Viren und Archaeen verbessern 4,5. Obwohl die CF-Lunge Bedingungen und Selektionsdruck bietet, um das Wachstum und Überleben von Mikroorganismen zu begrenzen, sind Bakterien wie Pseudomonas aeruginosa in hohem Maße an diese rauen Umgebungen angepasst6. Dies ermöglicht sowohl die Besiedlung als auch das Überleben, was zur Bildung anhaltender chronischer Infektionen führt7.
Viele der Mikroben, die diese chronischen Infektionen verursachen, tun dies unter Verwendung einer phänotypischen Verschiebung von einem anfänglichen planktonischen zu einem Biofilm-Wachstumsstil, entweder oberflächengebunden oder in Aggregaten7. Diese Biofilme zeichnen sich durch dicht gepackte Bakteriengemeinschaften aus, die von einer Exopolysaccharid (EPS)-Matrix umhüllt sind, die aus einer Vielzahl von Komponenten besteht, darunter Polysaccharide, Proteine, Lipide und Umwelt-DNA (eDNA)8. Diese Matrix ist ein gemeinsames Merkmal von Mikroorganismen; Die Zusammensetzung kann jedoch unterschiedlich sein. Zum Beispiel sind die Hauptpolysaccharidkomponenten der bakteriellen P. aeruginosa-Matrix Psl, Pel und Alginat, im Gegensatz zu den Pilzen Candida albicans, deren Hauptbestandteile der Polysaccharidmatrix Mannane und Glucane sind9. In Monospezies-Biofilmen kann diese Matrix die Antibiotikatoleranz im Vergleich zu Plankton stark beeinflussen, indem sie das Eindringen von Antibiotika in den Biofilm verringert und deren Wirksamkeit verringert10. Die Art des Biofilmwachstums induziert auch die Bildung von Persisterzellen mit reduzierter Stoffwechselaktivität im Vergleich zu ihren planktonischen Gegenstücken und damit zu einer weiter verringerten Anfälligkeit für Antibiotika11. Diese Art des Wachstums ist auch durch die Hochregulierung einiger Antibiotikaresistenzmechanismen gekennzeichnet, wie z. B. Effluxpumpen und solche, die für den horizontalen Gentransfer erforderlich sind und den Austausch von Resistenzgenen ermöglichen 11,12,13. Darüber hinaus beeinflusst das krankheitsbedingte Umfeld des Wirts die mikrobielle Physiologie und die Art und Weise, wie er auf Antibiotika reagiert, sehr stark. Dazu gehören die erhöhte Mikroaerobiose im dicken Schleim sowie die Verfügbarkeit von nicht standardmäßigen Kohlenstoffquellen wie Aminosäuren und eDNA, die entweder vom Wirt stammen oder aus dem mikrobiellen Abbau von Lungenprodukten stammen 14,15,16.
Diese spezifischen Wechselwirkungen werden durch die polymikrobielle Natur von Biofilmen noch komplizierter, was eine zusätzliche Komplexitätsschicht mit komplexen Wechselwirkungen nicht nur zwischen Bakterien, sondern auch zwischen Bakterien und Pilzen mit sich bringt. Im Vergleich zu bakteriellen Biofilmen sind einige der Wechselwirkungen zwischen bakteriellen und pilzlichen Biofilmen weniger bekannt, obwohl C. albicans von über 75% der Menschen mit CF15 isoliert wurde. Im Allgemeinen sind die Wechselwirkungen zwischen C. albicans und Bakterien wie P. aeruginosa antagonistisch, können aber zu chronischeren und schwereren Infektionen führen18. Die Kombination von mikrobiellen Interaktionen, sowohl pathogenen als auch kommensalen, mit einer Reihe von CF-bedingten Umweltfaktoren kann letztendlich zu einer erhöhten Antibiotikatoleranz führen 19,20. Viele dieser Faktoren werden in präklinischen Antibiotikatests oft nicht berücksichtigt, obwohl sie in bestehenden Modellen auf eine erhöhte Antibiotikatoleranz zurückgeführt werden21.
Diese Zustände sind auch in vitro schwer zu rekapitulieren, und daher fehlen in vielen Modellen spezifische toleranzinduzierende Faktoren, die bei pwCF vorhanden sind, wie z. B. solche, die die Beta-Lactamase-Produktion in P. aeruginosaerhöhen 22, die Induktion kleiner Kolonievarianten bei Staphylococcus aureus und die Hemmung der Silbentrennung bei C. albicans; all dies wurde in CF-Sputum nachgewiesen23,24.
Es besteht daher eine große Diskrepanz zwischen den Bedingungen, die in den derzeitigen Antibiotika-Sensitivitätstestmethoden verwendet werden, die auf Plankton- oder Agarplattenkulturen in standardisierten Medien wie Mueller-Hinton mit Scheibendiffusions-, Etest- und CLSI-Bouillon-Mikrodilutionsassays basieren, und den Bedingungen, die in der Wirtsumgebungangetroffen werden 25. Dadurch kann die Antibiotika-Sensitivität häufig nicht genau bestimmtwerden 26. Dieses Problem wird durch die mangelnde Standardisierung bei der Untersuchung antimikrobieller Biofilme noch komplizierter, was es schwierig macht, die antimikrobielle Wirksamkeit genau vom Labor in die Klinik zu übertragen27,28.
Das polymikrobielle Modell, das wir hier entwickelt haben, zeigt eine erhöhte Toleranz von Pseudomonas aeruginosa gegenüber einer Reihe von antimikrobiellen Wirkstoffen, einschließlich Meropenem und Tobramycin. Dies verdeutlicht die große Variation zwischen den derzeitigen antimikrobiellen Tests mit Monospezies-Biofilmen und dem in dieser Studie entwickelten polymikrobiellen Biofilmmodell zur Bestimmung minimaler Hemmkonzentrationen. Dieses Modell behält auch einen relativ hohen Durchsatz und niedrige Kosten bei, die für antimikrobielle Tests wünschenswert sind. Das Modell kann auch verwendet werden, um den Einfluss einer antimikrobiellen Therapie auf die polymikrobielle Dynamik zu untersuchen und festzustellen, ob eine bestimmte Behandlung dazu führen kann, dass ein bestimmter Erreger dominant wird, was die Vorhersage weiterer Komplikationen ermöglicht. Obwohl dieses Modell den Aufbau komplexer Biofilme ermöglicht, erfordert sein Aufbau keine ausgeklügelte Laborausrüstung und bietet eine Plattform für eine breite Palette von klinischen und Forschungsergebnissen.
Protokoll
1. Vorbereitung von synthetischer Mukoviszidose media-2 (SCFM2) und Platten
- Stellen Sie SCFM2 nach dem in Palmer et al.16 beschriebenen Rezept in einer 500 mL Borosilikatglasflasche in 2x Konzentration mit einigen Modifikationen her.
- Die Bestände werden wie in Tabelle 1 dargestellt gewogen und vorbereitet. Bereiten Sie Aminosäurebrühen gemäß Tabelle 2 vor.
- Wiegen Sie 5 g Mucin (aus Schweinemagen (Typ II)) und 600 mg eDNA (aus Lachsspermien) in 100 ml dH2O in einer 200 mL Borosilikatglasflasche ab. Über Nacht bei 4 °C umrühren, bevor die Sterilisation durch Autoklavieren erfolgt.
- In eine sterilisierte 500-ml-Flasche werden 0,54 g Glukose, 6,06 g NaCl und 2,228 g KCl gegeben. Jeweils 10 ml der in Schritt 1.1.1 vorbereiteten Brühen werden hinzugefügt. Stellen Sie den pH-Wert mit 1 M NaOH oder HCl auf 6,9 ein.
- Man füge 10 mL dH2O hinzu, gefolgt von 10 mL 0,175 M CaCl2 und 0,0606 M MgCl2. Bereiten Sie 10 mL 0,93 M L-Milchsäure vor und fügen Sie sie hinzu. Unmittelbar vor Gebrauch 10 mL 0,36 mM Fe(III)SO47H2O zugeben
- Filtern Sie das SCFM2 mit einer Filtereinheit und einer Vakuumpumpe und fügen Sie dann das autoklavierte Mucin und die eDNA hinzu.
HINWEIS: Das vorbereitete SCFM2 kann bis zu 1 Monat bei 4 °C gelagert werden.
- Bereiten Sie SCFM2-Agarplatten wie unten beschrieben vor.
- 25 mL 2x SCFM2 in einem 50 mL Röhrchen in einem 55 °C Wasserbad erhitzen, bevor 25 mL vorgeschmolzener 3 % (w/v) technischer Agar gemischt werden, was zu einer Mischung aus 1x SCFM2 und 1,5 % (w/v) technischem Agar führt.
- Übertragen Sie mit einer serologischen Pipette 5 ml der Mischung in jede Vertiefung einer 6-Well-Platte.
- Bereiten Sie die gleiche Anzahl von 13 mm Polycarbonat-Scheiben mit einer Porengröße von 0,2 μm vor, indem Sie sie auf den Boden einer Petrischale legen und 600 s lang UV-C bei einer Wellenlänge von 254 nm und einer Frequenz von 60 Hz auftragen. Drehen Sie die Scheiben um und wiederholen Sie den UV-C-Vorgang.
- Sobald der technische Agar mit 1x SCFM2-1,5 % (w/v) ausgehärtet ist, wird mit einer sterilen Pinzette eine einzelne Polycarbonatscheibe auf die Oberfläche jeder Vertiefung der 6-Well-Platte aufgebracht.
2. Vorbereitung der Bakterien auf die Infektion
- Bereiten Sie vor dem Einrichten des Modells die Agarplatte(n) für die erforderlichen Bakterien- und Pilzstämme gemäß dem im Labor verwendeten Standard-Agar für P. aeruginosa PAO1, S. aureus SH1000 und C. albicans SC5314 vor (z. B. Lysogeny-Bouillon mit 1,2 % (w/v) Agar für Bakterien und Sabouraud-Dextrose-Agar für C. albicans).
HINWEIS: Zur Erstellung des Modells wurden spezifische Laborreferenzstämme verwendet. Dieses Modell wurde jedoch auch bei anderen Stämmen, einschließlich klinischer Isolate, verwendet, mit einigen Modifikationen, wie z. B. der Verlängerung der Zeit für die Etablierung von Biofilmen vor der Behandlung von langsamer wachsenden Stämmen. - Verwenden Sie eine 1-μl-Impfschleife, um eine einzelne Kolonie jedes Mikroorganismus zu entnehmen, um 5 ml Standard-Laborflüssigkeitsmedien zu impfen (z. B. Lysogeny-Bouillon für P. aeruginosa, S. aureus und Hefe-Pepton-Dextrose (YPD) für C. albicans). Inkubieren Sie diese Kulturen, bis sie die exponentielle Wachstumsphase bei 200 U/min, 37 °C für die Bakterien und 200 U/min, 30 °C für C. albicans erreichen.
HINWEIS: Die Belüftung hängt vom Schüttelbrutkasten und dem Rotationsradius ab
3. Einrichten des Festluft-Grenzflächenmodells
HINWEIS: Eine schematische Gesamtdarstellung des Modells ist in der ergänzenden Abbildung 1 zu sehen.
- 1 ml jeder Flüssigkultur in ein separates steriles 1,5-ml-Röhrchen umfüllen und 2 Minuten lang bei 8.000 x g zentrifugieren, um die Bakterien/Pilze zu pelletieren. Führen Sie diesen und alle folgenden Zentrifugationsschritte bei Raumtemperatur durch.
- Aspirieren Sie den Überstand mit einer Pipette und resuspendieren Sie die Pellets in 1 mL PBS, bevor Sie erneut 2 Minuten lang bei 8.000 x g zentrifugieren.
- Entfernen Sie den Überstand wie zuvor und suspendieren Sie die Pellets in 1 ml PBS. Die resuspendierten P . aeruginosa - und S. aureus-Zellen werden 1:10 in PBS verdünnt und die optische Dichte mit einem Spektralphotometer bei einer Wellenlänge von 600 nm gemessen.
- Die gewaschenen Proben werden auf 1 x 108 koloniebildende Einheiten (KBE)/ml eingestellt, basierend auf einer vorherigen Validierung, dass ein OD600 von 0,05 für P. aeruginosa und 0,1 für S. aureus 1 x 108 KBE/ml entspricht.
- Für C. albicans verdünnen Sie die gewaschenen Proben 1:100 in PBS, fügen Sie jeweils 10 μl zu zwei einzelnen Kammern eines Hämozytometers hinzu und verwenden Sie die festen Abmessungen der Kammer und die Verdünnung, die zur Berechnung von KBE/ml verwendet werden. Mit dem verbesserten Neubauer-Hämozytometer sieht die Berechnung wie folgt aus:
Anzahl der Zellen = durchschnittliche Zellzahl × 10.000 / (Verdünnungsfaktor) - Die berechneten KBE stellen C. albicans KBE/ml in der vorverdünnten Probe dar. Die C . albicans-Probe wird wie bei den Bakterienkulturen auf 1 x 108 KBE/ml in PBS eingestellt.
- Um das Inokulum für die Zugabe zum Biofilm vorzubereiten, stellen Sie das Inokulum von P. aeruginosa mit PBS auf 1 x 104 KBE/ml (2x 100-fache Verdünnungen), C. albicans auf 1 x 105 KBE/ml (100-fache Verdünnung, gefolgt von einer 10-fachen Verdünnung) und schließlich S. aureus auf 1 x 106 KBE/ml (100-fache Verdünnung) mit PBS ein.
- Bei Monospezies-Biofilmen werden 10 μl der verdünnten gewünschten Mikrobe in die Mitte der Polycarbonatscheibe gegeben, die zuvor auf den 1x SCFM2-1,5 % technischen Agar-Sechswellplatten platziert wurde, und vor der Behandlung oder Unterbrechung 24 Stunden lang statisch bei 37 °C inkubieren.
- Für polymikrobielle Biofilme geben Sie jeweils 10 μl von S. aureus und C. albicans übereinander auf dieselbe Scheibe. Inkubieren Sie die inokulierte Scheibe 24 h lang statisch bei 37 °C.
HINWEIS: Die Reihenfolge, in der die Inokula von S. aureus und C. albicans hinzugefügt werden, hat keinen Einfluss auf die Entwicklung des Modells und führt zu den gleichen Ergebnissen. - Anschließend werden die Polycarbonat-Scheiben mit einer sterilen Pinzette auf frische 1x SCFM2-1,5% technische Agarplatten umgefüllt und 10 μl der 1 x 104 KBE/ml-Verdünnung von P. aeruginosa auf den zuvor festgelegten S. aureus - C. albicans-Biofilm gegeben. Weitere 24 Stunden bei 37 °C inkubieren.
HINWEIS: P. aeruginosa wird 24 h nach S. aureus und C. albicans hinzugefügt, um die Reihenfolge widerzuspiegeln, in der diese Organismen die CF-Lunge bei Menschen mit CF häufig besiedeln. Dies verhindert auch, dass P. aeruginosa aus den beiden anderen Mikroorganismen herauswächst. Sobald P. aeruginosa hinzugefügt wurde, kann der Biofilm länger als 24 h gezüchtet werden; Die Polycarbonat-Scheibe mit dem Biofilm muss jedoch alle 24 Stunden auf ein frisches Medium übertragen werden. Vor dem Aufschluss können Biofilme für die Mikroskopie verwendet werden, wobei Stämme verwendet werden, die fluoreszierende Proteine, Fluoreszenzfarbstoffe oder stamm-/mikrobenspezifische Antikörper exprimieren. Es ist jedoch erwähnenswert, dass einige Farbstoffe die Polycarbonatscheibe verfärben.
4. Störung des Biofilms
- Geben Sie Keramikkügelchen mit einem Durchmesser von 2,8 mm auf die Platten und vernetzen Sie die Platten wie in Schritt 1.3 beschrieben. Geben Sie 5 Keramikkügelchen mit einer flammsterilisierten Pinzette in ein steriles 2-ml-Homogenisatorröhrchen. Pipettieren Sie dann 1 ml phosphatgepufferte Kochsalzlösung (PBS) in das 2-ml-Homogenisierungsröhrchen.
- Wirbeln Sie die Rohre mindestens 10 s lang oder bis der Biofilm von der Polycarbonatscheibe entfernt wurde, bestimmt durch Sichtprüfung, und entfernen Sie dann die Polycarbonatscheibe.
- Setzen Sie die Röhrchen mit den Kügelchen in den Homogenisator und schlagen Sie 2x für 10 s bei 6 m/s mit einem Abstand von 10 s zwischen den Schlägen.
HINWEIS: Diese Kombination aus Kügelchen, Schlagzeit und Homogenisatoreinsatz führt zu einer ausreichenden Aufschluss des Biofilms mit minimaler Lyse, wie durch Mikroskopie und koloniebildende Einheiten bestimmt. Dies kann je nach verwendetem Schläger variieren. - Gießen Sie den zerbrochenen Biofilm aus dem Homogenisatorröhrchen in ein 7-ml-Bijou, das 4 mL PBS enthält, was zu einem Endvolumen von 5 mL führt.
- Stellen Sie sicher, dass die gesamte Flüssigkeit aus den Homogenisatorröhrchen in das Bijou überführt wurde, indem Sie die Röhrchen 2 Minuten lang bei 8.000 x g zentrifugieren und die restliche Flüssigkeit in das 7 mL Bijou überführen.
HINWEIS: Aus diesen 5 ml zerbrochenem Biofilm kann eine Reihe von Assays durchgeführt werden, aber es ist wichtig, sich bei allen Berechnungen die PBS-Verdünnung von 1 ml Biofilm auf ein Endvolumen von 5 ml zu merken.
5. Prüfung der Stoffwechselaktivität
- Geben Sie 200 μl des aufgebrochenen Biofilms aus Schritt 4 auf eine schwarze 96-Well-Platte mit klarem Boden. Fügen Sie dann 10 μl 0,02 % (w/v) Resazurinlösung hinzu.
- Inkubieren Sie den mit Resazurin behandelten Biofilm mit einem Plattenleser und lesen Sie die Stoffwechselaktivität ab. Die Platte wird bei 37 °C inkubiert, wobei alle 30 Minuten 10 s bei 200 U/min orbitales Schütteln durchgeführt wird, bevor Fluoreszenzmessungen vorgenommen werden.
- Messen Sie die Fluoreszenz mit einer Anregung von 540 nm und einer Emission von 590 nm. Wiederholen Sie diesen 30-minütigen Zyklus für 4 Stunden mit einem Plattenleser.
HINWEIS: Es wird der 4-Stunden-Zeitpunkt verwendet, da er deutlichere Unterschiede zwischen behandelten und unbehandelten Proben und zwischen Proben mit unterschiedlichen Antibiotikakonzentrationen bietet.
6. Prüfung der Empfindlichkeit gegenüber antimikrobiellen Mitteln
- Um die antimikrobielle Verträglichkeit von Standardantibiotika zu testen, lösen Sie die Antibiotika Ihrer Wahl in ihrem jeweiligen Lösungsmittel auf und geben Sie dieses in flüssigem Zustand in das 1x SCFM2-1,5% (w/v) technische Agarmedium, bevor Sie 5 mL in jede Vertiefung einer Sechs-Well-Platte überführen. Nach dem Aushärten übertragen Sie den auf der Polycarbonat-Scheibe gewachsenen Biofilm mit einer sterilen Pinzette auf die Oberseite des antibiotikahaltigen Mediums und geben Sie einen 10-μl-Tropfen der entsprechenden Antibiotikakonzentration auf die Oberseite des Biofilms.
HINWEIS: Für diese Studie wurde eine 2-log-fache serielle Verdünnung zwischen 64 μg/ml und 0,125 μg/ml von Meropenem und Tobramycin verwendet, um klinisch relevante Konzentrationen widerzuspiegeln. Die Verabreichung von Antibiotika oder anderen antimikrobiellen/antivirulenzartigen Behandlungen ausschließlich auf dem Biofilm ist ausreichend, da ähnliche Reaktionen beobachtet wurden, wenn sie sowohl dem Medium als auch dem Biofilm zugesetzt wurden. - Nach 24 h Inkubation bei 37 °C mit der Behandlung bilden sich Bildbiofilme und stören wie in Schritt 4 beschrieben.
- Entnehmen Sie aus den 5 mL der gestörten Kultur ein Aliquot und führen Sie eine 10-fache serielle Verdünnung mit PBS durch. 10 μl Spots in dreifacher Ausfertigung auf Agar ausplattieren, um koloniebildende Einheiten mit der Miles- und Misra-Methode30 zu bestimmen.
HINWEIS: Das Volumen, das den Agarplatten zugesetzt wird, wirkt sich auf die Nachweisgrenze aus, größere Volumina verringern die Nachweisgrenze, erfordern jedoch mehr Proben- und Agarplatten. - Für Monospezies-Biofilme streifen P. aeruginosa und S. aureus auf LB-Agarplatten und C. albicans auf SDA-Agar.
- Bei polymikrobiellen Biofilmen werden Aliquots auf drei verschiedene selektive Medien mit Antibiotika aufgebracht, um jede der drei Spezies zu isolieren und die Bestimmung von KBE zu ermöglichen. Dazu gehören Pseudomonas-Isolationsagar und Mannitolsalz-Agar, die Nystatin in 10 μg/ml enthalten, um P. aeruginosa bzw . S. aureus zu isolieren, sowie Sabouraud-Dextrose-Agar mit Tetracyclin in 125 μg/ml, um C. albicans zu isolieren. Inkubieren Sie die Platten von S. aureus bei 37 °C über Nacht und die Platten von P. aeruginosa und C. albicans bei 30 °C.
HINWEIS: Inkubieren Sie P. aeruginosa-Platten bei 30 °C, um das Überwachsen der Kolonie zu minimieren und das Schwärmen zu reduzieren.
Ergebnisse
Die Einfachheit des Fest-Luft-Grenzflächen-Biofilmmodells ermöglicht das Screening einer großen Anzahl von Antibiotika unter verschiedenen klinisch relevanten Bedingungen gleichzeitig. Dieses Modell ermöglicht die Bewertung der Wirksamkeit von Antibiotika anhand von KBE-Zahlen und metabolischen Assays sowohl in mono- als auch in polymikrobiellen Biofilmen, die innerhalb einer Woche durchgeführt werden können. Aufgrund der Beschaffenheit des Modells ermöglicht es auch eine einfache Manipulation der Umgebungsbedingungen, wie z. B. die Änderung der Medienzusammensetzung und das Platzieren der Biofilme unter sauerstoffreduzierten und anaeroben Bedingungen. Mit diesem Modell haben wir Veränderungen in der Toleranz gegenüber zwei häufig bei pwCF verwendeten Antibiotika zwischen P. aeruginosa gezeigt, die in Monospezies- und polymikrobiellen Biofilmen gezüchtet wurden (Abbildung 1). Darüber hinaus konnten wir feststellen, wie sich eine antimikrobielle Behandlung auf die Populationsdynamik innerhalb polymikrobieller Biofilme auswirken kann (Abbildung 2).
Zur Durchführung dieser Versuche wurde das erste Inokulum wie im obigen Protokoll spezifiziert vorbereitet, was zur Bildung stabiler Monospezies und polymikrobieller Biofilme mit reproduzierbarer KBE/ml und geringen Standardabweichungen führte, die durch ein- oder bidirektionale ANOVA bestimmt wurden. Dieses Modell unterstreicht auch das konsistente Wachstum und die KBE Rückgewinnung und ermöglicht die Möglichkeit, diese klassischen mikrobiologischen Techniken mit anderen Techniken zu kombinieren, die in dieser Studie nicht durchgeführt wurden, ohne dass eine ausgeklügelte Verarbeitung erforderlich ist. Dazu gehören Bildgebung wie Leben/Tot, Matrixvisualisierung und molekulare Analyse, die andere dreidimensionale Modelle in dieser Form nicht so zugänglich anbieten können (Abbildung 1)31. Es ist zu beachten, dass die erwartete Standardabweichung für KBE/ml zunimmt, wenn die Daten als Prozentsatz des Überlebens im Vergleich zu CFU/ml angezeigt werden (Abbildung 1). Beim Vergleich der Wirksamkeit der Antibiotikabehandlung zwischen Monospezies und polymikrobiellen Biofilmen ergab sich ein signifikanter Anstieg aller Antibiotikakonzentrationen, die erforderlich sind, um eine Abtötung von 50 % zu erreichen. Um dieses Abtötungsniveau für Meropenem und Tobramycin zu erreichen, war eine Erhöhung der Antibiotikakonzentration um 2 log erforderlich (Abbildung 1). Es gab auch eine allgemeine Verlängerung des Überlebens von P. aeruginosa in Gegenwart von S. aureus und C. albicans im polymikrobiellen Biofilm, wenn es mit 64 μg/ml Tobramycin behandelt wurde, während das Gegenteil für Meropenem der Fall war.
Die Methode zur Bestimmung der Stoffwechselaktivität misst die Gesamtaktivität des gesamten polymikrobiellen Biofilms, ohne den individuellen Beitrag jeder Spezies unterscheiden zu können. Aus diesem Grund werden nur metabolische Veränderungen von Monospezies gezeigt, um die Verwendung von Stoffwechselaktivitätsassays für dieses Modell zu demonstrieren. Bei Monospezies-Biofilmen gab es einen starken Zusammenhang zwischen dem Überleben von P. aeruginosa und der Stoffwechselaktivität sowohl für Meropenem als auch für Tobramycin (Abbildung 2). Die Verwendung sowohl der metabolischen Aktivität als auch der KBE-Zahl aus denselben Proben ermöglicht die einfache Identifizierung sowohl bakteriostatischer als auch bakterizider Wirkungen. Bei polymikrobiellen Biofilmen kann die Bestimmung der metabolischen Aktivität darauf hinweisen, wie das Targeting auf eine Spezies eine allgemeine Zunahme der metabolischen Aktivität des Biofilms induzieren und somit möglicherweise das mikrobielle Wachstum anderer Spezies erhöhen kann.
Obwohl KBE der Goldstandard für antimikrobielle Sensitivitätstests sind, ermöglichen sie in polymikrobiellen Biofilmen auch die Beurteilung der Spezieszusammensetzung innerhalb des Biofilms. Durch die Behandlung von P. aeruginosa in polymikrobiellen Biofilmen können wir nicht nur den erhöhten MBEC50-Spiegel für diesen Organismus nachweisen, sondern auch den Effekt auf die anderen co-isolierten Spezies nachweisen (Abbildung 3). Dies ist z. B. bei Meropenem (antipseudomonal) der Fall, bei dem die Reduktion von P. aeruginosa mit einer Zunahme von S. aureus und C. albicans KBE einhergeht, was dazu führt, dass S. aureus bei Exposition gegenüber bestimmten Antibiotikakonzentrationen zum am weitesten verbreiteten Organismus wird (Abbildung 3A). Dies unterstreicht, wie wichtig es ist, die polymikrobielle Natur des Biofilms zu berücksichtigen, da die Populationsgröße anderer krankheitserregender Spezies bei der Behandlung eines bestimmten Krankheitserregers zunehmen kann.
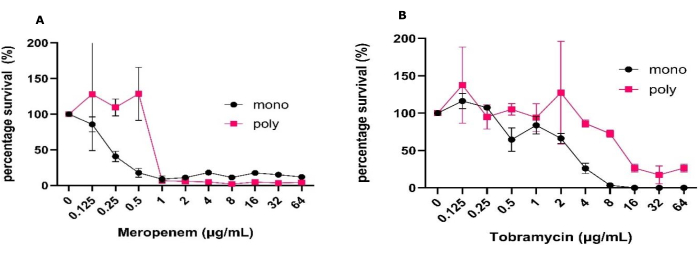
Abbildung 1: Variation der antimikrobiellen Toleranz von P. aeruginosa gegenüber Meropenem und Tobramycin zwischen mono- und polymikrobiellen Biofilmen, die im Fest-Luft-Grenzflächenmodell gezüchtet wurden. P. aeruginosa PAO1, gezüchtet in mono- oder polymikrobiellen Biofilmen mit S. aureus und C. albicans , wurde unter Verwendung der Feststoff-Luft-Grenzflächenmodelle für 24 h auf SCFM2 ermittelt. Die Biofilme wurden mit einem Bereich von (A) Meropenem- oder (B) Tobramycin-Konzentrationen von 0,125 μg/ml bis 64 μg/ml zusammen mit einer Antibiotika-Kontrollen behandelt. Die KBE/ml jedes Biofilms wurde bestimmt. P. aeruginosa Die KBE wurden in PIA bestimmt und in das prozentuale Überleben umgerechnet, indem die Negativkontrolle als 100%-Überleben verwendet wurde. Fehlerbalken bezeichnen die Standardabweichung, und jeder Datenpunkt wird aus 3 biologischen Wiederholungen mit jeweils drei technischen Wiederholungen abgeleitet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Variation der metabolischen Aktivität von P . aeruginosa-Biofilmen in Gegenwart von Meropenem oder Tobramycin, die im Fest-Luft-Grenzflächenmodell gezüchtet wurden. P. aeruginosa PAO1-Biofilme wurden auf SCFM2 im Fest-Luft-Grenzflächenmodell gezüchtet, und eine Reihe von (A) Meropenem- oder (B) Tobramycin-Konzentrationen von 0,125 μg/ml bis 64 μg/ml wurden zusammen mit einer Kontrolle ohne Antibiotika hinzugefügt. Die Platte wurde fluoreszierend bei einer Anregung von 540 nm und einer Emission von 590 nm alle 30 min abgelesen. Die prozentuale metabolische Aktivität wurde berechnet, indem der Prozentsatz dieser Aktivität bei jeder mit Antibiotika behandelten Probe im Vergleich zur Kontrolle ohne Antibiotika bestimmt wurde. Fehlerbalken bezeichnen die Standardabweichung, und jeder Datenpunkt wird aus 3 biologischen Wiederholungen mit jeweils drei technischen Wiederholungen abgeleitet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
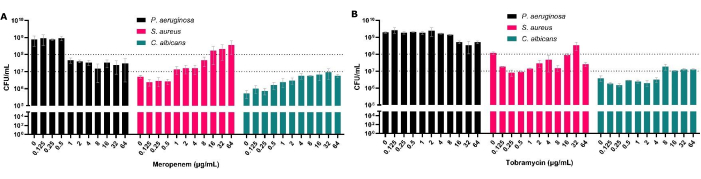
Abbildung 3: Auswirkungen auf die Gesamt-KBE von P. aeruginosa, S. aureus und C. albicans , die nach einer antimikrobiellen Behandlung aus dem polymikrobiellen Fest-Luft-Grenzflächenmodell gewonnen wurden. S. aureus und C. albicans wurden auf SCFM2 auf dem Fest-Luft-Grenzflächenmodell gezüchtet. P. aeruginosa wurde hinzugefügt, und ein Bereich von 0,125 μg/ml bis 64 μg/ml-Konzentrationen von (A) Meropenem oder (B) Tobramycin wurden den Biofilmen zusammen mit einer nicht-antibiotischen Kontrolle zugesetzt. Die KBE/ml jedes Biofilms wurde bestimmt. Fehlerbalken bezeichnen die Standardabweichung, und jeder Datenpunkt wurde aus 3 biologischen Wiederholungen mit jeweils drei technischen Wiederholungen abgeleitet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Abbildung 1: Etablierung und Arbeitsablauf des Fest-Luft-Grenzflächenmodells. Grafische Darstellung der Etablierung des Fest-Luft-Grenzflächenmodells, der Biofilmzerstörung und der Ergebnisse. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Das hier beschriebene Biofilmmodell ermöglicht es uns, einige Aspekte der Umgebung von CF-Lungeninfektionen nachzuahmen, indem wir eine Reihe gängiger polymikrobieller Wechselwirkungen auf eine zugänglichere Weise einbeziehen als derzeit verwendete antimikrobielle In-vitro-Testmethoden wie den CDC-Reaktor und das Lubbock-Biofilmmodell31. Dies ist wichtig, wenn eine Reihe von Faktoren, einschließlich der physikalischen Eigenschaften des Biofilms, der Nährstoffverfügbarkeit und der molekularen Wechselwirkungen zwischen den Spezies innerhalb des Biofilms, sowohl die Aktivität des antimikrobiellen Mittels als auch die Reaktion der Mikroben auf das antimikrobielle Mittel beeinflussen können21. Durch die Einbeziehung dieser Faktoren konnten wir ein äußerst vielseitiges Modell entwickeln, das eine genauere Vorhersage der Ergebnisse antimikrobieller Behandlungen ermöglicht als aktuelle Modelle, die die oben genannten Faktoren nicht berücksichtigen.
P. aeruginosa, S. aureus und C. albicans wurden als repräsentative grampositive, gramnegative und Pilze ausgewählt, die häufig in CF32 isoliert werden. Wir konnten jedoch auch zeigen, dass dieses Modell für eine Reihe anderer CF-relevanter Mikroben verwendet werden kann, darunter Burkholderia cenocepacia, Burkholderia multivorans, Aspergillus fumigatus, und wird derzeit für den Einsatz mit anaeroben Bakterien wie Prevotella melaninogenica angepasst. Dies zeigt die Vielseitigkeit des Modells, da eine breite Palette von Mikroben integriert werden konnte, was die Anpassungsfähigkeit dieses Modells für antimikrobielle Tests gegen eine Vielzahl von mikrobiellen Krankheitserregern unterstreicht. Das Modell kann auch leicht an eine Reihe von Umgebungsbedingungen angepasst werden, um personalisiertere antimikrobielle Sensitivitätstests zu ermöglichen, einschließlich des Ersatzes von SCFM2 durch Sputum des Patienten.
Die Berücksichtigung polymikrobieller Biofilmmodelle für die Erprobung neuer antimikrobieller Wirkstoffe ist ebenfalls von größter Bedeutung, da sie eine bestehende Lücke in der antimikrobiellen Entwicklungspipeline schließen können, bevor sie in Tier- oder Humanstudien eingesetzt werden33,34. Darüber hinaus behält das Modell einen relativ hohen Durchsatz bei und ermöglicht eine Reihe von Ergebnissen, die sowohl für die Industrie als auch für die Forschung relevant sind.
Obwohl wir in dieser Methodenarbeit nur KBEs und metabolische Aktivität veranschaulicht haben, haben wir auch den Nutzen des Modells bei der Analyse des Biofilm-Exometaboloms mit Hilfe der Massenspektrometrie der Flüssigkeitsextraktion und Oberflächenanalyse (LESA) demonstriert. Dies ermöglicht es, einige der molekularen Mechanismen zu untersuchen, die der antimikrobiellen Toleranz zugrunde liegen35. Zusätzlich zu diesen anderen Ergebnissen haben wir auch die Verwendung dieses Modells zum Testen neuartiger antimikrobieller Verabreichungssysteme demonstriert. Dazu gehören Polymer-Ciprofloxacin-Konjugate, um die Penetration dieses Antibiotikums innerhalb des Biofilms zu verbessern und gleichzeitig seine Aktivität zu erhöhen und die Entwicklung von Resistenzen zu verringern36.
Kritische Schritte und Überlegungen
Es ist wichtig, die Sterilität aufrechtzuerhalten, insbesondere bei der Herstellung von SCFM2 und Medien, da eine Kontamination zur Einführung nicht natürlich vorkommender Spezies führen kann, die die Ergebnisse beeinträchtigen können. Unterschiedliche Stämme derselben Mikrobe und verschiedene Mikroben können unterschiedliche Umweltbedingungen für ihr Wachstum benötigen oder unterschiedliche Wachstumseigenschaften aufweisen. Daher muss das Modell optimiert werden, bevor Antibiotika-Sensitivitätstests durchgeführt werden. Wir empfehlen, eine kleine Pilotstudie von 24 h und 48 h für Monospezies und polymikrobiellen Biofilm durchzuführen, um das Wachstum unter Standardmodellbedingungen zu bewerten. Wir haben festgestellt, dass verschiedene P. aeruginosa-Stämme und bestimmte Antibiotikakonzentrationen ein Schwärmen von der Polycarbonatscheibe auslösen können. Dies kann leicht durch die Verwendung von größeren Scheiben überwunden werden, die im Handel erhältlich sind. Auch die Auflockerung des Biofilms kann je nach Bead-Beater und den verwendeten Mikroben eine Optimierung erfordern. Wir optimierten die Aufschlussmethode, indem wir die KBE/ml einer bekannten Probe vor und nach dem Bead-Schlagen bestimmten. Wir verwendeten auch Mikroskopie, um die Bead-Beat-Proben zu visualisieren, um zu visualisieren, wie gut der Biofilm aufgebrochen wurde.
Eine der Einschränkungen dieses CF-imitierenden Biofilmmodells ist die Bindung an eine feste Oberfläche, die Polycarbonat enthält, das in der CF-Lunge nicht vorkommt, anstatt frei schwebende Aggregate innerhalb des CF-Sputumszu sein 37 . Das hier vorgestellte Fest-Agar-Modell schließt jedoch aus, dass die Biofilme gewaschen werden müssen, um planktonische Mikroben zu entfernen, und stellt sicher, dass die Ergebnisse nur Mikroben enthalten, die aus dem Biofilm gewonnen wurden, im Gegensatz zu einer gemischten planktonischen Biofilmkultur. Wir haben auch festgestellt, dass das Eindringen einiger Antibiotika in die tieferen Bereiche dieses Biofilmmodells reduziert werden kann, und die Verwendung von DNase kann erforderlich sein, um die Rheologie der extrazellulären Matrix zu verändern und die elektrostatischen Wechselwirkungen mit der eDNA zu verringern.
Wir glauben, dass das Modell aufgrund seiner Vielseitigkeit, Anpassungsfähigkeit und der Einbeziehung von Umweltfaktoren, die viele bestehende Modelle nicht berücksichtigen, sehr gut für antimikrobielle Tests geeignet ist. Sein Einsatz in der Grundlagenforschung und bei präklinischen antimikrobiellen Sensitivitätstests kann einen klinisch relevanteren Ansatz für die AST klinischer Proben und die Entwicklung neuartiger Therapeutika bieten.
Offenlegungen
Für diese Arbeit wird kein Interessenkonflikt oder konkurrierende finanzielle Interessen deklariert.
Danksagungen
Diese Arbeit wurde vom National Biofilms Innovation Centre (NBIC) finanziert, einem Innovations- und Wissenszentrum, das vom Biotechnology and Biological Sciences Research Council, Innovate UK und dem Hartree Centre [Auszeichnungen BB/R012415/1 und BB/X002950/1] sowie vom UK CF Trust und dem USA CF Foundation Strategic Research Centre: "An evidence-based preclinical framework for the development of antimicrobial therapeutics in cystic fibrosis" (PIPE-CF) finanziert wird [Auszeichnung SRC022].
Materialien
| Name | Company | Catalog Number | Comments |
| 1 µL inoculation loops | |||
| 13 mm 0.2 µm pore size polycarbonate discs | Isopore | GTTP01300 | Larger discs are also available |
| 2 mL reinforced tubes | Thermofisher | 15545809 | |
| 2.5 mL ceramic beads | Qiagen | 13114-325 | |
| 500 mL borosilicate glass Duran bottle | Sigma Aldrich | Z305197 | larger bottles available in larger volumes are desired |
| 6-well culture plates | Greiner | 657165 | |
| 7 mL Bijou | Thermofisher | 129B | |
| 96-well plates | Thermofisher | 167008 | for serial dilutions in CFU assay |
| Agar plates for preparing plates of P. aerugnisa, S. aureus, and C. albicans | LB miller for P. aeruginosa and S. aureus and Sabouraud dextrose agar for C. albicans | ||
| Bead beater - suitable for 2 mL tubes | Fisherbrand | 15515799 | Thermofisher bead mill 24 |
| bench top centrifuge | must be capable of at least 8000 x g | ||
| Black clear bottom 96 well plates | Costar | 3603 | |
| Bunsen Burner | |||
| Containers for disposing of contaminated equipment and material according to the institutes health and safety regulations. | |||
| deionised water | |||
| eDNA | Sigma Aldrich | 31149 | |
| Filter unit | Fisherbrand | FB12566504 | Interchangeable depending on the vacuum pump used but must have a pore size of 0.2 µm |
| Haemocytometer and cover slip | Hawksley | HC001 | Haemocytometers may differ in size and volume. Double check and adjust CFU calculations accordingly |
| LB broth | oxoid | 1.46813 | |
| Mannitol salt agar | Oxoid | CM0085B | |
| meropenem | abcr | Ab243429 | |
| Mucin from porcine stomach Type II | Sigma Aldrich | M2378 | |
| Nystatin | Millipore | 1003352658 | |
| petri dishes | SLS | SLS2000 | |
| Phosphate buffered saline | |||
| Pseudomonas isolation agar | Millipore | 17208 | |
| Resazurin sodium salt | Sigma Aldrich | 199303 | |
| Sabouraud dextrose agar | Oxoid | CM0041 | |
| selection of forceps | fine tipped and tissue forceps with teeth for transferring ceramic beads | ||
| serological pipette | |||
| shaking and static incubators | must be temperature controlled | ||
| Sparks microtitre plate reader | Tecan | For Resazurin assay the microtitre plate reader must have the appropriate filters or be a monochromator for detecting flourescence. | |
| spectrophotometer | |||
| Technical agar (Agar Technical No.2 ) | Oxoid | LP0012B | |
| tetracycline | Sigma Aldrich | T7660 | |
| UV crosslinker | Spectroline | 11-992-89 | |
| vacuum pump/ flask | Fisherbrand | FB12566504 | |
| water bath | must be capable of maintaining 55 °C | ||
| YPD broth | Millipore | Y1375 | Can be bought pre-made or made using the base ingredients |
Referenzen
- . 2021 Cf foundation patient registry highlights report Available from: https://cysticfibrosis.msu.edu/index.php/welcome/news-and-events/39-2021-cf-foundation-patient-registry-highlights-report (2022)
- Guo, J., Garratt, A., Hill, A. Worldwide rates of diagnosis and effective treatment for cystic fibrosis. J Cyst Fibros. 21 (3), 456-462 (2022).
- Mandal, V., Ghosh, N. N., Mitra, P. K., Mandal, S., Mandal, V. Production and characterization of a broad-spectrum antimicrobial 5-butyl-2-pyridine carboxylic acid from Aspergillus fumigatus nhf-01. Sci Rep. 12 (1), 6006 (2022).
- Françoise, A., Héry-Arnaud, G. The microbiome in cystic fibrosis pulmonary disease. Genes. 11 (5), 536 (2020).
- Grasemann, H., Ratjen, F. Cystic fibrosis. New Engl J Med. 389 (18), 1693-1707 (2023).
- Camus, L., Vandenesch, F., Moreau, K. From genotype to phenotype: Adaptations of pseudomonas aeruginosa to the cystic fibrosis environment. Microb Genom. 7 (3), mgen000513 (2021).
- Rossi, E., et al. Pseudomonas aeruginosa adaptation and evolution in patients with cystic fibrosis. Nat Rev Microbiol. 19 (5), 331-342 (2021).
- Flemming, H. -. C., Wingender, J. The biofilm matrix. Nat Rev Microbiol. 8 (9), 623-633 (2010).
- Grainha, T., Jorge, P., Alves, D., Lopes, S. P., Pereira, M. O. Unraveling pseudomonas aeruginosa and candida albicans communication in coinfection scenarios: Insights through network analysis. Front Cell Infect Microbiol. 10, 550505 (2020).
- Flemming, H. C., et al. Biofilms: An emergent form of bacterial life. Nat Rev Microbiol. 14 (9), 563-575 (2016).
- Høiby, N., Bjarnsholt, T., Givskov, M., Molin, S., Ciofu, O. Antibiotic resistance of bacterial biofilms. Int J Antimicrob Agents. 35 (4), 322-332 (2010).
- Lorusso, A. B., Carrara, J. A., Barroso, C. D. N., Tuon, F. F., Faoro, H. Role of efflux pumps on antimicrobial resistance in Pseudomonas aeruginosa. Int J Mol Sci. 23 (24), 15779 (2022).
- Michaelis, C., Grohmann, E. Horizontal gene transfer of antibiotic resistance genes in biofilms. Antibiotics .(Basel). 12 (2), 328 (2023).
- Worlitzsch, D., et al. Effects of reduced mucus oxygen concentration in airway pseudomonas infections of cystic fibrosis patients. J Clin Invest. 109 (3), 317-325 (2002).
- König, J., Schreiber, R., Voelcker, T., Mall, M., Kunzelmann, K. The cystic fibrosis transmembrane conductance regulator (CFTR) inhibits ENaC through an increase in the intracellular Cl- concentration. EMBO Reps. 2 (11), 1047-1051 (2001).
- Palmer, K. L., Mashburn, L. M., Singh, P. K., Whiteley, M. Cystic fibrosis sputum supports growth and cues key aspects of Pseudomonas aeruginosa physiology. J Bacteriol. 187 (15), 5267-5277 (2005).
- Valenza, G., et al. Prevalence and antimicrobial susceptibility of microorganisms isolated from sputa of patients with cystic fibrosis. J Cyst Fibros. 7 (2), 123-127 (2008).
- Alam, F., Catlow, D., Di Maio, A., Blair, J. M. A., Hall, R. A. Candida albicans enhances meropenem tolerance of Pseudomonas aeruginosa in a dual-species biofilm. J Antimicrob Chemother. 75 (4), 925-935 (2020).
- Burmølle, M., Ren, D., Bjarnsholt, T., Sørensen, S. J. Interactions in multispecies biofilms: Do they actually matter. Trends Microbiol. 22 (2), 84-91 (2014).
- Lories, B., Belpaire, T. E. R., Smeets, B., Steenackers, H. P. Competition quenching strategies reduce antibiotic tolerance in polymicrobial biofilms. NPJ Biofilms Microbiomes. 10 (1), 23 (2024).
- Nabb, D. L., et al. Polymicrobial interactions induce multidrug tolerance in Staphylococcus aureus through energy depletion. Front Microbiol. 10, 2803 (2019).
- Cornforth, D. M., et al. Pseudomonas aeruginosa transcriptome during human infection. Proc Natl Acad Sci U S A. 115 (22), E5125-E5134 (2018).
- Wolter, D. J., et al. Staphylococcus aureus small-colony variants are independently associated with worse lung disease in children with cystic fibrosis. Clin Infect Dis. 57 (3), 384-391 (2013).
- Takagi, J., et al. Mucin o-glycans are natural inhibitors of Candida albicans pathogenicity. Nat Chem Biol. 18 (7), 762-773 (2022).
- Cinical and Laboratory Standard Institute. . Methods for dilution susceptibility tests for bacteria that grow aerobically. 35 (2), (2015).
- Roberts, A. E., Kragh, K. N., Bjarnsholt, T., Diggle, S. P. The limitations of in vitro experimentation in understanding biofilms and chronic infection. J Mol Biol. 427 (23), 3646-3661 (2015).
- Macia, M. D., Rojo-Molinero, E., Oliver, A. Antimicrobial susceptibility testing in biofilm-growing bacteria. Clin Microbiol Infect. 20 (10), 981-990 (2014).
- Coenye, T., Goeres, D., Van Bambeke, F., Bjarnsholt, T. Should standardized susceptibility testing for microbial biofilms be introduced in clinical practice. Clin Microbiol Infect. 24 (6), 570-572 (2018).
- Turner, K. H., Wessel, A. K., Palmer, G. C., Murray, J. L., Whiteley, M. Essential genome of Pseudomonas aeruginosa in cystic fibrosis sputum. Proc Natl Acad Sci U S A. 112 (13), 4110-4115 (2015).
- Miles, A. A., Misra, S. S., Irwin, J. O. The estimation of the bactericidal power of the blood. J Hyg. 38 (6), 732-749 (1938).
- Robertson, S. N., Romero, M., Fenn, S., Kohler Riedi, P. L., Cámara, M. Development, characterization, and evaluation of a simple polymicrobial colony biofilm model for testing of antimicrobial wound dressings. J Appl Microbiol. 135 (3), lxae042 (2024).
- Rumpf, C., Lange, J., Schwartbeck, B., Kahl, B. C. Staphylococcus aureus and cystic fibrosis-a close relationship. What can we learn from sequencing studies. Pathogens. 10 (9), 1177 (2021).
- Tay, W. H., Chong, K. K. L., Kline, K. A. Polymicrobial-host interactions during infection. J Mol Biol. 428 (17), 3355-3371 (2016).
- Orazi, G., O'toole, G. A. "It takes a village": Mechanisms underlying antimicrobial recalcitrance of polymicrobial biofilms. J Bacteriol. 202 (1), e00530-e00519 (2019).
- Arjes, H. A., et al. Three-dimensional biofilm colony growth supports a mutualism involving matrix and nutrient sharing. eLife. 10, e64145 (2021).
- Kasza, K., et al. Ciprofloxacin poly(β-amino ester) conjugates enhance antibiofilm activity and slow the development of resistance. ACS Appl Mater Interf. 16 (5), 5412-5425 (2024).
- Martin, I., Waters, V., Grasemann, H. Approaches to targeting bacterial biofilms in cystic fibrosis airways. Int J Mol Sci. 22 (4), 2155 (2021).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten